扩展功能
文章信息
- 刘明艳, 马嘉晗, 李瑜, 刘文霞, 秦鸿娟, 高配科
- LIU Ming-Yan, MA Jia-Han, LI Yu, LIU Wen-Xia, QIN Hong-Juan, GAO Pei-Ke
- 16S rRNA基因高变区V4和V3-V4及测序深度对油藏细菌菌群分析的影响
- Influence of 16S rRNA gene V4 and V3-V4 sequencing and sequencing depth on unraveling bacterial communities inhabiting oil reservoirs
- 微生物学通报, 2020, 47(2): 440-449
- Microbiology China, 2020, 47(2): 440-449
- DOI: 10.13344/j.microbiol.china.190251
-
文章历史
- 收稿日期: 2019-03-28
- 接受日期: 2019-08-15
- 网络首发日期: 2019-09-30
微生物在碳循环、氮循环、磷循环和硫循环等生物地球化学过程、营养盐再生、环境污染物降解、能源勘探开发和能量传递等过程中发挥着重要作用[1-2]。特定环境微生物的群落组成、多样性及其生态功能是微生物生态学研究的核心问题。微生物群落结构决定了其生态功能,研究微生物群落有助于人类更好地驾驭这些微生物净化环境、勘探开发矿藏、生产合成医药等。
微生物群落分析已从最初的分离培养发展到分子水平[3-4],分子检测技术主要包括荧光原位杂交[5-6]、克隆文库[7]、基因芯片[8-9]、基于高通量测序的16S rRNA基因序列分析[10-12]、宏基因组[13]和宏转录组[14]等,其中16S rRNA基因测序在研究微生物群落组成及其分布过程中得到广泛应用。
油藏孕育着丰富的微生物类群,这些微生物及其代谢产物能够一定程度上改善油藏环境和原油物性,提高原油采收。油藏微生物群落分析是石油微生物学的一个重要研究方向,对微生物采油技术的应用具有重要指导意义。454焦磷酸测序开创了16S rRNA基因高通量测序的先河,2014年前后454焦磷酸测序在油藏微生物群落解析方面发挥了重要作用[12, 15],但要达到较高测序深度,不可避免地要增加测序成本。MiSeq测序技术的出现使得同样测序成本情况下测序深度大大提高。现阶段,基于MiSeq测序平台的16S rRNA基因V4区测序被广泛用于各环境(含油藏)微生物群落研究[10, 16-17]。基于V3−V4区测序可提供更丰富的序列信息,同样测序深度下测序成本略有提高,该测序区域在油藏环境的应用鲜有报道。鉴于此,本文基于MiSeq测序平台探究测序区域(V4区和V3−V4区)及测序深度对油藏细菌菌群分析的影响。
油藏是一个高压缺氧的极端环境,微生物数量远远低于地表土壤和湖泊等环境,基于待分析油水样微生物16S rRNA基因拷贝数,设置V4区和V3−V4区MiSeq测序所需深度。分析内容包括:(1)不同测序区域和深度对油藏细菌菌群多样性指数分析的影响;(2)不同测序区域和深度对菌群组成分析的影响;(3)不同测序区域和深度下相对丰度之间具有显著性差异的微生物类群分析。本文研究结果可为基于16S rRNA基因高通量测序研究油藏微生物群落提供一定参考。
1 材料与方法 1.1 主要试剂和仪器AxyPrep™ Genomic DNA Miniprep Kit,康宁生命科学(吴江)有限公司;FastStart Universal SYBR Green Master (ROX)试剂盒,上海罗氏制药有限公司;大肠杆菌DH5α和pEASY-T载体,北京全式金生物技术有限公司。NanoDrop分光光度计,赛默飞世尔科技公司;PCR仪,Bio-Rad公司;MiSeq测序仪,Illumina公司。
1.2 微生物菌体收集与总DNA提取实验所用油水样取自新疆克拉玛依油田六中区克下组采油井T6890产出液。该油藏区块已经历近30年水驱,截至取样时采油井综合含水92.5%。油藏平均深度480 m,地层温度22.6 ℃,地层压力7.2 MPa。菌体收集过程为:将2 L油水样置于60 ℃下静置30 min,利用无菌分液漏斗进行油水相分离;水相经0.22 μm孔径滤膜过滤收集菌体;油相加入3倍体积异辛烷,混合均匀后12 000 r/min离心20 min收集菌体,同时将离心后的油相经0.22 μm孔径滤膜过滤收集菌体;将油水相所得菌体混合用于基因组提取。基因组提取采用玻璃珠研磨和Axygen基因组提取试剂盒相结合的方法[15]:将1 mL TE缓冲液(Tris 80 mmol/L,EDTA 40 mmol/L,pH 8.0)加入上述菌体,加入0.2 g 0.1 mm玻璃珠2 500 r/min振荡1 min,此过程重复3次,期间置于冰上冷却1 min。后续基因组提取过程用Axygen基因组提取试剂盒完成。
1.3 16S rRNA基因数量定量分析以16S rRNA基因通用引物8F (5′-AGAGTTT GAT(CT)(AC)TGGCTC-3′)和338R (5′-GCTGCCT CCCGTAGGAGT-3′)为定量PCR引物,检测样品基因组中16S rRNA基因数量[18]。回收纯化16S rRNA基因扩增子,将目的基因片段连接入pEASY-T载体,进而克隆转化到大肠杆菌DH5α中(具体操作按照试剂盒说明书进行)。选取阳性克隆于液体肉汤培养基中37 ℃、160 r/min培养过夜,收集菌体提取质粒。用NanoDrop分光光度计检测所提质粒DNA浓度,计算质粒数量的公式:样品浓度(ng/μL)=OD260×50 ng/μL×稀释倍数;样品摩尔质量=碱基对数×660 g/mol;样品基因数量(copies/μL)=6.02×1023×待测样品浓度(ng/μL)× 10−9/样品摩尔质量;核酸最高吸收波长为260 nm;1 OD相当于50 μg/mL的dsDNA。将质粒进行10倍比梯度稀释至102 copies/μL,用于标准曲线绘制。荧光定量采用SYBR Green I染料法,PCR反应体系:Fast Start Universal SYBR Green Master 10 μL,引物8F和338R各6 pmol/L,模板DNA 1 μL,ddH2O补足至20 μL。PCR反应条件:95 ℃ 10 min;95 ℃ 20 s,60 ℃ 1 min采集荧光信号,40个循环;以每循环0.5 ℃由60 ℃上升至95 ℃,每循环保留10 s,获得溶解曲线。根据标准曲线计算待测样品中16S rRNA基因数。
1.4 16S rRNA基因MiSeq测序及序列处理16S rRNA基因V4区测序使用Illumina MiSeq PE250测序平台(单端最大读长250 bp),16S rRNA基因V3−V4区测序使用MiSeq PE300测序平台(单端最大读长300 bp)。V4区PCR扩增所用引物为515f (5′-GTGCCAGCMGCCGCGGTAA-3′)和806r (5′-GGACTACHV GGGTWTCTAAT-3′),扩增子长度约290 bp[10];V3–V4区PCR扩增所用引物为338f (5′-ACTCCTACGGGAGGCAGCAG-3′)和806r (5′-GGACTACHVGGGTWTCTAAT-3′),扩增子长度约460 bp。PCR反应体系(20 µL):5×TransStart® FastPfu Buffer 4 µL,dNTPs (2.5 mmol/L) 2 µL,TransStart® FastPfu DNA Polymerase (2.5 U/µL) 0.5 µL,正、反向引物(10 μmol/L)各0.5 µL,样品DNA 2 µL,ddH2O 10.5 µL。PCR反应条件:94 ℃ 4 min;94 ℃ 30 s,50 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 5 min。测序由上海美吉生物医药科技有限公司完成。
测序数据处理通过Quantitative insights into microbial ecology (QIIME)软件完成。PE reads首先根据Overlap关系进行拼接,对低质量的数据进行过滤;根据Barcode序列将序列按照样品来源进行分拣,对每个样品的序列数目进行统计;在97%相似度水平上,通过Usearch方法对序列进行操作分类单元(operational taxonomic units,OTU)划分,在聚类过程中去除嵌合体,得到OTU的代表序列,并生产OTU表格;采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析;基于OTU计算物种多样性指数,如菌群丰度指数Ace、菌群多样性指数Shannon和Simpson、测序深度指数Good’s coverage。
1.5 统计分析采用相关性分析、方差分析和Duncan检验、基于Bray-Curtis算法的非度量多维尺度分析(non-metric multidimensional scaling,NMDS)和ANOSIM分析对各测序方法得到的微生物群落组成异同进行比较分析。差异性微生物类群分析基于Welch’s t test,所用软件为Statistical analysis of metagenomic profiles (STAMP 2.1.3)[19]。
2 结果与分析 2.1 采油井产出液微生物群落16S rRNA基因数荧光定量PCR结果表明,阴性对照组扩增无信号,扩增效率达到90%,标准曲线R达到0.99,经计算本文所用油水样中微生物16S rRNA基因数量为(6.51±0.56)×108/L。
2.2 16S rRNA基因不同测序区域和深度测序概况及多样性指数所用样品V4区测序1−2万条、V4区测序10万条和V3−V4测序1−2万条,其文库覆盖率均达到99.6%以上;同一测序区域(V4),随测序深度增加,OTU数量明显增加(表 1);同V4区测序相比,V3−V4测序获得的OTU数量明显较少(表 1)。多样性指数分析表明,V4区测序1−2万和10万时,菌群Shannon和Simpson指数受测序深度影响不显著;与V4区测序相比,V3−V4区测序Shannon指数偏低,Simpson指数偏高。以上数据表明,对于同一测序区域,即使文库覆盖率已达到99.6% (如V4区测序,表 1),增加测序深度仍会有相当数量OTU被检测到;与V4区测序相比,在相近测序深度下(V3−V4区测序条带数略少),V3−V4区测序检测到的OTU数量明显减少,菌群Shannon多样性指数明显降低,但Simpson多样性指数和文库覆盖率更高。
| 测序方法 Sequencing method |
条数 Reads |
Coverage | OTUs | Shannon | Simpson | |
| V4区测序10万条 V4 with sequencing depth of 100 000 |
130 750 (4 858) | 0.999 6 | 440 (18)* | 3.085 (0.262)a | 0.161 (0.025)b | |
| V4区测序1−2万条 V4 with sequencing depth of 10 000−20 000 |
22 492 (5 494) | 0.996 2 | 340 (23)** | 3.153 (0.130)a | 0.150 (0.017)b | |
| V3−V4区1−2万条 V3−V4 with sequencing depth of 10 000−20 000 |
17 276 (6 635) | 0.999 6 | 145 (4) | 2.533 (0.050)b | 0.236 (0.012)a | |
| 注:以上数据均为平均值,括号中为标准偏差;V4区测序10万条2个平行,V4区和V3−V4区测序1−2万条各3个平行;*表示含16个古菌OTU (相对丰度为0.45%),**表示含15个古菌OTU (相对丰度为1.2%);Duncan多重比较结果的字母标记参考文献[20],小写字母表示α < 0.05水平. Note: The above data are averages of the parallel samples, with standard deviation in brackets; V4 with sequencing depth of 100 000 included two parallel samples, V4 and V3−V4 with sequencing depth of 10 000−20 000 included three parallel samples, respectively; * indicated that 16 archaeal OTUs were detected, ** indicated that 15 archaeal OTUs were detected; Letter markers for multiple comparison result refer to references [20], the lower-case letters represent α < 0.05. | ||||||
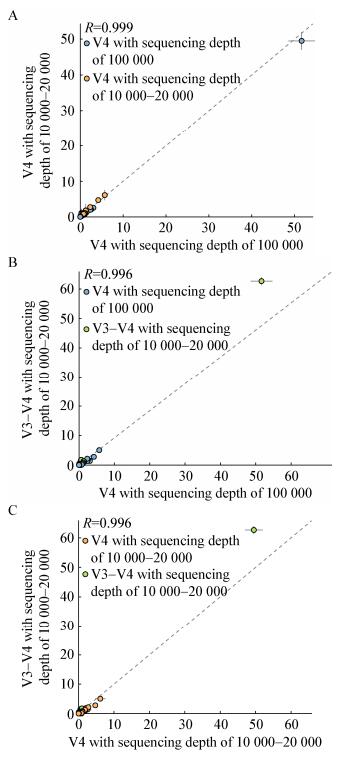
在纲水平上,V4区测序1−2万、V4区测序10万和V3−V4区测序1−2万3种测序方式获得的菌群中共有微生物类群所占百分比分别为98.15%、98.99%和99.5% (图 1A)。在属水平上,3种测序方式获得的菌群中共有微生物类群在V4区测序1−2万中占94.06%,在V4区测序10万中占94.61%,在V3−V4区测序1−2万中占98.13% (图 1B)。在属水平上对上述3种测序方式获得的菌群进行Pearson线性相关性分析表明,3种测序方式获得的菌群中共有微生物类群具有极为相近的相对丰度(图 2)。以上数据表明,3种测序方式获得的菌群中优势微生物类群(纲和属水平)及其相对丰度具有较高的一致性。

|
| 图 1 V4和V3−V4区不同测序深度获得菌群中共有微生物类群和特有微生物类群在各菌群纲水平(A)和属水平(B)中所占百分比 Figure 1 The abundances of different and shared microbial populations at class (A) and genus (B) level in the microbial communities obtained by 16S rRNA gene V4 and V3−V4 sequencing |
|
|
|
| 图 2 V4和V3−V4区不同测序深度获得菌群的Pearson线性相关性分析 Figure 2 The Pearson analysis for the microbial populations obtained by 16S rRNA gene V4 and V3−V4 sequencing |
|
|
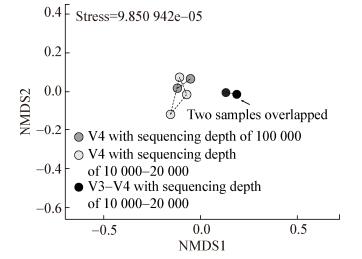
菌群β多样性分析表明,同一测序体系重复测序获得的菌群仍具有一定差异,但整体上优于不同测序体系获得的菌群,在NMDS图中表现为具有更近的距离(图 3)。相同测序区域(V4)不同测序深度(1−2万和10万条)在NMDS图中并未明显分开,不同测序区域(V4和V3−V4区)相同测序深度(1−2万条)获得的菌群在NMDS图中明显分开。即便如此,V4和V3−V4区测序获得的菌群间并未表现出显著性差异(ANOSIM分析,P=0.1)。
|
| 图 3 V4和V3−V4区不同测序深度获得菌群的NMDS分析 Figure 3 The NMDS analysis for the microbial communities obtained by 16S rRNA V4 and V3−V4 sequencing |
|
|
如表 2所示,两两显著性差异分析表明,在纲水平上,V4区测序10万与V4区测序1−2万两种测序深度下几乎不存在相对丰度具有显著性差异的微生物类群(图 4A);V4区测序10万与V3−V4区测序1−2万两种测序下优势微生物类群Deltaproteobacteria、Gammaproteobacteria和Betaproteobacteria相对丰度间存在显著性差异(图 4B,P < 0.05);V4区测序1−2万与V3−V4区测序1−2万两种测序下优势微生物类群Epsilonproteobacteria、Deltaproteobacteria、Alphaproteobacteria和Deferribacteres相对丰度间存在显著性差异(图 4C,P < 0.05)。在属水平上V4区测序10万与V4区测序1−2万两种测序深度下不存在相对丰度具有显著性差异的优势微生物类群(图 5A);V4区测序10万与V3−V4区测序1−2万两种测序下Desulfobotulus、Arcobacter、Sulfurospirillum、Desulfomicrobium、Desulfococcus、Geobacter、Desulfotignum和Desulfocapsa等微生物类群相对丰度间存在显著性差异(图 5B,P < 0.05),但这些微生物类群在群落中占比在5%以下;V4区测序1−2万与V3−V4区测序1−2万两种测序下相对丰度间存在显著差异的微生物类群数量增多(图 5C,P < 0.05),其中包含优势微生物Sulfurovum (V4区49.49%,V3−V4区62.67%),其他具有显著性差异的微生物类群在群落中占比在5%以下。
| 纲水平 Class level |
V4区测序10万 V4 with sequencing depth of 100 000 |
V4区测序1−2万 V4 with sequencing depth of 10 000−20 000 |
V3−V4区测序1−2万 V3−V4 with sequencing depth of 10 000−20 000 |
| Epsilonproteobacteria | 53.96 | 51.37 | 64.23 |
| Deltaproteobacteria | 17.99 | 17.96 | 11.40 |
| Gammaproteobacteria | 2.75 | 2.31 | 1.43 |
| Deferribacteres | 1.37 | 1.90 | 0.99 |
| Sphingobacteriia | 1.16 | 1.22 | 0.69 |
| Betaproteobacteria | 0.83 | 0.66 | 0.46 |
| Alphaproteobacteria | 0.12 | 0.12 | 0.05 |

|
| 图 4 V4和V3−V4区不同测序深度获得菌群中相对丰度具有显著差异的微生物类群(纲水平) Figure 4 The microbial populations that showed significant difference between the communities obtained by 16S rRNA gene V4 and V3−V4 sequencing (at the class level) |
|
|

|
| 图 5 V4和V3−V4区不同测序深度获得菌群中相对丰度具有显著差异微生物类群(属水平) Figure 5 The microbial populations that showed significant differences between the communities obtained by 16S rRNA gene V4 and V3−V4 sequencing (at the genus level) |
|
|
鉴于Illumina MiSeq高通量测序平台读长及测序成本等问题,基于引物515f和806r扩增的V4区测序被广泛使用,是发起于2010年的地球微生物组计划(Earth Microbiome Project,http://www.earthmicrobiome.org/)建议使用的测序引物及区域。引物515f和806r由Caporaso等设计,对微生物覆盖度高,同时也可以检测到部分古菌类群[10]。然而Parada等发现该引物对会对Gammaproteobacteria造成高估,因此对其进行了优化[21]。这一现象在本文中也有所体现,如同一测序深度下,V4区测序获得的菌群中Gammaproteobacteria的相对丰度是2.31%,高于V3−V4区测序获得的菌群中Gammaproteobacteria相对丰度是1.43%。基于引物338f和806r对V3−V4区进行扩增,可获得约460 bp扩增子。更长的序列信息意味着可用于反映种属亲缘关系的位点数增多,能够更准确地进行物种分类分析。与此同时,对测序平台的读长要求也相应提高,测序成本相应增加,如相对V4区测序(约290 bp),单端测序150 bp的读长即可满足分析需求,而V3−V4区单端测序读长至少要达到250 bp。现阶段Illumina测序平台读长已经可以达到400 bp,测序成本也在不断降低。
16S rRNA基因测序区域和测序深度对菌群α多样性指数分析具有明显影响。就本文所用样本,V4区测序深度由1−2万条增加至10万条,文库覆盖率仅由99.62%增加至99.96%,但被检测到的OTU数量由340个(含15个古菌OTU)增加到440个(含16个古菌OTU)。多出来的OTU大多仅出现一次,这表明增加测序深度可以检测到样本中极低丰度微生物类群。由于测序平台、序列质控、序列拼接及OTU划分等过程完全相同,因此测序深度增加是导致OTU数量大幅度增加的根本原因。由于共同检测到的微生物类群在菌群中所占比重高(在属水平上,本文涉及到的3种测序方式获得的菌群中共有微生物类群在V4区测序1−2万中占94.06%,在V4区测序10万中占94.61%),差异微生物类群在菌群中所占比重极低,测序深度对菌群β多样性并未表现出显著影响。在测序深度略高的情况下,V4区测序获得了340个OTU (含15个古菌OTU),明显高于V3−V4区测序的145个OTU;V4区测序Shannon多样性指数显著高于V3−V4区测序,但Simpson多样性指数和文库覆盖率却明显低于V3−V4区测序。上述现象表明,V4区测序可能会导致对菌群的α多样性指数高估。值得关注的是,V4区测序10万计算出的α多样性指数比V4区测序1−2万更接近于V3−V4区测序1−2万。
无论是纲水平还是属水平,V4区测序10万与1−2万两种测序深度下几乎不存在相对丰度具有显著性差异的微生物类群,这应该与此两种测序深度下文库覆盖率分别达到99.96%和99.62%相关。此外,由于测序平台、测序引物、数据处理方法等完全相同,可以排除系统误差。因此,只要达到相应的测序深度,获得的菌群数据可以有效反映整个菌群组成。V3−V4区测序与V4区测序相比,同样测序深度下,相对丰度具有显著性差异的微生物类群数量增多,其中包含优势微生物类群Epsilonproteobacteria (51.37%:64.23%)和Deltaproteobacteria (17.96%:11.40%)。
本文数据反映了16S rRNA基因不同测序区域和测序深度对同一测序样本菌群分析的影响:对于特定样品,测序深度达到一定水平(送样测序前检测样本菌群基因组16S rRNA拷贝数,根据拷贝数确定测序深度),不同测序深度对菌群组成分析的影响十分有限,但增加测序深度会一定程度影响菌群α多样性指数;V4区与V3−V4区测序获得的菌群α多样性指数明显不同,且存在相对丰度具有显著差异的优势微生物类群。鉴于测序读长的提升和测序成本降低,以及在更低的测序深度下V3−V4区测序文库覆盖率更高,而且可提供更多可用于反映物种亲缘关系的16S rRNA碱基信息,V3−V4区测序可作为当下菌群分析的首选区域。此外,不同领域微生物群落分析在满足测序深度需要的前提下均应采用统一的测序平台、测序引物和数据处理方法,从而有助于不同研究者对不同采样位点产生的实验数据进行Meta分析,在更大的时空尺度上揭示特定环境(如油藏)微生物群落组成特征及演替规律。
本文实验设计之初考虑到V4区测序扩增子长度约290 bp,V3−V4区测序扩增子长度约460 bp,因此,V4区测序由MiSeq PE250测序平台(单端测序长度可达250 bp)完成,V3−V4区测序由MiSeq PE300测序平台(单端测序长度可达300 bp)完成。尽管MiSeq PE250和MiSeq PE300测序准确度都较高,但依旧无法排除因测序平台不同可能对菌群分析造成的影响。
| [1] |
Youssef N, Elshahed MS, McInerney MJ. Microbial processes in oil fields: culprits, problems, and opportunities[J]. Advances in Applied Microbiology, 2009, 66: 141-251. DOI:10.1016/S0065-2164(08)00806-X |
| [2] |
Zheng P. Environmental Microbiology[M]. 2nd ed. Hangzhou: Zhejiang University Press, 2012. (in Chinese) 郑平. 环境微生物学[M]. 2版. 杭州: 浙江大学出版社, 2012. |
| [3] |
Li H, Mu BZ. Recent advances in molecular microbial ecology of petroleum rese[J]. Microbiology China, 2008, 35(5): 803-808. (in Chinese) 李辉, 牟伯中. 油藏微生物多样性的分子生态学研究进展[J]. 微生物学通报, 2008, 35(5): 803-808. DOI:10.3969/j.issn.0253-2654.2008.05.027 |
| [4] |
Shendure J, Ji H. Next-generation DNA sequencing[J]. Nature Biotechnology, 2008, 26(10): 1135-1145. DOI:10.1038/nbt1486 |
| [5] |
Raap AK. Advances in fluorescence in situ hybridization[J]. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 1998, 400(1/2): 287-298. |
| [6] |
Hu Q, Qi HY, Zhang HX. Fluorescence in situ hybridization (FISH) and its applications in microbial ecology[J]. Acta Ecologica Sinica, 2004, 24(5): 1048-1054. (in Chinese) 呼庆, 齐鸿雁, 张洪勋. 荧光原位杂交技术及其在微生物生态学中的应用[J]. 生态学报, 2004, 24(5): 1048-1054. DOI:10.3321/j.issn:1000-0933.2004.05.028 |
| [7] |
Zhao LX, Ma T, Gao PK, et al. Characterization of microbial diversity and community in water flooding oil reservoirs in China[J]. World Journal of Microbiology and Biotechnology, 2012, 28(10): 3039-3052. DOI:10.1007/s11274-012-1114-2 |
| [8] |
He ZL, Deng Y, van Nostrand JD, et al. GeoChip 3.0 as a high-throughput tool for analyzing microbial community composition, structure and functional activity[J]. The ISME Journal, 2010, 4(9): 1167-1179. DOI:10.1038/ismej.2010.46 |
| [9] |
Sagaram US, DeAngelis KM, Trivedi P, et al. Bacterial diversity analysis of Huanglongbing pathogen-infected citrus, using PhyloChip arrays and 16S rRNA gene clone library sequencing[J]. Applied and Environmental Microbiology, 2009, 75(6): 1566-1574. DOI:10.1128/AEM.02404-08 |
| [10] |
Caporaso JG, Lauber CL, Walters WA, et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(Suppl 1): 4516-4522. |
| [11] |
Xiao M, Sun SS, Zhang ZZ, et al. Analysis of bacterial diversity in two oil blocks from two low-permeability reservoirs with high salinities[J]. Scientific Reports, 2016, 6: 19600. DOI:10.1038/srep19600 |
| [12] |
Lenchi N, İnceoğlu Ö, Kebbouche-Gana S, et al. Diversity of microbial communities in production and injection waters of Algerian oilfields revealed by 16S rRNA gene amplicon 454 pyrosequencing[J]. PLoS One, 2013, 8(6): e66588. DOI:10.1371/journal.pone.0066588 |
| [13] |
An DS, Caffrey SM, Soh J, et al. Metagenomics of hydrocarbon resource environments indicates aerobic taxa and genes to be unexpectedly common[J]. Environmental Science & Technology, 2013, 47(18): 10708-10717. |
| [14] |
Liu YF, Galzerani DD, Mbadinga SM, et al. Metabolic capability and in situ activity of microorganisms in an oil reservoir[J]. Microbiome, 2018, 6: 5. DOI:10.1186/s40168-017-0392-1 |
| [15] |
Li GQ, Gao PK, Wu YQ, et al. Microbial abundance and community composition influence production performance in a low-temperature petroleum reservoir[J]. Environmental Science & Technology, 2014, 48(9): 5336-5344. |
| [16] |
Song YT, Hu J, Wu XL, et al. Stability of microbial community structure in reservoir water samples at room temperature[J]. Chinese Journal of Applied & Environmental Biology, 2017, 23(3): 495-501. (in Chinese) 宋永亭, 胡婧, 吴晓玲, 等. 室温条件下油藏采出液微生物群落结构稳定性[J]. 应用与环境生物学报, 2017, 23(3): 495-501. |
| [17] |
Hu J, Sun GZ, Liu T, et al. Spatial distribution of indigenous microbial communities under simulated reservoir conditions[J]. Microbiology China, 2019, 46(5): 1008-1017. (in Chinese) 胡婧, 孙刚正, 刘涛, 等. 模拟油藏条件下内源微生物群落空间分布规律[J]. 微生物学通报, 2019, 46(5): 1008-1017. |
| [18] |
Gittel A, Sørensen KB, Skovhus TL, et al. Prokaryotic community structure and sulfate reducer activity in water from high-temperature oil reservoirs with and without nitrate treatment[J]. Applied and Environmental Microbiology, 2009, 75(22): 7086-7096. DOI:10.1128/AEM.01123-09 |
| [19] |
Parks DH, Tyson GW, Hugenholtz P, et al. STAMP: statistical analysis of taxonomic and functional profiles[J]. Bioinformatics, 2014, 30(21): 3123-3124. DOI:10.1093/bioinformatics/btu494 |
| [20] |
Qiu NW, Liu Q, Liu H. Multiple comparisons of biological experimental data in statistical analysis[J]. Journal of Biomathematics, 2015, 30(3): 535-541. (in Chinese) 邱念伟, 刘倩, 刘慧. 生物学实验数据统计分析中的多重比较法[J]. 生物数学学报, 2015, 30(3): 535-541. |
| [21] |
Parada AE, Needham DM, Fuhrman JA. Every base matters: assessing small subunit rRNA primers for marine microbiomes with mock communities, time series and global field samples[J]. Environmental Microbiology, 2016, 18(5): 1403-1414. DOI:10.1111/1462-2920.13023 |
 2020, Vol. 47
2020, Vol. 47




