扩展功能
文章信息
- 张志东, 朱静, 唐琦勇, 顾美英, 楚敏
- ZHANG Zhi-Dong, ZHU Jing, TANG Qi-Yong, GU Mei-Ying, CHU Min
- 不同扩增引物对高通量测序分析盐角草内生真菌多样性的影响
- Effects of different amplification primers on diversity analysis of endophytic fungi in Salicornia europaea by the high-throughput sequencing
- 微生物学通报, 2020, 47(2): 411-419
- Microbiology China, 2020, 47(2): 411-419
- DOI: 10.13344/j.microbiol.china.190448
-
文章历史
- 收稿日期: 2019-05-23
- 接受日期: 2019-09-20
- 网络首发日期: 2019-10-22
植物内生菌(plant endophyte)是指与植物体共存,但不引起植物产生明显病症的一类微生物。自20世纪初首次报道植物内生真菌以来,几乎在所有研究的植物组织内都发现了内生真菌,且具有丰富的种群多样性,存在植物类群、属种间分布的偏好性[1-3]。近年来,利用PCR技术扩增真菌ITS序列的某一区间,采用高通量测序技术可更方便和全面地认知目标植物内生菌群落组成和多样性。然而研究表明多种因素会影响高通量测序分析样本真菌的群落丰度和多样性,如起始DNA浓度、真菌核糖体拷贝数及扩增引物,其中部分扩增引物存在明显真菌种群偏好性[4]。Taylor等[5]设计了一对ITS引物,可以最大限度地覆盖真菌界,同时最少地扩增非目标真核生物;de Filippis等[6]使用不同扩增引物对一个模拟群落和几个样本进行了分析,证明使用不同PCR引物扩增会影响真菌微生物群落分析的结果,并认为扩增ITS序列分析会导致实验结果中真菌多样性高估;然而Řezáčová等[7]分别对菌根真菌的SSU、LSU和ITS目的序列扩增,却发现采用ITS引物仅能获得样本的25%的OTU,显著低于其它引物所得结果。目前,国内学者在高通量测序中也会选用不同的引物扩增[8]。
盐角草(Salicornia europaea)又名欧洲海蓬子,藜科一年生草本植物,是地球上最耐盐的陆生高等植物之一,可以在含盐浓度高达6.5%的潮湿盐碱地中正常生长,广泛分布在海滩、盐碱滩涂沙地[9]。盐角草属多种植物可被食用或药用[10-11],同时也是植物耐盐机制、耐盐基因及转基因植物操作等研究的热点[12-13],常作为盐碱地改良和重金属修复植物[14]。Petrini等[15]最早从Salicornia perenni的茎中分离出32株内生真菌,分析发现在植株的新生茎和老茎组织中内生真菌定殖有显著差异。国内相继从该属的海芦笋(Salicornia bigelovii)中分离出Phoma、Fusarium、Cunninghamella等属内生真菌[16-18],表明盐角草属植物内生真菌存在着一定的分布差异,并可产生多种功能代谢产物。
本研究利用Illumina MiSeq高通量测序技术,对新疆达板城盐湖的盐角草(S. europaea)内生真菌群落组成进行了分析,同时对不同引物扩增下盐角草(S. europaea)内生真菌高通量测序分析的影响进行了解析,为进一步挖掘盐角草(S. europaea)蕴藏的内生真菌提供了研究基础,也为相关植物内生真菌的高通量测序研究提供了科学借鉴。
1 材料与方法 1.1 材料采样区域位于天山山脉中段天格尔山北麓,乌鲁木齐市东南,达坂城盐湖东岸沿岸盐渍区域(N 43.402 080°,E 88.121 986°)。采样地点海拔1 066 m,土质上层5 cm为泥土,下层为砂石,盐渍区域内除盐角草(S. europaea)分布外,还有沙葱、芦苇等耐盐植物分布。
主要试剂和仪器:E.Z.N.A.® Fungal DNA Kit提取试剂盒,Qmega Biotek有限公司;乙醇、次氯酸钠,北京化学试剂公司。高速离心机、PCR仪,Eppendorf公司。
1.2 样品采集及方法盐角草(S. europaea)样品采集于2018年7月4日,采取蛇形取样,每个地点采集3个重复,每个点间距离大约20-50 m。选择生长状况相近的健康个体,采用垂直挖掘法,尽可能地保证植株的完整性。所采集样品装入灭菌的牛皮纸袋内,并放入车载冰箱(4 ℃)后迅速带回实验室开展相关样品的处理。
1.3 样品表面消毒和处理将完整植株分别使用自来水冲洗10 min,晾干。采用全株表面消毒方式,将植株依次使用70%乙醇浸泡1 min,3.3%次氯酸钠浸泡5 min,75%乙醇浸泡0.5 min,再经无菌水反复冲洗后,用无菌滤纸吸干,并收集冲洗后的无菌水进行消毒效果验证[19]。
1.4 基因组DNA提取、ITS-PCR扩增和测序将消毒后的植株液氮冷冻10 min,然后进行组织研磨。采用E.Z.N.A.® Fungal DNA Kit提取内生真菌总DNA。样品DNA通过分别提取再合并混匀后进行PCR扩增。采用ITS1-5F引物对(ITS5:5′-GGAAGTAAAAGTCGTAACAAGG-3′,ITS2:5′-GCTGCGTTCTTCATCGATGC-3′)扩增ITS1区序列[20],PCR反应条件[21]:94 ℃ 30 s;55 ℃ 30 s;72 ℃ 30 s,35个循环;采用ITS1-1F引物对(ITS1F:5′-CTTGGTCATTTAGAGGAAGTAA-3′,ITS2:5′-GCTGCGTTCTTCATCGATGC-3′)扩增ITS1区序列[20, 22],PCR反应条件[23]:94 ℃ 30 s;52 ℃ 45 s;72 ℃ 1 min,35个循环;采用ITS2引物对(ITS3:5′-GCATCGATGAAGAACGCAGC-3′,ITS4:5′-TC CTCCGCTTATTGATATGC-3′)扩增ITS2区序列[20],PCR反应条件[22]:94 ℃ 45 s;60 ℃ 45 s;72 ℃ 1 min,35个循环。上述PCR产物经过切胶纯化后由北京诺禾致源生物信息科技有限公司完成测序。
1.5 生物信息学分析从Illumina HiSeq测序平台得到的下机数据,进行拼接、质控和嵌合体过滤,得到可用于后续分析的有效数据,即Effective Tags。用UCLUST V1.1.579对所有样品的Effective Tags进行聚类[24],以97%的一致性(identity)将序列聚类成为操作分类单元(operational taxonomic units,OTU)构建稀释曲线。序列比对分别采用UNITE数据库(https://unite.ut.ee/analysis.php)和NCBI数据库(https://www.ncbi.nlm.nih.gov),并进行序列注释。不同ITS扩增区域所得OTU如注释相同,分别将相关序列与同一参比菌株序列进行手工比对,如两者与参比序列相似性均大于97%,视为1个OTU,反之则视为2个不同的OTU。其它相关数据结果处理使用Microsoft Excel 2013和DPS 9.05软件进行分析。
2 结果与分析 2.1 测序结果及序列深度验证通过使用不同引物对样品中内生真菌ITS序列进行扩增和测序,3个引物对扩增总计测得原始序列条数为240 434条,过滤掉低质量的序列,去冗余处理后,质控序列数为217 308条,在97%相似度下将其聚类用于物种分类的OTU,总计获得122个,再去除宿主及外源动植物的OTU,总计获得盐角草(S. europaea)内生真菌OTU 102个,总有效序列数为72 537条,其中采用ITS1-1F引物对扩增的有效序列数最高,占87.0%,而采用ITS2引物对扩增的有效序列数最低,仅为1.6%,因此,采用ITS1-1F引物对可以较好地避免宿主和外源动植物的影响。各引物对扩增后测序信息结果见表 1。
| 样品编号 Samples |
原始序列数 Raw number |
质控序列数 Clean number |
总OTU Total OTUs |
有效序列数 Effective number |
有效OTU Effective OTUs |
| ITS1-5F | 80 228 | 75 616 | 70 | 1 579 | 44 |
| ITS1-1F | 80 101 | 71 038 | 88 | 69 711 | 55 |
| ITS2 | 80 105 | 70 654 | 35 | 1 247 | 25 |
| Total | 240 434 | 217 308 | 122 | 72 537 | 102 |
稀释曲线反映了测序的深度,也可以用来评价测序量是否足以覆盖样品所有种群。从图 1可知,当测序数量达到25 000条时,3种处理的稀释曲线均基本趋于平缓。继续加大测序量,当测序数量达到70 000条时,采用ITS1-1F引物对扩增的样品,OTU仅增加6个,约占该样品中总OTU数的6.8%,而从47 000-70 000条间,OTU仅增加1个;其它处理OTU在相应区间变化更小,说明实验所得OTU数已接近最大可测数量,即使再加大测序量,也难以更多地反映真实环境中内生真菌菌落结构。
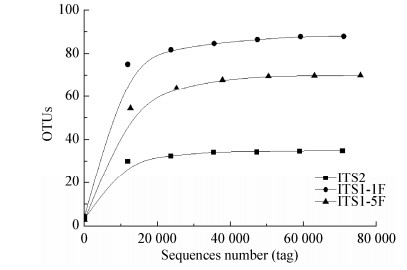
|
| 图 1 不同引物对扩增下样品内生真菌的稀释曲线 Figure 1 Rarefaction curves for the sequencing with different PCR primers 注:OTU数为所有测样所得OTU数,包括植物、昆虫等. Note:Total OTUs were showed including host plant and bugs. |
|
|
根据各引物对所得序列OTU注释结果分析,去除非真菌OTU后,合并分类相同的OTU,构建韦恩图(图 2)。不同引物所得序列共获得真菌OTU数为102个,其中以ITS1-1F引物对扩增获得55个OTU,以ITS1-5F引物对扩增获得44个OTU,以ITS2引物对扩增获得25个OTU。3种引物扩增均检测到的OTU为5个,占总共获得OTU的4.9%。ITS1-1F和ITS1-5F两种引物对扩增重叠检测出的OTU为13个,占总数的12.7%,这一结果表明采用不同引物进行扩增所得的盐角草(S. europaea)内生真菌信息存在明显的差异;而两者合计所得OTU为86个,占全部OTU数的84.3%,表明采用ITS1-1F和ITS1-5F两种引物对可基本反应盐角草(S. europaea)内生真菌的主要群落组成(表 2)。

|
| 图 2 不同引物对扩增下样品内生真菌OTU相关性 Figure 2 The similarity analysis of fungal communities |
|
|
| Primers | ITS1-1F | ITS1-5F | ITS2 | Rate (%) |
| ITS1-1F | 55 | 12.7% | 8.8% | 53.9 |
| ITS1-5F | 13 | 44 | 6.8% | 43.1 |
| ITS2 | 9 | 7 | 25 | 24.5 |
| 注:表格对角线下半部分数据为两两处理共有OTU数,上半部分百分数据是两两共有OTU数占全部的百分比. Note: The numbers below the diagonal line of the table were OTUs detected in two treatments; The data above the diagonal line were percentage of OTUs detected in two treatments to total OTUs. | ||||
采用α多样性指标中物种数、Simpson (J)、Shannon (H)、Brillouin指数对样品群落的异质性以及估计群落中物种总数,一般而言Simpson (J)指数对富集种更加敏感,Shannon (H)对稀疏种更为敏感。由表 3可以看出,ITS1-1F引物对扩增下OTU数明显高于ITS2、ITS1-5F,而Simpson (J)、Shannon (H)、Brillouin指数均低于后两者,表明采用ITS1-1F引物对能更多地获得内生真菌群落组成信息,但检出的物种间相对丰度差异较大,物种间均匀度降低。其中,Simpson (J)降低表明采用ITS1-1F引物对能明显增加优势菌群的检出率,而Shannon (H)降低表明该引物能更多覆盖低丰度的真菌种群。
| 引物 Primers |
OTUs | Simpson (J) | Shannon (H) | Brillouin |
| ITS2 | 25 | 0.855 9 | 3.305 9 | 3.255 0 |
| ITS1-1F | 55 | 0.669 4 | 2.486 6 | 2.483 4 |
| ITS1-5F | 44 | 0.892 4 | 4.004 0 | 3.918 3 |
通过对样品中102个OTU注释,依次进行门(phylum)、纲(class)、目(order)、科(family)、属(genus)等分类信息分析,进一步挖掘各引物对扩增对内生真菌群落组成分析的影响。研究发现,3种引物对所获得的102个OTU共涉及真菌界的隐真菌门(Rozellomycota)、毛霉菌门(Mucoromycota)、被孢霉门(Mortierellomycota)、单毛壶菌门(Monoblepharomycota)、梳霉门(Kickxellomycota)、球囊霉门(Glomeromycota)、担子菌门(Basidiomycota)、子囊菌门(Ascomycota)等8个菌门以及未分类菌群(图 3)。其中,子囊菌门(Ascomycota)占绝对优势,共计69个OTU,占总数的67.6%;其次为担子菌门(Basidiomycota),共计20个OTU,占总数的19.6%;再次被孢霉门(Mortierellomycota),总计有5个OTU,其它有5个门和未知菌群有8个OTU,合计占总数的7.8%。

|
| 图 3 盐角草内生真菌OTU在门水平分布情况 Figure 3 Frequency of phyla in fungi communities from sample |
|
|
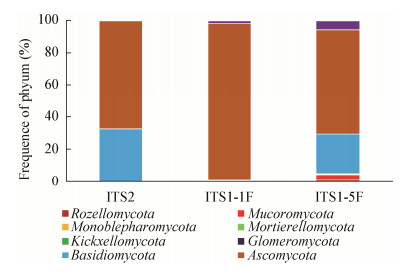
进一步对不同引物对扩增下测序结果中OTU在真菌门水平的分布情况进行分析(图 4),发现采用ITS2引物对扩增下仅获得子囊菌门(Ascomycota)的20个OTU和担子菌门(Basidiomycota)的5个OTU。而采用ITS1-5F引物对扩增能获得除毛霉门(Mucoromycota)外的其它菌门,包括子囊菌门(Ascomycota)的24个OTU、担子菌门(Basidiomycota)的12个OTU、隐真菌门(Rozellomycota)的1个OTU、被孢霉门(Mortierellomycota)的1个OTU、单毛壶菌门(Monoblepharomycota)的1个OTU、梳霉门(Kickxellomycota)的1个OTU、球囊霉门(Glomeromycota)的1个OTU和未分类菌群的3个OTUs;采用ITS1-1F引物对扩增获得子囊菌门(Ascomycota)的41个OTU、担子菌门(Basidiomycota)的6个OTU、毛霉门(Mucoromycota)的1个OTU、被孢霉门(Mortierellomycota)的5个OTU和未分类菌群的2个OTU。由于ITS1-5F和ITS2引物对扩增所得序列中均存在大量植物宿主和外源动植物序列干扰,而ITS1-1F较少扩增到宿主基因,建议在多样性分析中应以ITS1-1F为主,其它作为佐证。

|
| 图 4 不同引物扩增下门水平菌群OTU分布 Figure 4 Frequence of OTUs in phyla with different PCR primers |
|
|
另外,通过对分布在各真菌门OTU在各引物对扩增获得总OTU的比例分析,各种引物对扩增而担子菌门OTU,在采用不同引物对所得OTU比例变化比较明显,采用ITS1-5F引物对扩增所得的子囊菌门OTU均为绝对优势,其所占的比例按照ITS2、ITS1-1F和ITS1-5F逐步下降。ITS1-1F扩增,前者比例提高了1倍。被孢霉门(Mortierellomycota)仅在采用ITS1-1F和ITS1-5F引物对扩增获得,但二者相差近5倍。
进一步对各门所有OTU的序列数进行分析(图 5)可以看出,采用ITS1-1F引物对扩增测序中,子囊菌门(Ascomycota)占绝对优势,占总有效序列数的97.7%,而其余所占比例不足3%。采用引物对ITS2和ITS1-5F扩增,内生真菌中主要菌群序列数所占比例类似;ITS2引物对扩增仅获得2个门的OTU,两者所占比例分别为子囊菌门占67.2%和担子菌门32.7%;采用ITS1-5F引物对,子囊菌门占64.9%和担子菌门24.3%,其它占10.8%。
|
| 图 5 不同引物扩增下门水平菌群序列百分比 Figure 5 Phylum-level distribution of fungi with different PCR primers |
|
|
通过进一步分析,实验获得的102个OTU分布在真菌界的64个属及20个未分类属中,表明盐角草(S. europaea)存在着丰富多样的内生真菌资源,且存在着大量潜在新种属。采用不同引物扩增分析主要种属存在着明显的差异(表 4),Alternaria属在采用不同引物扩增情况分析下均为优势属(> 20%)。采用ITS1-1F、ITS1-5F引物对扩增分析表明,在所占比例排名前5的属序列数占各自总数的79.2%和64.3%,Alternaria、Cladosporium、Podospora均排名前5,是盐角草内生真菌的主要菌属。在种属分布中,除Coniophora、Mortierella、Podospora、Fusarium、Pichia、Penicillium、Stemphylium、Alternaria、Cladosporium等属有2个OTU外(表 5),其它属均只有1个OTU,表明盐角草内生真菌在种类分布上较均匀。
| ITS2 | Ratio (%) | ITS1-1F | Ratio (%) | ITS1-5F | Ratio (%) |
| Wallemia | 23.3 | Alternaria | 55.30 | Alternaria | 23.2 |
| Cutaneotrichosporon | 23.3 | Others | 19.46 | Inocybe | 15.6 |
| Alternaria | 22.1 | Podospora | 19.10 | Podospora | 13.1 |
| Candida | 10.3 | Aureobasidium | 3.90 | Others | 12.1 |
| Cladosporium | 5.90 | Cladosporium | 0.60 | Cladosporium | 8.3 |
| Issatchenkia | 3.50 | Stemphylium | 0.30 | Arthrocladium | 4.1 |
| Podospora | 2.50 | Saitozyma | 0.30 | Mortierella | 3.5 |
| Aureobasidium | 2.50 | Mortierella | 0.23 | Coniophora | 2.7 |
| Papiliotrema | 1.20 | Ascobolus | 0.20 | Neocosmospora | 2.6 |
| Pleospora | 0.86 | Penicillium | 0.20 | Hohenbuehelia | 2.5 |
| 属名 Genus |
OTUs | 注释菌种名称 Closet species |
序列数量 Sequences numbers |
| Podospora | 6 | Podospora cochleariformis | 31 |
| Podospora sp.1 | 5 730 | ||
| Podospora sp.2 | 3 420 | ||
| Podospora sp.3 | 1 589 | ||
| Podospora sp.4 | 405 | ||
| Podospora sp.5 | 2 166 | ||
| Alternaria | 4 | Alternaria alternata | 38 180 |
| Alternaria sp.1 | 958 | ||
| Alternaria sp.2 | 69 | ||
| Alternaria sp.3 | 48 | ||
| Mortierella | 3 | Mortierella humilis | 42 |
| Mortierella sp.1 | 96 | ||
| Mortierella sp.2 | 59 | ||
| Fusarium | 2 | Fusarium oxysporum | 29 |
| Fusarium proliferatum | 10 | ||
| Cladosporium | 2 | Cladosporium colombiae | 208 |
| Cladosporium sphaerospermum | 375 | ||
| Penicillium | 2 | Penicillium simplicissimum | 105 |
| Penicillium rubens | 30 | ||
| Stemphylium | 2 | Stemphylium solani | 156 |
| Stemphylium sp.1 | 79 | ||
| Coniophora | 2 | Coniophora olivacea | 20 |
| Coniophora sp.1 | 22 |
引物的选择对高通量分析真菌多样性研究具有较大的影响,尽管真菌的SSU、LSU和ITS目的片段序列均可以用来进行真菌群落组成分析,但国内有关内生真菌研究多数采用ITS序列进行群落分析。由于在提取内生真菌DNA过程中难免会混杂植株自身DNA,也必然要求我们在PCR扩增时对引物进行相应的选择。目前,有关真菌ITS序列扩增所用的引物较多,如国内利用ITS5-1737F/ITS1-2043R、BITS/B58S3、ITS1-F/ITS2等先后对沙冬青、白芨、手参等进行了内生真菌的高通量分析[25-27],尽管不同作者一般均会选择具有真菌扩增的特异引物,但是难以保证不同引物对高通量测序结果产生影响。本研究分别采用高通量测序公司常用真菌引物对ITS2、ITS1-1F、ITS1-5F等3对引物,通过PCR扩增和高通量测序,结果发现不同引物扩增对盐角草(S. europaea)内生真菌群落组成分析有着明显的影响。在实验全部所得的102个真菌OTU中,利用ITS1-1F引物对获得55个OTU,ITS1-5F引物对获得44个OTU,ITS2引物对仅获得25个OTU,但3种引物共同获得到的OTU数仅为5个,表明不同引物扩增所得的物种组成信息均存在不全面的现象;使用引物ITS2和ITS1-5F引物对扩增所得序列中均存在大量植物宿主序列和外源动植物序列。客观地说,在提取总DNA中难免会提取出植物宿主DNA,并在含量中占总DNA的主要部分,因此在采用上述两对对真菌无特异性引物时,扩增所得序列中绝大多数序列为植物宿主序列也是必然的,但也造成了内生真菌序列扩增和检测到的序列数量有限,但两者较传统培养法分析仍具有数量可观、物种多样的优点。而ITS1-1F引物对具有真菌扩增特异性,能有效避免宿主序列的干扰,但也存在样品中内生真菌种类检测有遗漏的现象。通过分析我们发现同时采用ITS1-1F和ITS1-5F两种引物对扩增,两者合计所得OTU数可占全部OTU数的84.3%,可基本反映盐角草内生真菌的主要群落组成。因此,基于上述结果,推荐在内生真菌多样性分析中以ITS1-1F引物对为主,ITS1-5F引物对为辅的分析策略,进行植物内生菌的群落组成分析。
多样性分析表明,采用ITS1-1F引物对扩增下内生真菌OTU数明显高于ITS2、ITS1-5F,而Simpson (J)、Shannon (H)、Brillouin指数均低于后两者,表明采用ITS1-1F引物对能更多地获得内生真菌群落组成信息,但检出的物种间相对丰度差异变大,物种间均匀度降低。其中,Simpson (J)降低表明采用ITS1-1F引物能明显增加优势菌群的检出,而Shannon (H)降低表明该引物能更多覆盖低丰度的真菌种群。但就本实验而言,盐角草(S. europaea)内生真菌不同物种间丰度是未知的,采用ITS1-1F引物是否会导致高估了其内生真菌丰度间的差异,还有待通过更多的不同目的区间片段扩增,同时采用模拟系统进行评估。
3.2 盐角草内生真菌组成和多样性盐角草属植物作为地球上最耐盐的植物之一,目前国内外有关其内生真菌的研究仅有零星报道。Petrini等[15]在1986年首次对盐角草属植物S. perenni内生真菌进行研究,从其茎中分离到32株内生真菌,发现各类真菌在新旧植物组织中分布存在显著差异,Pleospora salicorniae在茎中都有定殖,而Pleospora bjorlingii主要存在于老的植物组织中,Stagonospora主要存在于新的植物组织中。国内学者[16-18]从海芦笋(S. bigelovii)中分离出Cunninghamella、Phoma、Fusarium等属的内生真菌,并表现出多种功能活性,发现并发表了真菌新物种Cunninghamella bigelovii。本研究通过高通量测序技术对盐角草(S. europaea)内生真菌进行了研究,实验共获得102个内生真菌OTU,涉及真菌界的8个菌门以及未分类菌群,其中子囊菌门(Ascomycota)占绝对优势,其次为担子菌门(Basidiomycota)。在属层次水平上,盐角草内生真菌共涉及64个属及20个未分类属,而Alternaria、Cladosporium、Podospora等3个属为优势菌属,也发现了已报道的盐角草属植物内生菌Pleospora、Stagonospora、Cunninghamella、Phoma、Fusarium等属OTU的存在,并预示着可能存在多个潜在真菌新种属,为进一步挖掘盐角草(S. europaea)丰富的内生真菌资源提供了科学依据。
| [1] |
Hao XJ. Plant Endophyte[M]. Beijing: China Agricultural University Press, 2010: 1-10. (in Chinese) 郝晓娟. 植物内生菌[M]. 北京: 中国农业科学技术出版社, 2010: 1-10. |
| [2] |
Akutse KS, Maniania NK, Fiaboe KKM, et al. Endophytic colonization of Vicia faba and Phaseolus vulgaris (Fabaceae) by fungal pathogens and their effects on the life-history parameters of Liriomyza huidobrensis (Diptera: Agromyzidae)[J]. Fungal Ecology, 2013, 6(4): 293-301. DOI:10.1016/j.funeco.2013.01.003 |
| [3] |
Rodriguez RJ, White Jr JF, Arnold AE, et al. Fungal endophytes: diversity and functional roles[J]. New Phytologist, 2009, 182(2): 314-330. DOI:10.1111/j.1469-8137.2009.02773.x |
| [4] |
Bokulich NA, Mills DA. Improved selection of internal transcribed spacer-specific primers enables quantitative, ultra-high-throughput profiling of fungal communities[J]. Applied and Environmental Microbiology, 2013, 79(8): 2519-2526. DOI:10.1128/AEM.03870-12 |
| [5] |
Taylor DL, Walters WA, Lennon NJ, et al. Accurate estimation of fungal diversity and abundance through improved lineage-specific primers optimized for illumina amplicon sequencing[J]. Applied and Environmental Microbiology, 2016, 82(24): 7217-7226. DOI:10.1128/AEM.02576-16 |
| [6] |
de Filippis F, Laiola M, Blaiotta G, et al. Different amplicon targets for sequencing-based studies of fungal diversity[J]. Applied and Environmental Microbiology, 2017, 83(17): e00905-17. |
| [7] |
Řezáčová V, Gryndler M, Bukovská P, et al. Molecular community analysis of arbuscular mycorrhizal fungi—contributions of PCR primer and host plant selectivity to the detected community profiles[J]. Pedobiologia, 2016, 59(4): 179-187. DOI:10.1016/j.pedobi.2016.04.002 |
| [8] |
Chen ZB, Xia TY, Wang DK, et al. Diversity analysis of endophytic fungi in Mentha using Illumina MiSeq high-throughput sequencing technology[J]. Medicinal Plant, 2016, 7(S1): 46-50. |
| [9] |
Zhao HM. Eco-physiological characteristics of halophyte salt-worts and their potential utilization in environmental mediation and germplasm improvement[J]. Bulletin of Science and Technology, 2004, 20(2): 167-171. (in Chinese) 赵惠明. 盐生植物盐角草的资源特点及开发利用[J]. 科技通报, 2004, 20(2): 167-171. DOI:10.3969/j.issn.1001-7119.2004.02.018 |
| [10] |
Liu XF, Feng X, Wang QZ, et al. Recent study on the chemistry and pharmacology of Salicornia plant[J]. Chinese Wild Plant Resources, 2012, 31(2): 8-11, 16. (in Chinese) 刘晓峰, 冯煦, 王奇志, 等. 盐角草属植物化学成分和药理研究进展[J]. 中国野生植物资源, 2012, 31(2): 8-11, 16. DOI:10.3969/j.issn.1006-9690.2012.02.003 |
| [11] |
Lee H, Choi J, Kim Y, et al. Effect of dietary intake of Salicornia herbacea L. hot water extract on anti-obesity in diet-induced obese rats[J]. Journal of the Korean Society of Food Science and Nutrition, 2012, 41(7): 950-956. DOI:10.3746/jkfn.2012.41.7.950 |
| [12] |
Yi LP, Wang ZW. Root system characters in growth and distribution among three littoral halophytes[J]. Acta Ecologica Sinica, 2011, 31(5): 1195-1202. (in Chinese) 弋良朋, 王祖伟. 盐胁迫下3种滨海盐生植物的根系生长和分布[J]. 生态学报, 2011, 31(5): 1195-1202. |
| [13] |
Wu F, Yu M, Lu ML, et al. Cloning and functional characterization of three superoxide dismutases genes from halophyte Salicornia europaea and Thellungiella halophila[J]. Acta Botanica Boreali-Occidentalia Sinica, 2012, 32(10): 1957-1964. (in Chinese) 吴凡, 余梅, 鲁茂龙, 等. 盐角草和盐芥3个超氧化物歧化酶基因的克隆和功能分析[J]. 西北植物学报, 2012, 32(10): 1957-1964. DOI:10.3969/j.issn.1000-4025.2012.10.004 |
| [14] |
Gao H. Study on several halophytes for improvement of secondary salinization in soils[D]. Hefei: Master's Thesis of Anhui Agricultural University, 2018 (in Chinese) 高涵.几种盐生植物对改善土壤次生盐渍化的研究[D].合肥: 安徽农业大学硕士学位论文, 2018 http://cdmd.cnki.com.cn/Article/CDMD-10364-1018318642.htm |
| [15] |
Petrini O, Fisher PJ. Fungal endophytes in Salicornia perennis[J]. Transactions of the British Mycological Society, 1986, 87(4): 647-651. DOI:10.1016/S0007-1536(86)80109-7 |
| [16] |
Zhao YH, Yang HY, Xin ZH. Anti-tumor components from fermentation products of fungus Salcoli6 derived from Salicornia herbacea[J]. Food Science, 2011, 32(21): 6-11. (in Chinese) 赵育卉, 杨海燕, 辛志宏. 盐生海芦笋来源真菌Salcoli6发酵产物抗肿瘤活性成分研究[J]. 食品科学, 2011, 32(21): 6-11. |
| [17] |
Zhan DR, Meng XL, Li LQ, et al. Isolation and identification of endophytic fungal strain Salicorn 35 from Salicornia bigelovii and optimization of its fermentation conditions for production of antioxidants[J]. Food Science, 2013, 34(19): 158-165. (in Chinese) 湛东锐, 孟晓露, 李连强, 等. 盐生海芦笋内生真菌Salicorn 35的分离鉴定与抗氧化发酵条件优化[J]. 食品科学, 2013, 34(19): 158-165. DOI:10.7506/spkx1002-6630-201319034 |
| [18] |
Guo J, Wang H, Liu D, et al. Isolation of Cunninghamella bigelovii sp. nov. CGMCC 8094 as a new endophytic oleaginous fungus from Salicornia bigelovii[J]. Mycological Progress, 2015, 14(3): 11. DOI:10.1007/s11557-015-1029-z |
| [19] |
Wang L, Sun X, Wei JG, et al. A new endophytic fungus Neofabraea illicii isolated from Illicium verum[J]. Mycoscience, 2015, 56(3): 332-339. DOI:10.1016/j.myc.2014.10.002 |
| [20] |
White TJ, Bruns T, Lee S, et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[A]//Innis MA, Gelfand DH, Sninsky JJ. PCR Protocols: A Guide to Methods and Applications[M]. San Diego: Academic Press, 1990: 315-322
|
| [21] |
Lu LH, Yin SX, Liu X, et al. Fungal networks in yield-invigorating and -debilitating soils induced by prolonged potato monoculture[J]. Soil Biology and Biochemistry, 2013, 65: 186-194. DOI:10.1016/j.soilbio.2013.05.025 |
| [22] |
Gardes M, Bruns TD. ITS primers with enhanced specificity for basidiomycetes – application to the identification of mycorrhizae and rusts[J]. Molecular Ecology, 1993, 2(2): 113-118. DOI:10.1111/j.1365-294X.1993.tb00005.x |
| [23] |
Xiong W, Zhao QY, Xue C, et al. Comparison of fungal community in black pepper-vanilla and vanilla monoculture systems associated with vanilla Fusarium wilt disease[J]. Frontiers in Microbiology, 2016, 7: 117. |
| [24] |
Edgar RC. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [25] |
Wang S, Wei J, Yang Y, et al. Diversity and community structure of entophytic fungi in roots of Ammopiptanthus mongolicus in different recession degrees[J]. Mycosystema, 2018, 37(4): 411-421. (in Chinese) 王珊, 魏杰, 杨岳, 等. 不同衰退等级蒙古沙冬青根内真菌多样性与群落结构[J]. 菌物学报, 2018, 37(4): 411-421. |
| [26] |
Xi GJ, Li JB, Shi J, et al. Diversity of endophytic fungi in Bletilla striata[J]. Acta Agriculturae Zhejiangensis, 2017, 29(12): 2077-2083. (in Chinese) 席刚俊, 李警保, 史俊, 等. 白芨内生真菌的多样性[J]. 浙江农业学报, 2017, 29(12): 2077-2083. DOI:10.3969/j.issn.1004-1524.2017.12.16 |
| [27] |
Chen YH, Xing XK, Guo SX. The endophytic fungal community composition of Gymnadenia conopsea in Beijing[J]. Mycosystema, 2018, 37(1): 35-42. (in Chinese) 陈艳红, 邢晓科, 郭顺星. 北京地区手参内生真菌的区系组成分析[J]. 菌物学报, 2018, 37(1): 35-42. |
 2020, Vol. 47
2020, Vol. 47




