扩展功能
文章信息
- 李书廷, 洪坤强, 汪保卫, 陈聪, 陈涛, 王智文
- LI Shu-Ting, HONG Kun-Qiang, WANG Bao-Wei, CHEN Cong, CHEN Tao, WANG Zhi-Wen
- 大肠杆菌乙酸耐受性菌株的构建及其耐受机制研究进展
- Advances in construction of acetic acid tolerance Escherichia coli
- 微生物学通报, 2020, 47(12): 4250-4259
- Microbiology China, 2020, 47(12): 4250-4259
- DOI: 10.13344/j.microbiol.china.200048
-
文章历史
- 收稿日期: 2020-01-17
- 接受日期: 2020-04-02
- 网络首发日期: 2020-04-16
2. 天津大学系统生物工程教育部重点实验室 天津 300350
2. Key Laboratory of Systems Bioengineering, Ministry of Education, Tianjin University, Tianjin 300350, China
乙酸是一种二元弱酸,是微生物发酵生产多种化学品时常见的副产物,也存在于木质纤维素水解液等非粮发酵原料中。近年来,廉价的乙酸作为原料逐渐被开发应用于高附加值产品的生产,如聚羟基脂肪酸[1]、异丙醇[2]、乙醇酸[3]等。然而,在以乙酸为原料时,高浓度的乙酸或乙酸盐会抑制细胞生长,从而降低生物量,影响目标产品的产量和产率。为解决乙酸毒性问题,研究者采用多种物理化学方法脱毒,包括活性炭吸附[4]、电渗析[5]和添加碱性物质(CaCO3、NaOH和NH4OH)中和等[6-7],这些方法虽可降低游离乙酸产生的负面影响,但却不能转化利用乙酸而造成碳源浪费。因此,研究乙酸耐受性机制,改进菌株的乙酸耐受性,构建具有高乙酸耐受性工程菌株,对于含乙酸原料的利用或以乙酸为碳源的微生物发酵生产具有重要意义。
大肠杆菌是一种重要的工业微生物菌株,具有碳源利用谱广、生长速度快、遗传背景清晰、基因操作便捷等优势,被广泛用于生产大宗化学品及高附加值产品。目前,提高大肠杆菌乙酸耐受性的方法有多种,包括化学/紫外诱变[8]、代谢工程[9-11]、实验室适应性进化[12-13]以及基于文库设计的多种基因组尺度方法,如全局转录机器工程(global transcription machinery engineering,gTME)[14]、结合文库富集定量分析法(scalar analysis of library enrichments,SCALES)[15]、可追踪的多元重组(trackable multiplex recombineering,TRMR)[16]、基于CRISPR可追踪基因组工程[CRISPR(clustered regularly interspaced short palindromic repeats) enabled trackable genome engineering,CREATE][17]等。随着基因组学、转录组学、蛋白组学、代谢组学等系统生物学研究方法的快速发展,研究工作也从最初的乙酸耐受性菌株的选育,拓展到乙酸耐受机制的解析。本文围绕大肠杆菌乙酸耐受性菌株的构建、大肠杆菌乙酸耐受机制及其应用展开综述和展望,以期对乙酸耐受性菌株构建和乙酸耐受机制的深入研究提供启发。
1 乙酸耐受性大肠杆菌的构建构建乙酸耐受性大肠杆菌工程菌的方法随着科研水平的发展而不断被开发。传统的代谢工程方法可以实现目的基因的准确编辑,但是近些年新兴发展的高效技术可以更加高效地实现乙酸耐受性大肠杆菌的筛选与构建。目前,构建乙酸耐受性大肠杆菌的方法主要包括:代谢工程、实验室适应性进化(adaptive laboratory evolution,ALE)、全局转录机器工程和基于CRISPR可追踪基因组工程等(表 1)。
| 菌株 Strain |
方法策略 Method |
胁迫条件 Stress |
表型 Phenotype |
参考文献 References |
| E.coli JW3973 | metAI229T, metAN267 | 1.8 g/L aceticacid, M9 | Greater cell density | [9] |
| E.coli JW3973 | metE(V39A, R46C, T106I, C645A, K713E) | 1.7 g/L acetate, M9 | 18.5% increase of specific growth rate | [10] |
| E.coli W, E. coli K-12 | Δ(evgA-ptsP-yciW-ackA-typA-yobF-rfe) | 15 g/L acetate, M9 | Signifcantly higher growth rates | [18] |
| E.coli JM109 | pMV24-uvrA | 5.2 g/L acetic acid shock | 10.2–17.5fold increase of the survival rates | [11] |
| E.coli MG1655 | IdhA::pssA | 1.8 g/L acetate, MOPS | 21% increase of specific growth rate | [19] |
| E.coli CL3 | GM(ΔxylFGH, ΔldhA, PpflB::pdcZm-adhBZm), ALE | 10-40 g/L acetate, AM1 | Obtain an acetate tolerant strain MS04 | [12] |
| E.coli DH5α | GREACE (PQ-lib, ALE) | 1.0 g/L acetate, LB | 8-foldincrease of OD600 in mutant AR5 cells | [20] |
| E.coli ATCC8739 | ALE | 7.1 g/L acetate defined medium | 25%increase of growth ratein mutant MEC136 | [13] |
| E. coli DH5α Δcrp | gTME, crp | 15 g/L acetate, M9 | 5-foldincrease of growth rate in the variant A2 | [21] |
| E.coli DH5α Δcrp | gTME, crp | Acetic acid (finalpH 5.35) | Obtainan acetate tolerant strain ACM1 | [22] |
| E.coli K-12 | SCALES | 1.75 g/L acetate, MOPS | Enrichedhigh fitness genes for acetate tolerance | [23] |
| E.coli K-12 | TRMR | 16 g/L acetate, MOPS | 50%increase of growth rate in the variant | [24] |
| E.coli MG1655 | CREATE | 10 g/L acetate, M9 | Enrichedhigh fitness genes for acetate (fis, fadR, rho, and fnr) | [17] |
| BGgxk4P4 | iCREATE | 30 g/L acetate, HM | Stillgrew in reconstruction strain | [25] |
| Note: GM: Gene manipulation; GREACE: Genome replication engineering assisted continuous evolution; gTME: Global transcription machinery engineering; SCALES: Scalar analysis of library enrichments; TRMR: Trackable multiplex recombineering; CREATE: CRISPR enabled trackable genome engineering; iCREATE: Iterative CRISPR enabled trackable genome engineering. | ||||
通过代谢工程构建大肠杆菌乙酸耐受性菌株是基于乙酸耐受性机制的研究,定向修饰与大肠杆菌乙酸耐受性相关的基因,达到乙酸耐受性提高的目的。Han等根据乙酸会抑制甲硫氨酸的合成且可通过补充甲硫氨酸提高菌株乙酸耐受性的原理[26],分别对E. coli JW3973的甲硫氨酸合成酶MetA[9]和MetE[10]进行定点突变,获得乙酸耐受菌株。Xiao等[27]从乙酸代谢角度出发,在E. coli BL21中通过lac启动子诱导过表达乙酰辅酶A合成酶(acetyl-CoA synthetase,ACS),获得的突变菌株在8.3 g/L乙酸钠胁迫条件下表现出更好的乙酸耐受性。此外,Zheng等[11]将来源于A. pasteurianus AC2005的uvrA (编码核酸切除酶复合物ABC的亚基A)克隆到E. coli JM109中,获得的工程菌在5.2 g/L乙酸刺激处理20 min和40 min条件下,存活率分别提高17.5倍和10.2倍。Jensen等[18]通过分别同时敲除E. coli W和E. coli K-12的evgA-ptsP-yciW-ackA-yobF,突变株均在15 g/L乙酸钠胁迫条件下表现出更好的耐受性。然而,由于耐受性通常是由多基因协同控制的复杂表型,基于耐受性相关基因的过表达或敲除所实现的表型搜索空间有限。
1.2 实验室适应性进化构建大肠杆菌乙酸耐受性菌株实验室适应性进化是在人为设置的选择压力下模拟自然进化中的变异和选择过程,并通过富集筛选获得优良性状目的菌株的技术。Fernández-Sandoval等[12]在以木糖为筛选条件下,对乙醇生产菌株E. coli CL3进行代谢进化,提升菌株的乙酸钠耐受和利用能力,获得的菌株E. coli MS04在10 g/L乙酸钠条件下,与不添加乙酸钠相比,比生长速率(0.28±0.013 h-1)提高了75%,且乙醇产率提高近6倍;此外,当比生长速率同时减少50% (IC50)时,菌株E. coli MS04的乙酸钠补加浓度为40 g/L,而对照菌株仅为10 g/L。Rajaraman等[13]将大肠杆菌ATCC8739菌株在以2.9 g/L乙酸盐为唯一碳源的培养条件下进行适应性进化,获得了3株乙酸钠耐受突变菌株,且在以7.1 g/L乙酸盐为碳源的培养条件下,3株突变株中最大的比生长速率高达0.50±0.01 h-1,比对照提高21.9%。Luan等[20]开发了一种新型进化工具GREACE (genome replication engineering assisted continuous evolution)即通过引入一组缺失校正功能的DNA聚合酶复合物实现细胞连续诱变,可达到应激条件下加速进化的一种新型进化工具,该方法中诱变和筛选同时进行,可极大地减少迭代操作和冗长的人为干预。Luan等[20]应用GREACE进化工具即通过易错PCR构建了dnaQ (编码DNA聚合酶ε亚基)的突变文库PQ-lib并将其导入E. coli DH5α细胞中,依次提高乙酸盐浓度进行转接培养,成功进化出在1 g/L乙酸盐浓度下OD600提高了8倍的AR5突变菌群。虽然实验室适应性进化取得一些成效,但由于基因组的突变空间很大,也很难评估每种突变对特定进化表型的贡献,因而需进一步开发缩小突变范围的适应性进化筛选方法。
1.3 利用全局转录机器工程构建大肠杆菌乙酸耐受性菌株2006年,Alper等[28]报道了全局转录机器工程技术,即通过易错PCR (error-prone polymerase chain reaction)或DNA重排(DNA shuffling)的方法突变菌株的全局转录调控因子,从而改变转录因子与其众多基因启动子序列的结合状态,以达到基因组尺度的转录扰动,进一步结合一定的筛选压力,定向获得目标表型菌株。Alper等[14]成功地利用全局转录机器工程策略构建rpoD突变文库,筛选到大肠杆菌乙酸耐受性菌株。Chong等[21]利用全局转录机器工程策略构建crp突变文库,在5-10 g/L乙酸钠条件下筛选大肠杆菌乙酸耐受突变菌株A2,且在15 g/L乙酸钠测试条件下,突变菌株的生长速率(0.083 h-1)比对照菌株提高418.75%。基于同样的策略,Basak等[22]也利用全局转录机器工程方法改造crp转录调控因子,并获得了乙酸耐受性菌株ACM1。Chen等[29]利用全局转录机器工程方法构建了来自耐辐射奇球菌(Deinococcus radiodurans)的全局调控因子IrrE突变文库并转化到大肠杆菌中,在含有0.7 g/L乙酸盐的LB培养基中筛选得到乙酸耐受性菌株A15,该菌株也表现出耐低pH的特性。全局转录机器工程将调控目标拓展到全局转录因子,克服有关乙酸耐受性表型与基因型复杂关系认识的不足,实现从全局水平上强化微生物的目标性能,是一种在基因和细胞水平上改造微生物细胞的新途径。
1.4 利用基于CRISPR可追踪基因组工程构建大肠杆菌乙酸耐受性菌株基于CRISPR可追踪基因组工程[17]是Gill课题组在CRISPR基因组编辑技术的基础上,结合文库富集定量分析法[15, 23]和可追踪的多元重组[16, 24]构建的可追踪基因组工程技术;该技术通过突变文库理性自动设计、突变库芯片高通量合成、基于CRISPR原理基因组尺度快速高通量突变修饰、胁迫条件下的耐受性筛选和适应度分析,实现突变型-表型高通量快速关联分析。Gill课题组[17]利用CREATE技术构建了含有35个基因的52 356个突变的突变文库,将突变文库导入E. coli MG1655,并在10 g/L乙酸盐浓度条件下富集筛选,最终富集到了与乙酸耐受性相关的高适应度基因如fis、metA、fadR、marA、rho等。Liu等[25]通过对该系统的优化得到了iCREATE (iterative CRISPR enabled trackable genome engineering),实现gRNA质粒的消除;基于该系统构建靶向30个基因的(约40 000个)突变文库,获得了既耐受糠醛又耐受乙酸盐的突变菌株。本课题组[30]通过CREATE技术突变了大肠杆菌20个全局调控因子,成功构建了含有34 340个位点饱和突变的突变文库,并在10-30 g/L乙酸钠条件下,筛选获得了高耐受乙酸的大肠杆菌CRP-G142I;在30 g/L乙酸钠条件下,该耐受性菌株的比生长速率提高了313.82%。CREATE可以单次实现数万个位点的突变,即仅通过一轮质粒转化便可完成对多个细胞靶点的编辑,通过测序追踪,缩短了突变构建与分析的周期,大大节约了时间和成本。CREATE系统的可追踪性,为高通量鉴定突变基因与进化表型之间的关系提供了有利的支持。
2 大肠杆菌乙酸耐受性机制研究及相关应用随着生物技术(基因组学、转录组学、蛋白质组学和代谢组学等)的发展,研究者发现耐乙酸的大肠杆菌会进化出多种不同机制来应对乙酸的毒性。主要包括:乙酸同化代谢;氨基酸依赖型代谢;离子转运系统调节;细胞膜成分修饰(图 1)。
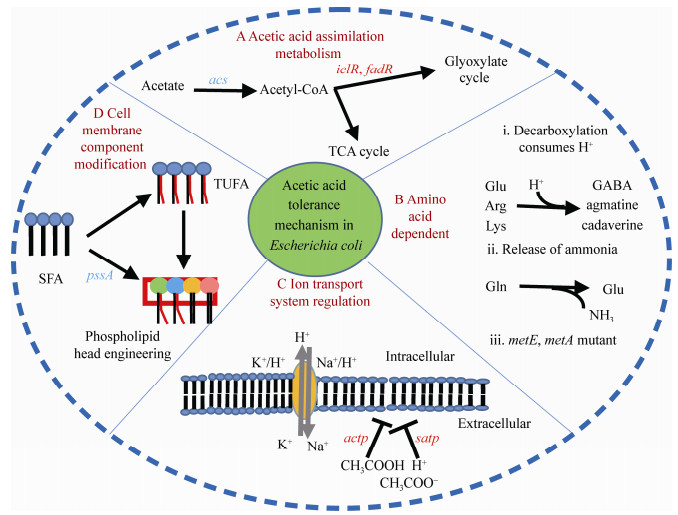
|
| 图 1 大肠杆菌的乙酸耐受性机制 Figure 1 The different mechanisms of acetic acid tolerance in E. coli 注:A:乙酸同化代谢[31-35];B:氨基酸依赖型代谢[9-10, 36];C:离子转运系统调节[37-39];D:细胞膜成分修饰[19, 40].蓝色斜体字代表过表达,红色斜体字表示敲除. acs:编码乙酰辅酶A合成酶;iclR:编码抑制乙醛酸转录调控因子;fadR:编码DNA结合转录双调控因子;GABA:谷氨酸-γ-氨基丁酸;metE:编码同型半胱氨酸转甲基酶;metA:编码高丝氨酸琥珀酰转移酶;actp:编码乙酸/乙醛酸:阳离子转运蛋白;satp:编码乙酸/琥珀酸:H+同向转运蛋白;SFA:饱和脂肪酸;TUFA:反式不饱和脂肪酸;pssA:编码磷脂酰丝氨酸合酶. Note: A: Acetic acid assimilation metabolism[31-35]; B: Amino acid dependent metabolismt[9-10, 36]; C: Iontransport system regulation[37-39]; D: Cell membrane component modification[19, 40]. Overexpression/knockout gene in blueItalic and red italic. acs: Encoding acetyl-CoA synthetase; iclR: Encoding DNA-binding transcriptional repressor; fadR: DNA-binding transcriptional dual regulator; GABA: γ-aminobutyric acid; metE: Encoding homocysteine transmethylase; metA: Encoding homoserine O-succinyltransferase; actp: Encoding acetate/glycolate: cation symporter; satp: Encoding acetate/succinate: H+ symporter; SFA: Saturated fatty acids; TUFA: Trans unsaturated fatty acids; pssA: Encoding phosphatidylserine synthase. |
|
|
大肠杆菌天然存在乙酸的同化代谢途径。乙酸为碳源通过中心碳代谢途径转化为生物量,但与糖利用途径相比效率低很多,所以对乙酸同化代谢途径的改造是加强乙酸利用和减少乙酸积累的重要方式。在大肠杆菌中,最重要的一条乙酸利用途径是ACS途径即通过乙酰辅酶A合成酶(acs编码)将乙酸转化为乙酰辅酶A,然后进入三羧酸(TCA)循环或者乙醛酸循环等被消耗(图 1A)[31]。acs编码的乙酰辅酶A合成酶是参与乙酸同化吸收的关键酶[27, 32]。Lin等[33]在大肠杆菌中过表达acs获得的工程菌株,在以乙酸盐为唯一碳源的基本盐培养基(M9培养基,乙酸盐浓度5.3 g/L)和丰富培养基(LB培养基,乙酸盐浓度10.3 g/L)培养条件下,乙酸盐的消耗速率、生长速率以及生物量的积累均显著高于对照菌株。此外,通过敲除乙醛酸循环表达的负调控因子iclR[34]和fadR[35]也可提高乙酸的消耗。这些研究均表明激活ACS途径有利于提高乙酸的消耗,但对于可逆途径PTA-ACKA [通过磷酸乙酰转移酶(phosphate acetyltransferase,PTA)和乙酸激酶(acetate kinase,ACKA)将乙酸转化为乙酰辅酶A]的影响却并不清楚。研究发现过表达ackA和pta的大肠杆菌在乙酸盐利用过程中不仅没有提高乙酸利用,反而生长速度明显减慢[27, 34]。综上,通过乙酸的利用来缓解乙酸毒性是有效的策略,但仍需要进一步研究更高效的乙酸利用途径。
2.2 氨基酸依赖型代谢氨基酸的正常代谢与合成是微生物维持正常活动的必要条件。大肠杆菌可通过氨基酸合成和代谢的调节来应对外界环境的胁迫与压力,从而提高对不利条件乙酸的耐受性。目前研究者对大肠杆菌氨基酸依赖型的耐酸性机制分析部分适用于乙酸耐受性研究,主要包括4个依赖于氨基酸脱羧反应的氨基酸(谷氨酸、精氨酸、赖氨酸、鸟氨酸)依赖型耐酸系统和1个依赖于氨基酸脱氨基反应的谷氨酰胺依赖型的耐酸系统(图 1B)。这些耐酸性系统的调控和作用机制在陈卓逐等[36]和Guan等[41]的综述中有详细阐述,主要是包括脱羧或脱氨和转运两个过程,氨基酸脱羧或脱氨反应将乙酸进入细胞发生解离产生的H+消耗,转运是将产物输出细胞交换新的氨基酸进入细胞,如此循环,提高细胞内pH,缓解乙酸引起的细胞质酸化,防止细胞致死[42-45]。此外,研究发现乙酸会抑制大肠杆菌甲硫氨酸的合成,积累有毒的中间体同型半胱氨酸,影响大肠杆菌的生长[46]。Han等[26]和Sandoval等[23]也证明补加甲硫氨酸可提高大肠杆菌的乙酸耐受性。Mordukhova等对甲硫氨酸合成相关酶MetA[9]和MetE[10]进行改造,促进同型半胱氨酸转化为甲硫氨酸,缓解乙酸对甲硫氨酸合成的抑制,提高了大肠杆菌的乙酸耐受性。
2.3 离子转运系统调节通过离子转运系统来缓解乙酸的毒性主要涉及两个方面:一方面是阻止乙酸/乙酸盐进入胞内,另一方面是通过离子通道或者流体泵将细胞内的H+或者阴离子(CH3COOH-)排出细胞。乙酸能以乙酸小分子的形式自由扩散进入细胞,也能以阴离子(CH3COOH-)的形式借助转运蛋白[乙酸/乙醇酸转运蛋白(acetate/glycolate: cation symporter,ActP)、琥珀酸/乙酸盐转运蛋白(acetate/succinate: H+ symporter,SATP)等]进入细胞(图 1C),所以阻止乙酸进入细胞的一个潜在有效策略就是通过修饰这些转运蛋白来影响乙酸盐的摄取。Gimenez等[37]发现敲除actP的突变菌株只能降低大肠杆菌对乙酸的摄取能力,因此推测还存在另外的转运体。Sá-Pessoa等[38]证明了SATP与ActP一起影响乙酸作为碳源的利用,同时敲除这2个转运蛋白可阻止大肠杆菌对乙酸的转运和摄取。通过控制质子的输入输出细胞来维持胞内pH的稳态,也是大肠杆菌耐受乙酸的重要组成部分。阳离子转运体K+/H+和Na+/H+转运蛋白,可将质子排出细胞并吸收阳离子,进而减少胞内质子的积累和维持细胞膜电势的稳定,提高大肠杆菌的乙酸耐受性[39]。有研究报道,过表达ABC (ATP-binding cassett)转运家族的外膜蛋白也有助于提高大肠杆菌的乙酸耐受性,这可能与减少乙酸进入细胞相关,从而降低乙酸的毒害[47-48]。
2.4 细胞膜成分修饰细胞膜是大肠杆菌抵御乙酸胁迫的第一道屏障,大肠杆菌会通过调整细胞膜中脂肪酸组成和含量的变化改变细胞膜的流动性、稳定性等来应答乙酸环境胁迫,增加对乙酸的抵抗力(图 1D)。在E. coli K-12 CSH1中,乙酸浓度增加至2.5×10-2 g/L导致脂肪酸从C16:1转变为C18:1,表明脂肪酸链延长有助于提高大肠杆菌的乙酸耐受性[49]。Choi等[50]通过在E. coli BL21(DE3)中过表达来自嗜盐单胞菌(Halomonas socia)的环丙烷脂肪酸酰基磷脂合成酶(cyclopropane fatty acid,CFA),证明提高大肠杆菌细胞膜内环丙烷脂肪酸的含量有助于提高大肠杆菌的乙酸耐受性。据报道,顺式和反式不饱和脂肪酸的异构化对于提高大肠杆菌的乙酸耐受性具有重要的作用[51]。Tan等[40]在大肠杆菌中过表达来自铜绿假单胞菌(Pseudomonas aeruginosa)的顺反异构合成酶(cis-trans isomerase,Cti),增加了大肠杆菌细胞膜中反式不饱和脂肪酸(trans unsaturated fatty acids,TUFA)的含量,使得细胞膜的流动性降低、细胞膜的刚性提高,从而获得在2.5 g/L乙酸盐胁迫条件下比生长速率提高5.6%的大肠杆菌突变菌株。Tan等[19]进一步通过磷脂酰丝氨酸合酶(phosphatidylserine synthase,PSSA)过表达增加了磷脂头部磷酸乙醇胺(phosphoethanolamine,PE)的含量,获得大肠杆菌乙酸耐受性菌株;此外,该菌株也同时具有辛酸、低pH和糠醛等多重耐受性。磷脂工程设计的成功应用表明通过改变细胞膜中磷脂极性头部基团成分的含量可增加细胞膜的稳定性,从而降低乙酸对大肠杆菌的伤害。
2.5 大肠杆菌乙酸耐受性菌株的应用大肠杆菌乙酸耐受性菌株可以抵抗乙酸胁迫,促进大肠杆菌在以乙酸为唯一碳源或者是富含乙酸的纤维素水解液等非粮发酵原料碳源的利用,提高目标产品的生产。Noh等[34]通过将不同遗传背景的大肠杆菌菌株(W、BL21、W3110和MG1655)置于含10 g/L乙酸盐的培养基中,以比生长速率和乙酸盐的消耗速率为指标,筛选到乙酸耐受性最强的W菌株,然后异源过表达来自土曲霉(A. terreus)的顺式脱羧酶基因(cad)和乙酸同化途径基因(acs),敲除抑制乙醛酸途径基因(iclR),获得可以快速摄取乙酸和高乙酸耐受性的工程菌株WCIAG4,该工程菌株在以乙酸盐为唯一碳源的培养基中,发酵88 h后生产了3.57 g/L的衣康酸。Xiao等[27]在大肠杆菌BL21中通过过表达acs和tesA以及敲除fadE构建的乙酸耐受性菌株,在以乙酸为唯一碳源的培养基中生产了1 g/L的脂肪酸,进一步将该菌株在富含乙酸的木质纤维素酸性水解液和厌氧消化的污泥中发酵并分别获得0.43 g/L和0.17 g/L的脂肪酸。最近,Liu等[25]将通过iCREATE方法筛选到的乙酸和糠醛高耐受性的组合突变位点(SoxR S26G,SoxR E32V)引入产3-羟基丙酸(3-hydroxypropionic acid,3-HP)的菌株中获得突变菌株,该菌株分别在含有糠醛、乙酸和木糖的培养基中发酵,3-HP的最终产量是对照菌株的7-8倍。Jo等[52]通过代谢工程改造乙酸耐受性菌株的乙醛酸碳流分布,获得酪氨酸产量提高1.6-1.8倍的突变菌株。总之,大肠杆菌乙酸耐受菌株的开发不仅提高了菌株对乙酸胁迫的适应性,也为大肠杆菌以乙酸为唯一碳源高产高附加值产品成为可能。除了大肠杆菌外,在恶臭假单胞菌[53]、酵母[54]等菌株的发酵生产中,也成功获得并应用乙酸耐受性菌株,加速对乙酸的利用,进而获得高附加值产品。
3 结语与展望目前关于构建大肠杆菌乙酸耐受性菌株及相关耐受机理解析的研究很多,在实际应用研究上也取得了突破。在乙酸耐受性菌株构建方面,代谢工程大肠杆菌乙酸耐受性菌株具有靶标明确的优点,但是由于乙酸的耐受性是多基因控制的复杂表型,因而存在自身的局限性。全局转录机器工程将靶向的调控目标拓展到全局转录因子,达到对多基因控制的性状进行有效的改造,避免了单个基因操作的局限性,使其成为当前研究的热点之一。基于CRISPR可追踪基因组工程的提出大大缩短了突变构建与分析的周期,但是可追踪基因组工程操作手段要求很高、普及性差。因此,迫切需要开发一种可追踪或可视化的基因组工程操作技术。在自然界中有一些菌株能够天然耐受乙酸/乙酸盐,甚至高效利用乙酸。例如,在微生物发酵生产食醋过程中用到的葡糖醋杆菌属具有较高的耐酸性,可以达到100 g/L以上[55]。研究者发现在醋酸杆菌的Gluconacetobacter中存在群体感应调控系统(quorum sensing,QS),菌群通过QS可以增强微生物体系对乙酸的耐受[56]。由于大肠杆菌的基因操作方法比较成熟,被广泛应用于生产化学品。因此,构建大肠杆菌QS系统应用于提高菌群对于乙酸耐受以及乙酸的转化率也是一种可借鉴的方法,同时也启发我们可以进一步挖掘自然界中天然的大肠杆菌乙酸耐受菌株。
大肠杆菌通过乙酸同化代谢、氨基酸依赖型代谢、离子转运系统的调节以及细胞膜成分修饰等响应机制来应答乙酸胁迫条件的迫害,增加了对乙酸的耐受性。随着对大肠杆菌耐乙酸作用机制的深入了解,也引出了新的问题。微生物细胞代谢与耐受通常是多方面共同作用的结果,而目前耐受性机制的解析通常是从单一角度解析,对于不同模块之间的相互作用以及整个细胞的耐受性机制网络的调控机制尚不清楚,所以需要进一步从系统水平、全局调控等角度进行进一步研究。大肠杆菌乙酸耐受性菌株可以高效利用富含乙酸的发酵原料甚至以乙酸为唯一碳源来生产高附加值产品,而乙酸也是微生物利用CO2、甲烷和甲醇等一碳化合物生长和发酵生产化学品过程的中间体,因此,构建大肠杆菌乙酸耐受性菌株对提高大肠杆菌对一碳单位化合物的利用与实现可持续生产具有重要意义。
| [1] |
Chen J, Li W, Zhang ZZ, et al. Metabolic engineering of Escherichia coli for the synthesis of polyhydroxyalkanoates using acetate as a main carbon source[J]. Microbial Cell Factories, 2018, 17(1): 102. DOI:10.1186/s12934-018-0949-0 |
| [2] |
Yang H, Zhang C, Lai NY, et al. Efficient isopropanol biosynthesis by engineered Escherichia coli using biologically produced acetate from syngas fermentation[J]. Bioresource Technology, 2020, 296: 122337. DOI:10.1016/j.biortech.2019.122337 |
| [3] |
Li W, Chen J, Liu CX, et al. Microbial production of glycolate from acetate by metabolically engineered Escherichia coli[J]. Journal of Biotechnology, 2019, 291: 41-45. DOI:10.1016/j.jbiotec.2018.12.012 |
| [4] |
Carvalho GB, Mussatto SI, Cândido EJ, et al. Comparison of different procedures for the detoxification of eucalyptus hemicellulosic hydrolysate for use in fermentative processes[J]. Journal of Chemical Technology and Biotechnology, 2016, 81(2): 152-157. |
| [5] |
Wong M, Wright M, Woodley JM, et al. Enhanced recombinant protein synthesis in batch and fed-batch Escherichia coli fermentation based on removal of inhibitory acetate by electrodialysis[J]. Journal of Chemical Technology and Biotechnology, 2009, 84(9): 1284-1291. DOI:10.1002/jctb.2174 |
| [6] |
Abdel-Rahman MA, Tashiro Y, Sonomoto K. Recent advances in lactic acid production by microbial fermentation processes[J]. Biotechnology Advances, 2013, 31(6): 877-902. DOI:10.1016/j.biotechadv.2013.04.002 |
| [7] |
Wang HW, Wang FQ, Wang W, et al. Improving the expression of recombinant proteins in E. coli BL21(DE3) under acetate stress: an alkaline pH shift approach[J]. PLoS One, 2014, 9(11): e112777. DOI:10.1371/journal.pone.0112777 |
| [8] |
Zhu CQ, Ye Q. Selection of acetate-tolerant mutants from Escherichia coli DH5α and the metabolic properties of mutant DA19[J]. Acta Microbiologica Sinica, 2003, 43(4): 460-465. (in Chinese) 朱才庆, 叶勤. 大肠杆菌DH5α耐乙酸突变株的选育及其代谢特性研究[J]. 微生物学报, 2003, 43(4): 460-465. DOI:10.3321/j.issn:0001-6209.2003.04.010 |
| [9] |
Mordukhova EA, Lee HS, Pan JG. Improved thermostability and acetic acid tolerance of Escherichia coli via directed evolution of homoserine o-succinyltransferase[J]. Applied and Environmental Microbiology, 2008, 74(24): 7660-7668. DOI:10.1128/AEM.00654-08 |
| [10] |
Mordukhova EA, Pan JG. Evolved cobalamin-independent methionine synthase (MetE) improves the acetate and thermal tolerance of Escherichia coli[J]. Applied and Environmental Microbiology, 2013, 79(24): 7905-7915. DOI:10.1128/AEM.01952-13 |
| [11] |
Zheng Y, Chen XJ, Wang J, et al. Expression of gene uvrA from Acetobacter pasteurianus and its tolerance to acetic acid in Escherichia coli[A]//Zhang TC, Nakajima M. Advances in Applied Biotechnology[M]. Berlin, Heidelberg: Springer, 2015: 163-169
|
| [12] |
Fernández-Sandoval MT, Huerta-Beristain G, Trujillo-Martinez B, et al. Laboratory metabolic evolution improves acetate tolerance and growth on acetate of ethanologenic Escherichia coli under non-aerated conditions in glucose-mineral medium[J]. Applied Microbiology and Biotechnology, 2012, 96(5): 1291-1300. DOI:10.1007/s00253-012-4177-y |
| [13] |
Rajaraman E, Agarwal A, Crigler J, et al. Transcriptional analysis and adaptive evolution of Escherichia coli strains growing on acetate[J]. Applied Microbiology and Biotechnology, 2016, 100(17): 7777-7785. DOI:10.1007/s00253-016-7724-0 |
| [14] |
Alper H, Stephanopoulos G. Global transcription machinery engineering: a new approach for improving cellular phenotype[J]. Metabolic Engineering, 2007, 9(3): 258-267. DOI:10.1016/j.ymben.2006.12.002 |
| [15] |
Lynch MD, Warnecke T, Gill RT. SCALEs: multiscale analysis of library enrichment[J]. Nature Methods, 2007, 4(1): 87-93. DOI:10.1038/nmeth946 |
| [16] |
Mansell TJ, Warner JR, Gill RT. Trackable multiplex recombineering for gene-trait mapping in E. coli[A]//Alper HS. Systems Metabolic Engineering: Methods and Protocols[M]. Totowa, NJ: Humana Press, 2013: 223-246
|
| [17] |
Garst AD, Bassalo MC, Pines G, et al. Genome-wide mapping of mutations at single-nucleotide resolution for protein, metabolic and genome engineering[J]. Nature Biotechnology, 2017, 35(1): 48-55. DOI:10.1038/nbt.3718 |
| [18] |
Jensen SI, Lennen RM, Herrgard MJ, et al. Seven gene deletions in seven days: fast generation of Escherichia coli strains tolerant to acetate and osmotic stress[J]. Scientific Reports, 2015, 5(1): 17874. |
| [19] |
Tan ZG, Khakbaz P, Chen YX, et al. Engineering Escherichia coli membrane phospholipid head distribution improves tolerance and production of biorenewables[J]. Metabolic Engineering, 2017, 44: 1-12. DOI:10.1016/j.ymben.2017.08.006 |
| [20] |
Luan GD, Cai Z, Li Y, et al. Genome replication engineering assisted continuous evolution (GREACE) to improve microbial tolerance for biofuels production[J]. Biotechnology for Biofuels, 2013, 6(1): 137. DOI:10.1186/1754-6834-6-137 |
| [21] |
Chong HQ, Yeow J, Wang I, et al. Improving acetate tolerance of Escherichia coli by rewiring its global regulator cAMP receptor protein (CRP)[J]. PLoS One, 2013, 8(10): e77422. DOI:10.1371/journal.pone.0077422 |
| [22] |
Basak S, Geng HF, Jiang RR. Rewiring global regulator cAMP receptor protein (CRP) to improve E. coli tolerance towards low pH[J]. Journal of Biotechnology, 2014, 173: 68-75. DOI:10.1016/j.jbiotec.2014.01.015 |
| [23] |
Sandoval NR, Mills TY, Zhang M, et al. Elucidating acetate tolerance in E. coli using a genome-wide approach[J]. Metabolic Engineering, 2011, 13(2): 214-224. DOI:10.1016/j.ymben.2010.12.001 |
| [24] |
Sandoval NR, Kim JYH, Glebes TY, et al. Strategy for directing combinatorial genome engineering in Escherichia coli[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(26): 10540-10545. DOI:10.1073/pnas.1206299109 |
| [25] |
Liu RM, Liang LY, Garst AD, et al. Directed combinatorial mutagenesis of Escherichia coli for complex phenotype engineering[J]. Metabolic Engineering, 2018, 47: 10-20. DOI:10.1016/j.ymben.2018.02.007 |
| [26] |
Han K, Hong J, Lim HC. Relieving effects of glycine and methionine from acetic acid inhibition in Escherichia coli fermentation[J]. Biotechnology and Bioengineering, 1993, 41(3): 316-324. DOI:10.1002/bit.260410305 |
| [27] |
Xiao Y, Ruan ZH, Liu ZG, et al. Engineering Escherichia coli to convert acetic acid to free fatty acids[J]. Biochemical Engineering Journal, 2013, 76: 60-69. DOI:10.1016/j.bej.2013.04.013 |
| [28] |
Alper H, Moxley J, Nevoigt E, et al. Engineering yeast transcription machinery for improved ethanol tolerance and production[J]. Science, 2006, 314(5805): 1565-1568. DOI:10.1126/science.1131969 |
| [29] |
Chen TJ, Wang JQ, Yang R, et al. Laboratory-evolved mutants of an exogenous global regulator, IrrE from Deinococcus radiodurans, enhance stress tolerances of Escherichia coli[J]. PLoS One, 2011, 6(1): e16228. |
| [30] |
Wang ZW, Li ST, Chen C, et al. Escherichia coli resistant to sodium acetate, sodium chloride and isobutanol and construction method thereof: CN, 110564659A[P]. 2019-12-13 (in Chinese) 王智文, 李书廷, 陈聪, 等.耐乙酸钠、氯化钠和异丁醇的大肠杆菌及其构建方法: 中国, 110564659A[P]. 2019-12-13 |
| [31] |
Wolfe AJ. The acetate switch[J]. Microbiology and Molecular Biology Reviews, 2005, 69(1): 12-50. DOI:10.1128/MMBR.69.1.12-50.2005 |
| [32] |
Novak K, Flöckner L, Erian AM, et al. Characterizing the effect of expression of an acetyl-CoA synthetase insensitive to acetylation on co-utilization of glucose and acetate in batch and continuous cultures of E. coli W[J]. Microbial Cell Factories, 2018, 17(1): 109. DOI:10.1186/s12934-018-0955-2 |
| [33] |
Lin H, Castro NM, Bennett GN, et al. Acetyl-CoA synthetase overexpression in Escherichia coli demonstrates more efficient acetate assimilation and lower acetate accumulation: a potential tool in metabolic engineering[J]. Applied Microbiology and Biotechnology, 2006, 71(6): 870-874. DOI:10.1007/s00253-005-0230-4 |
| [34] |
Noh MH, Lim HG, Woo SH, et al. Production of itaconic acid from acetate by engineering acid-tolerant Escherichia coli W[J]. Biotechnology and Bioengineering, 2018, 115(3): 729-738. DOI:10.1002/bit.26508 |
| [35] |
Peng LF, Shimizu K. Effect of fadR gene knockout on the metabolism of Escherichia coli based on analyses of protein expressions, enzyme activities and intracellular metabolite concentrations[J]. Enzyme and Microbial Technology, 2006, 38(3/4): 512-520. |
| [36] |
Chen ZZ, Kan JQ, Shi H. Recent progress in molecular mechanism of acid resistance in Escherichia coli[J]. Food Science, 2015, 36(21): 273-278. (in Chinese) 陈卓逐, 阚建全, 石慧. 大肠杆菌耐酸分子机制研究进展[J]. 食品科学, 2015, 36(21): 273-278. DOI:10.7506/spkx1002-6630-201521051 |
| [37] |
Gimenez R, Nuñez MF, Badia J, et al. The gene yjcG, cotranscribed with the gene acs, encodes an acetate permease in Escherichia coli[J]. Journal of Bacteriology, 2003, 185(21): 6448-6455. DOI:10.1128/JB.185.21.6448-6455.2003 |
| [38] |
Sá-Pessoa J, Paiva S, Ribas D, et al. SATP (YaaH), a succinate-acetate transporter protein in Escherichia coli[J]. Biochemical Journal, 2013, 454(3): 585-595. DOI:10.1042/BJ20130412 |
| [39] |
Booth IR. Regulation of cytoplasmic pH in bacteria[J]. ACS Sustainable Chemistry & Engineering, 1985, 49(4): 359-378. |
| [40] |
Tan ZG, Yoon JM, Nielsen DR, et al. Membrane engineering via trans unsaturated fatty acids production improves Escherichia coli robustness and production of biorenewables[J]. Metabolic Engineering, 2016, 35: 105-113. DOI:10.1016/j.ymben.2016.02.004 |
| [41] |
Guan NZ, Liu L. Microbial response to acid stress: mechanisms and applications[J]. Applied Microbiology and Biotechnology, 2020, 104(1): 51-65. DOI:10.1007/s00253-019-10226-1 |
| [42] |
Richard H, Foster JW. Escherichia coli glutamate- and arginine-dependent acid resistance systems increase internal pH and reverse transmembrane potential[J]. Journal of Bacteriology, 2004, 186(18): 6032-6041. |
| [43] |
Iyer R, Williams C, Miller C. Arginine-agmatine antiporter in extreme acid resistance in Escherichia coli[J]. Journal of Bacteriology, 2003, 185(22): 6556-6561. DOI:10.1128/JB.185.22.6556-6561.2003 |
| [44] |
Moreau PL. The lysine decarboxylase CadA protects Escherichia coli starved of phosphate against fermentation acids[J]. Journal of Bacteriology, 2007, 189(6): 2249-2261. DOI:10.1128/JB.01306-06 |
| [45] |
Lu PL, Ma D, Chen YL, et al. L-glutamine provides acid resistance for Escherichia coli through enzymatic release of ammonia[J]. Cell Research, 2013, 23(5): 635-644. DOI:10.1038/cr.2013.13 |
| [46] |
Roe AJ, O'Byrne C, McLaggan D, et al. Inhibition of Escherichia coli growth by acetic acid: A problem with methionine biosynthesis and homocysteine toxicity[J]. Microbiology, 2002, 148(7): 2215-2222. DOI:10.1099/00221287-148-7-2215 |
| [47] |
Nakano S, Fukaya M, Horinouchi S. Putative ABC transporter responsible for acetic acid resistance in Acetobacter aceti[J]. Applied and Environmental Microbiology, 2006, 72(1): 497-505. DOI:10.1128/AEM.72.1.497-505.2006 |
| [48] |
Linton KJ, Higgins CF. The Escherichia coli ATP-binding cassette (ABC) proteins[J]. Molecular Microbiology, 1998, 28(1): 5-13. |
| [49] |
Ingram LO. Adaptation of membrane lipids to alcohols[J]. Journal of Bacteriology, 1976, 125(2): 670-678. DOI:10.1128/JB.125.2.670-678.1976 |
| [50] |
Choi TR, Song HS, Han YH, et al. Enhanced tolerance to inhibitors of Escherichia coli by heterologous expression of cyclopropane-fatty acid-acyl-phospholipid synthase (cfa) from Halomonas socia[J]. Bioprocess and Biosystems Engineering, 2020, 43: 909-918. |
| [51] |
Heipieper HJ, Weber FJ, Sikkema J, et al. Mechanisms of resistance of whole cells to toxic organic solvents[J]. Trends in Biotechnology, 1994, 12(10): 409-415. DOI:10.1016/0167-7799(94)90029-9 |
| [52] |
Jo M, Noh MH, Lim HG, et al. Precise tuning of the glyoxylate cycle in Escherichia coli for efficient tyrosine production from acetate[J]. Microbial Cell Factories, 2019, 18(1): 57. DOI:10.1186/s12934-019-1106-0 |
| [53] |
Yang SY, Li SH, Jia XQ. Production of medium chain length polyhydroxyalkanoate from acetate by engineered Pseudomonas putida KT2440[J]. Journal of Industrial Microbiology & Biotechnology, 2019, 46(6): 793-800. |
| [54] |
Xu Q, Bai CX, Liu YQ, et al. Modulation of acetate utilization in Komagataella phaffii by metabolic engineering of tolerance and metabolism[J]. Biotechnology for Biofuels, 2019, 12(1): 61. DOI:10.1186/s13068-019-1404-0 |
| [55] |
Xia K, Zhu JL, Liang XL. Advances in acid resistant mechanism of acetic acid bacteria and related quorum sensing system[J]. Acta Microbiologica Sinica, 2017, 57(3): 321-332. (in Chinese) 夏凯, 朱军莉, 梁新乐. 醋酸菌耐酸机理及其群体感应研究新进展[J]. 微生物学报, 2017, 57(3): 321-332. |
| [56] |
Iida A, Ohnishi Y, Horinouchi S. Control of acetic acid fermentation by quorum sensing via N-acylhomoserine lactones in Gluconacetobacter intermedius[J]. Journal of Bacteriology, 2008, 190(7): 2546-2555. DOI:10.1128/JB.01698-07 |
 2020, Vol. 47
2020, Vol. 47