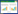扩展功能
文章信息
- 马广强, 李伟, 韩飞, 黄小英, 章常华, 廖永翠, 万红娇, 丁成华
- MA Guang-Qiang, LI Wei, HAN Fei, HUANG Xiao-Ying, ZHANG Chang-Hua, LIAO Yong-Cui, WAN Hong-Jiao, DING Cheng-Hua
- 黄腻舌苔和薄白舌苔微生物种群差异
- Differences of microbial populations between yellow greasy and thin white tongue coating in healthy population
- 微生物学通报, 2020, 47(12): 4229-4239
- Microbiology China, 2020, 47(12): 4229-4239
- DOI: 10.13344/j.microbiol.china.200575
-
文章历史
- 收稿日期: 2020-06-08
- 接受日期: 2020-09-07
- 网络首发日期: 2020-09-23
2. 江西中医药大学药学院 江西 南昌 330004;
3. 江西中医药大学江西省中医院 江西 南昌 330004
2. College of Medicine, Jiangxi University of Traditional Chinese Medicine, Nanchang, Jiangxi 330004, China;
3. Jiangxi Hospital of Traditional Chinese Medicine, Jiangxi University of Traditional Chinese Medicine, Nanchang, Jiangxi 330004, China
舌苔是指舌头上覆盖的一层物质,一般由食物残渣、微生物群、唾液以及舌头角化的上皮细胞组成[1]。根据舌苔的厚度、颜色等苔质情况来鉴别病人的身体状况,是“望”诊的组成部分之一,是中医专家对病人辨证施治的主要依据之一[2]。中医诊断学根据舌苔颜色的苔质情况,将舌苔的表型分为薄白苔、黄腻苔、无根苔、有根苔、润苔、燥苔等几十种舌苔表型[3-4]。
健康人群的舌苔薄白而润,是为薄白苔,黄腻苔是指舌苔发黄、苔质粘腻,是中医痰湿病人常有的一种舌苔,而痰湿现一般认为与肥胖有关[5-6]。目前有些中医专家认为中医的痰湿和西医的糖尿病合并高脂血症大部分相同,可以合并治疗[5-7]。
为了了解薄白舌苔和黄腻舌苔在微生物种群上的差异,本团队以中医硕士研究生和本科生为主要研究对象,采集薄白舌苔和黄腻舌苔样本,提取总DNA后,用原核微生物核糖体16S rRNA基因通用引物PCR扩增16S rRNA基因后送上海美吉生物医药科技有限公司进行全基因测序,然后通过微生物群落分析软件获得黄腻舌苔和薄白舌苔微生物种群的差异情况。
1 材料与方法 1.1 材料口腔拭子基因组DNA提取试剂盒,天根生化科技(北京)有限公司。
采样人群:2017级江西中医药大学中医学院硕士研究生(男24,女26)和2017级生物工程专业本科生(男40,女34);采样标准:黄腻舌苔:取苔色黄而粘腻、颗粒紧密胶粘的舌苔样本;薄白舌苔:取舌色淡红鲜明、表面滋润的舌苔样本(表 1)。
| 样品名称 Sample name |
舌苔样品Tongue coating samples | 年龄分布Age range | 总计Total | |
| 男生Boy students | 女生Girl students | |||
| 薄白舌苔 Thin white tongue coating |
A1, A2, A4, A8 | A3, A5, A7, A6, A9, A10, A11 | 19-25 | 11 |
| 黄腻舌苔 Yellow greasy tongue coating |
B1, B2, B3, B4, B5, B11 | B6, B7, B8, B9, B10, B12 | 19-25 | 12 |
采样方法:采样对象早晨起床后未进食和刷牙情况下,先用灭菌生理盐水漱口后,一次性手套取灭菌刮舌板,用一次性棉拭子刮取舌苔样本0.2 mL左右,用5 mL一次性注射器吸取2 mL无菌生理盐水冲洗,冲洗液保存于5 mL采样离心管中,冰盒保存[1]。-80 ℃冰箱保存样本。
样品DNA的提取方法:取样品于12 000 r/min离心5 min后,去除上清,用1 mL灭菌水重悬菌液,然后转入口腔拭子基因组DNA提取试剂盒中1.5 mL的离心管中。按试剂盒操作步骤提取总DNA,-20 ℃冰箱保存。所有样品统一送上海美吉生物医药科技有限测序公司测序分析。
1.3 16S rRNA基因的PCR扩增方法利用16S rRNA基因高变区V3区设计通用引物338F (5′-ACTCCTACGGGAGGCAGCAG-3′)和806R (5′-GGACTACHVGGGTWTCTAAT-3′)进行PCR扩增,预计PCR扩增片段长度为468 bp。PCR反应体系(20 μL):5×FastPfu Buffer 4 μL,dNTPs (2.5 mmol/L) 2 μL,正向引物(5 μmol/L) 0.8 μL,反向引物(5 μmol/L) 0.8 μL,FastPfu Polymerase (5 U/μL) 0.4 μL,基因组DNA模板10 ng。PCR反应条件:95 ℃ 30 s;55 ℃ 30 s,72 ℃ 30 s,30个循环。用PCR产物制备MiSeq PE文库,进行Illumina MiSeq高通量测序[8],采用生信云在线软件进行生物信息学分析,包括α多样性分析、β多样性分析、菌群结构分析、KEGG功能预测等。
1.4 统计学分析采用SPSS软件进行卡方检验、t检验、非参数检验、相关性分析等,双侧检验P < 0.05表示显著性差异,P < 0.01表示差异极显著。
2 结果与分析 2.1 舌苔样本采集19-25岁本科生或者硕士研究生薄白舌苔样本(表 1),筛选薄白舌苔11个,黄腻舌苔12个(表 1,图 1)。黄腻舌苔苔厚、色黄、有异味;而薄白舌苔苔薄、舌苔白、无异味。
|
| 图 1 舌苔图片 Figure 1 Samples of tongue coating 注:BB:薄白舌苔;HN:黄腻舌苔. Note: BB: Thin white tongue coating; HN: Yellow greasy tongue coating. |
|
|
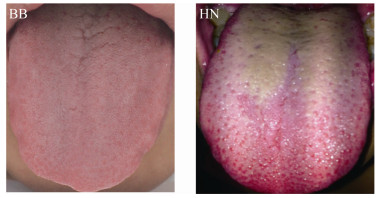
舌苔样本提取总DNA后,用16S rRNA基因的通用引物进行PCR,扩增舌苔原核微生物核糖体高变区468 bp片段,1%琼脂糖凝胶电泳结果见图 2。PCR扩增产物送上海美吉生物医药科技有限公司高通量测序。
|
| 图 2 PCR扩增舌苔样品16S rRNA基因片段 Figure 2 PCR products of 16S rRNA gene from samples 注:1-11:黄腻舌苔(HN);12-23:薄白舌苔(BB); M: DL2000 DNA Marker. Note: 1-11: Yellow greasy tongue coating (HN); 12-23: Thin white tongue coating (BB); M: DL2000 DNA Marker. |
|
|
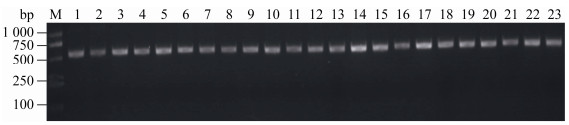
通过生信云Venn图统计分析软件分析舌苔样本中的群落组成,发现舌苔上共有11个门17个纲37个目66个科128个属229种细菌;薄白舌苔和黄腻舌苔在门和纲的细菌组成上没有差异。在目、科、属、种的组成上有一定的差异(表 2,图 3)。薄白舌苔在属水平上特有的菌属有:Blautia、Caulobacteraceae、Sphingomonas、Sphingobium、Curvibacter、Mesorhizobium、Bradyrhizobium、Muribaculaceae、Chitinophagaceae、Cupriavidus和Pseudoramibacter;黄腻舌苔上特有的菌种有Zymomonas、Bacillus、Brevundimonas、Bifidobacterium、Moryella、Parascardovia、Enterococcus、Bacteroides、Peptoanaerobacter、Pantoea、Leuconostoc、Howardella、Acinetobacter和Weissella。在种水平上差异更大,除了191种共有菌群外,薄白舌苔有14种特有菌种,黄腻舌苔有24种特有菌种(表 2)。
| 分类 Taxonomy |
薄白舌苔特有菌群 Specific flora of thin white tongue coating |
共有菌群 Common flora |
黄腻舌苔特有菌群 Specific flora of yellow greasy tongue coating |
| 门Phylum | 0 | 11 | 0 |
| 纲Class | 0 | 17 | 0 |
| 目Order | 2 | 34 | 1 |
| 科Family | 5 | 56 | 5 |
| 属Genus | 11 | 101 | 14 |
| 种Species | 14 | 191 | 24 |
|
| 图 3 舌苔样品聚类分析(A)和Venn分析图片(B) Figure 3 Results of OTU analysis (A) and Venn analysis (B) 注:HN:黄腻舌苔;BB:薄白舌苔. Note: HN: Yellow greasy tongue coating; BB: Thin white tongue coating. |
|
|
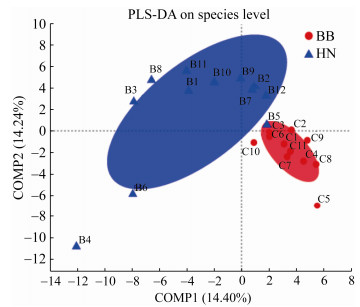
对所采集的舌苔样本进行组间相似性分析,检验组间差异的显著性。通过PLS-DA分析,即偏最小二乘法判别分析(partial least squares discriminant analysis)来判断所采集舌苔样品分类是否符合区分要求。结果发现:在种水平上,黄腻舌苔和薄白舌苔样品分区情况很好(图 4),黄腻舌苔和薄白舌苔被很好地区分到不同区域范围。
|
| 图 4 PLS-DA组间相似性分析结果 Figure 4 PLS-DA results of similarity analysis among groups 注:HN:黄腻舌苔;BB:薄白舌苔. Note: HN: Yellow greasy tongue coating; BB: Thin white tongue coating. |
|
|
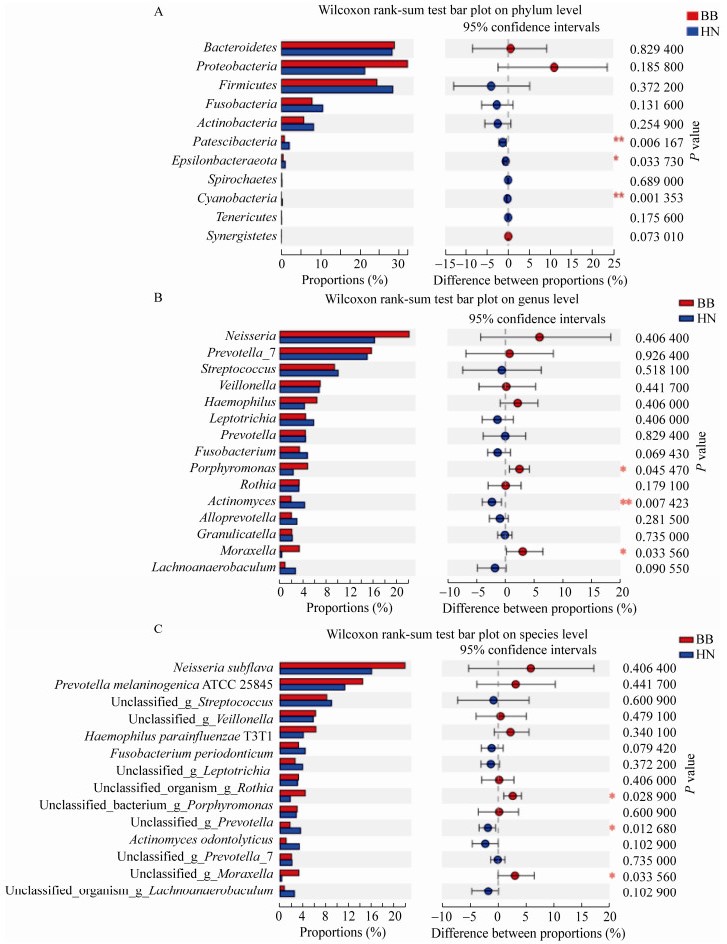
利用生信云平台对舌苔分组样品进行Wilcoxon rank-sum组间丰度差异性检测发现,在门水平上(表 3):黄腻舌苔和薄白舌苔主要组成菌群是变形菌门、拟杆菌门、厚壁菌门、梭杆菌门以及放线菌门,含量都在5%以上;但是变形菌门有较大的差异;变形菌门在黄腻舌苔含量为32%,而在薄白舌苔上只有21.89%,但是变形菌门在两组样品中差异不显著(P > 0.05),没有统计学意义。SPSS组间差异显著性分析发现,Patescibacteria和蓝细菌门(Cyanobacteria)在黄腻舌苔和薄白舌苔上具有极显著差异(P < 0.01),Epsilonbacteraeota具有显著差异(P < 0.05) (图 5A)。菌群丰度在属水平上差异显著性分析发现,大部分属的细菌在黄腻舌苔和薄白舌苔上菌群丰度没有明显差异,但是放线菌属(Actinomyces)的组成具有极显著差异(P < 0.01),黄腻舌苔中放线菌含量明显高于薄白舌苔;而薄白舌苔中的卟啉单胞菌属(Porphyromonas)和莫拉氏菌属(Moraxella)的含量明显高于黄腻舌苔,差异显著(P < 0.05) (图 5B)。在种水平上,薄白舌苔和黄腻舌苔在放线菌、卟啉单胞菌和莫拉氏菌也差异显著(P < 0.05) (图 5C),此结果和属水平上的差异显著性一致。这表明黄腻舌苔和薄白舌苔的菌群分布在放线菌、卟啉单胞菌和莫拉氏菌这3个种群上有显著差异,可能是导致舌苔表型不同的主要原因之一。
| 菌群Flora | 薄白舌苔组 Thinwhite tongue coating (%) |
黄腻舌苔组 Yellowgreasy tongue coating (%) |
组成差异分析P值 P value of composition difference analysis |
| 变形菌门Proteobacteria | 21.89 | 32 | 0.185 8 |
| 拟杆菌门Bacteroidetes | 27.91 | 28.20 | 0.829 4 |
| 梭杆菌门Fusobacteria | 10.42 | 8.23 | 0.131 8 |
| 厚壁菌门Firmicutes | 28.15 | 24.80 | 0.372 2 |
| 放线菌门Actinobacteria | 8.29 | 5.49 | 0.254 9 |
| Patescibacteria | 2.03 | 0.006 2** | |
| 蓝细菌门Cyanobacteria | 0.001 4** | ||
| Epsilonbacteraeota | 0.003 4* | ||
| Note: *: P < 0.05; **: P < 0.01. | |||
|
| 图 5 组间菌群群落组成差异显著性分析 Figure 5 Significance analysis of the difference of microbial community composition between groups 注:HN:黄腻舌苔;BB:薄白舌苔. A:门水平上差异显著性分析;B:属水平上差异显著性分析;C:种水平上差异显著性分析. Note: HN: Yellow greasy tongue coating; BB: Thin white tongue coating. A: Significance analysis of the difference on phylum level; B: Significance analysis of the difference on genus level; C: Significance analysis of the difference on species level. |
|
|
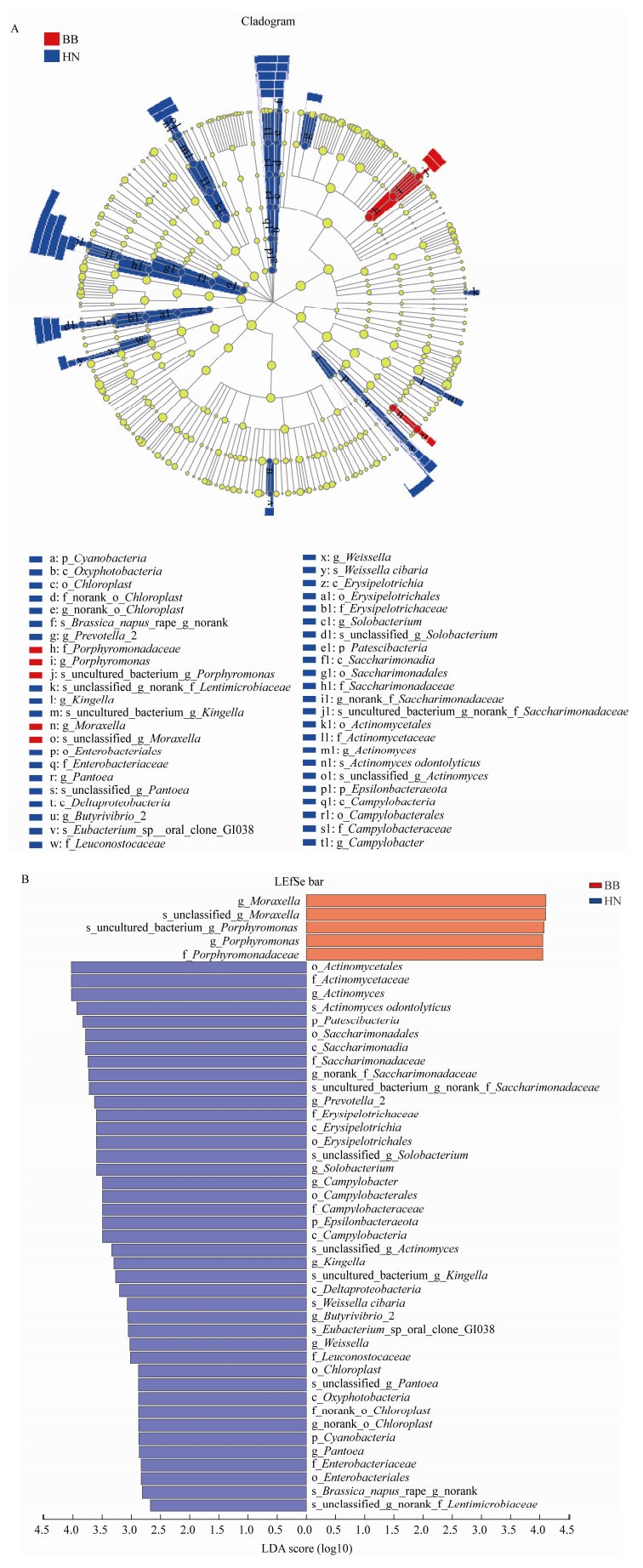
使用生信云平台LEfSe多级物种组间差异显著性分析黄腻舌苔和薄白舌苔样本菌群差异,主要从门、纲、目、科、属、种水平对各级群落丰度水平进行差异检验。由图 6可以判断出,黄腻舌苔主要特有细菌是放线菌属(Actinomyces);而薄白舌苔主要特有菌是莫拉氏菌属(Moraxella)和卟啉单胞菌属(Porphyromonas)。此结果和Wilcoxon rank-sum的菌群差异分析结果一致。
|
| 图 6 LEfSe多级分析组间菌群差异 Figure 6 Microflora differences among LEfSe multi-level analysis groups 注:HN:黄腻舌苔;BB:薄白舌苔. A:LEfSe多级物种层级树图;B:LDA判别柱形图. Note: HN: Yellow greasy tongue coating; BB: Thin white tongue coating. A: LEfSe hierarchical tree; B: LDA discriminant histogram. |
|
|
舌苔的形成原因复杂,微生物菌群有可能是舌苔形成的主要原因之一。本实验采集薄白舌苔和黄腻舌苔样本后,经16S rRNA基因的高通量测序,利用上海美吉生物云交互分析平台分析发现:微生物菌群组成对舌苔苔色的形成具有重要的影响作用。微生物菌群的组成不同有可能是导致舌苔形成的主要原因。
首先,利用生物云平台PLS-DA分析方法对采集的舌苔样本进行了菌群组间相似性分析,发现黄腻舌苔样本和薄白舌苔样本明显区分为2个区域,说明样本采集过程质量控制严,这也表明黄腻舌苔和薄白舌苔上微生物菌群组成具有明显差异(图 4)。
其次,我们通过生信云平台Wilcoxon rank-sum组间丰度差异性检测比较样本菌群组成,在门水平上分析舌苔样本原核微生物的组成,结果发现:门水平上舌苔优势菌群相对丰度从高到低为:梭杆菌门(Fusobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)是舌苔微生物的主要组成菌群。这和孙娴等2018年的研究结果相差不多[9]。虽然黄腻舌苔和薄白舌苔在这些主要菌群组成上有一定差异,但是差异不显著,与肠道菌群相比,舌苔中的梭杆菌和拟杆菌含量明显少很多。这可能和口腔含氧量明显比肠道多有关。黄腻舌苔上的Patescibacteria和蓝细菌门细菌在舌苔微生物组成上明显比薄白舌苔多,具有极显著差异(P < 0.01) (图 5A);Patescibacteria是一种与其他细菌共生的基因组较小的自养微生物,目前没有相应的培养基培养此细菌[10]。推测其主要通过氧化氮、硫等无机物发酵产能,Patescibacteria在正常人肠道内很难发现,主要在地下水和污泥中大量存在[11-12]。Patescibacteria菌群可能是造成黄腻舌苔异味的主要原因之一,但是这种细菌的生理、生化代谢途径还不是十分清楚,有待进一步研究发现。蓝细菌是一类含有叶绿素、可进行光合作用产能的原核细胞型微生物[13]。蓝细菌是肠道中常见的微生物群,但其含量不多,不是肠道微生物的主要类群,主要通过食物进入人体内。蓝细菌由于具有叶绿素,所以一般都能产生色素,这有可能是黄腻舌苔形成的部分原因。
在属水平上,通过Wilcoxon rank-sum丰度差异性检测和LEfSe多级物种差异分析,本研究发现,黄腻舌苔上的放线菌属明显高于薄白舌苔,而且差异极显著(P < 0.01) (图 5B);在种水平上也得到了相类似的结果(图 5C)。LEfSe多级物种差异也发现放线菌在黄腻舌苔中显著高于薄白舌苔(图 5),这说明放线菌有可能是黄腻舌苔上的一个标志性菌群,有可能是导致黄腻舌苔形成的原因之一。放线菌是一种丝状原核细胞型微生物,革兰氏阳性菌,广泛分布于土壤等环境中;多数是需氧型腐生菌,能产生色素、抗生素等次级代谢产物[9]。放线菌也是一种口腔常见的共生菌[14-15],属于口咽、胃肠道和泌尿生殖道的人类共生菌群[16-17];有些放线菌可以导致侵袭性细菌性疾病,比如:牛放线菌可通过口腔或牙龈伤口侵入组织内部引发疾病,衣放线菌在人体免疫力下降或者抵抗力降低的时候容易诱发慢性脓肿为特征的疾病,这些疾病统称放线菌病[16]。因此,健康人群出现黄腻苔,可在一定程度上提示体内潜在的炎性预警。
此外,在属和种水平上,薄白舌苔上的莫拉氏菌属(Moraxella)和卟啉单胞菌属(Porphyromonas)显著比黄腻舌苔上的多(P < 0.05) (图 5B、C);莫拉氏菌属(Moraxella)是一种革兰氏阴性非发酵菌,属于口腔常见菌群,是一种对人无致病性的口腔、呼吸道寄居菌。一些特殊的莫拉氏菌属细菌可以导致疾病,比如亚特兰大莫拉氏菌(M. atlantae)可以导致败血症[18-19];卟啉单胞菌属细菌是一种小型厌氧革兰氏阴性非运动球菌属,通常为短棒状,能产生光滑、灰色至黑色的菌落,菌落大小因种类而异;其作为正常菌群的一部分存在于人类的口咽部,包括牙龈沟、阴道和肠道;模式种为细沙卟啉单胞菌(P. asaccharolytica),也是口腔中特别是牙齿上常见的一种正常菌群[20-21]。
综上所述,放线菌菌群的增多可能是黄腻舌苔形成的主要原因之一,黄腻舌苔在中医上分析为由邪热与痰涎湿浊交结而形成[3];临床多见于急慢性胃肠炎、胆囊炎等病人的舌象[9]。因此,健康人群出现黄腻苔可能是体内潜在炎性病证的早期信号,可进一步追踪观察其变化,酌情给予干预防治。
4 结论薄白舌苔和黄腻舌苔在微生物群落组成上有明显差异,黄腻舌苔苔厚、色黄、有异味,有可能是Patescibacteria、蓝细菌门和放线菌群含量过高所致。值得注意的是:通过菌群差异显著性分析发现,黄腻舌苔上的放线菌菌群在属和种水平上都显著高于薄白舌苔,这说明放线菌有可能是黄腻舌苔形成的主要菌群之一。有些放线菌可以导致侵袭性细菌性疾病,称作放线菌病[18]。因此,健康人出现黄腻苔可在一定程度上提示体内潜在的炎性预警。
| [1] |
Zhang HL, Tian KX, Zhou DR, et al. Application of PCR-DGGE in the analysis of structure of bacteria colony from tongue coating in gastritis patients[J]. Life Science Research, 2011, 15(2): 125-128. (in Chinese) 张红琳, 田凯旋, 周东蕊, 等. PCR-DGGE法在慢性胃炎病人舌苔菌群结构研究中的应用[J]. 生命科学研究, 2011, 15(2): 125-128. |
| [2] |
Pan CH, Yang DC, Yin QY, et al. Literature research and thinking on tongue coating flora and diagnosis of TCM syndromes[J]. Chinese Journal of Microecology, 2019, 31(12): 1456-1459, 1464. (in Chinese) 潘辰慧, 杨德才, 尹沁怡, 等. 舌苔菌群与中医病证诊断的文献研究与思考[J]. 中国微生态学杂志, 2019, 31(12): 1456-1459, 1464. |
| [3] |
Lei SY. Memory on Tongue Coating[M]. Beijing: Truth Seeking Press, 2010. (in Chinese) 雷抒雁. 舌苔上的记忆[M]. 北京: 求真出版社, 2010. |
| [4] |
Chen ZL. Tongue Coating and Disease[M]. Beijing: People's Health Press, 1991. (in Chinese) 陈泽霖. 舌苔与疾病[M]. 北京: 人民卫生出版社, 1991. |
| [5] |
Zhang F. Treating diabetes with hyperlipidemia from phlegm and dampness[J]. Hebei Journal of Traditional Chinese Medicine, 2013, 35(10): 1488-1489. (in Chinese) 张芳. 从痰湿论治糖尿病合并高脂血症[J]. 河北中医, 2013, 35(10): 1488-1489. DOI:10.3969/j.issn.1002-2619.2013.10.026 |
| [6] |
Tan Y, Wang F, Zhang Y, et al. Research progress on the correlation between hyperlipidemia and phlegm-dampness constitution[J]. Chinese Medicine Modern Distance Education of China, 2018, 16(5): 155-157. (in Chinese) 谭玉, 王飞, 章莹, 等. 高脂血症与痰湿质的相关性研究进展[J]. 中国中医药现代远程教育, 2018, 16(5): 155-157. DOI:10.3969/j.issn.1672-2779.2018.05.069 |
| [7] |
Zhang XQ, Sun DJ. Treating hypertension from phlegm and dampness[J]. Chinese Modern Doctor, 2012, 50(8): 33. (in Chinese) 张小庆, 孙丹杰. 从痰湿论治高血压[J]. 中国现代医生, 2012, 50(8): 33. DOI:10.3969/j.issn.1673-9701.2012.08.013 |
| [8] |
Qi CC, Li W, Ding CH, et al. Research on the flora differences of yellow-greasy fur in phylum and genus level[J]. China Journal of Traditional Chinese Medicine and Pharmacy, 2018, 33(8): 3455-3458. (in Chinese) 齐城成, 李伟, 丁成华, 等. 黄腻苔舌面菌群门与属水平上的差异性研究[J]. 中华中医药杂志, 2018, 33(8): 3455-3458. |
| [9] |
Sun X, Xu J, Hua ZL, et al. Effects of pungent dispersion bitter purgation method on bacterial flora of tongue coating in 24 patients with precancerous lesions of upper gastrointestinal[J]. China Journal of Traditional Chinese Medicine and Pharmacy, 2018, 33(8): 3322-3326. (in Chinese) 孙娴, 徐静, 华召来, 等. 辛开苦降法对24例上消化道癌前病变患者舌苔菌群的影响[J]. 中华中医药杂志, 2018, 33(8): 3322-3326. |
| [10] |
Shen P, Chen XD. Microbiology[M]. Beijing: Higher Education Press, 2009. (in Chinese) 沈萍, 陈向东. 微生物学[M]. 北京: 高等教育出版社, 2009. |
| [11] |
Lemos LN, Medeiros JD, Dini-Andreote F, et al. Genomic signatures and co-occurrence patterns of the ultra-small Saccharimonadia (phylum CPR/Patescibacteria) suggest a symbiotic lifestyle[J]. Molecular Ecology, 2019, 28(18): 4259-4271. DOI:10.1111/mec.15208 |
| [12] |
Sánchez-Osuna M, Barbé J, Erill I. Comparative genomics of the DNA damage-inducible network in the Patescibacteria[J]. Environmental Microbiology, 2017, 19(9): 3465-3474. DOI:10.1111/1462-2920.13826 |
| [13] |
Herrmann M, Wegner CE, Taubert M, et al. Predominance of Cand. Patescibacteria in groundwater is caused by their preferential mobilization from soils and flourishing under oligotrophic conditions[J]. Frontiers in Microbiology, 2019, 10: 1407. DOI:10.3389/fmicb.2019.01407 |
| [14] |
Hyun DW, Shin NR, Kim MS, et al. Actinomyces haliotis sp. nov., a bacterium isolated from the gut of an abalone, Haliotis discus hannai[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(2): 456-461. |
| [15] |
Cimmino T, Metidji S, Labas N, et al. Genome sequence and description of Actinomyces polynesiensis str. MS2 sp. nov. isolated from the human gut[J]. New Microbes and New Infections, 2016, 12: 1-5. DOI:10.1016/j.nmni.2016.02.014 |
| [16] |
Valour F, Sénéchal A, Dupieux C, et al. Actinomycosis: etiology, clinical features, diagnosis, treatment, and management[J]. Infection and Drug Resistance, 2014, 7: 183-197. |
| [17] |
Egal A, Etienney I, Beate H, et al. Diagnosis and management of a cryptoglandular actinomycotic fistula-in-ano: an update on 7 new cases and a review of the literature[J]. Annals of Coloproctology, 2018, 34(3): 152-156. DOI:10.3393/ac.2017.07.23 |
| [18] |
Carbonell-Muñoz C, Zhilina S, Barbosa-Ventura A, et al. Isolation of Moraxella atlantae on the valve of a patient with native infective endocarditis[J]. Revista Española de Quimioterapia, 2018, 31(6): 550-551. |
| [19] |
Barash A, Chou TY. Moraxella atlantae keratitis presenting with an infectious ring ulcer[J]. American Journal of Ophthalmology Case Reports, 2017, 7: 62-65. DOI:10.1016/j.ajoc.2017.06.003 |
| [20] |
Moe KK, Yano T, Misumi K, et al. Detection of antibodies against Fusobacterium necrophorum and Porphyromonas levii-like species in dairy cattle with papillomatous digital dermatitis[J]. Microbiology and Immunology, 2010, 54(6): 338-346. DOI:10.1111/j.1348-0421.2010.00220.x |
| [21] |
Lockhart JS, Buret AG, Ceri H, et al. Mixed species biofilms of Fusobacterium necrophorum and Porphyromonas levii impair the oxidative response of bovine neutrophils in vitro[J]. Anaerobe, 2017, 47: 157-164. DOI:10.1016/j.anaerobe.2017.05.008 |
 2020, Vol. 47
2020, Vol. 47