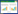扩展功能
文章信息
- 张玲莉, 王建峰, 魏华, 于纪棉, 费红军, 岑叶平
- ZHANG Ling-Li, WANG Jian-Feng, WEI Hua, YU Ji-Mian, FEI Hong-Jun, CEN Ye-Ping
- 铜绿假单胞菌PAO1 ku基因缺失菌株的构建及其对生物被膜耐药性的影响
- Construction of Pseudomonas aeruginosa ku gene deletion mutant and its effects on antibiotic resistance of biofilm
- 微生物学通报, 2020, 47(12): 4219-4228
- Microbiology China, 2020, 47(12): 4219-4228
- DOI: 10.13344/j.microbiol.china.200033
-
文章历史
- 收稿日期: 2020-01-12
- 接受日期: 2020-06-22
- 网络首发日期: 2020-08-31
2. 宁波检验检疫科学技术研究院 浙江 宁波 315012
2. Ningbo Academy of Science and Technology for Inspection and Quarantine, Ningbo, Zhejiang 315012, China
铜绿假单胞菌(Pseudomonas aeruginosa,PA)是一种临床上重要的条件致病菌,常引起严重的医院获得性感染,导致呼吸道感染、菌血症、肺炎、烧伤感染、泌尿系统感染和囊性纤维化继发感染等多种疾病。PA易形成生物被膜,这是细菌附着于惰性或活性实体表面,为了适应生存环境,繁殖、分化并分泌多糖基质包裹菌体群落而形成的细菌聚集体膜状物。成熟的生物被膜形成后,生物被膜深层的细菌被多糖蛋白复合物包围,与外界沟通机会少,处于低营养物质、低氧分压的微环境[1]。这些环境因素导致细菌生长速率减慢,产生综合广泛性的应激反应以及使多药物外排泵基因表达上调,致使生物被膜具有极强的适应性与耐药性[2-3]。
非同源末端连接(non-homologous end joining,NHEJ)途径是DNA双链损伤修复(double-strand break,DSB)的主要途径之一,修复DSB过程中无需同源模板,直接将断裂缺口连接,因此常引起基因突变。Ku蛋白作为NHEJ途径的关键组分之一,在NHEJ修复途径中,可识别并稳定DSB双链末端,保护末端不被核酸酶降解,并招募LigD连接酶进行断裂末端的修复[4-5]。NHEJ途径广泛存在于真核细胞中,但仅在包括PA在内的部分细菌种类中发现[6-10]。研究结果显示PA生物被膜内部细菌具有极高的基因突变率,而PA生物被膜耐药性的提高或获得往往与某些相关基因的突变密切相关,如外排泵MexAB-OprM及MexCD-OprJ的表达上调可分别由它们各自的调控基因mexR及nfxB突变引起[11-13],药物靶基因gyrA、gyrB的突变可引起对喹诺酮类药物耐药性增强[14-15]等。究竟是何种机制导致PA生物被膜基因突变率增强和耐药性提高呢?本课题通过构建ku基因的缺失突变菌株及其回补株,针对Ku对PA生物被膜耐药性的作用展开研究。
1 材料与方法 1.1 材料 1.1.1 菌株、质粒及培养条件研究涉及的菌株与质粒见表 1。大肠杆菌、铜绿假单胞菌及其突变菌株在37 ℃的LB培养基中培养,MIC测定使用MH培养基。
| 材料 Materials |
基因型及相关特征 Genotype and relevant characteristics |
来源 Source |
| 菌株Strains | ||
| E. coli | ||
| DH5α λpir | F-φ80 lac ZΔM15Δ(lacZYA-arg F) LAMpirU169 endA1 recA1 hsdR17(rk–, mk+) supE44λ– thi –1 gyrA96 relA1 phoA | This Lab |
| β2163 | F–, RP4-2-Tc::Mu ΔdapA::(erm-pir) | [16] |
| Pseudomonas aeruginosa | ||
| PAO1 | Wild type | This Lab |
| Δku | PAO1, knockout of ku gene | This study |
| Δku::ku | PAO1 Δku::ku | This study |
| 质粒Plasmids | ||
| pLP12 | oriTRP4 oriVR6Kvmi480 PBAD, Cmr | [17] |
| pLP12-ku | pLP12 derivative containing homologousarms of ku gene of PAO1 | This study |
| pBAD33 | pACYC184 ori, araC ParaBAD, Cmr | [18] |
| pBAD33-ku | pBAD33 derivative containing kugene | This study |
实验所用培养基及抗生素均购自北京索莱宝科技有限公司;2, 6-二氨基庚二酸(DAP)、D-Glucose和L-阿拉伯糖,Sigma-Aldrich公司;PrimeSTAR Max DNA Polymerase和限制性内切酶EcoR I、Nhe I、Spe I及T4 DNA ligase,TaKaRa公司;PCR产物纯化试剂盒、细菌基因组提纯试剂盒、细菌总RNA提取试剂盒,天根生化科技(北京)有限公司;无缝克隆试剂盒ClonExpress II,南京诺唯赞生物科技有限公司;EasyScript First-Strand cDNA Synthesis SuperMix和SYBR Green Select Master Mix,北京全式金生物技术有限公司。96孔全自动梯度PCR扩增仪,Bio-Rad公司;实时荧光定量PCR仪,Applied Biosystems公司;MultiskanTM FC酶标仪,Thermo Scientific公司;引物合成和测序由生工生物工程(上海)股份有限公司完成。
1.2 方法 1.2.1 铜绿假单胞菌ku缺失突变株的构建铜绿假单胞菌ku缺失突变株的构建方法参照Luo等[17]方法,所用引物见表 2。根据细菌基因组提纯试剂盒使用说明提取铜绿假单胞菌PAO1基因组DNA作为模板,使用PrimeSTAR Max DNA Polymerase,以ku-EcoR I-MF1和ku-MR1为引物扩增ku基因上游同源臂A片段(531 bp),以ku-MF2和ku-Nhe I-MR2为引物扩增ku基因下游同源臂B片段(705 bp)。PCR反应体系(50 μL):DNA模板(150 mg/L) 1 μL,引物F/R (10 μmol/L)各1 μL,2×PrimeSTAR Max Premix 25 μL,ddH2O 22 μL。PCR反应条件:98 ℃ 1.5 min;98 ℃ 10 s,56 ℃ (A片段)/60 ℃ (B片段) 20 s,72 ℃ 1 min,30个循环;72 ℃ 7 min。以经PCR产物纯化后的A、B片段为模板,以ku-EcoR I-MF3和ku-Spe I-MR3为引物,通过交叠PCR扩增获得包含A、B片段DNA序列以及EcoR I、Spe I酶切位点的融合片段。PCR反应体系(50 μL):DNA模板A片段(100 mg/L) 0.5 μL,B片段(100 mg/L) 0.5 μL,引物F/R (10 μmol/L)各1 μL,2×PrimeSTAR Max Premix 25 μL,ddH2O 22 μL。PCR反应条件:98 ℃ 1 min;98 ℃ 10 s,68 ℃ 20 s,72 ℃ 30 s,7个循环;98 ℃ 10 s,58 ℃ 20 s,72 ℃ 1 min,30个循环。融合片段纯化后通过EcoR I/Spe I双酶切,得到的片段与经过双酶切的自杀载体pLP12连接。连接产物转化至大肠杆菌DH5α λpir感受态细胞,通过LB培养基中添加12 mg/L盐酸四环素和0.3% D-葡萄糖双重抗性以及PCR扩增筛选获得重组质粒pLP12-ku。
| 引物 Primers |
序列 Sequences (5′→3′) |
来源出处 Source |
| ku-EcoR I-MF1 | TGACGATGAATTCCACCATTCTCAAGGCAGGTCGGG | This study |
| ku-MR1 | ATTTACCCAGCGGCTCGCCTCACGGTATATGCACCAGGCCGAAGCT | This study |
| ku-MF2 | AGCTTCGGCCTGGTGCATATACCGTGAGGCGAGCCGCTGGGTAAAT | This study |
| ku-Nhe I-MR2 | TGTCAGAGCTAGCGGCCGACCAGACGCAACAGTTC | This study |
| ku-EcoR I-MF3 | TGACGATGAATTCCAGGTCGGGAAAGCCGGGTCA | This study |
| ku-Spe I-MR3 | TAGTCGTACTAGTATGCTTGGCACGCAATTCGCG | This study |
| ku-RF | TGGGCTAGCGAATTCGAGCTAGGAGGAATTCACCATGGCGCGTGCGATC | This study |
| ku-RR | TGCCTGCAGGTCGACTCTAGTCAGGCCTTGCGCCG | This study |
| pBAD-ZF | CTAGAGTCGACCTGCAGGCA | [19] |
| pBAD-ZR | AGCTCGAATTCGCTAGCCCA | [19] |
| pBAD-mcf-TF | CCATAAGATTAGCGGATCCTACCT | [19] |
| pBAD-mcf-TR | CTTCTCTCATCCGCCAAAACAG | [19] |
| ku-F | GGCTACAAGCGGGTGAACAA | This study |
| ku-R | GGCTATCGACGAAAGCGAAGA | This study |
| 16s-F | GCGCAACCCTTGTCCTTAGTT | [20] |
| 16s-R | TGTCACCGGCAGTCTCCTTAG | [20] |
| 注:下划线序列:限制性内切酶识别序列. Note: Underlined sequences: Restriction endonuclease recognition sequences. |
||
重组质粒pLP12-ku电转化至大肠杆菌β2163感受态细胞,β2163因营养缺陷需在含有DAP的培养基上才能生长,通过12 mg/L盐酸四环素和0.3 mmol/L DAP筛选获得β2163/pLP12-ku菌株。β2163/pLP12-ku菌株作为供体菌,铜绿假单胞菌PAO1作为受体菌,分别在37 ℃、150 r/min振荡培养过夜,各取培养物100 μL混合后于37 ℃、200 r/min条件下振荡培养6 h,进行接合实验。先用含盐酸四环素(30 mg/L)和0.3% D-葡萄糖的LB培养基进行初筛,待菌落长成,提取细菌基因组DNA,以ku-EcoR I-MF3/ku-Spe I-MR3为引物对扩增鉴定;然后复筛:挑取鉴定正确的单克隆菌落于含0.3% D-葡萄糖的LB液体培养基中37 ℃培养过夜,取培养液涂布于含0.4% L-阿拉伯糖的LB平板中37 ℃培养过夜,挑取平板菌落,以ku-EcoR I-MF1/ku-Nhe I-MR2为引物对进行PCR扩增检测,获得缺失突变克隆,PCR产物纯化后送至生工生物工程(上海)股份有限公司进行测序验证。突变株用终浓度为20%的甘油于–80 ℃保存,命名为Δku。
1.2.2 铜绿假单胞菌ku基因敲除回补株的构建以铜绿假单胞菌PAO1基因组为模板,ku-RF/ku-RR为引物对扩增获得ku基因表达片段(936 bp),PCR反应体系(50 μL):DNA模板(150 mg/L) 1 μL,引物F/R (10 μmol/L)各1 μL,2×PrimeSTAR Max Premix 25 μL,ddH2O 22 μL。PCR反应条件:98 ℃ 3 min;98 ℃ 10 s,58 ℃ 20 s,72 ℃ 1 min,30个循环;72 ℃ 7 min。以pBAD33为模板,pBAD-ZF/pBAD-ZR为引物对扩增获得pBAD33载体片段(5 529 bp)。PCR反应体系(50 μL):DNA模板(200 mg/L) 1 μL,引物F/R (10 μmol/L)各1 μL,2×PrimeSTAR Max Premix 25 μL,ddH2O 22 μL。PCR反应条件:98 ℃ 3 min;98 ℃ 10 s,60 ℃ 20 s,72 ℃ 3 min,30个循环;72 ℃ 7 min。用ClonExpress II将纯化的ku基因表达片段与pBAD33载体片段进行无缝克隆连接,构建重组质粒pBAD33-ku;将pBAD33-ku转化于大肠杆菌DH5α感受态细胞,转化产物涂布于含10 μg/mL氯霉素的LB固体培养基,37 ℃培养过夜,并筛选重组克隆以pBAD-mcf-TF/pBAD- mcf-TR为引物进行PCR鉴定(1 040 bp);提取重组阳性克隆质粒转化至大肠杆菌β2163感受态细胞,转化产物涂布于含10 μg/mL氯霉素和0.3 mmol/L DAP的LB固体培养基,以筛选获得β2163/ pBAD33-ku菌株。以β2163/pBAD33-ku菌株作为供体菌,与缺失株PAO1Δku进行接合培养,培养物涂布于含有10 μg/mL氯霉素的LB抗性平板筛选克隆,以pBAD-mcf-TF/pBAD-mcf-TR为引物对进行PCR检测(1 040 bp),并将PCR产物纯化后送至生工生物工程(上海)股份有限公司测序验证。回补株命名为Δku::ku。
1.2.3 生物被膜的培养向24孔细胞培养板每个反应孔内置入1片灭菌细菌片(直径14 mm),并加入2 mL LB液体培养基和100 μL稀释细菌悬液(终浓度为105 CFU/mL)混匀,37 ℃培养。每块培养板设置一个阴性对照孔(不加入菌悬液)。每24 h更换一次培养液(吸去1 mL菌悬液,加入1 mL LB液体培养基)。PAO1、Δku以及Δku::ku菌株在24孔细胞培养板中按上述方法进行生物被膜培养。取出细菌爬片,在无菌生理盐水中轻轻漂洗,收集漂洗后的生理盐水为浮游菌液。用细胞刮刀刮取细菌爬片上的细菌溶于生理盐水中,制得生物被膜菌液。
1.2.4 生物被膜形成能力的检测过夜培养PAO1、Δku以及Δku::ku菌株,将各菌液稀释至终浓度为105 CFU/mL,取200 μL加入于96孔板,每个菌株3个平行,等量LB液体培养基为阴性对照,于37 ℃培养,每24 h更换一次培养液。分别对培养3 d和6 d的96孔板用蒸馏水冲洗3次,洗去浮游菌,用甲醛固定5 min,结晶紫染色30 min,再用蒸馏水冲洗3次,放置至干燥后用400 μL 95%乙醇脱色,所得有色溶液用分光光度计测590 nm处吸光值。
1.2.5 生物被膜基因突变率的检测取100 μL培养1、3、6、9 d的各菌株生物被膜菌液及PAO1浮游菌液稀释后,分别涂布于LB固体培养基和含100 mg/L利福平的LB固体培养基,37 ℃培养18 h后进行平板菌落计数,按照Rosche等[21]方法计算基因突变率。
1.2.6 生物被膜菌耐药性的检测按照上述方法培养PAO1、Δku及Δku::ku菌株的生物被膜,分别取培养2、6、12 d的生物被膜溶于MH培养基中制得生物被膜菌液。采用微量稀释法测定环丙沙星、头孢他啶和庆大霉素的MIC。于96孔微量滴定板各孔中加入100 μL MH培养基,配制抗生素溶液,第一孔内加入浓度为128 mg/L的抗生素溶液100 μL,逐孔2倍稀释至浓度为0.5 mg/L,于每孔中加入稀释至0.5麦氏单位的生物被膜菌液,MH液体培养基为阴性对照,不加抗生素溶液的菌液为阳性对照,37 ℃培养20 h后观察结果,测得MIC。
1.2.7 基因转录水平检测分别取培养1、2、4、6 d的PAO1生物被膜菌液和浮游菌液,根据天根RNA抽提试剂盒说明书,分别抽提总RNA,取300 ng总RNA,通过EasyScript First-Strand cDNA Synthesis SuperMix反转录合成cDNA,反转录体系为20 μL。使用SYBR Green Select Master Mix,以cDNA为模板,ku-F/ku-R为特异性引物对,采用qPCR检测ku基因在浮游状态和生物被膜状态的转录水平。以16S rRNA基因为内参基因,所用引物见表 2。qPCR的反应体系(10 μL):cDNA 1 μL (cDNA使用前进行稀释:检测ku基因稀释10倍,检测16S rRNA基因稀释100倍),上、下游引物(10 μmol/L)各0.5 μL,SYBR Green Select Master Mix 5 μL,超纯水3 μL。qPCR反应条件:95 ℃ 1 min;95 ℃ 10 s,55 ℃ 30 s,72 ℃ 30 s,40个循环。采用2-ΔΔCt (Livak)法分析数据,计算不同培养时间生物被膜菌中ku基因的转录水平。
2 结果与分析 2.1 铜绿假单胞菌ku缺失突变菌株的构建根据引物的设计,以PAO1基因组DNA为模板,扩增ku基因(PA2150)上游片段(A)和下游片段(B),通过交叠PCR的方法将A片段和B片段融合为AB片段,并构建至载体pLP12中,获得重组自杀质粒(pLP12-ku)。将pLP12-ku电转化至大肠杆菌β2163中,获得供体菌β2163/pLP12-ku。供体菌与受体菌PAO1混合培养后,通过盐酸四环素和D-葡萄糖进行初筛,获得重组自杀质粒插入受体菌基因组的单交换同源重组克隆。单交换同源重组克隆经过L-阿拉伯糖诱导,vmi480致死基因反向筛选获得双交换同源重组克隆。以ku-EcoR I-MF1/ku-Nhe I-MR2为引物对,通过PCR扩增检测双交换克隆,获得ku缺失突变菌株。其中,回复突变克隆扩增片段为2 060 bp,ku缺失突变克隆扩增片段为1 236 bp (图 1A)。最后通过PCR产物测序验证证实ku缺失突变菌株Δku构建成功。
|
| 图 1 ku基因缺失株及其回补株的PCR验证 Figure 1 PCR verification of ku gene deletion mutant strains and its complementary strains 注:M:DL2000 DNA Marker;A:1为野生菌株PAO1,2-6为ku缺失突变克隆Δku;B:1-6为回补株,7为阳性对照β2163/pBAD33-ku,8为阴性对照Δku. Note: M: DL2000 DNA Marker; A: Wild-type PAO1 in lane 1, ku gene deletion clones Δku in lanes 2-6; B: Complementary strains in lanes 1-6, positive control β2163/pBAD33-ku in lane 7, negative control Δku in lane 8. |
|
|
根据引物设计,分别扩增获得ku基因及pBAD33载体片段,两者通过无缝克隆连接构建重组表达质粒pBAD33-ku,将重组质粒电转化至大肠杆菌β2163中,获得供体菌β2163/pBAD33-ku。供体菌与受体菌Δku接合培养获得回补株Δku::ku。以突变株为阴性对照,β2163/pBAD33-ku为阳性对照,pBAD-mcf-TF/pBAD-mcf-TR为引物对进行PCR检测,阳性克隆PCR条带大小为1 040 bp (图 1B)。
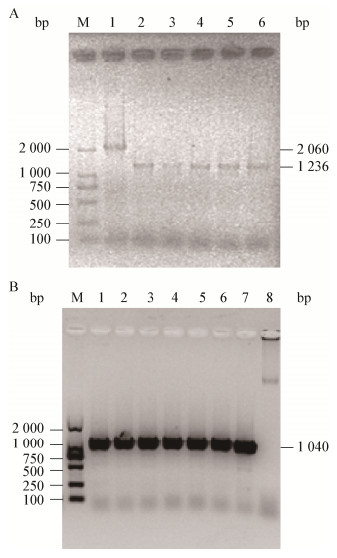
2.3 ku缺失对铜绿假单胞菌生物被膜形成能力的影响比较PAO1、Δku以及Δku::ku菌株生物被膜的形成能力,结果显示,培养3 d和6 d时,突变菌株与野生菌株形成的生物被膜厚度无显著差异(图 2)。
|
| 图 2 ku基因对生物被膜菌形成能力的影响 Figure 2 Effect of ku gene on biofilm-forming ability of PAO1 at different cultivation times |
|
|
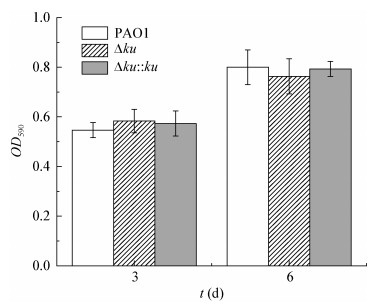
以PAO1浮游菌液为对照,比较PAO1、Δku及Δku::ku这3种菌株在培养1、3、6、9 d时生物被膜中细菌的基因突变率。PAO1浮游菌的基因突变率在培养1 d时为0.6×10-11,培养9 d时上升至1.06×10-11,而生物被膜中的PAO1基因突变率显著高于浮游菌(图 3),这与Driffield等[22]的研究结果相符。另外,生物被膜状态下,PAO1基因突变率随着培养时间的不同发生着变化,在第6天时,基因突变率达到最高值,而Δku的基因突变率在不同培养时间几乎没有变化,此时两者基因突变率差异达到最高,Δku基因突变率降低至PAO1约1/5处(P < 0.01)。回补株Δku::ku与野生菌株无显著差异(P > 0.05)。
|
| 图 3 ku基因对生物被膜中的PAO1基因突变率的影响 Figure 3 Effect of ku gene on mutation rate of PAO1 within biofilms grown at different cultivation times 注:PL:浮游菌;BF:生物被膜. Note: PL: Planktonic bacteria; BF: Biofilm. |
|
|
比较PAO1、Δku以及Δku::ku生物被膜菌对环丙沙星、头孢他啶和庆大霉素的耐药性,发现两者的耐药性随着生物被膜培养时间的延长皆有所增强。生物被膜培养6 d和12 d时,Δku对环丙沙星以及庆大霉素的耐药性比PAO1下降了50%,而两种菌株对头孢他啶的耐药性并无明显差别。回补株生物被膜对各抗菌药物耐药性与野生菌株无差异(表 3)。
| 抗生素 Antibiotics |
培养时间 Time (d) |
菌株 Strains |
||
| PAO1 | Δku | Δku::ku | ||
| 环丙沙星 Ciprofloxacin |
2 | 0.5 | 0.5 | 0.5 |
| 6 | 2.0 | 1.0 | 2.0 | |
| 12 | 8.0 | 4.0 | 8.0 | |
| 庆大霉素 Gentamicin |
2 | 1.0 | 1.0 | 1.0 |
| 6 | 4.0 | 2.0 | 4.0 | |
| 12 | 8.0 | 4.0 | 8.0 | |
| 头孢他啶 Ceftazidime |
2 | 0.5 | 0.5 | 0.5 |
| 6 | 2.0 | 2.0 | 2.0 | |
| 12 | 4.0 | 4.0 | 4.0 | |
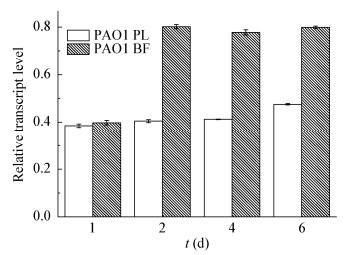
通过qPCR比较PAO1浮游菌与PAO1生物被膜细菌在不同培养时间ku基因的转录水平,结果表明,培养2 d后,PAO1生物被膜ku基因转录水平比PAO1浮游菌增强了2倍,这样的差异并未随着生物被膜培养时间的延长而有所改变(图 4)。
|
| 图 4 不同培养时间下ku基因转录水平 Figure 4 Transcription level of ku gene in PAO1 biofilms grown at different cultivation times compared to PAO1 planktonic bacteria 注:PL:浮游菌;BF:生物被膜. Note: PL: Planktonic bacteria; BF: Biofilm. |
|
|
铜绿假单胞菌是临床常见条件致病菌,生物被膜的形成极大地增强了其耐药性。生物被膜的耐药机制由多种因素引起,包括外排泵机制上调、免疫逃逸、物理屏障以及低营养微环境等[23]。另外,表型变异在生物被膜特殊的环境因素中产生,也使得细菌对多种抗菌药物耐药[24-25]。研究显示,DNA错配修复途径(DNA mismatch repair)的失活可引起基因突变频率增加,从而引起PA生物被膜对抗菌药物耐药水平的提高[26-28],这表明基因突变与生物被膜耐药性密切相关。实际上,即使在非人工诱导的情况下,野生型PA生物被膜的基因突变率可达浮游菌的100倍[22]。究竟是何种机制导致PA生物被膜基因突变率增加以及PA生物被膜的耐药作用增强目前仍不清楚。
NHEJ是DSB的主要修复机制之一,修复过程无需同源模板,直接将断裂缺口连接,常导致DNA发生序列改变。研究表明恶臭假单菌NHEJ与稳定生长期的细菌变异相关[29],铜绿假单胞菌NHEJ途径可促进染色体片段缺失[30]。细菌的NHEJ修复途径主要由Ku蛋白和LigD连接酶参与,Shen等的研究显示Δku突变体与ΔkuΔligD双缺失突变体丢失染色体片段的概率下降,而Δku突变体的ku基因回补提高了染色体片段缺失的概率[30],这些数据表明了Ku蛋白在NHEJ修复途径中的关键性。因此,我们认为ku基因缺失可导致NHEJ途径的失活。
NHEJ可以作用于细胞周期的任何时期,但在细菌中NHEJ的作用主要体现在生长稳定期、饥饿状态或孢子等营养低、生长慢的环境中[31-32],这与生物被膜特殊的微环境状况非常相似。据谢轶等[33]的研究表明,PA细菌悬液培养1 d后未形成生物被膜,培养3 d后开始形成生物被膜,培养6 d可形成很厚的生物被膜。本研究显示,ku基因表达水平在PA生物被膜培养2 d后有所提高,说明NHEJ的作用在生物被膜形成早期得到了一定程度的激活,但是生物被膜形成能力检测结果表明ku基因并不影响生物被膜的形成。我们由此推测在PA中,生物被膜的形成不受NHEJ调节,但其是NHEJ作用的主要环境。
本研究中,通过筛选利福平耐药突变体检测生物被膜中PA野生菌株及ku缺失菌株的基因突变率,结果显示,随着生物被膜的形成和成熟,PA基因突变率不断提高,在培养6 d时达到最大值,但ku缺失突变株并未显示基因突变率的变化,这一结果表明NHEJ对生物被膜中PA基因突变率的提高有一定的促进作用。文献报道利福平耐药主要是因为rpoB基因发生突变引起,包括点突变、短的插入和缺失突变[34-35],而NHEJ修复DSB引起的突变以短的插入和缺失为主[36]。我们由此推测ku缺失导致NHEJ途径被抑制,致使rpoB基因发生突变的频率降低,PA基因突变率下降。为验证这一推测,我们下一步的工作将对PA利福平耐药突变体进行rpoB基因测序,分析其突变类型,进一步明确ku介导的突变与生物被膜中PA基因突变率上升的联系。
PA对抗菌药物的耐药性可由多种耐药机制介导,而研究发现耐药机制可由基因突变激活或调节,如外排泵MexAB-OprM因mexR、nalB、nalC或nalD基因突变而上调,增强了对β-内酰胺类和喹诺酮类药物的耐受力[37];喹诺酮类药物靶基因gyrA、gyrB或parC、parE以及氨基糖苷类药物靶基因rplY的突变降低了PA对该药物的敏感性[37-38];ampC基因突变可增强β-内酰胺酶表达量,大大增强PA对头孢菌素的耐药性[39]。本研究发现ku基因对生物被膜状态中的PA环丙沙星和庆大霉素的耐药性有一定的正调控作用,这极有可能源于NHEJ途径在修复DNA的过程中产生的突变涉及了PA耐药机制的相关基因而影响了其活性,但NHEJ产生的突变影响了何种耐药机制以及其如何引起耐药机制活性变化,这一系列问题还有待进一步的研究。
综上所述,我们通过本研究发现PA在生物被膜环境下NHEJ途径活性增强,引起基因突变率升高,对某些抗菌药物耐药性增强。我们将在未来的工作中进一步探讨NHEJ引起基因突变率和耐药性增强的机制。
| [1] |
Costerton JW, Stewart PS, Greenberg EP. Bacterial biofilms: a common cause of persistent infections[J]. Science, 1999, 284(5418): 1318-1322. DOI:10.1126/science.284.5418.1318 |
| [2] |
Häussler S. Multicellular signalling and growth of Pseudomonas aeruginosa[J]. International Journal of Medical Microbiology, 2010, 300(8): 544-548. DOI:10.1016/j.ijmm.2010.08.006 |
| [3] |
Kostakioti M, Hadjifrangiskou M, Hultgren SJ. Bacterial biofilms: development, dispersal, and therapeutic strategies in the dawn of the postantibiotic era[J]. Cold Spring Harbor Perspectives in Medicine, 2013, 3(4): a010306. |
| [4] |
Della M, Palmbos PL, Tseng HM, et al. Mycobacterial Ku and ligase proteins constitute a two-component NHEJ repair machine[J]. Science, 2004, 306(5696): 683-685. DOI:10.1126/science.1099824 |
| [5] |
de Ory A, Zafra O, de Vega M. Efficient processing of abasic sites by bacterial nonhomologous end-joining Ku proteins[J]. Nucleic Acids Research, 2014, 42(21): 13082-13095. DOI:10.1093/nar/gku1029 |
| [6] |
Aravind L, Koonin EV. Prokaryotic homologs of the eukaryotic DNA-end-binding protein Ku, novel domains in the Ku protein and prediction of a prokaryotic double-strand break repair system[J]. Genome Research, 2001, 11(8): 1365-1374. DOI:10.1101/gr.181001 |
| [7] |
Weller GR, Kysela B, Roy R, et al. Identification of a DNA nonhomologous end-joining complex in bacteria[J]. Science, 2002, 297(5587): 1686-1689. DOI:10.1126/science.1074584 |
| [8] |
Bowater R, Doherty AJ. Making ends meet: repairing breaks in bacterial DNA by non-homologous end-joining[J]. PLoS Genetics, 2006, 2(2): e8. DOI:10.1371/journal.pgen.0020008 |
| [9] |
Pitcher RS, Brissett NC, Doherty AJ. Nonhomologous end-joining in bacteria: a microbial perspective[J]. Annual Review of Microbiology, 2007, 61: 259-282. DOI:10.1146/annurev.micro.61.080706.093354 |
| [10] |
Shuman S, Glickman MS. Bacterial DNA repair by non-homologous end joining[J]. Nature Reviews Microbiology, 2007, 5(11): 852-861. DOI:10.1038/nrmicro1768 |
| [11] |
Poole K, Tetro K, Zhao Q, et al. Expression of the multidrug resistance operon mexA-mexB-oprM in Pseudomonas aeruginosa: mexR encodes a regulator of operon expression[J]. Antimicrobial Agents and Chemotherapy, 1996, 40(9): 2021-2028. DOI:10.1128/AAC.40.9.2021 |
| [12] |
Jeannot K, Elsen S, Köhler T, et al. Resistance and virulence of Pseudomonas aeruginosa clinical strains overproducing the MexCD-OprJ efflux pump[J]. Antimicrobial Agents and Chemotherapy, 2008, 52(7): 2455-2462. DOI:10.1128/AAC.01107-07 |
| [13] |
Wang SC, Yang ZM, Cai XY, et al. Expression of MexAB-OprM, MexCD-OprJ, MexEF-OprN and MexXY-OprM in multidrug-resistant Pseudomonas aeruginosa and their relationship with drug resistance phenotypes in a Third Class A Hospital in Hainan[J]. Practical Pharmacy and Clinical Remedies, 2020, 23(3): 247-252. (in Chinese) 王生成, 杨祚明, 蔡潇阳, 等. 海南某三甲医院多重耐药铜绿假单胞菌MexAB-OprM、MexCD-OprJ、MexEF-OprN、MexXY-OprM表达及与耐药表型的关系研究[J]. 实用药物与临床, 2020, 23(3): 247-252. |
| [14] |
Lee JK, Lee YS, Park YK, et al. Alterations in the GyrA and GyrB subunits of topoisomerase Ⅱ and the ParC and ParE subunits of topoisomerase IV in ciprofloxacin-resistant clinical isolates of Pseudomonas aeruginosa[J]. International Journal of Antimicrobial Agents, 2005, 25(4): 290-295. DOI:10.1016/j.ijantimicag.2004.11.012 |
| [15] |
Zhang GD, Zeng ZR, Wang Y, et al. Mechanisms of gyrA, gyrB and efflux system jointly mediating quinolone resistance in clinical isolates of Pseudomonas aeruginosa[J]. Chinese Journal of Infection and Chemotherapy, 2014, 14(3): 224-228. (in Chinese) 张国栋, 曾章锐, 王莹, 等. gyrA、gyrB和外排系统共同介导铜绿假单胞菌对喹诺酮类耐药机制研究[J]. 中国感染与化疗杂志, 2014, 14(3): 224-228. |
| [16] |
Demarre G, Guérout AM, Matsumoto-Mashimo C, et al. A new family of mobilizable suicide plasmids based on broad host range R388 plasmid (IncW) and RP4 plasmid (IncPα) conjugative machineries and their cognate Escherichia coli host strains[J]. Research in Microbiology, 2005, 156(2): 245-255. DOI:10.1016/j.resmic.2004.09.007 |
| [17] |
Luo P, He XY, Liu QT, et al. Developing universal genetic tools for rapid and efficient deletion mutation in Vibrio species based on suicide T-vectors carrying a novel counterselectable marker, vmi480[J]. PLoS One, 2015, 10(12): e0144465. DOI:10.1371/journal.pone.0144465 |
| [18] |
Guzman LM, Belin D, Carson MJ, et al. Tight regulation, modulation, and high-level expression by vectors containing the arabinose PBAD promoter[J]. Journal of Bacteriology, 1995, 177(14): 4121-4130. DOI:10.1128/JB.177.14.4121-4130.1995 |
| [19] |
Wang WJ, Xu J, Gao HX, et al. Construction of baeSR gene knock-out in Salmonella typhimurium and analysis of its sensitivity to antibiotics[J]. Microbiology China, 2019, 46(11): 3013-3021. (in Chinese) 王文静, 徐军, 高海侠, 等. 鼠伤寒沙门菌baeSR基因缺失株的构建及其对抗生素敏感性分析[J]. 微生物学通报, 2019, 46(11): 3013-3021. |
| [20] |
Zhao JM, Jiang HD, Cheng W, et al. The role of quorum sensing system in antimicrobial induced ampC expression in Pseudomonas aeruginosa biofilm[J]. Journal of Basic Microbiology, 2015, 55(5): 671-678. DOI:10.1002/jobm.201300987 |
| [21] |
Rosche WA, Foster PL. Determining mutation rates in bacterial populations[J]. Methods, 2000, 20(1): 4-17. |
| [22] |
Driffield K, Miller K, Bostock JM, et al. Increased mutability of Pseudomonas aeruginosa in biofilms[J]. Journal of Antimicrobial Chemotherapy, 2008, 61(5): 1053-1056. DOI:10.1093/jac/dkn044 |
| [23] |
Hall-Stoodley L, Stoodley P. Evolving concepts in biofilm infections[J]. Cellular Microbiology, 2009, 11(7): 1034-1043. DOI:10.1111/j.1462-5822.2009.01323.x |
| [24] |
Giraud A, Radman M, Matic I, et al. The rise and fall of mutator bacteria[J]. Current Opinion in Microbiology, 2001, 4(5): 582-585. DOI:10.1016/S1369-5274(00)00254-X |
| [25] |
Macià MD, Pérez JL, Molin S, et al. Dynamics of mutator and antibiotic-resistant populations in a pharmacokinetic/pharmacodynamic model of Pseudomonas aeruginosa biofilm treatment[J]. Antimicrobial Agents and Chemotherapy, 2011, 55(11): 5230-5237. DOI:10.1128/AAC.00617-11 |
| [26] |
Smania AM, Segura I, Pezza RJ, et al. Emergence of phenotypic variants upon mismatch repair disruption in Pseudomonas aeruginosa[J]. Microbiology, 2004, 150(5): 1327-1338. DOI:10.1099/mic.0.26751-0 |
| [27] |
Plasencia V, Borrell N, Maciá MD, et al. Influence of high mutation rates on the mechanisms and dynamics of in vitro and in vivo resistance development to single or combined antipseudomonal agents[J]. Antimicrobial Agents and Chemotherapy, 2007, 51(7): 2574-2581. DOI:10.1128/AAC.00174-07 |
| [28] |
Luján AM, Maciá MD, Yang L, et al. Evolution and adaptation in Pseudomonas aeruginosa biofilms driven by mismatch repair system-deficient mutators[J]. PLoS One, 2011, 6(11): e27842. DOI:10.1371/journal.pone.0027842 |
| [29] |
Paris Ü, Mikkel K, Tavita K, et al. NHEJ enzymes LigD and Ku participate in stationary-phase mutagenesis in Pseudomonas putida[J]. DNA Repair, 2015, 31: 11-18. DOI:10.1016/j.dnarep.2015.04.005 |
| [30] |
Shen MY, Zhang HD, Shen W, et al. Pseudomonas aeruginosa MutL promotes large chromosomal deletions through non-homologous end joining to prevent bacteriophage predation[J]. Nucleic Acids Research, 2018, 46(9): 4505-4514. DOI:10.1093/nar/gky160 |
| [31] |
Pitcher RS, Green AJ, Brzostek A, et al. NHEJ protects mycobacteria in stationary phase against the harmful effects of desiccation[J]. DNA Repair, 2007, 6(9): 1271-1276. DOI:10.1016/j.dnarep.2007.02.009 |
| [32] |
Moeller R, Reitz G, Li ZF, et al. Multifactorial resistance of Bacillus subtilis spores to high-energy proton radiation: role of spore structural components and the homologous recombination and non-homologous end joining DNA repair pathways[J]. Astrobiology, 2012, 12(11): 1069-1077. DOI:10.1089/ast.2012.0890 |
| [33] |
Xie Y, Yang WQ, Jia WX, et al. Change of intrinsic drug resistance in the formation of Pseudomonas aeruginosa biofilms[J]. Chinese Journal of Microbiology and Immunology, 2005, 25(4): 314-318. (in Chinese) 谢轶, 杨维青, 贾文祥, 等. 铜绿假单胞菌耐药性在生物被膜形成过程中的变化[J]. 中华微生物学和免疫学杂志, 2005, 25(4): 314-318. DOI:10.3760/j:issn:0254-5101.2005.04.019 |
| [34] |
Farhat MR, Sultana R, Iartchouk O, et al. Genetic determinants of drug resistance in Mycobacterium tuberculosis and their diagnostic value[J]. American Journal of Respiratory and Critical Care Medicine, 2016, 194(5): 621-630. DOI:10.1164/rccm.201510-2091OC |
| [35] |
Jatsenko T, Tover A, Tegova R, et al. Molecular characterization of Rifr mutations in Pseudomonas aeruginosa and Pseudomonas putida[J]. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 2010, 618(1/2): 106-114. |
| [36] |
Bertrand C, Thibessard A, Bruand C, et al. Bacterial NHEJ: a never ending story[J]. Molecular Microbiology, 2019, 111(5): 1139-1151. DOI:10.1111/mmi.14218 |
| [37] |
Pang Z, Raudonis R, Glick BR, et al. Antibiotic resistance in Pseudomonas aeruginosa: mechanisms and alternative therapeutic strategies[J]. Biotechnology Advances, 2019, 37(1): 177-192. DOI:10.1016/j.biotechadv.2018.11.013 |
| [38] |
El'Garch F, Jeannot K, Hocquet D, et al. Cumulative effects of several nonenzymatic mechanisms on the resistance of Pseudomonas aeruginosa to aminoglycosides[J]. Antimicrobial Agents and Chemotherapy, 2007, 51(3): 1016-1021. DOI:10.1128/AAC.00704-06 |
| [39] |
Berrazeg M, Jeannot K, Ntsogo Enguene VY, et al. Mutations in β-lactamase AmpC increase resistance of Pseudomonas aeruginosa isolates to antipseudomonal cephalosporins[J]. Antimicrobial Agents and Chemotherapy, 2015, 59(10): 6248-6255. DOI:10.1128/AAC.00825-15 |
 2020, Vol. 47
2020, Vol. 47