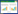扩展功能
文章信息
- 范婷文, 范耿文, 侯艾琦, 陈倩媚, 钞亚鹏, 孙艳
- FAN Ting-Wen, FAN Geng-Wen, HOU Ai-Qi, CHEN Qian-Mei, CHAO Ya-Peng, SUN Yan
- 基于分子动力学模拟的高γ-环糊精专一性环糊精糖基转移酶理性改造
- Rational engineering of high γ-cyclodextrin specific cyclodextrin glycosyltransferase based on molecular dynamics simulation
- 微生物学通报, 2020, 47(12): 4172-4181
- Microbiology China, 2020, 47(12): 4172-4181
- DOI: 10.13344/j.microbiol.china.200072
-
文章历史
- 收稿日期: 2020-02-01
- 接受日期: 2020-04-06
- 网络首发日期: 2020-05-09
2. 中国科学院微生物研究所 传感技术联合国家重点实验室 北京 100101;
3. 华北电力大学控制与计算机工程学院 北京 102206;
4. 北京航空航天大学生物医学工程高精尖创新中心 北京 102402
2. State Key Laboratory of Transducer Technology, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
3. School of Control and Computer Engineering, North China Electric Power University, Beijing 102206, China;
4. Beijing Advanced Innovation Centre for Biomedical Engineering, Beihang University, Beijing 102402, China
环糊精(cyclodextrin)是直链淀粉在由芽孢杆菌产生的环糊精糖基转移酶(cyclodextrin glycosyltransferase,CGTase,EC 2.4.1.19)作用下生成的一系列环状低聚糖的总称,在医药业、食品、电化学分析、农业等行业具有广泛的应用[1-3],主要由CGTase催化产生,但常得到是由α-、β-和γ-CD三种环糊精组成的混合物,如何提高CGTase产物专一性是生产中亟待解决的课题。研究者们大多使用的突变方法是易错PCR方法[4-5],以及通过总结文献找出的特定位点进行尝试性的定点突变以及饱和突变[6]。研究证明使用点突变的方法虽然能达到一定的效果[7],但效率仍不高。
分子动力学主要优势就是把大量的实验数据统一进行归纳分析,找出其中关键的有规律特征,深化了对物质的性质-结构关系以及化学物理变化动态演化过程的认识[8-11]。如今,在化学、物理学、生命科学等多个领域的科学家都在研究通过模拟计算指导实验,以减少实验的盲目性,节约成本和时间[12-13]。CGTase是一类以淀粉为底物偶联水解和环化过程的复杂催化反应,应用已有技术模拟其催化过程有一定的难度。有研究学者使用分子动力学研究了环糊精糖基转移酶的热稳定性[14-15],但是对于用分子动力学探究CGTase催化专一性机理的研究不多。本实验利用分子动力学模拟实验,通过模拟比较3种代表类型的环糊精糖基转移酶与底物的对接方式,预测CGTase口袋附近的重要位点,并指导定点突变对酶蛋白进行改造。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒菌种Bacillus sp. 602-1、重组质粒pET-22b(+)- cgt、携带该质粒的Escherichia coli BL21和E.coli DH5α均由本实验室构建并保藏。
1.1.2 主要试剂和仪器及培养基快速定点突变试剂盒,天根生化科技(北京)有限公司;引物、质粒小量提取试剂盒、DNA胶回收试剂盒,北京擎科生物科技有限公司;限制性内切酶Xho I、BamH I、T4 DNA连接酶,北京全式金生物技术公司;α-CD、β-CD、γ-CD标准品,美伦生物技术公司。高效液相色谱仪,Waters公司;PCR仪,Biometra公司;恒温摇床,上海福玛实验设备有限公司。LB培养基、TB培养基,北京酷来搏科技有限公司。
1.2 环糊精糖基转移酶与底物的分子对接与动力学模拟将蛋白质与小分子糖链利用autdock软件进行柔性对接,得到最初的对接位相结构。在对接中,将产生至少30个对接结果,挑选能量最好的位相进行提取,用于后续的分子动力学分析。用AMBER 14软件做分子动力学模拟实验,整个蛋白体系都采用gaff和ff14SB力场,以蛋白为中心,加10 Å的立方水盒子,加Na+使体系呈电中性,保存拓扑和坐标文件,然后进行模拟[16]。通过限制蛋白体系使整个体系水分子能量最小化。采用郎之万控温和各向同性的Berendsen控压方法分别达到平衡体系升温过程和升压平衡过程。最后采用无限自由模拟阶段进行动力学模拟,控压控温方法与前一步一致,范德华能和短程静电能的截断距离为10 Å,采用Particle mesh ewald (PME)方法计算长程静电能。
1.3 基于模拟结果的定点突变使用快速定点突变试剂盒进行实验,设计的引物信息见表 1。PCR反应体系(50 μL):5×Fast Alteration Buffer 10 μL,Fast Alteration DNA Polymerase (1 U/μL) 1 μL,正、反向引物(10 μmol/L)各2 μL,Template DNA 1 μL,超纯水补足至50 μL。PCR反应条件:95 ℃ 2 min;94 ℃ 20 s,55 ℃ 10 s,68 ℃ 2.5 min,30个循环;68 ℃ 5 min。进行定点突变实验之后将PCR反应产物加入1 μl的Dpn I,37 ℃进行消化实验1.5 h。加入350 mL的LB培养基于37 ℃、150 r/min振荡培养1 h,使菌体复苏,然后转化到E.coli DH5α感受态细胞中。待菌落长出后,进行突变克隆筛选。
| Primer | Primers sequence (5′→3′) | Size (bp) | (G+C)mol% | Tm | Gene Tm |
| Y262L-Forward | GGAATGGCTCCTTGGCGCGGATCAAACC | 28 | 61 | 73.3 | 62.0 |
| Y262L-Reverse | CCAAGGAGCCATTCCCCGAACGTAAATACCG | 31 | 55 | 71.7 | 63.6 |
| 注:带下划线的序列表示突变氨基酸的编码序列. Note: Underlined sequences denote the coding sequence of the mutated amino acid. | |||||
从平板上挑取单菌落接入5 ml LB培养基中(含100 μl/ml氨苄青霉素),37 ℃、200 r/min摇床培养12 h。将培养好的种子培养基按照1% (体积比)接种量接入50 ml TB培养基(含100 μl/ml氨苄青霉素)中进行发酵培养,在37 ℃、200 r/min培养至OD600为0.6−0.8时在冰上冷却10 min,添加IPTG至终浓度为0.01 mmol/L,转至16 ℃、200 r/min摇床培养,24 h后加入氯化钙和甘氨酸至终浓度分别为20 mmol/L和150 mmol/L,继续诱导培养72 h。
1.5 突变体酶的酶活测定将试管放入40 ℃水浴锅中进行预热,每支试管加入0.4 mL的0.25% (质量体积比)淀粉溶液在40 ℃水浴锅中静置10 min;在对照组中加入1.5 mL的0.1 mol/L盐酸溶液;实验组和对照组均加入0.1 mL已经稀释的酶液,40 ℃静置10 min;取出试管向实验组中加入1.5 mL的0.1 mol/L盐酸溶液以终止反应。实验组和对照组同时加入3 mL 1 mmol/L的I2-KI以及5 mL蒸馏水,摇匀。样品在700 nm波长的分光光度计下测定吸光度值,对照蒸馏水调零,计算酶活性。酶活性=[(对照组−实验组)/对照组]×100×稀释倍数。
1.6 突变体酶的产物专一性测定配制质量分数为1%的可溶性淀粉,加入400 U/g突变酶,在40 ℃水浴锅中转化24 h后,将样品煮沸10 min后终止反应,在12 000 r/min下离心10 min。将上清液收集并用0.22 μm的滤膜过滤,用高效液相色谱检测:色谱柱:Supersil NH2 (250 mm×4.6 mm×5 μm);流动相:乙腈/水(65/35,体积比);柱温:40 ℃;样品室温度:4 ℃;流速:1.0 mL/min;进样体积:20 μL;分析时间:20 min。
2 结果与分析 2.1 分子动力学模拟实验选取产α-、β-和γ-环糊精专一性最高的环糊精基糖转移酶结构作为代表。3种结构分别经过以下处理:α-环糊精的PDB号为3WMS,菌种为Paenibacillus macerans,用Pymol进行突变将Y167点的加入3个组氨酸;β-环糊精的PDB号为1DTU,菌种为Bacillus circulans,用Pymol进行突变将P146变为S146、将位点Y89位点变为F89;γ-环糊精的PDB号为4JCM,菌种为Bacillus clarkii。
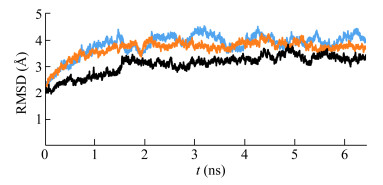
将处理完成的环糊精转移酶与十糖链进行分子对接以及分子动力学模拟,图 1显示了3种复杂度的均方根偏差(root mean square deviation,RMSD)值随时间的变化是通过分子动力模拟获得的。在经过2 ns的模拟时间后,这3种配合物的结构是稳定的,达到平衡后,每种配合物的平均RMSD波动值分别为3.0、3.5和3.5 Å。基于最终的6 ns MDs轨迹,从复杂的最终平均结构的最后1 ns轨迹中提取每个复杂结构的1 000张快照。最后选取模拟结果如图 2所示,其中图 2A和图 2B的环糊精糖基转移酶和十糖链在S1区域呈现Closed的形态,图 2C的环糊精糖基转移酶和十糖链在S1区域呈现更易于生成γ-环糊精的Open形态。模拟得出α-CGTase、β-CGTase和γ-CGTase与糖链对接体系的吉布斯自由能分别为−14.26、−12.13和−15.36 kcal/mol,表明γ-CGTase-糖链体系的结合能最高。
|
| 图 1 分子动力学模拟轨迹在6 ns内的RMSD值 Figure 1 The RMSD values obtained during 6 ns of molecular dynamic simulation for complex 注:蓝色:α-CGTase;红色:β-CGTase;黑色:γ-CGTase. Note: blue: α-CGTase; red: β-CGTase; black: γ-CGTase. |
|
|
|
| 图 2 三种环糊精糖基转移酶与十糖链的分子对接和分子动力学模拟结构的概览 Figure 2 Molecular docking and molecular dynamics simulation of three cyclodextrin glycosyltransferases with decasaccharide chains Note: A: α-CGTase; B: β-CGTase; C: γ-CGTase. |
|
|
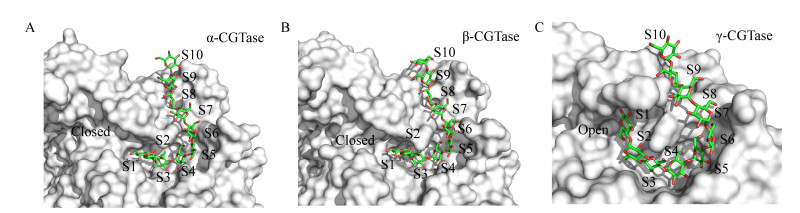
经过分析α-CGTase氨基酸和十糖链间相互作用,由于环糊精糖基转移酶的氨基酸与糖链间的氢键结构复杂,将十糖链分成3部分进行分析,以便分析清楚每一个氨基酸的氢键作用,即S10−S8区域、S7−S4区域和S3−S1,如图 3所示。通过分析得出,产α-CD最多的环糊精糖基转移酶与十糖链间重要作用力的氨基酸分别是R146、D147、H168、D145、N195、G182、Y89、W101、D374、H140、Y197、H235、K234、W261、Q267、Y262、E260、D331、H330、D374和R378等。
|
| 图 3 α-CGTase氨基酸和十糖链间相互作用三维立体图 Figure 3 Three dimensional diagram of interaction between α-CGTase and decasaccharide chain 注:A:十糖链和α-CGTase氨基酸之间的氢键图;B:糖链中的S10−S8与α-CGTase氨基酸之间的氢键放大图;C:S7−S4;D:S3−S1. Note: A: Hydrogen bond diagram between decasaccharide chain and α-CGTase amino acids; B: Enlarged diagram of hydrogen bond between S10−S8 and α-CGTase amino acids in sugar chain; C: S7−S4; D: S3−S1. |
|
|
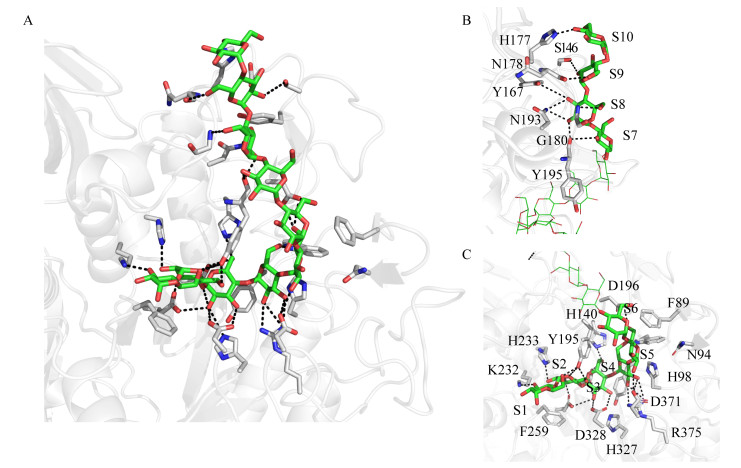
经过分析β-CGTase氨基酸和十糖链间相互作用,由于环糊精糖基转移酶的氨基酸与糖链间的氢键结构复杂,将十糖链分成两部分进行分析,以便于分析清楚每一个氨基酸的氢键作用,即S10−S7区域和S6−S1区域,如图 4所示。通过分析得知,产β-CD最多的环糊精糖基转移酶与十糖链间重要作用力的氨基酸分别是H177、S146、N178、Y167、N193、G180、Y195、H140、D196、H233、K232、F259、D328、H327、R375、D371、H98、N94、F89等。
|
| 图 4 β-CGTase氨基酸和十糖链间相互作用三维立体图 Figure 4 Three dimensional diagram of interaction between β-CGTase and decasaccharide chain 注:A:十糖链和β-CGTase氨基酸之间的氢键图;B:糖链中的S10−S7与β-CGTase氨基酸之间的氢键放大图;C:S6−S1. Note: A: Hydrogen bond diagram between decasaccharide chain and β-CGTase amino acids; B: Enlarged diagram of hydrogen bond between S10−S7 and β-CGTase amino acids in sugar chain; C: S6−S1. |
|
|
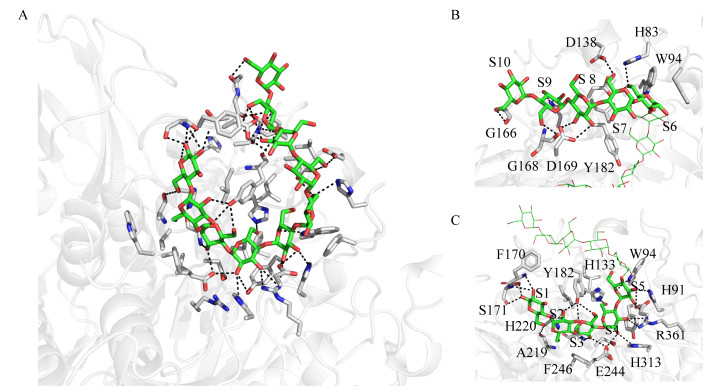
经过分析γ-CGTase氨基酸和十糖链间相互作用,由于环糊精糖基转移酶的氨基酸与糖链间的氢键结构复杂,将十糖链分成两部分进行分析,以便分析清楚每一个氨基酸的氢键作用,即S10−S6区域和S5−S1区域,如图 5所示。通过分析得知,产γ-CD最多的环糊精糖基转移酶与十糖链间重要作用力的氨基酸分别是G166、G168、D169、Y182、W94、H83、D138、H133、F170、S171、H220、A219、F246、E244、H313、R361、H91、D357等。
|
| 图 5 γ-CGTase氨基酸和十糖链间相互作用三维立体图 Figure 5 Three dimensional diagram of interaction between γ-CGTase and decasaccharide chain 注:A:十糖链和γ-CGTase氨基酸之间的氢键图;B:糖链中的S10−S6与γ-CGTase氨基酸之间的氢键放大图;C:糖链中的S5−S1与γ-CGTase氨基酸之间的氢键. Note: A: Hydrogen bond diagram between decasaccharide chain and γ-CGTase amino acids; B: Enlarged diagram of hydrogen bond between S10−S6 and γ-CGTase amino acids in sugar chain; C: Enlarged diagram of hydrogen bond between S5−S1 and γ-CGTase amino acids in sugar chain. |
|
|
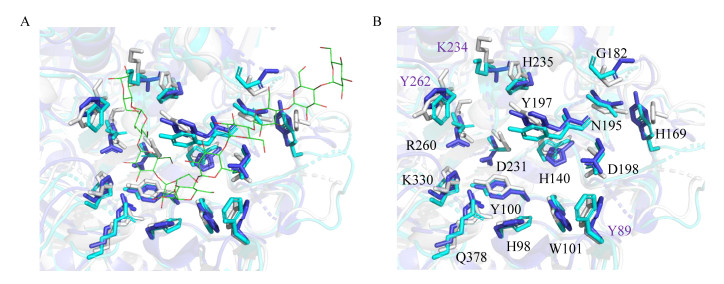
通过将3种环糊精糖基转移酶的所有氢键进行重合,选取3种CGTase氨基酸与十糖链在相同位置都存在氢键的氨基酸,如图 6所示。经过分析得出3种CGTase与十糖链重合的位置共有17个位点,其中14个位点上的氨基酸种类一致,3个位点上的氨基酸种类不一致。对应氨基酸一致的14个位点对应α-CGTase位点为H98、Y100、W101、H140、H169、G182、N195、Y197、D198、D231、H235、E260、H330和R378,对应氨基酸不一致的3个位点对应α-CGTase位点为Y89、D234和Y262。可以通过表 2直观看出3种环糊精糖基转移酶之间的氨基酸位置关系。
|
| 图 6 三种突变体与十糖链位置重合的氢键氨基酸 Figure 6 Hydrogen bond amino acids of three mutants overlapped with decasaccharide chain 注:A:相同位置氨基酸与十糖链之间的氨基酸位置关系,白色表示α-CGTase,浅蓝色表示β-CGTase,深蓝色表示γ-CGTase;B:重合的关键氢键氨基酸在α-CGTase的具体氨基酸名称和位点,黑色字体表示氨基酸类型是一样的,紫色字体表示氨基酸类型不同. Note: A: Amino acid positional relationship between the amino acid at the same position and the decasaccharide chain. White indicates α-CGTase, light blue indicates β-CGTase, and dark blue indicates γ-CGTase. B: The key hydrogen-bonding amino acids that overlap are in α-CGTase. The specific amino acid name and position, the black font indicates that the amino acid types are the same, and the purple font indicates that the amino acid types are different. |
|
|
| No. | α-CGTase | β-CGTase | γ-CGTase |
| 1 | Y89 | F89 | H83 |
| 2 | H98 | H98 | H91 |
| 3 | Y100 | Y100 | Y93 |
| 4 | W101 | W101 | W94 |
| 5 | H140 | H140 | H133 |
| 6 | H169 | Y167 | Y154 |
| 7 | G182 | G180 | G167 |
| 8 | N195 | N193 | N180 |
| 9 | Y197 | Y195 | Y182 |
| 10 | D198 | D196 | D183 |
| 11 | D231 | D229 | D216 |
| 12 | K234 | K232 | A219 |
| 13 | H235 | H233 | H220 |
| 14 | E260 | E257 | E244 |
| 15 | Y262 | F259 | F246 |
| 16 | H330 | H327 | H313 |
| 17 | R378 | R375 | R361 |
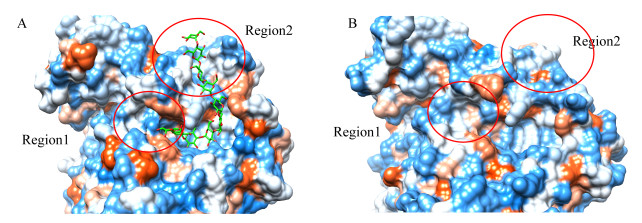
通过对α-CGTase与γ-CGTase亲疏水作用力的研究发现,在区域1和区域2部分两者的差别较大。α-CGTase相对于γ-CGTase的区域一来说,有一面亲水作用力较大的几个氨基酸组成了一面“亲水墙”。根据图 7和表 2分析得出,区域一位置的亲水位点分别对应的是K234和Y262。在表 2中,α-CGTase的K234和Y262分别对应的γ-CGTase位点为A219和F246。234位点在α-CGTase中的氨基酸为赖氨酸,属于碱性极性亲水氨基酸,在γ-CGTase该位置变为疏水性的丙氨酸;262位点在α-CGTase中的氨基酸为酪氨酸,也属于亲水氨基酸,在γ-CGTase该位置变为疏水性的苯丙氨酸。所以本实验预测如果将262位点从亲水性的酪氨酸变成疏水性的氨基酸,可能会对CGTase的产物方向有大的改变。
|
| 图 7 CGTase表面亲疏水作用图 Figure 7 Hydrophilic and hydrophobic effects on the surface of CGTase 注:A:α-CGTase的亲疏水作用;B:γ-CGTase的亲疏水作用;Region1和Region2表示α-CGTase与γ-CGTase亲疏水作用力差距较大的区域. Note: A: the hydrophilic and hydrophobic effects of α-CGTase; B: the hydrophobic and hydrophobic effects of γ-CGTase; the Region1 and Region2 show the regions with different affinity forces between α-CGTase and γ-CGTase. |
|
|
通过定点突变实验以Paenibacillus macerans sp. 602-1作为模版,将262位点亲水性的酪氨酸突变为疏水性的亮氨酸,突变体酶Y262L与野生菌酶的活性进行比较(表 3),发现Y262L相对于野生菌而言酶活性降低。经过高效液相色谱测得突变酶Y262L相较于野生酶总产量有所减少;其中产生α-CD的比例有明显降低,从原来的51.5%降低到16.7%,总体降低了2/3;野生菌Paenibacillus macerans sp. 602-1产生的γ-CD占总CD产量的比例为13.7% (质量比),而改造后的Y262L突变体产生的γ-CD占总CD产量的比例达到39.9% (质量比),突变体相对于野生菌产γ-CD的比例增加了3倍(表 4)。实验还研究了针对K234位点的定点突变,突变体酶K234A的α-、β-和γ-CD产物比为64:25:9 (质量比),酶活性与野生酶相比变化不大。Y89位点的突变酶已有文献报道[17-18],取代不同的氨基酸会导致产物专一性的变化较大。
| Mutants | Enzymatic activity (U/ml) | Protein concentration (mg/ml) | Specific activity (U/mg) |
| Wild | 4 856±24 | 0.547±0.014 | 8 289±43 |
| Y262L | 2 922±13* | 0.667±0.011 | 4 381±19* |
| 注:*:p < 0.01,与野生组进行对比,n=3. Note: *: p < 0.01, compared with the wild group, n=3. | |||
| 24 h | Mutants | Product percentage (%) | Yield (g) | ||
| α | β | γ | |||
| 1 | Wild | 51.5±1.2 | 34.8±1.4 | 13.7±0.3 | 3.64±0.11 |
| 2 | Y262L | 16.7±1.4* | 43.5±3.1 | 39.9±0.9* | 2.10±0.06* |
| 注:*:p < 0.01,与野生组进行对比,n=3. Note: *: p < 0.01, compared with the wild group, n=3. | |||||
Sonnendecker等[19-20]改变Bacillus sp. G-825-6菌的Y183位点和D358位点后产生的CD8−CD12的产率大大增加。然而Sonnendecker等[19-20]的Y183和D358对应的本文中4JCM的位点为Y182和D357,这两个氨基酸均与十糖链存在氢键作用力。经过表 1比对发现,Y182位点相对应改造后的α-CGTase和β-CGTase对应的是Y197和Y195,Chen等[17]通过改造Paenibacillus macerans的Y195位点可以产生更多的γ-CD。对于α-、β-和γ-CGTase来说,结构框架基本相同,无论是Sonnendecker等[19-20]改造Y183位点还是Chen等[17]改造Y195位点,都是处于不同类型CGTase的同一关键位置。这一结果表明,无论是否属于同一种CGTase,只要相对应改造的氨基酸处于同一关键位置,便会引起不同CGTase的酶产物专一性产生相近的变化。
本研究通过比较3种CGTase口袋附近与底物存在氢键连接的氨基酸,发现这些氨基酸对酶产物专一性有重要的影响。Li等[18]发现突变体Y89R有利于将α-CD产率从野生菌的41.8%提升到58.2%;Kim等[21]表明突变体Y89F和Y100F有利于产生β-CD;Leemhuis等[22]对位点Y167F、G179L、G180L、N193G、N193L和G179L/G180L做了研究,发现改造G180位点会引起环化反应和偶联反应,这些位点都与表 1中的相关位点对应,如G180和N193对应的是α-CGTase的G182和N195位点。根据本实验的分析得出对接口袋附近的这些关键位点对环糊精基糖转移酶的改造有重要意义,今后可以通过对这些关键位点进行理性改造,有可能会获得较为理想的结果。本研究对Y262位点进行定点突变和产物专一性实验,结果显示经过分子动力学预测的Y262L有助于提高产γ-CD专一性,从野生酶的13.7%提高到39.9%,γ-CD产物比例提高了3倍。研究证实了分子动力学模拟结果对于指导环糊精糖基转移酶的专一性内在机理具有正向的指导意义,所以如果需要CGTase进行更深催化机理的研究,分子动力学模拟将是重要的突破研究手段。
| [1] |
Szejtli J. Introduction and general overview of cyclodextrin chemistry[J]. Chemical Reviews, 1998, 98(5): 1743-1754. DOI:10.1021/cr970022c |
| [2] |
Loftsson T, Duchêne D. Cyclodextrins and their pharmaceutical applications[J]. International Journal of Pharmaceutics, 2007, 329(1/2): 1-11. |
| [3] |
Brewster ME, Vandecruys R, Verreck G, et al. Supersaturating drug delivery systems: Effect of hydrophilic cyclodextrins and other excipients on the formation and stabilization of supersaturated drug solutions[J]. Pharmazie, 2008, 63(3): 217-220. |
| [4] |
van der Veen BA, Uitdehaag JCM, Penninga D, et al. Rational design of cyclodextrin glycosyltransferase from Bacillus circulans strain 251 to increase α-cyclodextrin production[J]. Journal of Molecular Biology, 2000, 296(4): 1027-1038. DOI:10.1006/jmbi.2000.3528 |
| [5] |
Chen SD, Li ZF, Gu ZB, et al. Leu600 mutations decrease product inhibition of the β-cyclodextrin glycosyltransferase from Bacillus circulans STB01[J]. International Journal of Biological Macromolecules, 2018, 115: 1194-1201. DOI:10.1016/j.ijbiomac.2018.05.006 |
| [6] |
Ling K, Zhang HB, Liu PF, et al. Site-directed mutation of cyclodextrin glycosyltransferase and analysis of its product specificity[J]. Food Science, 2016, 37(17): 133-138. (in Chinese) 凌凯, 张洪斌, 刘鹏飞, 等. 环糊精糖基转移酶定点突变及其产物特异性分析[J]. 食品科学, 2016, 37(17): 133-138. DOI:10.7506/spkx1002-6630-201617022 |
| [7] |
Ishii N, Haga K, Yamane K, et al. Crystal structure of asparagine 233-replaced cyclodextrin glucanotransferase from alkalophilic Bacillus sp. 1011 determined at 1.9 Å resolution[J]. Journal of Molecular Recognition, 2000, 13(1): 35-43. DOI:10.1002/(SICI)1099-1352(200001/02)13:1<35::AID-JMR481>3.0.CO;2-J |
| [8] |
Madani SH, Sabour MH, Fadaee M. Molecular dynamics simulation of vibrational behavior of annular graphene sheet: Identification of nonlocal parameter[J]. Journal of Molecular Graphics and Modelling, 2018, 79: 264-272. DOI:10.1016/j.jmgm.2017.11.008 |
| [9] |
Xu XD, Cai L, Zheng XL, et al. Molecular dynamics simulations of solvent-exfoliation and stabilization of graphene with the assistance of compressed carbon dioxide and pyrene-polyethylene glycol[J]. Physical Chemistry Chemical Physics, 2017, 19(24): 16062-16070. DOI:10.1039/C7CP01277B |
| [10] |
Huang J, MacKerell AD Jr. Force field development and simulations of intrinsically disordered proteins[J]. Current Opinion in Structural Biology, 2018, 48: 40-48. DOI:10.1016/j.sbi.2017.10.008 |
| [11] |
Robustelli P, Piana S, Shaw DE. Developing a molecular dynamics force field for both folded and disordered protein states[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(21): E4758-E4766. DOI:10.1073/pnas.1800690115 |
| [12] |
Carvalho HF, Barbosa AJM, Roque ACA, et al. Integration of molecular dynamics based predictions into the optimization of De Novo protein designs: Limitations and Benefits[A]//Samish I. Computational Protein Design. Methods in Molecular Biology[M]. New York: Humana Press, 2017, 1529: 181-201
|
| [13] |
Huber RG, Marzinek JK, Holdbrook DA, et al. Multiscale molecular dynamics simulation approaches to the structure and dynamics of viruses[J]. Progress in Biophysics and Molecular Biology, 2017, 128: 121-132. DOI:10.1016/j.pbiomolbio.2016.09.010 |
| [14] |
Jia XB, Guo YH, Lin XJ, et al. Fusion of a family 20 carbohydrate-binding module (CBM20) with cyclodextrin glycosyltransferase of Geobacillus sp. CHB1 improves catalytic efficiency[J]. Journal of Basic Microbiology, 2017, 57(6): 471-480. DOI:10.1002/jobm.201600628 |
| [15] |
Lee YS, Woo JB, Ryu SI, et al. Glucosylation of flavonol and flavanones by Bacillus cyclodextrin glucosyltransferase to enhance their solubility and stability[J]. Food Chemistry, 2017, 229: 75-83. DOI:10.1016/j.foodchem.2017.02.057 |
| [16] |
Liao SM, Shen NK, Liang G, et al. Inhibition of α-amylase activity by Zn2+: Insights from spectroscopy and molecular dynamics simulations[J]. Medicinal Chemistry, 2019, 15(5): 510-520. DOI:10.2174/1573406415666181217114101 |
| [17] |
Chen FJ, Xie T, Yue Y, et al. Molecular dynamic analysis of mutant Y195I α-cyclodextrin glycosyltransferase with switched product specificity from α-cyclodextrin to γ-cyclodextrin[J]. Journal of Molecular Modeling, 2015, 21(8): 208. DOI:10.1007/s00894-015-2734-x |
| [18] |
Li ZF, Zhang JY, Wang M, et al. Mutations at subsite -3 in cyclodextrin glycosyltransferase from Paenibacillus macerans enhancing α-cyclodextrin specificity[J]. Applied Microbiology and Biotechnology, 2009, 83(3): 483-490. DOI:10.1007/s00253-009-1865-3 |
| [19] |
Sonnendecker C, Melzer S, Zimmermann W. Engineered cyclodextrin glucanotransferases from Bacillus sp. G-825-6 produce large-ring cyclodextrins with high specificity[J]. Microbiology Open, 2019, 8(6): e00757. |
| [20] |
Sonnendecker C, Wei R, Kurze E, et al. Efficient extracellular recombinant production and purification of a Bacillus cyclodextrin glucanotransferase in Escherichia coli[J]. Microbial Cell Factories, 2017, 16(1): 87. DOI:10.1186/s12934-017-0701-1 |
| [21] |
Kim YH, Bae KH, Kim TJ, et al. Effect on product specificity of cyclodextrin glycosyltransferase by site-directed mutagenesis[J]. Biochemistry and Molecular Biology International, 1997, 41(2): 227-234. |
| [22] |
Leemhuis H, Uitdehaag JCM, Rozeboom HJ, et al. The remote substrate binding subsite -6 in cyclodextrin- glycosyltransferase controls the transferase activity of the enzyme via an induced-fit mechanism[J]. Journal of Biological Chemistry, 2002, 277(2): 1113-1119. DOI:10.1074/jbc.M106667200 |
 2020, Vol. 47
2020, Vol. 47