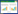扩展功能
文章信息
- 殷鑫欢, 鲁令华, 王进疆, 谢勇, 朱玲, 周莉媛, 陈弟诗, 徐志文
- YIN Xin-Huan, LU Ling-Hua, WANG Jin-Jiang, XIE Yong, ZHU Ling, ZHOU Li-Yuan, CHEN Di-Shi, XU Zhi-Wen
- 2018-2019年四川省伪狂犬病毒的流行病学调查及gC、gE、TK基因遗传进化分析
- Epidemiological investigation and gC, gE, TK genetic characteristics of pseudorabies virus in Sichuan province of China between 2018-2019
- 微生物学通报, 2020, 47(12): 4142-4154
- Microbiology China, 2020, 47(12): 4142-4154
- DOI: 10.13344/j.microbiol.china.191089
-
文章历史
- 收稿日期: 2019-12-30
- 接受日期: 2020-02-18
- 网络首发日期: 2020-03-24
伪狂犬病毒(pseudorabies virus,PRV)是伪狂犬病(pseudorabies,PR)的病原,是疱疹病毒(Herpeviriade) α疱疹病毒科(Alpha Herpesvirinae)的成员[1]。PRV基因组约为143 kb,包含至少72个编码蛋白的基因,这些蛋白参与了病毒衣壳、间质和包膜的形成[2-3]。其中胸苷激酶基因TK和糖蛋白E基因gE是PRV主要的毒力基因[4]。gE/TK基因缺失的毒株常被用于疫苗研发[5-8]。此外,TK和gE基因常被用于监测野毒株的遗传进化[1, 9-10]。糖蛋白C基因gC位于病毒包膜内,主要参与诱导机体细胞和体液免疫应答[11]。据报道,gC基因遗传进化分析可以将PRV毒株分为两种基因型,从而将中国毒株与国外毒株区分开[12]。
PRV可以感染多种动物,包括牛、啮齿动物、貂、狗等[13-15],2018−2019年间曾有报道关于从事畜牧行业的人员感染PRV后引起脑炎和眼炎的案例[16-17],但猪是该病毒的自然宿主[18-19]。感染了PRV的猪在外周神经系统中终生携带病毒,不同生长阶段的猪只感染PRV表现出不同的症状[20]。新生仔猪主要表现为神经症状和致死性脑炎;育肥猪只感染后以呼吸系统障碍为特征;而该病毒常常导致怀孕母猪流产,产木乃伊胎等[21]。中国首例PRV感染病例可以追溯到20世纪50年代,20世纪70年代中国从匈牙利引进了Bartha-K61疫苗,直到80年代Bartha-K61疫苗才在中国得到广泛应用,为疾病控制提供了有利条件[22]。然而2011年底,在部分Bartha-K61免疫的猪场暴发了以神经症状和新生仔猪高死亡率为特征的PR,并分离到多株流行毒株,包括HNX、ZJ01、HeN1、SMX、TJ、JS、JS-2012和HN1201等[21, 23-24]。遗传进化显示这些毒株与传统毒株相比存在变异。
PRV在四川省内的流行情况较少报道,为了了解四川省PRV流行毒株的流行特征,本研究对四川省2018年1月至2019年8月的PR流行病学进行了调查,共分离到3株PRV毒株,并对这些毒株的gC、gE和TK基因序列进行扩增和测序,构建遗传进化树。以本实验室保存的2016年PRV分离株(PRV-XJ株)为参考毒株,分析近年来四川省PRV毒株的流行变异情况。
1 材料 1.1 样品采集及处理2018−2019年本实验室收集了四川省12个地区86个养猪场384头以流产、神经系统疾病、呼吸系统疾病为特征的猪只组织样本。其中,春、夏、秋、冬收集的疑似阳性样品数目分别是33、130、127、94份;与呼吸系统、神经系统、母猪流产及死胎、公猪生殖系统感染相关的病例分别有67、35、84、198份。死胎及神经症状猪只采集其小脑,流产胎儿采集胎衣,呼吸系统障碍猪只采集其肺脏,种公猪通过采集精液处理样品中的蛋白。
1.2 主要试剂和仪器2×GC buffer、LA Taq® Hot Start Version、DL2000 DNA Marker、反转录试剂盒(PrimeScriptTM RT Reagent Kit),Takara公司;病毒基因组DNA/RNA提取试剂盒(TIANamp Virus DNA/RNA Kit),天根生化科技(北京)有限公司;SanPrep柱式DNA胶回收试剂盒,生工生物工程(上海)股份有限公司;胎牛血清、DMEM (Dulbecco’s Modified Eagle Medium)、营养液,Gibco公司;BHK-21 (Baby Hamster Kidney Cell-21)细胞,本实验室保存。PCR仪、凝胶成像系统,Bio-Rad公司。
2 方法 2.1 PRV的检测对384头患病猪只的小脑、肺脏、胎衣等组织研磨,液氮反复冻融3次后12 000 r/min离心5 min收集上清;公猪精液首先通过破乳去除过多的蛋白然后12 000 r/min离心5 min收集上清。根据传统的酚-氯仿法抽提组织中的DNA,参照国家PRV-gE基因的检测引物gE1-F/R (表 1)对抽提的DNA样品进行PCR扩增。PCR反应体系(10 μL):2×GC buffer 5 μL,ddH2O 2 μL,正、反向引物(100 μmol/L)各0.5 μL,dNTPs (2.5 mmol/L) 1 μL,Taqhas DNA聚合酶(5 U/μL) 0.1 μL,DNA模板0.9 μL。PCR反应条件:95 ℃ min;95 ℃ 30 s,60 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 7 min。同时按照Trizol法抽提组织中的RNA,用PrimeScriptTM RT Reagent Kit合成cDNA。同时利用PCR和RT-PCR技术对常见的猪瘟病毒(classical swine fever virus,CSFV)、猪繁殖与呼吸障碍病毒(porcine reproductive and respiratory syndrome virus,PRRSV)、猪细小病毒(porcine parvovirus,PPV)、猪圆环病毒2型(porcine circovirus 2,PCV2)、猪圆环病毒3型(porcine circovirus 3,PCV3)病原进行检测,引物见表 1。PCR反应结束后,取5 μL产物进行1%的琼脂糖凝胶电泳,最终若PRV-gE的产物条带约534 bp,则证明是PRV阳性感染猪只。
| 序号 No. |
引物名称 Primers name |
引物序列 Primers sequence (5′→3′) |
片段长度 Fragments (bp) |
功能 Effects |
| 1 | PCV2-F | CGGATATTGTAGTCCTGGTCG | 481 | 样品检测Sample detection |
| PCV2-R | ACTGTCAAGGCTACCACAGTCA | |||
| 2 | PCV3-F | TACCGGGCAGTGGATGATGAAG | 639 | 样品检测Sample detection |
| PCV3-R | CGCAACCTGGAGGACCAATAAA | |||
| 3 | CSFV-F | GACACTAGYGCAGGCAAYAG | 449 | 样品检测Sample detection |
| CSFV-R | AGTGGGTTCCAGGARTACAT | |||
| 4 | PRRSV-F | AATTCCCGCACCTCGCGGAACTGT | 260 | 样品检测Sample detection |
| PRRSV-R | ATGTCCGAGATTTCACTCAGG | |||
| 5 | PPV-F | AACACACTTCCATACACACCAGCAG | 380 | 样品检测Sample detection |
| PPV-R | CTAGTATAATTTTCTTGG | |||
| 6 | gE1-F | ATCTGGACGTTCCTGCCC | 534 | 样品检测Sample detection (GB/T 18641-2002) |
| gE1-R | GTAGATGCAGGGCTCGTACA | |||
| 7 | gE2-F | CCTGGGCACCCCCGCGAGTCTCGCA | 896 | 扩增N端Amplifying the N-terminal of gE |
| gE2-R | TGCAGGGCTCGTACACGTAGTACA | |||
| 8 | gE3-F | CCGCCACGCTGGACTGGTACTACGC | 1 001 | 扩增C端Amplifying the C-terminal of gE |
| gE3-R | GCCGGGCATGTCGGAATGCGGGCG | |||
| 9 | gC1-F | CACTAGCATTAAATCCGTTTCCTGA | 952 | 扩增N端Amplifying the N-terminal of gC |
| gC2-R | GTCCACCGGGTGCTCGTCCGCGAA | |||
| 10 | gC2-F | GTGTGCGTCGTCCGCGACTACTAC | 739 | 扩增C端Amplifying the C-terminal of gC |
| gC2-R | ACCGGACGCGATGGCTCGGTTCA | |||
| 11 | TK-F | CGGCGCACCCCGAGGTTGACTTCAA | 1 278 | 扩增全基因Amplifying the whole TK gene |
| TK-R | CACGGACGACGCGGGCATGGTGAC |
取1 mL的组织研磨液上清加入青链霉素(100 μg/ml)和左氧氟杀星(100 μg/mL) 4 ℃过夜,将BHK-21细胞传代至T25细胞瓶,当细胞密集程度达到90%时将上清接种单层细胞。37 ℃吸附1 h后加入含2% FBS的DMEM维持液,置于37 ℃ 5% CO2的培养箱中培养3−4 d,观察细胞病变(cytopathic effect,CPE)。当细胞达到80% CPE时,置于−80 ℃反复冻融3次,5 000 r/min离心10 min收集病毒液。
2.3 gC、gE、TK基因的扩增和序列分析根据病毒DNA/RNA抽提试剂盒提取PRV基因DNA,利用NCBI的primerblast (https://www.ncbi.nlm.nih.gov/tools/primer-blast/)功能区设计针对gC、gE、TK基因的引物,由生工生物工程(上海)股份有限公司合成(表 1)。参照2.1的反应体系扩增病毒gC、gE、TK基因的编码区(coding sequence,CDS),将PCR产物通过SanPrep柱式DNA胶回收试剂盒回收,并送至北京擎科生物科技有限公司进行测序。利用DNAStar 7.1软件拼接病毒gC、gE、TK基因核苷酸序列并上传至Genbank获得相应的登录号(表 2)。从Genbank数据库下载已公布的PRV毒株序列作为参考序列(表 2),最后通过MEGAX软件,建立针对gC、gE、TK基因的遗传进化树。
| 分离株 Isolates |
上传年份 Submission year to NCBI |
国家 Country |
登录号 Genbank accession No. |
| Becker | − | USA | JF797219.1 (complete genome) |
| Kaplan | − | Hungary | JF797218.1 (complete genome) |
| Bartha | − | Hungary | JF797217.1 (complete genome) |
| NIA3 | − | United Kingdom | KU900059.1 (complete genome) |
| SC | 1986 | China | KT809429.1 (complete genome) |
| Ea | 1993 | China/Hubei | KX423960.1 (complete genome) |
| LA | 1997 | China | KU552118.1 (complete genome) |
| Kolchis | 2010 | Greece | KT983811.1 (complete genome) |
| Fa | 2012 | China/ Hubei | KM189913.1 (complete genome) |
| JS-2012 | 2012 | China/Jiangsu | KP257591.1 (complete genome) |
| HeN1 | 2012 | China/Henan | KP098534.1 (complete genome) |
| ZJ01 | 2012 | China/Zhejiang | KM061380.1 (complete genome) |
| TJ | 2012 | China/Tianjin | KJ789182.1 (complete genome) |
| HNX | 2012 | China/Henan | KM189912.1 (complete genome) |
| HNB | 2012 | China/Henan | KM189914.3 (complete genome) |
| HN1201 | 2012 | China/Henan | KP722022.1 (complete genome) |
| Xiang A | 2012 | China/Hunan | KF711991.1 (TK); KJ463839.1 (gE) |
| GX-GL | 2013 | China/Guangxi | KU323910.1 (gC); KT936471.1 (gE) |
| LN10-13 | 2013 | China/Liaoning | KY398785.1 (gE); KY398814.1 (TK) |
| BJ/RD | 2013 | China/Beijing | KF017273.1 (gC); KF017275.1 (gE) |
| DL/1408 | 2014 | China/Jilin | KU360259.1 (complete genome) |
| YY | 2014 | China/Hunan | KF997101.1 (gC); KF130885.1 (gE); KP259815.1 (TK) |
| FJN5-14 | 2014 | China/Fujian | KY398743.1 (gC); KY398773.1 (gE); KY398807.1 (TK) |
| XJ-14 | 2014 | China/Shandong | KY398767.1 (gC); KY398801.1 (gE); KY398830.1 (TK) |
| HLJ8 | 2015 | China/Heilongjiang | KT824771.1 (complete genome) |
| RC1 | 2016 | Japan | LC342744.1 (complete genome) |
| XJ | 2016 | China/Sichuan | KY498321 (gC); KY498323 (gE); MN255325 (TK) |
| JL1 | 2017 | China/Jilin | MG733280.1 (gE); MG733282.1 (TK) |
| SD HZ01 | 2018 | China/Shandong | MH521042.1 (gE) |
| DJY | 2018 | China/Sichuan | MN240561 (gC); MN240564 (gE); MN255324 (TK) |
| SN | 2018 | China/Sichuan | MN240562 (gC); MN240565 (gE); MN262476 (TK) |
| CD | 2019 | China/Sichuan | MN240560 (gC); MN240563 (gE); MN240566 (TK) |
| 注:−:时间不详. Note: −: The date is not definite. | |||
使用GraphPad Prism 8.0和IBM SPSS Statistics,V22.0软件对数据进行统计学分析。利用DNAStar 7.1和MEGAX (https://www.megasoftware.net/)软件进行序列及遗传进化分析。PRV感染与猪只临床症状的相关性采用卡方测试(χ2=∑[(实际观测数−理论频数)2/理论频数]),所有分析的显著性水平为5%,置信区间设置为95%。
3 结果与分析 3.1 2018−2019 PRV的流行病学调查对2018−2019年四川省12个地区86个猪场198头种猪、84头流产胎儿、67头呼吸系统病猪、35头神经系统病猪样品进行了PCR检测,具体的临床症状及病理观察见图 1。最终PRV个体阳性率为9.9% (38/384);阳性猪场比率为16.3% (14/86);流产胎儿中PRV的阳性率为32.1% (27/84);种公猪的阳性率为2.0% (4/198);神经系统感染阳性率为11.4% (4/35);呼吸系统感染阳性率为4.5% (3/67) (图 2A)。所有的阳性病例中有一例流产是由PRRSV和PRV混合感染引起的,其余阳性病例均为PRV单一感染。经统计学分析,PRV感染与母猪和公猪生殖系统相关(P < 0.01),而呼吸系统和神经系统感染与PRV无明显相关性(P > 0.05) (表 3)。

|
| 图 1 变异PRV毒株感染猪只后的临床症状及病理观察 Figure 1 Clinical signs and pathological observation in pigs infected by variant PRV 注:A:流产胎儿;B:木乃伊胎;C:神经症状的仔猪大脑;D:肾脏表面出血点;E/F:肝脏和脾脏表面局灶性坏死. Note: A: Stillborn foetus; B: Mummified fetus; C: Cerebral hemorrhage; D: Hemorrhagic spots on renal surface; E/F: Small white focal areas of necrosis on liver and spleen. |
|
|

|
| 图 2 不同感染症状猪只(A)及不同季节(B)的PRV阳性率 Figure 2 PRV positive rates in pigs with different infection symptoms (A) and different seasons (B) |
|
|
PRV感染率最高的季节是冬季(12月、1月、2月),阳性率为33.0% (31/94),其次是春季(3月、4月、5月),阳性率为9.1% (3/33)。夏季(6月、7月、8月)和秋季(9月、10月、11月)感染较低,分别为1.5% (2/130)和1.6% (2/127) (图 2B)。
| Variables | PRV (n=384) | P value | OR | 95% CI | ||
| Positive (n=38) | Negative (n=346) | |||||
| Reproductive system of sows | RSS | 27 | 57 | 0.000 | 12.445 | 5.841−26.516 |
| NRSS | 11 | 289 | ||||
| Reproductive system of boars | RSB | 4 | 194 | 0.000 | 0.092 | 0.032−0.265 |
| NRSB | 34 | 152 | ||||
| Respiratory system | RS | 3 | 64 | 0.103 | 0.378 | 0.113−1.266 |
| NRS | 35 | 282 | ||||
| Neurological system | RES | 4 | 31 | 0.750 | 1.195 | 0.398−3.590 |
| NRES | 34 | 315 | ||||
| 注:OR:比值比;CI:置信区间;RSS:母猪生殖系统;RSB:公猪生殖系统;RS:呼吸系统;NES:神经系统. Note: OR: Odds ratio; CI: Confidence interval; RSS: Reproductive system of sows; RSB: Reproductive system of boars; RS: Respiratory system; NES: Neurological system. | ||||||
对照组的BHK-21细胞没有出现CPE (图 3A),而病毒感染BHK-21细胞24 h后,CPE明显,视野中大多数细胞变圆(图 3B)。抽提病毒液的基因组DNA,利用PRV-gE的检测引物gE1-F/R (表 1)进行PCR扩增,结果显示阳性样品均可扩增到长度约为534 bp (图 3C)的条带。随后使用gE的全基因引物gE2-F/R和gE3-F/R (表 1)进行PCR扩增并测序,将序列上传至NCBI数据库进行核甘酸BLAST,结果显示与PRV基因组相似性为100%,证实分离的病毒是PRV。2018−2019年共分离到3株PRV毒株,分别命名为PRV-SN、PRV-CD、PRV-DJY。

|
| 图 3 BHK-21细胞中PRV的分离 Figure 3 Isolation of PRV in BHK-21 cells 注:A:BHK-21细胞对照组;B:PRV接种BHK-21细胞24 h后的CPE;C:PRV-gE的PCR鉴定;M:DL2000 DNA marker;1、2、3:分离的毒株;+:阳性对照;−:阴性对照. Note: A: No CPE appeared in control BHK-21 cells; B: Obvious CPE appeared in BHK-21 cells 24 h post inoculation; C: PCR identification of PRV-gE gene; M: DL2000 DNA marker; 1, 2, 3: Isolated viruses; +: Positive control; −: Negative control. |
|
|
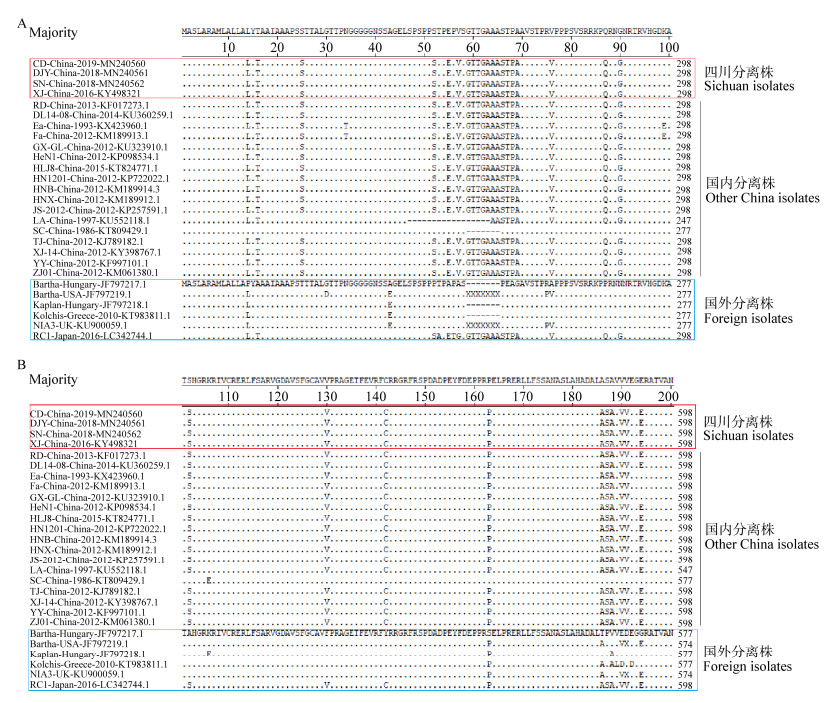
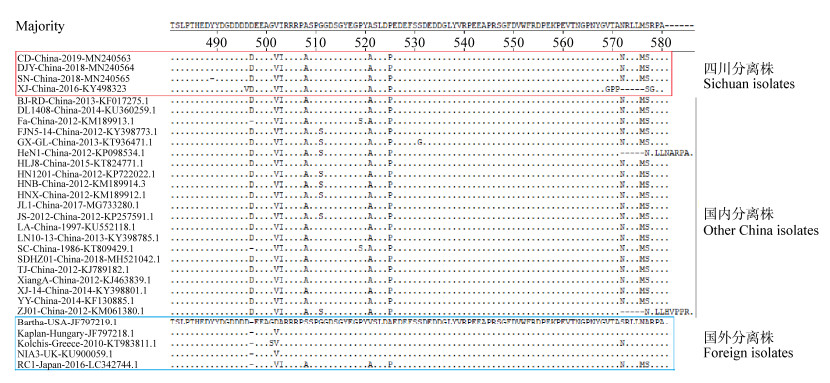
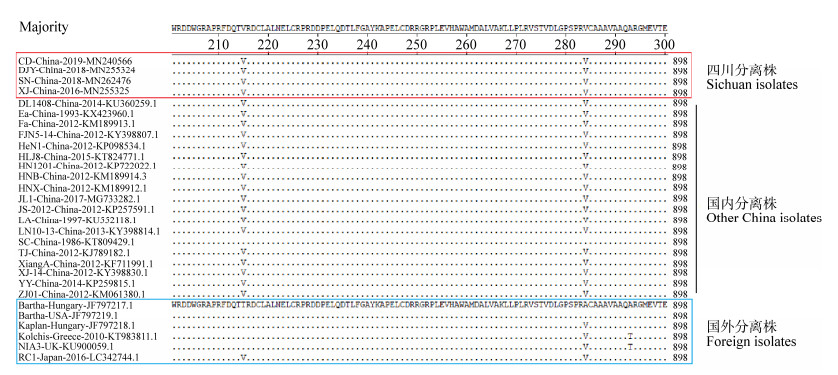
测序结果表明gC、gE、TK基因的CDS全长分别为1 464、1 740和963 bp,将分离的病毒核苷酸序列分别上传至Genbank并获得相应的登录号(表 2)。gC基因核苷酸序列分析发现PRV-SN、PRV-DJY、PRV-CD和国内分离株亲缘性很近,并无额外的缺失或突变位点(图 4)。gE基因核苷酸序列分析显示PRV-SN株全长为1 737 bp,其在第489位点存在一个氨基酸(amino acid,aa)的缺失(Y→−)。PRV-XJ是本实验室2016年在四川地区分离到的一株变异毒株,除特定位点的突变外,PRV-XJ在第252 (D→H)、282 (E→K)、496 (D→V)位点均存在aa的替换。此外,PRV-XJ与ZJ01和HeN1毒株在572−576 aa处都存在一个连续的缺失(图 5)。从TK基因的序列比对结果可以看出,中国毒株与国外毒株相比出现了一些点突变,如215 (T→V)、284 (A→V) (图 6)。总体上,TK基因相对于gC和gE基因更加保守。
|
| 图 4 四川毒株与GenBank数据库中其他PRV毒株gC氨基酸序列的比对 Figure 4 Comparison of PRV gC amino acid sequence with other PRV strains available in the GenBank database 注:L:亮氨酸;T:苏氨酸;S:丝氨酸;R:精氨酸;H:组氨酸;A:丙氨酸;V:缬氨酸;Q:谷氨酰胺;G:甘氨酸;C:半胱氨酸;E:谷氨酸;P:脯氨酸;I:异亮氨酸;M:甲硫氨酸;D:天冬氨酸. Note: L: Leucine; T: Threonine; S: Serine; R: Arginine; H: Histidine; A: Alanine; V: Valine; Q: Glutamine; G: Glycine; C: Cysteine; E: Glutamic acid; P: Proline; I: Isoleucine; M: Methionine; D: Aspartic acid. |
|
|
|
| 图 5 四川毒株与GenBank数据库中其他PRV毒株gE氨基酸序列的比对 Figure 5 Comparison of PRV gE amino acid sequence with other PRV strains available in the GenBank database 注:L:亮氨酸;T:苏氨酸;S:丝氨酸;R:精氨酸;H:组氨酸;A:丙氨酸;V:缬氨酸;Q:谷氨酰胺;N:天冬酰胺;P:脯氨酸;I:异亮氨酸;M:甲硫氨酸;D:天冬氨酸. Note: L: Leucine; T: Threonine; S: Serine; R: Arginine; H: Histidine; A: Alanine; V: Valine; Q: Glutamine; N: Asparagine; P: Proline; I: Isoleucine; M: Methionine; D: Aspartic acid. |
|
|
|
| 图 6 四川毒株与GenBank数据库中其他PRV毒株TK氨基酸序列的比对 Figure 6 Comparison of PRV TK amino acid sequence with other PRV strains available in the GenBank database 注:A:丙氨酸;T:苏氨酸;V:缬氨酸. Note: A: Alanine; T: Threonine; V: Valine. |
|
|
gC、gE、TK等基因的遗传进化分析显示,四川地区的PRV毒株处于同一分支上(图 7),与国外分离株有明显区别。研究表明,gC基因序列分析可以将PRV毒株分为两个基因型,中国毒株属于基因Ⅱ型,而国外毒株Bartha、Becker、NIA3、Kaplan、Kolchis等属于基因Ⅰ型[10]。本研究中四川的4个毒株均属于基因Ⅱ型(图 7A)。与此同时,gE基因(图 7B)和TK基因(图 7C)的遗传进化分析结果类似。然而基于TK基因建立的进化树,Bartha、Becker、Kaplan与中国毒株属于同一分支,NIA3与Kolchis属于单独的分支。
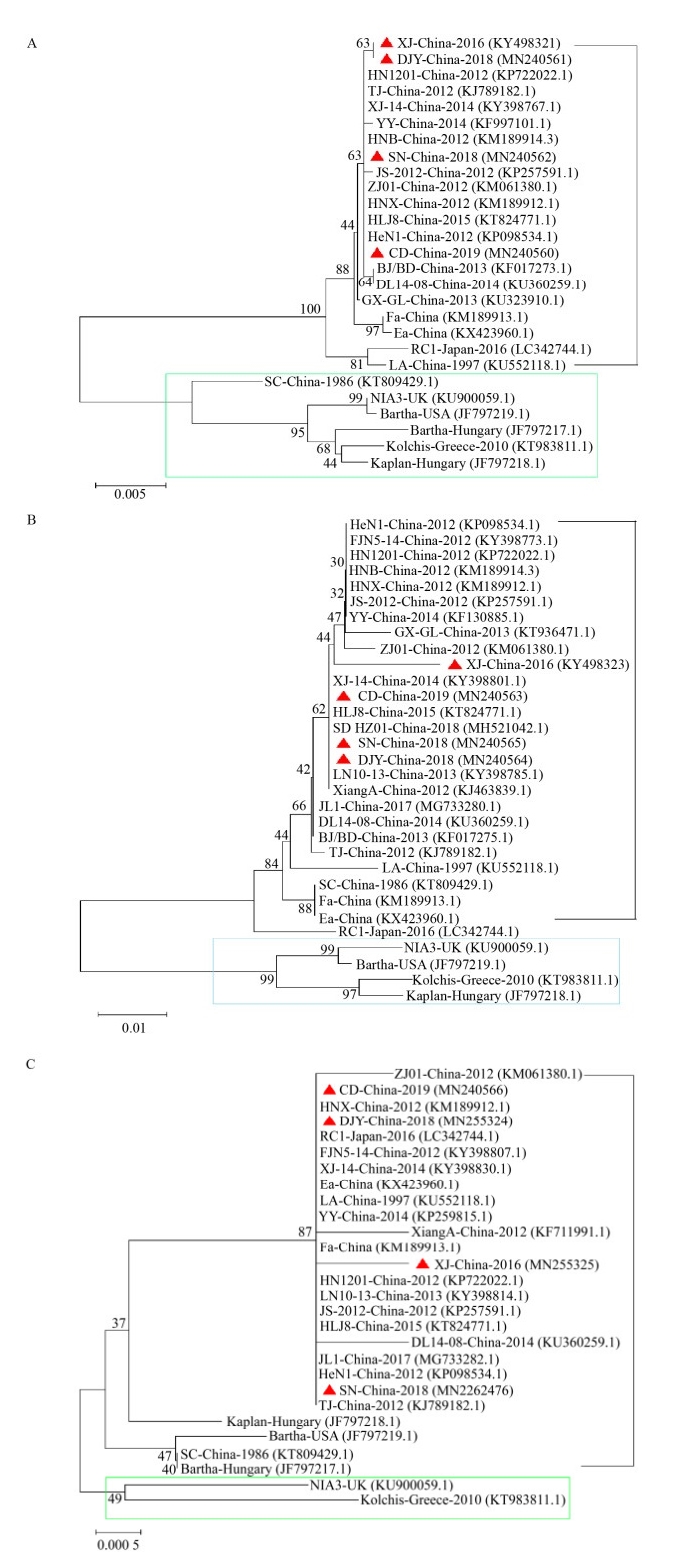
|
| 图 7 基于gC(A)、gE(B)和TK(C)的PRV分离株遗传进化分析 Figure 7 Phylogenetic analysis of PRV isolates based on gC (A), gE (B) and TK (C) 注:图中对分离的毒株进行了标记,且对其进行分区. Note: The isolated strains are labeled and artificially partitioned. |
|
|
PRV是危害养猪生产的重要病原,传统认为Bartha-K61对该病的防控是必不可少的[25]。2011年以来,病毒的遗传进化和变异降低了疫苗的保护效率[26-28]。然而,zhou等[22]将Bartha-K61免疫育肥阶段的猪后,利用分离的PRV-XJ5变异毒株进行攻毒试验,发现Bartha-K61可以刺激育肥猪只产生足够的免疫保护,从而抵抗变异毒株的感染。2018年Sun等调查了2012−2017年期间中国27个省份的PRV分布,发现全国PRV的阳性率平均为8.27%[10],而在我们的研究中,四川省PRV的阳性率略高于他们。本研究针对PRV的gE基因设计引物,对成都、绵阳、德阳、广元、南充、巴中、遂宁、雅安、乐山、宜宾、眉山、内江等12个地区86个大型养殖场的384份组织样本进行了检测,阳性率约为9.9% (38/384)。在所有地区中,成都地区阳性样本最多,阳性率为12.3% (15/122),其中PRV-XJ、PRV-DJY、PRV-CD均在成都分离。我们发现PRV在春季、冬季更容易暴发,其中冬春季节检出的阳性大都是因母猪产木乃伊胎、死胎以及新生仔猪脑膜炎引起的。在检测的所有流产性案例中,有32.1% (27/84)都是由PRV感染引起的,仅有一例是PRV和PRRSV混合感染。统计学分析显示种猪生殖障碍与PRV感染之间存在着相关性(P < 0.01),因此养殖场应该定期监测妊娠母猪和种公猪体内gB、gE抗体水平,适当地加强免疫并及时淘汰阳性猪只进而净化猪群。表现为神经症状的猪只随着生长日龄的增加,PRV阳性的概率逐渐降低,在我们检测的35份病料中仅有4例是PRV阳性,且都是1周龄以内的仔猪,而其余的保育猪、育肥猪大都是由链球菌、金黄色葡萄球菌单一或混合感染引起的,因此养殖场应该加强猪群环境的消毒。由于2019年非洲猪瘟病毒(African swine fever virus,ASFV)传入中国,可采集的猪只组织样本量较少,并且PRV的检出率较低,本实验室2019年仅仅分离到PRV-CD这一株PRV毒株。
基于gC、gE、TK等基因的遗传进化分析发现,四川株与近几年国内的分离株相似,但与国外毒株亲缘性较远。gC蛋白是PRV的重要中和抗原,在病毒进入细胞的过程中起重要作用[29]。与其他国内毒株一样,四川株gC蛋白在63−69 aa位点存在一个VSGTTGA的插入[10]。这个区域正是gC的B细胞抗原线性表位,2019年Zhang等[30]鉴定了134−138 aa、143−153 aa也都是gC重要的B细胞抗原表位。gE是PRV的主要毒力基因,但对病毒复制不是必需的,gE蛋白的抗原表位区位于52−238 aa[23]。与国外毒株相比,中国株在该区域有13个氨基酸突变,而PRV-SN株在第489位点处还存在一个额外的aa缺失。ZJ01毒株具有高致病性,可引起14日龄仔猪和80日龄猪只多脏器病变,死亡率为100%[7]。考虑到PRV-XJ和PRV-ZJ01的相似性,我们正在尝试构建基于PRV-XJ的候选疫苗[31]。TK蛋白是一种酶,参与病毒在中枢神经系统中的复制和传播[32]。序列分析发现TK蛋白的氨基酸突变较少,说明TK基因在一定程度上比较保守。综上,四川流行的PRV仍属于变异毒株,因此筛选具有代表性的变异毒株并在此基础上研发新的基因缺失疫苗十分必要。
| [1] |
Zhou MM, Wu XJ, Jiang DD, et al. Characterization of a moderately pathogenic pseudorabies virus variant isolated in China, 2014[J]. Infection, Genetics and Evolution, 2019, 68: 161-171. DOI:10.1016/j.meegid.2018.12.016 |
| [2] |
Nauwynck H, Glorieux S, Favoreel H, et al. Cell biological and molecular characteristics of pseudorabies virus infections in cell cultures and in pigs with emphasis on the respiratory tract[J]. Veterinary Research, 2007, 38(2): 229-241. DOI:10.1051/vetres:200661 |
| [3] |
Pomeranz LE, Reynolds AE, Hengartner CJ. Molecular biology of pseudorabies virus: impact on neurovirology and veterinary medicine[J]. Microbiology and Molecular Biology Reviews, 2005, 69(3): 462-500. DOI:10.1128/MMBR.69.3.462-500.2005 |
| [4] |
Wang JC, Song ZC, Ge AM, et al. Safety and immunogenicity of an attenuated Chinese pseudorabies variant by dual deletion of TK & gE genes[J]. BMC Veterinary Research, 2018, 14: 287. DOI:10.1186/s12917-018-1536-7 |
| [5] |
Zhang CL, Guo LH, Jia XR, et al. Construction of a triple gene-deleted Chinese pseudorabies virus variant and its efficacy study as a vaccine candidate on suckling piglets[J]. Vaccine, 2015, 33(21): 2432-2437. DOI:10.1016/j.vaccine.2015.03.094 |
| [6] |
Cong X, Lei JL, Xia SL, et al. Pathogenicity and immunogenicity of a gE/gI/TK gene-deleted pseudorabies virus variant in susceptible animals[J]. Veterinary Microbiology, 2016, 182: 170-177. DOI:10.1016/j.vetmic.2015.11.022 |
| [7] |
Dong J, Bai J, Sun T, et al. Comparative pathogenicity and immunogenicity of triple and double gene-deletion pseudorabies virus vaccine candidates[J]. Research in Veterinary Science, 2017, 115: 17-23. DOI:10.1016/j.rvsc.2017.01.011 |
| [8] |
Hu RM, Zhou Q, Song WB, et al. Novel pseudorabies virus variant with defects in TK, gE and gI protects growing pigs against lethal challenge[J]. Vaccine, 2015, 33(43): 5733-5740. DOI:10.1016/j.vaccine.2015.09.066 |
| [9] |
Sozzi E, Moreno A, Lelli D, et al. Genomic characterization of pseudorabies virus strains isolated in Italy[J]. Transboundary and Emerging Diseases, 2014, 61(4): 334-340. DOI:10.1111/tbed.12038 |
| [10] |
Sun Y, Liang W, Liu QY, et al. Epidemiological and genetic characteristics of swine pseudorabies virus in mainland China between 2012 and 2017[J]. PeerJ, 2018, 6: e5785. DOI:10.7717/peerj.5785 |
| [11] |
Ober BT, Teufel B, Wiesmuller KH, et al. The porcine humoral immune response against pseudorabies virus specifically targets attachment sites on glycoprotein gC[J]. Journal of Virology, 2000, 74(4): 1752-1760. DOI:10.1128/JVI.74.4.1752-1760.2000 |
| [12] |
Ye C, Zhang QZ, Tian ZJ, et al. Genomic characterization of emergent pseudorabies virus in China reveals marked sequence divergence: evidence for the existence of two major genotypes[J]. Virology, 2015, 483: 32-43. DOI:10.1016/j.virol.2015.04.013 |
| [13] |
Cheng ZL, Kong ZJ, Liu P, et al. Natural infection of a variant pseudorabies virus leads to bovine death in China[J]. Transboundary and Emerging Diseases, 2019. DOI:10.1111/tbed.13427 |
| [14] |
Wang GS, Du YJ, Wu JQ, et al. Vaccine resistant pseudorabies virus causes mink infection in China[J]. BMC Veterinary Research, 2018, 14(1): 20. DOI:10.1186/s12917-018-1334-2 |
| [15] |
Laval K, Vernejoul JB, van Cleemput J, et al. Virulent pseudorabies virus infection induces a specific and lethal systemic inflammatory response in mice[J]. Journal of Virology, 2018, 92(24): e01614-18. |
| [16] |
Wang YW, Nian H, Li ZW, et al. Human encephalitis complicated with bilateral acute retinal necrosis associated with pseudorabies virus infection: a case report[J]. International Journal of Infectious Diseases, 2019, 89: 51-54. DOI:10.1016/j.ijid.2019.09.019 |
| [17] |
Yang HN, Han H, Wang H, et al. A case of human viral encephalitis caused by pseudorabies virus infection in China[J]. Frontiers in Neurology, 2019, 10: 534. DOI:10.3389/fneur.2019.00534 |
| [18] |
Klupp BG, Lomniczi B, Visser N, et al. Mutations affecting the UL21 gene contribute to avirulence of pseudorabies virus vaccine strain Bartha[J]. Virology, 1995, 212(2): 466-473. DOI:10.1006/viro.1995.1504 |
| [19] |
Müller T, Hahn EC, Tottewitz F, et al. Pseudorabies virus in wild swine: a global perspective[J]. Archives of Virology, 2011, 156(10): 1691-1705. DOI:10.1007/s00705-011-1080-2 |
| [20] |
Tong W, Liu F, Zheng H, et al. Emergence of a pseudorabies virus variant with increased virulence to piglets[J]. Veterinary Microbiology, 2015, 181(3/4): 236-240. |
| [21] |
Gu J, Hu D, Peng T, et al. Epidemiological investigation of pseudorabies in Shandong province from 2013 to 2016[J]. Transboundary and Emerging Diseases, 2018, 65(3): 890-898. DOI:10.1111/tbed.12827 |
| [22] |
Zhou JZ, Li S, Wang XB, et al. Bartha-K61 vaccine protects growing pigs against challenge with an emerging variant pseudorabies virus[J]. Vaccine, 2017, 35(8): 1161-1166. DOI:10.1016/j.vaccine.2017.01.003 |
| [23] |
Wang YB, Qiao SL, Li XW, et al. Molecular epidemiology of outbreak-associated pseudorabies virus (PRV) strains in central China[J]. Virus Genes, 2015, 50(3): 401-409. DOI:10.1007/s11262-015-1190-0 |
| [24] |
Yu ZQ, Tong W, Zheng H, et al. Variations in glycoprotein B contribute to immunogenic difference between PRV variant JS-2012 and Bartha-K61[J]. Veterinary Microbiology, 2017, 208: 97-105. DOI:10.1016/j.vetmic.2017.07.019 |
| [25] |
An TQ, Peng JM, Tian ZJ, et al. Pseudorabies virus variant in Bartha-K61-vaccinated pigs, China, 2012[J]. Emerging Infectious Diseases, 2013, 19(11): 1749-1755. DOI:10.3201/eid1911.130177 |
| [26] |
Luo YZ, Li N, Cong X, et al. Pathogenicity and genomic characterization of a pseudorabies virus variant isolated from Bartha-K61-vaccinated swine population in China[J]. Veterinary Microbiology, 2014, 174(1/2): 107-115. |
| [27] |
Wu R, Bai CY, Sun JZ, et al. Emergence of virulent pseudorabies virus infection in northern China[J]. Journal of Veterinary Science, 2013, 14(3): 363-365. DOI:10.4142/jvs.2013.14.3.363 |
| [28] |
Yu XL, Zhou Z, Hu DM, et al. Pathogenic pseudorabies virus, China, 2012[J]. Emerging Infectious Diseases, 2014, 20(1): 102-104. DOI:10.3201/eid2001.130531 |
| [29] |
Karger A, Schmidt J, Mettenleiter TC. Infectivity of a pseudorabies virus mutant lacking attachment glycoproteins C and D[J]. Journal of Virology, 1998, 72(9): 7341-7348. DOI:10.1128/JVI.72.9.7341-7348.1998 |
| [30] |
Zhang PP, Lv L, Sun HF, et al. Identification of linear B cell epitope on gB, gC, and gE proteins of porcine pseudorabies virus using monoclonal antibodies[J]. Veterinary Microbiology, 2019, 234: 83-91. DOI:10.1016/j.vetmic.2019.05.013 |
| [31] |
Mao XY, Zhou XK, Yin XH, et al. Construction of recombinant pseudorabies virus strain expressing PEDV S and PoRV VP7 protein[J]. Microbiology China, 2019, 46(12): 3345-3354. (in Chinese) 毛汐语, 周雪珂, 殷鑫欢, 等. 表达PEDV S和PoRV VP7蛋白的重组伪狂犬毒株构建[J]. 微生物学通报, 2019, 46(12): 3345-3354. |
| [32] |
Ferrari M, Gualandi GL, Corradi A, et al. Experimental infection of pigs with a thymidine kinase negative strain of pseudorabies virus[J]. Comparative Immunology, Microbiology and Infectious Diseases, 1998, 21(4): 291-303. DOI:10.1016/S0147-9571(98)00012-5 |
 2020, Vol. 47
2020, Vol. 47