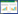扩展功能
文章信息
- 张龙, 曹梅, 孙慕溱, 汪小桐, 孔娜娜, 肖林林, 魏取好
- ZHANG Long, CAO Mei, SUN Mu-Zhen, WANG Xiao-Tong, KONG Na-Na, XIAO Lin-Lin, WEI Qu-Hao
- 细菌染色体第1类整合子捕获耐药性基因盒反应模型的构建
- Construction of reaction model for class 1 integron capturing antibiotic resistance gene cassettes on chromosome of bacteria
- 微生物学通报, 2020, 47(11): 3768-3776
- Microbiology China, 2020, 47(11): 3768-3776
- DOI: 10.13344/j.microbiol.china.191034
-
文章历史
- 收稿日期: 2020-05-15
- 接受日期: 2020-09-03
- 网络首发日期: 2020-09-09
2. 上海市奉贤区中心医院检验科 上海 201499
2. Department of Laboratory Medicine, Fengxian District Central Hospital, Shanghai 201499, China
随着抗菌药物在医疗、农业及水产养殖业的广泛使用,形成了抗菌药物的选择压力,造成耐药菌株不断出现,如耐碳青酶烯类肠杆菌、耐万古霉素肠球菌、耐甲氧西林金黄色葡萄球菌等检出率逐年升高[1-4]。耐药菌株的出现和播散对人类生命健康和社会经济发展造成了极大的影响。通过基因的水平转移获得耐药性基因是细菌耐药性产生和播散的重要途径[5]。整合子可通过位点特异性重组捕获并表达外源性耐药基因盒,在细菌耐药性产生和播散中起着“抗菌药物解码器”的作用[6-8]。同时,整合子常可以搭载于质粒或者转座子上,随着质粒和转座子的移动造成耐药基因的水平传播[7]。
整合子根据整合酶蛋白一级结构的不同进行分类,其中第1类整合子在临床菌株中分布最为广泛[7]。第1类整合子的基本结构包括编码整合酶的基因intI1、整合子重组位点attI1以及整合子可变区启动子Pc,有的整合子在整合子重组位点attI1位置附近还有启动子P2[6-7, 9]。整合子可变区会携带数量不等的基因盒,基因盒由一个读码框和一个基因盒重组位点attC组成,大多数基因盒自身不带有启动子,其表达依赖整合子可变区启动子Pc或P2[9-11]。由于质粒构建经济、简便、快速,人们对于整合子捕获耐药性基因盒机制方面的研究都是构建质粒模型,通过接合试验或实时定量PCR来分析基因盒整合频率或剪切频率[10-13]。然而,上述研究中质粒在单个细菌内都是以多拷贝的形式存在,除了整合酶介导的位点特异性重组外,质粒之间的同源重组、质粒拷贝数的变化等均影响基因盒整合频率或剪切频率的检测,特别是在研究宿主细菌相关蛋白对整合子捕获基因盒调控机制中,需要设定特殊的对照或检测其他参数来排除质粒拷贝数或同源重组的干扰,增加了工作量,同时影响结果的准确性[14-16]。本研究通过同源重组的方法,在细菌染色体中构建第1类整合酶介导基因盒位点特异性重组模型,将质粒模型中的多拷贝变为染色体模型中的单拷贝,单个菌体中排除质粒模型多拷贝的相互干扰,单个细菌即可代表一个分子的反应,通过该模型可更加精确地研究整合子中整合酶介导基因盒位点特异性重组反应、基因盒表达及其调控机制。
1 材料与方法 1.1 菌株和质粒大肠埃希菌JM109工程菌株、质粒pACYC184由本实验室保存,质粒pHSint和pLACaadA5C由复旦大学附属华山医院检验医学科构建,本实验室保存[11, 17],质粒pKOBEG由复旦大学附属金山医院李刚教授惠赠。
1.2 主要试剂和仪器PrimeSTAR HS DNA聚合酶、LA Taq DNA聚合酶、DL2000 DNA Marker和1 kb DNA Ladder,宝生物工程(大连)有限公司;阿拉伯糖、阿普霉素、氯霉素、链霉素、氨苄西林、柱式细菌基因组DNA抽提试剂盒、DNA胶回收试剂盒、细菌质粒抽提试剂盒、纯水(HPLC级)、溴化乙锭(EB)和异丙基硫代半乳糖苷(IPTG),生工生物工程(上海)股份有限公司;胰蛋白胨和酵母提取物,Oxoid公司。PCR仪,Applied Biosystems公司;凝胶成像仪,基因公司。寡核苷酸引物及DNA测序由生工生物工程(上海)股份有限公司完成,引物序列如表 1所示。
| 名称 Name | 序列 Sequences (5′→3′) | 靶序列 Target sequence |
| USF800 | ATTGCAGGTAGGCATCGGAA | US片段上游rffT基因 rffT gene upstream of US fragment |
| USF | AACTGACGTAGTACGCGAGA | US片段 US fragment |
| USR | TATGTCGTTGGCAAAAGCGT | |
| CMF | ACGCTTTTGCCAACGACATATTAAGGGCACCAATAAC | CM片段 CM fragment |
| CMR | CTGATGCCGCATAGTTAAGCCCTGTGACGGAAGATCAC | |
| LacA5CF | GGCTTAACTATGCGGCATCAG | LacA5片段 LacA5 fragment |
| LacA5CR | GTTGCAGGGCAGTTTGCCCCAATACGCAAACCGCCTC | |
| PcSF | GCAAACTGCCCTGCAAC | PcS片段 PcS fragment |
| PcSR | GACGATGCGTGGAGACC | |
| DSF | GGTCTCCACGCATCGTCTCAGGAAAAGTAGTTCAACA | DS片段 DS fragment |
| DSR | CTAACCGCAGCCAGTTCTTC |
使用柱式细菌基因组DNA抽提试剂盒抽提大肠埃希菌JM109基因组DNA作为模板,使用PrimeSTAR HS DNA聚合酶,用引物USF和USR扩增插入位点上游500 bp同源臂,命名为US片段;以JM109基因组DNA为模板,用引物DSF和DSR扩增插入位点下游500 bp同源臂,命名为DS片段;以质粒pACYC184为模板,用引物CMF和CMR扩增cat基因,命名为CM片段,作为氯霉素耐药筛选标志;以质粒pLACaadA5C为模板,用引物LacA5CF和LacA5CR扩增插入链霉素耐药aadA5基因盒的lacα段,命名为LacA5片段;以质粒pLACaadA5C为模板,用引物PcSF和PcSR扩增含强可变区启动子PcS和attI1位点的片段,命名为PcS片段。PCR反应体系(50 μL):5×PrimeSTAR缓冲液10 μL,dNTPs (2.5 μmol/L) 6 μL,上、下游引物(10 μmol/L)各1 μL,模板(约200 ng/μL) 1 μL,PrimeSTAR HS DNA聚合酶(2.5 U/μL) 0.5 μL,纯水(HPLC级) 30.5 μL。PCR反应条件:94 ℃ 4 min;98 ℃ 10 s,55 ℃ 5 s,72 ℃ 2 min,共30个循环;72 ℃ 5 min。各个片段结构及相应引物位置见图 1。
|
| 图 1 整合子反应模型的结构及整合反应示意图 Figure 1 The structure of the integration reaction model and the schematic diagram of the integration reaction |
|
|
将上述US片段、CM片段、LacA5片段、PcS片段和DS片段等摩尔比混合作为模板,用引物USF和DSR,通过重叠延伸PCR扩增出上述5个片段的连接体。PCR反应体系:5×PrimeSTAR缓冲液10 μL,dNTPs (2.5 μmol/L) 6 μL,引物USF和DSR (10 μmol/L)各1 μL,混合模板(约200 ng/μL) 1 μL,PrimeSTAR HS DNA聚合酶(2.5 U/μL) 0.5 μL,纯水(HPLC级) 30.5 μL,共50 μL。PCR反应条件:94 ℃ 4 min;98 ℃ 10 s,55 ℃ 5 s,72 ℃ 4 min,共30个循环;72 ℃ 5 min。扩增的连接体片段命名为UCAPSD片段,即为打靶片段(图 1)。
1.5 通过同源重组将UCAPSD片段插入大肠埃希菌JM109染色体中先将pKOBEG质粒(阿普霉素抗性)转化入大肠埃希菌JM109中,制备经30 mmol/L阿拉伯糖诱导的大肠埃希菌JM109电转化感受态细菌,通过电转化,将上述打靶片段电转入携带pKOBEG质粒的大肠埃希菌JM109中,用含25 μg/mL的氯霉素LB琼脂平板筛选插入成功菌株。
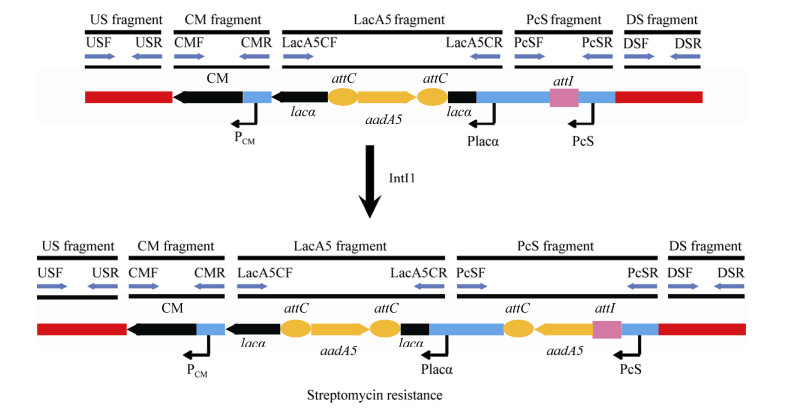
1.6 插入成功菌株的鉴定以上述氯霉素平板上筛选出的单克隆菌株基因组DNA为模板,用插入位点上游800 bp rffT基因的引物USF800和CM片段的反向引物CMR进行PCR扩增,PCR扩增出1 700 bp左右大小的产物为插入成功菌株,无PCR产物则为插入失败。同时用USF和DSR进行PCR扩增,插入菌株由于有3 000 bp模型片段的插入,PCR扩增出4 000 bp左右大小的产物为插入成功菌株,扩增出1 000 bp左右大小的产物为插入失败。同时以大肠埃希菌JM109基因组DNA作为阴性对照,以纯水作为空白对照。PCR产物经测序验证,将插入成功菌株命名为CLJM109Ⅰ。
1.7 链霉素平板筛选发生整合的菌株将上述插入成功的模型菌株CLJM109Ⅰ制备成为感受态细菌,转入高表达第1类整合酶的质粒pHSint (氨苄西林抗性)。将携带pHSint质粒的CLJM109Ⅰ细菌单克隆接种至100 μg/mL氨苄西林、25 μg/mL氯霉素和0.1 mmol/L IPTG的LB液体培养基中,37 ℃、200 r/min振荡培养过夜后,涂布于含30 μg/mL链霉素的LB平板上,37 ℃培养过夜,筛选发生整合的菌株。
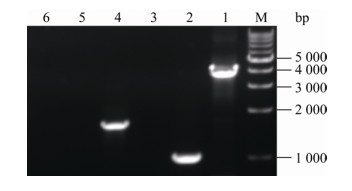
1.8 通过PCR和测序验证发生整合的菌株随机选取上述链霉素平板上生长的单克隆菌株,抽取细菌基因组DNA作为模板,用引物PcSF和PcSR进行PCR扩增,同时以含pUC19质粒的CLJM109Ⅰ菌株基因组DNA作为阴性对照。由于aadA5基因盒(约900 bp)的插入,扩增出1 400 bp左右大小的产物并经测序验证正确,即可认为是发生整合菌株,扩增出500 bp左右大小的产物,则为未发生整合菌株。
2 结果与分析 2.1 UCAPSD打靶片段的构建PCR成功扩增出CM片段、LacA5片段、PcS片段和两侧同源臂,经重叠延伸PCR成功扩增出其融合片段,PCR产物长度约为4 000 bp,经测序验证,片段大小和序列与理论一致,UCAPSD打靶片段构建成功。
2.2 插入菌株CLJM109Ⅰ的验证以氯霉素平板上筛选的待验证菌株CLJM109Ⅰ基因组DNA为模板,以引物USF800和CMR进行PCR扩增,成功扩增出长度约为1 700 bp的产物,而相应的对照大肠埃希菌JM109基因组DNA无PCR产物条带(图 2)。同时以引物USF和DSR对前述待验证菌株进行PCR扩增,成功扩增出长度约为4 000 bp的产物,而相应的对照大肠埃希菌JM109仅扩增出长度约为1 000 bp的产物(图 2),与预期结果一致。测序结果表明插入片段成功插入大肠埃希菌JM109染色体wzxE和rffA基因之间的骨架中,序列与理论序列一致,无碱基插入、缺失和错配,插入菌株CLJM109Ⅰ构建成功。
|
| 图 2 插入菌株验证PCR结果 Figure 2 Identification of knock-in strains by PCR 注:M:1 kb DNA Ladder;1−3:引物US和DS扩增产物;4−6:引物US800和CMR扩增产物. 1:CLJM109Ⅰ (约4 000 bp);2:JM109 (约1 000 bp);3:空白对照;4:CLJM109Ⅰ (约1 700 bp);5:JM109;6:空白对照. Note: M: 1 kb DNA Ladder; 1−3: Amplification products of primer US and DS; 4−6: Amplification products of primer US800 and CMR. 1: CLJM109 I (about 4 000 bp); 2: JM109 (about 1 000 bp); 3: Blank control; 4: CLJM109 Ⅰ (about 1 700 bp); 5: JM109; 6: Blank control. |
|
|
以链霉素平板中筛选出的14株待验证发生整合菌株基因组DNA为模板,使用引物PcSF和PcSR进行扩增,PCR鉴定结果如图 3所示,所有待验证菌株均成功扩增出长度约为1 400 bp的产物,经测序验证均为链霉素耐药基因盒aadA5整合入attI1位点发生整合的菌株。然而对照含pUC19质粒的CLJM109Ⅰ菌株仅扩增出约500 bp的产物(图 3、图 4)。
|
| 图 3 整合菌株的PCR鉴定 Figure 3 Identification of integrated strains by PCR 注:M:DL2000 DNA Marker;1−14:待验证整合菌株;N:阴性对照(含pUC19的CLJM109Ⅰ). Note: M: DL2000 DNA Marker; 1−14: Strains to be tested for integration; N: Negative control (CLJM109Ⅰcontaining pUC19). |
|
|
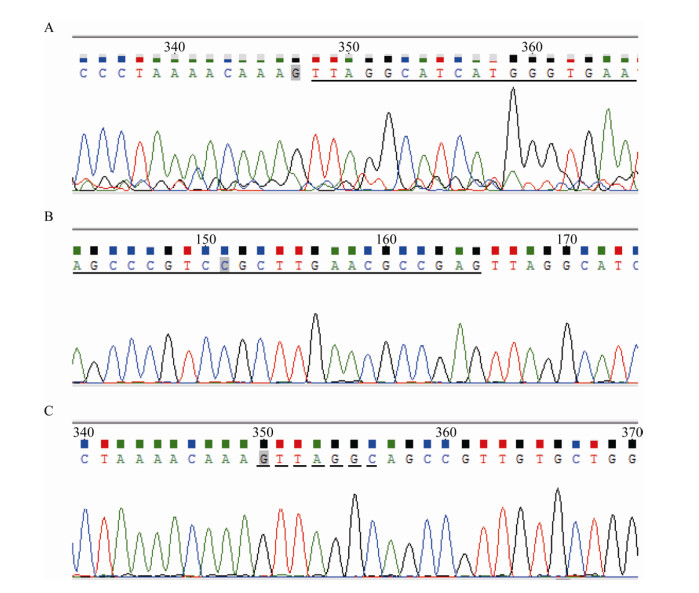
|
| 图 4 整合菌株测序鉴定 Figure 4 Identification of integrated strains by sequencing 注:A和B:下划实线为基因盒aadA5整合入attI1位点附近序列;C:下划虚线为未发生整合的attI1位点的核心位点序列. Note: The solid underlines in A and B indicate the sequences near the integration of gene cassette aadA5 into attI1 sites; The dotted underline in C indicates the core region sequence of the unintegrated attI1 sites. |
|
|
整合子因其在细菌耐药性产生和播散中的重要作用而受到研究者的关注,目前关于整合子的研究主要集中在整合子可变区基因盒分子流行病学研究、整合酶与重组位点attI、attC的相互作用、整合酶和基因盒表达调控及相互作用。通过整合子分子流行病学研究可发现一些新的基因盒及耐药机制,并发现一些整合子分布播散规律[18-22]。近年来,法国巴斯德研究所整合子研究小组在整合子捕获与表达耐药性基因盒调控机制方面的研究取得了一些进展,如通过体外实验证实第1类整合酶蛋白主要与双链attI1位点结合,与attC底链单链结合,并且attC底链发夹结构中的3个非配对结构可影响链的选择及整合频率[23-27];他们还发现整合酶的表达受SOS应激反应机制的调控[28-29];而且,不同可变区启动子Pc强度不同,与相应整合酶的活性及表达水平呈负相关[10, 30]。法国巴斯德研究所整合子研究小组在现有的研究基础上提出了整合酶整合与剪切基因盒的反应模式框架图,但截至目前第1类整合子剪切与整合耐药性基因盒的体外反应体系尚未成功建立,其具体的反应过程还不甚明了。
通过位点特异性重组对基因盒进行整合和剪切是整合子最重要的特征。研究者尝试采用不同的方法来评估整合子整合和剪切基因盒的效率,国外研究者通过接合试验和表型筛选的方法计算基因盒整合频率,该方法费时费力,需要2−3 d时间[10, 12]。Yang等通过构建包含attI1位点和基因盒的质粒,结合荧光定量PCR的方法,建立了一种检测基因盒整合频率的方法,使检测的灵敏度和实验周期大大缩短[13]。我们前期尝试采用检测转座子转座效率的“乳头状试验”,根据单个菌落生长过程中出现蓝色斑点数量来检测基因盒剪切效率[17]。该方法可以在同一块平板上对上百个菌落进行评估,可以高通量检测基因盒的剪切频率,但整合酶介导的基因盒剪切效率远远低于转座酶介导的转座频率,形成的蓝色斑点数较少,有待于进一步完善。
上述研究者构建的整合子模型均位于质粒上,由于质粒并非单拷贝,单个菌体中,所有质粒的整合和剪切并非同步进行,同时除了整合酶介导的位点特异性重组外,质粒之间的同源重组、质粒拷贝数的变化等均影响基因盒整合频率或剪切频率的检测。本研究中,我们采用同源重组的方法将构建的指示基因盒剪切与整合的模型片段成功插入大肠埃希菌JM109工程菌wzxE和rffA基因之间的染色体骨架中,排除了质粒多拷贝的干扰。该模型片段的插入没有破坏细菌染色体中的基因,并且构建模型片段中氯霉素启动子PCM、β半乳糖苷酶的α片段基因启动子Placα和整合子可变区强启动子PcS的方向都朝向插入位点US片段方向,与插入位点附近6 000−7 000 bp中的9个基因启动子方向一致,将模型片段的插入对宿主细菌功能基因表达的影响降到最低。
本研究中插入细菌染色体的LacA5片段来源于前期的乳头状试验中所用的质粒pLACaadA5C,基因盒aadA5在整合酶的介导下剪切后可以表达出具有β半乳糖苷酶的α片段活性的蛋白,在大肠埃希菌JM109中可发生α互补现象,在含有5-溴-4-氯-3-吲哚(X-gal)的平板上菌落会显蓝色,即通过蓝色菌落指示基因盒aadA5的剪切[17]。启动子PCM、Placα和PcS与基因盒中链霉素耐药基因aadA5读码框方向相反,同时aadA5读码框上游无启动子,因而不会表达,即未发生整合菌株不会表现出对链霉素耐药。当基因盒aadA5在整合酶的作用下整合入attI1位点时,aadA5可在可变区强启动子PcS的驱动下得以表达,赋予大肠埃希菌JM109对链霉素产生耐药性,即用链霉素耐药来指示基因盒aadA5的整合。
近年来,整合子捕获与表达耐药性基因盒调控机制方面的研究虽然取得了一定的进展,但其具体的反应机制仍未明了。本研究的另一个目的是研究整合子捕获耐药性基因盒的具体反应过程,我们将第1类整合酶剪切与整合耐药性基因盒这一复杂的反应过程简化为耐药基因盒是否必须先剪切下来才能发生整合,将其反应过程分为“剪切-粘贴”模式(基因盒先剪切下来再整合入attI1位点)和“复制-粘贴”模式(基因盒通过复制而不是剪切,即可整合入attI1位点),便于设计实验进行研究,以明确整合子捕获耐药性基因盒是否依赖或者独立于剪切反应。本研究将耐药性基因盒及整合子重组位点插入细菌染色体上,排除了多拷贝质粒的干扰,一个细菌就是一个反应容器,代表一个分子的反应。后续试验中计划用高表达第1类整合酶蛋白的温度敏感质粒转化进入该染色体反应模型菌内,第1类整合酶蛋白经诱导表达介导基因盒的整合与剪切后,通过在高温条件下培养传代消除高表达整合酶的质粒以终止整合与剪切反应,菌液涂布于相应筛选琼脂平板上,检测一定数量菌落(代表一定数量的单个细菌,即一定数量单个分子的反应)中不同反应模式(“剪切-粘贴”模式和“复制-粘贴”模式)是否存在及两者的比例,可为后续揭示整合子捕获耐药性基因盒反应机制提供线索。
本研究构建的染色体反应模型也有需要改进的地方,本模型中待整合的基因盒与attI1位点相邻,需要构建一定数量基因盒与attI1之间距离不同的反应模型来明确不同距离是否影响整合子捕获耐药性基因盒的效率及反应模式;同时需要分别构建与染色体复制方向相同和相反的反应模型,研究插入染色体不同方向是否影响整合子捕获耐药性基因盒的效率及反应模式,这些都将在后续的研究中加以完善。
总之,我们通过同源重组的方法在大肠埃希菌染色体上成功构建了第1类整合子捕获耐药性基因盒反应模型,该反应模型排除了质粒多拷贝的干扰,一个细菌就是一个反应容器,代表一个分子的反应,为进一步揭示整合子捕获耐药性基因盒的反应机制奠定了基础。
| [1] |
Smibert O, Satlin MJ, Nellore A, et al. Carbapenem-resistant Enterobacteriaceae in solid organ transplantation: management principles[J]. Current Infectious Disease Reports, 2019, 21(7): 26. DOI:10.1007/s11908-019-0679-4 |
| [2] |
Woodford N, Wareham DW, Guerra B, et al. Carbapenemase-producing Enterobacteriaceae and non-Enterobacteriaceae from animals and the environment: an emerging public health risk of our own making?[J]. Journal of Antimicrobial Chemotherapy, 2014, 69(2): 287-291. DOI:10.1093/jac/dkt392 |
| [3] |
Tzavaras I, Siarkou VI, Zdragas A, et al. Diversity of vanA-type vancomycin-resistant Enterococcus faecium isolated from broilers, poultry slaughterers and hospitalized humans in Greece[J]. Journal of Antimicrobial Chemotherapy, 2012, 67(8): 1811-1818. DOI:10.1093/jac/dks166 |
| [4] |
Neradova K, Jakubu V, Pomorska K, et al. Methicillin-resistant Staphylococcus aureus in veterinary professionals in 2017 in the Czech Republic[J]. BMC Veterinary Research, 2020, 16(1): 4. DOI:10.1186/s12917-019-2223-z |
| [5] |
Partridge SR, Kwong SM, Firth N, et al. Mobile genetic elements associated with antimicrobial resistance[J]. Clinical Microbiology Reviews, 2018, 31(4): e00088-17. DOI:10.1128/CMR.00088-17 |
| [6] |
Boucher Y, Labbate M, Koenig JE, et al. Integrons: mobilizable platforms that promote genetic diversity in bacteria[J]. Trends in Microbiology, 2007, 15(7): 301-309. DOI:10.1016/j.tim.2007.05.004 |
| [7] |
Cambray G, Guerout AM, Mazel D. Integrons[J]. Annual Review of Genetics, 2010, 44: 141-166. DOI:10.1146/annurev-genet-102209-163504 |
| [8] |
Wei QH, Xiao LL, Hu QF, et al. Structural analysis of atypical class 1 integrons in clinical Proteus isolates[J]. Chinese Journal of Microbiology and Immunology, 2018, 38(6): 440-445. (in Chinese) 魏取好, 肖林林, 胡庆丰, 等. 临床分离变形杆菌不典型第1类整合子结构分析[J]. 中华微生物学和免疫学杂志, 2018, 38(6): 440-445. |
| [9] |
Collis CM, Hall RM. Expression of antibiotic resistance genes in the integrated cassettes of integrons[J]. Antimicrobial Agents and Chemotherapy, 1995, 39(1): 155-162. DOI:10.1128/AAC.39.1.155 |
| [10] |
Jové T, da Re S, Denis F, et al. Inverse correlation between promoter strength and excision activity in class 1 integrons[J]. PLoS Genetics, 2010, 6(1): e1000793. DOI:10.1371/journal.pgen.1000793 |
| [11] |
Wei QH, Jiang XF, Li M, et al. Transcription of integron-harboured gene cassette impacts integration efficiency in class 1 integron[J]. Molecular Microbiology, 2011, 80(5): 1326-1336. DOI:10.1111/j.1365-2958.2011.07648.x |
| [12] |
Demarre G, Frumerie C, Gopaul DN, et al. Identification of key structural determinants of the IntI1 integron integrase that influence attC×attI1 recombination efficiency[J]. Nucleic Acids Research, 2007, 35(19): 6475-6489. DOI:10.1093/nar/gkm709 |
| [13] |
Yang ZH, Jiang XF, Wei QH, et al. A novel and rapid method for determining integration frequency catalyzed by integron integrase intI1[J]. Journal of Microbiological Methods, 2009, 76(1): 97-100. DOI:10.1016/j.mimet.2008.09.002 |
| [14] |
Baharoglu Z, Bikard D, Mazel D. Conjugative DNA transfer induces the bacterial SOS response and promotes antibiotic resistance development through integron activation[J]. PLoS Genetics, 2010, 6(10): e1001165. DOI:10.1371/journal.pgen.1001165 |
| [15] |
Cagle CA, Shearer JES, Summers AO. Regulation of the integrase and cassette promoters of the class 1 integron by nucleoid-associated proteins[J]. Microbiology, 2011, 157(Pt 10): 2841-2853. |
| [16] |
Lacotte Y, Ploy MC, Raherison S. Class 1 integrons are low-cost structures in Escherichia coli[J]. The ISME Journal, 2017, 11(7): 1535-1544. DOI:10.1038/ismej.2017.38 |
| [17] |
Wei QH, Jiang XF, Li M, et al. Establishment of a "papillary test" based method for detecting the gene cassette excision frequency mediated by integrase[J]. Chinese Journal of Microbiology and Immunology, 2012, 32(10): 871-873. (in Chinese) 魏取好, 蒋晓飞, 李敏, 等. 基于"乳头状试验"检测整合酶介导基因盒剪切频率方法的建立[J]. 中华微生物学和免疫学杂志, 2012, 32(10): 871-873. |
| [18] |
Wei QH, Jiang XF, Li M, et al. Diversity of gene cassette promoter variants of class 1 integrons in uropathogenic Escherichia coli[J]. Current Microbiology, 2013, 67(5): 543-549. DOI:10.1007/s00284-013-0399-1 |
| [19] |
Couvé-Deacon E, Jové T, Afouda P, et al. Class 1 integrons in Acinetobacter baumannii: a weak expression of gene cassettes to counterbalance the lack of LexA-driven integrase repression[J]. International Journal of Antimicrobial Agents, 2019, 53(4): 491-499. DOI:10.1016/j.ijantimicag.2018.11.012 |
| [20] |
Panopoulou M, Alepopoulou E, Ikonomidis A, et al. Emergence of VIM-12 in Enterobacter cloacae[J]. Journal of Clinical Microbiology, 2010, 48(9): 3414-3415. DOI:10.1128/JCM.00843-10 |
| [21] |
Wei QH, Jiang XF, Yang ZH, et al. dfrA27, a new integron-associated trimethoprim resistance gene from Escherichia coli[J]. Journal of Antimicrobial Chemotherapy, 2009, 63(2): 405-406. |
| [22] |
Wei QH, Hu QF, Li SS, et al. A novel functional class 2 integron in clinical Proteus mirabilis isolates[J]. Journal of Antimicrobial Chemotherapy, 2014, 69(4): 973-976. DOI:10.1093/jac/dkt456 |
| [23] |
Francia MV, Zabala JC, de La Cruz F, et al. The IntI1 integron integrase preferentially binds single-stranded DNA of the attC site[J]. Journal of Bacteriology, 1999, 181(21): 6844-6849. DOI:10.1128/JB.181.21.6844-6849.1999 |
| [24] |
Loot C, Parissi V, Escudero JA, et al. The integron integrase efficiently prevents the melting effect of Escherichia coli single-stranded DNA-binding protein on folded attC sites[J]. Journal of Bacteriology, 2014, 196(4): 762-771. DOI:10.1128/JB.01109-13 |
| [25] |
Grieb MS, Nivina A, Cheeseman BL, et al. Dynamic stepwise opening of integron attC DNA hairpins by SSB prevents toxicity and ensures functionality[J]. Nucleic Acids Research, 2017, 45(18): 10555-10563. DOI:10.1093/nar/gkx670 |
| [26] |
Nivina A, Escudero JA, Vit C, et al. Efficiency of integron cassette insertion in correct orientation is ensured by the interplay of the three unpaired features of attC recombination sites[J]. Nucleic Acids Research, 2016, 44(16): 7792-7803. DOI:10.1093/nar/gkw646 |
| [27] |
Mukhortava A, Pöge M, Grieb MS, et al. Structural heterogeneity of attC integron recombination sites revealed by optical tweezers[J]. Nucleic Acids Research, 2019, 47(4): 1861-1870. DOI:10.1093/nar/gky1258 |
| [28] |
Guerin É, Cambray G, Sanchez-Alberola N, et al. The SOS response controls integron recombination[J]. Science, 2009, 324(5930): 1034. DOI:10.1126/science.1172914 |
| [29] |
Hocquet D, Llanes C, Thouverez M, et al. Evidence for induction of integron-based antibiotic resistance by the SOS response in a clinical setting[J]. PLoS Pathogens, 2012, 8(6): e1002778. DOI:10.1371/journal.ppat.1002778 |
| [30] |
Guérin E, Jové T, Tabesse A, et al. High-level gene cassette transcription prevents integrase expression in class 1 integrons[J]. Journal of Bacteriology, 2011, 193(20): 5675-5682. DOI:10.1128/JB.05246-11 |
 2020, Vol. 47
2020, Vol. 47