扩展功能
文章信息
- 赵佳怡, 甄世军, 张翠云, 殷密英, 张胜, 何泽, 宁卓
- ZHAO Jia-Yi, ZHEN Shi-Jun, ZHANG Cui-Yun, YIN Mi-Ying, ZHANG Sheng, HE Ze, NING Zhuo
- 深部热水硫酸盐还原菌微滴数字PCR检测技术的建立与应用
- Development and application of a droplet digital PCR technique for detection of sulfate-reducing bacteria in deep geothermal water
- 微生物学通报, 2020, 47(11): 3756-3767
- Microbiology China, 2020, 47(11): 3756-3767
- DOI: 10.13344/j.microbiol.china.191034
-
文章历史
- 收稿日期: 2019-12-12
- 接受日期: 2020-05-08
- 网络首发日期: 2020-06-04
2. 中国地质科学院研究生院 北京 100037;
3. 中国地质调查局/河北省地下水污染机理与修复重点实验室 河北 石家庄 050061
2. Graduate University of Chinese Academy of Geological Sciences, Beijing 100037, China;
3. Key Laboratory of Groundwater Contamination and Remediation, China Geological Survey & Hebei Province, Shijiazhuang, Hebei 050061, China
硫酸盐还原菌(sulfate-reducing bacteria,SRB)是一类可将硫酸盐还原为硫化氢的厌氧菌。这些厌氧菌主要是细菌[1]。SRB在硫循环、有机物和金属污染修复等方面发挥着重要作用[2],而SRB活动产生的硫化氢是一种有毒的侵蚀性气体,会造成很多环境和经济问题,如危害工人健康、降低煤和天然气及石油的品质[3]、与金属结合会腐蚀堵塞地下管井等[4]。定量检测SRB对了解其对环境变化的响应和制定适当的处理方案非常重要。
人们常用的SRB定量检测技术主要是MPN (most probable number)培养技术和免培养的分子生物学技术。MPN培养技术费时、费力、培养时间长[5],且由于实验室很难模拟自然环境条件,只有部分微生物被检测[6],导致检测结果偏低,检测结果意义有限。免培养的分子生物学技术不但能检测到可培养菌,而且能检测到不可培养菌,特别是定量PCR (quantitative PCR,qPCR)技术,可通过检测样品微生物DNA中的特定基因片段快速定量微生物,这种特定的基因片段通常是16S rRNA基因或者是功能基因[7-8]。在SRB中最重要的功能基因是编码异化亚硫酸盐还原酶Dsr (dissimilatory sulfate reduction)的基因,该酶是硫酸盐还原过程的一个关键酶,是硫酸盐还原过程的最后一步将亚硫酸盐还原为硫化氢的催化酶[9],编码该酶的基因至少包括2个亚基基因dsrA和dsrB,其中dsrB基因被发现于所有已知的SRB中[10],是检测SRB的标记功能基因,利用qPCR可检测环境中硫酸盐还原菌的dsrB含量[11-12]。该技术需要标准品制成标准曲线,而获取标准曲线的过程费时费力。
微滴数字PCR (droplet digital PCR,ddPCR)技术是新一代基因检测定量技术,该技术不需要标准曲线就可实现基因的绝对定量。其原理是采用油包水技术将样品分散为上万个微滴,每个微滴或不包含目标基因,或包含1个或数个目标基因,然后让每个微滴进行PCR扩增,扩增后逐个检测微滴,有扩增荧光信号的微滴判读为1,没有扩增荧光信号的微滴判读为0,最后根据泊松分布计算出目标基因的起始拷贝数或浓度[13]。由于该技术是统计有无PCR扩增微滴个数,即使部分扩增也不影响阳性微滴统计的个数,因此该技术较qPCR技术更能耐受PCR扩增抑制[14]。由于该技术的原理与qPCR的本质差异,其检测灵敏度更高,可检测含量很低的样品。目前该技术主要应用于医疗、食品领域[15-16],而在自然环境应用较少[17-18],特别是应用于深部热水环境鲜见报道。
地壳深部蕴藏着巨量热能,通过钻探抽取深部热水可用于地面供热和发电,地热能已被认为是一种绿色能源。冀中地热区位于河北省中部地区,地热能开发历史悠久,目前主要开采1 800 m以上蓟县系雾迷山组上段岩溶-裂隙热储层,而对深部蓟县系雾迷山组下段和高于庄组热储层地热资源状况不清。中国地质调查局自2018年以来在冀中地热区实施了一批深部地热科学钻探,用于获取蓟县系雾迷山组和高于庄组热储层的地质、水文地质和地热基础数据,评价深部新储层地热资源[19]。地下存在一个深部生物圈[20],其生物量超过地球总生物量的50%[21]。深部沉积岩、玄武岩、花岗岩和变质岩等岩性环境的微生物群落已被调查[22-23],而地下深部碳酸盐岩岩溶-裂隙热储层微生物群落特征仍然不清。地热科学钻探为深部热水微生物研究提供了获取深部样品的机会,我们选取了部分钻探孔用于探索深部热水微生物群落特征。深部热水含盐量高、基质复杂、抑制物多、微生物含量低,常规的方法难于准确定量。SRB是地下深部频繁检出的微生物[24]。鉴于ddPCR技术优势,本文旨在建立以dsrB基因为检测目标,快速、准确定量深部热水SRB的ddPCR技术,并将该技术用于实际样品的检测,与浅层水和土壤对比探究深部热水SRB的丰富性,提高对深部生物圈的认识,为深部热水合理开发利用提供技术支撑和科学依据。
1 材料与方法 1.1 样品采集和预处理在冀中地热区(115°55′46″E,38°58′45″N),从区域地下水流系统角度来看,上部第四系100 m以上的地下水属浅层水,而地表 1 km以下的碳酸盐岩岩溶-裂隙热水属深部热水。2019年3-5月,7组深部热水样分别采自7个冀中地热区地热科学钻探孔,采样方法是钻探孔完工后抽水试验时最后阶段的抽出水,井台泵口采样;同时采集每个钻孔附近大田农灌井水(浅层水)和土壤样品,作为参照样品。采样深度和现场测试指标分别是:深部热水采样深度变化在1 500-3 853 m之间[泵口温度56-105 ℃,pH 6.77-8.75,总溶解性固体(total dissolved solids,TDS)含量1 990-2 677 mg/L];浅层水样变化在70-100 m之间(水温14-17 ℃,pH 7.50-7.87,TDS含量202-548 mg/L);土壤样品采样深度均为0.5 m (原位温度8-13 ℃,土水体积比1:2.5,水溶液pH 8.55-8.73,TDS含量63-178 mg/L)。水样采集容器为18 L耐高温的无菌塑料桶,样品采集后运回实验室立即定量(2 L或4 L)过滤到孔径为0.22 μm的多片聚四氟乙烯过滤膜上,将带有过滤物的过滤膜保存在-70 ℃冰箱中备用。土样采集使用洛阳铲取芯,采用无菌小刀从岩芯中部挖取土样,装入专用无菌塑料袋中,现场冰块保温箱保存,尽快运回实验室也在-70 ℃冰箱中保存备用。
1.2 主要试剂和仪器土壤DNA提取试剂盒(DNeasy® PowerSoil® Kit),QIAGEN公司;染料法ddPCR定量试剂盒:SuperMix预混液、微滴生成油、微滴分析油、微滴生成卡槽、微滴生成卡槽胶垫、锡纸膜以及96孔PCR板,QX200 Droplet Digital PCR系统:微滴生成仪、热循环仪和微滴分析仪,Bio-Rad公司。
1.3 样品总DNA提取水样和土样按DNA提取试剂盒说明书定量提取样品总DNA,其中水样每次取一片过滤膜剪碎,最后按定量过滤的多片膜提取的DNA合并为一个样品溶于100 μL TE溶液中;土样称取0.3-0.5 g左右,同法提取DNA,溶于100 μL TE溶液中。采用超微量紫外分光光度计测定DNA质量和数量。将100 μL DNA溶液分装2份,一份35 μL,另一份65 μL,前者用于条件试验,后者用于实际样品测试,-20 ℃保存备用。
1.4 SRB质粒的构建根据SRB dsrB基因的引物序列DSRp2060F (5′-CAACATCGTYCAYACCCAGGG-3′)和DSR4R (5′-GTGTAGCAGTTACCGCA-3′)[25-26],在NCBI Primer-BLAST操作平台上搜索dsrB基因序列,将获得的与引物对完全匹配的SRB dsrB基因序列[KY464856.1,Uncultured bacterium clone D4050-3-1 dissimilatory sulfite reductase beta subunit (dsrB) gene]提交给上海捷瑞生物工程有限公司,进行dsrB (约350 bp)的PCR扩增并构建重组质粒。PCR反应体系(50 μL):Pfu-MasterMix 25 μL,上、下游引物(10 μmol/L)各2 μL,DNA模板5 μL,ddH2O 16 μL。PCR反应条件:94 ℃ 2 min;94 ℃ 20 s,55 ℃ 20 s,72 ℃ 40 s,共20个循环;72 ℃ 2 min;4 ℃保存。重组质粒以pGH为克隆载体,转化到大肠杆菌DH5α感受态细胞中,筛选阳性克隆子并测序,序列正确的阳性克隆子利用试剂盒提取质粒作为标准品,最终获得SRB质粒干粉。根据获得的质粒干粉质量和分子量,加入一定体积的TE制成溶液,再换算成质粒标准品中硫酸盐还原菌dsrB基因片段拷贝数,其浓度为1.1×1010 copies/μL。
1.5 实验方法 1.5.1 SRB ddPCR检测方法采用染料法反应体系QX200 ddPCR EvaGreen Supermix,以人工合成的SRB质粒或实际样品为模板进行SRB ddPCR反应。该反应步骤包括:体系配制、微滴生成、扩增和信号读取分析4个步骤[27]。根据仪器生产厂家推荐,20 μL ddPCR反应体系为:SuperMix 10 μL,上、下游引物(10 μmol/L)各0.2 μL,ddH2O 8.6 μL,DNA模板1 μL。将20 μL反应液加入微滴生成卡DG8中间一排,再将70 μL微滴生成油加入DG8的最底一排,最后在微滴生成卡上覆盖专用胶垫,置于微滴生成仪,自动生成反应微滴。待微滴生成完毕后,采用8通道移液枪从微滴生成卡的最上一排吸取生成的全部微滴(约30 μL),加入96孔反应板内,用预热好的PX1热封仪对其进行封膜,封膜运行程序为180 ℃ 10 s。封膜后将96孔板放入PCR仪进行两步法扩增。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,退火温度60 s,共40个循环;4 ℃ 5 min,90 ℃ 5 min,4 ℃保存。全程升降温速度均小于等于2 ℃/s。扩增结束后,将96孔板置于微滴读取仪中进行信号读取,并使用软件QuantaSoft V1.7.4分析实验数据,获得检测结果并分析。除非特别说明,每次运行每个样品2个重复,且带空白对照。
1.5.2 SRB ddPCR技术的条件优化ddPCR反应体系中,退火温度作为反应体系中的一个重要参数,温度过高或者过低都会导致液滴生成数少或非特异性扩增。为了获得切合实际样品的退火温度,选择实际样品深部热水样、浅层水样和土样进行梯度退火温度试验,温度梯度为50、50.7、52、53.9、56.3、58.3、59.4和60 ℃。通过酶的活性对温度的要求、生成的阳性微滴数以及阴阳微滴荧光信号值差异情况等确定最佳退火温度。
1.5.3 SRB ddPCR方法的线性范围和灵敏性试验以人工构建的SRB质粒为标准品,浓度为1.1×1010 copies/μL,10倍梯度稀释,选取1.1×100-1.1×106 copies/μL-DNA浓度范围,编号依次为Z0、Z1、Z2、Z3、Z4、Z5和Z6,采用优化的退火温度进行ddPCR扩增,评估该方法的线性关系和灵敏性。
1.5.4 SRB ddPCR方法的特异性试验以双蒸水代替模板为空白,以细菌16S rRNA基因(LC435077.1)、古菌16S rRNA基因(KT461011.1)和产甲烷菌基因mcrA (KP742467.1)特异性插入片段合成的质粒为阴性对照,对空白、深部热水样和阴性对照进行SRB ddPCR检测,评估该方法的特异性。
1.5.5 SRB ddPCR方法的重复性试验为了评估实际样品的重复性,选取高(33 350 copies/μL-DNA)、中(13 540 copies/μL-DNA)、低(9 590 copies/μL-DNA)这3个浓度的深部热水样,进行SRB ddPCR检测,每个样品设立6孔重复,计算该方法的相对标准差,评估该方法的重复性。
1.6 实际样品检测以双蒸水代替模板为空白,采用优化的SRB ddPCR检测方法分别对深部热水样、浅层水样和土样进行检测,分析对比检测结果,说明深部热水硫酸盐还原菌的丰富性。数据处理时首先检查微滴总数,若两个重复微滴总数大于1万个,则取均值;若两个重复有1个大于1万个,可视情况取大于1万者或重做;若两个重复小于1万个,则需重做。
2 结果与分析 2.1 SRB ddPCR条件的优化微滴数字PCR技术的关键指标是形成的微滴总数。理论上20 μL反应液可形成2万个微滴,一般大于1万个微滴可满足要求。通过反复实践,当使用细枪头每次10 μL分两次加样且加入槽中的样品无气泡时,形成的微滴数一般大于1万个。如图 1所示,微滴数变化在14 075-15 259个之间,均大于1万个,为后续试验结果提供了可靠的保证。
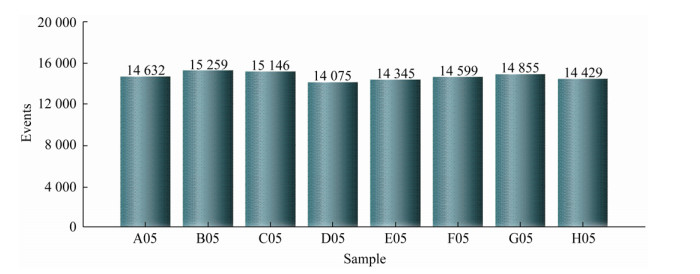
|
| 图 1 SRB ddPCR退火温度优化生成的微滴总数 Figure 1 Total droplet number generated from annealing temperatures optimization of SRB ddPCR 注:A05-H05为浅层水样品E5-2. Note: A05-H05: Shallow water sample E5-2. |
|
|
以两步法为PCR反应程序,对反应退火温度进行优化,温度梯度范围为50-60 ℃。深部热水、浅层水和土壤检测结果(图 2A-C)显示,退火温度在50-60 ℃之间均可检测到扩增荧光信号,而且在50-53.8 ℃时扩增荧光信号强度最大。综合考虑酶的活性、特异性、生成的阳性微滴数以及阴阳微滴荧光信号值差异情况等因素,确定退火温度为54 ℃。SRB的染料法ddPCR最终优化后的反应体系(20 μL):SuperMix 10 μL,上、下游引物(10 μmol/L)各0.2 μL,ddH2O 8.6 μL,模板DNA 1 μL。优化后的PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,54 ℃ 60 s,40个循环;4 ℃ 5 min,90 ℃5 min,4 ℃保存;全程升降温速度均≤2 ℃/s。
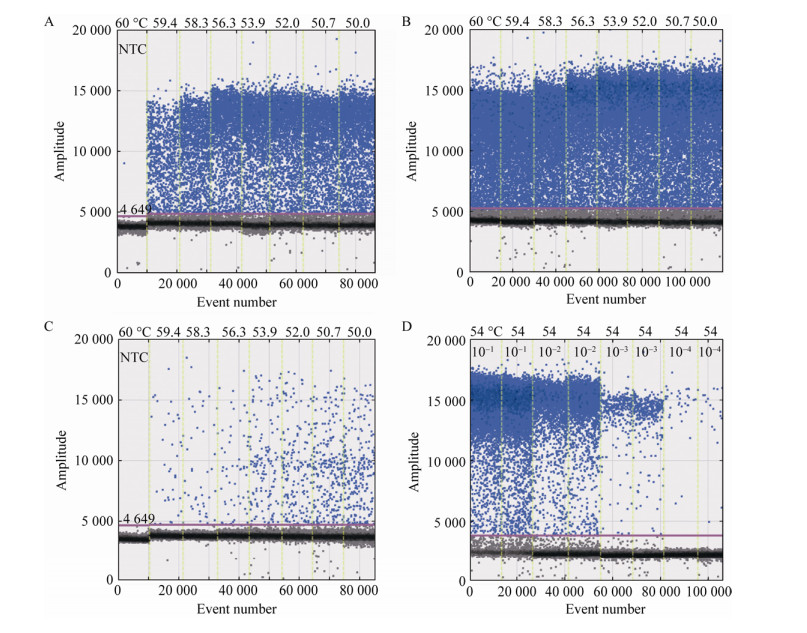
|
| 图 2 SRB ddPCR方法不同退火温度(A-C)和深部热水系列稀释(D)检测结果 Figure 2 Results of SRB ddPCR assay from different annealing temperatures (A-C) and from serial dilutions of deep geothermal water (D) 注:A:深部热水E5-1;B:浅层水E5-2;C:土壤E5-3;D:深部热水E4-1系列稀释(10-1、10-2、10-3和10-4). Note: A: Deep geothermal water E5-1; B: Shallow water E5-2; C: Soil E5-3; D: Serial dilutions (10-1, 10-2, 10-3 and 10-4) of deep geothermal water sample E4-1. |
|
|
图 2A-C还显示中间荧光强度的“雨点”较多。为了探明其原因,将深部样品E4-1系列稀释(10-1、10-2、10-3和10-4),图 2D结果显示,随着稀释度的增加,中间荧光强度的微滴越来越少,稀释到10-4时,中间荧光强度的微滴很少了,说明样品稀释可减少中间荧光强度的微滴。
2.2 SRB ddPCR方法的线性范围和灵敏度以人工构建的SRB质粒为标准品,10倍梯度稀释,选取1.1×100-1.1×106 copies/μL-DNA浓度范围,编号依次为Z0、Z1、Z2、Z3、Z4、Z5和Z6,采用优化的反应条件进行SRB ddPCR扩增。图 3结果显示,已知浓度1.1×106 copies/μL-DNA对应的Z6明显偏离相关直线,而在1.1×100- 1.1×105 copies/μL-DNA浓度范围内,已知值与检测值线性关系良好,R2为0.996 0。该技术最高检出限为1.1×105 copies/μL-DNA,检测的线性范围为4个数量级;最低定量限为1.1 copies/μL-DNA,相应地,在20 μL反应液中阳性微滴数为1,说明在20 μL反应液中只要有1个dsrB分子,该技术也能检测出来。因此,该技术的检出限或者灵敏度为1 copy/μL-DNA。
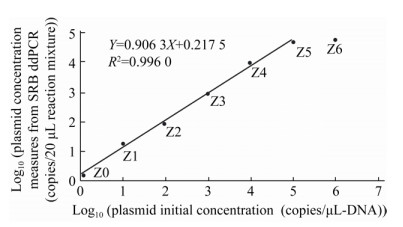
|
| 图 3 SRB质粒已知浓度与SRB ddPCR检测浓度的线性关系 Figure 3 Linear regression of known concentrations and measured concentrations from SRB ddPCR assay for SRB plasmid |
|
|
深部热水样E6-1、E5-1和E2-1进行SRB ddPCR检测,每个浓度6次重复测定。检测结果除了E5-1有一个重复样微滴总数小于1万个外,其余重复样微滴总数均大于1万个。去掉微滴总数小于1万个重复样后进行统计分析。表 1结果显示,相对标准差(relative standard deviation,RSD)均优于9%,远低于推荐的允许误差25%[28],说明建立的SRB ddPCR检测方法具有很好的重复性。
| 样品编号 Sample ID | 各次测定值(copies/20 μL反应液) Measured values (copies/20 μL reaction mixture) | 平均值 Average | 标准差 Standard deviation | 相对标准差 RSD (%) | |||||
| 1 | 2 | 3 | 4 | 5 | 6 | ||||
| E6-1 | 32 320 | 33 920 | 31 160 | 34 940 | 34 120 | 34 360 | 33 670 | 1 431 | 4 |
| E5-1 | 14 280 | 13 680 | 13 420 | 14 440 | 13 340 | - | 13 832 | 501 | 4 |
| E2-1 | 10 200 | 8 740 | 10 180 | 10 160 | 9 540 | 8 320 | 9 523 | 819 | 9 |
| 注:-:微滴数小于1万的检测值去除. Note: -: A measured value removed due to less than 1×104 of droplet number. | |||||||||
以ddH2O代替模板为空白,对深部热水样E6-1和构建的细菌16S rRNA基因、古菌16S rRNA基因、产甲烷菌基因mcrA特异性片段质粒进行SRB ddPCR检测。图 4结果显示,除了深部热水样品检出大量阳性微滴外,其余各样品几乎未检出阳性微滴,说明建立的SRB ddPCR方法具有很高的特异性。
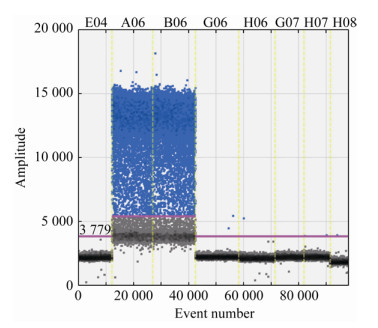
|
| 图 4 SRB ddPCR方法特异性检测结果 Figure 4 Specificity tests of SRB ddPCR assay 注:E04:NTC;A06、B06:深部热水E6-1;G06、H06:细菌16S rRNA基因;G07、H07:古菌16S rRNA基因;H08:产甲烷菌基因mcrA. Note: E04: NTC; A06, B06: Deep geothermal water sample E6-1; G06, H06: Bacterial 16S rRNA gene; G07, H07: Archaeal 16S rRNA gene; H08: Methanogenic Archaeal gene mcrA. |
|
|
在冀中地热区采集深部热水(1 500-3 853 m)及其附近浅层水(70-100 m)和土壤(0.5 m)样品各7组,定量过滤和DNA提取,分别定容于100 μL TE溶液中。加DNA模板1 μL进行SRB ddPCR检测,最终结果水土样分别换算为单位copies/mL和copies/g-dw表示。结果显示,深部热水样硫酸盐还原菌dsrB基因含量变化在11.2×100- 2.3×104 copies/mL之间,浅层水dsrB含量变化在6.4×100-9.4×102 copies/mL之间,而土壤dsrB含量
变化在4.7×102-3.5×103 copies/g-dw之间(表 2),相应的均值分别为(4.0±8.4)×103 copies/mL、(1.6±3.5)×102 copies/mL和(1.5±1.2)×103 copies/g-dw。深部热水硫酸盐还原菌dsrB基因含量均值分别较浅层水和土壤高。由此可见,深部热水不但检出硫酸盐还原菌,而且含量较浅层水(70-100 m)和土壤(0.5 m)更高,这与采样时深部热水散发出刺鼻的臭鸡蛋味(H2S味)的实际情况是一致的。
| 样品编号 Sample ID | 钻孔深度 Drilling depth (m) | 测定均值(copies/20 μL反应液) Measured mean (copies/20 μL reaction mixture) | dsrB含量 dsrB content |
| 深部热水 Deep geothermal water | (copies/mL) | ||
| E1-1 | 2 511.0 | 4.0×104 | 2.0×103 |
| E2-1 | 2 500.0 | 9.6×103 | 4.8×102 |
| E3-1 | 2 520.8 | 9.9×102 | 2.0×102 |
| E4-1 | 2 608.5 | 4.6×105 | 2.3×104 |
| E5-1 | 2 506.0 | 1.4×104 | 6.8×102 |
| E6-1 | 2 518.2 | 3.3×104 | 1.7×103 |
| E7-1 | 3 853.0 | 56 | 11.2 |
| 浅层水 Shallow water | (copies/mL) | ||
| E1-2 | 100 | 6.7×102 | 16.7 |
| E2-2 | 100 | 8.2×102 | 40.9 |
| E3-2 | 70 | 1.3×102 | 6.4 |
| E4-2 | 100 | 1.0×103 | 50.8 |
| E5-2 | 100 | 1.9×104 | 9.4×102 |
| E6-2 | 70 | 1.3×102 | 6.6 |
| E7-2 | 100 | 6.2×102 | 31.2 |
| 土壤 Soil | (copies/g-dw) | ||
| E1-3 | 0.5 | 2.7×102 | 6.5×102 |
| E2-3 | 0.5 | 2.3×102 | 5.0×102 |
| E3-3 | 0.5 | 1.3×102 | 4.7×102 |
| E4-3 | 0.5 | 7.0×102 | 2.4×103 |
| E5-3 | 0.5 | 2.4×102 | 8.6×102 |
| E6-3 | 0.5 | 6.0×102 | 2.1×103 |
| E7-3 | 0.5 | 8.4×102 | 3.5×103 |
本研究的目的是建立快速、准确定量深部热水硫酸盐还原菌的ddPCR检测技术,并与第四系浅层水和地表土壤对比,说明深部热水硫酸盐还原菌的丰富性。ddPCR技术的关键是微滴数的生成,理论上20 μL反应体系可生成2万个微滴,微滴数大于1万的试验可接受,否则试验必须重做。本研究通过操作技术改进,当使用细枪头分两次加样且加入微滴生成槽的样品无气泡时,生成的微滴数很高(图 1),均大于1万个,与同类试验结果相似[29],为后续试验结果提供了可靠的保证。退火温度是ddPCR扩增的关键参数,退火温度过高会低估基因的拷贝数,过低又会产生非特异性扩增,高估基因的拷贝数。本研究的退火温度(50-60 ℃)的试验结果(图 2A-C)显示,在50-60 ℃均有阳性微滴检出,温度越高,检测值越低;尽管50 ℃时检测值最大,54 ℃时次之,比58 ℃检测值高1.4倍,但是综合考虑酶的活性、特异性、生成的阳性微滴数以及阴阳微滴荧光信号值差异情况等因素,最终确定退火温度为54 ℃,该值与qPCR的退火温度(55 ℃)[23]相近。图 2A-C还显示属中间荧光强度的“雨点”较多,尽管曾试验过三步法,或增加退火/延伸时间到2 min等,但是“雨点效应”仍未得到改善。图 2D的结果显示,实际样品E4-1稀释到10-4时,“雨点效应”得到很大的改善,说明样品稀释可减少中间荧光强度的微滴。“雨点”多的原因可能是高浓度样品杂质多引起的。杂质多,影响PCR扩增的抑制物多,导致一些微滴部分扩增,出现中间荧光强度的“雨点效应”。通过稀释,抑制物浓度降低,稀释到10-4时,微滴PCR扩增基本不受影响,“雨点效应”没有了。尽管DNA高浓度时有“雨点效应”,由于ddPCR检测结果是统计PCR扩增后的阳性和阴性微滴数,PCR部分扩增微滴也属阳性微滴,而qPCR阈值Ct值受PCR扩增效率的影响,会影响目标基因的准确定量。因此ddPCR技术较qPCR技术更能耐受抑制物,仍然能够准确估计样品的基因拷贝数,这与文献[14]结果一致。另外,“雨点”多的原因也不排除其他原因,如操作过程中阳性微滴破裂,因为随着稀释倍数的增加,阳性微滴和雨点都在同时减少,阳性微滴减少,破裂的阳性微滴也会减少,从而导致“雨点”较少。
本研究利用人工构建的SRB质粒进行SRB ddPCR方法的线性范围和灵敏度试验,结果(图 3)显示,该技术检测的线性范围为4个数量级,最低定量限为1.1 copies/μL-DNA,灵敏度为1 copy/μL- DNA。仪器生产厂家说明书建议提前酶切,但是有研究表明,增加酶切对检测结果不会有很大影响[30],且复杂化了检测技术,增加了成本和污染的风险,因此,本研究没有考虑提前酶切。尽管没有提前酶切,该技术最高检出限(1.1×105)却大大超过仪器生产厂家规定的最高检出限(2×104),能满足实际需要。3个不同浓度实际样品的6次重复性实验结果显示,相对标准差(RSD)均优于9%(表 1)。在线性范围内,SRB质粒2个重复的系列稀释检测结果统计发现,RSD变化在0-11%之间。实际样品和dsrB质粒的RSD均远低于规定的允许误差25%。特异性试验结果(图 4)显示,该技术具有很好的特异性。完成一次96孔板样品的SRB ddPCR分析仅需5.5 h。与同类实验相比[18, 29],本研究建立的SRB ddPCR技术速度快、灵敏度高、重复性好、特异性强。与经典的qPCR相比,初步探索发现由于深部热水与浅层水、土壤样品的dsrB序列碱基不尽相同,融解曲线峰值位置不同,这需要用实际样品构建含有各自dsrB插入片段的质粒制作各自的标准曲线才能准确定量,这个过程费时费力,而ddPCR不需要标准曲线,体现了其优势。
实际样品的检测结果显示,深部热水存在硫酸盐还原菌,其dsrB含量变化在11.2×100- 2.3×104 copies/mL之间,平均值为4.0×103 copies/mL (SD=8.4×103 copies/mL)。标准偏差(standard deviation,SD)较均值大,说明7个样品的检测值变化大,是深部取样环境条件不尽相同所致。如最小值11.2 copies/mL为钻孔最深样品E7-1,深达3 853 m,符合取样深度越大微生物含量越低的规律。平均值与qPCR获得的油田水结果(2.79×102-1.26×104 copies/mL)[24]相似,但是较采自油水分离罐的油田水(105 copies/mL)[11]低,较芬兰东部2 260 m深处基岩裂隙水(10-102 copies/mL)[31]高,SRB含量的高低与环境条件密切相关。
浅层水SRB dsrB含量很低,变化在6.4×100-9.4×102 copies/mL之间,均值为(1.6±3.5)×102 copies/mL。其标准偏差也较均值大,同样说明7个样品的检测值变化大,是浅部取样环境条件不尽相同所致。dsrB拷贝数也代表SRB菌数量,因为大部分SRB菌每个细胞只有1个dsrB基因[32],这样dsrB基因拷贝数含量可与MPN检测结果的细胞数对比。李政红等[33]在相似深度采用MPN培养法检测地下水SRB结果显示,9个大田农灌井水样只有3个样品检出硫酸盐还原菌,最高为2.5 cells/mL,说明MPN培养法检测的大田农灌井地下水SRB含量也很低。但是本次研究SRB ddPCR方法对每个浅层水样即大田农灌井地下水均检出SRB,与以前的MPN培养法检测结果相比,平均高了近400倍,符合免培养法较培养法检测结果高的规律。大田农灌井地下水SRB含量低,除了与该环境营养水平低有关外,可能与该环境不是完全封闭的厌氧环境有关。
土壤SRB dsrB含量较浅层水高,含量变化在4.7×102-3.5×103 copies/g-dw之间,均值(1.5±1.2)×103 copies/g-dw。尽管其标准偏差(SD)较均值小,但是相对标准差(RSD)较大,达78%,同样说明7个样品的检测值变化较大,也是样品所处环境条件不尽相同所致。若土壤的容重取值1.5 g/cm3,则土壤dsrB检测结果可表示为每cm3的dsrB拷贝数,平均为996.5 copies/cm3,这个值较深部热水的平均值低4.6倍,但是较浅层水的平均值高6.3倍。土样SRB ddPCR检测结果较以前同一深度(50 cm)土样的MPN培养法检测结果(11.3 cells/g-dw)[33]平均高132倍,也符合免培养法较培养法检测结果高的规律,但是与20 cm深处水稻土SRB qPCR检测结果(108 copies/g-dw)[34]相比低许多,可能是20 cm深处水稻土有机质等营养更丰富和淹水条件下更易形成厌氧环境,有利于SRB生长繁殖的原因。本次研究50 cm处土壤的SRB ddPCR检测结果较浅层水高,可能是50 cm处土壤有机质相对于地下水更丰富及更易形成SRB生存的厌氧环境有关。
与浅层水和土壤相比,深部热水SRB含量更高,平均值分别高29.4倍和4.6倍,说明深部热水富含SRB菌,这与采样时深部热水散发出刺鼻的臭鸡蛋味(H2S味)的实际情况是一致的。
综上所述,本研究建立的SRB ddPCR检测技术不需要标准曲线,具有灵敏度高、特异性强、重复性好等特点。该技术有许多优点,也有一些缺点,如试剂成本较高、操作技术要求高、形成微滴前样品不能有气泡,但是该技术一旦开发成功,经验不断积累,操作熟练,这些缺点就不成问题了。因此,该技术具有很大的应用推广价值。下一步将开发深部热水其他菌的ddPCR检测技术,为深部热水微生物检测和定量提供新的技术支撑。
4 结论本研究进行了SRB ddPCR分析的条件优化,考察了其线性范围、灵敏度、重复性和特异性,建立了检测SRB的ddPCR技术,该技术具有速度快、灵敏度高、重复性好、特异性强等特点,为深部热水微生物准确定量检测提供了新的技术方法,同时为其他指示菌检测技术的建立提供了参考。利用该技术对深部热水、浅层水和土壤样品进行了检测,平均含量分别为(4.0±8.4)×103 copies/mL、(1.6±3.5)×102 copies/mL和(1.5±1.2)×103 copies/g-dw。与浅层水和土壤相比,深部热水富含SRB菌。
致谢: 本文工作得到中国地质科学院水文地质环境地质研究所王贵玲研究员、张薇副研究员等大力支持和帮助,特此致谢。| [1] |
Barton LL, Fauque GD. Biochemistry, physiology and biotechnology of sulfate-reducing bacteria[J]. Advances in Applied Microbiology, 2009, 68: 41-98. DOI:10.1016/S0065-2164(09)01202-7 |
| [2] |
Li YF, Liu XY, Yang CP. Bacteriology of Sulfate Reducing Bacteria[M]. Harbin: Northeast Forestry University Press, 2013. (in Chinese) 李永峰, 刘晓烨, 杨传平. 硫酸盐还原菌细菌学[M]. 哈尔滨: 东北林业大学出版社, 2013. |
| [3] |
Cord-Ruwisch R, Kleinitz W, Widdel F. Sulfate-reducing bacteria and their activities in oil production[J]. Journal of Petroleum Technology, 1987, 39(1): 97-106. DOI:10.2118/13554-PA |
| [4] |
Westphal A, Eichinger F, Eichinger L, et al. Change in the microbial community of saline geothermal fluids amended with a scaling inhibitor: effects of heat extraction and nitrate dosage[J]. Extremophiles, 2019, 23(3): 283-304. DOI:10.1007/s00792-019-01080-0 |
| [5] |
Zhang CY, Zhang S, He Z, et al. Assessment of microbial process potential for natural attenuation of contaminants in the thick vadose zone beneath the wastewater-irrigated area of southern Shijiazhuang City, Hebei Province[J]. Acta Geoscientica Sinica, 2014, 35(2): 223-229. (in Chinese) 张翠云, 张胜, 何泽, 等. 河北省石家庄市南部污灌区厚层包气带污染物自然衰减的微生物作用潜力评价[J]. 地球学报, 2014, 35(2): 223-229. |
| [6] |
Amann RI, Ludwig W, Schleifer KH. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiological Reviews, 1995, 59(1): 143-169. DOI:10.1128/MMBR.59.1.143-169.1995 |
| [7] |
Stubner S. Quantification of Gram-negative sulphate-reducing bacteria in rice field soil by 16S rRNA gene-targeted real-time PCR[J]. Journal of Microbiological Methods, 2004, 57(2): 219-230. DOI:10.1016/j.mimet.2004.01.008 |
| [8] |
Liu YC, He Z, Zhang S, et al. Development and application of a fluorescent quantitative real-time PCR technique for detection of methane-oxidizing bacteria in oil and gas field soil[J]. Microbiology China, 2014, 41(6): 1071-1081. (in Chinese) 刘雅慈, 何泽, 张胜, 等. 油气田土壤甲烷氧化菌实时荧光定量PCR检测技术的建立与应用[J]. 微生物学通报, 2014, 41(6): 1071-1081. |
| [9] |
Odom JM, Peck HD Jr. Hydrogenase, electron-transfer proteins, and energy coupling in the sulfate-reducing bacteria Desulfovibrio[J]. Annual Review of Microbiology, 1984, 38: 551-592. DOI:10.1146/annurev.mi.38.100184.003003 |
| [10] |
Muyzer G, Stams AJM. The ecology and biotechnology of sulphate-reducing bacteria[J]. Nature Reviews Microbiology, 2008, 6(6): 441-454. DOI:10.1038/nrmicro1892 |
| [11] |
Agrawal A, Lal B. Rapid detection and quantification of bisulfite reductase genes in oil field samples using real-time PCR[J]. FEMS Microbiology Ecology, 2009, 69(2): 301-312. DOI:10.1111/j.1574-6941.2009.00714.x |
| [12] |
Liu H, Yu T, Liu Y. Sulfate reducing bacteria and their activities in oil sands process-affected water biofilm[J]. Science of the Total Environment, 2015, 536: 116-122. DOI:10.1016/j.scitotenv.2015.06.135 |
| [13] |
Hindson BJ, Ness KD, Masquelier DA, et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number[J]. Analytical Chemistry, 2011, 83(2): 8604-8610. |
| [14] |
Rački N, Dreo T, Gutierrez-Aguirre I, et al. Reverse transcriptase droplet digital PCR shows high resilience to PCR inhibitors from plant, soil and water samples[J]. Plant Methods, 2014, 10(1): 42. DOI:10.1186/s13007-014-0042-6 |
| [15] |
Wu XL, Xiao L, Song Y, et al. A novel high-sensitivity droplet digital PCR (ddPCR) for detection of African swine fever virus[J]. Microbiology China, 2017, 44(12): 2839-2846. (in Chinese) 邬旭龙, 肖璐, 宋勇, 等. 非洲猪瘟病毒微滴数字PCR (ddPCR)方法的建立及应用[J]. 微生物学通报, 2017, 44(12): 2839-2846. |
| [16] |
Floren C, Wiedemann I, Brenig B, et al. Species identification and quantification in meat and meat products using droplet digital PCR (ddPCR)[J]. Food Chemistry, 2015, 173: 1054-1058. DOI:10.1016/j.foodchem.2014.10.138 |
| [17] |
Kim TG, Jeong SY, Cho KS. Comparison of droplet digital PCR and quantitative real-time PCR for examining population dynamics of bacteria in soil[J]. Applied Microbiology and Biotechnology, 2014, 98(13): 6105-6113. DOI:10.1007/s00253-014-5794-4 |
| [18] |
Ma W, Kong YJ, Wu WJ, et al. The establishment of quantitative detection of total coliforms in water based on ddPCR technology[J]. Environmental Protection Science, 2019, 45(1): 58-66. (in Chinese) 马薇, 孔义军, 吴伟健, 等. 水中大肠菌群微滴数字PCR定量检测方法的建立[J]. 环境保护科学, 2019, 45(1): 58-66. |
| [19] |
Wang GL, Li J, Wu AM, et al. A study of the thermal storage characteristics of Gaoyuzhuang Formation, a new layer system of thermal reservoir in Rongcheng uplift area, Hebei Province[J]. Acta Geoscientica Sinica, 2018, 39(5): 533-541. (in Chinese) 王贵玲, 李郡, 吴爱民, 等. 河北容城凸起区热储层新层系-高于庄组热储特征研究[J]. 地球学报, 2018, 39(5): 533-541. |
| [20] |
Escudero C, Oggerin M, Amils R. The deep continental subsurface: the dark biosphere[J]. International Microbiology, 2018, 21(1): 3-14. |
| [21] |
Whitman WB, Coleman DC, Wiebe WJ. Prokaryotes: the unseen majority[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(12): 6578-6583. DOI:10.1073/pnas.95.12.6578 |
| [22] |
Wang YL, Xia Y, Dong XZ, et al. Quantitative analysis of two uncultured bacterial species from the terrestrial deep subsurface by real-time PCR[J]. Acta Petrologica Sinica, 2006, 22(7): 2103-2106. (in Chinese) 王远亮, 夏莹, 东秀珠, 等. 中国大陆科学钻探(CCSD)深部地层微生物研究--两株未培养地下微生物菌株原位含量的分析[J]. 岩石学报, 2006, 22(7): 2103-2106. |
| [23] |
Dutta A, Dutta Gupta S, Gupta A, et al. Exploration of deep terrestrial subsurface microbiome in Late Cretaceous Deccan traps and underlying Archean basement, India[J]. Scientific Reports, 2018, 8(1): 17459. DOI:10.1038/s41598-018-35940-0 |
| [24] |
Tian HM, Gao PK, Chen ZH, et al. Compositions and abundances of sulfate-reducing and sulfur-oxidizing microorganisms in water-flooded petroleum reservoirs with different temperatures in China[J]. Frontiers in Microbiology, 2017, 8: 143. |
| [25] |
Geets J, Borremans B, Diels L, et al. DsrB gene-based DGGE for community and diversity surveys of sulfate-reducing bacteria[J]. Journal of Microbiological Methods, 2006, 66(2): 194-205. DOI:10.1016/j.mimet.2005.11.002 |
| [26] |
Wagner M, Roger AJ, Flax JL, et al. Phylogeny of dissimilatory sulfite reductases supports an early origin of sulfate respiration[J]. Journal of Bacteriology, 1998, 180(11): 2975-2982. DOI:10.1128/JB.180.11.2975-2982.1998 |
| [27] |
Pinheiro LB, Coleman VA, Hindson CM, et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification[J]. Analytical Chemistry, 2012, 84(2): 1003-1011. DOI:10.1021/ac202578x |
| [28] |
Marchesi U, Mazzara M, Broll H, et al. European Network of GMO Laboratories. Definition of minimum performance requirements for analytical methods of GMO testing[R]. Ispra Italy: JRC Science Hub, 2015
|
| [29] |
Dong LH, Zhang L, Jiang J, et al. Development of droplet digital polymerase chain reaction for quantifying Escherichia coli O157: H7[J]. Chinese Journal of Analytical Chemistry, 2015, 43(3): 319-324. (in Chinese) 董莲华, 张玲, 姜君, 等. 大肠杆菌O157: H7微滴数字PCR定量方法的建立[J]. 分析化学, 2015, 43(3): 319-324. |
| [30] |
Grelewska-Nowotko K, Żurawska-Zajfert M, Żmijewska E, et al. Optimization and verification of droplet digital PCR even-specific methods for the quantification of GM maize DAS1507 and NK603[J]. Applied Biochemistry and Biotechnology, 2018, 185(3): 207-220. |
| [31] |
Purkamo L, Bomberg M, Nyyssönen M, et al. Dissecting the deep biosphere: retrieving authentic microbial communities from packer-isolated deep crystalline bedrock fracture zones[J]. FEMS Microbiology Ecology, 2013, 85(2): 324-337. DOI:10.1111/1574-6941.12126 |
| [32] |
Kondo R, Nedwell DB, Purdy KJ, et al. Detection and enumeration of sulphate-reducing bacteria in estuarine sediments by competitive PCR[J]. Geomicrobiology Journal, 2004, 21(3): 145-157. DOI:10.1080/01490450490275307 |
| [33] |
Li ZH, Zhang S, Yin MY, et al. Distribution and ecological effects of sulfate-reducing bacteria in soil and groundwater of sewage irrigation areas in Shijiazhuang[J]. Journal of China Hydrology, 2008, 28(5): 48-51. (in Chinese) 李政红, 张胜, 殷密英, 等. 石家庄污灌区土体和地下水中硫酸盐还原菌的分布及生态效应[J]. 水文, 2008, 28(5): 48-51. |
| [34] |
Liu XZ, Zhang LM, Prosser JI, et al. Abundance and community structure of sulfate reducing prokaryotes in a paddy soil of southern China under different fertilization regimes[J]. Soil Biology and Biochemistry, 2009, 41(4): 687-694. DOI:10.1016/j.soilbio.2009.01.001 |
 2020, Vol. 47
2020, Vol. 47
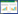


 ,
,