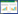扩展功能
文章信息
- 李严, 戴春晓, 杨婧, 杨冰玉, 曲媛媛
- LI Yan, DAI Chun-Xiao, YANG Jing, YANG Bing-Yu, QU Yuan-Yuan
- 吲哚的微生物代谢及其作为新型信号分子的研究进展
- Microbial metabolism of indole and its role as a novel signal molecule: a review
- 微生物学通报, 2020, 47(11): 3622-3633
- Microbiology China, 2020, 47(11): 3622-3633
- DOI: 10.13344/j.microbiol.china.191063
-
文章历史
- 收稿日期: 2019-12-19
- 接受日期: 2020-04-28
- 网络首发日期: 2020-05-26
吲哚是一种典型的氮杂环化合物,在畜牧废水和焦化废水中广泛存在,因其难以生物降解而受到广泛关注。研究表明,高浓度的吲哚可引起动物产生溶血、血红蛋白尿症、暂时性皮肤刺激、肿瘤以及抑制植物色素生成等效应[1-2]。更重要的是,大量文献表明吲哚在人体肠道与免疫系统中含量很高(约250−1 100 μmol/L),与几种人类疾病如腹泻、糖尿病等密切相关[3]。微生物能够合成、降解及转化吲哚[4],自20世纪以来,吲哚的微生物代谢就一直受到关注。近年来,相关研究证明了吲哚是一种多功能的种间交互信号分子,在微生物群落中发挥着重要作用[3]。吲哚对产吲哚和不产吲哚微生物的孢子形成、质粒稳定性、细胞分裂、抗生素耐受性和生物膜形成等均可产生一定影响[4]。因此,阐明吲哚在自然环境中的合成—转化—降解机制,了解吲哚在自然环境中的行为归趋,是揭示吲哚生态学意义的关键。本文系统地总结了吲哚的微生物代谢途径及菌株资源,介绍了其作为信号分子的重要意义,并对有关吲哚-微生物相互作用的研究趋势进行了展望。
1 吲哚的微生物代谢研究历程1866年,Baeyer在靛蓝的还原过程中首次发现了吲哚[5]。在1883年,Baeyer的学生Fischer等成功合成了吲哚,并发明了经典的Fischer吲哚合成法[6]。1897年,Simth指出大肠杆菌(Escherichia coli)和霍乱弧菌(Vibrio cholerae)能在细胞生长稳定期产生吲哚[7]。1972年,Watanabe等发现目前唯一的细菌产吲哚的途径,即色氨酸能够在色氨酸酶催化下可逆地转化为吲哚、丙酮酸和氨气[8]。2010年,Lee等报道超过85种革兰氏阴性和阳性菌可以利用色氨酸产吲哚[4]。此后,能否合成吲哚已经成为了常用的细菌生理生化指标之一。
吲哚降解的研究始于19世纪20年代,1921年,Raistrick等首次证明了绿脓杆菌(Bacillus pyocyaneus)、荧光极毛杆菌(B. fluorescens)和灵杆菌(B. prodigiosus)可以转化吲哚并释放氨气[9]。1928年,Gray等从能够氧化吲哚产生靛蓝的污泥中分离筛选出一株假单胞菌(Pseudomonas indoloxidans),但这株菌不能以吲哚为唯一碳源进行生长[10]。1953年,Sakamoto等开始详细分析吲哚的好氧降解途径[11]。2015年,Fukuoka等首次报道了贪铜杆菌(Cupriavidus) sp. KK10通过苯环开环降解吲哚的途径[12]。2017年,Sadauskas等与本课题组几乎同时报道了吲哚加氧酶基因簇[13]。同时,本课题组分别对处理市政污水或焦化废水的活性污泥在降解吲哚过程中发挥重要作用的微生物群落进行研究,结果发现微生物群落中丛毛单胞菌(Comamonas)、假单胞菌(Pseudomonas)、产碱杆菌(Alcaligenes)和伯克霍尔德菌(Burkholderia)是降解吲哚的优势菌属[14]。然而关于实际废水处理污泥中的分子机制仍需要进一步研究。
此外,菌株在降解吲哚的过程中能够转化吲哚生成一些具有高附加价值的化合物,所以近年对生物转化的关注越来越多。生物转化吲哚最早源于1928年,Gray首次报道了吲哚在污泥中转化为靛蓝的现象[10]。随后在1983年,Claus等分离得到了产碱杆菌Alcaligenes sp. In3,并找到一种新的吲哚转化中间体靛红[15]。1997年,Doukyu等报道假单胞菌P. sp. ST-200降解吲哚生成的中间体主要为靛红和靛红酸[16]。本课题组在2015年从吲哚驯化样品中获得了两株命名为IDO和SHE的贪铜杆菌(Cupriavidus)菌株,将它们分别在无机盐培养基中培养,在30 C和150 r/min条件下,它们均可以利用吲哚为唯一碳源并在1 d内完全降解100 mg/L吲哚并部分转化为靛蓝[17-18]。同年,Lin等首次证明了菌株鲍氏不动杆菌(Acinetobacter baumannii) ATCC19606可以通过将吲哚转化为无毒的靛蓝来降低吲哚的毒性效应[19]。2017年,Yang等从焦化废水中分离出一株不动杆菌A. sp. L1并鉴定出吲哚降解的中间体为4-(3-羟基-1H-吡咯-2-基)-2-氧代-丁-3-烯酸和靛红[20]。2018年,Hsu等在Nature上报道了利用葡萄糖基转移酶合成靛蓝的新途径[21]。图 1为吲哚的生物代谢研究历程。
|
| 图 1 吲哚的生物代谢研究历程 Figure 1 The research course of biological metabolism of indole |
|
|
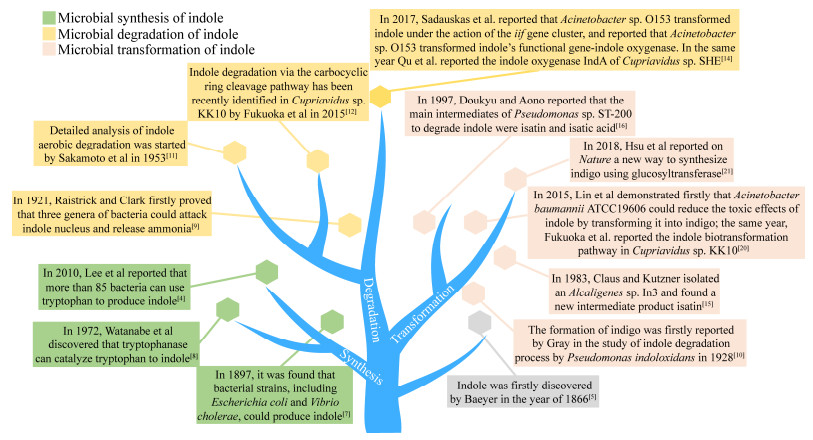
大肠杆菌(E. coli)和霍乱弧菌(V. cholerae)在LB培养基中培养时吲哚浓度可以达到0.5−0.6 mmol/L[3, 22]。吲哚的产生量受到环境和生物因素的影响,其中外源性色氨酸的浓度是最重要的因素。吲哚由色氨酸合成(图 2)。Li等发现大肠杆菌(E. coli)可以将5 mmol/L以下的色氨酸转化为等量的吲哚,转化过程主要是依赖色氨酸酶TnaA;色氨酸能够在色氨酸酶的催化下转化为吲哚、丙酮和氨气;然而,继续加入色氨酸不能使吲哚产量增加,原因可能是过量的吲哚抑制了TnaA的活性和色氨酸的转运过程;同时,较高的pH和温度可以促进吲哚的产生[23]。在pH 4.0−9.0的范围内吲哚的产量随着pH的升高而增加,这是因为较低的pH抑制了TnaA的表达;在50 ℃条件下每单位OD600的大肠杆菌细胞虽然停止了生长,但是可以产生大约7 mmol/L的吲哚,产量为37 ℃下的89倍;当大肠杆菌(E. coli)暴露在抗生素如氨苄青霉素和卡那霉素中时,吲哚的产量也有显著的增加,这说明吲哚可能影响细胞抗生素抗性[24]。然而吲哚的产生降低了产酶溶杆菌(Lysobacter enzymogenes)对氨苄青霉素、卡那霉素等多种抗生素的抗性[25]。因此,吲哚的合成在微生物中的生理意义仍值得进一步探讨。
|
| 图 2 吲哚的微生物合成—转化—降解途径汇总 Figure 2 Summary pathways of microbial synthesis—transformation—degradation of indole |
|
|
表 1中总结了吲哚降解的菌株资源及其对应的降解特性。1953年,Sakamoto等开始详细分析吲哚的好氧降解途径[11],他们发现吲哚会遵循以下途径进行降解:吲哚→吲哚酚→靛红→N-甲酰基邻氨基苯甲酸→邻氨基苯甲酸→水杨酸→邻苯二酚。之后,Fujioka等于1986年分离出一株革兰氏阳性菌并外源表达了二羟基吲哚加氧酶,重组菌可以直接催化二羟基吲哚生成邻氨基苯甲酸[35]。1983年,Claus等分离得到了产碱杆菌(Alcaligenes) sp. In3,并提出了吲哚→吲哚酚→靛红→邻氨基苯甲酸→龙胆酸的降解途径,但其中邻氨基苯甲酸被转化为龙胆酸盐的过程仍然未知[15]。1997年,Doukyu等报道称假单胞菌(Pseudomonas) sp. ST-200能够降解溶解在有机溶剂中的高浓度吲哚,并且检测到靛红和靛红酸是主要的中间产物;但是,菌株ST-200不能利用靛红作为生长底物,这表明其通过另一种降解途径转化吲哚[16]。自从2009年开始,本课题组便开始进行吲哚降解菌株的筛选工作,分别得到贪铜杆菌(Cupriavidus) sp. SHE[17]、(Cupriavidus) sp. IDO[18]、产碱杆菌(Alcaligenes) sp. YBY[36]、普罗维登斯菌(Providencia) sp. DCX[37]等菌株,它们能够高效降解吲哚,通过对吲哚的降解途径进行总结,我们发现靛红和邻氨基苯甲酸是它们降解过程中的关键产物[14]。然而,对不动杆菌(Alcaligenes) sp. O153的研究表明,邻氨基苯甲酸可以不通过靛红直接由二羟基吲哚或3-羟基-吲哚啉-2-酮形成[13]。此外,有报道称贪铜杆菌(Cupriavidus) sp. KK10和不动杆菌(Alcaligenes) sp. L1通过苯环裂解途径来降解吲哚[12, 20],该途径的具体分子机制仍需要进一步研究。
| 微生物 Microbial |
降解特性 Degradation characteristic |
参考文献 references |
| Bacillus spp. | 首次报道能够降解吲哚 First report on indole degradation |
[9] |
| Pseudomonas indoloxidans | 能够在降解吲哚的过程中产生靛蓝 Production of indigo in indole degradation |
[10] |
| Alcaligenes faecalis IITR89 | [26] | |
| Alcaligenes sp. In3 | 好氧降解,靛红为降解中间体 Aerobic degradation, isatin is an intermediate product |
[15] |
| Pseudomonas sp. ST-200 | [16] | |
| Alcaligenes sp. B5 | [27] | |
| Arthrobacter sp. B1 | [27] | |
| Acinetobacter pittii L1 | [20] | |
| Cupriavidus sp. IDO | 高效降解吲哚 Degradation of indole rapidly |
[18] |
| Pseudomonas aeruginosa | [28] | |
| Acinetobacter baumannii ATCC19606 | 首次报道了iif基因簇 Identification of an iif gene cluster for the first time |
[19] |
| Acinetobacter sp. O153 | 鉴定iif基因簇的功能 Identification of the function of the iif gene cluster |
[13] |
| Agrobacterium tumefaciens | 高效降解吲哚,能够转化色氨酸为吲哚-3-乙酸 Degradation of indole rapidly, transforming tryptophan to indole-3-acetic acid |
[3] |
| Acinetobacter sp. SPG | 产生吲哚-3-乙酸、吲哚-3-乙醛酸和吲哚-3-甲醛Production of indole-3-acetic acid, indole-3-glyoxylic acid, and indole-3-aldehyde | [29] |
| Burkholderia unamae CK43B | 能够在邻苯三酚型多酚存在下降解吲哚 Degradation of indole in the presence of pyrogallol-type polyphenols |
[30] |
| Cupriavidus sp. KK10 | 首次报道吲哚苯环裂解途径 First report of indole carbocyclic-aromatic ring cleavage pathway |
[12] |
| Cupriavidus sp. SHE | 鉴定ind基因簇的功能Identification the function of ind gene cluster | [17] |
| Pseudomonas sp. Gs | 降解过程产生两种新的代谢产物Production of two novel metabolites | [31] |
| Thauera sp. Q4/Q20-C/3–35 | 能够好氧降解吲哚、苯酚和甲酚 Degradation of indole, phenol, and methylphenol under aerobic conditions |
[32] |
| Desulfobacterium indolicum In04 | 厌氧降解吲哚 Degradation of indole under anoxic condition |
[33] |
| P. sp. LPA11/LPB11/LPC24 | [34] |
关于吲哚降解或转化的分子机制研究,前期多集中于其他芳烃加氧酶羟化吲哚的过程。直到2017年,Sadauskas等[13]首次报道了不动杆菌(Alcaligenes) sp. O153中的吲哚加氧酶;在吲哚加氧酶(iif基因簇)的作用下,吲哚代谢的第一步开始进行,吲哚加氧酶中的两个组分IifC和IifD氧化吲哚生成吲哚-2, 3-二氢二醇,该物质能够继续被氧化生成吲哚酮或在空气中自发合成靛蓝,同时也能够在脱氢酶IifB的作用下进一步生成3-羟基-吲哚啉-2-酮,进而在氧化还原酶IifA的作用下生成邻氨基苯甲酸。同时,2017年本课题组也从土壤样品中筛选得到一株吲哚高效降解菌(Cupriavidus) sp. SHE,通过对菌株SHE的基因组结合比较蛋白组技术研究了吲哚代谢过程中菌株SHE显著变化的蛋白;结合产物分析和基因分析的结构,本课题组首次在分子水平上揭示了贪铜杆菌属代谢吲哚的机制:吲哚首先在基因簇I中的吲哚加氧酶IndA作用下生成吲哚酚,吲哚酚会继续氧化生成靛红,靛红会发生水解反应,也会继续生成典型的中间产物邻氨基苯甲酸,邻氨基苯甲酸在基因簇Ⅱ的作用下生成邻氨基苯甲酸辅酶A,进入β氧化途径[38]。图 2对已报道的吲哚降解途径进行了总结。
吲哚厌氧降解研究主要集中在微生物群落上,初步研究可追溯到20世纪80年代。Balba等在1980年研究了色氨酸的产甲烷发酵过程,研究中发现吲哚是关键中间体,能够进一步转化为邻氨基苯甲酸、水杨酸和苯甲酸[39]。Wang等在1984年的研究中表明吲哚可以在厌氧条件下被降解生成甲烷和二氧化碳[40]。厌氧降解吲哚发生在产甲烷和硫酸盐还原条件下,1987年,Berry等用接种污泥的培养液证实了吲哚可以在产甲烷条件下被降解[41]。Shanker等在硫酸盐还原条件下也观察到吲哚完全降解的现象,且该途径与产甲烷过程较为相似[42]。相比而言,吲哚的好氧和厌氧降解途径具有一定的相似性,吲哚的羟基化过程均被认为是其代谢的关键一步;对于厌氧和好氧最本质的区别在于好氧条件下吲哚首先在2号碳或3号碳或者同时在2号碳、3号碳位置被氧化,而在厌氧条件下吲哚通常在2号碳位羟基化从而产生2-羟基吲哚,由此推测,吲哚厌氧降解的功能酶不同于好氧降解,厌氧酶选择性地攻击2号碳而不是3号碳[43]。
2.3 生物转化途径在吲哚的转化过程中发现了很多具有高附加价值的中间产物,如靛红、靛玉红以及靛蓝等。其中,靛蓝是最古老的颜料之一,被广泛用应于染色、食品、制药、半导体等行业。靛蓝最初是从植物中提取而来的,自从Baeyer等首次在实验室中成功合成靛蓝后,化学合成靛蓝很快占据了主导地位[44]。然而化学合成过程中不可避免地会使用有毒化学物质(如苯胺、亚硫酸钠等),会腐蚀设备并造成环境问题。然而微生物合成靛蓝具有环境友好、反应条件温和等特点,在过去几十年中引起了极大的关注。吲哚酚是靛蓝的前体物质,因此吲哚氧化生成吲哚酚是生物合成靛蓝的关键步骤。图 2对吲哚的转化途径进行了总结。据报道,许多加氧酶都能够氧化吲哚,这些酶主要分为双加氧酶和单加氧酶。典型的加氧酶包括萘双加氧酶[45](naphthalene dioxygenase,NDO)、多组分苯酚羟化酶[46](multicomponent phenol hydroxylase,mPH)、细胞色素P450单加氧酶[47]和黄素单加氧酶[48](flavin monooxygenase,FMO)等。表 2中总结了转化吲哚的微生物及酶资源。虽然微生物法可以生产高纯度靛蓝,但分析表明,生物法产靛蓝的生产效率仍远低于化学法[61]。因此,如何提高靛蓝产量、减少生物合成靛蓝的经济成本值得进一步研究。本课题组筛选得到了一些能够将吲哚转化为靛蓝的菌株,如丛毛单胞菌(Comamonas) sp. MQ[62]、丛毛单胞菌(Comamonas) sp. IDO2[63]、伯克霍尔德菌(Burkholderia) sp. IDO3[64],并对固定化联苯双加氧酶产靛蓝进行了一定的研究[65]。随后本课题组从丛毛单胞菌(Comamonas) sp. MQ中获取了ndo基因并重组表达,发现重组菌能够在不同底物的催化下产生靛蓝类色素[51]。
| 酶 enzyme |
微生物 Microorganisms |
参考文献 References |
| 萘双加氧酶 Naphthalene dioxygenase |
Pseudomonas putida PpG7 | [45] |
| Pseudomonas sp. NCIB9816-4 | [49] | |
| Pseudomonas putida RKJ1 | [50] | |
| Comamonas sp. MQ | [51] | |
| 苯酚羟化酶 Phenol hydroxylase |
Acinetobacter sp. ST-550 | [52] |
| Pseudomonas sp. KL33 | [53] | |
| Pseudomonas sp. KL28 | [53] | |
| Arthrobacter sp. W1 | [54] | |
| 细胞色素P450单加氧酶 Cytochrome P450 monooxygenase |
Bacillus megaterium | [55-56] |
| 苯乙烯单加氧酶 Styrene monoxygenase |
Pseudomonas putida S12 | [57] |
| Pseudomonas putida CA-3 | [57] | |
| Pseudomonas putida B4 | [58] | |
| 黄素单加氧酶 Flavin monooxygenase |
Methylophaga aminisulfidivorans MPT | [48, 59] |
| Corynebacterium glutamicum ATCC13032 | [60] | |
| 吲哚加氧酶/IifC Indole oxygenase/IifC |
Acinetobacter baumannii ATCC19606 | [19] |
| Acinetobacter sp. O153 | [13] | |
| 吲哚加氧酶/IndA Indole oxygenase/IndA |
Cupriavidus sp. SHE | [38] |
NDO是吲哚转化为靛蓝领域报道最多的加氧酶。1983年,Ensley等首次从菌株恶臭假单胞菌(P. putida) PpG7中成功克隆了NDO基因,并在大肠杆菌中表达,得到的重组菌株能够合成大量靛蓝,但是仍然达不到工业化应用的标准[45]。随后在2002年,为了提高生物靛蓝的产量,Berry等基于代谢工程优化了发酵过程,成功研发出一株靛蓝高产量菌株,命名为FM5/pBMW, pTacFd911[66]。除了吲哚外,NDO被报道可以将各种吲哚衍生物转化为不同的靛蓝类色素,包括甲基吲哚、甲氧基吲哚、硝基吲哚、氯吲哚和溴吲哚[49, 51]。
相比于NDO,细胞色素P450酶能够催化吲哚转化生成更多新的产物。2000年,Gillam等报道了P450 2A6在吲哚生物转化中具有很好的活性,并且可以产生靛蓝、靛玉红和新产物6H-恶唑并[3, 2-a:4, 5-b′]二吲哚[47]。2010年,Hu等通过随机诱变和饱和诱变得到了几株具有更高吲哚氧化性能的突变体,突变体D168W可以通过P450 BM3酶产生靛玉红,为靛玉红生物生产提供了新的途径[56]。
1990年,mPH在假单胞菌Pseudomonas sp. CF600降解苯酚和2-甲基苯酚的过程中被发现,该种酶由基因簇dmpKLMNOP编码[46]。由mPH主导的羟化吲哚的位点主要包含3种,即2号碳、3号碳和7号碳[55]。据报道,许多其他苯酚降解菌,包括假单胞菌(Pomamonas) sp. KL33、(Pomamonas) sp. CF600和(Pomamonas) sp. QM等能将吲哚转化为深紫色或红紫色产物;鉴定中发现紫色产物可能含有2-[7-氧代-1H-吲哚-6(7H)-亚基]二氢吲哚-3-酮,该物质可能通过吲哚酮和7-羟基吲哚转化合成[46, 53, 67]。本课题组在前期研究中,通过表达节杆菌(Arthrobacter) sp. W1中的mPH,成功将吲哚转化为3种不同产物,即靛蓝、靛玉红和2-[7-氧代-1H-吲哚-6(7H)-亚基]二氢吲哚-3-酮[54]。
此外,2003年,在菌株M. aminisulfidivorans MPT中发现了一种新酶——FMO;表达fmo因的重组大肠杆菌可在色氨酸培养基中产生160 mg/L的靛蓝[48]。另外,Han等[59]发现添加半胱氨酸可以诱导导入fmo的大肠杆菌(E. coli)转化吲哚生成2-羟基吲哚,显著增加了靛玉红的产量,这一发现为工业上生产靛玉红提供了思路。
3 吲哚作为新型种间及跨界信号分子的研究进展生态系统中微生物都以复杂的群落形式存在,而在群落中各种微生物可通过物理、化学等不同的方式进行信息的交流,有研究发现部分微生物能够合成小分子化合物作为信号分子进行信息交流,以便于更好地适应外界环境的变化[4]。除了微生物,玉米等植物能够合成和利用吲哚来抵御害虫的侵袭,而蝴蝶和蚊虫等动物也能够感知吲哚来调节自己的产卵行为[68]。此外,吲哚已被证实具有提升肠道上皮细胞功能、降低肠道炎症的发生、调节肠促胰岛素分泌以及保持动物年轻健康的作用,其在人类健康与疾病治疗中的潜力已受到大量关注[69-71]。近年来,除了研究较多的酰基高丝氨酸内酯、寡肽类分子及AI-2信号分子等物质,吲哚也被证实了能够作为种间及跨界信号分子,在信号调节中具有重要作用。对吲哚作为信号分子的研究最早可以追溯到1993年,Gerth等首次提出吲哚可作为标桩菌(Stigmatella aurantiaca)菌属的自诱导剂[72],随后的研究确认了吲哚为激活astD、gabT和tnaB所需的细胞外信号分子[73]。早年间的研究结果对吲哚是否为细胞间信号分子一直存在争议,然而,最近的研究已经证明了吲哚满足细胞-细胞信号分子的4个标准[74],表 3列出了信号分子的判定标准以及吲哚作为信号分子的依据。
| 信号分子判定标准 judgement standard of signal molecule |
吲哚的判定依据 Judgement basis for indole |
| 信号分子在特定阶段出现 Signal molecules are produced at specific stages |
吲哚主要在对数生长期产生,在稳定期大量累积 Indole mainly occurs in the logarithmic phase and accumulates in a large amount during the stationary phase |
| 信号分子必须在细胞外累积并被特定感受器识别 Signal molecules must accumulate outside the cell and be recognized by specific receptors |
吲哚具有疏水性,能扩散穿过细胞膜,AcrEF、Mtr蛋白分别负责吲哚的进入与外排 Indole is hydrophobic and can diffuse across cell membrane. AcrEF and Mtr proteins are responsible for indole entry and efflux, respectively |
| 信号分子必须具有累积效应并调控某一生理活动 Signal molecules must have an accumulation effect and regulate a certain physiological activity |
吲哚可延迟细胞分裂,影响抗生素抗性等 Indole can delay cell division and affect antibiotic resistance, etc |
| 信号分子必须引起超出代谢或解毒信号所需的生理变化 The signaling molecule must cause physiological changes beyond what is required for metabolic or detoxification signals |
吲哚可控制生物膜的形成与细胞分裂等 Indole can control biofilm formation and cell division, etc |
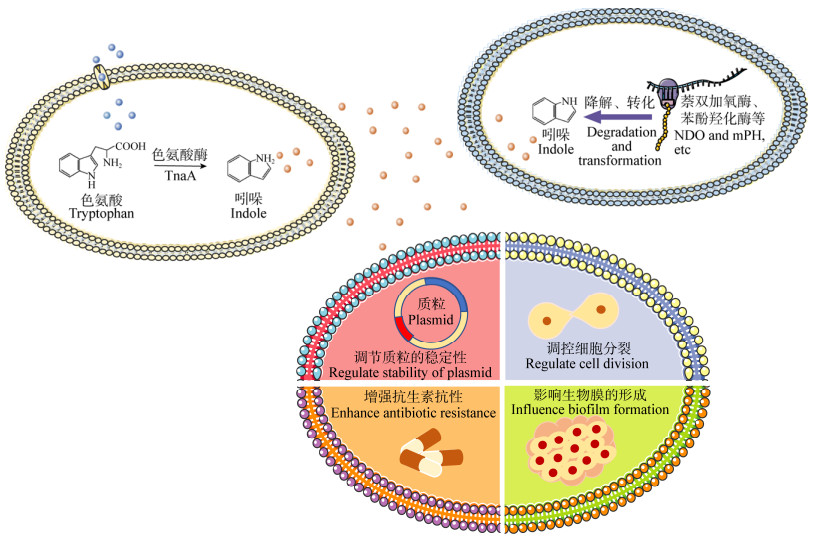
吲哚作为新型种间及跨界信号分子,在微生物群落物种间相互作用方面具有重要意义。目前Lee和Woods团队针对吲哚信号分子开展了一系列研究[4, 13]。研究表明,吲哚能够调节大肠杆菌(E. coli)中质粒的稳定性、调控细胞分裂、增强大肠杆菌(E. coli)抗生素抗性、降低大肠杆菌(E. coli)毒性,还能够影响大肠杆菌(E. coli)和霍乱弧菌(V. cholerae)生物膜的形成等;此外,吲哚能够降低致病菌鳗弧菌(V. anguillarum)的毒性、增强致病菌迟缓爱德华菌(Edwardsiella tarda)的抗生素抗性[68]。吲哚在吲哚产生菌中的含量十分稳定,而一些非吲哚产生菌则会编码萘双加氧酶、P450单加氧酶和苯酚羟化酶等加氧酶来转化或者降解吲哚,如图 3所示。吲哚能够控制非吲哚产生菌的多种生理活动,如抑制细菌群体感应、细胞膜的生成以及毒性蛋白的表达等[74]。研究证明,大肠杆菌(E. coli) O157:H7分泌的大量吲哚会被其他菌株如洋葱伯克霍尔德菌(B. cepacian) G4羟化生成羟基吲哚,然后反向调控大肠杆菌(E. coli) O157:H7生物膜的形成[4]。然而,吲哚的受体系统及吲哚在微生物群体协调行为中的作用仍需进一步研究。目前部分学者推测吲哚产生菌通过分泌吲哚来减弱其他细菌或真核生物的竞争,而非吲哚产生菌则可以通过降解吲哚来抵御这种影响。但吲哚在不同菌属间作为信号分子的生态学机制尚不完善,尤其是吲哚在其产生菌及非产生菌之间发挥信号分子作用的调节机制仍需深入研究。
|
| 图 3 吲哚作为引号分子的功能概述 Figure 3 Overview of the function of indole as a signal molecule |
|
|
吲哚作为种间及跨界信号分子的功能一直是近年来的研究热点,尤其是对细菌耐药性和毒性影响的研究[4, 14]。微生物、动植物以及人类活动都可以产生大量的吲哚并释放到环境中,但是吲哚与微生物、动植物之间的相互作用机制仍未被揭示。因此,未来对吲哚可在以下几个方面进行更深层次的研究:
(1) 吲哚微生物降解的分子机制仍需进一步探究。除吲哚加氧酶外,在一些细菌中专用于吲哚氧化的酶仍然未知,且吲哚的下游降解机制及相关酶功能仍需深入探索。此外,厌氧条件下的吲哚降解机制也值得更深层次的研究。结合分子生物学手段对未知的功能酶进行表达和功能测定是接下来吲哚降解机制解析研究的重点与难点。
(2) 吲哚对人体健康的影响。吲哚通过微生物转化食物中的色氨酸而得到,动物和人类肠道中富含吲哚。有研究证明,吲哚会对某些疾病产生影响,如炎症性肠病和糖尿病[69, 71]。但有关吲哚对人体健康的影响仍需证实。因此,以毒理学评价模型探究吲哚对于人类健康的风险是接下来研究需要关注的重点。
(3) 废水中吲哚的处理。生活污水及工业废水中都存在吲哚类物质,因此,吲哚类废水的处理工艺可以结合微生物代谢绿色无害等优势,针对氮杂环化合物进行细化调控。同时,废水中的吲哚作为信号分子具有群感淬灭的效应,因此,其对于实际活性污泥及膜生物处理体系的作用仍需要进一步研究。
(4) 后续研究可以进一步借助快速发展的高通量测序、基因组、蛋白组及代谢组等组学技术对微生物转化吲哚的机制进行更深入、更详细的阐述,也进一步从分子以及个体层面上对吲哚这一新型信号分子的作用方式及机理进行更全面细致的解析。
| [1] |
Collin G, Höke H.Indole[A]//Ullmann's Encyclopedia of Industrial Chemistry[M].Washington: John Wiley & Sons, 2000, 19: 75-77
|
| [2] |
Arora PK, Sharma A, Bae H. Microbial degradation of indole and its derivatives[J]. Journal of Chemistry, 2015, 2015: 129159. |
| [3] |
Lee JH, Kim YG, Baek KH, et al. The multifaceted roles of the interspecies signalling molecule indole in Agrobacterium tumefaciens[J]. Environmental Microbiology, 2015, 17(4): 1234-1244. DOI:10.1111/1462-2920.12560 |
| [4] |
Lee JH, Lee J. Indole as an intercellular signal in microbial communities[J]. FEMS Microbiology Reviews, 2010, 34(4): 426-444. DOI:10.1111/j.1574-6976.2009.00204.x |
| [5] |
Baeyer A. Ueber die reduction aromatischer Verbindungen mittelst zinkstaub[J]. European Journal of Organic Chemistry, 1866, 140(3): 295-296. |
| [6] |
Fischer E, Jourdan F. Ueber die hydrazine der brenztraubensäure[J]. Berichte der Deutschen Chemischen Gesellschaft, 1883, 16(2): 2241-2245. DOI:10.1002/cber.188301602141 |
| [7] |
Smith T. A modification of the method for determining the production of indol by bacteria[J]. The Journal of Experimental Medicine, 1897, 2(5): 543-547. DOI:10.1084/jem.2.5.543 |
| [8] |
Watanabe T, Snell EE. Reversibility of the tryptophanase reaction:synthesis of tryptophan from indole, pyruvate, and ammonia[J]. Proceedings of the National Academy of Sciences of the United States of America of the United States of America, 1972, 69(5): 1086-1090. DOI:10.1073/pnas.69.5.1086 |
| [9] |
Raistrick H, Clark AB. Studies on the cycloclastic power of bacteria:Part Ⅱ.A quantitative study of the aerobic decomposition of tryptophan and tyrosine by bacteria[J]. Biochemical Journal, 1921, 15(1): 76-82. |
| [10] |
Gray PHH. The formation of indigotin from indole by soil bacteria[J]. Proceedings of the Royal Society B:Biological Sciences, 1928, 102(717): 263-280. |
| [11] |
Sakamoto Y, Uchida M, Ichihara K. The bacterial decomposition of indole.I.Studies on its metabolic pathway by successive adaptation[[J]. Medical Journal of Osaka University, 1953, 3: 477-486. |
| [12] |
Fukuoka K, Tanaka K, Ozeki Y, et al. Biotransformation of indole by Cupriavidus sp.strain KK10 proceeds through N-heterocyclic-and carbocyclic-aromatic ring cleavage and production of indigoids[J]. International Biodeterioration & Biodegradation, 2015, 97: 13-24. |
| [13] |
Sadauskas M, Vaitekūnas J, GasparavičiūtėR, et al. Indole biodegradation in Acinetobacter sp.strain O153:genetic and biochemical characterization[J]. Applied and Environmental Microbiology, 2017, 83(19): e01453-17. DOI:10.1128/AEM.01453-17 |
| [14] |
Ma Q, Zhang XW, Qu YY. Biodegradation and biotransformation of indole:advances and perspectives[J]. Frontiers in Microbiology, 2018, 9: 2625. DOI:10.3389/fmicb.2018.02625 |
| [15] |
Claus G, Kutzner HJ. Degradation of indole by Alcaligenes spec[J]. Systematic and Applied Microbiology, 1983, 4(2): 169-180. DOI:10.1016/S0723-2020(83)80046-0 |
| [16] |
Doukyu N, Aono R. Biodegradation of indole at high concentration by persolvent fermentation with Pseudomonas sp.ST-200[J]. Extremophiles, 1997, 1(2): 100-105. DOI:10.1007/s007920050021 |
| [17] |
Qu YY, Shen E, Ma Q, et al. Biodegradation of indole by a newly isolated Cupriavidus sp.SHE[J]. Journal of Environmental Sciences, 2015, 34: 126-132. DOI:10.1016/j.jes.2015.01.023 |
| [18] |
Ma Q, Qu YY, Zhang ZJ, et al. Genome sequence of an efficient indole-degrading bacterium, Cupriavidus sp.strain IDO, with potential polyhydroxyalkanoate production applications[J]. Genome Announcements, 2015, 3(2): e00102-15. DOI:10.1128/genomeA.00102-15 |
| [19] |
Lin GH, Chen HP, Shu HY. Detoxification of indole by an indole-induced flavoprotein oxygenase from Acinetobacter baumannii[J]. PLoS One, 2015, 10(9): e0138798. DOI:10.1371/journal.pone.0138798 |
| [20] |
Yang ZY, Zhou JH, Xu YB, et al. Analysis of the metabolites of indole degraded by an isolated Acinetobacter pittii L1[J]. BioMed Research International, 2017, 2017: 2564363. |
| [21] |
Hsu TM, Welner DH, Russ ZN, et al. Employing a biochemical protecting group for a sustainable indigo dyeing strategy[J]. Nature Chemical Biology, 2018, 14(3): 256-261. DOI:10.1038/nchembio.2552 |
| [22] |
Kobayashi A, Hirakawa H, Hirata T, et al. Growth phase-dependent expression of drug exporters in Escherichia coli and its contribution to drug tolerance[J]. Journal of Bacteriology, 2006, 188(16): 5693-5703. DOI:10.1128/JB.00217-06 |
| [23] |
Li G, Young KD. Indole production by the tryptophanase TnaA in Escherichia coli is determined by the amount of exogenous tryptophan[J]. Microbiology, 2013, 159(2): 402-410. |
| [24] |
Han TH, Lee JH, Cho MH, et al. Environmental factors affecting indole production in Escherichia coli[J]. Research in Microbiology, 2011, 162(2): 108-116. DOI:10.1016/j.resmic.2010.11.005 |
| [25] |
Han Y, Wang Y, Yu YM, et al. Indole-induced reversion of intrinsic multiantibiotic resistance in Lysobacter enzymogenes[J]. Applied and Environmental Microbiology, 2017, 83(17): e00995-17. DOI:10.1128/AEM.00995-17 |
| [26] |
Regar RK, Gaur VK, Mishra G, et al. Draft genome sequence of Alcaligenes faecalis strain IITR89, an indole-oxidizing bacterium[J]. Genome Announcements, 2016, 4(2): e00067-16. DOI:10.1128/genomeA.00067-16 |
| [27] |
Kim M, Lee JH, Kim E, et al. Isolation of indole utilizing bacteria Arthrobacter sp.and Alcaligenes sp.from livestock waste[J]. Indian Journal of Microbiology, 2016, 56(2): 158-166. DOI:10.1007/s12088-016-0570-z |
| [28] |
Lee J, Attila C, Cirillo SLG, et al. Indole and 7-hydroxyindole diminish Pseudomonas aeruginosa virulence[J]. Microbial Biotechnology, 2009, 2(1): 75-90. |
| [29] |
Arora PK, Bae H. Identification of new metabolites of bacterial transformation of indole by gas chromatography-mass spectrometry and high performance liquid chromatography[J]. International Journal of Analytical Chemistry, 2014, 2014: 239641. |
| [30] |
Kim D, Rahman A, Sitepu IR, et al. Accelerated degradation of exogenous indole by Burkholderia unamae strain CK43B exposed to pyrogallol-type polyphenols[J]. Bioscience, Biotechnology, and Biochemistry, 2013, 77(8): 1722-1727. DOI:10.1271/bbb.130282 |
| [31] |
Yin B, Gu JD, Wan NS. Degradation of indole by enrichment culture and Pseudomonas aeruginosa Gs isolated from mangrove sediment[J]. International Biodeterioration & Biodegradation, 2005, 56(4): 243-248. |
| [32] |
Mao YJ, Zhang XJ, Xia X, et al. Versatile aromatic compound-degrading capacity and microdiversity of Thauera strains isolated from a coking wastewater treatment bioreactor[J]. Journal of Industrial Microbiology & Biotechnology, 2010, 37(9): 927-934. |
| [33] |
Bak F, Widdel F. Anaerobic degradation of indolic compounds by sulfate-reducing enrichment cultures, and description of Desulfobacterium indolicum gen.nov., sp.nov[J]. Archives of Microbiology, 1986, 146(2): 170-176. DOI:10.1007/BF00402346 |
| [34] |
Li P, Tong L, Liu K, et al. Indole degrading of ammonia oxidizing bacteria isolated from swine wastewater treatment system[J]. Water Science and Technology, 2009, 59(12): 2405-2410. DOI:10.2166/wst.2009.312 |
| [35] |
Fujioka M, Wada H. The bacterial oxidation of indole[J]. Biochimica et Biophysica Acta, 1968, 158(1): 70-78. DOI:10.1016/0304-4165(68)90073-1 |
| [36] |
Yang BY, Lin YX, Dai CX, et al. Characterization of an indole-degrading bacterium Alcaligenes sp.YBY[J]. Environmental Science & Technology, 2018, 41(12): 1-6. (in Chinese) 杨冰玉, 林宇星, 戴春晓, 等. Alcaligenes sp.YBY降解吲哚的特性研究[J]. 环境科学与技术, 2018, 41(12): 1-6. |
| [37] |
Dai CX, Yang J, Fang H, et al. Degradation characteristics of indole and biosynthesis of indigo by a novel bacteria Providencia sp[J]. Acta Scientiae Circumstantiae, 2018, 38(9): 3590-3596. (in Chinese) 戴春晓, 杨婧, 房皓, 等. 一株新颖的Providencia sp.菌降解吲哚及合成靛蓝的特性研究[J]. 环境科学学报, 2018, 38(9): 3590-3596. |
| [38] |
Qu YY, Ma Q, Liu ZY, et al. Unveiling the biotransformation mechanism of indole in a Cupriavidus sp.strain[J]. Molecular Microbiology, 2017, 106(6): 905-918. DOI:10.1111/mmi.13852 |
| [39] |
Balba MT, Evans WC. Methanogenic fermentation of the naturally occurring aromatic amino acids by a microbial consortium[J]. Biochemical Society Transactions, 1980, 8(5): 625-627. DOI:10.1042/bst0080625 |
| [40] |
Wang YT, Suidan MT, Pfeffer JT. Anaerobic biodegradation of indole to methane[J]. Applied and Environmental Microbiology, 1984, 48(5): 1058-1060. DOI:10.1128/AEM.48.5.1058-1060.1984 |
| [41] |
Berry DF, Madsen EL, Bollag JM. Conversion of indole to oxindole under methanogenic conditions[J]. Applied and Environmental Microbiology, 1987, 53(1): 180-182. |
| [42] |
Shanker R, Bollag JM. Transformation of indole by methanogenic and sulfate-reducing microorganisms isolated from digested sludge[J]. Microbial Ecology, 1990, 20(1): 171-183. DOI:10.1007/BF02543875 |
| [43] |
Madsen EL, Francis AJ, Bollag JM. Environmental factors affecting indole metabolism under anaerobic conditions[J]. Applied and Environmental Microbiology, 1988, 54(1): 74-78. |
| [44] |
Huxtable RJ. The mutability of blue[J]. Molecular Interventions, 2001, 1(3): 141-144. |
| [45] |
Ensley BD, Ratzkin BJ, Osslund TD, et al. Expression of naphthalene oxidation genes in Escherichia coli results in the biosynthesis of indigo[J]. Science, 1983, 222(4620): 167-169. DOI:10.1126/science.6353574 |
| [46] |
Nordlund I, Powlowski J, Shingler V. Complete nucleotide sequence and polypeptide analysis of multicomponent phenol hydroxylase from Pseudomonas sp.strain CF600[J]. Journal of Bacteriology, 1990, 172(12): 6826-6833. DOI:10.1128/JB.172.12.6826-6833.1990 |
| [47] |
Gillam EMJ, Notley LM, Cai HL, et al. Oxidation of indole by cytochrome P450 enzymes[J]. Biochemistry, 2000, 39(45): 13817-13824. DOI:10.1021/bi001229u |
| [48] |
Choi HS, Kim JK, Cho EH, et al. A novel flavin-containing monooxygenase from Methylophaga sp.strain SK1 and its indigo synthesis in Escherichia coli[J]. Biochemical and Biophysical Research Communications, 2003, 306(4): 930-936. DOI:10.1016/S0006-291X(03)01087-8 |
| [49] |
Kim JY, Lee K, Kim Y, et al. Production of dyestuffs from indole derivatives by naphthalene dioxygenase and toluene dioxygenase[J]. Letters in Applied Microbiology, 2003, 36(6): 343-348. DOI:10.1046/j.1472-765X.2003.01279.x |
| [50] |
Bhushan B, Samanta SK, Jain RK. Indigo production by naphthalene-degrading bacteria[J]. Letters in Applied Microbiology, 2000, 31(1): 5-9. |
| [51] |
Zhang XW, Qu YY, Ma Q, et al. Cloning and expression of naphthalene dioxygenase genes from Comamonas sp.MQ for indigoids production[J]. Process Biochemistry, 2013, 48(4): 581-587. DOI:10.1016/j.procbio.2013.02.008 |
| [52] |
Doukyu N, Toyoda K, Aono R. Indigo production by Escherichia coli carrying the phenol hydroxylase gene from Acinetobacter sp.strain ST-550 in a water-organic solvent two-phase system[J]. Applied Microbiology and Biotechnology, 2003, 60(6): 720-725. DOI:10.1007/s00253-002-1187-1 |
| [53] |
Kim JY, Kim JK, Lee SO, et al. Multicomponent phenol hydroxylase-catalysed formation of hydroxyindoles and dyestuffs from indole and its derivatives[J]. Letters in Applied Microbiology, 2005, 41(2): 163-168. DOI:10.1111/j.1472-765X.2005.01734.x |
| [54] |
Qu YY, Shi SN, Zhou H, et al. Characterization of a novel phenol hydroxylase in indoles biotranformation from a strain Arthrobacter sp.W1[J]. PLoS One, 2012, 7(9): e44313. DOI:10.1371/journal.pone.0044313 |
| [55] |
Lu Y, Mei LH. Co-expression of P450 BM3 and glucose dehydrogenase by recombinant Escherichia coli and its application in an NADPH-dependent indigo production system[J]. Journal of Industrial Microbiology & Biotechnology, 2007, 34(3): 247-253. |
| [56] |
Hu S, Huang J, Mei LH, et al. Altering the regioselectivity of cytochrome P450 BM-3 by saturation mutagenesis for the biosynthesis of indirubin[J]. Journal of Molecular Catalysis B:Enzymatic, 2010, 67(1/2): 29-35. |
| [57] |
O'Connor KE, Dobson AD, Hartmans S. Indigo formation by microorganisms expressing styrene monooxygenase activity[J]. Applied Microbiology and Biotechnology, 1997, 63(11): 4287-4291. |
| [58] |
Cheng L, Yin S, Chen M, et al. Enhancing indigo production by over-expression of the styrene monooxygenase in Pseudomonas putida[J]. Current Microbiology, 2016, 73(2): 248-254. DOI:10.1007/s00284-016-1055-3 |
| [59] |
Han GH, Gim GH, Kim W, et al. Enhanced indirubin production in recombinant Escherichia coli harboring a flavin-containing monooxygenase gene by cysteine supplementation[J]. Journal of Biotechnology, 2013, 164(2): 179-187. |
| [60] |
America SPL, Jung HS, Kim HS, et al. Characterization of a flavin-containing monooxygenase from Corynebacterium glutamicum and its application to production of indigo and indirubin[J]. Biotechnology Letters, 2015, 37(8): 1637-1644. DOI:10.1007/s10529-015-1824-2 |
| [61] |
Philp JC, Ritchie RJ, Allan JEM. Biobased chemicals:the convergence of green chemistry with industrial biotechnology[J]. Trends in Biotechnology, 2013, 31(4): 219-222. DOI:10.1016/j.tibtech.2012.12.007 |
| [62] |
Li SY, Yuan Q, Huang YY, et al. Biosynthesis of indigo by naphthalene-degrading strain Comamonas sp.MQ[J]. Acta Scientiae Circumstantiae, 2012, 32(8): 1807-1813. (in Chinese) 李诗阳, 袁琪, 黄益养, 等. 萘降解菌MQ合成靛蓝的研究[J]. 环境科学学报, 2012, 32(8): 1807-1813. |
| [63] |
You SN, Shen WL, Pei XF, et al. Characteristics of indigo biosynthesis by Comamonas sp.IDO2[J]. Acta Scientiae Circumstantiae, 2017, 37(6): 2069-2075. (in Chinese) 由胜男, 沈文丽, 裴晓芳, 等. Comamonas sp.IDO2合成靛蓝的特性研究[J]. 环境科学学报, 2017, 37(6): 2069-2075. DOI:10.13671/j.hjkxxb.2016.0419 |
| [64] |
Liu ZY, Ma Q, You SN, et al. Cloning, expression and application of an indigo-producing gene from Burkhoideria sp.IDO3[J]. Microbiology China, 2017, 44(11): 2634-2643. (in Chinese) 刘紫嫣, 马桥, 由胜男, 等. Burkholderia sp.IDO3中靛蓝合成基因的克隆表达及其合成特性[J]. 微生物学通报, 2017, 44(11): 2634-2643. |
| [65] |
Qu YY, Xu BW, Cong P, et al. Research on characteristics of biotransformation of indigo by immobilized whole cell expressing biphenyl dioxygenase in biphasic system[J]. Journal of Dalian University of Technology, 2013, 53(3): 340-345. (in Chinese) 曲媛媛, 许炳雯, 丛培, 等. 两相体系中固定化联苯双加氧酶全细胞产靛蓝特性研究[J]. 大连理工大学学报, 2013, 53(3): 340-345. |
| [66] |
Berry A, Dodge TC, Pepsin M, et al. Application of metabolic engineering to improve both the production and use of biotech indigo[J]. Journal of Industrial Microbiology and Biotechnology, 2002, 28(3): 127-133. DOI:10.1038/sj.jim.7000228 |
| [67] |
Ma Q, Qu YY, Tang HZ, et al. Genome sequence of a novel indigo-producing strain, Pseudomonas monteilii QM[J]. Journal of Bacteriology, 2012, 194(16): 4459-4460. DOI:10.1128/JB.00867-12 |
| [68] |
Lee JH, Wood TK, Lee J. Roles of indole as an interspecies and interkingdom signaling molecule[J]. Trends in Microbiology, 2015, 23(11): 707-718. DOI:10.1016/j.tim.2015.08.001 |
| [69] |
Bansal T, Alaniz RC, Wood TK, et al. The bacterial signal indole increases epithelial-cell tight-junction resistance and attenuates indicators of inflammation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(1): 228-233. DOI:10.1073/pnas.0906112107 |
| [70] |
Sonowal R, Swimm A, Sahoo A, et al. Indoles from commensal bacteria extend healthspan[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(36): E7506-E7515. DOI:10.1073/pnas.1706464114 |
| [71] |
Chimerel C, Emery E, Summers DK, et al. Bacterial metabolite indole modulates incretin secretion from intestinal enteroendocrine L cells[J]. Cell Reports, 2014, 9(4): 1202-1208. DOI:10.1016/j.celrep.2014.10.032 |
| [72] |
Gerth K, Metzger R, Reichenbach H. Induction of myxospores in Stigmatella aurantiaca(myxobacteria):inducers and inhibitors of myxospore formation, and mutants with a changed sporulation behaviour[J]. Journal of General Microbiology, 1993, 139(4): 865-871. DOI:10.1099/00221287-139-4-865 |
| [73] |
Wang DD, Ding XD, Rather PN. Indole can act as an extracellular signal in Escherichia coli[J]. Journal of Bacteriology, 2001, 183(14): 4210-4216. DOI:10.1128/JB.183.14.4210-4216.2001 |
| [74] |
Kim J, Park W. Indole:a signaling molecule or a mere metabolic byproduct that alters bacterial physiology at a high concentration?[J]. Journal of Microbiology, 2015, 53(7): 421-428. DOI:10.1007/s12275-015-5273-3 |
 2020, Vol. 47
2020, Vol. 47