扩展功能
文章信息
- 黄旭华, 龙江琼, 蒋满贵, 何强, 黄深惠, 董战旗, 夏青, 李田, 潘敏慧, 潘志新
- HUANG Xu-Hua, LONG Jiang-Qiong, JIANG Man-Gui, HE Qiang, HUANG Shen-Hui, DONG Zhan-Qi, XIA Qing, LI Tian, PAN Min-Hui, PAN Zhi-Xin
- 一株家蚕异型微孢子虫感染特征和分类地位
- Infection characteristics and classification of a microsporidian isolated from silkworm
- 微生物学通报, 2020, 47(11): 3577-3585
- Microbiology China, 2020, 47(11): 3577-3585
- DOI: 10.13344/j.microbiol.china.190986
-
文章历史
- 收稿日期: 2019-11-26
- 接受日期: 2020-03-31
- 网络首发日期: 2020-04-17
2. 西南大学家蚕基因组生物学国家重点实验室 重庆 400715
2. State Key Laboratory of Silkworm Genome Biology, Southwest University, Chongqing 400715, China
家蚕微粒子病是由微孢子虫感染家蚕引起的一种病害,微孢子虫对家蚕既能够食下传染,也能够胚种传染,是蚕种质量的重要检验对象,行业法规规定所有生产蚕种必须通过检疫合格后才能应用于蚕业生产[1]。家蚕微孢子虫(Nosema bombycis,Nb)是引发家蚕微粒子病的主要病原,但在蚕种质量检验中经常发现一些与Nb孢子形态有差异的异型微孢子虫,这些异型微孢子虫感染力和分类地位未明确,极大影响蚕种检疫和防病工作[2]。因此,在蚕种繁育过程中调查一些异型微孢子虫来源,并掌握这些微孢子虫的感染力和分类地位,对蚕种生产控制家蚕微粒子病具有重要指导作用。
微孢子虫寄主非常广泛,可感染从无脊椎动物到脊椎动物的几乎所有动物类群[3],至今已经发现的微孢子虫超过150个属,有1 400多种[4-5]。同时,微孢子虫是一种嗜昆虫的病原,据记载可感染各个目的昆虫,其中双翅目和鳞翅目的昆虫感染危害严重,很多种昆虫受到一种以上微孢子虫感染[6-7]。家蚕病原性微孢子虫种类多、来源复杂,如20世纪60–70年代日本科学家相继从家蚕体内分离到具褶孢虫属的M25、M27和泰罗汉孢虫M32及变形孢虫M12等异型微孢子虫[8];国内学者也对不同蚕区感染家蚕的异型微孢子虫进行了研究,如:方定坚等(1991)研究了MG1和MG2微孢子虫[9];万永继等(1995)研究了SCM6和SCM7微孢子虫[10];沈中元等(2001)研究了MZ1微孢子虫[11];潘敏慧等(2002)研究了SCM8微孢子虫[12];时连根等(2002)研究了SLN1微孢子虫[13];罗梅兰等(2012)研究了GXM1微孢子虫[14];Meng等(2018)研究了Vairimorpha necatrix BM,发现其对家蚕没有胚种传染性[15]。其中,万永继[16]研究了SCM6和SCM7微孢子虫,发现这2种微孢子虫的胚种传染力很低或没有,提议蚕种检疫中对SCM6和SCM7微孢子虫进行分类检验处理,累计挽回蚕种损失110多万张。
因此,深入调查蚕种质量检验发现的异型微孢子虫,调查其感染性和遗传系统发育情况,可深入了解家蚕病原性微孢子虫对蚕种生产的危害,可以提高蚕业生产中家蚕微粒子病的防控技术水平。广西是一个新兴蚕区,在蚕业生产中分布着一些家蚕病原性微孢子虫,但有关广西蚕区家蚕异型微孢子虫的研究较少;而蚕种微粒子病检疫是生产过程的关键环节,跟踪广西蚕种检疫发现的异型微孢子虫,将有助于完善家蚕微粒子病病原种类和数据库。本文研究了一株从广西蚕种检疫样品中分离获得的微孢子虫的感染性和遗传系统发育情况,有助于了解家蚕病原性微孢子虫对蚕种生产的危害性,掌握家蚕微粒子病病原来源,为蚕业生产中控制家蚕微粒子病的危害提供参考依据。
1 材料与方法 1.1 供试微孢子虫来源和纯化从广西蚕种检疫样品(母蛾)中分离获得一株异型微孢子虫,编号为GXM15;家蚕微孢子虫(Nosema bombycis,Nb)由广西蚕业技术推广总站提供,分别取GXM15和Nb微孢子虫感染2龄起蚕,饲养12 d后(5龄第2天),并分别取发病蚕体进行提取、纯化与精制GXM15微孢子虫和Nb[17]。
1.2 主要试剂和仪器植物基因组DNA提取试剂盒、pEASY®-T1 Simple Cloning Kit,天根生化科技(北京)有限公司;HiFi酶,北京普凯瑞生物科技有限公司;PCR引物,重庆华大基因科技有限公司;回收胶试剂盒(gel extraction kit 200),上海研拓生物科技有限公司。PCR仪,Applied Biosystems公司;珠磨式研磨器,MPBIO公司;高速冷冻离心机,eppendorf公司;相差显微镜,尼康公司;透射电子显微镜,日立公司。
1.3 供试家蚕品种试验应用的家蚕品种为932×芙蓉。
1.4 GXM15微孢子虫对家蚕食下感染和胚种传染 1.4.1 食下感染力测定将GXM15微孢子虫配制成1.0×108个/mL的孢子悬浮液,再利用无菌水以10倍梯度稀释成5个系列稀释液(107、106、105、104和103个/mL),分别取各种浓度的孢子悬浮液0.2 mL,均匀涂抹于1片的桑叶片(面积为16.0 cm2)上,饲喂一区2龄起蚕(30头) 12 h,重复2区。各区蚕儿吃完含有孢子的桑叶后更换蚕座纸片,改用洁净桑叶饲养12 d(5龄第3天),对各区每头家蚕进行显微镜检验,计算各区家蚕的GXM15微孢子虫感染率,以Nb作为对照[14]。2种微孢子虫对家蚕的半数感染浓度(IC50)用Reed Muench法计算[1]。
1.4.2 胚种传染力测定将GXM15微孢子虫配制成3.0×105个/mL的孢子悬浮液,取洁净桑叶浸泡5 min到孢子悬浮液中,稍微晾干,饲喂给5龄起蚕12 h,再利用洁净桑叶饲育;当家蚕结茧、出蛾后,获得感染GXM15微孢子虫的蛾,使雌雄蛾交配,雌蛾产下蚕卵;利用显微镜检验产卵的雌蛾,将严重感染GXM15微孢子虫的雌蛾(每个视野的微孢子虫检出密度 > 100个)所产的蚕卵,进行即时浸酸处理、催青、孵化蚁蚕,饲养至3龄起蚕,再利用显微镜检验各头家蚕的GXM15微孢子虫感染情况,以Nb作为对照,分别计算GXM15微孢子虫和Nb对家蚕的胚种传染率[14]。
1.5 GXM15微孢子虫形态观察分别将GXM15微孢子虫和Nb悬浮液做成水玻片标本,利用相差显微镜以600倍观察,并利用测微尺软件分别测量2种微孢子虫的长轴与短轴,计算各种微孢子虫的体积,比较其形状差异[14]。
1.6 GXM15微孢子虫感染家蚕组织观察将GXM15微孢子虫配制成浓度为1×107个/mL的孢子悬浮液,取洁净桑叶浸泡到孢子悬浮液中,稍微晾干,饲喂给2龄起蚕添食12 h,再利用洁净桑叶饲育至5龄期第3天。在立体显微镜下解剖蚕体,用镊子轻取家蚕中肠、丝腺、马氏管、性腺等组织,在无菌水中冲洗数次,再分别涂抹玻片,利用显微镜(600倍)观察有无GXM15微孢子虫[14]。
1.7 GXM15微孢子虫内部结构观察微孢子虫样品处理过程[18]:(1)预处理:取1.0 mL的GXM15微孢子虫孢子悬浮液(浓度为5.0×108个/mL),3 000 r/min离心10 min,去掉上清,加入1.0 mL戊二醛固定液(6%,体积比)溶解,在4 ℃下放置24 h,再次离心后去掉上清,加入适量琼脂液(2.5%)预包埋成块状。(2)固定:用戊二醛固定液(6%)浸泡12 h,再用0.1 mol/L二甲胂酸钠缓冲液冲洗6次;用1%锇酸浸泡处理2 h,再用0.1 mol/L二甲胂酸钠缓冲液冲洗6次;用0.5%高锰酸钾浸泡2 h,再用9 g/L NaCl溶液冲洗6次。(3)脱水:依次利用浓度为50%、70%、80%和90%的乙醇溶液处理。(4)包埋和切片:利用环氧树脂渗透、包埋,制成超薄切片。(5)染色:利用醋酸铀-柠檬酸铅进行双重电子染色,再利用透射电子显微镜观察孢子内部结构。
1.8 GXM15微孢子虫分子系统发育分析 1.8.1 GXM15微孢子虫基因组提取取1.0 mL GXM15微孢子虫孢子悬浮液(浓度约1×109个/mL),12 000 r/min离心5 min;加入700 μL植物基因组DNA提取试剂盒内的GP1悬浮,转入专用破碎管中(装有0.2 g大玻璃珠和0.2 g小玻璃珠),利用组织破碎仪破碎3次(40 s/次)。然后按照试剂盒说明书提取GXM15微孢子虫基因组DNA[19]。
1.8.2 GXM15微孢子虫SSU rRNA基因和ITS序列的PCR扩增、克隆与测序[20]选择引物18f/1537r用于GXM15微孢子虫的SSU rRNA扩增(表 1)。PCR反应体系(50 μL):dNTPs (10 mmol/L) 1 μL,18f和1537r引物(10 mmol/L)各2 μL,10×HiFi buffer 5 μL,HiFi酶(5 u/μL) 1 μL,模板DNA 2 μL,ddH2O 37 μL。PCR反应条件:94 ℃ 5 min;94 ℃ 45 s,55 ℃ 45 s,72 ℃ 1 min 10 s,循环30次;72 ℃ 10 min。通过1.5%琼脂糖凝胶电泳,利用回收胶试剂盒对目的条带进行回收;再利用pEASY®-T1 Simple Cloning Kit对目的产物进行克隆,取阳性菌落的菌液送华大基因公司测序。微孢子虫rRNA ITS序列的扩增采用引物ILSUF/S33R (表 1),PCR反应体系同SSU rRNA扩增(除引物外)。PCR反应条件:94 ℃ 5 min;94 ℃ 45 s,56 ℃ 45 s,72 ℃ 45 s,循环30次;72 ℃ 10 min。同样进行目的序列回收、克隆和测序。
| Gene name | Primers name | Primers sequence (5′→3′) |
| SSU rRNA | 18f | CACCAGGTTGATTCTGCC |
| 1537r | TTATGATCCTCCTAATGGTTC | |
| ITS | ILSUF | TGGGTTTAGACCGTCGTGAG |
| S33R | ATAGCGTCTACGTCAGGCAG |
以测序获得的GXM15微孢子虫SSU rRNA序列与GenBank上属于Nosema、Amblyospora、Endoreticulatus、Vairimorpha、Pleistophora和Glugea属的已知微孢子虫SSU rRNA序列进行比较分析,利用MEGA 5.0软件构建系统发育树;再利用DNAstar软件分别对GXM15微孢子虫SSU rRNA基因与ITS片段序列进行同源序列相似性和分化度分析[21]。
2 结果与分析 2.1 GXM15微孢子虫对家蚕的致病性 2.1.1 对家蚕的半数感染浓度(IC50)分别利用GXM15微孢子虫和Nb的梯度浓度孢子悬浮液感染家蚕2龄起蚕,经计算GXM15微孢子虫的IC50为8.29×104个/mL,Nb的IC50为3.63×104个/mL,GXM15微孢子虫的IC50是Nb微孢子虫的2.28倍(表 2),说明GXM15微孢子虫对家蚕也具有较强的食下感染力,但比Nb的感染力偏低。
| Dilution factor of microsporidia (spores/mL) | Number of silkworm | Infected rate of GXM15 (%) | Infected rate of Nb (%) |
| 107 | 90 | 100.00 | 100.00 |
| 106 | 90 | 93.33 | 100.00 |
| 105 | 90 | 51.11 | 68.89 |
| 104 | 90 | 13.33 | 21.11 |
| 103 | 90 | 0 | 3.33 |
| IC50 (sproes/mL) | 8.29×104 | 3.63×104 |
利用GXM15微孢子虫感染家蚕5龄起蚕,检测感染GXM15微孢子虫严重的母蛾产下的蚕卵孵化的后代感染孢子情况。结果表明GXM15微孢子虫对家蚕的胚种传染率为3.60%,而同时期Nb微孢子虫的胚种传染率为41.60%,证明GXM15微孢子虫对家蚕具有较低胚种传染力。
2.2 GXM15微孢子虫的基本形态通过显微镜观察出GXM15微孢子虫(图 1A)明显比Nb微孢子虫(图 1B)偏大,对两种微孢子虫的长轴和短轴进行测量(表 3),GXM15微孢子虫体积为7.20 μm3,是Nb孢子体积的2.19倍,与Nb差异明显。GXM15微孢子虫与Nb微孢子虫具有相似的折光性。

|
| 图 1 GXM15微孢子虫和Nb的孢子形态(600×) Figure 1 Morphology of GXM15 and Nb spores (600×) 注:A:GXM15微孢子虫;B:家蚕微孢子虫(Nb). Note: A: GXM15 microsporidian; B: Nosema bombycis. |
|
|
| 微孢子虫 Microsporidian |
GXM15 | Nb |
| 形状Shape | 短卵圆形Short oval | 卵圆形Oval |
| 大小Size (μm) | (2.05±0.20)×(3.25±0.30) | (1.48±0.18)×(2.84±0.25) |
| 体积Volume (μm3) | 7.20 | 3.29 |
| 长短轴比 Length/width |
1.59 | 1.91 |
| 注:V=π/6×长轴×短轴2. Note: V=π/6×Length×Width2. | ||
利用显微镜观察感染GXM15微孢子虫的家蚕的各种组织,发现家蚕丝腺有大量GXM15微孢子虫,尤其是前部丝腺最多;而家蚕气管上皮肌肉组织也有较多GXM15微孢子虫;家蚕中肠、马氏管、生殖腺、脂肪组织等组织有少量GXM15微孢子虫。说明GXM15微孢子虫能够对家蚕全身性感染。
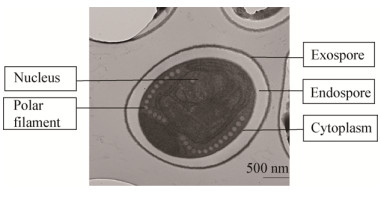
2.4 GXM15微孢子虫的超微结构利用透射电子显微镜观察GXM15微孢子虫的内部结构,GXM15微孢子虫纵剖面呈马蹄形;孢子壁结构清晰,由外壁、内壁、原生质膜3层组成;极丝13圈,极丝倾斜角约为45º;孢子具双核(图 2),核周围有大量的粗糙内质网,属于典型的Nosema属微孢子虫特征。
|
| 图 2 GXM15微孢子虫的超微结构(30 000×) Figure 2 Ultrastructure of microsporidian spores of GXM15 (30 000×) |
|
|
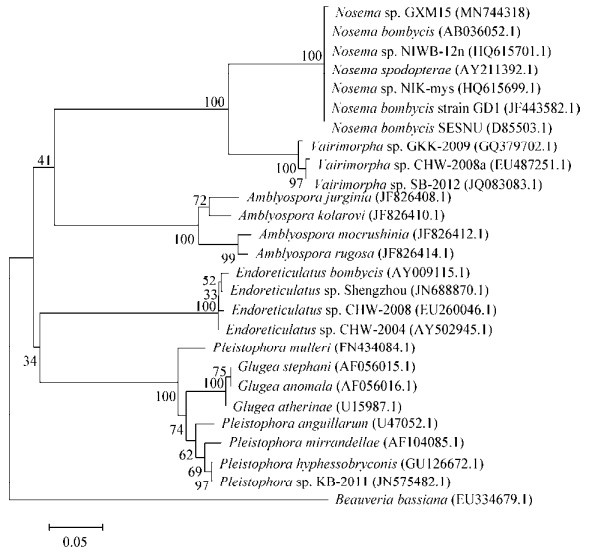
通过PCR扩增、T克隆和测序获得GXM15微孢子虫的SSU rRNA基因的部分序列(GenBank登录号MN744318)和ITS片段序列(GenBank登录号MN747890)。GXM15微孢子虫的SSU rRNA序列长度为1 110 bp,片段的(G+C)mol%含量为33.96%;ITS序列长度为339 bp,片段的(G+C)mol%含量为39.53%。基于SSU rRNA基因序列,利用MEGA 5.0软件对GXM15微孢子虫和已知的25株微孢子虫一起构建系统发育树,以白僵菌(Beauveria bassiana) (EU334679)作为外群。结果发现GXM15微孢子虫位于Nosema属的类群中(图 3),说明GXM15微孢子虫是一株Nosema属微孢子虫,与超微结构观察等传统分类方法一致。采用DNAstar软件的MegAlign对GXM15微孢子虫微孢子虫和其他14株已知微孢子虫的SSU rRNA基因序列进行相似性和分化度分析(表 4),发现GXM15微孢子虫的SSU rRNA与其他微孢子虫的SSU rRNA序列的最小分化度为0.8,最高相似度为95.7%,说明GXM15微孢子虫与其他微孢子虫有一定差异性。另外,基于微孢子虫ITS基因序列进行相似性和遗传距离分析结果表明,GXM15微孢子虫和熊蜂微孢子虫(MF998087)相似度最高达到97.1%,遗传距离也只有1.8,而GXM15微孢子虫与其他微孢子虫相似度很低,均低于50%,遗传距离也在12.0以上(表 5),说明GXM15微孢子虫是一种与熊峰微孢子虫亲缘关系较近的微孢子虫,暂时命名为Nosema sp. GXM15。
|
| 图 3 GXM15微孢子虫与其他微孢子虫基于SSU rRNA基因序列的系统发育进化树 Figure 3 Phylogenetic tree of SSU rRNA gene sequences of GXM15 and other microsporidian strains 注:分支点上的数字表示构建系统树时1 000次计算形成该节点的百分比;标尺长度代表 1%的SSU rRNA基因序列的进化差异. Note: The bootstrap values (%) presented at the branch points were calculated from 1 000 replications; The ruler length means a 1% SSU rRNA sequence difference. |
|
|
| No. | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 |
| 1 | 94.1 | 93.9 | 94.0 | 93.5 | 95.7 | 93.5 | 93.0 | 93.0 | 91.9 | 90.3 | 71.7 | 46.6 | 42.1 | 37.0 | |
| 2 | 0.9 | 99.8 | 99.9 | 99.4 | 99.4 | 99.4 | 99.0 | 99.4 | 97.1 | 96.3 | 76.6 | 45.8 | 43.3 | 36.3 | |
| 3 | 1.1 | 0.2 | 99.8 | 99.3 | 99.2 | 99.3 | 98.8 | 99.3 | 96.9 | 96.3 | 76.6 | 45.7 | 43.4 | 36.0 | |
| 4 | 1.0 | 0.1 | 0.2 | 99.5 | 99.5 | 99.5 | 99.1 | 99.5 | 97.2 | 96.4 | 76.5 | 45.8 | 43.3 | 36.3 | |
| 5 | 1.0 | 0.1 | 0.2 | 0.0 | 99.9 | 99.9 | 99.5 | 99.9 | 97.6 | 96.8 | 76.9 | 47.0 | 42.3 | 36.3 | |
| 6 | 0.8 | 0.1 | 0.3 | 0.0 | 0.0 | 99.9 | 99.4 | 99.9 | 98.1 | 96.6 | 75.8 | 45.3 | 43.5 | 37.5 | |
| 7 | 1.0 | 0.2 | 0.3 | 0.1 | 0.1 | 0.0 | 99.4 | 99.9 | 97.6 | 96.8 | 76.9 | 47.2 | 42.3 | 36.3 | |
| 8 | 1.5 | 0.6 | 0.7 | 0.5 | 0.5 | 0.5 | 0.6 | 99.5 | 97.2 | 96.2 | 76.9 | 46.7 | 42.1 | 36.0 | |
| 9 | 2.3 | 0.1 | 0.2 | 0.0 | 0.1 | 0.0 | 0.1 | 0.5 | 94.3 | 96.8 | 76.0 | 46.3 | 39.8 | 18.0 | |
| 10 | 2.6 | 0.5 | 0.7 | 0.4 | 0.5 | 0.3 | 0.5 | 0.7 | 3.3 | 94.6 | 74.3 | 47.7 | 39.3 | 16.8 | |
| 11 | 3.1 | 2.0 | 2.0 | 1.9 | 1.9 | 2.0 | 2.0 | 2.4 | 1.9 | 2.2 | 78.2 | 44.7 | 42.9 | 36.4 | |
| 12 | 16.7 | 15.8 | 15.9 | 15.9 | 15.9 | 16.7 | 16.0 | 15.9 | 15.9 | 16.3 | 15.3 | 51.2 | 45.8 | 37.9 | |
| 13 | 52.4 | 52.2 | 53.0 | 52.2 | 52.2 | 51.4 | 52.1 | 51.8 | 48.0 | 47.3 | 55.5 | 52.3 | 50.1 | 33.1 | |
| 14 | 56.4 | 56.1 | 56.1 | 56.5 | 56.5 | 55.5 | 56.8 | 56.8 | 49.1 | 49.9 | 56.6 | 57.0 | 43.6 | 34.5 | |
| 15 | 107.9 | 105.7 | 105.7 | 105.2 | 105.2 | 107.2 | 105.4 | 105.1 | 98.0 | 96.3 | 107.3 | 120.4 | 114.6 | 119.6 | |
| Note: 1: Nosema sp. GXM15; 2: Nosema sp. PA (EU864528.1); 3: Nosema bombycis strain GD8 (JF443589.1); 4: Nosema trichoplusiae (U09282.1); 5: Nosema spodopterae (AY211392); 6: Nosema bombycis (AB036052); 7: Nosema bombycis (D85503); 8: Nosema antheraeae (EU864526); 9: Nosema heliothidis (FJ772435); 10: Nosema fumiferanae (HQ457432); 11: Nosema furnacalis (U26532); 12: Vairimorpha sp. (JQ083083); 13: Endoreticulatus sp. (AY502945); 14: Amblyospora jurginia (JF826408); 15: Beauveria bassiana (EU334679). Data in upper right half of the table were of sequence similarity (%) and the bottom left half were of genetic distance. | |||||||||||||||
| No. | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 |
| 1 | 97.1 | 24.5 | 25.0 | 28.3 | 27.6 | 25.5 | 26.1 | 23.0 | 27.7 | 40.1 | |
| 2 | 1.8 | 26.0 | 25.5 | 29.3 | 28.1 | 24.5 | 26.1 | 23.5 | 28.7 | 37.8 | |
| 3 | 13.9 | 13.9 | 96.9 | 90.6 | 95.8 | 88.0 | 85.6 | 87.2 | 71.9 | 91.7 | |
| 4 | 13.9 | 13.9 | 3.2 | 91.6 | 96.9 | 88.5 | 85.6 | 88.2 | 73.4 | 90.6 | |
| 5 | 13.9 | 13.9 | 6.6 | 5.4 | 94.8 | 87.4 | 86.7 | 86.6 | 73.8 | 84.3 | |
| 6 | 13.9 | 13.9 | 4.3 | 3.2 | 2.7 | 87.5 | 85.1 | 87.2 | 72.9 | 89.6 | |
| 7 | 13.9 | 13.9 | 3.2 | 2.7 | 6.6 | 3.8 | 88.3 | 84.0 | 77.6 | 81.8 | |
| 8 | 16.1 | 16.1 | 4.5 | 4.5 | 5.0 | 4.4 | 3.9 | 92.0 | 76.6 | 86.7 | |
| 9 | 16.1 | 16.1 | 5.0 | 3.9 | 7.4 | 5.0 | 5.1 | 5.0 | 74.9 | 85.0 | |
| 10 | 16.1 | 16.1 | 7.5 | 4.2 | 9.8 | 5.2 | 3.1 | 6.4 | 4.2 | 71.3 | |
| 11 | 14.8 | 13.5 | 6.6 | 7.7 | 11.4 | 8.9 | 7.8 | 6.2 | 6.7 | 8.3 | |
| Note: 1: Nosema sp. GXM15; 2: Tubulinosema sp. Bpas2 (MF998087); 3: Nosema bombycis strain GD5 (JF443623); 4: Nosema bombycis clone 9 (EU350402); 5: Nosema bombycis strain GX2 (JF443601); 6: Nosema bombycis strain YN18 (JF443664); 7: Nosema bombycis clone NA6 (GQ334403); 8: Nosema bombycis strain GX18 (JF443617); 9: Nosema sp. HA (EU864524); 10: Nosema antheraeae (EU864521); 11: Nosema sopdopterae (AY747307). Data in upper right half of the table were of sequence similarity (%) and the bottom left half were of genetic distance. | |||||||||||
微孢子虫传统生物学分类标准主要以孢子形态及大小、内部结构、生活史以及感染性等特征为依据,其中极丝是微孢子虫的重要标志,Nosema属微孢子虫具有主要感染寄主细胞的细胞质、成熟孢子为卵圆形、有双核等特征[22]。微孢子虫SSU rRNA基因和ITS序列是开展微孢子虫分子分类研究的重要标志DNA序列,其中SSU rRNA基因适合用于分析微孢子虫属间的亲缘关系,而ITS序列适用于微孢子虫种的水平上的分类研究[23]。
本实验利用微孢子虫传统生物学分类方法与分子分类信息相结合对GXM15微孢子虫进行分类研究。通过感染试验发现GXM15微孢子虫对家蚕具有较强的食下感染力,其IC50为8.29×104个/mL,与Nb处于同一级别,但比Nb微孢子虫稍低;GXM15微孢子虫对家蚕只有轻微胚种传染力,明显低于Nb;而且GXM15微孢子虫对家蚕具有全身性感染情况,因此说明GXM15微孢子虫对家蚕具有一定感染性,是一种家蚕病原性微孢子虫。GXM15微孢子虫形态呈短卵圆形,体积明显比Nb微孢子虫大,是一种大孢子;GXM15微孢子虫超微结构有极丝(13圈)和双核结构,有Nosema属微孢子虫典型结构特征。基于SSU rRNA和ITS基因序列进行系统发育分析进一步确定GXM15微孢子虫是一株新的Nosema属微孢子虫,而且GXM15微孢子虫与熊蜂微孢子虫具有很高的亲缘关系。同时根据蚕种生产方式以及病原性和分类地位分析推测GXM15微孢子虫的原始寄主不是家蚕,而是某种野外昆虫,是否是熊蜂等昆虫携带GXM15微孢子虫交叉感染家蚕还有待进一步的分析研究。
蚕业法规规定所有蚕种必须进行微粒子病检疫,而蚕种生产中家蚕经常受到一些异型微孢子虫的感染,这些异型微孢子虫感染力不同,可以进行微孢子虫分类判定处理,如广东蚕种检疫部门也依据MG1、MG2、MG4微孢子虫的生物学和病理学特性,对这些异型微孢子虫采取3−4个有毒点作为1点或全部不计入毒率的方式进行分类处理,为挽回蚕种损失起到很好的作用[24]。因此,根据GXM15微孢子虫对家蚕病原性和分类地位,在桑蚕一代杂交种的蚕种检疫样品中检查GXM15微孢子虫可以采取适当的分类判定技术措施,以达到降低蚕种淘汰率的目的;同样地,也可以对其他一系列异型微孢子虫进行病原性和分类地位研究,建立分类检验判定技术,这将对蚕种生产防控家蚕微粒子病具有重要意义。
4 结论GXM15微孢子虫是一株感染家蚕的异型微孢子虫,其形态偏大,与家蚕微孢子虫(Nb)形态差异明显;GXM15微孢子虫对家蚕具有较强食下感染力,但对家蚕胚种传染力较低;分类鉴定表明GXM15微孢子虫是一株新的Nosema属微孢子虫;在桑蚕一代杂交种蚕种检疫中GXM15微孢子虫适用于分类检验判定技术进行检疫判断,将有利于减少蚕种生产损失。
| [1] |
Zhejiang University. Histopathology of Silkworm[M]. Beijing: China Agriculture Press, 2000: 148-173. (in Chinese) 浙江大学. 家蚕病理学[M]. 北京: 中国农业出版社, 2000: 148-173. |
| [2] |
Lu XM, Shao YQ. A review on current status and development trend of pebrine prevention and control technology[J]. Science of Sericulture, 2016, 42(6): 945-952. (in Chinese) 鲁兴萌, 邵勇奇. 家蚕微粒子病防控技术研究的发展现状与趋势[J]. 蚕业科学, 2016, 42(6): 945-952. |
| [3] |
Mathis A. Microsporidia:emerging advances in understanding the basic biology of these unique organisms[J]. International Journal for Parasitology, 2000, 30(7): 795-804. DOI:10.1016/S0020-7519(00)00064-3 |
| [4] |
Zhou ZY. Genome Biology of Nosema Bombycis[M]. Beijing: Science Press, 2014: 2-3. (in Chinese) 周泽扬. 家蚕微孢子虫基因组生物学[M]. 北京: 科学出版社, 2014: 2-3. |
| [5] |
Franzen C. Microsporidia:a review of 150 years of research[J]. The Open Parasitology Journal, 2008, 2(1): 1-34. |
| [6] |
Liu JP, Zeng L. An overview of research on the microsporidian biodiversity[J]. Chinese Bulletin of Entomology, 2006, 43(2): 153-158. (in Chinese) 刘吉平, 曾玲. 微孢子虫生物多样性研究的述评[J]. 昆虫知识, 2006, 43(2): 153-158. |
| [7] |
Chen SL, Gao X, Zhang JX, et al. Pathogenicity of microsporidium isolated from Pieris rapae L.on silkworm in Yunnan[J]. Southwest China Journal of Agricultural Sciences, 2015, 28(2): 913-917. (in Chinese) 陈世良, 高翔, 张金祥, 等. 云南菜粉蝶微孢子虫感染家蚕的病原性研究[J]. 西南农业学报, 2015, 28(2): 913-917. |
| [8] |
Kawarabata T. Biology of microsporidians infecting the silkworm, Bombyx mori, in Japan[J]. Journal of Insect Biotechnology and Sericology, 2003, 72(1): 1-32. |
| [9] |
Fang DJ, Liao ST, Zheng XM, et al. Studies on morphology, pathogenicity and infection path of new Nosema bombysis MG1 and MG.Ⅰ.Morphology, pathogenicity and infection path[J]. Guangdong Agricultural Sciences, 1991(2): 35-38. (in Chinese) |
| [10] |
Wan YJ, Zhang L, Chen ZP, et al. Study of a pathogenic microsporidium SCM7(Endoreticulatus sp.) isolated from the larvae of silkworm, Bombyx mori[J]. Acta Sericologica Sinica, 1995, 21(3): 168-172. (in Chinese) 万永继, 张琳, 陈祖佩, 等. 家蚕病原性微孢子虫SCM7(Endoreticulatus sp.)的分离和研究[J]. 蚕业科学, 1995, 21(3): 168-172. |
| [11] |
Shen ZY, Xu L. Studies on the pathogenicity of a microsporidium MZ1(Nosema sp.) to the silkowrm, Bombyx mori[J]. Science of Sericulture, 2001, 27(3): 197-199. (in Chinese) 沈中元, 徐莉. 从家蚕蛾中分离的微孢子虫MZ1(Nosema sp.)对家蚕的病原性研究[J]. 蚕业科学, 2001, 27(3): 197-199. |
| [12] |
Pan MH, Wan YJ, Zhu RY. Studied on a microsporidium (SCM8) pathogenic for the silkworm, Bombyx mori[J]. Science of Sericulture, 2002, 28(2): 115-119. (in Chinese) 潘敏慧, 万永继, 祝仁英. 家蚕病原性微孢子虫SCM8的研究[J]. 蚕业科学, 2002, 28(2): 115-119. |
| [13] |
Shi LG, Liu SM, Xu JL. Study on a new microsporidium SLN1 isolated from the silkworm, Bombyx mori[J]. Acta Sericologica Sinica, 2002, 28(3): 211-214. (in Chinese) 时连根, 刘淑梅, 徐俊良. 从家蚕体分离的一种新微孢子虫SLN1的研究[J]. 蚕业科学, 2002, 28(3): 211-214. |
| [14] |
Luo ML, Huang XH, Pan ZX, et al. Biological characters and molecular phylogenetic analysis of a microsporidian isolate pathogenic to Bombyx mori[J]. Science of Sericulture, 2012, 38(5): 856-863. (in Chinese) 罗梅兰, 黄旭华, 潘志新, 等. 一株家蚕病原性微孢子虫的生物学特性与分子系统发育分析[J]. 蚕业科学, 2012, 38(5): 856-863. |
| [15] |
Meng XZ, Luo B, Tang XY, et al. Pathological analysis of silkworm infected by two microsporidia Nosema bombycis CQ1 and Vairimorpha necatrix BM[J]. Journal of Invertebrate Pathology, 2018, 153: 75-84. DOI:10.1016/j.jip.2017.12.005 |
| [16] |
Wan YJ.Study and control on several population of pathogenic microsporidian in the silkworm, Bombyx mori (Bombycidae, Lepidoptera)[D].Beijing: Doctoral Dissertation of University of China Agriculture, 2005: 66-72(in Chinese) 万永继.家蚕病原性微孢子虫若干种群的研究与控制[D].北京: 中国农业大学博士学位论文, 2005: 66-72 |
| [17] |
Huang XH, Qi GJ, Tang QK, et al. Biological characters and molecular phylogenetic analysis of a microsporidium strain isolated from Cnaphalocrocis medinalis[J]. Science of Sericulture, 2013, 39(3): 543-550. (in Chinese) 黄旭华, 祁广军, 汤庆坤, 等. 从稻纵卷叶螟分离的一株微孢子虫的生物学特性与分子系统发育分析[J]. 蚕业科学, 2013, 39(3): 543-550. |
| [18] |
Huang XH, Qi GJ, Tang QK, et al. Biological characters and molecular phylogenesis of five microspo-ridian isolates from field insects[J]. Science of Sericulture, 2012, 38(5): 864-872. (in Chinese) 黄旭华, 祁广军, 汤庆坤, 等. 从野外昆虫体内分离的5株微孢子虫的生物学特性与分子系统发育研究[J]. 蚕业科学, 2012, 38(5): 864-872. |
| [19] |
Cai SF, He XY, He XK, et al. A protocol for fast and efficient preparation of genomic DNA and total proteins of Nosema bombycis[J]. Science of Sericulture, 2011, 37(6): 1019-1024. (in Chinese) 蔡顺风, 何欣怡, 何祥康, 等. 一种快速高效制备家蚕微孢子虫基因组DNA和总蛋白的方法[J]. 蚕业科学, 2011, 37(6): 1019-1024. |
| [20] |
Huang WF, Tsai SJ, Lo CF, et al. The novel organization and complete sequence of the ribosomal RNA gene of Nosema bombycis[J]. Fungal Genetics and Biology, 2004, 41(5): 473-481. DOI:10.1016/j.fgb.2003.12.005 |
| [21] |
Dong SN, Zhu F, Xu L, et al.Sequence and phylogenetic analysis of SSU rRNA gene of seven microsporidia[A]//Proceedings of Sixth Youth Academic[C].Yangling: Chinese Society of Sericultural Science, 2009: 1-23(in Chinese) 董世楠, 朱峰, 徐莉, 等.七种微孢子虫核糖体小亚基基因(SSU rRNA)的克隆及其系统发育分析[A]//中国蚕学会第六届青年学术研讨会论文集[C].杨凌: 中国蚕学会, 2009: 1-23 |
| [22] |
Kakuda T, Shiki M, Kubota S, et al. Phylogeny of benign Theileria species from cattle in Thailand, China and the U.S.A.based on the major piroplasm surface protein and small subunit ribosomal RNA genes[J]. International Journal for Parasitology, 1998, 28(8): 1261-1267. DOI:10.1016/S0020-7519(98)00113-1 |
| [23] |
Niu QL, Luo JX, Yin H. Advances and application of rDNA-ITS in the molecular taxonomy of parasite[J]. Chinese Journal of Veterinary Parasitology, 2008, 16(4): 41-47. (in Chinese) 牛庆丽, 罗建勋, 殷宏. 转录间隔区(ITS)在寄生虫分子生物学分类中的应用及其进展[J]. 中国兽医寄生虫病, 2008, 16(4): 41-47. |
| [24] |
Zheng XM, Yang Q, Huang BH, et al. Study on a microsporidian Vairimorpha MG4 separated from silkworm, Bombyx mori[J]. Acta Sericologica Sinica, 1999, 25(4): 225-229. (in Chinese) 郑祥明, 杨琼, 黄炳辉, 等. 家蚕分离的微孢子虫Vairimorpha sp.MG4的研究[J]. 蚕业科学, 1999, 25(4): 225-229. |
 2020, Vol. 47
2020, Vol. 47