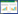扩展功能
文章信息
- 宋哲玮, 杜海, 聂尧, 徐岩
- SONG Zhe-Wei, DU Hai, NIE Yao, XU Yan
- 酱香型白酒发酵过程中核心酵母的鉴别及其功能
- Identification of core yeasts and their functions in the fermentation process of Maotai-flavor Baijiu
- 微生物学通报, 2020, 47(11): 3504-3514
- Microbiology China, 2020, 47(11): 3504-3514
- DOI: 10.13344/j.microbiol.china.191046
-
文章历史
- 收稿日期: 2019-12-14
- 接受日期: 2020-02-12
- 网络首发日期: 2020-04-23
真核微生物是自然界中普遍存在的微生物[1],它主要包括酵母、霉菌以及藻类等各种微生物[2],尤其在食品发酵、自然生态以及医疗保健方面有很多的应用[3-5]。其中酵母可以通过利用多种糖原产生多种醇类物质[6],调节发酵过程中风味代谢产物的种类和含量,影响发酵食品的品质。作为一种具有独特发酵工艺和复杂微生物群落的传统固态发酵产物,中国白酒是由酵母、霉菌、细菌等多种微生物构成的发酵微生物群落在自然环境和人为调节双重影响下通过天然发酵形成的风味代谢产物[7]。酵母是白酒发酵中最重要的真核微生物,其群落的结构和功能对白酒品质控制有很重要的影响[8]。因此,解析白酒发酵过程中酵母群落的结构和功能对于提高白酒品质具有积极意义。
虽然绝大部分酵母菌株可以通过可培养的方法获得,但是对于酵母在微生物群落中所占的比例以及发挥的功能还有很多未知之处。目前,内转录间隔区(internal transcribed spacer,ITS)扩增子测序技术可以深入分析微生物群落结构[9],这一成熟的技术已经大规模应用于土壤、海洋、发酵微生物群落中结构的解析[10-12],但是这种研究手段存在引物特异性和偏好性,可能会特异性扩增某些微生物的基因片段,从而导致发酵过程中鉴别的微生物种类和数量出现偏差[13]。随着高通量测序技术的进一步发展,宏转录(metatranscriptomic)测序技术可以更深入地分析群落中微生物所有功能基因的表达情况,确定每种微生物在群落中发挥作用的占比[14],现在这一技术开始应用于分析肠道、淡水、发酵微生物群落中核心微生物的功能[15-17]。
酱香型白酒是一种典型的固态发酵产物,其发酵过程受到多种微生物的驱动,最终形成自发稳定的微生物群落[18]。与浓香型和清香型白酒发酵过程相比,酱香型白酒发酵过程中微生物种类和数量相对复杂[19],来源也不清楚。Wu等[20]通过可培养法检测并分离出酱香型白酒微生物群落中含有的Saccharomyces cerevisiae、Zygosaccharomyces bailii、Pichia membranifaciens、Pichia kudriavzevii和Schizosaccharomyces pombe等酵母。然而,由于单一方法对于酵母群落研究的局限性,目前对于酱香型白酒发酵过程中核心酵母种类和功能的具体研究并不深入。
本研究通过运用未培养(ITS扩增子和宏转录组高通量测序技术)和可培养(菌种筛选和模拟发酵实验)技术,探究了酱香型白酒发酵过程中核心酵母的种类和功能,解析了核心酵母在发酵过程中发挥的作用,为深入探究核心酵母在白酒发酵过程中的作用提供了重要的科学理论依据。
1 材料与方法 1.1 样品收集样品收集于贵州省某酒厂提供的典型酱香型白酒发酵酒醅。酱香型白酒具有堆积发酵和入池发酵两个阶段,所以跟踪取样了酱香型白酒某一轮次的整个发酵阶段。选择堆积和入池发酵酒醅的不同位置和不同发酵时间进行了取样,每个位置大约取样200 g。入池发酵的时间大约为30 d,每隔5 d取样,时间分别为第0天(发酵初始)、第5天、第10天、第15天、第20天、第25天和第30天,所有收集好的样品在−80 ℃冰箱中保存。
1.2 主要试剂和仪器磷酸二氢钠、磷酸氢二钠、10 mmol/L Tris-HCl (pH 8.0)、氯化钠、三氯甲烷、异戊醇、乙醇购自国药集团化学试剂(北京)有限公司;月桂酸钠和TRIzol试剂购自西格玛奥德里奇(中国)有限公司;饱和酚溶液购自生工生物工程(上海)股份有限公司。高速冷冻离心机,Eppendorf公司;NanoDrop 8000蛋白核酸测定分光光度计,Thermo Fisher Scientific公司;Illumina MiSeq PE300和HiSeq测序平台,Illumina公司;StepOnePlus实时PCR系统,Applied Biosystems公司。
1.3 核酸提取及测序将所有样品用无菌磷酸盐缓冲液(PBS,0.1 mol/L)处理,10 000×g离心10 min,然后用液氮研磨沉淀细胞,用月桂酸钠缓冲液(月桂酸钠10 g/L,Tris-HCl 0.1 mol/L,NaCl 0.1 mol/L,乙二胺四乙酸0.02 mol/L)对样品基因组总DNA进行提取。利用2%的琼脂糖凝胶电泳和NanoDrop仪器检测基因组总DNA的纯度和浓度,将提取好的基因组总DNA保存在−40 ℃的冰箱中以备后续实验使用。采用Trizol法对样本的宏转录组总RNA进行提取,利用1%的琼脂糖凝胶电泳和NanoDrop仪器检测转录组总RNA的纯度和浓度,将提取好的转录组总RNA保存在−80 ℃的冰箱中以备后续实验使用。
对于样品的基因组总DNA,采用的引物为its1 (5′-CTTGGTCATTTAGAGGAAGTAA-3′)和its2 (5′-TGCGTTCTTCATCGATGC-3′)。每个样本进行3次重复的PCR扩增,PCR的反应体系和反应条件参考Hertz等[21]的方法。将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测。之后构建MiSeq文库,使用Illumina MiSeq PE300测序平台进行测序,获得样品中真核微生物群落的ITS扩增子基因序列。
对于样品的宏转录组总RNA,通过试剂盒去除rRNA。随后将mRNA打断成短片段,以mRNA为模板,用六碱基随机引物合成一链cDNA,然后加入缓冲液、dNTPs和DNA polymerase I和RNase H合成双链cDNA,再用AMPure XP beads纯化双链cDNA。纯化的双链cDNA先进行末端修复、加A尾并连接测序接头,再用AMPure XP beads进行片段大小选择,最后进行PCR扩增,PCR的反应体系和反应条件参考Hampton-Marcell等[22]的方法,并用AMPure XP beads纯化PCR产物,得到最终的文库进行Illumina HiSeq测序。
1.4 生物信息学分析将样品的真核微生物群落ITS扩增子基因序列通过QIIME软件进行分析处理,去掉质量不好以及含嵌合体的序列,并将相似度大于97%的序列聚类为一个操作分类单元(operational taxonomic unit,OTU)。将具有代表性的OTU基因序列通过NCBI的BLAST软件与其数据库中的序列进行比对,获得OTU的物种信息。然后,将高质量的基因序列也通过NCBI的BLAST软件与其数据库中的序列进行比对,获得基因序列的物种信息,并且通过MEGA软件构建酱香型白酒微生物群落的系统发育树。同时,将样品的宏转录组基因序列通过Trimmomatic软件进行处理,去除核糖体RNA,去掉质量不好的序列,然后将高质量的基因序列与KEGG (kyoto encyclopedia of genes and genomes)数据库中的序列进行比对,获得宏转录组基因序列的基因物种和功能的信息。
1.5 模拟发酵实验首先从酱香型白酒发酵过程中筛选核心酵母,使用酱香型白酒生产用的高粱浸出液作为培养基,将长出的酵母类似菌株分离纯化后转移至试管斜面于4 ℃下鉴定及保藏,最后采用真核微生物通用正向引物ITS1 (5′-TCCGTAGGTGAACCTGCGG-3′)和反向引物ITS4 (5′-TCCTCCGCTTATTGATATG C-3′)进行PCR分子鉴定。PCR反应体系(50 μL):Taq DNA Polymerase (5 U/μL) 0.5 μL,dNTP Mix (10 mmol/L) 1 μL,上、下游引物(10 μmol/L)各2 μL,10×Taq Buffer (Mg2+ plus) 5 μL,基因组模板DNA 10 ng,超纯水补足至50 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 15 s,55 ℃ 30 s,72 ℃ 45 s,共27个循环;72 ℃ 10 min。将PCR分子鉴定结果与NCBI网站系统中模式菌株的ITS扩增子序列进行对比,得到目标菌株的具体信息。最终获得4株核心酵母S. cerevisiae C-3、P. kudriavzevii C-16、Schi. pombe C-11和Z. bailii C-7。
为了探究酱香型白酒发酵过程中这4种核心酵母在酵母群落中的作用,将4种酵母组合在一起,设置了不同产物胁迫的模拟实验。首先将4株酵母于30 ℃、200 r/min的条件下在种子培养基中(高粱浸出液)培养24 h[23]。然后将每种酵母所属种子液的10%接种到发酵培养基(高粱浸出液)中,初始细胞密度达到8×106个细胞/mL,于30 ℃、200 r/min的条件下再混合培养72 h,未培养的高粱提取物也作为阴性对照进行培养。所有实验均独立重复3次。
1.6 实时荧光定量PCR通过实时定量PCR测定体外发酵实验中的4种核心酵母S. cerevisiae C-3、P. kudriavzevii C-16、Schi. pombe C-11和Z. bailii C-7。如表 1所示,使用通过primer-BLAST工具提供的算法设计引物。为了确定扩增特异性,在最后一个循环后进行另外的解离曲线分析,在所有情况下均显示一个单峰。所有基因组DNA都有3个平行,对每个分析的样品进行3次重复实时定量PCR反应。
| 引物名称 Primers name |
引物序列 Primers sequence (5′→3′) |
长度 Size (bp) |
参考文献 References |
| PkF | GTTTGAGCGTCGTTTCCATC | 20 | [24] |
| PkR | AGCTCCGACGCTCTTTACAC | 20 | |
| ScF | GTGCGCGGTCTTGCTAGGCT | 20 | 本研究Our research |
| ScR | TACCTCTGGGCCCCGATTGC | 20 | |
| SpF | AGTGAAGCGGGAAAAGCTCA | 20 | 本研究Our research |
| SpR | ATCGACCAAAGACGGGGTTC | 20 | |
| ZbF | CATGGTGTTTTGCGCC | 16 | [25] |
| ZbR | CGTCCGCCACGAAGTGGTAGA | 21 |
原位和模拟实验中采用高效液相色谱法测定乙醇含量,采用超高效液相色谱法测定有机酸含量[18]。
2 结果与分析 2.1 酱香型白酒发酵过程中主要代谢产物变化在酒精饮料的工业化生产中,乙醇产生的含量和速率是确定发酵过程是否正常、稳定的重要指标。在白酒生产过程中,出酒率作为极其重要的指标更受到生产者的关注[17]。在白酒发酵过程中,微生物会通过代谢产生一定种类和数量的有机酸类物质,能够使酒的味道更丰富、口感更好。在有机酸代谢产物的形成中,乳酸是主要的酸类代谢产物[7]。如图 1A所示,发酵前期(发酵初始到发酵第15天)乙醇含量呈上升趋势,在发酵第25天达到40.43±0.21 g/kg发酵酒醅,到发酵后期又有轻微下降。如图 1B所示,乳酸浓度含量在发酵前期基本维持不变,发酵后期(发酵第15天到发酵结束)有小幅上升,达到26.03±3.12 g/kg发酵酒醅,乳酸不易挥发的特性有利于乳酸含量的不断积累。这些结果表明酱香型白酒发酵过程中微生物群落具有一定的乙醇和乳酸耐受性。
|
| 图 1 酱香型白酒发酵过程中主要产物的变化过程 Figure 1 The profiles of major metabolites during Maotai-flavor Baijiu production 注:A:乙醇含量变化情况;B:乳酸含量变化情况. Note: A: The change of ethanol content; B: The change of lactic acid content. |
|
|
通过对ITS扩增子的测序结果进行序列比对发现,在整个酱香型白酒发酵过程中酵母属微生物是发酵过程中的绝对真核微生物,包括Pichia、Saccharomyces、Saccharomycopsis、Schizosaccharomyces、Zygosaccharomyces等10个属,共计13种酵母(表 2)。
| 酵母(属水平) Yeast (genus) |
近似种属 Similar strains |
GenBank 登录号 GenBank accession No. |
相似度 Similarity (%) |
| Galactomyces | Galactomyces candidum | JN974289.1 | 99 |
| Kazachstania | Kazachstania humilis | KY705010.1 | 100 |
| Komagataella | Komagataella phaffii | LT962476.1 | 100 |
| Naumovozyma | Naumovozyma castellii | HE576754.1 | 100 |
| Pichia | Pichia fermentans | KY104544.1 | 100 |
| Pichia kudriavzevii | KY104586.1 | 100 | |
| Pichia manshurica | KY104603.1 | 100 | |
| Pichia membranifaciens | KY104630.1 | 100 | |
| Saccharomyces | Saccharomyces cerevisiae | KY109257.1 | 100 |
| Saccharomycopsis | Saccharomycopsis fibuligera | KP132599.1 | 100 |
| Schizosaccharomyces | Schizosaccharomyces pombe | KY105378.1 | 99 |
| Wickerhamomyces | Wickerhamomyces anomalus | KY105896.1 | 100 |
| Zygosaccharomyces | Zygosaccharomyces bailii | KY106025.1 | 89 |
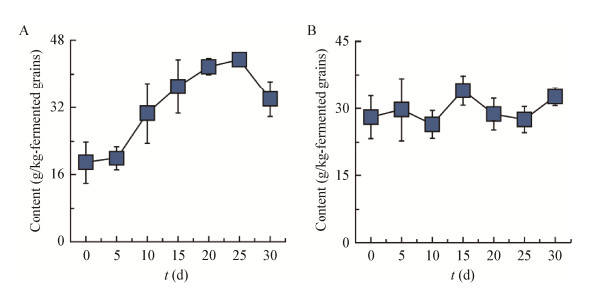
选取平均相对丰度大于0.2%的代表性OTU基因序列与NCBI数据库中的序列进行比对后,通过邻接(neighbor-joining)法建构主要真核微生物的系统发育树(图 2)。由图 2可知,酱香型白酒发酵过程中潜在的核心酵母可能有5种,分别是P. fermentans、P. kudriavzevii、S. cerevisiae、Saccharomycopsis fibuligera和Schi. pombe。
|
| 图 2 酱香型白酒发酵过程中核心酵母的系统发育树 Figure 2 Phylogenetic tree of core yeasts during Maotai-flavor Baijiu production 注:括号内为GenBank登录号;分支上的数字为1 000次重复后获得的置信值;标尺表示100个核苷酸中有10个被替换. Note: Numbers in parentheses are GenBank accession numbers; Numbers at the branches are bootstrap values obtained after 1 000 replicates; The scale bar represents 10 substitutions per 100 nucleotide positions. |
|
|
在之前的研究中,酵母中的P. kudriavzevii和S. cerevisiae在酱香型白酒的乙醇生成过程中起着决定性的作用[26]。P. kudriavzevii不仅能够产生乙醇,还能够产生苯乙醇等多种对白酒风味有重要贡献的代谢产物[27]。随着对乙醇发酵过程的深入研究,研究者对于Schi. pombe的认识也进一步加深[28]。在葡萄酒发酵过程中,Schi. pombe消耗的初级氨基氮较少,产生的尿素和丙酮酸较多,而且能够产生大量的乙醇和乙酸[29]。
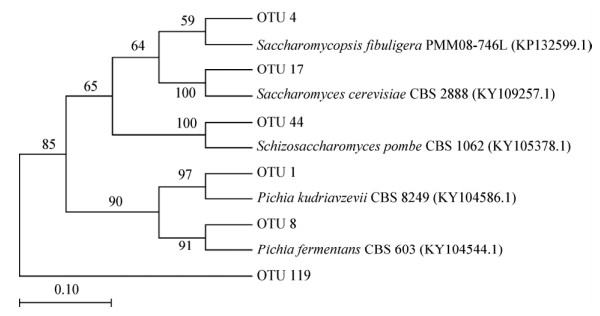
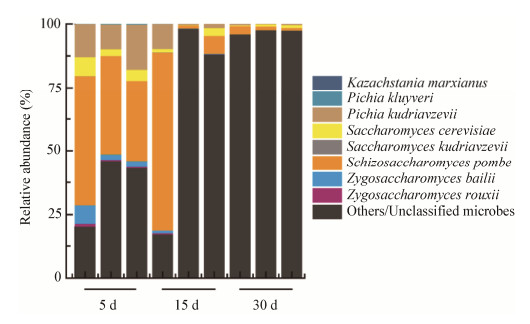
2.3 酱香型白酒发酵过程中酵母群落结构和功能的变化规律在酱香型白酒生产整个过程中酵母属占据了真核微生物群落中的绝大部分丰度(图 3)。在整个发酵阶段Pichia属占真核群落总数的90%以上,其次是Saccharomyces属和Saccharomycopsis属。随着发酵的进行,P. kudriavzevii的相对丰度逐渐下降,其他的主要真核微生物Sac. fibuligera、S. cerevisiae、Schi. pombe和P. fermentans的相对丰度都有显著上升。从整个入池发酵过程来看,除了P. kudriavzevii,其他酵母也逐渐发挥自己在微生物群落中的作用。通过ITS扩增子测序技术可以鉴定出在酱香型白酒发酵前15天,P. kudriavzevii酵母是酱香型白酒发酵微生物群落中的核心酵母(相对丰度 > 89%)。之前的研究表明P. kudriavzevii具有良好的耐受性,能够在极端环境下快速生长[30]。因此,P. kudriavzevii保证了微生物群落和发酵过程的稳定性[31],使得酱香型白酒发酵过程具有了自发性以及可重复性的特征。
|
| 图 3 酱香型白酒发酵过程中主要酵母群落的结构分布 Figure 3 The structural distributions in the yeast microbiota during Maotai-flavor Baijiu production |
|
|
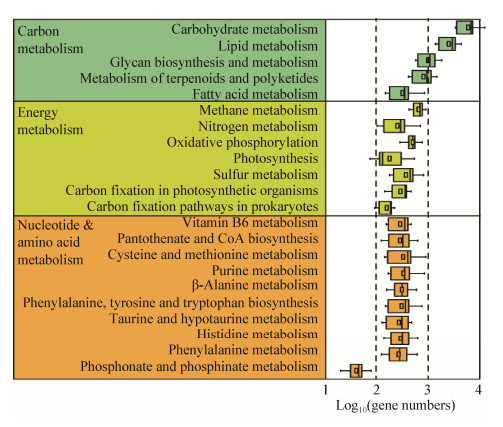
通过对酱香型白酒发酵微生物群落的宏转录组进行分析,主要的代谢途径集中在碳代谢、能量代谢以及核酸氨基酸代谢途径中(图 4)。其中碳代谢中碳水化合物相关代谢途径基因的种类和数量表达得最多。在碳水化合物代谢中,丙酮酸代谢是最主要的代谢途径。在酱香型白酒中产生最多的乙醇和乳酸是丙酮酸代谢的主要代谢产物。同时宏转录组中的所有表达基因与KEGG数据库进行比对,发现在酱香型白酒发酵酒醅中Schi. pombe、P. kudriavzevii、S. cerevisiae和Z. bailii这4种酵母的功能基因序列表达量(fragments per kilobase of transcript per million fragments mapped,FPKM)占微生物群落总功能基因序列表达量的30%以上,其中Schi. pombe占表达量21%以上,其他的基因表达序列主要是细菌、古菌以及其他未知微生物等(图 5)。在酱香型白酒发酵的前15天,Schi. pombe的表达基因在真核群落中占绝对优势,说明酱香型白酒发酵中Schi. pombe是核心酵母,可能也是主要的产醇酵母。在之前的研究中发现Schi. pombe不仅具有良好的产醇能力,还能够降低乙酸、苹果酸等酸类代谢产物[32]及一些有害物质对于微生物群落中其他酵母的影响[3]。因此,在酱香型白酒发酵前15天Schi. pombe能够调控主要醇类风味代谢产物尤其是乙醇的产生,对酱香型白酒的产醇能力有巨大的影响。
|
| 图 4 酱香型白酒发酵过程中微生物群落的主要代谢途径 Figure 4 The major metabolism in the microbiota during Maotai-flavor Baijiu production |
|
|
|
| 图 5 酱香型白酒发酵过程中主要酵母群落的功能基因表达变化 Figure 5 The changes of gene expression in the yeast microbiota during Maotai-flavor Baijiu production |
|
|
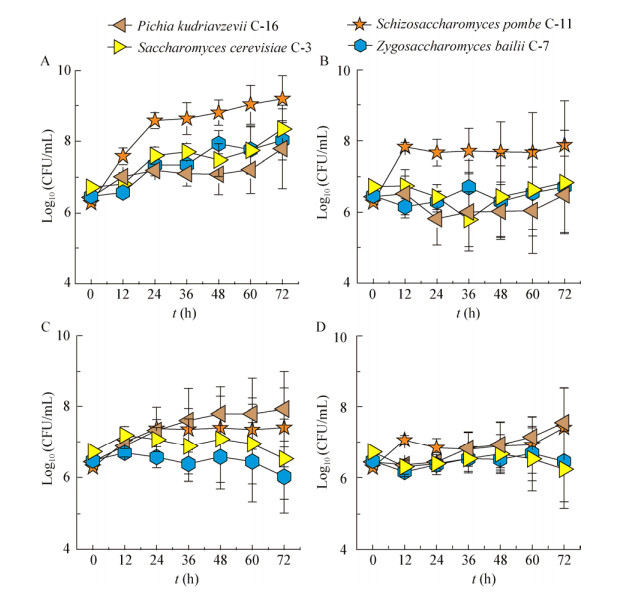
为了探索Schi. pombe和P. kudriavzevii两种核心酵母在酱香型白酒酵母群落中的具体作用,通过筛选获得的P. kudriavzevii C-16、S. cerevisiae C-3、Schi. pombe C-11和Z. bailii C-7构建了模拟发酵的酵母群落。根据之前对主要代谢产物乙醇和乳酸含量的测定,研究将模拟发酵实验分成4组:第1组为对照组,不添加乙醇和乳酸;第2组在培养基中有初始含量为40 g/L的乙醇;第3组在培养基中有初始含量为30 g/L的乳酸;第4组在培养基中有初始含量为40 g/L的乙醇和30 g/L的乳酸(图 6)。研究发现不同的初始条件下4种核心酵母菌株的生长情况是不一样的。在不添加乙醇和乳酸的对照组中,所有酵母都有比较好的生长。在初始含量为40 g/L乙醇的实验组中,只有Schi. pombe C-11的生长情况比较好,其他3株酵母的生长都受到了抑制;在初始含量为30 g/L乳酸的实验组中,只有P. kudriavzevii C-16的生长情况比较好,其他3株酵母的生长都受到了不同程度的抑制;在初始含量为40 g/L乙醇和30 g/L乳酸的实验组中,P. kudriavzevii C-16和Schi. pombe C-11的生长情况虽然都受到了一定抑制,但还维持了基本的微生物数量。综上所述,P. kudriavzevii C-16可能在酵母群落中主要有一定的耐乳酸特性,而Schi. pombe C-11可能在酵母群落中主要有一定的耐乙醇特性。
|
| 图 6 模拟发酵实验中4种核心酵母的耐受差异 Figure 6 The tolerant divergence of 4 core functional yeasts in the simulated experiments 注:A:不添加初始含量为40 g/L乙醇和30 g/L乳酸的对照组;B:添加初始含量为40 g/L乙醇的实验组;C:添加初始含量为30 g/L乳酸的实验组;D:同时添加初始含量为40 g/L乙醇和30 g/L乳酸的实验组. Note: A: Control group without initial content of 40 g/L ethanol and 30 g/L lactic acid; B: Test group with initial content of 40 g/L ethanol; C: Test group with initial content of 30 g/L lactic acid; D: Test group with initial content of 40 g/L ethanol and 30 g/L lactic acid. |
|
|
在过去的研究中,由于技术和分析手段的局限,通常认为P. kudriavzevii是发酵过程中的绝对优势结构和功能酵母[32],对于Schi. pombe在酱香型白酒发酵中的作用基本还是未知[17]。然而通过模拟发酵实验发现,P. kudriavzevii和Schi. pombe在酱香型白酒酵母群落中有不同的作用,这表明了酱香白酒发酵微生物群落虽然具有多样性和复杂性的特征,但P. kudriavzevii和Schi. pombe这两种酵母在群落中具有核心功能,能够发挥着决定性的作用。
3 讨论与结论在白酒发酵过程中,酵母是不可或缺的主要微生物。但长期以来,大多数研究者只通过ITS扩增子测序对酱香型白酒发酵真核微生物群落结构进行研究,而没有可靠的办法确定群落中真核微生物发挥的功能大小[31]。随着宏转录组等新组学技术的成熟[17]以及和可培养技术的联用[3],对酱香型白酒发酵微生物群落中酵母功能的研究越来越深入。
本研究联合使用未培养(ITS扩增子和宏转录组高通量测序技术)和可培养(菌种筛选和模拟发酵实验)技术,跟踪研究了酱香型白酒发酵过程中的酵母群落结构和功能,从菌群结构和功能表达两方面阐述了酱香型白酒发酵过程中酵母的变化情况。P. kudriavzevii和Schi. pombe是酱香型白酒发酵过程中的优势酵母。这两类酵母有各自不同的功能,P. kudriavzevii随着发酵的进行能够耐受其他微生物的产物胁迫(如乳酸积累),在发酵过程中起到了群落骨架的作用,保证了酱香型白酒多轮次发酵的稳定性。此外,Schi. pombe不仅能够耐受乳酸积累,还对酱香型白酒的产醇能力有很大的影响。本研究进一步加深了研究者对于酱香型白酒核心微生物,尤其是加深了核心微生物对于整个生产过程贡献大小的认识,这会促使生产者加强白酒发酵过程中对核心微生物的控制以及对发酵进程的调节,最终调控酱香型白酒风味代谢产物的种类和含量,提升酱香型白酒的品质。
| [1] |
Martin SG. Editorial overview: Eukaryotic microbes: models and beyond[J]. Current Opinion in Microbiology, 2015, 28: v-vi. DOI:10.1016/j.mib.2015.08.008 |
| [2] |
Zou W, Zhao CQ, Luo HB. Diversity and function of microbial community in Chinese strong-flavor baijiu ecosystem: a review[J]. Frontiers in Microbiology, 2018, 9: 671. DOI:10.3389/fmicb.2018.00671 |
| [3] |
Du H, Song ZW, Xu Y. Ethyl carbamate formation regulated by lactic acid bacteria and nonconventional yeasts in solid-state fermentation of Chinese Moutai-flavor liquor[J]. Journal of Agricultural and Food Chemistry, 2018, 66(1): 387-392. DOI:10.1021/acs.jafc.7b05034 |
| [4] |
Bochdansky AB, Clouse MA, Herndl GJ. Eukaryotic microbes, principally fungi and labyrinthulomycetes, dominate biomass on bathypelagic marine snow[J]. The ISME Journal, 2017, 11(2): 362-373. DOI:10.1038/ismej.2016.113 |
| [5] |
Berg G, Krause R, Mendes R. Cross-kingdom similarities in microbiome ecology and biocontrol of pathogens[J]. Frontiers in Microbiology, 2015, 6: 1311. |
| [6] |
Marsit S, Dequin S. Diversity and adaptive evolution of Saccharomyces wine yeast: a review[J]. FEMS Yeast Research, 2015, 15(7): fov067. DOI:10.1093/femsyr/fov067 |
| [7] |
Jin GY, Zhu Y, Xu Y. Mystery behind Chinese liquor fermentation[J]. Trends in Food Science & Technology, 2017, 63: 18-28. |
| [8] |
Wang XS, Du H, Zhang Y, et al. Environmental microbiota drives microbial succession and metabolic profiles during Chinese liquor fermentation[J]. Applied and Environmental Microbiology, 2018, 84(4): e02369-17. |
| [9] |
Ji L, Liu CJ, Zhang L, et al. Variation of rDNA internal transcribed spacer sequences in Rhizoctonia cerealis[J]. Current Microbiology, 2017, 74(7): 877-884. DOI:10.1007/s00284-017-1258-2 |
| [10] |
Schoch CL, Seifert KA, Huhndorf S, et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for fungi[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(16): 6241-6246. DOI:10.1073/pnas.1117018109 |
| [11] |
Risi MM, Macdonald AHH. Molecular examination of rocky shore brachycnemic zoantharians (Anthozoa: Hexacorallia) and their Symbiodinium symbionts (Dinophyceae) in the southwest Indian Ocean[J]. Marine Biodiversity, 2016, 46(1): 113-127. DOI:10.1007/s12526-015-0331-y |
| [12] |
Hu YL, Dun YH, Li SN, et al. Changes in microbial community during fermentation of high-temperature Daqu used in the production of Chinese 'Baiyunbian' liquor[J]. Journal of the Institute of Brewing, 2017, 123(4): 594-599. DOI:10.1002/jib.455 |
| [13] |
Anderson IC, Campbell CD, Prosser JI. Potential bias of fungal 18S rDNA and internal transcribed spacer polymerase chain reaction primers for estimating fungal biodiversity in soil[J]. Environmental Microbiology, 2003, 5(1): 36-47. DOI:10.1046/j.1462-2920.2003.00383.x |
| [14] |
Bailly J, Fraissinet-Tachet L, Verner MC, et al. Soil eukaryotic functional diversity, a metatranscriptomic approach[J]. The ISME Journal, 2007, 1(7): 632-642. DOI:10.1038/ismej.2007.68 |
| [15] |
Gosalbes MJ, Durbán A, Pignatelli M, et al. Metatranscriptomic approach to analyze the functional human gut microbiota[J]. PLoS One, 2011, 6(3): e17447. DOI:10.1371/journal.pone.0017447 |
| [16] |
Edwardson CF, Hollibaugh JT. Metatranscriptomic analysis of prokaryotic communities active in sulfur and arsenic cycling in Mono Lake, California, USA[J]. The ISME Journal, 2017, 11(10): 2195-2208. DOI:10.1038/ismej.2017.80 |
| [17] |
Song ZW, Du H, Zhang Y, et al. Unraveling core functional microbiota in traditional solid-state fermentation by high-throughput amplicons and metatranscriptomics sequencing[J]. Frontiers in Microbiology, 2017, 8: 1294. DOI:10.3389/fmicb.2017.01294 |
| [18] |
McGovern PE, Zhang J, Tang J, et al. Fermented beverages of pre- and proto-historic China[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(51): 17593-17598. DOI:10.1073/pnas.0407921102 |
| [19] |
Xu Y, Ji K. Moutai (Maotai): production and sensory properties[A]//Piggott J. Alcoholic Beverages[M]. Oxford: Woodhead Publishing, 2012: 315-330
|
| [20] |
Wu Q, Chen LQ, Xu Y. Yeast community associated with the solid state fermentation of traditional Chinese Maotai-flavor liquor[J]. International Journal of Food Microbiology, 2013, 166(2): 323-330. DOI:10.1016/j.ijfoodmicro.2013.07.003 |
| [21] |
Hertz M, Jensen IR, Jensen LØ, et al. The fungal community changes over time in developing wheat heads[J]. International Journal of Food Microbiology, 2016, 222: 30-39. DOI:10.1016/j.ijfoodmicro.2016.01.018 |
| [22] |
Hampton-Marcell JT, Moormann SM, Owens SM, et al. Preparation and metatranscriptomic analyses of host-microbe systems[J]. Methods in Enzymology, 2013, 531: 169-185. DOI:10.1016/B978-0-12-407863-5.00009-5 |
| [23] |
Meng X, Wu Q, Wang L, et al. Improving flavor metabolism of Saccharomyces cerevisiae by mixed culture with Bacillus licheniformis for Chinese Maotai-flavor liquor making[J]. Journal of Industrial Microbiology & Biotechnology, 2015, 42(12): 1601-1608. |
| [24] |
Zott K, Claisse O, Lucas P, et al. Characterization of the yeast ecosystem in grape must and wine using real-time PCR[J]. Food Microbiology, 2010, 27(5): 559-567. DOI:10.1016/j.fm.2010.01.006 |
| [25] |
Xu Y, Zhi Y, Wu Q, et al. Zygosaccharomyces bailii is a potential producer of various flavor compounds in Chinese Maotai-flavor liquor fermentation[J]. Frontiers in Microbiology, 2017, 8: 2609. DOI:10.3389/fmicb.2017.02609 |
| [26] |
Chen B, Wu Q, Xu Y. Filamentous fungal diversity and community structure associated with the solid state fermentation of Chinese Maotai-flavor liquor[J]. International Journal of Food Microbiology, 2014, 179: 80-84. DOI:10.1016/j.ijfoodmicro.2014.03.011 |
| [27] |
Xu D, Fan GS, Fu ZL, et al. Screening of a high-yield β-phenylethanol yeast and optimization of its cultural conditions[J]. Science and Technology of Food Industry, 2017, 38(5): 151-158. (in Chinese) 许岱, 范光森, 富志磊, 等. 一株高产β-苯乙醇酵母菌的筛选、鉴定及其发酵条件优化[J]. 食品工业科技, 2017, 38(5): 151-158. |
| [28] |
Tura A, Fontana RC, Camassola M. Schizosaccharomyces pombe as an efficient yeast to convert sugarcane bagasse pretreated with ionic liquids in ethanol[J]. Applied Biochemistry and Biotechnology, 2018, 186(4): 960-971. DOI:10.1007/s12010-018-2788-1 |
| [29] |
Benito S, Palomero F, Morata A, et al. New applications for Schizosaccharomyces pombe in the alcoholic fermentation of red wines[J]. International Journal of Food Science & Technology, 2012, 47(10): 2101-2108. |
| [30] |
Wang XD, Pang B, Lu AM, et al. Study on the yeast change trend in the process of accumulation of Maotai-flavor Liquor[J]. The Food Industry, 2014, 35(12): 42-44. (in Chinese) 王晓丹, 庞博, 陆安谋, 等. 酱香型白酒堆积过程中酵母变化趋势研究[J]. 食品工业, 2014, 35(12): 42-44. |
| [31] |
Yuangsaard N, Yongmanitchai W, Yamada M, et al. Selection and characterization of a newly isolated thermotolerant Pichia kudriavzevii strain for ethanol production at high temperature from cassava starch hydrolysate[J]. Antonie van Leeuwenhoek, 2013, 103(3): 577-588. DOI:10.1007/s10482-012-9842-8 |
| [32] |
Benito Á, Jeffares D, Palomero F, et al. Selected Schizosaccharomyces pombe strains have characteristics that are beneficial for winemaking[J]. PLoS One, 2016, 11(3): e0151102. DOI:10.1371/journal.pone.0151102 |
 2020, Vol. 47
2020, Vol. 47