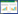扩展功能
文章信息
- 韩如梦, 柳鑫燕, 林文萱, 李翔飞, 杨套伟, 徐美娟, 邵明龙, 张显, 饶志明
- HAN Ru-Meng, LIU Xin-Yan, LIN Wen-Xuan, LI Xiang-Fei, YANG Tao-Wei, XU Mei-Juan, SHAO Ming-Long, ZHANG Xian, RAO Zhi-Ming
- 枯草芽孢杆菌氧化还原感应全局调控因子Rex对乙偶姻合成的影响
- Effect of global redox-sensing transcriptional regulator Rex on acetoin synthesis of Bacillus subtilis
- 微生物学通报, 2020, 47(11): 3494-3503
- Microbiology China, 2020, 47(11): 3494-3503
- DOI: 10.13344/j.microbiol.china.191048
-
文章历史
- 收稿日期: 2019-12-16
- 接受日期: 2020-03-04
- 网络首发日期: 2020-04-22
乙偶姻广泛存在于苹果、可可、草莓和玉米等物质中,具有特殊的奶油香味。乙偶姻是我国明确规定的食品添加剂[1],通常被添加到烘焙食物、糖果、乳制品和饮料等食品中。同时,乙偶姻也是多种微生物的次级代谢产物,许多微生物可以利用葡萄糖合成乙偶姻或2, 3-丁二醇,而乙偶姻和2, 3-丁二醇是中性物质,可调节胞内酸碱平衡[2-3]。
枯草芽孢杆菌是被美国食品和药物管理局(Food and Drug Administration,FDA)认可的Generally recognized as safe (GRAS)菌株,遗传背景清晰,具有良好的可操作性和安全性,是一种重要的工业生产菌株。前期研究发现,枯草芽孢杆菌中乙偶姻和其下游产物2, 3-丁二醇存在着可逆转化的关系,该可逆过程需要辅酶NADH/NAD+的参与[4-5]。研究表明,枯草芽孢杆菌体内含有一种可响应胞内氧化还原水平的因子,称之为氧化还原感应全局调控因子Rex (由基因ydiH编码)[6]。Rex通过感知NADH/NAD+比率的变化来调节胞内氧化还原平衡[7],并与许多基因的启动子区域结合,抑制基因表达,该过程可被NADH抑制并由过量的NAD+解除抑制。这些可与Rex结合的基因大多参与碳和能量代谢,包括NADH氧化、产氢、ATP合成及乳酸和琥珀酸的形成[8-9]。
研究发现,基因簇acuABC和acoABCLR与乙偶姻的降解有关[10]。王萌敲除B. subtilis 168中的基因bdhA和acoA后,弱化了2, 3-丁二醇的合成和乙偶姻的降解。B. subtilis 168ΔbdhAΔacoA的乙偶姻得率较B. subtilis 168提高了10%,B. subtilis 168ΔacoA中乙偶姻几乎不会降解[11]。康艳方对B. licheniformis WX-02ΔbudC进一步敲除acoR,在一定程度上减弱了乙偶姻的分解代谢,B. licheniformis WX-02ΔbudCΔacoR的乙偶姻产量相对于B. licheniformis WX-02ΔbudC提高了11.27%[12]。
前期研究发现Rex通过调控胞内辅因子代谢来影响胞内氧化还原平衡。本实验以Bacillus subtilis 168作为底盘细胞,结合代谢工程手段和基因组编辑技术,研究Rex的全局调控功能,解析在不同发酵阶段Rex影响胞内氧化还原平衡的整体发生规律及其与乙偶姻合成的相关性(图 1)。
|
| 图 1 枯草芽孢杆菌中乙偶姻的生物合成途径 Figure 1 Acetoin biosynthetic pathway in Bacillus subtilis 注:“×”表示敲除;红色基因为转录组差异较为显著的基因;蓝色实线箭头所指基因为乙偶姻降解相关的基因. Note: "×" means knock out; Red genes are genes with significant transcriptome differences; The genes indicated by the blue solid arrows are genes involved in the degradation of acetoin. |
|
|
研究用到的微生物菌株和载体质粒均列于表 1中,敲除菌株所用到的引物均列于表 2中。
| 菌株和质粒 Strains and plasmids |
特征 Characteristics |
来源 Source |
| Strains | ||
| Bacilus subtilis | ||
| B. subtilis 168 | trpC2 | Our lab |
| B. subtilis 168/ΔydiH | ydiH knockout strain | This study |
| B. subtilis 168/ΔacuA | acuA knockout strain | This study |
| B. subtilis 168/ΔacoC | acoC knockout strain | This study |
| Plasmids | ||
| p7Z6 | pMD18-T containing lox71-zeo-lox66 cassette | [13] |
| pTSC | pTS containing Pspac-cre expression cassette | [13] |
| 引物名称 Primers name |
引物序列 Primers sequence (5′→3′) |
| P1(ydiH-L-F) | CATACGAAAGCTGGCCTGCGATAATTTTAAGGAGCGTGGATTTTC |
| P2(ydiH-L-R) | AATCATGGTCTTTTGGTCCTCCAAATTATACTCGGATAGTTCTCTTTTAAAGTCACCG |
| P3(ydiH-Z-F) | CCGAGTATAATTTGGAGGACCAAAAGACCATGATTACGAATTCGAGCTCGGTACCCGG |
| P4(ydiH-Z-R) | GCATATTTGGGCTCCTCCTTTCCCTTGTAAAACGACGGCCAGTGCCAAGCT |
| P5(ydiH-R-F) | GGCACTGGCCGTCGTTTTACAAGGGAAAGGAGGAGCCCAAATATGCCGAT |
| P6(ydiH-R-F) | AGCGTAAAATACGCATACTTTCTGATTTTCGCCAAGAACATCGGT |
| P7(acuA-L-F) | TATAGTGAAGCAGAAAGCCGTCTT |
| P8(acuA-L-R) | CGAGCTCGAATTCGTAATCATGGTCTAATTCACCGTCCCATCAATTCTTATCA |
| P9(acuA-Z-F) | TAAGAATTGATGGGACGGTGAATTAGACCATGATTACGAATTCGAGCTC |
| P10(acuA-Z-R) | ATTTTTTCCCCTTTGTCAGTGTCAGTGTAAAACGACGGCCAGTGC |
| P11(acuA-R-F) | GCTTGGCACTGGCCGTCGTTTTACACTGACACTGACAAAGGGGAAA |
| P12(acuA-R-F) | AAGGGATGCTCCTGATGAAACATATAGG |
| P13(acoC-L-F) | ATCAACCGGTTTGAGACCGATTG |
| P14(acoC-L-R) | CGAGCTCGAATTCGTAATCATGGTCTTGTGTCCCCCCTTTAATTCAATGC |
| P15(acoC-Z-F) | GCATTGAATTAAAGGGGGGACACAAGACCATGATTACGAATTCGAGCTC |
| P16(acoC-Z-R) | GTCATGTCGTTTTCACCTGCTTTTCTGTAAAACGACGGCCAGTGC |
| P17(acoC-R-F) | GCTTGGCACTGGCCGTCGTTTTACAGAAAAGCAGGTGAAAACGACATGAC |
| P18(acoC-R-F) | CACATAATCGGCCTTCGTTTTAAACTC |
质粒小量制备试剂盒、琼脂糖凝胶DNA回收试剂盒和细菌基因组DNA快速提取试剂盒,上海捷瑞生物工程有限公司;2×Phanta Max Master Mix、FastPure Cell/Tissue Total RNA Isolation Kit、HiScript Q Setect RT SuperMix for qPCR和ChamQ Universal SYBR qPCR Master Mix,南京诺唯赞生物科技有限公司;卡那霉素,生工生物工程(上海)股份有限公司;博来霉素(Zeor),北京索莱宝科技有限公司;工具酶试剂,宝生物工程(大连)有限公司;引物合成和核酸测序由苏州金唯智生物科技有限公司完成。实时荧光定量PCR仪,Applied Biosystems公司。
液体LB培养基:大肠杆菌和枯草芽孢杆菌的活化培养基及发酵所用的种子培养基;固体培养基为添加20 g/L琼脂粉的液体LB培养基。
发酵培养基(g/L):酵母浸膏5.0,尿素2.0,玉米浆6.0,葡萄糖100.0。
1.3 感受态细胞的制备与转化枯草芽孢杆菌感受态细胞的制备和转化方法参照参考文献[14]。
1.4 转录组测序技术(RNA-seq)挑取B. subtilis 168平板上的单菌落,接种于10 mL LB培养基中,37 ℃、180 r/min培养10 h;转接6%菌液于50 mL种子培养基中,37 ℃、180 r/min培养10 h;转接6%菌液于50 mL发酵培养基中,37 ℃、180 r/min培养96 h。取24 h (命名BS24h)和96 h (命名BS96h)的发酵液,8 000 r/min离心10 min,去除上清液,收集菌体,经液氮速冻10 min,送往南京诺唯赞生物科技有限公司进行转录组测序。
1.5 ydiH、acuA和acoC的敲除从NCBI网站公布的B. subtilis 168基因组序列中确定要敲除的目的基因的序列位置,并下载目的基因前后各800 bp大小的一段DNA片段作为上、下游同源臂。以B. subtilis 168基因组为模板,PCR扩增获得目的基因的上、下游同源臂;以p7Z6质粒为模板,PCR扩增获得抗性筛选标记表达盒lox71-zeo-lox66[13]。PCR反应体系(50 μL):2×Phanta Max Master Mix 25 μL,上、下游引物(10 μmol/L)各0.5 μL,模板DNA (100 ng/μL) 1 μL,ddH2O 23 μL。PCR反应条件:98 ℃ 3 min;98 ℃ 30 s,59 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 5 min。利用重叠延伸PCR技术将3条纯化回收后的DNA片段融合扩增得到一条2.2 kb左右的重组DNA片段。PCR反应体系(50 μL):2×Phanta Max Master Mix 25 μL,上游同源臂的上游引物(10 μmol/L) 1 μL,下游同源臂的下游引物(10 μmol/L) 1 μL,上游同源臂片段(100 ng/μL) 2.5 μL,lox71-zeo-lox66片段(100 ng/μL) 2.5 μL,下游同源臂片段(100 ng/μL) 2.5 μL,ddH2O 15.5 μL。PCR反应条件除延伸时间改为90 s,其余设置和上一步相同。PCR反应产物的纯化参照琼脂糖凝胶DNA回收试剂盒的说明书。
按照1.3所述的转化方法将纯化回收后的重组DNA片段导入B. subtilis 168感受态细胞中,涂布于含有30 mg/L博来霉素的LB抗性平板上,于37 ℃培养箱中培养16 h,获得敲除目的基因的Zeor转化子。将温敏型质粒pTSC转入Zeor转化子的感受态细胞中,涂布于含有50 mg/L卡那霉素的LB抗性平板上,可获得无博来霉素抗性的转化子;再将所得到的转化子挑到无抗LB平板上,51 ℃培养48 h,除去质粒pTSC,获得无抗且不含pTSC的目的基因敲除菌株。
1.6 发酵参数分析方法OD600和葡萄糖的测定方法见参考文献[15]。乙偶姻含量的测定见参考文献[16]。
1.7 RNA提取和RT-qPCR分析收集不同发酵阶段的细胞,根据FastPure Cell/Tissue Total RNA Isolation Kit的使用说明书提取RNA。使用HiScript Q Setect RT SuperMix for qPCR试剂盒,将RNA (1 pg−1 μg)反转录成cDNA。使用实时荧光定量PCR仪对待测样品中特定DNA序列进行定量分析。RT-qPCR反应体系(20 μL):2×ChamQ Universal SYBR qPCR Master Mix 10 μL,上、下游引物(10 μmol/L)各0.4 μL,引物序列如表 3所示,cDNA (0.1 g/L) 1 μL,RNase-free ddH2O 8.2 μL。RT-PCR反应条件参照ChamQ Universal SYBR qPCR Master Mix说明书。以RNase-free ddH2O作为阴性对照,验证Mix和引物有无污染。以基因组DNA作为阳性对照,验证反应体系、条件和过程的正确性。
| 引物名称 Primers name |
引物序列 Primers sequence (5′→3′) |
| alsR F | TATCGCAGTAGCCGAAGAGC |
| alsR R | CTTTTCAGAAGCGTAACTCCG |
| alsS F | ACAGCGAACACTGAAGGAGA |
| alsS R | TCCAAAGATTGATGTGTCCGT |
| alsD F | AGACCGTTCACCGTTCTGTT |
| alsD R | GCGTGTCATTTTCGCATCAA |
| bdhA F | GTGTTACGACAAGCCATCCAG |
| bdhA F | ACCTTTTTCCCAAATGCTGACG |
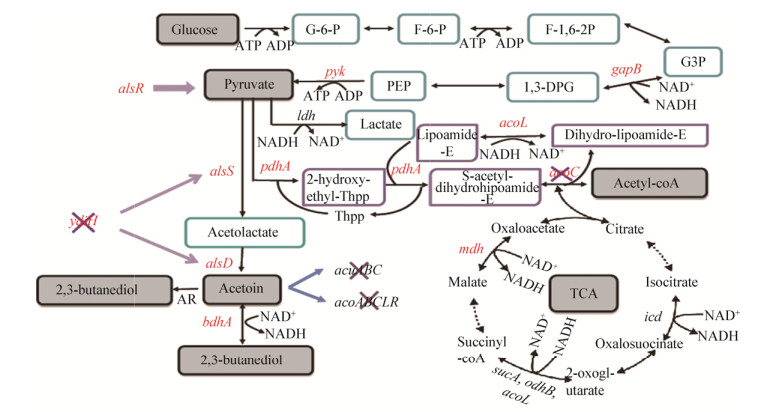
通过对转录组学测序结果进行分析发现,B. subtilis 168胞内与乙偶姻合成以及辅酶NADH/NAD+和ADP/ATP合成相关的部分基因的转录水平在发酵前期和后期有比较明显的变化,如图 2所示。发酵后期基因的转录水平和发酵前期相比,EMP途径中的gapB和pyk发生了明显的下调;TCA循环相关的基因中,mdh和pdhABCD发生了明显的上调;乙偶姻合成相关的基因中,alsR发生了下调,alsS和alsD发生了显著上调;乙偶姻降解相关的基因中,acuABC和acoABCL发生了明显的下调,bdhA和acoR发生了轻微上调。除此之外,氧化还原感应全局的调控因子编码基因ydiH的转录水平发生了轻微下调现象。通过NCBI检索发现,acuABC和acoABCLR分别为2个基因簇。研究表明其中的AcuA蛋白通过乙酰化残基Lys549控制枯草芽孢杆菌中乙酰辅酶A合成酶(AcsA)的活性[17]。然而acoC直接参与乙酰CoA的合成,与乙偶姻合成途径竞争消耗丙酮酸,如图 1所示。结合以上的分析结果,我们决定深入研究ydiH、acuA和acoC这3个基因与乙偶姻合成之间的关联性。
|
| 图 2 乙偶姻代谢途径相关基因表达水平 Figure 2 Acetoin metabolic pathway related gene expression levels 注:BS24h:B. subtilis 168发酵24 h基因的转录水平变化值;BS96h:B. subtilis 168发酵96 h基因的转录水平变化值. Log2(BS96h/BS24h) > 0表示B. subtilis 168发酵后期基因转录水平相较于发酵前期上调;Log2(BS96h/BS24h) < 0表示B. subtilis 168发酵后期基因转录水平相较于发酵前期下调. Note: BS24h: The transcription level change value of B. subtilis 168 fermentation 24 h gene; BS96h: The transcription level change value of B. subtilis 168 fermentation 96 h gene. Log2(BS96h/BS24h) > 0 means that the gene transcription level of B. subtilis 168 during the late fermentation period is increased compared with the pre-fermentation period; Log2(BS96h/BS24h) < 0 means that the gene transcription level of B. subtilis 168 during the late fermentation period is lower than the pre-fermentation period. |
|
|
以B. subtilis 168为出发菌,按照1.5所述方法得到ydiH的敲除菌株(含有基因zeo)。利用上游同源臂的上游引物ydiH-L-F和下游同源臂的下游引物ydiH-R-R进行菌落PCR验证。根据序列分析可知,ydiH的敲除菌株(含有基因zeo)经PCR扩增后可获得2 197 bp的DNA片段。将温敏型质粒pTSC导入该突变株后,可扩增获得1 768 bp的DNA片段。PCR验证结果如图 3A所示,表明基因ydiH已经敲除成功,获得无抗转化子。提取该突变株的基因组,经测序后进一步验证了ydiH敲除成功,命名该突变株为B. subtilis 168/ΔydiH。acuA和acoC敲除菌株的构建方法同上,分别命名为B. subtilis 168/ΔacuA和B. subtilis 168/ΔacoC,验证结果如图 3B和图 3C所示。

|
| 图 3 ydiH (A)、acuA (B)和acoC (C)敲除菌株PCR验证图 Figure 3 PCR verification of ydiH (A), acuA (B) and acoC (C) knockout strains 注:A:M:DL10000 DNA Marker;1:菌株B. subtilis 168/ΔydiH (Zeor)的PCR产物;2:菌株B. subtilis 168/ΔydiH的PCR产物. B:M:DL10000 DNA Marker;1:菌株B. subtilis 168/ΔacuA (Zeor)的PCR产物;2:菌株B. subtilis 168/ΔacuA的PCR产物. C:M:DL2000 DNA Marker;1:菌株B. subtilis 168/ΔacoC (Zeor)的PCR产物;2:菌株B. subtilis 168/ΔacoC的PCR产物. Note: A: M: DL10000 DNA Marker; 1: PCR product of B. subtilis 168/ΔydiH (Zeor); 2: PCR product of B. subtilis 168/ΔydiH. B: M: DL10000 DNA Marker; 1: PCR product of B. subtilis 168/ΔacuA (Zeor); 2: PCR product of B. subtilis 168/ΔacuA. C: M: DL2000 DNA Marker; 1: PCR product of B. subtilis 168/ΔacoC (Zeor); 2: PCR product of B. subtilis 168/ΔacoC. |
|
|
以往研究表明,Rex可通过NADH/NAD+比率的变化来感知胞内的氧化还原水平,并能够在不同比例NADH/NAD+条件下与DNA发生不同程度的结合,进而影响基因的表达[7]。因此,有必要研究敲除Rex编码基因后枯草芽孢杆菌合成乙偶姻的变化水平。除此之外,本研究还分别探究了acuA和acoC基因的缺失对乙偶姻合成的影响。
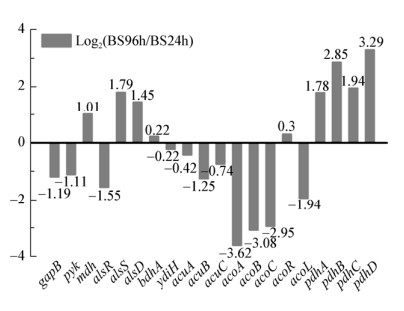
敲除菌株构建成功后,摇瓶发酵96 h,发酵参数分析结果如图 4所示。ydiH敲除菌耗糖速度明显降低,acuA和acoC敲除菌株的耗糖速率与原始菌株B. subtilis 168相比变化不大。发酵72 h,B. subtilis 168、B. subtilis 168/ΔacuA和B. subtilis 168/ΔacoC菌株将发酵液中的葡萄糖完全耗尽,而B. subtilis 168/ΔydiH菌株发酵96 h才将葡萄糖完全耗尽。发酵前期,B. subtilis 168/ΔydiH的菌体量增加缓慢,明显低于原始菌株,而acuA和acoC敲除菌株的菌体量与原始菌株相比变化不是很明显。由此可见,枯草芽孢杆菌中ydiH显著影响着菌体生长,而acuA和acoC并不是枯草芽孢杆菌生长所必需的。发酵72 h后,B. subtilis 168、B. subtilis 168/ΔacuA和B. subtilis 168/ΔacoC的菌体量急剧下降,菌体衰亡明显;而B. subtilis 168/ΔydiH的菌体量下降缓慢,菌体衰亡不太明显。3株敲除菌株与原始菌株相比,acuA和acoC的敲除对乙偶姻产量影响不明显;ydiH的敲除在发酵前期对乙偶姻产量影响较大,在发酵后期无明显优势。总而言之,ydiH的敲除虽影响了菌体的生长速率,但乙偶姻的产量高于另外3株菌。
|
| 图 4 原始菌和敲除菌发酵过程曲线 Figure 4 Fermentation process curve of original and knockout strains 注:黑色实线为发酵液中葡萄糖的剩余量;蓝色虚线为OD600;红色实线为乙偶姻的产量.发酵结果显示为3次重复的平均值±标准差. Note: The black solid line is the remaining amount of glucose in the fermentation broth; The blue dotted line is OD600; The red solid line is the yield of acetoin. The results of the fermentation are shown as the mean of three replicates±standard deviation. |
|
|
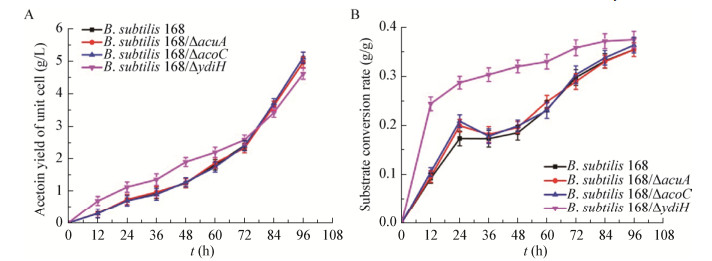
除了以上发酵参数外,还对乙偶姻单位细胞产量(乙偶姻产量/OD600)和底物转化率(乙偶姻产量/葡萄糖消耗量)进行了简要分析。如图 5A所示,发酵前期,B. subtilis 168/ΔydiH的乙偶姻单位细胞产量明显高于其他菌株。可见在发酵前期,ydiH的敲除对乙偶姻的生成很有利。除此之外,相较于其他3株菌,ydiH的敲除对底物转化率影响也很大(图 5B)。发酵前期,B. subtilis 168/ΔydiH的底物转化率增长较快,72 h后趋于平稳。可见,ydiH敲除菌株可在耗糖速率较低的情况下产生较多的乙偶姻。
|
| 图 5 乙偶姻单位细胞产量和底物转化率的曲线图 Figure 5 Graph of acetoin yield of unit cell and substrate conversion rate 注:A:乙偶姻单位细胞产量;B:底物转化率.发酵结果显示为3次重复的平均值±标准差. Note: A: Acetoin yield of unit cell; B: Substrate conversion rate. The results of the fermentation are shown as the mean of three replicates±standard deviation. |
|
|
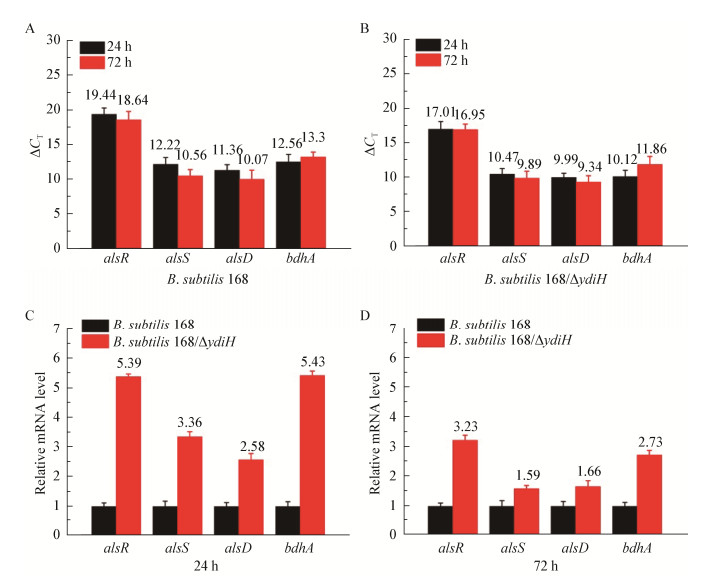
由图 4菌体量变化趋势可知,菌体在发酵72 h后开始进入衰亡期。为了探究在不同生理状态条件下枯草芽孢杆菌胞内基因的转录水平差异,选取24 h和72 h两个发酵点进行RT-qPCR分析。图 6A和图 6B分别为B. subtilis 168和B. subtilis 168/∆ydiH发酵前期和发酵后期alsR、alsS、alsD和bdhA转录水平的对比图。分析图 6A和图 6B可知,敲除ydiH不会影响枯草芽孢杆菌中alsR、alsS、alsD和bdhA发酵前后的转录趋势。不管枯草芽孢杆菌中是否含有Rex,发酵后期与发酵前期相比,alsR、alsS和alsD的转录水平都是上调,bdhA都是下调。发酵前期,bdhA转录水平较强,乙偶姻主要被用于合成2, 3-丁二醇。与发酵前期相比,发酵后期的bdhA转录水平下调,alsS和alsD的转录水平上调,表观上呈现出乙偶姻逐渐被积累的趋势。结合图 6C和图 6D分析,ydiH被敲除后,与原始菌株相比,alsR、alsS、alsD和bdhA的转录水平在发酵前后均发生不同程度的上调,且发酵前期(24 h)转录水平上调程度明显高于发酵后期(72 h),因此从发酵图 5中可以看出在敲除ydiH后,发酵前期乙偶姻单位细胞产量和底物转化率有明显的提高,而发酵后期敲除菌株与原始菌株差异不大。
|
| 图 6 B. subtilis 168和B. subtilis 168/∆ydiH的转录水平分析 Figure 6 Transcriptional level analysis of B. subtilis 168 and B. subtilis 168/∆ydiH 注:A:B. subtilis 168发酵后期(72 h)和发酵前期(24 h)转录水平分析;B:B. subtilis 168/∆ydiH发酵后期(72 h)和发酵前期(24 h)转录水平分析;C:发酵前期(24 h) B. subtilis 168和B. subtilis 168/∆ydiH的转录水平分析;D:发酵后期(72 h) B. subtilis 168和B. subtilis 168/∆ydiH的转录水平分析.结果显示为3次重复的平均值±标准差. Note: A: The transcriptional level analysis of B. subtilis 168 during the late fermentation (72 h) and pre-fermentation (24 h); B: The transcriptional level analysis of B. subtilis 168/∆ydiH during the late fermentation (72 h) and pre-fermentation (24 h); C: The transcriptional level analysis of B. subtilis 168 and B. subtilis 168/∆ydiH in the pre-fermentation period (24 h); D: The transcriptional level analysis of B. subtilis 168 and B. subtilis 168/∆ydiH at the late fermentation stage (72 h). Results are shown as the mean of three replicates±standard deviation. |
|
|
研究表明,Rex通过感知辅酶NADH/NAD+比例变化来调节胞内氧化还原水平,可与辅酶NADH/NAD+合成相关基因启动子区域结合并抑制基因的表达,如ldh (乳酸脱氢酶基因)[18]和alsSD[7]。本实验通过转录组数据分析,找到了一些与乙偶姻代谢相关且具有显著差异的基因(图 1中红色标记基因)。这些基因大多与辅酶NADH/NAD+或ADP/ATP合成有关,可能会受到Rex调控。通过Cre/lox敲除技术,实验敲除了B. subtilis 168中ydiH、acuA和acoC。经摇瓶发酵显示,ydiH的敲除可提高枯草芽孢杆菌中乙偶姻的合成和底物转化率;acuA和acoC的敲除对枯草芽孢杆菌中乙偶姻的合成和底物转化率影响较小。除此之外,ydiH的敲除还影响了枯草芽孢杆菌的生长速率。B. subtilis 168/∆ydiH菌体的生长速率发生明显的降低,可能与敲除ydiH破坏了胞内稳态环境有关。虽然敲除Rex在一定程度上影响了枯草芽孢杆菌的生长速率,但与此同时改变了碳流的分配,提高了乙偶姻的产量和底物转化率。
通过RT-qPCR分析可知,ydiH被敲除虽然未影响枯草芽孢杆菌中alsR、alsS、alsD和bdhA在发酵前后的转录趋势,但显著提高了基因的转录水平。可见枯草芽孢杆菌中的Rex显著影响着乙偶姻代谢相关基因的表达,其中alsS和alsD转录水平的提高可能与Rex能结合alsSD启动子区域有关。敲除ydiH后,Rex对alsS和alsD表达能力的阻遏作用得到解除,使得alsS和alsD的转录能力得到提高。通过NCBI检索发现,alsR和alsSD的启动子区域有158 bp碱基重叠,该重叠区域恰好包含Rex与alsSD启动子区域的结合位点[7]。由此推测,Rex能够与alsR启动子区域结合并抑制alsR表达,敲除Rex可解除对alsR转录水平的抑制,进而引起alsR转录水平大幅度的提高。有关Rex能够与alsR启动子区域结合的猜测,还需进一步的实验验证。枯草芽孢杆菌中的Rex通过感知NADH/NAD+比率的变化来调节胞内氧化还原平衡。敲除Rex后,破坏了胞内氧化还原的稳定,改变了胞内辅酶NADH/NAD+的比例。为保持胞内NADH/NAD+水平的稳定,bdhA的转录水平发生了显著上调。可见枯草芽孢杆菌中Rex可通过感知胞内辅酶NADH/NAD+水平的变化,调控alsR、alsS、alsD和bdhA的转录,进而影响乙偶姻的合成和转化。
敲除枯草芽孢杆菌中的Rex后显著影响了bdhA的转录,bdhA转录水平的上调并不利于乙偶姻的积累,反而会使乙偶姻更多地被转化成2, 3-丁二醇。课题组前期的研究已经证明敲除枯草芽孢杆菌中的bdhA并引入可调节NADH/NAD+平衡的NADH氧化酶YODC,可显著提高乙偶姻的产量[15],我们可对B. subtilis 168/∆ydiH进一步实施代谢改造,更好地完成胞内碳流的分配。本研究借助于枯草芽孢杆菌乙偶姻发酵这一过程,探索Rex是如何通过调控相关基因的转录来影响胞内氧化还原稳态的,以期在利用枯草芽孢杆菌作为工业生物底盘细胞进行代谢改造时,为提高枯草芽孢杆菌工业化生产强度和底物转化率提供借鉴。
| [1] |
Zhang XZ, Zeng CY, Ren XQ. Progress on synthesis of acetoin[J]. Jiangsu Chemical Industry, 2001, 29(2): 29-31. (in Chinese) 张小舟, 曾崇余, 任晓乾. 乙偶姻合成研究现状及展望[J]. 江苏化工, 2001, 29(2): 29-31. |
| [2] |
Speck EL, Freese E. Control of metabolite secretion in Bacillus subtilis[J]. Journal of General Microbiology, 1973, 78(2): 261-275. DOI:10.1099/00221287-78-2-261 |
| [3] |
Tsau JL, Guffanti AA, Montville TJ. Conversion of pyruvate to acetoin helps to maintain pH homeostasis in Lactobacillus plantarum[J]. Applied and Environmental Microbiology, 1992, 58(3): 891-894. DOI:10.1128/AEM.58.3.891-894.1992 |
| [4] |
Bao T, Zhang X, Rao ZM, et al. Efficient whole-cell biocatalyst for acetoin production with NAD+ regeneration system through homologous co-expression of 2, 3-butanediol dehydrogenase and NADH oxidase in engineered Bacillus subtilis[J]. PLoS One, 2014, 9(7): e102951. DOI:10.1371/journal.pone.0102951 |
| [5] |
Bao T, Zhang X, Zhao XJ, et al. Regulation of the NADH pool and NADH/NADPH ratio redistributes acetoin and 2, 3-butanediol proportion in Bacillus subtilis[J]. Biotechnology Journal, 2015, 10(8): 1298-1306. DOI:10.1002/biot.201400577 |
| [6] |
Pagels M, Fuchs S, Pané-Farré J, et al. Redox sensing by a Rex-family repressor is involved in the regulation of anaerobic gene expression in Staphylococcus aureus[J]. Molecular Microbiology, 2010, 76(5): 1142-1161. DOI:10.1111/j.1365-2958.2010.07105.x |
| [7] |
Wang E, Bauer MC, Rogstam A, et al. Structure and functional properties of the Bacillus subtilis transcriptional repressor Rex[J]. Molecular Microbiology, 2008, 69(2): 466-478. DOI:10.1111/j.1365-2958.2008.06295.x |
| [8] |
Wietzke M, Bahl H. The redox-sensing protein Rex, a transcriptional regulator of solventogenesis in Clostridium acetobutylicum[J]. Applied Microbiology and Biotechnology, 2012, 96(3): 749-761. DOI:10.1007/s00253-012-4112-2 |
| [9] |
Hu LJ, Huang HY, Yuan HX, et al. Rex in Clostridium kluyveri is a global redox-sensing transcriptional regulator[J]. Journal of Biotechnology, 2016, 233: 17-25. DOI:10.1016/j.jbiotec.2016.06.024 |
| [10] |
Huang M, Oppermann-Sanio FB, Steinbüchel A. Biochemical and molecular characterization of the Bacillus subtilis acetoin catabolic pathway[J]. Journal of Bacteriology, 1999, 181(12): 3837-3841. DOI:10.1128/JB.181.12.3837-3841.1999 |
| [11] |
Wang M. Preliminary study on metabolic engineering of Bacillus subtilis for acetoin production[D]. Tianjin: Master's Thesis of Tianjin University, 2012 (in Chinese) 王萌.产乙偶姻枯草芽孢杆菌代谢工程改造的初步研究[D].天津: 天津大学硕士学位论文, 2012 |
| [12] |
Kang YF. The function of budC gene of Bacillus licheniformis and construction of strains for enhanced production of acetoin[D]. Wuhan: Master's Thesis of Huazhong Agricultural University, 2013 (in Chinese) 康艳方.地衣芽胞杆菌budC基因的功能及高产乙偶姻菌株构建[D].武汉: 华中农业大学硕士学位论文, 2013 |
| [13] |
Yan X, Yu HJ, Hong Q, et al. Cre/lox system and PCR-based genome engineering in Bacillus subtilis[J]. Applied and Environmental Microbiology, 2008, 74(17): 5556-5562. DOI:10.1128/AEM.01156-08 |
| [14] |
Zhang X, Zhang RZ, Bao T, et al. Moderate expression of the transcriptional regulator ALsR enhances acetoin production by Bacillus subtilis[J]. Journal of Industrial Microbiology & Biotechnology, 2013, 40(9): 1067-1076. |
| [15] |
Zhang X, Zhang RZ, Bao T, et al. The rebalanced pathway significantly enhances acetoin production by disruption of acetoin reductase gene and moderate-expression of a new water-forming NADH oxidase in Bacillus subtilis[J]. Metabolic Engineering, 2014, 23: 34-41. DOI:10.1016/j.ymben.2014.02.002 |
| [16] |
Yang TW, Rao ZM, Zhang X, et al. Fermentation of biodiesel-derived glycerol by Bacillus amyloliquefaciens: effects of co-substrates on 2, 3-butanediol production[J]. Applied Microbiology and Biotechnology, 2013, 97(17): 7651-7658. DOI:10.1007/s00253-013-5048-x |
| [17] |
Gardner JG, Escalante-Semerena JC. Biochemical and mutational analyses of AcuA, the acetyltransferase enzyme that controls the activity of the acetyl coenzyme a synthetase (AcsA) in Bacillus subtilis[J]. Journal of Bacteriology, 2008, 190(14): 5132-5136. DOI:10.1128/JB.00340-08 |
| [18] |
Larsson JT, Rogstam A, von Wachenfeldt C. Coordinated patterns of cytochrome bd and lactate dehydrogenase expression in Bacillus subtilis[J]. Microbiology, 2005, 151(10): 3323-3335. DOI:10.1099/mic.0.28124-0 |
 2020, Vol. 47
2020, Vol. 47