扩展功能
文章信息
- 孙成成, 李俊德, 闵军, 胡晓珂
- SUN Cheng-Cheng, LI Jun-De, MIN Jun, HU Xiao-Ke
- 多氯联苯降解菌Burkholderia xenovorans LB400研究进展
- Advances in polychlorinated biphenyls degrading bacteria of Burkholderia xenovorans LB400
- 微生物学通报, 2020, 47(10): 3393-3406
- Microbiology China, 2020, 47(10): 3393-3406
- DOI: 10.13344/j.microbiol.china.200541
-
文章历史
- 收稿日期: 2020-06-02
- 接受日期: 2020-08-21
- 网络首发日期: 2020-09-09
2. 青岛海洋科学与技术试点国家实验室 海洋生物学与生物技术功能实验室 山东 青岛 266237;
3. 中国科学院大学 北京 100049
2. Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao, Shandong 266237, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
进入化学合成时代后,大量人工合成的化合物进入到自然环境中。微生物具有极强的环境适应能力,随着这些人工合成化合物的引入,微生物经过长期的适应可以进化出降解这些污染物的基因和酶系。同时,复杂而又特殊的污染环境也提供了一个局部筛选功能微生物的生境。许多学者相继从被多氯联苯(polychlorinated biphenyls,PCBs)污染的原位环境中发现了具有这类污染物降解能力的微生物。这些微生物的种类、遗传和生态功能的多样性,构成了PCBs生物修复的基础。LB400菌株(16S rRNA基因登录号NR_074325.2)是从纽约州北部PCBs污染的土壤中分离出来,能以联苯为唯一碳源和能源生长的细菌,以好氧降解多氯联苯而著称[1]。最初其被描述为一种假单胞菌,但当时尚未确定这种独特的多氯联苯降解菌的分类学地位。Yabuuchi于1992年创建新Burkholderia属,从而与以前的假单胞菌有所区分,随后菌株LB400被重新鉴定为Burkholderia xenovorans LB400[2]。传统意义上,伯克霍尔德菌属的菌株被称为土壤细菌。然而,Coenye等研究发现伯克霍尔德菌属的成员属于多功能微生物,占据了广泛的生态位,可以在土壤、水(包括海水)、植物根际等环境中生存,这类细菌常被用于生物防治、生物修复和植物生长等方面[3]。
多氯联苯(PCBs)是一类人工合成的有机污染物,该类化合物具有高毒性、环境持久性、生物累积性、半挥发性、可以远距离迁移等特点;在2001年,其成为《斯德哥尔摩公约》首批公布的12类持久性有机污染物之一[4-5]。由于多氯联苯的电绝缘性和不可燃性,许多多氯联苯及其衍生物在工业和商业领域被广泛应用,例如作为变压器油、塑料以及树脂、防火剂的添加剂和增塑剂等[5]。由于自然界中缺乏有效的酶降解系统,多氯联苯及其类似物在环境中能长久滞留。因此,筛选与PCBs相关的降解菌、设计高效酶降解系统是控制和处理多氯联苯污染的重要手段[6]。目前已经从环境中分离出部分多氯联苯降解菌,如Pseudomonas sp. P-6-5[7]等。其中,Burkholderia xenovorans LB400是截至目前发现的最好的好氧降解多氯联苯降解菌之一,引起广泛关注。研究表明,Burkholderia xenovorans LB400的联苯代谢途径也可以引发多氯联苯的降解;同时,其具有降解20多种多氯联苯(如PCB11、PCB12等)以及某些多氯联苯结构类似物的能力,如二苯并呋喃等[8]。考虑到对于Burkholderia xenovorans LB400的研究主要集中于多氯联苯的降解上,因此本文以其对PCBs的降解为核心,结合基因、酶、酶与底物相互作用的氨基酸残基、酶催化过程中酶与底物反应的化学基团4个层面展开论述。通过综述其代谢途径、代谢相关基因和酶系以及修复应用等方面的研究进展,以期为深入探讨Burkholderia xenovorans LB400的应用以及其他多氯联苯降解菌株的代谢途径、相关基因和酶系提供借鉴。
1 Burkholderia xenovorans LB400对多氯联苯的降解途径自Lunt和Ahmed分离出能够在联苯上生长的菌株以来,许多和多氯联苯降解相关的菌株相继被发现;这些菌株当中既有革兰氏阳性菌,也有革兰氏阴性菌,包括Burkholderia、Comamonas、Cupriavidus、Sphingomonas、Acidovorax、Rhodooccus和Bacillus等菌属[9-13]。按照好氧和厌氧条件以及降解机制的不同,其分类如表 1所示。在厌氧条件下,PCBs的代谢主要通过细菌还原脱氯来完成,生成低氯代的PCBs,并不能将其矿化[11, 17]。在好氧条件下,真菌通过木质素分解机制和单加氧反应将多氯联苯氧化和矿化,细菌则通过由双氧化反应起始的联苯代谢途径将其氧化降解[13-14, 16]。Burkholderia xenovorans LB400就是在好氧条件下,通过双氧化反应实现对PCBs的起始降解[14]。值得注意的是,已发现的好氧菌株如P. pseudoalcaligenes KF707,在双氧化反应中联苯双加氧酶大多只表现出了2, 3位的双加氧酶活性,而且只能对低氯代多氯联苯实现有效降解;然而Burkholderia xenovorans LB400联苯双加氧酶不仅具有2, 3位的双加氧酶活性,还能实现3, 4位的双加氧反应;在底物降解范围上,也可以实现对五氯、六氯等多氯代多氯联苯的代谢[12, 14]。但是,Burkholderia xenovorans LB400联苯双加氧酶这种宽松的区域特异性及其对于多氯代多氯联苯代谢有关的结构和氨基酸残基尚不十分明确,有待于进一步的探讨。
| 反应条件 Reaction condition |
代谢机制 Metabolic pathway |
相关代谢酶 Related metabolic enzymes |
菌名称 Bacteria name |
参考文献 References |
| 好氧条件 Aerobic condition |
联苯代谢机制 Metabolic mechanism of biphenyl |
联苯双加氧酶、二氢二醇脱氢酶、二羟基双加氧酶、二烯酸水解酶 Biphenyl dioxygenases, dihydrodiol, dehydrogenase, 2, 3-dihydroxybiphenyl dioxygenase, 2-hydroxy-6-oxo-6-phenyl-hexa- 2, 4-dienoic acid hydrolase |
红球菌属、芽孢杆菌属、假单胞菌属、伯克霍尔德菌属 Rhodooccus, Bacillus, Pseudomonas, Burkholderia |
[12, 14-15] |
| 木质素代谢机制和单加氧反应 Metabolic mechanism of lignin |
木质素过氧化物酶、锰依赖型过氧化物酶、漆酶 Manganese-dependent peroxidase, lignin peroxidase, laccase |
金丝孢杆菌、白腐真菌、丝状真菌 P. chrysosporium, Whiterot fungi, Cunninghamella elegans | [13, 16] | |
| 厌氧条件 Anaerobic condition |
还原脱氯反应 Reductive dechlorination reaction |
还原酶 Reductase | 脱盐杆菌、脱盐球虫菌、脱盐螺菌 Desulfitobacterium, Dehalococcoides, Dehalospirillum | [11, 17] |
同Pandoraea pnomenusa B356、Pseudomonas pseudoalcaligenes KF707等好氧降解菌株一样,菌株LB400将联苯转化为相应的苯甲酸盐需要经历4个酶反应的步骤,苯甲酸盐随后通过其他途径降解[8, 14]。联苯代谢途径的起始反应是联苯双加氧酶的催化反应,该酶在芳香环的2个相邻的碳上引入1个氧分子,产生二羟基的产物,随后代谢产物如顺位的2, 3-二氢-2, 3-二羟基联苯被2, 3-二氢-2, 3-二羟基联苯-2, 3-脱氢酶重新芳香化;邻苯二酚代谢物被2, 3-二羟基联苯-1, 2-双加氧酶裂解生成2-羟基-6-氧-6-苯基-2, 4-二烯酸(2-hydroxy-6-oxo-6-phenyl- hexa-2, 4-dienoic acid,HOPDA),进而在二烯酸水解酶的催化下水解生成苯甲酸[12]。PCBs代谢产物的鉴定以及相关基因的分析已经阐明了由联苯代谢途径起始多氯联苯的转化,通过联苯代谢上游途径可将其降解为氯苯甲酸。以单环氯代联苯为例,其代谢途径如图 1所示。目前,与bph操纵子相关的其他基因在PCBs降解中的明确作用尚未阐明,例如bphK被认为与PCBs脱卤有关[15]。
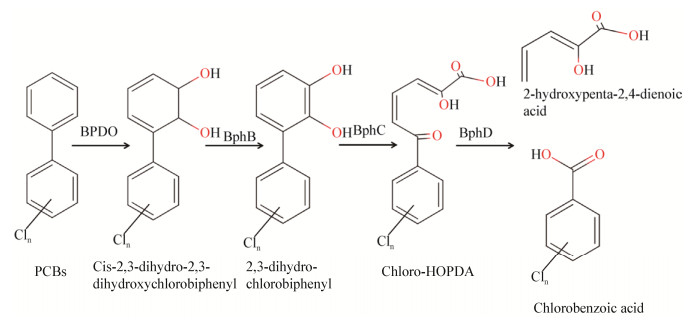
|
| 图 1 多氯联苯的代谢途径 Figure 1 Metabolic pathway of PCBs |
|
|
芳香族化合物的降解是通过脱卤或氧化芳香环起始的,从而使其更易于被环裂解。质粒、转座子和整合遗传元件(通过水平基因转移产生)上大量的芳香族降解基因导致了芳香族降解途径的进化,这使得微生物能够通过典型降解途径代谢多种芳香族化合物。通过使用基因组学和蛋白质组学方法,研究者已报道了一系列多氯联苯降解菌株的概况,如表 2所示。其中,LB400菌株降解PCBs的途径已在遗传和生化水平得到了广泛的鉴定。在分析Burkholderia xenovorans LB400的转录组时也揭示了推定的解毒机制和不同环境条件(例如碳源和生长期)对Burkholderia xenovorans LB400降解效率的影响[28]。
| 菌株 Bacteria name |
分类 Classification |
降解途径 Metabolic pathway |
降解基因 Degradation genes |
降解基因位置 Location of degradation genes |
底物范围 Substrate range |
参考文献 References |
| Burkholderia xenovorans LB400 | 伯克霍尔德属 Burkholderia |
联苯降解途径 Metabolic mechanism of biphenyl |
联苯双加氧酶基因、脱氢酶基因、二羟基联苯双加氧酶基因、二烯酸水解酶基因等 Biphenyl dioxygenase genes (bphAE, bphF, bphG), dihydrodiol dehydrogenase gene (bphB), 2.3-dihydroxybiphenyl dioxygenase gene (bphC), 2-hydroxy-6-phenyl-6-oxohexa-2, 4-dieneoate hydrolase gene(bphD), etc |
位于巨型质粒 Plasmid |
含有1-6个氯取代基的多氯联苯以及二苯并呋喃等多氯联苯结构类似物 Chlorinated biphenyl congeners containing from one to six chlorine substituents and dibenzofuran |
[8, 12, 18-19] |
| Pseudomonas pseudoalcaligenes KF707 | 假单胞菌属 Pseudomonas |
联苯降解途径 Metabolic mechanism of biphenyl |
联苯双加氧酶基因、脱氢酶基因、二羟基苯双加氧酶基因、二烯酸水解酶基因等 Biphenyl dioxygenase gene (bphAl, bphA2, bphA3, bphA4), dihydrodiol dehydrogenase gene (bphB), 2.3- dihydroxybiphenyl dioxygenase gene (bphC), 2-hydroxy-6-phenyl-6-oxohexa-2, 4-dieneoate hydrolase gene (bphD), etc |
位于染色体 Chromosome |
1-3个氯取代基的多氯联苯(2, 4, 5-CB, 2, 3-CB, 2, 4'-CB等)以及几种氟代联苯 Chlorinated biphenyl congeners containing from one to three chlorine substituents and fluorinated biphenyl |
[19-20] |
| Dehalococcoides sp. strain CBDB1 | 脱卤球菌属 Dehalococcoides |
还原脱氯 Reductive dechlorination reaction |
氯苯还原脱卤酶基因、四氯乙烯还原脱卤酶基因等 Chlorobenzene reductive dehalogenase gene (cbrA), tetrachloroethylene reductive dehalogenase gene (pceA), etc |
位于染色体 Chromosome |
绝大多数高氯联苯、高氯代苯酚以及几种氯代二恶英 Chlorinated dioxins, most of PCBs and fluorophenol |
[11, 21-22] |
| Rhodococcus sp. strain RHAl | 红球菌属 Rhodococcus |
联苯降解途径 Metabolic mechanism of biphenyl |
联苯双加氧酶基因、脱氢酶基因、二羟基联苯双加氧酶基因、二烯酸水解酶基因等 Biphenyl dioxygenase gene (bphAl, bphA2, bphA3, bphA4), dihydrodiol dehydrogenase gene (bphB), 2.3-dihydroxybiphenyl dioxygenase gene (bphC), 2-hydroxy-6-phenyl-6-oxohexa-2, 4-dieneoate hydrolase gene (bphD), etc | 位于线性质粒 Plasmid |
高氯代多氯联苯如六氯联苯、七氯联苯等 PCBs, such as hexachlorobiphenyl, heptachlorobiphenyl |
[10, 23-24] |
| Achromobacter Georgiopolitanum KKS102 | 无色杆菌属 Achromobacter |
联苯降解途径 Metabolic mechanism of biphenyl |
联苯双加氧酶基因、脱氢酶基因、二羟基联苯双加氧酶基因、二烯酸水解酶基因等 Biphenyl dioxygenase gene (bphAl, bphA2, bphA3, bphA4), dihydrodiol dehydrogenase gene (bphB), 2.3-dihydroxybiphenyl dioxygenase gene (bphC), 2-hydroxy-6-phenyl-6-oxohexa-2, 4-dieneoate hydrolase gene (bphD), etc |
主要位于质粒 Plasmid |
1-5个氯取代基的多氯联苯 Chlorinated biphenyl congeners containing from one to five chlorine substituents |
[25-26] |
| Phanerocheate chrysosporium RP78 | 平革菌属 Phanerocheate |
木质素代谢机制和单加氧反应 Metabolic mechanism of lignin |
木质素过氧化物酶基因、锰依赖型过氧化物酶基因等 Lignin peroxidase gene (lip1),Manganese-dependent peroxidase gene (mnp5), etc | 位于染色体 Chromosome |
木质素及相关芳香族化合物 Lignin and related aromatic compounds |
[13, 27] |
LB400基因组的大小为9.73 Mb,包括2个环形染色体(分别为4.90 Mb和3.36 Mb)和环形巨大质粒(1.47 Mb)[18]。在众多好氧降解菌株中,Rhodococcus sp. strain RHA1与其基因组大小最为相近。RHA1基因组大小为9.70 Mb,包括1条染色体(7.80 Mb)和3个质粒(分别为1.12 Mb的pRHL1、0.44 Mb的pRHL2和0.33 Mb的pRHL3)[18, 24]。然而,两个菌株中与多氯联苯代谢相关的基因分布有较大差异。在菌株LB400基因组中,与多氯联苯代谢相关的基因位于巨型质粒的bph操纵子上,如图 2所示。操纵子包含编码联苯双加氧酶(biphenyl dioxygenase,BPDO)、顺式-2, 3-二氢-2, 3-二羟基联苯脱氢酶(BphB)、2, 3-二氢联苯-1, 2-双加氧酶(BphC)的基因和二烯酸水解酶(BphD)的基因;其中bphC和bphD之间还有3.5 kb大小的序列区,包含编码谷胱甘肽转移酶(BphK)的基因和其他与PCBs代谢相关的基因[18, 29]。在Rhodococcus sp. RHA1基因组中,主要的bph基因簇位于pRHL1上,由bphA1A2A3A4-bphC-bphB组成,bphD1以及其他基因则位于pRHL2等质粒上[29]。随着越来越多的基因组被测定,可以发现这种基因分布上的差异并非个例,在好氧降解菌株中广泛存在。例如Rhodococcus globerulus P6的bph操纵子的组织结构与菌株LB400十分相似,但该菌株被证明存在多个bphC基因;在红球菌TA421中,7个bphC基因中有3个位于同一个质粒上[30]。从进化角度来看,这种分布上的差异可能与外源性bph基因的插入有关。
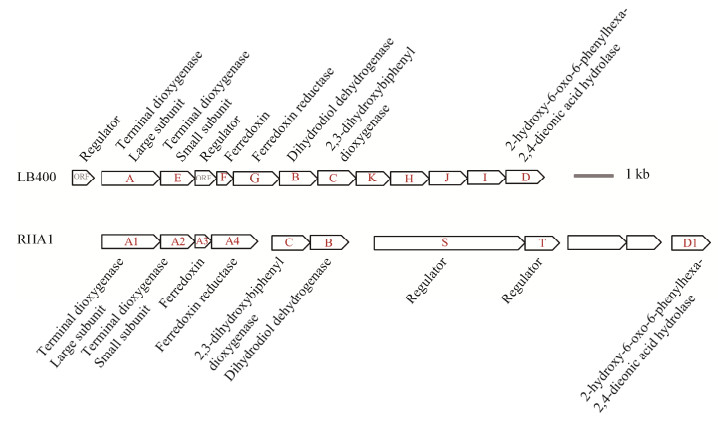
|
| 图 2 菌株LB400和RHA1降解基因分布 Figure 2 Distribution of degradation genes for strain LB400 and RHA1 |
|
|
研究发现,Burkholderia xenovorans LB400与Rhodococcus sp. strain RHA1和P. pseudoalcaligenes KF707等好氧降解菌株的bph基因簇均具有降解多种芳香族化合物的潜力,并且基因组序列显示出异常高的代谢通用性;同时,LB400菌株的基因组近20%的基因被证明是通过水平基因转移获得[18, 24, 29]。其他测序的芳香降解细菌,如恶臭假单胞菌KT2440,其编码联苯降解途径中酶的基因位于移动性遗传元件中,而恶臭假单胞菌KF715含有的bphABCD基因簇被认为是从LB400型基因簇进化而来[31-33]。这些研究表明,Burkholderia xenovorans LB400等好氧降解菌株中bph基因并非独立进化,而是拥有共同的起源。bph基因在质粒或转座子等移动遗传元件上的存在表明这些基因能够在基因组之间移动,从而使微生物群落适应PCBs。总体而言,我们对涉及PCBs代谢的酶和基因的知识仍然不够深入,并且细菌降解途径的多样性被大大低估。
3 Burkholderia xenovorans LB400中的酶系统正确理解多氯联苯降解的生物化学过程是成功构建和优化多氯联苯降解菌的关键。下面将阐明联苯分解代谢途径中每一种酶在PCBs降解中的作用,并对这些酶的生物催化特性和人工进化的现有研究进行总结。
3.1 联苯双加氧酶联苯双加氧酶(BPDO)对于PCBs的代谢至关重要。一方面,它们的双氧化区域专一性决定了随后代谢途径的攻击位点;另一方面,它们的底物特异性决定了生物体可以转化的多氯联苯的范围[34]。菌株LB400的联苯双加氧酶包含3个组分:(1)末端氧化酶(BphAE),其是一个六聚体,如图 3所示,由3个α亚基(Mr=51 000)和3个β亚基(Mr=22 000)组成;(2)铁氧还原蛋白(BphF,Mr=12 000);(3)铁氧还蛋白还原酶(BphG,Mr=43 000),BphAE直接与底物相互作用,实现联苯的二羟基化催化反应[35]。应该注意的是,同类降解菌中的BphAE,即末端氧化酶可分为两类,一类末端双加氧酶为同源多聚体,而另一类则是由大亚基(α)和小亚基(β)组成的异源多聚体。在酶与底物反应过程中,电子由辅酶因子NAD(P)H经还原酶链转移至末端氧化酶[35]。
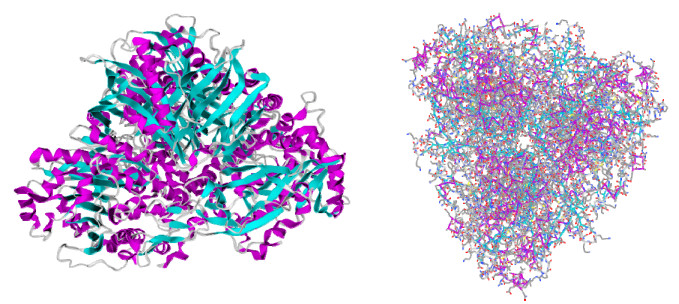
|
| 图 3 联苯双加氧酶模型 Figure 3 The model of biphenyl dioxygenase |
|
|
通过序列分析可以发现,Pseudomonas pseudoalcaligenes KF707与菌株LB400的BPDO基因核苷酸序列几乎完全相同(仅在α亚基上存在20个氨基酸的不同);然而,这两种菌株对PCBs的降解能力存在较大差异,菌株LB400氧化PCBs同系物的范围比KF707氧化范围宽得多,而KF707联苯双加氧酶对几种双对位取代的多氯联苯有更大的活性[12, 34, 36]。这2个菌株在PCBs降解过程中所表现出的差异说明α亚基可能与底物特异性有关。菌株LB400的BphAE的α亚基被认为在反应中起关键作用,共分为4个区域。在LB400联苯双加氧酶与底物结合过程中,Ⅰ、Ⅱ区域中某些氨基酸会发生移动,其移动距离随着底物不同而不同,如283位的丝氨酸;Ⅲ、Ⅳ区域的某些氨基酸被证明具有底物识别的作用,比如Ⅲ区域氨基酸顺序为Thr335Phe336Asn337Asn338Ile339Arg340Ile341的片段;同时BphAELB400的氨基酸Asn377Phe378、Phe384、Gln255Ile258和Ala268也被证明对双加氧酶的催化性能有影响[12, 36-37]。
研究表明,联苯分子的2个苯环在反应时并不是平行的,而是在某些好氧菌株如Rhodococcus jostii RHA1和Pandorae pnomenusa双加氧酶的催化口袋内发生了倾斜[38]。这一结果引起关注,因为已知毒性最强的邻-对位取代同系物是共面构象的。BphAELB400对二苯并呋喃(共面构象分子)的氧化效率不高,然而,将BphAELB400的2个氨基酸残基Thr335Phe336突变为Ala335Met336即得到突变体BphAEp4。相比于BphAELB400,BphAEp4的底物谱范围扩大,能够代谢更多的持久性污染物,如2, 6-二氯联苯;其中,Ala取代Thr335是BphAEp4获得新特性的关键[38-39]。LB400中BphAE的Thr335并不与底物直接接触,但通过极性作用限制催化腔里的Val320Gly321Gln322段的移动;Thr335突变为Ala可以缓解分子段内的约束,允许在底物结合过程中这些残基段的显著移动,从而增加了可用空间,以容纳体积较大的底物,如2, 6-二氯联苯、二苯并呋喃等[38]。因此,联苯双加氧酶的底物范围可以通过突变催化腔内的氨基酸残基调节催化腔的可塑性来改变,从而创造出容纳结构上具有不同构象底物所需的空间。随后,将BphAEp4的Asn338Ile341Leu409转化为Gln338Val341Phe409得到突变体BphAERR41,后者比前者能够更有效地氧化二苯并呋喃[38, 40]。结构和生化分析表明位于邻近亚基Rieske结构域界面催化域的几个蛋白结构参与亚基组装和催化反应(电子转移和质子化)[38]。研究发现BphAERR41对二苯并呋喃的代谢能力优于BphAEp4,这是由Gln取代Asn338及Phe取代Leu409的双重取代所导致;从晶体结构来看,这2个残基离基体很远,但它们一起影响亚基组装和底物诱导构象,从而影响催化反应[38, 41-42]。
综上所述,催化腔内的Ⅲ、Ⅳ区氨基酸残基在固定非反应联苯环的过程中起着至关重要的作用,改变这一区域是增强联苯双加氧酶对共平面的二苯并呋喃及其类似物降解活性的一条有效途径。然而,与以上2个区域相比,人们对Ⅰ、Ⅱ区域氨基酸残基的功能知之甚少。
3.2 2, 3-二氢-2, 3-二羟基联苯脱氢酶联苯分解代谢途径的第二步是由脱氢酶催化生成二羟基代谢物。2, 3-二氢-2, 3-二羟基联苯脱氢酶(BphB)由四聚体组成,如图 4所示。氨基酸序列分析显示Pseudomonas putida OU83、B. xenovorans LB400和P. pseudoalcaligenes KF707的bphB基因表现出高度同源性;作为短链还原酶家族的一员,BphB与其他短链还原酶在催化三联体的折叠和定位方面非常相似,仅在辅因子结合位点上有微小的差异[43]。B. xenovorans LB400中BphB的结构已经被确定,其X射线结构表明,142位的丝氨酸、159位的赖氨酸、去质子化的155位的酪氨酸和辅酶NAD+ (烟酰胺腺嘌呤二核苷酸)形成BphB的活性位点;同时,通过氢键等结合底物多卤代联苯来实现底物和酶构象的改变,从而降低反应的活化能[43]。Zhang等[44]通过量子力学和分子力学的方法解释了BphB催化反应的过程,BphB对底物的催化属于碱催化,由去质子化的155位的酪氨酸引起催化反应,酪氨酸的O原子可以与底物羟基上的H原子形成氢键(键长为1.64 Å),而氢键形成的同时也伴随着底物羟基O-H键的断裂;同时,离开的H原子与充当催化碱的酪氨酸的O原子共价结合,底物上与羟基成键的C原子上的H原子被辅酶NAD+的C原子接受形成σ键,这与实验数据也相吻合。量子力学和分子力学法在酶学方面的应用,提供了研究催化机制的一种思路,将生理生化数据与微观力学分析相结合,对于明确关键氨基酸残基在催化过程中的作用具有重要意义。
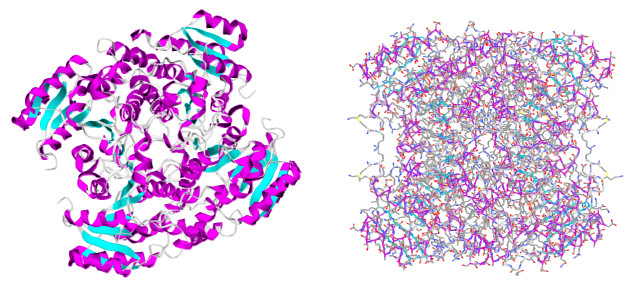
|
| 图 4 2, 3-二氢-2, 3-二羟基联苯脱氢酶模型 Figure 4 The model of BphB |
|
|
值得注意的是,BphB具有广泛的底物特异性,参与各种芳香化合物降解,包括3, 4-二氢-3, 4-二羟基联苯和其他由芳族和杂芳族化合物如二苯并呋喃、类黄酮双氧合制得的二氢二羟基产物;除了2, 6位取代的化合物外,B. xenovorans LB400的BphB基本可将所有在非氧化环带有氯原子的联苯2, 3-二氢二醇进行有效脱氢[43, 45]。因此,2, 3-二氢-2, 3-二羟基联苯脱氢酶并不是降解途径中的瓶颈,人工进化方面的研究较少。
3.3 2, 3-二羟基联苯-1, 2-双加氧酶联苯分解代谢途径的第3个反应是由二羟基双加氧酶(BphC)将2, 3-二羟基联苯转化为2-羟基-6-氧-6-苯基二烯酸(HOPDA),BphC是一种二羟基儿茶酚环裂解双加氧酶,如图 5所示,通过催化2个氧原子加入底物的邻苯二酚环,导致其裂解[12]。外源性双加氧酶的活性位点通常含有1个Fe(Ⅱ),Fe(Ⅱ)和酶之间可能以配位键相互结合[46]。Hatta等从萘降解菌Bacillus sp. JF8中分离纯化出一种耐热的Mn(Ⅱ)依赖型BphCJF8,该酶在80 ℃和75 ℃时的半衰期分别为30 min和81 min;尽管BphCJF8的一级结构与Rhodococcus globerulus P6和Burkholderia sp. LB400的BphC的序列相似性不到25%,但BphCJF8中活性位点残基极具保守性[46]。
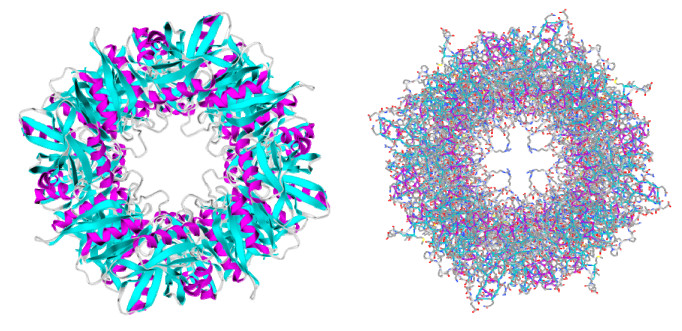
|
| 图 5 2, 3-二羟基联苯-1, 2-双加氧酶模型 Figure 5 The model of BphC |
|
|
从Burkholderia sp. LB400和Pseudomonas sp. KKS102中分离得到的BphC极易被氧化,在厌氧条件下测定其结构,需要进行厌氧纯化;BphC由八聚体组成,每个单体包含2个域;每个域有2个拷贝,由4个β链和1个α螺旋形成,顺序为β-α-β-β-β[47]。因此,每个单体在2个区域内包含该单元的4个拷贝,这表明该酶在进化过程中存在遗传复制。由于BphC对3-氯邻苯二酚的高敏感性,容易由邻氯二羟联苯代谢物引起底物抑制,以及它们不能裂解间对羟基二羟联苯代谢物,使其成为多氯联苯降解途径中的瓶颈[46-47]。随着Denome发现Pseudomonas sp. C18的同源酶1, 2-二羟基萘双加氧酶比BphC更有效地催化间对羟基化代谢产物的环裂解[48],使得其他联芳基化合物降解菌株成为了PCBs降解酶的重要来源。因此,考虑到二羟基双加氧酶的同源性,定向进化BphC或将成为克服由BphC局限性引起的瓶颈的有效手段。
3.4 2-羟基-6-氧-6-苯基-2, 4-二烯酸水解酶联苯代谢通路的最后一步由二烯酸水解酶(BphD)催化,将2-羟基-6-氧-6-苯基-2, 4-二烯酸(HOPDA)水解生成2-羟基-2, 4-二烯酸酯和苯甲酸[49]。来自Burkholderia sp. LB400的HOPDA水解酶是一种同型四聚体酶,如图 6所示,其亚基可以分为核心域和限制域,同所属水解酶家族其他成员相同的是其催化过程需利用Ser-His-Asp三联体[50]。李延伟的研究提供了BphD的底物辅助酰化过程的更多细节,BphD的催化过程属于共价催化,酶活性中心具有一个非极性和一个极性亚位点,分别用于容纳其天然底物HOPDA的苯基和二烯酸部分;酰化过程的产物(2-羟基-2, 4-二烯酸)被3个水分子代替,其中1个参与脱酰过程;同时,他们发现了可能加速BphD降解速率的突变靶标(如Phe239)[50]。
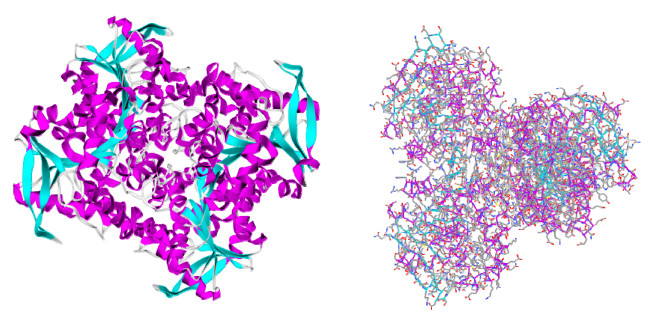
|
| 图 6 2-羟基-6-氧-6-苯基-2, 4-二烯酸水解酶模型 Figure 6 The model of BphD |
|
|
Seah等则比较了B. xenovorans LB400和R. globerulus P6 HOPDA水解酶的催化性能异同,分离自菌株LB400的HOPDA水解酶对苯环上带有氯原子的HOPDA具有较强的降解活性;同时,3-Cl-HOPDA和4-Cl-HOPDA对酶抑制作用较弱[49, 51]。然而,与LB400中的BphD不同,Rhodococcus globerulus P6中的BphD对3-Cl-HOPDA和4-Cl-HOPDA较为敏感[49-51]。这意味着HOPDA水解酶的底物特异性随酶结构的不同变化很大。通过对同源酶的比较研究,可以发现关键氨基酸残基的具体功能,帮助设计新的BphD工程酶,使其能够更有效地降解在2个环上都带有氯原子的同类物质。
4 Burkholderia xenovorans LB400对PCBs的降解应用环境中多氯联苯的迁移转化是由物理、化学和生物过程的相互作用所控制。物理过程是多氯联苯从土壤、地下、水体和大气等离开其来源的过程,而化学和生物过程决定了其转化的程度。同样地,多氯联苯污染的修复也对应着物理修复、化学修复和生物修复。物理和化学修复主要包括高温、高压、强碱性环境、光催化、金属催化、电化学催化等过程。然而在生物修复过程中,原位的土壤细菌是参与PCBs降解的主要贡献者;有学者对B. xenovorans LB400降解多氯联苯的底物谱和降解率做了研究,发现B. xenovorans LB400在1–1.7 mg/L的底物浓度下,能降解15种多氯联苯如表 3所示,而且对低氯代的联苯降解率高于多氯代的联苯[52]。同时,与另一株模式菌株Rhodococcus sp. strain RHA1相比,B. xenovorans LB400对PCBs代谢物的毒性表现出更高的耐受性[53]。鉴于商用PCBs多为三氯到六氯的多氯联苯,B. xenovorans LB400菌株在生物修复方面极具潜力。当前的研究结果表明,利用工程酶和细菌对持久性污染物及其类似物所造成的污染进行修复是可行的。联苯降解菌可以降解间位、对位取代、共平面、双邻位取代的持久性污染物以及通过厌氧降解来代谢高氯代联苯[53-54]。
| PCBs | OD600 | t (h) | Depl (%) | C0(mg/L) |
| 1 | 0.400 | 24.0 | 100±0 | 0.94 |
| 3 | 0.400 | 22.7 | 100±0 | 0.94 |
| 5 | 0.395 | 20.8 | 99±0 | 1.12 |
| 16 | 0.395 | 20.2 | 91±1 | 1.29 |
| 17 | 0.386 | 22.2 | 97±1 | 1.29 |
| 18 | 0.395 | 19.8 | 92±2 | 1.29 |
| 20 | 0.397 | 21.6 | 89±7 | 1.29 |
| 25 | 0.400 | 23.4 | 42±1 | 1.29 |
| 31 | 0.395 | 20.8 | 92±2 | 1.29 |
| 34 | 0.400 | 22.2 | 80±2 | 1.29 |
| 41 | 0.404 | 22.5 | 39±5 | 1.46 |
| 52 | 0.397 | 22.5 | 87±3 | 1.46 |
| 69 | 0.404 | 23.0 | 26±2 | 1.46 |
| 70 | 0.397 | 22.1 | 57±3 | 1.46 |
| 101 | 0.397 | 21.9 | 47±1 | 1.63 |
| 注:PCBs为多氯联苯;C0为多氯联苯起始浓度;t (h)为降解时间;Depl (%)为降解率. Note: PCBs indicates polychlorinated biphenylsl; C0 indicates initial concentration of polychlorinated biphenyls; t (h) indicates degradation time; Depl (%) indicates degradation rate. |
||||
另外,植物对多氯联苯污染的修复做出了积极的贡献。植物本身可以积累和代谢PCBs,但其代谢速率较慢并产生有毒代谢物[55-56]。然而,植物可以与根际细菌联合作用有效降解PCBs。有学者提出可以利用转基因植物表达降解PCBs的基因,诱导PCBs降解并将部分PCBs代谢物进行解毒代谢[57-58]。Mohammadi等的研究表明,在烟草植株短暂表达B. xenovorans LB400编码BPDO的基因实验中,每个组分(BphAE、BphF、BphG)都可以作为活性蛋白单独在植物中产生,这意味着利用转基因植物降解PCBs似乎是可行的[59-60]。未来或将通过转基因植物和根际细菌开发出一种高效绿色的修复多氯联苯污染的方法。
5 结论与展望作为研究多氯联苯降解的模式菌株,B. xenovorans LB400降解PCBs的途径已在遗传和生化水平得到了广泛的鉴定。参与起始代谢的联苯双加氧酶通常被认为是该途径的关键酶,其决定了底物范围和PCBs降解程度。然而由bphC编码的二羟基双加氧酶和bphD编码的水解酶被认为是PCBs降解中的瓶颈,需要不断优化。设计修饰酶以增强其对底物和底物类似物的活性,需要了解酶催化腔的氨基酸与底物相互作用中用以结合和定位的机制。通过比较不同的蛋白质进化策略,关于这些酶的作用机理已经取得了重大进展,其中还包括对谷胱甘肽转移酶(BphK)功能的阐述。
然而,总体来看,对于B. xenovorans LB400等多氯联苯降解菌株的研究仍存在很多值得探讨的地方,具体包括以下几个方面:(1)从基因层面来看,与PCBs相关降解基因的功能较为明确,但在调控基因方面仍有空白,如在菌株LB400的bph操纵子上bphB和bphC间的序列ORF调控作用仍不明确,有待分子生物学层面进一步的探究。(2)在降解酶方面,设计修饰酶以增强其对底物的活性,需要对酶的结构以及关键氨基酸残基的作用有详细的了解。但是,在催化腔中排列的氨基酸残基或那些影响底物定位以及影响催化中心构象的氨基酸残基的确切作用有的仍不清楚,如在菌株LB400联苯双加氧酶催化底物反应时,BphAE催化腔283位的丝氨酸发生明显的移动,但目前对于其作用知之甚少。因此,需要对能够与底物反应的野生型和进化型酶的晶体结构结合分子力学进行分析,以帮助探究这些酶扩大其底物特异性的机制。(3)在降解途径方面,B. xenovorans LB400等多氯联苯降解菌株对很多多氯联苯及其结构类似物的代谢过程都存在中心代谢途径。然而,转化转座等产生的新基因和基因转移、重组过程导致的遗传进化往往导致围绕中心代谢途径的旁支代谢途径的变化,这种旁支途径的变化仍需探究。(4)微生物并不是生物降解过程的唯一驱动因素,植物对PCBs污染的修复也有重要贡献。植物和微生物联合应用的根际修复成为近年来研究的热点。未来设计优化B. xenovorans LB400工程菌以及与植物联合作用或将成为控制多氯联苯污染的重要手段。
| [1] |
O'Sullivan LA, Mahenthiralingam E. Biotechnological potential within the genus Burkholderia[J]. Letters in Applied Microbiology, 2005, 41(1): 8-11. |
| [2] |
Achouak W, Christen R, Barakat M, et al. Burkholderia caribensis sp. nov., an exopolysaccharide-producing bacterium isolated from vertisol microaggregates in Martinique[J]. International Journal of Systematic Bacteriology, 1999, 49(2): 787-794. DOI:10.1099/00207713-49-2-787 |
| [3] |
Coenye T, Vandamme P. Diversity and significance of Burkholderia species occupying diverse ecological niches[J]. Environmental Microbiology, 2003, 5(9): 719-729. DOI:10.1046/j.1462-2920.2003.00471.x |
| [4] |
Li FH, Xu L, Zhang TH, et al. Degradation characteristics and fermentation conditions optimization of a PCBs-degrading strain[J]. Microbiology China, 2014, 41(7): 1299-1307. (in Chinese) 李方卉, 徐莉, 张腾昊, 等. 一株PCBs降解菌的降解特性及发酵条件优化[J]. 微生物学通报, 2014, 41(7): 1299-1307. |
| [5] |
Lu YT, Liu ML, Liu YZ, et al. Characteristics and health risk assessment of polychlorinated biphenyls in surface soil of the Yangtze River[J]. China Environmental Science, 2018, 38(12): 4617-4624. (in Chinese) 鲁垠涛, 刘明丽, 刘殷佐, 等. 长江表层土壤多氯联苯污染特征及风险评价[J]. 中国环境科学, 2018, 38(12): 4617-4624. |
| [6] |
L'Abbée JB, Tu YB, Barriault D, et al. Insight into the metabolism of 1, 1, 1-trichloro-2, 2-bis(4-chlorophenyl)ethane (DDT) by biphenyl dioxygenases[J]. Archives of Biochemistry and Biophysics, 2011, 516(1): 35-44. DOI:10.1016/j.abb.2011.09.016 |
| [7] |
Sun GT, Chen HY, Zhao LC, et al. Isolation, identification and characterization of a PCBs-degrading bacterium[J]. Biotechnology Bulletin, 2018, 34(6): 141-148. (in Chinese) 孙桂婷, 陈红云, 赵玲超, 等. 多氯联苯降解菌的筛选、鉴定及其降解特性研究[J]. 生物技术通报, 2018, 34(6): 141-148. |
| [8] |
Pham TTM, Sondossi M, Sylvestre M, et al. Metabolism of doubly para-substituted hydroxychlorobiphenyls by bacterial biphenyl dioxygenases[J]. Applied and Environmental Microbiology, 2015, 81(14): 4860-4872. DOI:10.1128/AEM.00786-15 |
| [9] |
Qiao C, Ren L, Fan SH, et al. Characterization of 2, 3-dihydroxybiphenyl-1, 2-dioxygenase BphC from biphenyl degrader Arthrobacter sp. YC-RL1[J]. Microbiology China, 2017, 44(7): 1639-1648. (in Chinese) 乔铖, 任磊, 樊双虎, 等. 联苯利用节杆菌YC-RL1中2, 3-二羟基联苯-1, 2-双加氧酶BphC功能验证[J]. 微生物学通报, 2017, 44(7): 1639-1648. |
| [10] |
Yang XQ, Yang L. Physiological function of regulatory protein RHOGL007659 involved in metabolism biphenyl/polychlorinated biphenyl in Rhodococcus sp. R04[J]. Acta Microbiologica Sinica, 2018, 58(5): 915-925. (in Chinese) 杨秀清, 杨琳. 红球菌R04联苯/多氯联苯代谢相关调控蛋白RHOGL007659的生理功能[J]. 微生物学报, 2018, 58(5): 915-925. |
| [11] |
Smidt H, de Vos WM. Anaerobic microbial dehalogenation[J]. Annual Review of Microbiology, 2004, 58(1): 43-73. DOI:10.1146/annurev.micro.58.030603.123600 |
| [12] |
Barriault D, Sylvestre M. Evolution of the biphenyl dioxygenase BphA from Burkholderia xenovorans LB400 by random mutagenesis of multiple sites in region Ⅲ[J]. Journal of Biological Chemistry, 2004, 279(46): 47480-47488. DOI:10.1074/jbc.M406805200 |
| [13] |
Stella T, Covino S, Čvančarová M, et al. Bioremediation of long-term PCB-contaminated soil by white-rot fungi[J]. Journal of Hazardous Materials, 2017, 324: 701-710. DOI:10.1016/j.jhazmat.2016.11.044 |
| [14] |
Kumar P, Gómez-Gil L, Mohammadi M, et al. Anaerobic crystallization and initial X-ray diffraction data of biphenyl 2, 3-dioxygenase from Burkholderia xenovorans LB400: addition of agarose improved the quality of the crystals[J]. Acta Crystallographica Section F, 2011, 67(1): 59-63. |
| [15] |
Tocheva EI, Fortin PD, Eltis LD, et al. Structures of ternary complexes of BphK, a bacterial glutathione S-transferase that reductively dechlorinates polychlorinated biphenyl metabolites[J]. Journal of Biological Chemistry, 2006, 281(41): 30933-30940. DOI:10.1074/jbc.M603125200 |
| [16] |
Périgon S, Massier M, Germain J, et al. Metabolic adaptation of fungal strains in response to contamination by polychlorinated biphenyls[J]. Environmental Science and Pollution Research, 2019, 26(15): 14943-14950. DOI:10.1007/s11356-019-04701-5 |
| [17] |
Xu Y, Fu DF. A review on microbial-catalyzed reductive dechlorination of polychlorinated biphenyls[J]. Environmental Chemistry, 2014, 33(6): 908-914. (in Chinese) 许妍, 傅大放. 多氯联苯微生物厌氧脱氯研究进展[J]. 环境化学, 2014, 33(6): 908-914. |
| [18] |
Chain PSG, Denef VJ, Konstantinidis KT, et al. Burkholderia xenovorans LB400 harbors a multi-replicon, 9.73-Mbp genome shaped for versatility[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(42): 15280-15287. DOI:10.1073/pnas.0606924103 |
| [19] |
Gibson DT, Cruden DL, Haddock JD, et al. Oxidation of polychlorinated biphenyls by Pseudomonas sp. strain LB400 and Pseudomonas pseudoalcaligenes KF707[J]. Journal of Bacteriology, 1993, 175(14): 4561-4564. DOI:10.1128/JB.175.14.4561-4564.1993 |
| [20] |
Murphy CD, Quirke S, Balogun O. Degradation of fluorobiphenyl by Pseudomonas pseudoalcaligenes KF707[J]. FEMS Microbiology Letters, 2008, 286(1): 45-49. DOI:10.1111/j.1574-6968.2008.01243.x |
| [21] |
Adrian L, Dudková V, Demnerová K, et al. "Dehalococcoides" sp. strain CBDB1 extensively dechlorinates the commercial polychlorinated biphenyl mixture Aroclor 1260[J]. Applied and Environmental Microbiology, 2009, 75(13): 4516-4524. DOI:10.1128/AEM.00102-09 |
| [22] |
Kube M, Beck A, Zinder SH, et al. Genome sequence of the chlorinated compound-respiring bacterium Dehalococcoides species strain CBDB1[J]. Nature Biotechnology, 2005, 23(10): 1269-1273. DOI:10.1038/nbt1131 |
| [23] |
Seto M, Kimbara K, Shimura M, et al. A novel transformation of polychlorinated biphenyls by Rhodococcus sp. strain RHA1[J]. Applied and Environmental Microbiology, 1995, 61(9): 3353-3358. DOI:10.1128/AEM.61.9.3353-3358.1995 |
| [24] |
Masai E, Sugiyama K, Iwashita N, et al. The bphDEF meta-cleavage pathway genes involved in biphenyl/polychlorinated biphenyl degradation are located on a linear plasmid and separated from the initial bphACB genes in Rhodococcus sp. strain RHA1[J]. Gene, 1997, 187(1): 141-149. DOI:10.1016/S0378-1119(96)00748-2 |
| [25] |
Fukuda M, Yasukochi Y, Kikuchi Y, et al. Identification of the bphA and bphB genes of Pseudomonas sp. strain KKS102 involved in degradation of biphenyl and polychlorinated biphenyls[J]. Biochemical and Biophysical Research Communications, 1994, 202(2): 850-856. DOI:10.1006/bbrc.1994.2008 |
| [26] |
Kimbara K, Hashimoto T, Fukuda M, et al. Isolation and characterization of a mixed culture that degrades polychlorinated biphenyls[J]. Agricultural and Biological Chemistry, 1988, 52(11): 2885-2891. |
| [27] |
Martinez D, Larrondo LF, Putnam N, et al. Genome sequence of the lignocellulose degrading fungus Phanerochaete chrysosporium strain RP78[J]. Nature Biotechnology, 2004, 22(6): 695-700. DOI:10.1038/nbt967 |
| [28] |
Parnell JJ, Denef VJ, Park J, et al. Environmentally relevant parameters affecting PCB degradation: carbon source- and growth phase-mitigated effects of the expression of the biphenyl pathway and associated genes in Burkholderia xenovorans LB400[J]. Biodegradation, 2010, 21(1): 147-156. DOI:10.1007/s10532-009-9289-4 |
| [29] |
McLeod MP, Warren RL, Hsiao WWL, et al. The complete genome of Rhodococcus sp. RHA1 provides insights into a catabolic powerhouse[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(42): 15582-15587. DOI:10.1073/pnas.0607048103 |
| [30] |
Kosono S, Maeda M, Fuji F, et al. Three of the seven bphC genes of Rhodococcus erythropolis TA421, isolated from a termite ecosystem, are located on an indigenous plasmid associated with biphenyl degradation[J]. Applied and Environmental Microbiology, 1997, 63(8): 3283-3285. |
| [31] |
Hayase N, Taira K, Furukawa K. Pseudomonas putida KF715 bphABCD operon encoding biphenyl and polychlorinated biphenyl degradation: cloning, analysis, and expression in soil bacteria[J]. Journal of Bacteriology, 1990, 172(2): 1160-1164. DOI:10.1128/JB.172.2.1160-1164.1990 |
| [32] |
Ohtsubo Y, Delawary M, Kimbara K, et al. BphS, a key transcriptional regulator of bph genes involved in polychlorinated biphenyl/biphenyl degradation in Pseudomonas sp. KKS102[J]. Journal of Biological Chemistry, 2001, 39: 36146-36154. |
| [33] |
Jimenéz JI, Miñambres B, García JL, et al. Genomic analysis of the aromatic catabolic pathways from Pseudomonas putida KT2440[J]. Environmental Microbiology, 2002, 4(12): 824-841. DOI:10.1046/j.1462-2920.2002.00370.x |
| [34] |
Vézina J, Barriault D, Sylvestre M. Diversity of the C-terminal portion of the biphenyl dioxygenase large subunit[J]. Journal of Molecular Microbiology and Biotechnology, 2008, 15(2/3): 139-151. |
| [35] |
Dhindwal S, Gomez-Gil L, Neau DB, et al. Structural basis of the enhanced pollutant-degrading capabilities of an engineered biphenyl dioxygenase[J]. Journal of Bacteriology, 2016, 198(10): 1499-1512. DOI:10.1128/JB.00952-15 |
| [36] |
Arnett CM, Parales JV, Haddock JD. Influence of chlorine substituents on rates of oxidation of chlorinated biphenyls by the biphenyl dioxygenase of Burkholderia sp. strain LB400[J]. Applied and Environmental Microbiology, 2000, 66(7): 2928-2933. DOI:10.1128/AEM.66.7.2928-2933.2000 |
| [37] |
Barriault D, Simard C, Chatel H, et al. Characterization of hybrid biphenyl dioxygenases obtained by recombining Burkholderia sp. strain LB400 bphA with the homologous gene of Comamonas testosteroni B-356[J]. Canadian Journal of Microbiology, 2001, 47(11): 1025-1032. DOI:10.1139/w01-108 |
| [38] |
Mohammadi M, Viger JF, Kumar P, et al. Retuning rieske-type oxygenases to expand substrate range[J]. Journal of Biological Chemistry, 2011, 286(31): 27612-27621. DOI:10.1074/jbc.M111.255174 |
| [39] |
Pham TTM, Tu YB, Sylvestre M. Remarkable ability of Pandoraea pnomenusa B356 biphenyl dioxygenase to metabolize simple flavonoids[J]. Applied and Environmental Microbiology, 2012, 78(10): 3560-3570. DOI:10.1128/AEM.00225-12 |
| [40] |
Mohammadi M, Sylvestre M. Resolving the profile of metabolites generated during oxidation of dibenzofuran and chlorodibenzofurans by the biphenyl catabolic pathway enzymes[J]. Chemistry & Biology, 2005, 12(7): 835-846. |
| [41] |
Mondello FJ, Turcich MP, Lobos JH, et al. Identification and modification of biphenyl dioxygenase sequences that determine the specificity of polychlorinated biphenyl degradation[J]. Applied and Environmental Microbiology, 1997, 63(8): 3096-3103. DOI:10.1128/AEM.63.8.3096-3103.1997 |
| [42] |
Kumar P, Mohammadi M, Viger JF, et al. Structural insight into the expanded PCB-degrading abilities of a biphenyl dioxygenase obtained by directed evolution[J]. Journal of Molecular Biology, 2011, 405(2): 531-547. DOI:10.1016/j.jmb.2010.11.009 |
| [43] |
Hülsmeyer M, Hecht HJ, Niefind K, et al. Crystal structure of cis-biphenyl-2, 3-dihydrodiol-2, 3-dehydrogenase from a PCB degrader at 2.0 Å resolution[J]. Protein Science, 1998, 7(6): 1286-1293. DOI:10.1002/pro.5560070603 |
| [44] |
Zhang RM, Zhuang T, Zhang QZ, et al. Effect of F, Cl, Br and I substitution on the BphB enzyme for the degradation of halogenated biphenyls, revealed by quantum and molecular mechanics[J]. Environmental Chemistry Letters, 2019, 17(2): 1167-1173. DOI:10.1007/s10311-019-00873-1 |
| [45] |
Jouanneau Y, Meyer C. Purification and characterization of an arene cis-dihydrodiol dehydrogenase endowed with broad substrate specificity toward polycyclic aromatic hydrocarbon dihydrodiols[J]. Applied and Environmental Microbiology, 2006, 72(7): 4726-4734. DOI:10.1128/AEM.00395-06 |
| [46] |
Hatta T, Mukerjee-Dhar G, Damborsky J, et al. Characterization of a novel thermostable Mn(Ⅱ)-dependent 2, 3-dihydroxybiphenyl 1, 2-dioxygenase from a polychlorinated biphenyl- and naphthalene-degrading Bacillus sp. JF8[J]. Journal of Biological Chemistry, 2003, 278(24): 21483-21492. DOI:10.1074/jbc.M210240200 |
| [47] |
Fortin PD, Lo ATF, Haro MA, et al. Evolutionarily divergent extradiol dioxygenases possess higher specificities for polychlorinated biphenyl metabolites[J]. Journal of Bacteriology, 2005, 187(2): 415-421. DOI:10.1128/JB.187.2.415-421.2005 |
| [48] |
Denome SA, Stanley DC, Olson ES, et al. Metabolism of dibenzothiophene and naphthalene in Pseudomonas strains: complete DNA sequence of an upper naphthalene catabolic pathway[J]. Journal of Bacteriology, 1993, 175(21): 6890-6901. DOI:10.1128/JB.175.21.6890-6901.1993 |
| [49] |
Seah SYK, Labbé G, Nerdinger S, et al. Identification of a serine hydrolase as a key determinant in the microbial degradation of polychlorinated biphenyls[J]. Journal of Biological Chemistry, 2000, 275(21): 15701-15708. DOI:10.1074/jbc.275.21.15701 |
| [50] |
Li YW. Theoretical study on the catalytic mechanisms of typical environment related enzyme systems[D]. Ji'nan: Doctoral Dissertation of Shandong University, 2014 (in Chinese) 李延伟.环境相关典型生物酶体系的催化反应机理研究[D].济南: 山东大学博士学位论文, 2014 |
| [51] |
Seah SYK, Labbé G, Kaschabek SR, et al. Comparative specificities of two evolutionarily divergent hydrolases involved in microbial degradation of polychlorinated biphenyls[J]. Journal of Bacteriology, 2001, 183(5): 1511-1516. DOI:10.1128/JB.183.5.1511-1516.2001 |
| [52] |
Rein A, Fernqvist MM, Mayer P, et al. Degradation of PCB congeners by bacterial strains[J]. Applied Microbiology and Biotechnology, 2007, 77(2): 469-481. DOI:10.1007/s00253-007-1175-6 |
| [53] |
Parnell JJ, Park J, Denef V, et al. Coping with polychlorinated biphenyl (PCB) toxicity: physiological and genome-wide responses of Burkholderia xenovorans LB400 to PCB-mediated stress[J]. Applied and Environmental Microbiology, 2006, 72(10): 6607-6614. DOI:10.1128/AEM.01129-06 |
| [54] |
Focht DD. Strategies for the improvement of aerobic metabolism of polychlorinated biphenyls[J]. Current Opinion in Biotechnology, 1995, 6(3): 341-346. DOI:10.1016/0958-1669(95)80057-3 |
| [55] |
Lovecká P, Macková M, Demnerová K, et al. Metabolic Products of PCBs in Bacteria and Plants-Comparison of Their Toxicity and Genotoxicity[M]. London, UK: Taylor and Francis Group, 2004: 134-145.
|
| [56] |
Passatore L, Rossetti S, Juwarkar AA, et al. Phytoremediation and bioremediation of polychlorinated biphenyls (PCBs): State of knowledge and research perspectives[J]. Journal of Hazardous Materials, 2014, 278: 189-202. DOI:10.1016/j.jhazmat.2014.05.051 |
| [57] |
Macek T, Kotrba P, Svatos A, et al. Novel roles for genetically modified plants in environmental protection[J]. Trends in Biotechnology, 2008, 26(3): 146-152. DOI:10.1016/j.tibtech.2007.11.009 |
| [58] |
Campanella BF, Bock C, Schröder P. Phytoremediation to increase the degradation of PCBs and PCDD/Fs[J]. Environmental Science and Pollution Research, 2002, 9(1): 73-85. DOI:10.1007/BF02987318 |
| [59] |
Mohammadi M, Chalavi V, Novakova-Sura M, et al. Expression of bacterial biphenyl-chlorobiphenyl dioxygenase genes in tobacco plants[J]. Biotechnology and Bioengineering, 2007, 97(3): 496-505. DOI:10.1002/bit.21188 |
| [60] |
Xu L, Teng Y, Li ZG, et al. Enhanced removal of polychlorinated biphenyls from alfalfa rhizosphere soil in a field study: The impact of a rhizobial inoculum[J]. Science of the Total Environment, 2010, 408(5): 1007-1013. DOI:10.1016/j.scitotenv.2009.11.031 |
 2020, Vol. 47
2020, Vol. 47




