扩展功能
文章信息
- 梁丛颖, 林璐
- LIANG Cong-Ying, LIN L
- 环境微生物介导的木质素代谢及其资源化利用研究进展
- Environmental microorganisms driven lignin biodegradation and their roles in lignin utilization
- 微生物学通报, 2020, 47(10): 3380-3392
- Microbiology China, 2020, 47(10): 3380-3392
- DOI: 10.13344/j.microbiol.china.200796
-
文章历史
- 收稿日期: 2020-07-31
- 接受日期: 2020-09-25
- 网络首发日期: 2020-09-30
木质素是自然界中含量丰富的有机碳库,是羟基苯丙烷单体通过脱氢缩合由各种醚键和碳碳键连接而成的一种高度交联的芳香族杂聚物,结构的复杂性和无规则性导致其难以降解[1]。自然界中多种微生物已进化出降解木质素的能力,从而驱动木质素分解代谢,完成全球碳循环。此外,考虑到木质素是地球上含量丰富的可再生的生物资源,微生物驱动木质素生物转化将是生物资源化利用的一条有效途径。
在自然界中,木质素降解是由真菌、细菌等生物因素以及环境等非生物因素(如光降解、热降解)共同作用的结果。其中,由于真菌降解木质素的能力较强,对其研究开展较早,研究得也更为广泛和深入。但真菌生长条件较为苛刻,极大限制了其在工业上的应用,如高pH值、缺氧、较高底物浓度等工业发酵较为常见的条件极大地抑制了真菌生长。此外,较复杂的遗传体系限制了其代谢工程研究[2]。近年来陆续发现来自不同生态环境的细菌均具有木质素代谢能力。与真菌相比,细菌生长所处的pH值、温度、氧气范围更加广泛,并易于遗传操作。因此,细菌是研究木质素转化和高值化利用的突破口。在木质素生物降解过程中,胞外木质素解聚酶催化是第一步也是最为关键的一步。本文将重点综述胞外木质素解聚酶系组成及其分泌机制,介绍木质素生物转化的最新研究进展,并探讨环境微生物降解木质素的应用前景和未来研究发展方向。
1 环境中参与分解代谢木质素的微生物 1.1 真菌真菌是迄今为止发现的参与木质素代谢的最活跃的微生物,其优势在于菌丝组织赋予其较强的渗透能力,并通过分泌胞外酶攻击植物细胞壁中的木质素,从而实现木质素的解聚。根据真菌对木质素的降解类型可以分为白腐菌、褐腐菌和软腐菌[3]。白腐菌和褐腐菌属于担子菌纲(Basidiomycetes),而软腐菌多属于子囊菌纲(Ascomycetes)[4]。白腐菌被认为是最主要的木质素降解微生物,其降解活动主要发生在次生代谢阶段,这主要是因为参与木质素生物降解过程的酶(即木质素降解酶系)通常在营养条件贫瘠时(如缺无机碳、氮、硫等)被诱导表达。此外,由于酶系较强的氧化活性以及较低的底物特异性,白腐真菌能将木质素完全分解代谢,生成二氧化碳[5]。其中Phanerochaete chrysosporium是研究较为深入的白腐菌模式菌株,2004年该菌的全基因组测序已完成,极大地推动了白腐真菌降解木质素的研究[6]。
1.2 细菌随着培养技术和高通量测序技术的发展,来自海洋、土壤、腐烂的木材、活性污泥、堆肥和动物肠道等不同生境的细菌均被发现能有效降解木质素。这些木质素降解菌主要集中在放线菌门(Actinobacteria)、变形菌门(Proteobacteria)和厚壁菌门(Firmicutes)等[7-8]。
放线菌是木质素降解能力较强的细菌,其中Streptomyces viridosporus T7A是目前研究较深入的菌株,该菌株在菌丝形态上与真菌相似,能产生胞外木质素降解酶将木质素降解为水溶性的聚合物[9]。此外,Rhodococcus sp. RHA也是一株研究较为深入、降解能力与S. viridosporus T7A相当的放线菌[7]。变形菌门是细菌中最大的一门,在木质素降解方面的研究也受到广泛关注。其中恶臭假单胞菌(Pseudomonas putida)具有木质素分解代谢能力,由于有较强的碳储存能力和环境适应能力以及较好的遗传操作系统,已成为变形菌门研究木质素代谢的代表菌株[10-11]。例如,我们团队发现恶臭假单胞菌(Pseudomonas putida)木质素分解代谢的独特特性,并建立木质素至聚羟基脂肪酸酯(polyhydroxyalkanoates,PHA)生物转化路线[12]。除此之外,以芽孢杆菌属(Bacillus)为代表的厚壁菌门的菌株也被报道出具有木质素降解能力,如B. puilus、B. atphaeus[13]、B. ligniniphilus[14]等。
2 微生物降解木质素的酶系组成及其分泌机制木质素生物降解第一步是通过胞外木质素解聚酶对木质素进行解聚反应。微生物通过过氧化物酶如木质素过氧化物酶(LiP)、锰过氧化物酶(MnP)、多功能过氧化物酶(VP)、染料脱色过氧化物酶(DyP)等[15]和漆酶以及一些辅助酶(图 1)[16-18],引发木质素内部的Cα-Cβ裂解、烷基-芳基裂解、去甲基化及环裂解等,将木质素解聚为小分子的芳烃化合物。

|
| 图 1 木质素解聚酶催化循环 Figure 1 Catalytic cycle of lignin degrading enzymes 注:QR:醌氧化还原酶;GLOX:乙二醛氧化酶;AAO:芳基醇氧化酶;PROD:吡喃糖氧化酶;HBT:1-羟基苯丙三唑. Note: QR: Quinone reductase; GLOX: Glyoxal oxidase; AAO: Aryl alcohol oxidase; PROD: Pyranose oxidase; HBT: 1-hydroxybenzotriazole. |
|
|
漆酶是一种多铜氧化酶,以分子氧作为电子受体,自生自由基,创造可持续的氧化还原环境,攻击木质素酚类化合物Cα-Cβ键,实现木质素解聚改性[19]。同时,漆酶可以通过介体系统来解除空间位阻和低氧化还原电势的局限性,从而有效降解大分子木质素和非酚型芳香族化合物。真菌漆酶的底物范围极其广泛,酚类及其衍生物、芳胺及其衍生物、芳香羧酸及其衍生物等都可被其催化氧化。除漆酶外,过氧化物酶也在真菌木质素降解中起着重要作用[3]。LiP以H2O2为电子受体直接从芳香族底物上捕获电子,将其氧化形成自由基(如苯氧自由基和VA+),经Cα-Cβ键断裂形成醌,参与到醌氧化循环过程中(图 1);真菌LiP催化氧化木质素需要黎芦醇(VA,真菌代谢产物)的参与,VA既是LiP的底物,又是其氧化还原介体,同时还可以保护LiP免受过量的H2O2破坏[20]。然而MnP以H2O2为电子受体氧化Mn2+生成Mn3+,但Mn3+是强氧化剂,在水相中极其不稳定,白腐真菌可以通过分泌草酸等有机酸形成Mn3+螯合物,作为可扩散的小分子氧化还原介质进攻酚型木质素结构(图 1)[21]。VP则结合了LiP和MnP的生物学催化特性,既能有效地氧化MnP特异性底物Mn2+,又能氧化降解一系列酚型和非酚型木质素底物[22]。DyP虽然在结构上不同于其他过氧化物酶,但其氧化机理与LiP和MnP类似[23],需要H2O2启动反应,通过自由基介导的氧化过程进行木质素的裂解。根据氨基酸序列特征将DyP分为A、B、C、D四类,其中D类DyP是真菌胞外酶的代表,表现出较高的过氧化物酶活性[24]。真菌来源的DyP可以氧化高还原电位的蒽醌类特异性底物,但不能以常用的过氧化物酶底物藜芦基醇和愈创木酚为电子供体[25]。
除上述几种重要酶外,真菌木质素降解过程中还需要葡萄糖氧化酶、芳基醇氧化酶和乙二醛氧化酶等辅助酶的参与[26]。这类氧化酶在分子氧的参与下氧化相应底物提供H2O2来激活过氧化物酶(LiP、VP、DyP、MnP),以启动木质素氧化反应,形成高度活性的自由基中间体,继而产生许多不同自由基,促使各种底物如非酚类芳香族、酚类物质的氧化[27-28]。
2.2 细菌木质素解聚酶 2.2.1 木质素解聚酶漆酶基因也广泛存在于细菌中,尤其是链霉菌、芽孢杆菌和假单胞菌[29]。与真菌漆酶相似,细菌漆酶也含有典型的4个铜结合位点,在分子氧存在时,木质素底物失去一个电子生成苯氧自由基,引起木质素酚型结构单元侧链断裂,包括Cα-Cβ键断裂以及Cα氧化(图 1)。多数细菌漆酶属于低氧化还原电势漆酶,通常只能作用于氧化还原电位更低的底物(如酚类木质素化合物),底物范围相对较窄[30]。但在构象上,细菌漆酶的底物结合腔较大,较真菌漆酶更能催化大分子木质素底物的氧化[31]。除此之外,细菌漆酶在热稳定性、耐受碱性条件等方面更有优势,而且结构较为简单,没有糖基化的修饰,这使得细菌漆酶可利用原核表达系统(如E. coli)实现高水平的表达[32]。
DyP在细菌中也广泛存在,细菌来源的DyP主要为A-C类,A类DyP蛋白酶通常含有典型的信号序列(Tat),分泌到胞外发挥作用[33],而B类、C类中的DyP不含有典型的分泌信号肽,被简单归类为胞内蛋白[2]。B类DyP通过攻击酚类木质素化合物Cα-Cβ键进行氧化还原反应,在木质素降解中发挥重要作用。Ahmad等[34]发现缺失DypB的R. jostii RHA1突变体对木质素的降解活性显著降低。Rahmanpour等[35]在Pseudomonas fluorescence Pf-5菌株中发现对Mn2+和硫酸盐木质素均有氧化活性的Dyp1B。Lin等[36]也从假单胞菌A514中鉴定出2个不含典型信号肽的具有独特木质素氧化活性的DypB酶。C类DyPs与A类和B类DyPs在亲缘关系上相差甚远,与D类较为接近[23]。Amycolatopsis sp. 75iv2的DyP2是C类DyPs的典型代表[23],参与木质素降解过程,其氧化模式同样依赖于Mn2+,而且底物范围更为广泛,对苯酚、偶氮染料和蒽醌染料均表现出过氧化物酶活性。
相比于漆酶和DyP,人们对细菌MnP和LiP的认知较少,虽然近年来已取得一定进展,但目前仅克隆出一种细菌LiP,并没有细菌MnP蛋白的结构信息[33],可能与产MnP和LiP酶菌株发现相对较少,酶活与真菌来源相差较多有关。此外,Lin等[12]从近海海域分离鉴定出不同表型的木质素降解菌株样品,在单一碳源(可溶性、不溶性木素)培养基中并未检测到MnP与LiP活性,缺乏MnP和LiP活性的海洋细菌仍能降解木质素,表明MnP与LiP的存在或许并非细菌降解木质素的必要条件。因此,关于MnP和LiP在细菌降解木质素中扮演的角色与发挥的作用还需要进一步阐明。
除上述介绍的几种典型的木质素解聚酶外,超氧化物歧化酶(MnSOD)、过氧化氢-过氧化物酶(KatGs)、细胞色素P450单加氧酶(P450s)等也被发现具有木质素降解作用。Rashid等[37]从Sphingobacterium sp. T2中鉴定出2个胞外MnSOD,这两种酶都能降解有机质和硫酸盐木质素以及不同的木质素模型底物,但它们是否属于典型的细菌木质素解聚酶,仍有待进一步确定。过氧化氢酶-过氧化物酶是一种以过氧化氢为供体释放氧气的氧化还原酶,Amyco1是由Amycolatopsis sp. 75iv2分泌的一种具有降解木质素相关化合物能力的细菌过氧化氢酶-过氧化物酶,其能形成过氧化物酶中典型的双电子和单电子氧化态,并能降解酚型木质素模型化合物,但对其甲基化衍生物不起作用[2, 38]。然而P450s是一类以亚铁血红素为反应活性中心、以分子氧(O2)为氧化剂的多功能氧化还原酶超家族,具有羟基化、环氧化、脱烷基化、硫氧化等多种生物催化活性[39]。此外,有学者发现P450s具有一定的木质素降解能力。例如,Amycolatopsis sp.中的细胞色素酶P450芳香族-O-脱甲基酶能够将木质素单体愈创木酚转化为邻苯二酚,然后进入β-酮己二酸途径,在细菌降解木质素过程中发挥重要作用[40]。
2.2.2 木质素解聚辅助酶除上述介绍的几种核心木质素解聚酶外,细菌中还有其他氧化还原酶类作为辅酶也参与到木质素的降解过程中(图 1),包括过氧化氢酶(CAT)、醌氧化还原酶(QR)、芳基醇氧化酶(AAO)、乙二醛氧化酶(GLOX)以及吡喃糖氧化酶(PROD)等。
在木质素降解过程中,LiP、MnP和DyP等酶类均需要H2O2启动酶促反应,而H2O2对细胞有一定的毒害作用,因此需要过氧化氢酶将其分解。过氧化氢酶通过分解H2O2释放新生态的O2,起到解毒和保护巯基的作用。Amycolatopsis sp. 75iv2分泌的过氧化氢酶Amyco2在木质素降解过程中参与保护机体免受氧化损伤[38]。
此外,木质素的降解过程还需要一些氧化还原酶类在胞外维持氧化还原环境,如醌氧化还原酶。研究表明,醌氧化还原酶具有还原醌和苯氧自由基的能力,在木质素的降解过程中起到还原中间产物、抑制再聚合发生的重要作用,从而有利于木质素的彻底降解。全基因组测序和生物信息学分析结果显示,内生细菌P. ananatis Sd-1中含有大量与木质素降解相关基因,其中包括过氧化氢酶和醌氧化还原酶等编码的基因[41]。然而吡喃糖氧化酶、芳基醇氧化酶、乙二醛氧化酶通过产生H2O2参与到木质素的降解过程中。乙二醛氧化酶能将特定底物乙二醛和甲基乙二醛氧化成相应的酸,同时生成H2O2以协助过氧化物酶完成催化循环(图 1);而芳基醇氧化酶能氧化一系列芳醇(如苯甲基醇)生成相应的醛,同时还原分子氧形成H2O2,并为Mn3+提供螯合剂-草酸[42]。
2.3 木质素解聚酶分泌机制真菌中木质素解聚酶分泌机制的研究较少。现有的研究表明,前导肽序列引导是真菌木质素解聚酶分泌到胞外的有效方式。例如黄孢原毛平革菌中的2个木质素过氧化物酶Li PH2[43]和Li PH8[44],在其N-端约有一个27-28个氨基酸残基的前导序列(leader sequence),编码分泌信号肽,其主要作用是引导分泌蛋白在细胞内沿着正确的途径转移到细胞外发挥作用。
细菌中蛋白质分泌机制的研究相对较为丰富,目前已发现的细菌分泌系统有7个类型,即Ⅰ-Ⅶ型,其中木质素解聚酶主要通过Ⅱ型分泌系统进行分泌。例如,A型DyP (SviDyP[45]、TfuDyP[46]、EfeB[47]) N端含Tat信号肽,通过Tat途径将完全折叠分泌到周浆间隙,经切割、加工,然后通过微孔蛋白穿越外膜分泌到胞外(图 2B)。Sec分泌途径[48-49]是木质素解聚酶分泌到胞外的另一种有效途径(图 2A),通过该途径将尚未发生折叠或部分折叠的蛋白质运送到周质空间,在周质空间折叠成有活性的蛋白质。

|
| 图 2 木质素解聚酶分泌机制[49-51] Figure 2 Secretion mechanism of ligninolytic enzymes[49-51] 注:A:Sec途径;B:Tet途径;C:纳米仓系统;D:OMVs. SP:信号肽;SRP:信号识别粒子;RNC:核糖体-新生肽链;PIP’s:翻译后相互作用的蛋白质;SPase:信号肽酶;CM:细胞质膜;RR:双精氨酸基序;pmf:质子动势;TatABC:双精氨酸易位蛋白ABC. Note: A: Sec pathway; B: Tet approach; C: Nanobin System; D: OMVs. SP: Signal peptides; SRP: Signal recognition particle; RNC: Ribosome-nascent chain; PIP's: Posttranslationally interacting proteins; SPase: Signal peptidase; CM: Cytoplasmic membrane; RR: Twin-arginine motif; pmf: Proton motive force; TatABC: The twin-arginine translocation ABC. |
|
|
除以上两种典型分泌途径外,细菌还能通过非典型的分泌方式将木质素解聚酶分泌到胞外。例如,以蛋白质为基础的纳米仓系统(图 2B)由多面体状的蛋白质亚基排列而成,其内部通常含有具有特定催化作用的酶。Rahmanpour等[50]报道R. jostii RHA1的B型DyP酶(RjDypB)的C端含有被封装素基因识别的信号肽,C端信号肽的所有结合位点都位于纳米室的内部,可以通过纳米舱系统运输到胞外。被封装后的DypB复合物在硝化木质素实验中的活性比单独的DypB高8倍,可能是纳米舱中的封装物有助于将DypB定位到木质素的疏水表面,从而导致其活性增加[2]。但DypB封装复合物的位置并未确定,包裹DypB酶的纳米仓如何输送到细胞外也有待进一步研究。此外,Salvachúa等[51]发现,在木质素底物下,假单胞菌可以通过分泌外膜囊泡(outer membrane vesicles,OMVs)的方式将木质素降解酶运输到胞外(图 2D),从而分解木质素衍生的芳香族化合物,许多已知对木质素衍生芳香族化合物表现出活性的酶(如β-酮己二酸途径中的原儿茶酸3, 4-双加氧酶)动态封装在OMVs中并保持活性。
除上述已知的分泌机制,也有报道显示不含典型信号肽的DyP (如Amycolatopsis sp. 75iv2的DyP2)仍能分泌到胞外发挥作用,表明还存在未知的分泌机制[23]。此外,Lin等[36]也发现P. putida A514中不含典型信号肽的DypB能分泌到细胞周质,但由于未检测到典型的信号肽,其分泌机制还有待进一步研究。
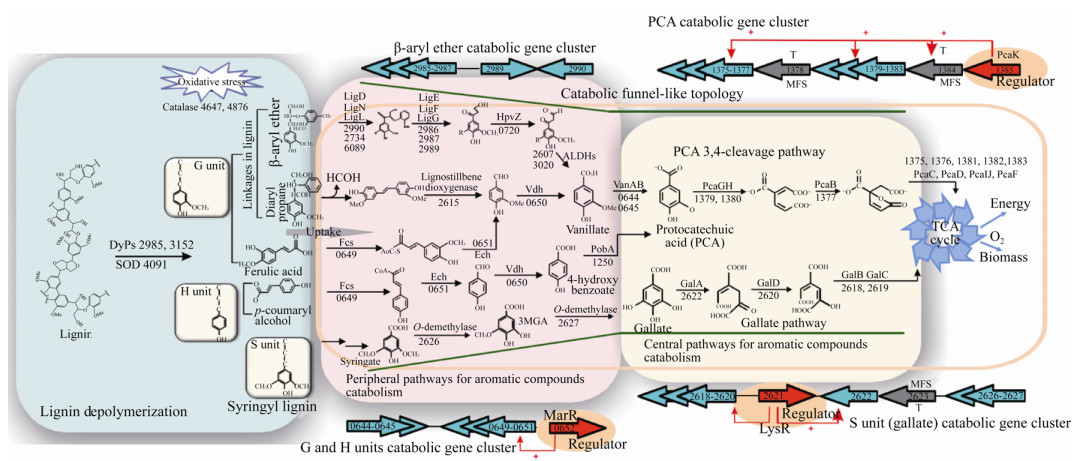
3 木质素衍生体的生物降解途径木质素在胞外进行解聚后产生各种小分子芳香化合物,运输到胞内经过一系列外围分解途径和中心代谢途径生成三羧酸循环中间体,进入三羧酸(TCA)循环为细胞供能。研究表明,芳香族化合物的分解代谢途径主要包括芳基醚的裂解途径、联苯代谢途径、阿魏酸代谢途径、没食子酸甲酯代谢途径、原儿茶酸代谢途径、二芳基丙烷代谢途径、香草酸代谢途径等[52]。但目前代谢途径的解析大多数集中在代表性木质素降解菌株上,例如Sphingomonas sp. SYK-6[53]和S. viridosporus T7A[54]等。近年来,对于其他木质素降解细菌代谢途径的研究也取得了一些积极的进展。Lin等[36]以P. putida A514为模式菌株,通过基因组与蛋白组学数据的分析解析了其木质素解聚途径(图 3),发现木质素衍生芳香族化合物经过5条外围分解代谢途径:β-芳基醚降解途径、二芳基丙烷降解途径、阿魏酸降解途径、对香豆醇降解途径、紫丁香基降解代谢途径;这些外围途径利用经典的依赖O2的环裂解策略来分解这些化合物,并将中间代谢产物汇聚到两条中心分解代谢途径-原儿茶酸3, 4-裂解途径和没食子酸途径,最后进入TCA循环参与细胞的生命活动。
将木质素转化为高价值的生物产品,有助于减少CO2排放、增加净能源、有效提高废物管理和生物燃料的成本效益。目前已知的木质素高值化产品有香草醛、聚羟基脂肪酸酯(polyhydroxyalkanoates,PHA)、脂质体、粘康酸、丙酮酸、乳酸等,下文将进行分类阐述。
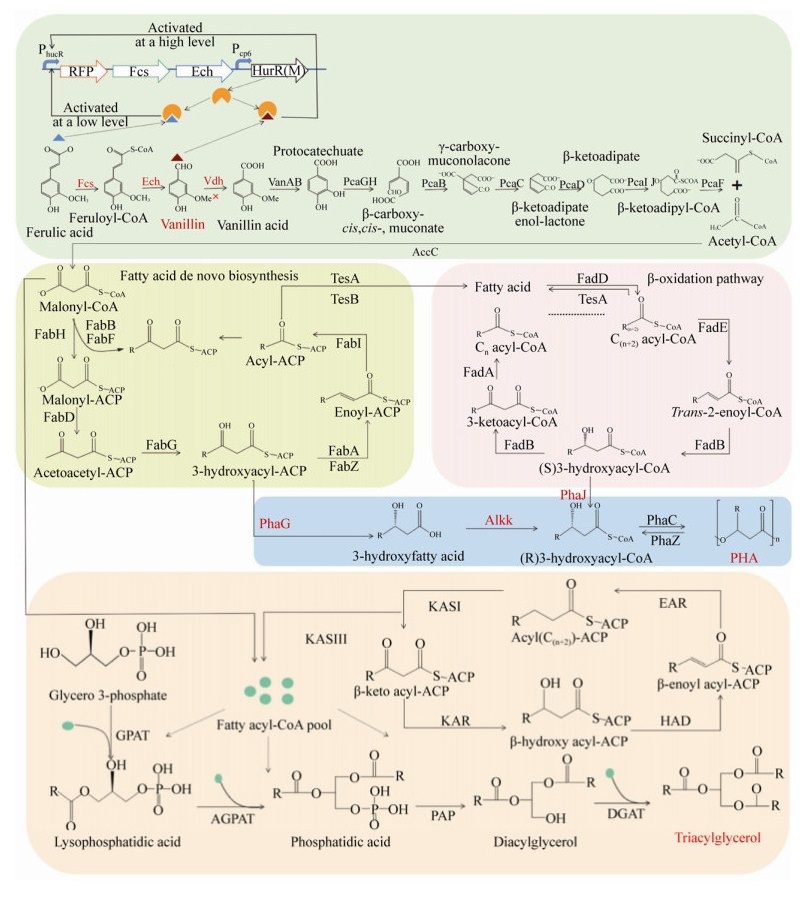
4.1 香草醛香草醛(4-羟基-3-甲氧基苯甲醛)是一类重要的芳香族化合物,目前已被广泛应用于食品、日用品、医药等领域。利用木质素衍生产品生产香草醛已得到广泛的研究[55-56]。香草醛是阿魏酸降解途径的醛类中间体,各种微生物,包括Pseudomonas sp.、E. coli、Rhodococcus sp.、Bacillus subtilis等均已被报道可由阿魏酸生产香草醛[57]。敲除香草脱氢酶(vdh)基因阻断香草醛的分解代谢是提高香草醛产量的最直接策略(图 4)[58-59]。在此基础上,Di Gioia等[63]进一步转入含fcs (编码阿魏酰辅酶A合成酶)和ech(编码烯酰辅酶A水合酶/醛缩酶)基因的低拷贝质粒,在优化培养条件后使香草醛产量得到进一步提高。但高浓度香草醛会破坏细胞生物膜的完整性,从而对细胞产生毒性[64-66]。通过生物合成途径的动态调控可以实现细胞生长和香草醛生产之间的平衡。PhucR是在Deinococcus radiodurans中发现的尿酸诱导启动子,尿酸能与HucR调控蛋白结合,促进PhucR的转录,但在没有尿酸存在的条件下,PhucR的表达被HucR所抑制(图 4)[60]。中国科学院微生物研究所唐双焱团队[60]将PhucR启动子调控的香草醛生物合成途径整合到E. coli基因组中,并对HucR底物口袋的关键位点进行饱和突变,筛选得到能够不同程度感应阿魏酸和香草醛的HucR突变体(HucR-V7),从而不同程度地激活fcs和ech的表达(图 4);在此基础上,对启动强度做精细调控,前期增加阿魏酸浓度启动香草醛生物合成途径低水平表达,以缓解生长前期的代谢负担;后期高浓度香草醛诱导合成途径较高水平表达,显著提高了生物合成的效率,有效地管理了细胞生长和香兰素生产之间的平衡。该研究也为天然产物生物合成途径的设计构建提供了新的思路和调控元件。
聚羟基脂肪酸酯(PHA),是微生物在营养失衡情况下合成的一种生物聚合物,作为自身的碳源和能源储备物质[67]。假单胞菌是目前研究较为丰富的能够进行木质素-PHA生物转化的微生物。Linger等[68]的研究表明,P. putida KT2440可以利用木质素及其衍生物合成PHA,作为储备碳源在细胞内积累。但木质素向PHA的生物转化过程中还存在2个瓶颈:(1) PHA的生物合成过程更倾向于以脂肪酸为底物,在木质素底物下其PHA产量相对较低[69-70]。当前多种基因编辑技术已被开发用于微生物的代谢工程,以克服木质素至PHA的生物转化产量低等难点[71-73]。例如,Zhou等[74]开发出一种适用于假单胞菌的高效的CRISPR-Cas9n基因编辑技术,借助该技术解析出与木质素衍生物阿魏酸分解代谢和PHA生物合成相关的9个关键基因,并将其组装至4个功能模块中,整合于P. putida KT2440基因组中,再经过培养条件的优化,使得阿魏酸至PHA的转化效率得到显著提高。(2)在木质素底物下,PHA通常在营养失衡的条件下(如缺氮条件)积累,但这种营养失衡条件通常会限制菌株生长(如细胞密度低、细胞干重轻或生长速度慢等)[59, 75]。因此,细胞生长和PHA生产之间的不平衡是木质素转化为PHA的另一障碍[76]。针对这一瓶颈,Wang等[61]对P. putida A514中木质素单体到PHA合成途径中的关键基因进行解析,发现phaG(编码3-羟酰基-ACP硫酯酶)和alkK(编码脂肪酸-CoA连接酶)是连接细胞生长和PHA合成的关键桥梁,在PHA生物合成和细胞生长中均发挥作用,提高phaG和alkK的表达水平可同时改善细胞生长和PHA的生物合成(图 4)。
4.3 脂质体脂质体是一种潜在的生物燃料。木质素在微生物体内通过β-酮己二酸途径转化为乙酰CoA,再通过脂肪酸合成途径将其转化为脂质体积累到细胞内[77]。红球菌属(Rhodococcus)是研究较为深入的产油微生物[78]。美国德州农工大学Joshua Yuan教授团队[79]发现当R.opacus PD630细胞与漆酶共同作用木质素时,木质素至脂质体产量增加了17倍,达到145 mg/L,这表明漆酶与细菌细胞在木质素降解过程中具有协同作用。在此基础上,该团队进一步开发出用于木质素的生物转化的漆酶-介质-微生物体系,该体系促进木质素降解,从而促进细胞生长和脂质体合成[77]。上述工作均是体外添加的酶系策略,极大地提高了木质素高值化利用的成本。考虑到体外添加的酶系催化条件和微生物发酵条件容易不一致,该团队[62]以R. opacus PD630为模式菌株构建漆酶胞外高效表达的体系,并以FASI (脂肪酸合成酶Ⅰ)作为关键酶驱动木质素至脂质体的转化,通过优化与整合,木质素至脂质体转化提高至950 mg/L (图 4)。
4.4 其他高值化合物除以上典型化合物外,木质素及其衍生物还能合成丙酮酸、乳酸和粘康酸等化合物。利用合成生物学方法可以有效地改造木质素衍生物的降解途径,提高目标产物的产量。例如,在P. putida KT2440和Sphingobium sp. SYK-6中使用外源的间位裂解途径替代内源的邻苯二酚邻位裂解途径来降解木质素衍生体儿茶酚和原儿茶酚,丙酮酸和乳酸的产量均得到显著提高[80]。粘康酸是生产尼龙的潜在平台化学品。木质素衍生物邻苯二酚可在catA(邻苯二酚-1, 2-双加氧酶)的催化下合成粘康酸,通过catA基因过表达或catB基因(编码粘康酸内酯酶)失活[81-82]等策略可以提高木质素至粘康酸的转化。
5 结论与展望木质素是自然界中含量丰富的芳香化合物,对其分解和转化过程的研究一直是全球碳循环和生物资源研究的热点。近年来在认识和理解木质素降解菌、降解机理以及木质素生物转化等方面取得了积极的进展,极大地丰富了我们对环境微生物驱动木质素有机碳分解代谢的认识,但仍存在以下两个方面的挑战:(1)微生物介导的木质素代谢机理尚不完全清楚。木质素由于结构的复杂性,其降解过程所涉及的酶系种类繁多。目前已对木质素降解酶系组成有了初步的认识[78-79],但仅仅依赖已知的木质素降解酶并不能完全阐明木质素的降解过程[78],推测可能存在未知的酶在木质素降解过程中发挥着重要作用[33]。因此,木质素降解酶系组成成员的进一步挖掘和表征是当前亟待解决的问题。借助高通量组学技术,如基因组、转录组、蛋白质组和代谢物组等,挖掘与木质素代谢相关的基因、潜在的酶系成员以及代谢产物,将有助于进一步阐明微生物降解木质素酶系及其作用机理。另外,胞外木质素降解酶分泌机制的认知也不完整。现有蛋白质分泌机制不能完全解释木质素降解酶如何离开胞内分泌到周质空间或胞外发挥作用。凭借分子生物学技术阐明木质素降解酶分泌机制,完善现有的酶胞外分泌机制是当前急需解决的问题。(2)木质素分解代谢产物如何高效转化成高价值的终端产品是制约木质素生物炼制发展的关键瓶颈[12, 62]。充分利用迅速发展的微生物基因组工程改造策略,对现有的基因组代谢网络进行重新整合和优化;或者通过对关键酶的改造再平衡基因组代谢网络,都将有效提高目标产物的合成水平。此外,借助ZFN或CRISPER-Cas介导的基因组编辑技术能够方便快捷地对微生物基因组进行大片段删除和修饰工作,使有价值的生物产品在一定条件下积累到更高的水平,也有助于实现木质素高值化利用[74]。
综上所述,认识环境微生物降解木质素机制为理解木质素代谢菌在全球碳循环中的作用提供理论依据。在此基础上,开发资源化利用策略将为建立环境有机质资源利用平台奠定基础。
| [1] |
Kamimura N, Sakamoto S, Mitsuda N, et al. Advances in microbial lignin degradation and its applications[J]. Current Opinion in Biotechnology, 2019, 56: 179-186. DOI:10.1016/j.copbio.2018.11.011 |
| [2] |
de Gonzalo G, Colpa DI, Habib MHM, et al. Bacterial enzymes involved in lignin degradation[J]. Journal of Biotechnology, 2016, 236: 110-119. DOI:10.1016/j.jbiotec.2016.08.011 |
| [3] |
Cabana H, Jones JP, Agathos SN. Elimination of endocrine disrupting chemicals using white rot fungi and their lignin modifying enzymes: a review[J]. Engineering in Life Sciences, 2007, 7(5): 429-456. DOI:10.1002/elsc.200700017 |
| [4] |
Liers C, Arnstadt T, Ullrich R, et al. Patterns of lignin degradation and oxidative enzyme secretion by different wood- and litter-colonizing basidiomycetes and ascomycetes grown on beech-wood[J]. FEMS Microbiology Ecology, 2011, 78(1): 91-102. DOI:10.1111/j.1574-6941.2011.01144.x |
| [5] |
Khatami S, Deng Y, Tien M, et al. Formation of water-soluble organic matter through fungal degradation of lignin[J]. Organic Geochemistry, 2019, 135: 64-70. DOI:10.1016/j.orggeochem.2019.06.004 |
| [6] |
Martinez D, Larrondo LF, Putnam N, et al. Genome sequence of the lignocellulose degrading fungus Phanerochaete chrysosporium strain RP78[J]. Nature Biotechnology, 2004, 22(6): 695-700. DOI:10.1038/nbt967 |
| [7] |
Bugg TDH, Ahmad M, Hardiman EM, et al. The emerging role for bacteria in lignin degradation and bio-product formation[J]. Current Opinion in Biotechnology, 2011, 22(3): 394-400. DOI:10.1016/j.copbio.2010.10.009 |
| [8] |
Ahmad M, Taylor CR, Pink D, et al. Development of novel assays for lignin degradation: comparative analysis of bacterial and fungal lignin degraders[J]. Molecular Biosystems, 2010, 6(5): 815-821. DOI:10.1039/b908966g |
| [9] |
Saini A, Aggarwal NK, Sharma A, et al. Actinomycetes: a source of lignocellulolytic enzymes[J]. Enzyme Research, 2015, 2015: 279381. |
| [10] |
Mazurkewich S, Brott AS, Kimber MS, et al. Structural and kinetic characterization of the 4-carboxy-2-hydroxymuconate hydratase from the gallate and protocatechuate 4, 5-cleavage pathways of Pseudomonas putida KT2440[J]. Journal of Biological Chemistry, 2016, 291(14): 7669-7686. DOI:10.1074/jbc.M115.682054 |
| [11] |
Sun J, Wang QZ, Jiang Y, et al. Genome editing and transcriptional repression in Pseudomonas putida KT2440 via the type Ⅱ CRISPR system[J]. Microbial Cell Factories, 2018, 17(1): 41. DOI:10.1186/s12934-018-0887-x |
| [12] |
Lin L, Cheng YB, Pu YQ, et al. Systems biology-guided biodesign of consolidated lignin conversion[J]. Green Chemistry, 2016, 18(20): 5536-5547. DOI:10.1039/C6GC01131D |
| [13] |
Huang XF, Santhanam N, Badri DV, et al. Isolation and characterization of lignin-degrading bacteria from rainforest soils[J]. Biotechnology and Bioengineering, 2013, 110(6): 1616-1626. DOI:10.1002/bit.24833 |
| [14] |
Zhu DC, Zhang PP, Xie CX, et al. Biodegradation of alkaline lignin by Bacillus ligniniphilus L1[J]. Biotechnology for Biofuels, 2017, 10(1): 44. DOI:10.1186/s13068-017-0735-y |
| [15] |
Chen Z, Wan CX. Biological valorization strategies for converting lignin into fuels and chemicals[J]. Renewable and Sustainable Energy Reviews, 2017, 73: 610-621. DOI:10.1016/j.rser.2017.01.166 |
| [16] |
Abdelaziz OY, Brink DP, Prothmann J, et al. Biological valorization of low molecular weight lignin[J]. Biotechnology Advances, 2016, 34(8): 1318-1346. DOI:10.1016/j.biotechadv.2016.10.001 |
| [17] |
Kumar A, Chandra R. Ligninolytic enzymes and its mechanisms for degradation of lignocellulosic waste in environment[J]. Heliyon, 2020, 6(2): e03170. DOI:10.1016/j.heliyon.2020.e03170 |
| [18] |
Palazzolo MA, Kurina-Sanz M. Microbial utilization of lignin: available biotechnologies for its degradation and valorization[J]. World Journal of Microbiology and Biotechnology, 2016, 32(10): 173. DOI:10.1007/s11274-016-2128-y |
| [19] |
Riva S. Laccases: blue enzymes for green chemistry[J]. Trends in Biotechnology, 2006, 24(5): 219-226. DOI:10.1016/j.tibtech.2006.03.006 |
| [20] |
Hofrichter M, Ullrich R, Pecyna MJ, et al. New and classic families of secreted fungal heme peroxidases[J]. Applied Microbiology and Biotechnology, 2010, 87(3): 871-897. |
| [21] |
Nayanashree G, Thippeswamy B. Biodegradation of natural rubber by laccase and manganese peroxidase enzyme of Bacillus subtilis[J]. Environmental Processes, 2015, 2(4): 761-772. DOI:10.1007/s40710-015-0118-y |
| [22] |
Salame TM, Knop D, Levinson D, et al. Inactivation of a Pleurotus ostreatus versatile peroxidase-encoding gene (mnp2) results in reduced lignin degradation[J]. Environmental Microbiology, 2014, 16(1): 265-277. DOI:10.1111/1462-2920.12279 |
| [23] |
Brown ME, Barros T, Chang MCY. Identification and characterization of a multifunctional dye peroxidase from a lignin-reactive bacterium[J]. ACS Chemical Biology, 2012, 7(12): 2074-2081. DOI:10.1021/cb300383y |
| [24] |
Yoshida T, Sugano Y. A structural and functional perspective of DyP-type peroxidase family[J]. Archives of Biochemistry and Biophysics, 2015, 574: 49-55. DOI:10.1016/j.abb.2015.01.022 |
| [25] |
Sugawara K, Nishihashi Y, Narioka T, et al. Characterization of a novel DyP-type peroxidase from Streptomyces avermitilis[J]. Journal of Bioscience and Bioengineering, 2017, 123(4): 425-430. DOI:10.1016/j.jbiosc.2016.12.001 |
| [26] |
Becker J, Wittmann C. A field of dreams: lignin valorization into chemicals, materials, fuels, and health-care products[J]. Biotechnology Advances, 2019, 37(6): 107360. DOI:10.1016/j.biotechadv.2019.02.016 |
| [27] |
Tang J, Duan CR, Huang YY, et al. Characteristics of ligninolytic enzymes of white-rot fungus and their interactions in the process of lignin degradation[J]. Biotechnology Bulletin, 2011(10): 32-36. (in Chinese) 唐菊, 段传人, 黄友莹, 等. 白腐菌木质素降解酶及其在木质素降解过程中的相互作用[J]. 生物技术通报, 2011(10): 32-36. |
| [28] |
Xu R, Zhang K, Liu P, et al. Lignin depolymerization and utilization by bacteria[J]. Bioresource Technology, 2018, 269: 557-566. DOI:10.1016/j.biortech.2018.08.118 |
| [29] |
Chandra R, Chowdhary P. Properties of bacterial laccases and their application in bioremediation of industrial wastes[J]. Environmental Science: Processes & Impacts, 2015, 17(2): 326-342. |
| [30] |
Mikolasch A, Schauer F. Fungal laccases as tools for the synthesis of new hybrid molecules and biomaterials[J]. Applied Microbiology and Biotechnology, 2009, 82(4): 605-624. DOI:10.1007/s00253-009-1869-z |
| [31] |
Dwivedi UN, Singh P, Pandey VP, et al. Structure-function relationship among bacterial, fungal and plant laccases[J]. Journal of Molecular Catalysis B: Enzymatic, 2011, 68(2): 117-128. DOI:10.1016/j.molcatb.2010.11.002 |
| [32] |
Ruijssenaars HJ, Hartmans S. A cloned Bacillus halodurans multicopper oxidase exhibiting alkaline laccase activity[J]. Applied Microbiology and Biotechnology, 2004, 65(2): 177-182. |
| [33] |
Chauhan PS. Role of various bacterial enzymes in complete depolymerization of lignin: a review[J]. Biocatalysis and Agricultural Biotechnology, 2020, 23: 101498. DOI:10.1016/j.bcab.2020.101498 |
| [34] |
Ahmad M, Roberts JN, Hardiman EM, et al. Identification of DypB from Rhodococcus jostii RHA1 as a lignin peroxidase[J]. Biochemistry, 2011, 50(23): 5096-5107. DOI:10.1021/bi101892z |
| [35] |
Rahmanpour R, Bugg TDH. Characterisation of Dyp-type peroxidases from Pseudomonas fluorescens Pf-5: oxidation of Mn(Ⅱ) and polymeric lignin by Dyp1B[J]. Archives of Biochemistry and Biophysics, 2015, 574: 93-98. DOI:10.1016/j.abb.2014.12.022 |
| [36] |
Lin L, Wang XP, Cao LF, et al. Lignin catabolic pathways reveal unique characteristics of dye-decolorizing peroxidases in Pseudomonas putida[J]. Environmental Microbiology, 2019, 21(5): 1847-1863. DOI:10.1111/1462-2920.14593 |
| [37] |
Rashid GMM, Taylor CR, Liu YQX, et al. Identification of manganese superoxide dismutase from Sphingobacterium sp. T2 as a novel bacterial enzyme for lignin oxidation[J]. ACS Chemical Biology, 2015, 10(10): 2286-2294. DOI:10.1021/acschembio.5b00298 |
| [38] |
Brown ME, Walker MC, Nakashige TG, et al. Discovery and characterization of heme enzymes from unsequenced bacteria: application to microbial lignin degradation[J]. Journal of the American Chemical Society, 2011, 133(45): 18006-18009. DOI:10.1021/ja203972q |
| [39] |
Tumen-Velasquez M, Johnson CW, Ahmed A, et al. Accelerating pathway evolution by increasing the gene dosage of chromosomal segments[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(27): 7105-7110. DOI:10.1073/pnas.1803745115 |
| [40] |
Mallinson SJB, Machovina MM, Silveira RL, et al. A promiscuous cytochrome P450 aromatic O-demethylase for lignin bioconversion[J]. Nature Communications, 2018, 9(1): 2487. DOI:10.1038/s41467-018-04878-2 |
| [41] |
Ma JS, Zhang KK, Liao HD, et al. Genomic and secretomic insight into lignocellulolytic system of an endophytic bacterium Pantoea ananatis Sd-1[J]. Biotechnology for Biofuels, 2016, 9: 25. DOI:10.1186/s13068-016-0439-8 |
| [42] |
Yamada Y, Wang JQ, Kawagishi H, et al. Improvement of ligninolytic properties by recombinant expression of glyoxal oxidase gene in hyper lignin-degrading fungus Phanerochaete sordida YK-624[J]. Bioscience, Biotechnology, and Biochemistry, 2014, 78(12): 2128-2133. DOI:10.1080/09168451.2014.946398 |
| [43] |
de Boer HA, Zhang YZ, Collins C, et al. Analysis of nucleotide sequences of two ligninase cDNAs from a white-rot filamentous fungus, Phanerochaete chrysosporium[J]. Gene, 1987, 60(1): 93-102. |
| [44] |
Andrawis A, Pease EA, Kuan IC, et al. Characterization of two lignin peroxidase clones from Phanerochaete chrysosporium[J]. Biochemical and Biophysical Research Communications, 1989, 162(2): 673-680. DOI:10.1016/0006-291X(89)92363-2 |
| [45] |
Yu WN, Liu WN, Huang HQ, et al. Application of a novel alkali-tolerant thermostable DyP-type peroxidase from Saccharomonospora viridis DSM 43017 in biobleaching of eucalyptus kraft pulp[J]. PLoS One, 2014, 9(10): e110319. DOI:10.1371/journal.pone.0110319 |
| [46] |
van Bloois E, Pazmiño DET, Winter RT, et al. A robust and extracellular heme-containing peroxidase from Thermobifida fusca as prototype of a bacterial peroxidase superfamily[J]. Applied Microbiology and Biotechnology, 2010, 86(5): 1419-1430. DOI:10.1007/s00253-009-2369-x |
| [47] |
Zamocký M, Hofbauer S, Schaffner I, et al. Independent evolution of four heme peroxidase superfamilies[J]. Archives of Biochemistry and Biophysics, 2015, 574: 108-119. DOI:10.1016/j.abb.2014.12.025 |
| [48] |
Sugano Y. DyP-type peroxidases comprise a novel heme peroxidase family[J]. Cellular and Molecular Life Sciences, 2009, 66(8): 1387-1403. DOI:10.1007/s00018-008-8651-8 |
| [49] |
Freudl R. Signal peptides for recombinant protein secretion in bacterial expression systems[J]. Microbial Cell Factories, 2018, 17(1): 52. DOI:10.1186/s12934-018-0901-3 |
| [50] |
Rahmanpour R, Bugg TDH. Assembly in vitro of Rhodococcus jostii RHA1 encapsulin and peroxidase DypB to form a nanocompartment[J]. The FEBS Journal, 2013, 280(9): 2097-2104. DOI:10.1111/febs.12234 |
| [51] |
Salvachúa D, Werner AZ, Pardo I, et al. Outer membrane vesicles catabolize lignin-derived aromatic compounds in Pseudomonas putida KT2440[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(17): 9302-9310. DOI:10.1073/pnas.1921073117 |
| [52] |
Xie CX, Sun JZ, Li CL, et al. Exploring the lignin degradation by bacteria[J]. Microbiology China, 2015, 42(6): 1122-1132. (in Chinese) 谢长校, 孙建中, 李成林, 等. 细菌降解木质素的研究进展[J]. 微生物学通报, 2015, 42(6): 1122-1132. |
| [53] |
Masai E, Katayama Y, Fukuda M. Genetic and biochemical investigations on bacterial catabolic pathways for lignin-derived aromatic compounds[J]. Bioscience, Biotechnology, and Biochemistry, 2007, 71(1): 1-15. DOI:10.1271/bbb.60437 |
| [54] |
Zeng J, Singh D, Laskar DD, et al. Degradation of native wheat straw lignin by Streptomyces viridosporus T7A[J]. International Journal of Environmental Science and Technology, 2013, 10(1): 165-174. DOI:10.1007/s13762-012-0085-z |
| [55] |
Chen P, Yan L, Zhang S, et al. Optimizing bioconversion of ferulic acid to vanillin by Bacillus subtilis in the stirred packed reactor using Box-Behnken design and desirability function[J]. Food Science and Biotechnology, 2017, 26(1): 143-152. DOI:10.1007/s10068-017-0019-0 |
| [56] |
Chakraborty D, Kaur B, Obulisamy K, et al. Agrowaste to vanillin conversion by a natural Pediococcus acidilactici strain BD16[J]. Environmental Technology, 2017, 38(13/14): 1823-1834. |
| [57] |
Banerjee G, Chattopadhyay P. Vanillin biotechnology: the perspectives and future[J]. Journal of the Science of Food and Agriculture, 2019, 99(2): 499-506. DOI:10.1002/jsfa.9303 |
| [58] |
Sainsbury PD, Hardiman EM, Ahmad M, et al. Breaking down lignin to high-value chemicals: the conversion of lignocellulose to vanillin in a gene deletion mutant of Rhodococcus jostii RHA1[J]. ACS Chemical Biology, 2013, 8(10): 2151-2156. DOI:10.1021/cb400505a |
| [59] |
Salvachua D, Karp EM, Nimlos CT, et al. Towards lignin consolidated bioprocessing: simultaneous lignin depolymerization and product generation by bacteria[J]. Green Chemistry, 2015, 17(11): 4951-4967. DOI:10.1039/C5GC01165E |
| [60] |
Liang CN, Zhang XX, Wu JY, et al. Dynamic control of toxic natural product biosynthesis by an artificial regulatory circuit[J]. Metabolic Engineering, 2020, 57: 239-246. DOI:10.1016/j.ymben.2019.12.002 |
| [61] |
Wang XP, Lin L, Dong JD, et al. Simultaneous improvements of Pseudomonas cell growth and polyhydroxyalkanoate production from a lignin derivative for lignin-consolidated bioprocessing[J]. Applied and Environment Microbiology, 2018, 84(18): e01469-18. |
| [62] |
Xie SX, Sun S, Lin FR, et al. Mechanism-guided design of highly efficient protein secretion and lipid conversion for biomanufacturing and biorefining[J]. Advanced Science, 2019, 6(13): 1801980. |
| [63] |
Di Gioia D, Luziatelli F, Negroni A, et al. Metabolic engineering of Pseudomonas fluorescens for the production of vanillin from ferulic acid[J]. Journal of Biotechnology, 2011, 156(4): 309-316. DOI:10.1016/j.jbiotec.2011.08.014 |
| [64] |
Cray JA, Stevenson A, Ball P, et al. Chaotropicity: a key factor in product tolerance of biofuel-producing microorganisms[J]. Current Opinion in Biotechnology, 2015, 33: 228-259. DOI:10.1016/j.copbio.2015.02.010 |
| [65] |
Wu WH, Liu F, Singh S. Toward engineering E. coli with an autoregulatory system for lignin valorization[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(12): 2970-2975. DOI:10.1073/pnas.1720129115 |
| [66] |
Ovsienko MV, Fedorova EN, Doroshenko VG. Vanillin resistance induced by BssS overexpression in Escherichia coli[J]. Applied Biochemistry and Microbiology, 2018, 54(1): 21-25. |
| [67] |
Wang Y, Yin J, Chen GQ. Polyhydroxyalkanoates, challenges and opportunities[J]. Current Opinion in Biotechnology, 2014, 30: 59-65. DOI:10.1016/j.copbio.2014.06.001 |
| [68] |
Linger JG, Vardon DR, Guarnieri MT, et al. Lignin valorization through integrated biological funneling and chemical catalysis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(33): 12013-12018. DOI:10.1073/pnas.1410657111 |
| [69] |
Salvachua D, Rydzak T, Auwae R, et al. Metabolic engineering of Pseudomonas putida for increased polyhydroxyalkanoate production from lignin[J]. Microbial Biotechnology, 2020, 13(1): 290-298. |
| [70] |
Meng DC, Chen GQ. Synthetic biology of polyhydroxyalkanoates (PHA)[A]//Zhao HM, Zeng AP. Synthetic Biology - Metabolic Engineering[M]. Cham: Springer, 2017: 147-174
|
| [71] |
Zhang X, Lin YN, Wu Q, et al. Synthetic biology and genome-editing tools for improving PHA metabolic engineering[J]. Trends in Biotechnology, 2020, 38(7): 689-700. DOI:10.1016/j.tibtech.2019.10.006 |
| [72] |
Chen GQ, Jiang XR. Engineering bacteria for enhanced polyhydroxyalkanoates (PHA) biosynthesis[J]. Synthetic and Systems Biotechnology, 2017, 2(3): 192-197. DOI:10.1016/j.synbio.2017.09.001 |
| [73] |
Zheng Y, Chen JC, Ma YM, et al. Engineering biosynthesis of polyhydroxyalkanoates (PHA) for diversity and cost reduction[J]. Metabolic Engineering, 2020, 58: 82-93. DOI:10.1016/j.ymben.2019.07.004 |
| [74] |
Zhou YY, Lin L, Wang H, et al. Development of a CRISPR/Cas9n-based tool for metabolic engineering of Pseudomonas putida for ferulic acid-to-polyhydroxyalkanoate bioconversion[J]. Communications Biology, 2020, 3(1): 98. DOI:10.1038/s42003-020-0824-5 |
| [75] |
Nikodinovic-Runic J, Flanagan M, Hume AR, et al. Analysis of the Pseudomonas putida CA-3 proteome during growth on styrene under nitrogen-limiting and non-limiting conditions[J]. Microbiology, 2009, 155(10): 3348-3361. DOI:10.1099/mic.0.031153-0 |
| [76] |
Zhuang QQ, Wang Q, Liang QF, et al. Synthesis of polyhydroxyalkanoates from glucose that contain medium-chain-length monomers via the reversed fatty acid β-oxidation cycle in Escherichia coli[J]. Metabolic Engineering, 2014, 24: 78-86. DOI:10.1016/j.ymben.2014.05.004 |
| [77] |
Xie SX, Sun QN, Pu YQ, et al. Advanced chemical design for efficient lignin bioconversion[J]. ACS Sustainable Chemistry & Engineering, 2017, 5(3): 2215-2223. |
| [78] |
Kosa M, Ragauskas AJ. Lignin to lipid bioconversion by oleaginous Rhodococci[J]. Green Chemistry, 2013, 15(8): 2070-2074. DOI:10.1039/c3gc40434j |
| [79] |
Zhao C, Xie S, Pu Y, et al. Synergistic enzymatic and microbial lignin conversion[J]. Green Chemistry, 2016, 18(5): 1306-1312. DOI:10.1039/C5GC01955A |
| [80] |
Johnson CW, Beckham GT. Aromatic catabolic pathway selection for optimal production of pyruvate and lactate from lignin[J]. Metabolic Engineering, 2015, 28: 240-247. DOI:10.1016/j.ymben.2015.01.005 |
| [81] |
Barton N, Horbal L, Starck S, et al. Enabling the valorization of guaiacol-based lignin: integrated chemical and biochemical production of cis, cis-muconic acid using metabolically engineered Amycolatopsis sp ATCC 39116[J]. Metabolic Engineering, 2018, 45: 200-210. DOI:10.1016/j.ymben.2017.12.001 |
| [82] |
Johnson CW, Salvachúa D, Khanna P, et al. Enhancing muconic acid production from glucose and lignin-derived aromatic compounds via increased protocatechuate decarboxylase activity[J]. Metabolic Engineering Communications, 2016, 3: 111-119. DOI:10.1016/j.meteno.2016.04.002 |
 2020, Vol. 47
2020, Vol. 47