扩展功能
文章信息
- 沙国萌, 陈冠军, 陈彤, 王禄山
- SHA Guo-Meng, CHEN Guan-Jun, CHEN Tong, WANG Lu-Shan
- 抗生素耐药性的研究进展与控制策略
- Strategies for controlling antibiotic resistance: a review
- 微生物学通报, 2020, 47(10): 3369-3379
- Microbiology China, 2020, 47(10): 3369-3379
- DOI: 10.13344/j.microbiol.china.200668
-
文章历史
- 收稿日期: 2020-06-30
- 接受日期: 2020-08-14
- 网络首发日期: 2020-08-22
2. 山东鲁抗中和环保科技有限公司 山东 济宁 272000
2. Shandong Lukang Zhonghe Environmental Protection Science and Technology Company Limited, Jining, Shandong 272000, China
抗生素作为20世纪最重要的医学发现之一,拯救了无数人的生命,为人类传染病的防治做出了重要贡献[1]。现在人类医学中使用的大约70%的抗菌药物都是在抗生素发现的黄金时期获得的,这些药物大部分从放线菌和真菌中分离得到。然而,在20世纪70年代以后,在实验室条件下通过微生物纯培养的方法已经很难从微生物代谢产物中获取具有开发价值的抗生素。目前抗菌药物的研发多集中于对已知化合物的改造或优化组合[2]。在抗生素发现越来越困难的时期,细菌对抗生素的耐药性却逐年增加[3-5]。抗生素耐药性能够使细菌耐受临床治疗浓度的抗生素,导致抗生素失去治疗的效力[6]。美国疾病控制和预防中心(Centers for Disease Control and Prevention,CDC)统计,美国每年有近2.3万人死于抗生素的耐药性[7]。据预测,到2050年,由多重耐药性造成的人类死亡数将增加到1 000万,超过癌症的死亡人数,成为全球人类死亡的主要原因之一[8]。同时世界卫生组织(World Health Organization,WHO)呼吁要特别注意Klebsiella pneumoniae、Mycobacterium tuberculosis和Neisseria gonorrhoeae等耐药菌的感染,以及血液中毒和食源性的疾病感染,这些感染将变得难以治疗甚至无法治疗[9]。此外,碳青霉烯类药物及粘菌素是目前用于治疗多重耐药菌如Acinetobacter baumannii的“最后手段”[10],但最近的研究也表明质粒携带的耐药基因NMD、mcr-1能够抵抗碳青霉烯类药物及粘菌素的效力,这意味着人类治疗细菌感染的最后一道防线被耐药性所破环[11]。因此,控制耐药性的发展迫在眉睫。
细菌是通过内在的耐药机制及获得性的耐药机制产生对所使用抗生素的耐药性,但是不同种类的抗生素诱发细菌产生的主要耐药机制不同,了解抗生素的研发历史及主要抗生素种类的耐药机制有助于我们针对特定耐药性进行靶向控制。基于此,本文综述了抗生素的研发历程、耐药性的发展及防控策略,这对进一步控制耐药性的发展具有一定的指导意义。
1 抗生素的研发历史1928年Fleming在污染的金黄色葡萄球菌培养基中发现了能抑制葡萄球菌生长的青霉菌所分泌的化学物质——青霉素[12],自此开启了发现抗生素的新时代(图 1)。但是由于缺乏恰当的纯化技术及产量低的问题,青霉素并没有在当时成为能临床应用的抗生素[13]。直至1940年,药理、病理学家Howard Florey和在牛津工作的生物化学家Ernst Chain发明了一种青霉素纯化方法,纯化后青霉素的量足以进行临床试验[14]。通过进一步改进菌株的发酵生产方法,最终在1945年实现了青霉素的工业生产[15]。同年,Dorothy Hodgkin解析了青霉素β-内酰胺的结构,这是一个重大的突破,可以使开发半合成的抗生素绕开青霉素的耐药性[2]。除了生物合成的抗生素外,1908年科研工作者用化学的方法成功合成了磺胺类药物,这是第一个真正起广谱作用的抗生素,并在1931年用于生产[16]。
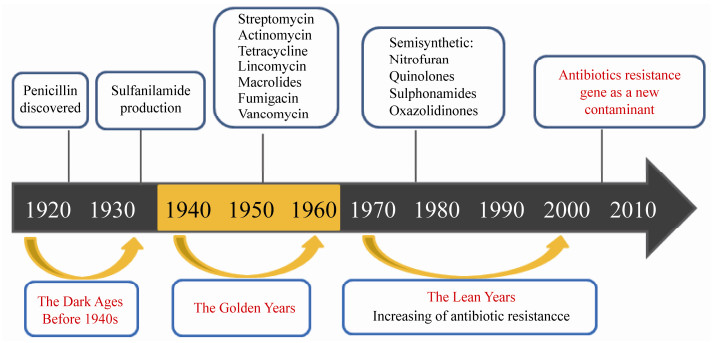
|
| 图 1 抗生素的发现简史 Figure 1 A brief history of the time of antibiotic discovery |
|
|
1940-1960年间是合成及使用抗生素的黄金时代。Waksman在1942年首次提出了抗生素的概念[17],并与他的学生Albert Schatz在土壤放线菌中分离得到了一种新的抗生素——链霉素,临床试验证明,链霉素对革兰氏阴性菌和结核分枝杆菌引起的传染病有效[18]。Waksman的开创性工作确定了链霉菌是许多天然产物或次级代谢产物的生产者,许多链霉菌的天然产物对细菌、真菌、病毒、线虫和昆虫都有活性,甚至有些放线菌产生的次级代谢产物也被用来开发成抗癌和免疫抑制药物[19]。Waksman的工作极大地促进了人们从土壤中分离抗生素的研究,仅仅20年的时间,利用Waksman所建立的分离平台,研究者从几十种细菌和真菌中分离得到了20多种抗生素,包括放线菌素、四环素、林可霉素、烟曲霉素、万古霉素及其他一些大环内酯类的药物[20],目前这些抗生素仍在用于临床治疗。
20世纪60年代以后,从土壤中获取新的抗生素变得越来越困难,人们开始转向基于已知抗生素的作用机制在体外合成新的抗生素分子,但合成出的新抗生素种类很少,仅有1953年的硝基呋喃、1960年的喹诺酮类和1987年的恶唑烷酮类的药物[20]。然而近年来利用基因组挖掘技术探索更多可能编码未知分子的生物合成基因簇的方法[21-23] (biosynthetic gene clusters,BGCs)及利用培养组学对除土壤外的复杂生态系统如植物根系、肠道微生物群的研究[24-26],使探索天然产物的领域重新焕发了活力。此外,还可利用合成生物学的方法,通过对不同基因元件的改造、组合及拼装得到新的微生物代谢途径和体系,以扩大许多天然产物的化学多样性,从而获得新的药物分子,如糖肽类抗生素即合成生物学的成功典例[27]。然而,合成生物学策略在扩大天然产物多样性方面仍然存在一些挑战,包括生物合成基因簇难以异源表达、不可预测的底物特异性及未知的外排系统将新衍生物质泵出细胞外的特异性等[28]。在这个研发新抗生素越发困难的时期,由于抗生素的滥用及其自然筛选功能使得微生物对抗生素的耐药性却逐年增加,有些新的抗生素在研发出1-2年的时间里就会有耐药性微生物的出现。此外,人们通过对细菌耐药性的研究也逐渐意识到基因水平转移在耐药性扩散中的重要作用,其中由质粒介导的耐药性转移已成为医学研究的主要焦点[29]。同时,在临床治疗过程中,由耐药的不动杆菌、肠杆菌、假单胞菌及多重耐药菌引起的感染是对临床医学的挑战[30]。因此,进一步了解耐药性的发展及主要类别抗生素的耐药机制对于有针对性地进行耐药性的靶向控制十分重要。
2 抗生素耐药性的发展及机制 2.1 耐药性的起源与发展抗生素仅在临床使用了80余年,包括多种致病菌在内的许多细菌就对其产生了耐药性,这将使人们在未来面对细菌性或真菌性的疾病感染时无药可用。研究表明抗生素的耐药性实际上是微生物进化过程的产物,许多编码蛋白质的普通基因因为要适应抗生素的存在或代谢抗生素而被激活,被命名为耐药性相关基因即耐药基因[31],其发生并存在于所有的生态位和地质时期。D’Costa等在研究中发现3万年前的永久冻土的宏基因组中存在对β-内酰胺、四环素类和糖肽抗生素基因编码的抗性,该样本中还包含着其他动物及植物的DNA[32]。Kashuba等在西伯利亚的永久冻土中分离得到的人类葡萄球菌基因组中也发现了一些耐药基因[33]。有研究也表明,西伯利亚永久冻土中存在耐药基因,功能宏基因组学显示这些耐药基因至少出现在5 000年前[34]。以上这些横跨近3万年的耐药基因的发现,证实了耐药基因的古老。同时,这些古老的DNA也证明,早在20世纪抗生素生产产业化前,细菌中就已经存在抗生素耐药基因。从生理学的角度分析,对于能够分泌抗生素的微生物来说,其耐药基因必须与抗生素的生物合成基因一样古老。产抗生素的微生物会在抗生素合成的同一基因簇或相邻基因簇上编码耐药基因,这样才能防止抗生素对自身的伤害。可以依据抗生素合成基因簇推测抗性出现的时间[35],目前关于耐药性的古老性问题的研究,可能对预测未来耐药性的发展具有巨大价值。
在过去的几十年里,抗生素的工业化生产及其在人类和动物身上的过度使用为耐药性的扩增、多样化和传播提供了巨大的选择压力,这种压力是抗生素过度使用前微生物所未曾经历的,不仅导致耐药性在污水处理厂的水体、医院废水、畜牧养殖场的固体废弃物中快速扩散,而且还渗入地下水和土壤等[36-39]。目前,对微生物耐药性研究最为广泛的是受人类活动污染的各类水体(如医院及生活污水)及含抗生素残留的固体废弃物(污泥、畜禽粪便及有机工业菌渣)[40-43]。据统计,在2005年时国内就已经建设了600多家污水处理厂,每年产生约500万t的污泥,同时作为畜禽养殖及抗生素的生产大国,每年产生超过40亿t的粪污及数百万吨的菌渣[44-46]。研究表明,污水处理厂的水体及活性污泥中含有大量的耐药基因及耐药微生物,这些耐药基因往往位于可转移质粒的基因组上,从而使得耐药基因在环境微生物中易于扩散和转移[47]。此外,由于抗生素可用于预防疾病及具有促生长功能,其在动物养殖场中往往过度使用,然而这些抗生素在动物体内不能被完全代谢,约60%-90%都会随粪便排出体外,从而使得畜禽粪便成为了耐药性传播的重要来源[48]。同样地,有机工业菌渣也因其未被提取完全的抗生素及产抗生素菌的存在被定义为危险废弃物[49]。但是这些固体废物(如污泥、畜禽粪便及有机工业菌渣)中含有丰富的有机物质,可作为土壤改良剂或肥料,从而改善土壤的物理特性,包括质地、持水性和土壤肥力[40]。固体废弃物中残留的抗生素若直接排放到环境中,将会使得耐药基因及耐药菌在环境中快速扩散,造成严重的环境污染。耐药性在环境中的快速扩散也是临床中病原微生物耐药性的根源。环境微生物在细菌分裂过程中不仅能从母体微生物中获得耐药基因(垂直基因转移),更多地可从整个群落中借助转移元件(质粒、整合子、转座子、插入序列、噬菌体等)获取耐药基因(水平基因转移,如氨基糖苷类的耐药性)[50]。因此,微生物群落通过基因水平转移获取和不断出现的新的耐药性使得环境成为了巨大的耐药基因储存库。
2.2 耐药性的机制如图 2所示,环境微生物,尤其是致病菌产生耐药性的机制包括内在和获得性机制,内在机制包括微生物对毒性物质产生的一系列反应:SOS的应激反应[51]、DNA复制过程中出现的易错聚合酶、广谱性的外排泵[52-53]、染色体上编码的抗生素失活酶(如β-内酰胺酶)和通透性的屏障(如革兰氏阴性菌的外膜和孔蛋白)[54];获得性的耐药机制指通过水平基因转移获取的耐药性,包括化合物特异性的外排泵、修饰靶点或抗生素分子的酶(图 2)。微生物可以通过转移元件(质粒、整合子、转座子、插入序列、噬菌体等)介导的水平基因转移加速耐药基因在环境中的传播,而且研究表明质粒及整合子Intl1在耐药基因的水平转移中起着更为重要的作用[55]。表 1列举了五类主要使用的天然抗生素(β-内酰胺类、氨基糖苷类、大环内酯类、四环素类和氯霉素类)及两类化学合成抗生素(磺胺类、喹诺酮类)的结构特征、作用模式及抗性类型[56-60]。但是微生物对不同种类抗生素产生的主要耐药机制不同,如氨基糖苷类的抗生素耐药性最广泛的传播途径是修饰酶的存在[61],而大环内酯类抗生素的耐药机制主要为药物外排和rRNA的甲基化[62]。此外,进一步的分析表明在抗生素的产生菌及致病菌中存在的耐药机制也不同[63],如氨基糖苷类抗生素产生菌的耐药机制根据不同的抗生素结构而不同,往往以修饰酶为主,而致病菌对氨基糖苷类抗生素的耐药机制包括修饰酶、核糖体靶基因的突变及外排泵[64];大环内酯类抗生素生产菌的主要耐药机制为药物外排和rRNA基因的甲基化,而在病原菌中为23S rRNA基因的突变、外排泵及磷酸转移酶和酯酶等钝化抗生素使其失去活性[65];四环素类抗生素产生菌的主要耐药机制为核糖体保护蛋白和转运蛋白,而在致病菌中至少存在4种已报道的耐药性,即结合位点的突变、核糖体保护蛋白、外排泵和抗生素的钝化酶[66]。氯霉素产生菌的耐药性为磷酸转移酶及水解酶的存在,而在致病菌中主要是氯霉素修饰酶、外排泵和靶点的修饰[67]。目前研究的挑战是进一步了解病原菌通过水平基因转移获取耐药基因的速率并确定耐药基因从抗生素产生菌转移到病原体的关键途径,这对我们思考环境微生物中耐药性向临床致病菌的转移及危害具有重要意义。
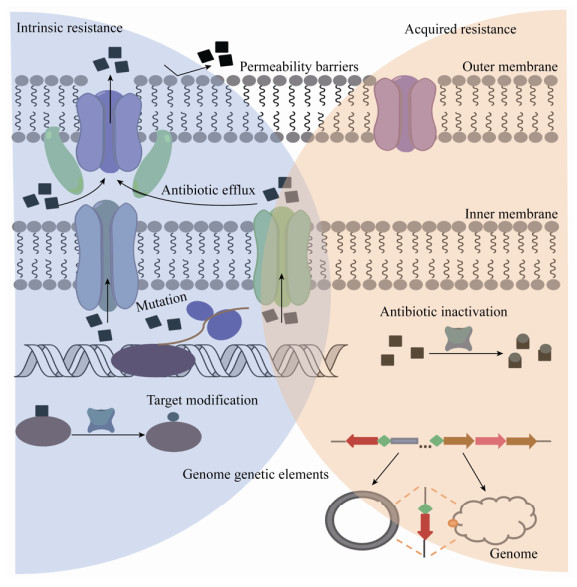
|
| 图 2 内在及获得性的耐药机制 Figure 2 The intrinsic and acquired antibiotic resistomes |
|
|
| 抗生素类型 Antibiotic class |
结构特征 Structural features |
成员 Members |
作用方式 Mode of action |
抗性类型 Resistance type |
| β-lactams | β-lactam ring | Penicillin Cephalosporins Monobactams | Peptidoglycan biosynthesis | β-lactamases efflux altered target |
| Aminoglycosides | Amino-sugars+2 deoxystreptamine (2-DOS) core | Spectinomycin Amikacin Gentamicin | Translation | Modification enzyme efflux altered target |
| Tetracycline | Basic skeleton containing tetraphenyl | Oxytetracycline Tetracycline Chlortetracycline | Translation | Monooxygenation efflux altered target |
| Macrolides | 12-16 carbolactone ring | Erythromycin Spiramycin Tylosin | Translation | Hydrolysis, altered target, glycosylation, efflux, phosphorylation |
| Chloramphenico | Nitrobenzene ring | Chloramphenicol Methiphenicol | Translation | Enzymatic inactivation |
| Quinolone | Possess a quinolone core | Norfloxacin Ofloxacin | DNA replication | Acetylation, efflux, altered target |
| Sulfonamide | Have an aryl sulfonamide moiety in common | Sulfadiazine Sulfamerazine | C1 metabolism | Efflux altered target |
目前人们已意识到微生物耐药性的发展最终会威胁人类的健康,但是新抗生素的发现也必然会伴随着耐药性的产生。因此,对于耐药性的扩散与细菌感染的控制,需要找到合理的平衡点,并通过药敏试验标准加以规范(CLSI/EUCAST)。多年来,WHO也为此给出了许多建议,包括严格控制人类使用抗生素,要求正确的处方,以及控制农业及畜牧业上抗生素的用量[15],即从源头加以控制。自2006年欧盟提出了禁止使用促生长的抗生素后,其他国家也采取了类似的措施以限制来自农业耐药性的发生和传播。此外,在第四届环境抗生素耐药性的国际讨论会上,讨论审议了有关农业和水产养殖中使用抗生素对环境的影响及抗生素生产排放的关键问题,强调减少农业排放抗生素的关键是抗生素的管理[68]。因此,可分别在全球和国家层面建立抗生素耐药性控制委员会,管理抗生素的生产、人畜使用并制定相关政策、规范和监督等[69]。
此外,控制耐药性发展的策略之一就是努力消除环境中残留的抗生素及耐药微生物。由于人类在抗生素生产、疾病医疗及生活中排放的固体废弃物或污水中含有残留的抗生素及丰富的耐药菌,因此对各类污水及固体废弃物进行处理并改进处理工艺来控制耐药性的发展十分重要。有研究表明经污水处理厂处理后的污水中仍然含有大量耐药基因,并在污泥中富集[41],而含有耐药基因的污泥及其他固废在用于农田前可进行进一步的预处理,包括堆肥、厌氧消化、干燥及巴氏杀菌。结果表明通过好氧堆肥处理后有机废物中耐药基因及转移元件的相对丰度显著降低[70-73]。好氧堆肥是一种自发热的微生物发酵过程,通过微生物的活动对有机质进行矿化和部分腐殖化,推动堆肥原料有机质向无机质转化[74]。好氧堆肥包含两个主要阶段:高温阶段及腐熟阶段。在高温阶段,有机物经微生物分解释放出二氧化碳和氨,并伴随热量的产生消除堆肥原料中的病原菌,高温阶段结束后,易于生物降解的部分被完全矿化;在腐殖阶段,另一部分生物降解低的有机物可作为一种新原料形成腐殖质,从而完成对物料的处理[42]。目前超高温堆肥技术在畜禽粪污及污泥堆肥过程去除抗生素残留、抗生素耐药基因及转移元件中显示出更大的优势,可能成为未来固体废弃物处理的主流方式。但目前也有些研究结果表明经过处理后有机废弃物中有些耐药基因减少了,但有些耐药基因反而增加了,对这其中变化的机制尚不清楚,有可能是这些废弃物中含有多种抗生素的残留对微生物的选择压力难以剖析耐药基因变化的真正原因。因此,可以分别以已知的单一抗生素及多种抗生素污染源为研究对象,开展不同种类的抗生素对微生物耐药性的影响,以揭示耐药基因丰度增加或减少的机制。从文献报道看,人们利用高温好氧堆肥的技术对污泥及畜禽粪便中抗生素残留、耐药微生物及耐药基因的研究比抗生素菌渣的研究更多,而对抗生素菌渣的处理仍然是集中于不经济且对环境不友好的方式如焚烧及填埋[75]。因此,找到合适处理抗生素菌渣的工艺来控制耐药性的发展应该是科研工作者予以解决的一个重要问题。现有的文献报道显示,对抗生素菌渣处理的研究大多数是在实验室规模下进行的,对抗生素耐药基因在大规模的商业堆肥过程中的变化了解较少[46]。因此,开展抗生素菌渣的大规模堆肥发酵,了解大生产下耐药微生物及耐药基因的动态变化,寻找能够使抗生素菌渣无害化的处理工艺,对消除抗生素菌渣对环境的污染及耐药基因的扩散具有重要的现实意义,也会对抗生素产业的发展具有深远的影响。
控制微生物耐药性发展的另一方面策略,就是从临床耐药菌株中去除耐药基因。由2种耐药机制(突变及水平基因转移)发展而来的针对细菌进化过程的耐药性抑制及基于CRISPR的基因编辑技术使得从临床菌株中去除耐药菌株的耐药基因成为可能[76]。细菌具有增加诱导突变的活性机制,因此诱导突变是细菌耐药性发生的主要驱动因素,尤其是在应激状态下,如活性氧的产生增加了突变几率[77]。此外,在细菌DNA的损伤反应中(如SOS反应),调节因子Lex A可通过整合子加速基因的交换或促进生物被膜形成耐受性[78]。因此,在医学治疗过程中阻止病原体突变可抑制抗生素耐药性的发生与发展。此外,基因编辑技术能作用于细菌通过基因水平转移获得的耐药性[79]。细菌拥有多种异源基因的防御机制,包括先天的免疫系统和适应性的免疫系统(如CRISPR-Cas),这2种免疫系统能够识别并摧毁外来的非自身DNA。使用这种方法的关键是开发有效的基因工程工具将CRISPR-Cas系统传递给致病菌,这就会使得控制临床耐药菌的扩散成为可能。除此之外,多药物联用也越来越多地用于临床医学来控制病原微生物耐药性的发展。如喹诺酮类药物与大环内酯或氨基糖苷或四环素类的药物联用可以克服病原微生物的耐药性,并且已经成功地用于癌症治疗和艾滋病感染等疾病的治疗[80]。值得注意的是,要确定2种药物联用时到达感染部位的最佳浓度范围十分关键。当然,避开现有耐药性的新药设计对抵抗病原微生物的耐药性也是十分重要的,但对于新型抗生素的使用,如何尽可能地延长其有效性则是新药物设计的重点。
研制高效疫苗或通过抑制细菌毒性来阻止疾病,从而消除对抗生素的需求可能是对某些难治疗耐药菌的最后策略。假想所有的传染病都具有有效疫苗,那抗生素的使用将大幅减少。肺炎球菌疫苗的成功是一个典范,这使我们可寄希望于努力研发有效的疫苗[81],如针对B组链球菌(group B streptococcus,GBS)疫苗可以减少母婴的许多感染,并减少抗生素耐药性[82]。尽管在研制疫苗方面有许多挑战,但对耐药性已经泛滥的许多疾病,疫苗可能是拯救生命最好的选择[83]。
此外,寻找抗生素的替代品也成为了控制耐药性发展的重要手段。目前常见的抗生素替代品包括免疫调节剂、噬菌体、抗菌肽、益生元、植物提取物等[84]。其中噬菌体是唯一的活体药物,与抗生素相比具有很多优势,例如相对安全、专一性强、不影响正常微生物群落的生长等[85],并且对于病原菌有足够的特异性和可选择的种类,解决了“菌变药不变”的抗药性本质[86]。当追踪流行菌株并筛选和组合裂解性噬菌体时,动态抑制病原菌就成为一种自然的生态控制策略。这能够与抗生素精准使用、生态环境及医疗中耐药菌的去除和疫苗预防组成综合的防控策略,使病原菌不易暴发和传播,抗药性得到遏制和降低,实现减抗降抗的战略目标。
4 总结抗生素是用于预防和治疗微生物感染的重要生物活性物质,它们在医学及食品生产中至关重要。目前,抗生素的功效正被细菌对这些药物的耐药性所削弱。抗生素耐药性将对全球经济产生负面影响。美国传染病学会(Infectious Diseases Society,IDS)表示,现有抗生素的耐药性不断上升以及正在开发的新抗生素的缺乏正威胁着公众的健康。因此,本文对抗生素的研发历程、耐药性的起源和发展、主要的分子机制及耐药性在致病菌中的扩散进行了综合性评述,由此可以看出,进一步开展耐药性防控工作是非常有必要的。同时,国家为遏制细菌耐药发展与蔓延、维护人民群众身体健康,也投入了大量的精力和资金用于新药研发和耐药性的控制,并于2016年联合制定了《遏制细菌耐药国家行动计划(2016-2020年)》。对于我们而言,合理谨慎地使用抗生素并处理好人类生活和生产产生的废弃物中耐药性的传播,切断传播途径可能是避免耐药性扩散的重要一环。
| [1] |
Davies J, Davies D. Origins and evolution of antibiotic resistance[J]. Microbiology and Molecular Biology Reviews, 2010, 74(3): 417-433. |
| [2] |
Hutchings MI, Truman AW, Wilkinson B. Antibiotics: past, present and future[J]. Current Opinion in Microbiology, 2019, 51: 72-80. DOI:10.1016/j.mib.2019.10.008 |
| [3] |
Katz L, Baltz RH. Natural product discovery: past, present, and future[J]. Journal of Industrial Microbiology and Biotechnology, 2016, 43(2): 155-176. |
| [4] |
Walsh CT, Wencewicz TA. Prospects for new antibiotics: A molecule-centered perspective[J]. Journal of Antibiotics, 2014, 67(1): 7-22. |
| [5] |
Landecker H. Antibiotic resistance and the biology of history[J]. Body and Society, 2016, 22(4): 19-52. DOI:10.1177/1357034X14561341 |
| [6] |
Lee JH. Perspectives towards antibiotic resistance: from molecules to population[J]. Journal of Microbiology, 2019, 57(3): 181-184. |
| [7] |
Kuehn BM. Proposals seek to reduce resistance, boost development of new antibiotics[J]. JAMA, 2011, 305(18): 1845-1846. DOI:10.1001/jama.2011.598 |
| [8] |
O'Neill J. Tackling a crisis for the health and wealth of nations[J]. Review on Antimicrobial Resistance, 2014, 20: 1-16. |
| [9] |
Bi DX, Jiang XF, Sheng ZK, et al. Mapping the resistance-associated mobilome of a carbapenem-resistant Klebsiella pneumoniae strain reveals insights into factors shaping these regions and facilitates generation of a 'resistance-disarmed' model organism[J]. Journal of Antimicrobial Chemotherapy, 2015, 70(10): 2770-2774. DOI:10.1093/jac/dkv204 |
| [10] |
Lean SS, Suhaili Z, Ismail S, et al. Prevalence and genetic characterization of carbapenem- and polymyxin-resistant Acinetobacter baumannii isolated from a tertiary hospital in Terengganu, Malaysia[J]. ISRN Microbiology, 2014, 2014: 953417. |
| [11] |
Liu YY, Wang Y, Walsh TR, et al. Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China: a microbiological and molecular biological study[J]. The Lancet Infectious Diseases, 2016, 16(2): 161-168. |
| [12] |
Fleming A. On the antibacterial action of cultures of a penicillium, with special reference to their use in the isolation of B. influenzae. 1929[J]. Bulletin of the World Health Organization, 2001, 79(8): 780-790. |
| [13] |
Dai JG, Zhang GQ, Huang XB, et al. A brief history of antibiotic science[J]. Chinese Journal of Medical History, 1999, 29(2): 88-91. (in Chinese) 戴纪刚, 张国强, 黄小兵, 等. 抗生素科学发展简史[J]. 中华医史杂志, 1999, 29(2): 88-91. |
| [14] |
Chain E, Florey HW, Gardner AD, et al. The classic: penicillin as a chemotherapeutic agent[J]. Clinical Orthopaedics and Related Research, 2005, 439: 23-26. DOI:10.1097/01.blo.0000183429.83168.07 |
| [15] |
Aminov RI. A brief history of the antibiotic era: lessons learned and challenges for the future[J]. Frontiers in Microbiology, 2010, 1: 134. |
| [16] |
Jeśman C, Młudzik A, Cybulska M. History of antibiotics and sulphonamides discoveries[J]. Polski Merkuriusz Lekarski, 2011, 30(179): 320-322. |
| [17] |
Aminov R. History of antimicrobial drug discovery: Major classes and health impact[J]. Biochemical Pharmacology, 2017, 133: 4-19. DOI:10.1016/j.bcp.2016.10.001 |
| [18] |
Lewis K. Antibiotics: Recover the lost art of drug discovery[J]. Nature, 2012, 485(7399): 439-440. DOI:10.1038/485439a |
| [19] |
Newman DJ, Cragg GM. Natural products as sources of new drugs from 1981 to 2014[J]. Journal of Natural Products, 2016, 79(3): 629-661. DOI:10.1021/acs.jnatprod.5b01055 |
| [20] |
Durand GA, Raoult D, Dubourg G. Antibiotic discovery: history, methods and perspectives[J]. International Journal of Antimicrobial Agents, 2019, 53(4): 371-382. DOI:10.1016/j.ijantimicag.2018.11.010 |
| [21] |
Cimermancic P, Medema MH, Claesen J, et al. Insights into secondary metabolism from a global analysis of prokaryotic biosynthetic gene clusters[J]. Cell, 2014, 158(2): 412-421. DOI:10.1016/j.cell.2014.06.034 |
| [22] |
Tracanna V, de Jong A, Medema MH, et al. Mining prokaryotes for antimicrobial compounds: from diversity to function[J]. FEMS Microbiology Reviews, 2017, 41(3): 417-429. DOI:10.1093/femsre/fux014 |
| [23] |
Walsh CJ, Guinane CM, Hill C, et al. In silico identification of bacteriocin gene clusters in the gastrointestinal tract, based on the Human Microbiome Project's reference genome database[J]. BMC Microbiology, 2015, 15: 183. DOI:10.1186/s12866-015-0515-4 |
| [24] |
Lagier JC, Dubourg G, Million M, et al. Culturing the human microbiota and culturomics[J]. Nature Reviews Microbiology, 2018, 16(9): 540-550. DOI:10.1038/s41579-018-0041-0 |
| [25] |
Qin ZW, Munnoch JT, Devine R, et al. Formicamycins, antibacterial polyketides produced by Streptomyces formicae isolated from African Tetraponera plant-ants[J]. Chemical Science, 2017, 8(4): 3218-3227. DOI:10.1039/C6SC04265A |
| [26] |
Chu J, Vila-Farres X, Inoyama D, et al. Discovery of MRSA active antibiotics using primary sequence from the human microbiome[J]. Nature Chemical Biology, 2016, 12(12): 1004-1006. DOI:10.1038/nchembio.2207 |
| [27] |
Yim G, Wang WL, Thaker MN, et al. How to make a glycopeptide: a synthetic biology approach to expand antibiotic chemical diversity[J]. ACS Infectious Diseases, 2016, 2(9): 642-650. DOI:10.1021/acsinfecdis.6b00105 |
| [28] |
Thaker MN, Wright GD. Opportunities for synthetic biology in antibiotics: expanding glycopeptide chemical diversity[J]. ACS Synthetic Biology, 2015, 4(3): 195-206. |
| [29] |
Acheampong G, Owusu M, Owusu-Ofori A, et al. Chromosomal and plasmid-mediated fluoroquinolone resistance in human Salmonella enterica infection in Ghana[J]. BMC Infectious Diseases, 2019, 19(1): 898. DOI:10.1186/s12879-019-4522-1 |
| [30] |
Gould K. Antibiotics: from prehistory to the present day[J]. The Journal of Antimicrobial Chemotherapy, 2016, 71(3): 572-575. DOI:10.1093/jac/dkv484 |
| [31] |
Rather PN. Origins of the aminoglycoside modifying enzymes[J]. Drug Resistance Updates, 1998, 1(5): 285-291. DOI:10.1016/S1368-7646(98)80044-7 |
| [32] |
D'Costa VM, McGrann KM, Hughes DW, et al. Sampling the antibiotic resistome[J]. Science, 2006, 311(5759): 374-377. DOI:10.1126/science.1120800 |
| [33] |
Kashuba E, Dmitriev AA, Kamal SM, et al. Ancient permafrost staphylococci carry antibiotic resistance genes[J]. Microbial Ecology in Health and Disease, 2017, 28(1): 1345574. DOI:10.1080/16512235.2017.1345574 |
| [34] |
Perron GG, Whyte L, Turnbaugh PJ, et al. Functional characterization of bacteria isolated from ancient arctic soil exposes diverse resistance mechanisms to modern antibiotics[J]. PLoS ONE, 2015, 10(3): e0069533. DOI:10.1371/journal.pone.0069533 |
| [35] |
Perry J, Waglechner N, Wright G. The prehistory of antibiotic resistance[J]. Cold Spring Harbor Perspectives in Medicine, 2016, 6(6): a025197. DOI:10.1101/cshperspect.a025197 |
| [36] |
Surette MD, Wright GD. Lessons from the environmental antibiotic resistome[J]. Annual Review of Microbiology, 2017, 71: 309-329. DOI:10.1146/annurev-micro-090816-093420 |
| [37] |
Stanton IC, Bethel A, Leonard AFC, et al. What is the research evidence for antibiotic resistance exposure and transmission to humans from the environment? A systematic map protocol[J]. Environmental Evidence, 2020, 9(1): 12. DOI:10.1186/s13750-020-00197-6 |
| [38] |
Bengtsson-Palme J, Milakovic M, Švecová H, et al. Industrial wastewater treatment plant enriches antibiotic resistance genes and alters the structure of microbial communities[J]. Water Research, 2019, 162: 437-445. DOI:10.1016/j.watres.2019.06.073 |
| [39] |
Yuan K, Wang XW, Chen X, et al. Occurrence of antibiotic resistance genes in extracellular and intracellular DNA from sediments collected from two types of aquaculture farms[J]. Chemosphere, 2019, 234: 520-527. DOI:10.1016/j.chemosphere.2019.06.085 |
| [40] |
Su JQ, Wei B, Ouyang WY, et al. Antibiotic resistome and its association with bacterial communities during sewage sludge composting[J]. Environmental Science and Technology, 2015, 49(12): 7356-7363. DOI:10.1021/acs.est.5b01012 |
| [41] |
Wang MY, Shen WT, Yan L, et al. Stepwise impact of urban wastewater treatment on the bacterial community structure, antibiotic contents, and prevalence of antimicrobial resistance[J]. Environmental Pollution, 2017, 231: 1578-1585. DOI:10.1016/j.envpol.2017.09.055 |
| [42] |
Ezzariai A, Hafidi M, Khadra A, et al. Human and veterinary antibiotics during composting of sludge or manure: Global perspectives on persistence, degradation, and resistance genes[J]. Journal of Hazardous Materials, 2018, 359: 465-481. DOI:10.1016/j.jhazmat.2018.07.092 |
| [43] |
Ren ST, Guo XL, Lu AQ, et al. Effects of co-composting of lincomycin mycelia dregs with furfural slag on lincomycin degradation, maturity and microbial communities[J]. Bioresource Technology, 2018, 265: 155-162. DOI:10.1016/j.biortech.2018.05.087 |
| [44] |
Chen ZQ, Wang Y, Wen QX, et al. Feasibility study of recycling cephalosporin C fermentation dregs using co-composting process with activated sludge as co-substrate[J]. Environmental Technology, 2016, 37(17): 2222-2230. DOI:10.1080/09593330.2016.1146340 |
| [45] |
Cai QY, Mo CH, Wu QT, et al. Bioremediation of polycyclic aromatic hydrocarbons (PAHs)-contaminated sewage sludge by different composting processes[J]. Journal of Hazardous Materials, 2007, 142(1/2): 535-542. |
| [46] |
Zhang M, He LY, Liu YS, et al. Variation of antibiotic resistome during commercial livestock manure composting[J]. Environment International, 2020, 136: 105458. DOI:10.1016/j.envint.2020.105458 |
| [47] |
Ju F, Beck K, Yin XL, et al. Wastewater treatment plant resistomes are shaped by bacterial composition, genetic exchange, and upregulated expression in the effluent microbiomes[J]. The ISME Journal, 2019, 13(2): 346-360. DOI:10.1038/s41396-018-0277-8 |
| [48] |
Gou M, Hu HW, Zhang YJ, et al. Aerobic composting reduces antibiotic resistance genes in cattle manure and the resistome dissemination in agricultural soils[J]. The Science of the Total Environment, 2018, 612: 1300-1310. DOI:10.1016/j.scitotenv.2017.09.028 |
| [49] |
Ren ST, Lu AQ, Guo XY, et al. Effects of co-composting of lincomycin mycelia dregs with furfural slag on lincomycin degradation, degradation products, antibiotic resistance genes and bacterial community[J]. Bioresource Technology, 2019, 272: 83-91. DOI:10.1016/j.biortech.2018.10.014 |
| [50] |
Stokes HW, Gillings MR. Gene flow, mobile genetic elements and the recruitment of antibiotic resistance genes into Gram-negative pathogens[J]. FEMS Microbiology Reviews, 2011, 35(5): 790-819. DOI:10.1111/j.1574-6976.2011.00273.x |
| [51] |
Culyba MJ, Mo CY, Kohli RM. Targets for combating the evolution of acquired antibiotic resistance[J]. Biochemistry, 2015, 54(23): 3573-3582. DOI:10.1021/acs.biochem.5b00109 |
| [52] |
Martinez JL, Sánchez MB, Martínez-Solano L, et al. Functional role of bacterial multidrug efflux pumps in microbial natural ecosystems[J]. FEMS Microbiology Reviews, 2009, 33(2): 430-449. DOI:10.1111/j.1574-6976.2008.00157.x |
| [53] |
Kobylka J, Kuth MS, Müller RT, et al. AcrB: a mean, keen, drug efflux machine[J]. Annals of the New York Academy of Sciences, 2020, 1459(1): 38-68. DOI:10.1111/nyas.14239 |
| [54] |
Nikaido H. Molecular basis of bacterial outer membrane permeability revisited[J]. Microbiology and Molecular Biology Reviews, 2003, 67(4): 593-656. DOI:10.1128/MMBR.67.4.593-656.2003 |
| [55] |
Huddleston JR. Horizontal gene transfer in the human gastrointestinal tract: potential spread of antibiotic resistance genes[J]. Infection and Drug Resistance, 2014, 7: 167-176. |
| [56] |
Sultan I, Rahman S, Jan AT, et al. Antibiotics, resistome and resistance mechanisms: a bacterial perspective[J]. Frontiers in Microbiology, 2018, 9: 2066. DOI:10.3389/fmicb.2018.02066 |
| [57] |
Sengupta S, Chattopadhyay MK, Grossart HP. The multifaceted roles of antibiotics and antibiotic resistance in nature[J]. Frontiers in Microbiology, 2013, 4: 47. |
| [58] |
Sheykhsaran E, Baghi HB, Soroush MH, et al. An overview of tetracyclines and related resistance mechanisms[J]. Reviews in Medical Microbiology, 2019, 30(1): 69-75. |
| [59] |
Mann J. Antibiotics: actions, origins, resistance[J]. Natural Product Reports, 2005, 22(2): 304-305. DOI:10.1039/b417591n |
| [60] |
Alekshun MN, Levy SB. Molecular mechanisms of antibacterial multidrug resistance[J]. Cell, 2007, 128(6): 1037-1050. DOI:10.1016/j.cell.2007.03.004 |
| [61] |
Wu LZ, Hu D, Qin M. Deciphering mechanisms of aminoglycoside antibiotics with enzymes that cause resistance to their action[J]. Acta Biophysica Sinica, 2013, 29(1): 15-25. (in Chinese) 武灵芝, 胡栋, 秦猛. 氨基糖苷类修饰酶引起的细菌耐药性机制的研究进展[J]. 生物物理学报, 2013, 29(1): 15-25. |
| [62] |
Franceschi F, Kanyo Z, Sherer EC, et al. Macrolide resistance from the ribosome perspective[J]. Current Drug Targets-Infectious Disorders, 2004, 4(3): 177-191. DOI:10.2174/1568005043340740 |
| [63] |
Ogawara H. Comparison of antibiotic resistance mechanisms in antibiotic-producing and pathogenic bacteria[J]. Molecules, 2019, 24(19): 3430. DOI:10.3390/molecules24193430 |
| [64] |
Costello SE, Deshpande LM, Davis AP, et al. Aminoglycoside-modifying enzyme and 16S ribosomal RNA methyltransferase genes among a global collection of gram-negative isolates[J]. Journal of Global Antimicrobial Resistance, 2019, 16: 278-285. DOI:10.1016/j.jgar.2018.10.020 |
| [65] |
Fong DH, Burk DL, Blanchet J, et al. Structural basis for kinase-mediated macrolide antibiotic resistance[J]. Structure, 2017, 25(5): 750-761. DOI:10.1016/j.str.2017.03.007 |
| [66] |
Thaker M, Spanogiannopoulos P, Wright GD. The tetracycline resistome[J]. Cellular and Molecular Life Sciences, 2010, 67(3): 419-431. DOI:10.1007/s00018-009-0172-6 |
| [67] |
Mosher RH, Ranade NP, Schrempf H, et al. Chloramphenicol resistance in Streptomyces: cloning and characterization of a chloramphenicol hydrolase gene from Streptomyces venezuelae[J]. Journal of General Microbiology, 1990, 136(2): 293-301. DOI:10.1099/00221287-136-2-293 |
| [68] |
Topp E, Larsson DGJ, Miller DN, et al. Antimicrobial resistance and the environment: assessment of advances, gaps and recommendations for agriculture, aquaculture and pharmaceutical manufacturing[J]. FEMS Microbiology Ecology, 2018, 94(3): fix185. |
| [69] |
Zhu YG, Ouyang WY, Wu N, et al. Antibiotic resistance: sources and mitigation[J]. Bulletin of the Chinese Academy of Sciences, 2015, 30(4): 509-516. (in Chinese) 朱永官, 欧阳纬莹, 吴楠, 等. 抗生素耐药性的来源与控制对策[J]. 中国科学院院刊, 2015, 30(4): 509-516. |
| [70] |
Zhang LL, Li LJ, Sha GM, et al. Aerobic composting as an effective cow manure management strategy for reducing the dissemination of antibiotic resistance genes: An integrated meta-omics study[J]. Journal of Hazardous Materials, 2020, 386: 121895. DOI:10.1016/j.jhazmat.2019.121895 |
| [71] |
Goulas A, Belhadi D, Descamps A, et al. How effective are strategies to control the dissemination of antibiotic resistance in the environment? A systematic review[J]. Environmental Evidence, 2020, 9(1): 4. DOI:10.1186/s13750-020-0187-x |
| [72] |
Diehl DL, LaPara TM. Effect of temperature on the fate of genes encoding tetracycline resistance and the integrase of class 1 integrons within anaerobic and aerobic digesters treating municipal wastewater solids[J]. Environmental Science & Technology, 2010, 44(23): 9128-9133. |
| [73] |
Liao HP, Lu XM, Rensing C, et al. Hyperthermophilic composting accelerates the removal of antibiotic resistance genes and mobile genetic elements in sewage sludge[J]. Environmental Science and Technology, 2018, 52(1): 266-276. DOI:10.1021/acs.est.7b04483 |
| [74] |
Zhang LL, Li LJ, Pan XG, et al. Enhanced growth and activities of the dominant functional microbiota of chicken manure composts in the presence of maize straw[J]. Frontiers in Microbiology, 2018, 9: 1131. DOI:10.3389/fmicb.2018.01131 |
| [75] |
van Luong H, Tai PH, Nishijo M, et al. Association of dioxin exposure and reproductive hormone levels in men living near the Bien Hoa airbase, Vietnam[J]. Science of the Total Environment, 2018, 628-629: 484-489. DOI:10.1016/j.scitotenv.2018.02.094 |
| [76] |
Bikard D, Euler CW, Jiang WY, et al. Exploiting CRISPR-Cas nucleases to produce sequence-specific antimicrobials[J]. Nature Biotechnology, 2014, 32(11): 1146-1150. DOI:10.1038/nbt.3043 |
| [77] |
Merrikh H, Kohli RM. Targeting evolution to inhibit antibiotic resistance[J]. The FEBS Journal, 2020. DOI:10.1111/febs.15370 |
| [78] |
Guerin E, Cambray G, Sanchez-Alberola N, et al. The SOS response controls integron recombination[J]. Science, 2009, 324(5930): 1034. DOI:10.1126/science.1172914 |
| [79] |
MacLean RC, Millan AS. The evolution of antibiotic resistance[J]. Science, 2019, 365(6458): 1082-1083. DOI:10.1126/science.aax3879 |
| [80] |
Baym M, Stone LK, Kishony R. Multidrug evolutionary strategies to reverse antibiotic resistance[J]. Science, 2016, 351(6268): aad3292. DOI:10.1126/science.aad3292 |
| [81] |
Theuretzbacher U. New drugs-will they solve the problem of resistance to antibiotics?[J]. Clinical Microbiology and Infection, 2017, 23(10): 695-696. DOI:10.1016/j.cmi.2017.08.013 |
| [82] |
Paynter J, Goodyear-Smith F, Morgan J, et al. Effectiveness of a group B outer membrane vesicle meningococcal vaccine in preventing hospitalization from gonorrhea in New Zealand: A retrospective cohort study[J]. Vaccines, 2019, 7(1): 5. |
| [83] |
Clift C, Salisbury DM. Enhancing the role of vaccines in combatting antimicrobial resistance[J]. Vaccine, 2017, 35(48): 6591-6593. DOI:10.1016/j.vaccine.2017.09.053 |
| [84] |
Cheng GY, Hao HH, Xie SY, et al. Antibiotic alternatives: the substitution of antibiotics in animal husbandry?[J]. Frontiers in Microbiology, 2014, 5: 217. |
| [85] |
Kutateladze M, Adamia R. Bacteriophages-alternative approach to treat infections caused by bacterial pathogens[J]. International Journal of Infectious Diseases, 2012, 16(S1): E443-E444. |
| [86] |
Zhang Q, Shang Y, Zhu JS, et al. Defense and anti-defense mechanisms of bacteriophages and host bacteria[J]. Shandong Agricultural Sciences, 2018, 50(7): 48-54. (in Chinese) 张庆, 商延, 朱见深, 等. 噬菌体与宿主细菌的攻防机制[J]. 山东农业科学, 2018, 50(7): 48-54. |
 2020, Vol. 47
2020, Vol. 47




