扩展功能
文章信息
- 毕晓琼, 高菡, 郭倩, 崔浪军, 李明珠
- BI Xiao-Qiong, GAO Han, GUO Qian, CUI Lang-Jun, LI Ming-Zhu
- 三种猕猴桃根腐病致病菌多重实时定量PCR检测技术的建立及应用
- Development and application of a multiplex real-time PCR assay for quantitative detection of three pathogens related to kiwifruit root rot disease
- 微生物学通报, 2020, 47(1): 311-321
- Microbiology China, 2020, 47(1): 311-321
- DOI: 10.13344/j.microbiol.china.190410
-
文章历史
- 收稿日期: 2019-05-10
- 接受日期: 2019-07-17
- 网络首发日期: 2019-09-16
疫霉菌(Phytophthora de Bary)是一类重要的植物病原菌,破坏性大且寄主范围广,对林木、农作物和花卉等都能造成非常大的危害[1]。据报道有多种疫霉菌,包括隐地疫霉(P. cryptogea)、柑橘褐腐疫霉(P. citrophthora)、掘氏疫霉(P. drechsleri)、棕榈疫霉(P. palmivora)、恶疫霉(P. cactorum)、樟疫霉(P. cinnamomi)、大雄疫霉(P. megasperma)、柑橘生疫霉(P. citricola)和雪松疫霉(P. lateralis)与猕猴桃病害密切相关[2-4]。1990年,在河南省首次从染病猕猴桃树的根中分离出恶疫霉、樟疫霉和雪松疫霉3种疫霉菌[5]。
从1993年至今,猕猴桃种植面积不断增加。自2013年以来,中国已成为世界上猕猴桃产量最多且种植面积最大的国家[6]。陕西省作为国内最大的猕猴桃生产基地,年产60万t以上,占据了国内猕猴桃生产总产量的一半[6]。王汝贤等曾对陕西省猕猴桃产区的病害进行过调查,发现猕猴桃疫霉病在大多数果园均有不同程度的发生,重病园发病率达到了20%-30%,死亡率达到10%-20%[7]。
疫霉菌卵孢子、厚壁孢子和菌丝体随病残体可在土壤中越冬。春末夏初随雨水或灌溉水传播,病害一旦发生往往难以控制。PCR技术已成为植物病害诊断和研究的重要工具。大多数PCR诊断技术是基于核编码核糖体DNA (rDNA)基因的内转录间隔区(internal transcribed spacer,ITS)或序列特征性扩增区(sequence characterized amplified regions,SCAR)建立的[1],但是ITS序列往往无法区分近缘种[8],同时SCAR引物的设计比较繁琐[9]。此外,线粒体cox1和cox2基因、β微管蛋白基因、elicitin基因和Ypt1 (ras-related protein gene)基因也逐渐被应用于疫霉菌分子标记的开发中[10-11]。其中Ypt1基因具有保守的外显子和高度可变的内含子,几乎适用于所有疫霉菌种特异性标记的开发[10]。
近年来一些新的等温扩增技术也相继出现,如环介导等温扩增(loop-mediated isothermal amplification,LAMP)[12]和重组酶聚合酶扩增(recombinase polymerase amplification,RPA)[13]。然而这些检测有一定的局限性,比如在实际操作中容易污染、成本高且无法进行多目标检测。
本研究建立了一种多重PCR实时定量检测技术,可以同时检测恶疫霉、樟疫霉和雪松疫霉3种猕猴桃致病疫霉,并成功将该技术应用于我国猕猴桃主产区疫霉病害的定量检测分析。
1 材料与方法 1.1 材料 1.1.1 主要试剂和仪器氯化苄、脱脂乳、醋酸钠,国药集团化学试剂(北京)有限公司;V8汁琼脂培养基(340 mL V8 juice,6.8 g碳酸钙,16 g/L琼脂)、玉米粉琼脂培养基(CMA),海博生物科技有限公司;马铃薯葡萄糖琼脂培养基(PDA),北京奥博星生物科技有限公司;MagExtractor Plant Genome Kit,Toyobo公司;TaKaRa Taq Hot Start Version和Probe qPCR Mix,TaKaRa公司;探针和引物均由TaKaRa公司合成。荧光定量PCR仪,Bio-Rad公司;Qbuit 3.0、PCR仪,Thermo Fisher Scientific公司;凝胶成像分析系统,天能公司;Field Scout pH 400 m,Spectrum Technologies公司。
1.1.2 供试菌株与菌株培养供试菌株:共50种不同菌种,其中44种疫霉菌和6种土传性病原菌,包括腐霉菌属、镰刀菌属、丝核菌属和轮枝菌属(表 1)。供试菌株来源于多个菌种资源保存机构,包括荷兰菌种保藏中心(Centraalbureau fur Schimmelcultures,the Netherlands,CBS)、世界疫霉遗传资源中心(World Phytophthora Genetic Resource Collection,USA,WPC)、日本农业部(Ministry of Agriculture,Forestry and Fisheries,Japan,MAFF)、日本NITE生物资源中心(NITE Biological Resource Centre,Japan,NBRC)和日本岐阜大学。其余菌株则从陕西省周至县和眉县的猕猴桃果园内分离所得。菌种放置于V8汁琼脂培养基(V8A)、玉米粉琼脂培养基(CMA)或马铃薯葡萄糖琼脂培养基(PDA)中于20 ℃黑暗中保存。
| 进化分支 Clade# |
模式菌株Type isolate |
菌种Species | 分离株Isolate & | 来源Origins | 地区Region | 特异性检测Specificity tests$ | ||
| Yph_cac_ R5/Yph1F_ mod2/cac_ Ypro |
Yph_cin_ R1/Yph1F_ mod2/cin_ Ypro |
Yph_lat_ R2/Yph1F_ mod2/lat_ Ypro |
||||||
| 1 | Phytophthora nicotianae | GF468 | Strawberry | Gifu, Japan | – | – | – | |
| 1a | P. cactorum | CH989A11 | Strawberry | Gifu, Japan | + | – | – | |
| ZZ017 | Kiwifruit | Shaanxi, China | + | – | – | |||
| * | P. hedraiandra | CBS111725 | Viburnum sp. | Netherlands | – | – | – | |
| * | P. idaei | WPC6767 | Rubus idaeus | UK | – | – | – | |
| * | P. pseudotsugae | WPC10339 | Pseudotsuga menziesii | USA | – | N | N | |
| 1b | P. clandestina | WPC3942 | Trifolium subterraneum | Australia | – | N | N | |
| * | P. iranica | CBS374.72 | Solanum melongena | Iran | – | – | – | |
| * | P. tentaculata | CBS552.96 | Chrysanthemum leucanthemum | Germany | – | N | N | |
| 1c | P. infestans | MAFF305586 | Potato | Hokkaido, Japan | – | N | N | |
| * | P. ipomoeae | WPC10225 | Ipomoea longipedunculata | Mexico | – | – | – | |
| P. mirabilis | WPC3005 | Mirabilis jalapa | Mexico | – | N | N | ||
| P. phaseoli | WPC10145 | Phaseolus lunatus | USA | – | N | N | ||
| 2 | P. citricola | WPC1321 | Rubus sp. | California, USA | – | – | – | |
| 2a | P. citrophthora | CBS950.87 | Citrus sp. | California, USA | – | – | – | |
| 2b | P. capsici | WPC1319 | Green bell pepper | California, USA | – | – | – | |
| 3 | P. pseudosyringae | CBS111772 | Quercus robur | Germany | – | – | – | |
| 5 | P. heveae | WPC1102 | Avocado | Guatemala | – | – | – | |
| 6 | P. humicola | WPC6701 | Citrus sp. | Taiwan | – | – | – | |
| P. megasperma | NBRC32176 | White trumpet lily | Yokohama, Japan | – | – | – | ||
| 7a | P. cambivora | WPC0592 | Abies procera | USA | – | – | – | |
| * | P. europaea | CBS109049 | Quercus rhizosphere | France | – | – | – | |
| * | P. fragariae | CBS209.46 | Fragaria sp. | England | N | – | N | |
| * | P. uliginosa | CBS109054 | Quercus robur | Poland | N | – | N | |
| 7b | P. cajani | WPC3105 | Cajanus cajan | India | – | – | – | |
| P. cinnamomi | WPC2160 | Grape | South Africa | – | + | – | ||
| ZZ029 | Kiwifruit | Shaanxi, China | – | + | – | |||
| * | P. parvispora | CBS411.96 | Beaucamea sp. | Germany | – | – | – | |
| * | P. melonis | WPC6870 | Cucumber | Japan | – | – | – | |
| P. niederhauserii | CH96HE1 | Hedera helix | Chiba, Japan | N | – | N | ||
| * | P. pistaciae | CBS137185 | Pistachia vera | Iran | N | – | N | |
| P. sojae | WPC7358 | Soybean | NA | – | – | – | ||
| P. vignae | Ph-9 | Adzuki bean | Hokkaido, Japan | – | – | – | ||
| 8a | P. cryptogea | WPC1088 | Callistephus chinensis | California, USA | – | – | – | |
| * | P. drechsleri | WPC1087 | Beet | California, USA | – | – | – | |
| P. medicaginis | WPC10138 | Medicago sativa | California, USA | N | N | – | ||
| P. sansomeana | WPC3163 | Silene latifolia subsp. alba | New York, USA | N | N | – | ||
| 8b | P. brassicae | CBS179.87 | Brassica oleracea | Netherlands | – | – | – | |
| P. primulae | CBS620.97 | Primula acaulis | Germany | N | N | – | ||
| 8c | P. syringae | MAFF645010 | Malus pumila | Aomori, Japan | – | – | – | |
| * | P. foliorum | WPC10974 | Azalea | Tennessee, USA | N | N | – | |
| P. hibernalis | CBS114104 | Citrus sinensis | Australia | – | – | – | ||
| * | P. lateralis | WPC3361 | Chamaecyparis lawsoniana | Oregon, USA | – | – | + | |
| * | P. ramorum | CBS101553 | Rhododendron catawbiense | Germany | – | – | – | |
| 9 | * | P. insolita | WPC6195 | Soil | Taiwan | – | – | – |
| 10 | P. kernoviae | NA | NA | NA | – | – | – | |
| Pythium vexans | MS6-10-8V | Forest soil | Gifu, Japan | – | – | – | ||
| Py. helicoides | NBRC100107 | Rose | Gifu, Japan | – | – | – | ||
| Py. irregulare | CBS263.30 | Nicotiana tabacum | USA | – | – | – | ||
| Fusarium oxysporum | MAFF727510 | NA | NA | – | – | – | ||
| Rhizoctonia solani | S02 | NA | NA | – | – | – | ||
| Verticillium alboatrum | Vaal 130308 | NA | NA | – | – | – | ||
| 注:*:模式菌株;#:分子系统发育进化枝[14];& :CBS:荷兰菌种保藏中心,荷兰;WPC:世界疫霉遗传资源保藏中心,美国;MAFF:日本农业部,日本;NBRC:NITE生物资源中心,日本;$:3个菌种均以Yph1F_mod2为正向引物;NA:信息不详;+:扩增;-:无扩增;N:未检测.
Note: *: Type isolate of species; #:Molecular phylogenetic clade according to Martin et al.[14]; & : International identification abbreviations; CBS: Centraalbureau fur Schimmelcultures, The Netherlands; WPC: World Phytophthora Genetic Resource Collection, USA; MAFF: Ministry of Agriculture, Forestry and Fisheries, Japan; NBRC: NITE Biological Resource Centre, Japan; $: Yph1F_mod2 was used as the forward primer for all 3 species; NA: Not available; +: Amplified; -: Not amplified; N: Not tested. | ||||||||
于2017年4月在陕西省眉县收集根际土壤样品20份;2017年6月在陕西省周至县和眉县分别收集土壤样品25份和5份;同年10月在周至县和眉县各收集24份和15份土壤样品;2018年5月在周至县和眉县各收集18份土壤样品;同年10月分别收集周至县和眉县土壤样品23份和18份。每份土壤样本均从表层土壤10 cm以下取土约500 g,储存于5 ℃保温箱中。使用Field Scout pH 400 m实地测量土壤pH值。土壤质地及结构则以International Society of Soil Science[15]为标准进行判定。
1.2 方法 1.2.1 菌丝及土壤DNA的提取供试疫霉菌采用V8液体培养基进行培养,挑取适量的菌丝,采用改良的磁珠提取法[16]提取菌丝DNA。土壤DNA的提取与纯化方法与菌丝DNA的提取方法相同。具体操作步骤为:2 mL的离心管中加入0.2 g土壤和0.2 g直径为1 mm的磁性玻璃珠,并加入250 μL的提取液(100 mmol/L pH为9.0的Tris-HCl,40 mmol/L的EDTA,10% SDS,0.8%脱脂乳),4 000 r/min旋涡振荡1 min,然后加入150 mL氯化苄并振荡混匀。60 ℃放置15 min后,在悬浮液中加入3 mol/L醋酸钠150 μL,轻微振荡混合,于冰上放置15 min后,15 000 r/min离心10 min后将上清液转移到新的1.5 mL离心管内。按照MagExtractor Plant Genome Kit的操作说明纯化土壤DNA,将提取所得的DNA保存于-20 ℃备用。
1.2.2 探针设计与PCR扩增采用Bi等[17]基于Ypt1基因序列设计恶疫霉、樟疫霉和雪松疫霉的特异性引物(表 2)。从NCBI数据库中获取50种疫霉菌和3种腐霉菌的Ypt1基因序列(表 3),利用软件BioEdit V7.0.0 (Ionis Pharmaceuticals,Dublin,Ireland)进行序列分析,使用软件Beacon Designer V7.51 (PREMIER Biosoft International,USA)设计TaqMan探针(表 2)。分别用HEX、FAM和ROX荧光染料标记恶疫霉、樟疫霉和雪松疫霉探针。
| 菌种 Species |
基因 Gene |
引物/探针 Primer/Probe |
引物类型 Primer type |
序列 Sequence (5′→3′) |
Tm (℃) |
扩增片段长度 Amplified fragment length (bp) |
参考文献 References |
| Phytophthora cactorum | Ypt1 | Yph1F_mod2 | Forward | CGACCATKGGTGTGGACTTTG | 54 | 112 | [17] |
| Yph_cac_R5 | Reverse | CTGGGCACAACCGCAATAAAGA | 55 | [17] | |||
| cac_ Ypro | (HEX)-TCTGCGCTAGGCGACCTT TGCGAGCT-(BHQ1) |
78.9 | This study | ||||
| P. cinnamomi | Ypt1 | Yph1F_mod2 | Forward | CGACCATKGGTGTGGACTTTG | 54 | 229 | [17] |
| Yph_cin_R1 | Reverse | CACTACAGCAGCACCATTTATTT | 52 | [17] | |||
| cin_Ypro | (FAM)-CTCCACGAACAGCTTCCA ACAGGCGAA-(BHQ1) |
75.1 | This study | ||||
| P. lateralis | Ypt1 | Yph1F_mod2 | Forward | CGACCATKGGTGTGGACTTTG | 54 | 189 | [17] |
| Yph_lat_R2 | Reverse | GGAAAAAATCTCCCGCAGACA | 52 | [17] | |||
| lat_Ypro | (ROX)-CGTACGGATTTTCTAAAT -(MGB-X) |
47.7 | This study | ||||
| 注:Tm值的计算参照文献[18].
Note: The Tm was calculated according to reference [18]. | |||||||
| 菌种 Species |
分离株 Isolates |
登录号 Accession No. |
| Phytophthora alni subsp. alni | SCRP2 | DQ162953 |
| P. boehmeriae | SCRP23 | DQ270324 |
| P. cactorum | IMI296524 | DQ162960 |
| CH03OKTYPE1 | HQ850000 | |
| P. cambivora | SCRP82 | DQ162956 |
| P. capsici | IMI352321 | DQ162972 |
| P. cinnamomi | CBS270.55 | DQ162959 |
| SCRP118 | DQ270317 | |
| P. citricola | SCRP143 | DQ162971 |
| P. citrophthora | IMI332632 | DQ162973 |
| P. clandestina | CBS347.86 | HQ850002 |
| P. cryptogea | IMI045168 | DQ162987 |
| P. drechsleri | ATCC46724 | DQ162989 |
| P. erythroseptica | SCRP240 | DQ162988 |
| P. europaea | SCRP622 | DQ162952 |
| P. foliorum | CBS121655 | KJ755148 |
| P. fragariae | SCRP245 | DQ162950 |
| P. hedraiandra | CBS111725 | HQ850003 |
| P. hibernalis | JKI906242 | KJ755160 |
| P. idaei | CBS971.95 | HQ850004 |
| P. ilicis | SCRP379 | DQ162963 |
| P. infestans | CBS368.51 | HQ850005 |
| P. insolita | IMI288805 | DQ162974 |
| P. inundata | SCRP649 | DQ162985 |
| P. ipomoeae | CBS122203 | HQ850006 |
| P. iranica | CBS374.72 | HQ850007 |
| P. katsurae | SCRP388 | DQ162980 |
| P. kernoviae | SCRP722 | DQ162975 |
| P. lateralis | IMI040503 | DQ162991 |
| US1-2 | KM975317 | |
| P. medicaginis | SCRP407 | DQ162990 |
| P. megakarya | P8517 | HQ850008 |
| P. megasperma | IMI133317 | DQ162986 |
| P. melonis | PMNJHG1 | EF649778 |
| P. mirabilis | CBS678.85 | HQ850009 |
| P. multivesiculata | CBS545.96 | HQ850010 |
| P. nemorosa | SCRP910 | DQ162965 |
| P. nicotianae | CH02FPK3 | HQ849999 |
| P. obscura | BBA 2/94-II-B | KJ755158 |
| P. palmivora | IPPc3 | HQ850011 |
| P. phaseoli | CBS120373 | HQ850012 |
| P. pistaciae | IMI386658 | DQ162957 |
| P. pseudosyringae | SCRP734 | DQ162967 |
| P. pseudotsugae | CBS444.84 | HQ850013 |
| P. psychrophila | SCRP630 | DQ162964 |
| P. quercina | SCRP550 | DQ162979 |
| P. ramorum | SCRP911 | DQ162992 |
| P. sansomeana | - | FJ966876 |
| P. sojae | SCRP555 | DQ162958 |
| P. syringae | CBS110161 | KF882681 |
| P. tentaculata | C45 | HQ850014 |
| P. uliginosa | BBA 12/02-1 | KJ755139 |
| P. vignae | BBA P3071 | KJ755145 |
| Pythium oedochilum | CBS597.68 | HQ850015 |
| P. helicoides | TCG3 | HQ850016 |
| P. ostracodes | CBS768.73 | HQ850017 |
| 注:-:无数据. Note: -: No data. | ||
PCR反应体系(25 μL):正、反向引物(25 μmol/L)各1 μL,HotStart Taq DNA Polymerase (5 U/μL) 0.125 μL,dNTP混合物(2.5 mmol/L) 2 μL,1×PCR Buffer 2.5 μL,Bovine serum albumin (BSA) (4 mg/mL) 2.5 μL,DNA模板2 μL,ddH2O 13.875 μL。PCR反应条件:95 ℃ 3 min;94 ℃ 30 s,62 ℃ 45 s,72 ℃ 30 s,共40个循环;72 ℃ 10 min。PCR扩增产物通过2 %琼脂糖凝胶电泳检测。
实时荧光定量PCR反应体系(20 μL):DNA样品2 μL,1×Premix Taq 10 μL,BSA (4 mg/mL) 2 μL,MgCl2 (25 mmol/L) 1.5 μL,分别加入对应的引物和探针:恶疫霉正向引物(20 μmol/L) 0.2 μL,反向引物(40 μmol/L) 0.1 μL,探针(40 μmol/L) 0.1 μL;樟疫霉正向引物(20 μmol/L) 1.6 μL,反向引物(80 μmol/L) 0.4 μL,探针(40 μmol/L) 0.4 μL;雪松疫霉正向引物(20 μmol/L) 0.8 μL,反向引物(40 μmol/L) 0.45 μL,探针(40 μmol/L) 0.45 μL。反应条件:95 ℃ 10 s;95 ℃ 10 s,60 ℃ 35 s,共40个循环。在每个PCR循环退火阶段监测荧光信号,通过Bio-Rad CFX Manager软件获取Ct值。
2 结果与分析 2.1 分子标记的特异性检测由于引物的特异性在Bi等[17]建立的常规PCR检测体系中已证实,本研究仅在实时荧光定量PCR实验中对引物及探针进行了特异性检验。选取了2个恶疫霉的菌株及Clade1[19]中的11个近缘种,2个樟疫霉的菌株及Clade7[19]中的11个近缘种,1个雪松疫霉的菌株及Clade8[19]中的13个近缘种,此外还有9个疫霉属其他进化分支菌种及6种常见土传性病原菌(包括腐霉菌、镰刀霉菌、立枯丝核菌及轮枝菌) (表 1)。结果显示,HEX的荧光信号只在含有恶疫霉DNA的样本中被检测到,FAM的荧光信号只在含有樟疫霉DNA的样本中被检测到,而ROX的荧光信号只在含有雪松疫霉DNA的样本中被检测到,在其他非目标菌株样本中均无荧光信号。
2.2 实时荧光定量PCR反应体系的优化为了优化恶疫霉、樟疫霉和雪松疫霉实时荧光定量PCR体系,共设计了21种不同浓度的引物和探针组合进行测试。结果显示,恶疫霉在0.2 μmol/L的引物和探针浓度下扩增效率最佳;樟疫霉在1.6 μmol/L的引物与0.8 μmol/L的探针浓度下扩增效率最佳;雪松疫霉在0.8 μmol/L的引物与探针浓度下扩增效率最佳。退火温度分别测试了58、60、62和65 ℃,其中退火温度为60 ℃时恶疫霉、樟疫霉和雪松疫霉的引物和探针的特异性和扩增效率都最佳。在多重实时荧光定量PCR中镁离子的浓度也是一个重要因素,通过在反应体系中加入不同浓度的镁离子进行测试,得出加入1.5 mmol/L的氯化镁时3种疫霉反应体系的扩增效率均最优。
2.3 多重实时荧光定量PCR的敏感度测试将恶疫霉CH989A11、樟疫霉WPC2160和雪松疫霉WPC3361的DNA等浓度混合,按10倍梯度稀释成6个浓度,即(1.0×103)-(1.0×10-2) μg/L再进行PCR检测。结果表明,3对特异性引物及探针在所建立的实时荧光定量PCR体系中对(1.0×103)-(1.0×10-1) μg/L浓度的目标菌种DNA均有扩增,恶疫霉、樟疫霉和雪松疫霉在多重反应体系中的灵敏度均为100 fg。
以不同稀释倍数的DNA浓度对数值为横坐标,反应循环数(Ct值)为纵坐标,构建标准曲线。标准曲线如图 1所示,标准曲线显示目标菌种DNA浓度和Ct值呈线性关系,恶疫霉的相关系数为0.996,扩增效率为104.84%;樟疫霉的相关系数为0.998 9,扩增效率为102.43%;雪松疫霉的相关系数为0.998 8,扩增效率为112.09%。

|
| 图 1 恶疫霉(A)、樟疫霉(B)和雪松疫霉(C)的敏感度检测、标准曲线、相关系数及扩增效率 Figure 1 Sensitivity tests, standard curves, correlation coefficients and amplification efficiencies assessed for Phytophthora cactorum (A), P. cinnamomi (B) and P. lateralis (C) 注:多重实时PCR扩增前将3个菌种的DNA等浓度混合在一起,连续稀释得到最终浓度为(1.0×103) pg/μL-(1.0×10–1) pg/μL的DNA样品. Note: Total DNA from the three species was mixed together and serially diluted to yield final concentrations ranging from 1.0×103 pg/μL to 1.0×10–1 pg/μL before multiplex real-time PCR amplification. |
|
|
在陕西省周至县2017年6、10月和2018年10月采集的土壤样品中同时检测出恶疫霉、樟疫霉和雪松疫霉3种疫霉;在2018年5月的样品中仅检测出恶疫霉。在眉县2017年4月的样品中只检测出恶疫霉,而在2017年6月和10月的样品中检测出恶疫霉和雪松疫霉2种疫霉;在2018年5月采集的样品中同时检测出3种疫霉菌,而在2018年10月的样品中检测出恶疫霉和樟疫霉2种疫霉(表 4)。
| 地区 Prefecture |
取样日期 Sampling date |
感染疫霉样本数/样本采集数 Samples with Phytophthora/ Samples collected |
土壤质地 Soil texture |
pH | 症状 Symptoms in field |
检测到的病原菌 Detected pathogens |
| 周至Zhouzhi | Jun-17 | 10/25 | L, SL, CL | 5.9-6.9 | Phytophthora rot, leaves yellowing |
P. cactorum, P. cinnamomi, P. lateralis |
| Oct-17 | 8/24 | N | 6.2-6.8 | Leaves yellowing | P. cactorum, P. cinnamomi, P. lateralis | |
| May-18 | 1/18 | N | 6.3-7.1 | Leaves yellowing, gray mold | P. cactorum | |
| Oct-18 | 9/23 | N | N | Root rot, leaves yellowing | P. cactorum, P. cinnamomi, P. lateralis | |
| 眉县Meixian | Apr-17 | 3/20 | L, SL, CL | 7.1-7.6 | Bacterial canker, leaves yellowing |
P. cactorum |
| Jun-17 | 1/5 | N | 6.8-7.7 | Root rot, leaves yellowing | P. cactorum, P. lateralis | |
| Oct-17 | 4/15 | N | 7.0-7.5 | Bacterial canker, leaves yellowing |
P. cactorum, P. lateralis | |
| May-18 | 7/18 | N | N | Bacterial canker | P. cactorum, P. cinnamomi, P. lateralis | |
| Oct-18 | 4/18 | N | N | Leaves yellowing, root rot | P. cactorum, P. cinnamomi | |
| 注:L:壤土;SL:沙质土;CL:黏土;N:没有检测. Note: L: Loam; SL: Sandy loam; CL: Clay loam; N: Not tested. | ||||||
周至县猕猴桃种植区恶疫霉、樟疫霉和雪松疫霉的分布为:共采集90份土壤,其中24份土壤中检测到恶疫霉,Ypt1基因的浓度范围为15-89 pg/g土壤,其中2017年6月检测出的样品数最多(图 2A);6份土壤中检测到樟疫霉,Ypt1基因的浓度范围为15-43 pg/g土壤,其中2018年10月检测出的样品数最多(图 2B);6份土壤中检测到雪松疫霉,Ypt1基因的浓度范围为25-56 pg/g土壤,2017年6、10月和2018年10月均有2个样品被检测出(图 2C)。

|
| 图 2 陕西省周至县恶疫霉(A)、樟疫霉(B)及雪松疫霉(C)的分布及Ypt1基因含量 Figure 2 Distributions and the Ypt1 amounts of Phytophthora cactorum (A), P. cinnamomi (B) and P. lateralis (C) in Zhouzhi Prefecture of Shaanxi Province |
|
|
眉县猕猴桃种植区恶疫霉、樟疫霉和雪松疫霉的分布为:共采集76份土壤,14份土壤中检测到恶疫霉,Ypt1基因的浓度范围为10-67 pg/g土壤,其中2018年5月检测出的样品数最多(图 3A);3份土壤中检测到樟疫霉,Ypt1基因的浓度范围为17-151 pg/g土壤,其中2018年5月检测出的样品数最多(图 3B);7份土壤中检测到雪松疫霉,Ypt1基因的浓度范围为9-59 pg/g土壤,2017年10月和2018年5月均有3个样品被检测到(图 3C)。
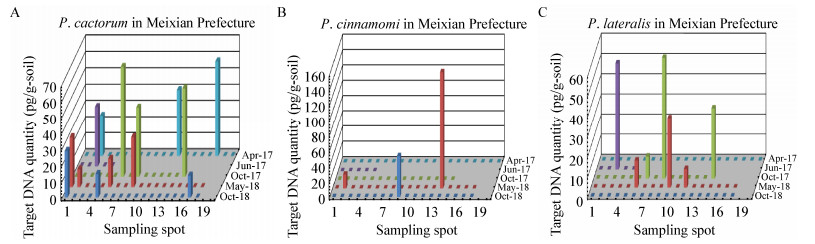
|
| 图 3 陕西省眉县恶疫霉(A)、樟疫霉(B)及雪松疫霉(C)的分布及Ypt1基因含量 Figure 3 Distributions and the Ypt1 amounts of Phytophthora cactorum (A), P. cinnamomi (B) and P. lateralis (C) in Meixian Prefecture of Shaanxi Province |
|
|
在本研究中我们建立了可以同时鉴定和定量3种猕猴桃根腐病致病疫霉(恶疫霉、樟疫霉和雪松疫霉)的方法。在设计雪松疫霉的探针时,由于可设计区域有限且(G+C)mol%含量低,无法满足TaqMan探针的设计要求,因此使用了TaqMan-MGB探针。MGB既可提高探针的Tm值[16],又可以保证其特异性,从而达到实现多个DNA片段同时扩增的目的。
引物及探针特异性对PCR的检测至关重要。在之前的研究中Li等共检测了9个DNA位点,包括rDNA的ITS区域、28S rRNA基因、核糖体蛋白60S的L10基因、β微管蛋白基因、延长因子1-α基因、烯醇酶基因、热休克蛋白90基因、tigA融合蛋白基因和Ypt1基因[15]。为了找出可以区别种间差异的特异性引物,对恶疫霉和其近缘种[20]的不同DNA序列进行分析,发现只有Ypt1基因适合恶疫霉特异性引物的设计[14]。
根据Henegariu等[21]关于多重PCR影响因素的研究结论,本实验共优化了5个因素:退火温度、退火时间、探针和引物浓度及镁离子浓度。此外,为了平衡Ypt1基因同时扩增的竞争性反应,本研究调整了不同种特异性引物和探针的浓度,最大程度地提高了多重实时定量PCR检测敏感度。
对于樟疫霉和雪松疫霉的分子检测,虽然已经报道了一些特异性标记[8, 20, 22],但大多数研究缺少对近缘种的检验。Kunadiya等提出,特异性分子标记在使用前应该至少对同一分支中亲缘关系密切的物种进行确认[19],并测试了8组已报道樟疫霉特异性引物,发现仅有3组是真正对樟疫霉具有特异性的。
rDNA基因为多拷贝基因,而Ypt1基因是单拷贝基因。虽然Ypt1基因的敏感度低于rDNA基因,但在敏感度测试中恶疫霉、樟疫霉和雪松疫霉均可达到100 fg,可以满足环境样品定量检测的要求。此外,单拷贝基因更有利于对单个繁殖体进行准确定量分析[23]。
Bi等[17]针对恶疫霉、樟疫霉及雪松疫霉建立的多重PCR检测方法为常规PCR检测技术,虽然引物特异性强,但是敏感度较低,因此难以检测出低浓度的样品。本研究在Bi等的基础上建立多重定量检测方法,敏感度提高25倍,不仅检测出更多的低浓度感染样品,还可以实现靶基因浓度的准确定量。
通过对陕西省猕猴桃栽培区的调查发现,虽然只有少数果园有疫霉病害史,但在一些没有疫霉病害史的果园中也检测到恶疫霉,这表明该地区猕猴桃果园可能不同程度地受到恶疫霉的侵染。从恶疫霉、樟疫霉和雪松疫霉在周至县和眉县不同时期Ypt1基因含量柱状图来看,致病菌数量与季节有一定联系。与4、5月份采集的样品相比,6、10月份的土壤样品中3种疫霉的DNA含量较高,这有可能与温度和降水量有关。
疫霉菌病害引起的症状与其他病原引起的症状非常相似,但防治方法有所不同。因此,正确的诊断对保护农田生态环境具有重要意义。本文介绍的方法对猕猴桃疫霉病害的鉴定及防治具有重要的科学意义和应用价值。
致谢: 感谢日本岐阜大学景山幸二教授提供了很多重要疫霉菌种,并且感谢周至和眉县植物保护站提供猕猴桃种植区土壤样本的信息。| [1] |
O'Brien PA, Williams N, Hardy GES. Detecting Phytophthora[J]. Critical Reviews in Microbiology, 2009, 35(3): 169-181. DOI:10.1080/10408410902831518 |
| [2] |
Akilli S, Serce CU, Katırcıoğlu YZ, et al. Involvement of Phytophthora citrophthora in kiwifruit decline in Turkey[J]. Journal of Phytopathology, 2011, 159(7/8): 579-581. |
| [3] |
Kurbetli İ, Ozan S. Occurrence of Phytophthora root and stem rot of kiwifruit in Turkey[J]. Journal of Phytopathology, 2013, 161(11/12): 887-889. |
| [4] |
Lee YH, Jee HJ, Cha KH, et al. Occurrence of Phytophthora root rot on kiwifruit in Korea[J]. The Plant Pathology Journal, 2001, 17(3): 154-158. |
| [5] |
Huang YJ, Qi PK. Studies on the cause of root rot of kiwifruit in Guangdong Province[J]. Journal of South China Agricultural University, 1998, 19(4): 19-22, 35. (in Chinese) 黄亚军, 戚佩坤. 广东省猕猴桃根腐病病因研究[J]. 华南农业大学学报, 1998, 19(4): 19-22, 35. |
| [6] |
Ferguson AR. Kiwifruit in the world – 2014[J]. Acta Horticulturae, 2015. DOI:10.17660/ActaHortic.2015.1096.1 |
| [7] |
Wang RX, Cao ZJ. Diagnosis of blight of Yangtao actinidia in Shaanxi Province[J]. Acta Universitatis Agriculturalis Boreali-Occidentalis, 1999, 27(4): 75-78. (in Chinese) 王汝贤, 曹张军. 陕西省猕猴桃疫霉病的诊断及病原鉴定[J]. 西北农业大学学报, 1999, 27(4): 75-78. DOI:10.3321/j.issn:1671-9387.1999.04.017 |
| [8] |
Engelbrecht J, Duong TA, van den Berg N. Development of a nested quantitative real-time PCR for detecting Phytophthora cinnamomi in Persea americana rootstocks[J]. Plant Disease, 2013, 97(8): 1012-1017. DOI:10.1094/PDIS-11-12-1007-RE |
| [9] |
Schena L, Nigro F, Ippolito A, et al. Real-time quantitative PCR: a new technology to detect and study phytopathogenic and antagonistic fungi[J]. European Journal of Plant Pathology, 2004, 110(9): 893-908. DOI:10.1007/s10658-004-4842-9 |
| [10] |
Chen Y, Roxby R. Characterization of a Phytophthora infestans gene involved in vesicle transport[J]. Gene, 1996, 181(1/2): 89-94. |
| [11] |
Wang Y, Meng JC. Rapid detection of Phytophthora nicotianae in infected tobacco tissues and soil samples based on its Ypt1 gene[J]. Journal of Phytopathology, 2010, 158(1): 1-7. DOI:10.1111/j.1439-0434.2009.01548.x |
| [12] |
Hansen ZR, Knaus BJ, Tabima JF, et al. Loop-mediated isothermal amplification for detection of the tomato and potato late blight pathogen, Phytophthora infestans[J]. Journal of Applied Microbiology, 2016, 120(4): 1010-1020. DOI:10.1111/jam.13079 |
| [13] |
Alejandro Rojas J, Miles TD, Coffey MD, et al. Development and application of qPCR and RPA genus- and species-specific detection of Phytophthora sojae and P. sansomeana root rot pathogens of soybean[J]. Plant Disease, 2017, 101(7): 1171-1181. DOI:10.1094/PDIS-09-16-1225-RE |
| [14] |
Martin FN, Blair JE, Coffey MD. A combined mitochondrial and nuclear multilocus phylogeny of the genus Phytophthora[J]. Fungal Genetics and Biology, 2014, 66: 19-32. DOI:10.1016/j.fgb.2014.02.006 |
| [15] |
Li MZ, Asano T, Suga H, et al. A multiplex PCR for the detection of Phytophthora nicotianae and P. cactorum, and a survey of their occurrence in strawberry production areas of Japan[J]. Plant Disease, 2011, 95(10): 1270-1278. DOI:10.1094/PDIS-01-11-0076 |
| [16] |
Kutyavin IV, Afonina IA, Mills A, et al. 3'-minor groove binder-DNA probes increase sequence specificity at PCR extension temperatures[J]. Nucleic Acids Research, 2000, 28(2): 655-661. DOI:10.1093/nar/28.2.655 |
| [17] |
Bi XQ, Hieno A, Otsubo K, et al. A multiplex PCR assay for three pathogenic Phytophthora species related to kiwifruit diseases in China[J]. Journal of General Plant Pathology, 2019, 85(1): 12-22. |
| [18] |
Panjkovich A, Norambuena T, Melo F. dnaMATE: a consensus melting temperature prediction server for short DNA sequences[J]. Nucleic Acids Research, 2005, 33(S2): W570-W572. |
| [19] |
Blair JE, Coffey MD, Park SY, et al. A multi-locus phylogeny for Phytophthora utilizing markers derived from complete genome sequences[J]. Fungal Genetics and Biology, 2008, 45(3): 266-277. DOI:10.1016/j.fgb.2007.10.010 |
| [20] |
Winton LM, Hansen EM. Molecular diagnosis of Phytophthora lateralis in trees, water, and foliage baits using multiplex polymerase chain reaction[J]. Forest Pathology, 2001, 31(5): 275-283. DOI:10.1046/j.1439-0329.2001.00251.x |
| [21] |
Henegariu O, Heerema NA, Dlouhy SR, et al. Multiplex PCR: critical parameters and step-by-step protocol[J]. BioTechniques, 1997, 23(3): 504-511. DOI:10.2144/97233rr01 |
| [22] |
O'Brien PA. PCR primers for specific detection of Phytophthora cinnamomi[J]. Australasian Plant Pathology, 2008, 37(1): 69-71. |
| [23] |
Kageyama K, Komatsu T, Suga H. Refined PCR protocol for detection of plant pathogens in soil[J]. Journal of General Plant Pathology, 2003, 69(3): 153-160. |
 2020, Vol. 47
2020, Vol. 47