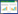扩展功能
文章信息
- 王艳, 舒健, 张宸, 李铮
- WANG Yan, SHU Jian, ZHANG Chen, LI Zheng
- 肠道微生物蛋白糖基化修饰的研究进展
- New progress of protein glycosylation modification in gut microbes
- 微生物学通报, 2020, 47(1): 253-262
- Microbiology China, 2020, 47(1): 253-262
- DOI: 10.13344/j.microbiol.china.190309
-
文章历史
- 收稿日期: 2019-04-11
- 接受日期: 2019-08-16
- 网络首发日期: 2019-09-11
肠道生态系统作为人体内最重要、最复杂的生态系统,包含并滋养了80%以上的微生物,该系统不仅是调节人体健康的发动机,更是连接膳食和宿主间的重要桥梁。正常生理条件下,肠道微生物与宿主之间存在着互利关系,后者为前者提供栖息地和营养物质,而前者则通过促进营养物质的代谢、协助消化碳水化合物以及合成宿主生长所需的维生素来调节肠道发育代谢并维持免疫稳态[1]。
在由肠道微生物所导致的各种病变之中,细菌鞭毛蛋白、菌毛蛋白以及一些黏附素的糖链对于细菌粘附、识别靶细胞相应受体至关重要。随着研究人员对传统的微生物蛋白质组和基因组学研究陷入瓶颈,研究者们亟需寻求一个新的突破口。糖组学(glycomics)是研究生物体内全部的糖链结构、表达、功能以及与疾病之间关系的一个新兴研究领域[2]。新兴的糖组学对于细菌的聚糖结构和糖基化途径及其重要功能的研究无疑是一个绝佳的切入点,微生物糖基化相关研究的重要性日益增强。近年来,国内外对于微生物糖基化的研究日益增多,尤其在关注肠道内细菌的糖基化途径和它的多种生理学功能中更是如此。人体肠道中的大部分细菌蛋白拥有糖基化修饰。原核生物糖基化蛋白的合成有以下两种方式:(1)糖基转移酶将寡糖残基依次连到目标蛋白上,例如有些鞭毛蛋白利用这种方式进行糖基化修饰;(2)寡糖链在脂质载体上完成组装,再由糖基转移酶将寡糖链转移并连接到相应目的蛋白上[3],例如空肠弯曲杆菌(Campylobacter jejuni,C. jejuni)、革兰氏阳性菌的S层蛋白均以该糖基化机制进行糖基化。
本文通过讨论并分析肠道微生物的组成、作用以及糖基化在肠道微生物中的研究进展,以期发现及归纳肠道微生物糖基化对宿主功能或活动的调节方式,为肠道微生物糖基化的研究及相关疾病的诊断、治疗和疫苗开发提供新的研究思路。
1 肠道微生物与糖基化 1.1 肠道微生物的组成与功能肠道微生物群作为已知存在的密度最高的微生物群落之一,是定殖于胃肠道中所有微生物的集合。人体肠道内的微生物超过99%都是细菌[4],其组成相对稳定,但由于个体基因背景、性别、饮食习惯、生活方式的差异以及滥用抗生素等因素影响,深刻改变了菌群的组成[5-6]。例如食用高脂肪食物会增加肠道中拟杆菌属的含量,而食用富含纤维的食物会增加普氏菌属的数量[7]。肠道菌群的失调会引起一些疾病,如2型糖尿病、肥胖症、高血压、肠道疾病[如炎性肠病(inflammatory bowel disease,IBD)、结肠直肠癌和肠易激综合症]等[8-9]。乳酸杆菌、双歧杆菌等益生菌能协助食物消化,并产生对维持肠上皮细胞和免疫细胞稳态起着至关重要作用的短链脂肪酸,此类脂肪酸还能有效抑制有害菌的生长;与此同时,机体中还存在着能产生有毒物质破坏肠道微环境或者直接影响免疫系统功能的致病菌,然而肠道中存在的益生菌与致病菌之间的界定并非绝对,某些条件致病菌便可能同时属于益生菌与致病菌,在正常情况下不会引起任何病变,但若机体稳态被破坏,条件致病菌便会导致极其严重的疾病。大肠杆菌便是最典型的条件致病菌,在一般情况下稳定存在,但如果异常无限增殖,或者从肠道转移到身体其他部位,就会导致一系列严重疾病例如败血症。脆弱拟杆菌是另一种典型的条件致病菌,若该细菌从肠道移位到腹膜,因受刺激所产生的调节免疫反应的抗炎细胞因子荚膜多糖(capsular polysaccharide,CPS)会引起明显的炎症并形成脓肿[10],虽然脓肿会限制疾病的传播,但若不及时治疗,就会造成细菌的进一步感染。
肠道内的致病菌大多通过自身表面物质(主要是菌毛或鞭毛)的糖链来识别并粘附宿主细胞相应受体,进而发挥毒力作用或引起免疫表达。例如结核分枝杆菌(Mycobacterium tuberculosis)黏附蛋白LpqH在苏氨酸残基上被糖基化修饰,从而识别宿主细胞表面的甘露糖受体,最终加速单核细胞将结核分枝杆菌吞噬[11]。此外,金黄色葡萄球菌富丝氨酸重复蛋白(serine-rich repeat proteins,SRRP)中的丝氨酸被糖基化修饰过后能促进其识别宿主细胞表面受体,最终达到侵染宿主的目的[12]。微生物细胞除了能合成细菌细胞壁的肽聚糖、脂多糖(lipopolysaccharides,LPS)等结构性多糖以外,还能合成与人体正常细胞表面糖链结构类似的糖链,以干扰免疫系统识别自身,进而逃避免疫系统的攻击并维持自身在宿主体内的持续存在。细菌不仅能通过其表面物质对宿主产生影响,更能通过菌体分泌的糖苷酶和糖基转移酶(glycosyltransferases,GT)来影响宿主细胞。例如中间型链球菌(Str. intermediusgroup)分泌的多底物糖苷酶A (multisubstrate glycosidase A,MsgA)能促进人体内α-1抗胰蛋白酶(α-1-antitrypsin,α-1-AT)的降解[13],进而使得机体正常细胞和器官受到损伤,最终打破机体内环境的平衡。
1.2 微生物糖基化糖基化修饰是最常见的蛋白质翻译后修饰现象之一[14]。很久以前人们普遍认为糖基化只存在于真核生物中,直到21世纪初细菌和古细菌的蛋白质糖基化修饰系统被发现才打破了这一观念[15],并由此意识到细菌聚糖的多样性。原核生物蛋白质糖基化发展的迟滞导致了目前只有少数肠道细菌表层结构(鞭毛蛋白、菌毛蛋白、黏附素等)的糖基化蛋白得到了充分的鉴定与研究(表 1)。
| 菌名 Bacteria |
糖蛋白 Glycoprotein |
糖蛋白结构 Glycan structure |
参考文献 References |
| Campylobacter jejuni 81-176 | FlaA | Pse5Ac7Ac, PseAm, Pse8OAc, PseAmGlnAc | [16] |
| Campylobacter coli VC167 | FlaA | Pse5Ac7Ac, PseAm, Pse/PseAm-deoxypentose, Leg5Ac7Ac, Leg5Am7Ac, Leg5AmNMe7Ac | [17] |
| Clostridium difficile 630 | FliC | HexNAc-Phosphate-methylated aspartic (398 Da) | [18] |
| Clostridium difficile Bi-1 | FliC | 204 (HexNAc), 146 (deoxyHex), 160 (methylated deoxyHex) and 192 (hept) | [19] |
| Escherichia coli | AIDA-1 | Heptose | [20] |
| TibA | Heptose | [21-22] | |
| Ag43 | Heptose | [23-24] | |
| Lactobacillus plantarum | Acm2 | Up to 3 HexNAc (GlcNAc according to sWGA lectin blot) | [23] |
| Bacteriocins | -C-GlcNAc | [25] | |
| Bacillus subtilis | Bacteriocins | -C-Glucose | [25] |
| Lactobacillus plantarum KW30 | GccF | Cys5/Cys28 and Cys12/Cys21 HexNAc, Ser18, Cys43 | [26] |
与真核生物类似,原核生物糖基化有两种常见的修饰方式:一种是聚糖与丝氨酸或苏氨酸中羟基相连的O-糖基化;一种是与特征序列Asn-X-Ser/Thr (X为除Pro以外任意的氨基酸)中天冬酰胺相连的N-糖基化[27]。此外,少数细菌如植物乳杆菌和枯草芽孢杆菌合成的细菌素上还存在聚糖与半胱氨酸巯基连接形成的S-糖基化[28]。
不同类型的蛋白糖基化修饰深刻影响着宿主的各项生命活动。例如N-糖基化在维持肠内稳态、调节肠上皮细胞反应等方面起着至关重要的作用。变形杆菌N-糖基化的鞭毛蛋白能通过与大肠黏膜表面一种高度糖基化的GPI锚定蛋白的Lypd8结构域结合并抑制其运动,进而阻止其对肠上皮细胞的侵染[29]。O-糖基化的重要功能之一是帮助细菌逃避宿主防御[21, 30]。例如结核分枝杆菌的O-甘露糖基化蛋白与宿主细胞表面的特定受体相互作用,能促进吞噬细胞将其吞噬,这一过程有利于细菌逃避宿主的杀伤作用[11, 31]。另一方面,O-甘露糖基化蛋白PstS-1还可以通过激活TLR2、TNF-α和FasL来诱导巨噬细胞的凋亡,进一步破坏人体免疫系统[32]。较为稀少的S-糖基化的关键作用是保持对酶的抗性。例如植物乳杆菌KW30分泌的GccF (glycocin F) C末端Cys43残基与N-乙酰基己糖胺相连接,这种修饰使其对糖苷酶或糖基转移酶具有一定程度的抗性[33]。
2 肠道微生物的糖基化修饰系统随着研究技术手段的不断发展,研究人员对于致病菌蛋白糖基化对人体各项生理功能的影响有了深刻了解,其重要地位也日益彰显。以下就肠道中几种常见的细菌糖基化修饰方式进行概括,以期总结并归纳出糖基化影响肠道微生物生命活动的调节方式。
2.1 C. jejuni的N-糖基化修饰系统N-糖基化修饰在肠道细菌蛋白中十分常见,这里以最具代表性的C. jejuni的蛋白糖基化修饰系统举例说明。C. jejuni是人类细菌性肠胃炎的主要病原体,可以侵入小肠和结肠黏膜,并引起腹痛、腹泻等症状[34],最严重的情况下可导致患者死亡。在发病机制研究中,C. jejuni是第一个被发现存在糖基化过程的细菌。有人发现C. jejuni的糖基化在其致病过程中起着非常重要的作用[35],其周质蛋白和少数外膜蛋白均存在N-糖基化修饰,这种修饰主要是由质膜内的活性糖苷UDP-GalNAc依次经过脱水酶PglF、氨基转移酶PglE、乙酰转移酶PglD催化生成UDP-diNAcBac,再由胞质内的糖基转移酶PglC、PglA、PglJ、PglH、PglI向其依次添加不同的单糖并分别形成寡糖前体[GalNAcα1, 4GalNAcα1, 4-(Glcβ1, 3)-GalNAcα1, 4GalNAcα1, 4GalNAcα1, 3Bac2, 4diNAcα1-PP-Und],随后经翻转酶PglK将之翻转至周质,最后再由寡糖基转移酶PglB将寡糖前体转运到底物蛋白特定序列的天冬氨酸残基上[36] (图 1)。参与以上过程的酶均由pgl基因簇编码,早在1999年,Szymanski等便在C. jejuni中发现了pgl基因簇,其中糖基转移酶(oligosaccharyltransferase,OST) PglB的发现为原核生物的N-糖基化提供了研究基础[37]。可以看出,PglB在细菌蛋白质N-糖基化过程中起着重要作用,其对底物特异性相对较低,可以协助不同种类的聚糖从脂质载体十一烯基焦磷酸(undecaprenylpyrophosphate,Und-PP)转移到底物蛋白上;另一方面,PglB还能催化折叠完全的蛋白质糖基化,这明显与真核生物中的寡糖基转移酶只能催化一些未折叠的蛋白糖基化有所不同[38]。值得注意的是,如果C6位前两个糖β1-4连接,尽管还原端第一个糖的第二位C原子被乙酰化修饰,但是不能被PglB催化,说明糖底物需同时满足以下条件:还原端第一个糖的C2位需要乙酰化修饰,并且C6位前两个糖的连接方式不能是β1-4连接[39-40]。PglB是否和真核生物中的寡糖基转移酶一样在催化位点具有相同的保守序列?研究表明,在细菌中PglB催化的N-糖基化位点为特征序列Asp-Glu-Y-Asn-X-Ser/Thr[40],这显然比真核生物的特征序列Asn-X-Ser/Thr更为保守。事实上,并不是所有的保守序列Asp-Glu-Y-Asn-X-Ser/Thr都能成为PglB的催化底物,蛋白质的高级结构也影响其催化活性,例如霍乱毒素B亚基尽管含有保守序列,但只能部分被糖基化[41]。而在另一些细菌的O-糖基化修饰中,PglL作为重要的寡糖基转移酶,其对底物的选择更加宽松,可以催化几乎全部类型的糖[42]。因此,深入了解细菌糖基化过程中的重要糖基转移酶如PglB和PglL的重要功能不失为一个新的研究思路,例如将这些特有的糖基转移酶作为治疗靶点,可以防止C. jejuni和其他细菌对宿主的感染。此外,以PglB为背景,研发还原端C2位乙酰化修饰并且C6位前两个糖的连接方式不是β1-4连接的糖链相关的糖蛋白疫苗。
|
| 图 1 C. jejuni糖基化增强蛋白的稳定性 Figure 1 C. jejuni glycosylation promotes protein stability |
|
|
除了N-糖基化修饰外,细菌中还普遍存在O-糖基化修饰,该糖基化修饰主要存在于鞭毛、菌毛、黏附素等。细菌O-糖基化根据修饰过程中需要的糖基转移酶分为两类:OTase依赖型和OTase非依赖型[43]。OTase依赖型糖基化通过寡糖基转移酶将寡糖完整转移至受体蛋白上,例如B. fragilis通过此方式完成O-糖基化。OTase非依赖型糖基化利用糖基转移酶依次将单糖添加至目标蛋白上,多数鞭毛和黏附素都是利用此种途径O-糖基化。
2.2.1 鞭毛和黏附素的O-糖基化C. jejuni NCTC11168的鞭毛糖基化蛋白是迄今为止被鉴定出的糖基化程度最高的蛋白质之一。其主要糖链结构为Pseudaminic acid [5, 7-diacetamido-3, 5, 7, 9-tetradeoxy-L-glycero-L-manno-nonulosonic acid,(Pse5Ac7Ac)]或Legionaminic acid (Leg)[44]。有研究表明,C. jejuni NCTC11168的鞭毛蛋白除了Pse5Am7AcPse之外,还存在Pse的二甲基甘油酸衍生物[16-17]。此外,C. jejuni NCTC11168主要外膜蛋白MOMP也存在O-糖基化修饰,其糖链结构为Gal(β1-3)-GalNAc(β1-4)- GalNAc(β1-4)-GalNAcα1-Thr(268)[45-46]。
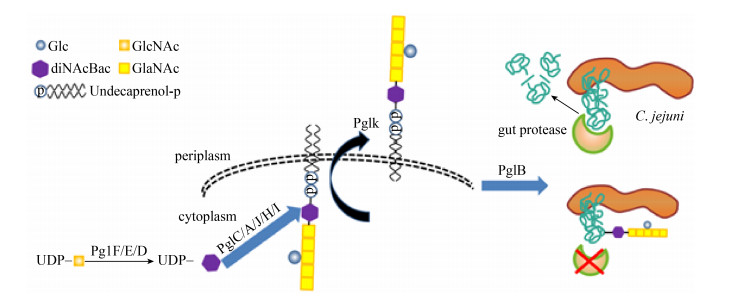
除了鞭毛蛋白和外膜蛋白外,菌体外表面的黏附素也被O-糖基化修饰。分别由弥散黏附性大肠杆菌(Diffuselyadherent E. coli,DAEC)、肠产毒性大肠杆菌(Enterotoxigenic E. coli,ETEC)、肠致病性大肠杆菌(Enteropathogenic E. coli,EPEC)所分泌的AIDA-I、TibA以及Ag43 (Antigen43)均存在O-糖基化修饰,这几种黏附素属于自转运蛋白家族[47]。自转运蛋白是一类特殊的细菌表面蛋白,其糖基化对自身毒力以及与宿主的相互作用密切相关。这类蛋白主要由3部分组成:N端可剪切的信号肽、中间的Passenger结构域和C端的β结构域,其中Passenger结构域是其重要的功能区域,它能够穿过膜锚定的桶状结构将一些毒力因子运送到细菌外表面[48]。该类蛋白最典型的代表之一AIDA-I能在细菌细胞质内被黏附素庚糖基转移酶AAH和AAH2庚糖基化[49],AAH和AAH2属于细菌自转运蛋白庚糖基转移酶BAHT (bacterial autotransporter heptosyltransferase),它们专一性修饰AIDA-I中的丝氨酸残基,在细菌细胞质中通过O-糖基化向AIDA-I上添加庚糖,该过程中所需的糖配体来自于LPS生物合成途径中的ADP-D-D-庚糖[50] (图 2)。有研究表明,存在于aidA基因上游的aah基因与tibA基因上游tibC编码的蛋白存在同源性,都与糖基化相关[22],TibC能催化TibA糖基化[51]。此外,没有专一性庚糖基转移酶参与Ag43糖基化途径,但是Ag43的糖基化途径增强了EPEC与宿主细胞的结合能力,而非糖基化的Ag43不具备这种能力[23, 47]。
|
| 图 2 AIDA-I糖基化促进DAEC对宿主细胞的粘附 Figure 2 AIDA-I glycosylation promotes adhesion of DAEC to host cells |
|
|
拟杆菌(Bacteroides)在人体肠道微生物中所占比重较大,且大多都能产生多种多样的聚糖结构,这些聚糖中含有岩藻糖,其O-糖基化特征序列为Asp-Ser/Thr-Ala/Ile/Leu/Val/Met/Thr,这显然与真核生物中N-乙酰半乳糖胺、半乳糖等与苏氨酸或丝氨酸相连的O-糖基化修饰不同[52]。拟杆菌属的B. fragilis所含有的多数糖基化蛋白均参与重要的蛋白质折叠和细胞分裂过程,与C. jejuni中pgl基因簇类似,B. fragilis中也含有编码多种寡糖基转移酶的脆弱糖基化的基因座(locus of fragilis glycosylation,lfg),其他拟杆菌成员也具有与lfg类似的区域[53]。
乳杆菌也是肠道内的高丰度益生菌群。植物乳杆菌(Lactobacillus plantarum)是乳酸菌的一种,它能有效抑制病原菌对肠道的感染、维持肠道内稳态并调节机体的免疫功能。L. plantarum WCFS1中的主要自溶素Acm2,它不仅在自溶中发挥着重要的作用,而且是菌体分裂过程中的重要酶。研究发现Acm2糖基化发生在富含丙氨酸、丝氨酸和组氨酸的N端结构域,也就是在丝氨酸或组氨酸残基上连有21个GlcNAc单体[54]。除了L. plantarum WCFS1外,鼠李糖乳杆菌(Lactobacillus rhamnosus GG)中主要分泌蛋白Msp1在Ser106-107残基处也被证明存在O-糖基化修饰,并且聚糖部分含有葡萄糖和甘露糖残基[55]。可以看出,不同种类的乳杆菌发生糖基化的蛋白有差异,体现出细菌糖基化的多样性,这无疑将成为原核生物蛋白糖基化领域内一个新的研究热点。
3 糖基化对肠道微生物功能或活动的调节 3.1 糖基化调控细菌蛋白的稳定性和活性糖基化修饰对于肠道微生物的各种生理行为都不可或缺,这种重要性在其膜表面的菌毛或鞭毛蛋白上尤为突出。有研究表明,C. jejuni可通过N-糖基化来增强自身适应性并保护自身免受肠蛋白酶的切割[56] (图 1),而其主要外膜蛋白MOMP的O-糖基化则能影响该蛋白自身的构象,这种构象的变化不仅能影响C. jejuni与组织血型抗原(blood group antigens,BgAg)的结合能力[57],还能通过表面暴露的聚糖促进与相邻细菌上的其他鞭毛聚糖自动凝聚[58],说明糖基化修饰能改变细菌蛋白的结构,进而影响自身的稳定性。细菌蛋白糖基化影响自身的稳定性还在口腔中的变形链球菌表面胶原结合蛋白Cnm上有所体现,其糖基化修饰过程的发生障碍会影响自身的稳定性[59]。此外,Mahdavi等将C. jejuni NCTC11168中flaA基因敲除,发现该突变体与BgAg的结合能力显著降低[25]。这说明了鞭毛糖基化蛋白FlaA可能影响C. jejuni与BgAg的相互结合。综上所述,蛋白糖基化修饰对于C. jejuni的鞭毛组装是不可或缺的[60],而鞭毛的组装与否能深刻影响包括C. jejuni在内的众多肠道微生物生物膜的形成以及其对宿主的侵袭能力。
多项研究[36, 55]表明,L. rhamnosus GG的蛋白Msp1的糖基化功能可能与流感嗜血杆菌粘附素HMW1的功能类似,都具有保护自身蛋白质对蛋白酶的抗性的功能;另一方面,Msp1表面的聚糖结构会降低Msp1与肠上皮细胞的相互作用,进而调节Akt信号传导的强度,这表明细菌表面蛋白的糖基化不仅能保持自身的稳定,还能影响相关区域下游分子的活性。此外,Rolain等首次发现L. plantarum WCFS1中Acm2的O-糖基化会控制该酶的活性,但是Acm2的N端结构域不发生糖基化反倒会提高它的酶活性,也许是因为O-糖基化减弱了Acm2结构域的能力,进而使得Acm2的N端结构域的功能发生障碍[61]。综上所述,包括L. rhamnosus GG和L. plantarum WCFS1在内的众多细菌蛋白的糖基化对于细菌有着一定程度的影响,进而影响其与宿主的相互作用。
3.2 糖基化对细菌粘附的重要影响菌体外表面黏附素的糖基化与其粘附能力紧密相关。如上文所述,未被糖基化修饰的TibA不能作为介质使ETEC粘附在宿主细胞上,而AIDA-I的庚糖基化最终决定了DAEC黏附在宿主细胞上(图 2)[22, 50]。有可能是糖基化影响TibA的寡聚化,未被糖基化的TibA被锁定在不适合粘附的寡聚体构像中[20],尽管这并不影响细菌自动凝聚和其生物膜的形成,但降低了ETEC对宿主的粘附能力[62]。同样地,AIDA-I被糖基化修饰后影响了自身的结构,最终影响了E. coli对宿主细胞的粘附能力[51]。值得注意的是,口腔中的副血链球菌上被糖基化修饰的菌毛蛋白Fap1也能促进其对口腔表皮细胞的粘附[63]。综上所述,细菌黏附素以及菌毛蛋白通过糖基化修饰有可能改变了自身的构象,进而增强了对宿主的粘附能力。
另有研究表明,B. fragilis的lfg区域既可以从肠上皮细胞中富集α-1, 2岩藻糖并将其作为碳源,也可以将其合并到自身糖蛋白中[64]。另一方面,lfg区域还与生物体蛋白质翻译重要基因metG连接,可以由此得出B. fragilis的O-糖基化与生物体蛋白质的合成有一定相关性。Coyne等敲除lfg区域的所有基因,不仅抑制了岩藻糖蛋白的合成,还影响了B. fragilis在小鼠肠道中的定殖[52]。说明B. fragilis的lfg区域对于自身的合成调节以及对宿主细胞的黏附有着至关重要的作用。
3.3 糖基化与免疫调节细菌表面蛋白的聚糖结构不仅能影响自身毒力、促进其对宿主细胞的粘附,还能调节宿主的免疫反应。其中最典型的例子如L. rhamnosus GG菌毛蛋白,其含有编码菌毛的SpaCBA基因簇,它编码的产物形成了以SpaA亚基为骨架,以SpaB亚基为底端,以SpaC亚基为尖端的杂聚体菌毛结构,其中含有甘露糖和岩藻糖残基[65]。L. rhamnosus GG糖基化菌毛中的聚糖结构被树突细胞表面的特异性受体树突状细胞特异性细胞间黏附分子-3-结合非整合素因子(dendritic cell-specific intercellular- adhesion-molecule-3-grabbing nonintergrin,DC-SIGN)所识别,诱导DC发生反应,进而调节炎症相关细胞因子白细胞介素10 (interleukin-10,IL-10)、IL-8、IL-6和肿瘤坏死因子(tumor necrosis factor,TNF)的表达[66-67],降低细胞的炎症反应(图 3)。另一项研究表明,嗜酸乳杆菌NCFM的表层蛋白A (SlpA)也可被DC-SIGN受体所识别,进而影响人DC和T细胞功能,最终调节IL-10和IL-12p70的表达[62]。说明细菌表面蛋白的聚糖结构影响相关免疫细胞的激活,进而促进相关区域下游分子的表达,最终引起免疫反应。
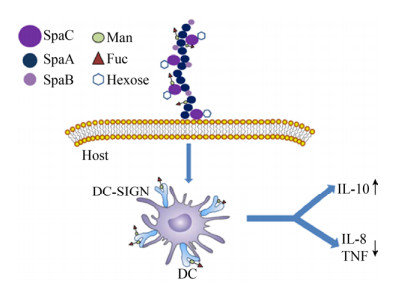
|
| 图 3 L. rhamnosus GG糖基化促进宿主细胞免疫应答 Figure 3 L. rhamnosus GG glycosylation promotes immune response in host cells |
|
|
多项研究表明,C. jejuni NCTC11168鞭毛蛋白FlaA除了能影响C. jejuni与BgAg的结合能力,还能促进宿主免疫应答[25, 37, 68]。研究表明,FlaA上有19个位点被糖基化,位点之上还连接有多个与唾液酸有关的九碳糖Pse或Leg及其衍生物[69]。FlaA结构中的Pse通过与宿主表面的Siglec-10相互作用,Pse和leg的核心环结构使其易于容纳在Siglec的sia口袋中,进而可通过P38依赖性途径调节MyD88介导的IL-10进行表达[70]。Howard等将flaA基因敲除后的鞭毛蛋白与野生型相比,IL-10表达水平明显下降[45],说明该结构可能会影响IL-10的表达。
此外,一些细菌的分泌蛋白能够对相关组织中的重要酶进行糖基化修饰,进而调节下游免疫分子的活性。例如肠出血性大肠杆菌(Enterohemhemorrhagic E. coli,EHEC)利用III型分泌系统将毒力效应因子NleB1递入宿主细胞,通过影响宿主细胞的各项生理功能来达到侵染宿主的目的[71-72]。与EHEC亲缘关系类似的柠檬酸杆菌(Citrobacter rodentium)分泌的毒力蛋白NleB具有N-乙酰葡糖胺转移酶活性,能够对GAPDH进行N-乙酰葡糖胺糖基化修饰,进而破坏其与肿瘤坏死因子受体相关因子2 (TNF receptor-associated factor 2,TRAF2)的相互作用,最终抑制了TRAF2的多聚泛素化和NF-κB的活性[73]。
4 总结与展望肠道微生物的蛋白质糖基化大多发生于鞭毛蛋白和黏附素等表层结构中,其糖基化修饰在肠道微生物的生命活动中发挥着重要作用。深入了解其影响机制一方面可以更好地阐明细菌糖类物质与宿主的生物学功能,另一方面可以应用于疫苗开发、疾病诊断以及药物治疗等。不同种类的细菌发生糖基化的蛋白有差异,体现出细菌糖基化的多样性,根据其多样性可以研发多种糖蛋白疫苗,或利用肠道宿主细胞与细菌特异性糖链之间的反应研发抗感染药物,这为肠道相关疾病的治疗提供参考信息。此外,肠道微生物异常糖基化也可能标志着某些肠道感染性疾病的发生,然而这些过程还有待进一步的研究。可以预见,将异常糖基化作为生物标志物寻找新的治疗靶标,为肠道有害菌致病机制的深入研究打下良好的基础。随着微生物蛋白组学和基因组学研究的快速发展,对于一些在细菌中尚未发现的蛋白质糖基化研究不断增多,但是这些现象下所潜藏机制的研究还不够深入。因此,未来的研究重点应集中在以下方面:有害菌致病机制的进一步阐明、肠道益生菌糖基化对宿主的详细保护机制、糖基化过程中蛋白质如何进行翻转和折叠、重要的糖基转移酶的特性以及编码糖基转移酶的基因等,这些都是研究者需要去解决的问题。
| [1] |
McDermott AJ, Huffnagle GB. The microbiome and regulation of mucosal immunity[J]. Immunology, 2014, 142(1): 24-31. DOI:10.1111/imm.12231 |
| [2] |
Li Z. Technology for Glycomics[M]. Beijing: Higher Education Press, 2015. (in Chinese) 李铮. 糖组学研究技术[M]. 北京: 高等教育出版社, 2015. |
| [3] |
Banerjee A, Wang R, Supernavage SL, et al. Implications of phase variation of a gene (pgtA) encoding a pilin galactosyl transferase in gonococcal pathogenesis[J]. Journal of Experimental Medicine, 2002, 196(2): 147-162. DOI:10.1084/jem.20012022 |
| [4] |
Kamada N, Seo SU, Chen GY, et al. Role of the gut microbiota in immunity and inflammatory disease[J]. Nature Reviews Immunology, 2013, 13(5): 321-335. DOI:10.1038/nri3430 |
| [5] |
Kashtanova DA, Popenko AS, Tkacheva ON, et al. Association between the gut microbiota and diet: fetal life, early childhood, and further life[J]. Nutrition, 2016, 32(6): 620-627. DOI:10.1016/j.nut.2015.12.037 |
| [6] |
Panda S, El Khader I, Casellas F, et al. Short-term effect of antibiotics on human gut microbiota[J]. PLoS One, 2014, 9(4): e95476. DOI:10.1371/journal.pone.0095476 |
| [7] |
Wu GD, Chen J, Hoffmann C, et al. Linking long-term dietary patterns with gut microbial enterotypes[J]. Science, 2011, 334(6052): 105-108. DOI:10.1126/science.1208344 |
| [8] |
Rodríguez JM, Murphy K, Stanton C, et al. The composition of the gut microbiota throughout life, with an emphasis on early life[J]. Microbial Ecology in Health and Disease, 2015, 26(1): 26050. |
| [9] |
Qin JJ, Li YR, Cai ZM, et al. A metagenome-wide association study of gut microbiota in type 2 diabetes[J]. Nature, 2012, 490(7418): 55-60. DOI:10.1038/nature11450 |
| [10] |
Surana NK, Kasper DL. The yin yang of bacterial polysaccharides: lessons learned from B. fragilis PSA[J]. Immunological Reviews, 2012, 245(1): 13-26. DOI:10.1111/j.1600-065X.2011.01075.x |
| [11] |
Diaz-Silvestre H, Espinosa-Cueto P, Sanchez-Gonzalez A, et al. The 19-kD antigen of Mycobacterium tuberculosis is a major adhesin that binds the mannose receptor of THP-1 monocytic cells and promotes phagocytosis of mycobacteria[J]. Microbial Pathogenesis, 2005, 39(3): 97-107. DOI:10.1016/j.micpath.2005.06.002 |
| [12] |
Bensing BA, Gibson BW, Sullam PM. The Streptococcus gordonii platelet binding protein GspB undergoes glycosylation independently of export[J]. Journal of Bacteriology, 2004, 186(3): 638-645. DOI:10.1128/JB.186.3.638-645.2004 |
| [13] |
Imaki H, Tomoyasu T, Yamamoto N, et al. Identification and characterization of a novel secreted glycosidase with multiple glycosidase activities in Streptococcus intermedius[J]. Journal of Bacteriology, 2014, 196(15): 2817-2826. DOI:10.1128/JB.01727-14 |
| [14] |
Qin XM, Guo YH, Du HQ, et al. Comparative analysis for glycopatterns and complex-type N-glycans of glycoprotein in sera from chronic hepatitis B- and C-infected patients[J]. Frontiers in Physiology, 2017, 8: 596. DOI:10.3389/fphys.2017.00596 |
| [15] |
Nothaft H, Szymanski CM. Protein glycosylation in bacteria: sweeter than ever[J]. Nature Reviews Microbiology, 2010, 8(11): 765-778. DOI:10.1038/nrmicro2383 |
| [16] |
Thibault P, Logan SM, Kelly JF, et al. Identification of the carbohydrate moieties and glycosylation motifs in Campylobacter jejuni flagellin[J]. Journal of Biological Chemistry, 2001, 276(37): 34862-34870. DOI:10.1074/jbc.M104529200 |
| [17] |
Logan SM, Kelly JF, Thibault P, et al. Structural heterogeneity of carbohydrate modifications affects serospecificity of Campylobacter flagellins[J]. Molecular Microbiology, 2002, 46(2): 587-597. DOI:10.1046/j.1365-2958.2002.03185.x |
| [18] |
Twine SM, Reid CW, Aubry A, et al. Motility and flagellar glycosylation in Clostridium difficile[J]. Journal of Bacteriology, 2009, 191(22): 7050-7062. DOI:10.1128/JB.00861-09 |
| [19] |
Aas FE, Vik Å, Vedde J, et al. Neisseria gonorrhoeae O-linked pilin glycosylation: functional analyses define both the biosynthetic pathway and glycan structure[J]. Molecular Microbiology, 2007, 65(3): 607-624. DOI:10.1111/j.1365-2958.2007.05806.x |
| [20] |
Charbonneau MÈ, Girard V, Nikolakakis A, et al. O-linked glycosylation ensures the normal conformation of the autotransporter adhesin involved in diffuse adherence[J]. Journal of Bacteriology, 2007, 189(24): 8880-8889. DOI:10.1128/JB.00969-07 |
| [21] |
Tan FYY, Tang CM, Exley RM. Sugar coating: bacterial protein glycosylation and host-microbe interactions[J]. Trends in Biochemical Sciences, 2015, 40(7): 342-350. DOI:10.1016/j.tibs.2015.03.016 |
| [22] |
Moormann C, Benz I, Schmidt MA. Functional substitution of the TibC protein of enterotoxigenic Escherichia coli strains for the autotransporter adhesin heptosyltransferase of the AIDA system[J]. Infection and Immunity, 2002, 70(5): 2264-2270. DOI:10.1128/IAI.70.5.2264-2270.2002 |
| [23] |
Knudsen SK, Stensballe A, Franzmann M, et al. Effect of glycosylation on the extracellular domain of the Ag43 bacterial autotransporter: enhanced stability and reduced cellular aggregation[J]. Biochemical Journal, 2008, 412(3): 563-577. DOI:10.1042/BJ20071497 |
| [24] |
Reidl S, Lehmann A, Schiller R, et al. Impact of O-glycosylation on the molecular and cellular adhesion properties of the Escherichia coli autotransporter protein Ag43[J]. International Journal of Medical Microbiology, 2009, 299(6): 389-401. DOI:10.1016/j.ijmm.2009.01.001 |
| [25] |
Mahdavi J, Pirinccioglu N, Oldfield NJ, et al. A novel O-linked glycan modulates Campylobacter jejuni major outer membrane protein-mediated adhesion to human histo-blood group antigens and chicken colonization[J]. Open Biology, 2014, 4(1): 130202. DOI:10.1098/rsob.130202 |
| [26] |
Siboo IR, Chambers HF, Sullam PM. Role of SraP, a serine-rich surface protein of Staphylococcus aureus, in binding to human platelets[J]. Infection and Immunity, 2005, 73(4): 2273-2280. DOI:10.1128/IAI.73.4.2273-2280.2005 |
| [27] |
Zhang JX, Zhong YG, Zhang PX, et al. Identification of abnormal fucosylated-glycans recognized by LTL in saliva of HBV-induced chronic hepatitis, cirrhosis, and hepatocellular carcinoma[J]. Glycobiology, 2019, 29(3): 242-259. DOI:10.1093/glycob/cwy108 |
| [28] |
Stepper J, Shastri S, Loo TS, et al. Cysteine S-glycosylation, a new post-translational modification found in glycopeptide bacteriocins[J]. FEBS Letters, 2011, 585(4): 645-650. DOI:10.1016/j.febslet.2011.01.023 |
| [29] |
Okumura R, Kurakawa T, Nakano T, et al. Lypd8 promotes the segregation of flagellated microbiota and colonic epithelia[J]. Nature, 2016, 532(7597): 117-121. DOI:10.1038/nature17406 |
| [30] |
Iwashkiw JA, Vozza NF, Kinsella RL, et al. Pour some sugar on it: the expanding world of bacterial protein O-linked glycosylation[J]. Molecular Microbiology, 2013, 89(1): 14-28. |
| [31] |
Yang SF, Liu X, Deng GY, et al. Advances in O-mannosylated proteins in Mycobacterium tuberculosis[J]. Journal of Microbiology, 2015, 35(4): 98-101. (in Chinese) 杨淑凤, 刘欣, 邓国英, 等. 结核分枝杆菌中O-甘露糖基化蛋白功能的研究进展[J]. 微生物学杂志, 2015, 35(4): 98-101. DOI:10.3969/j.issn.1005-7021.2015.04.017 |
| [32] |
de Araujo LS, de Queiroz Mello FC, de Bárbara Moreira da Silva N, et al. Evaluation of gamma interferon immune response elicited by the newly constructed PstS-1(285-374):CFP10 fusion protein to detect Mycobacterium tuberculosis infection[J]. Clinical and Vaccine Immunology, 2014, 21(4): 552-560. DOI:10.1128/CVI.00726-13 |
| [33] |
Todorov SD. Bacteriocins from Lactobacillus plantarum - production, genetic organization and mode of action[J]. Brazilian Journal of Microbiology, 2009, 40(2): 209-221. DOI:10.1590/S1517-83822009000200001 |
| [34] |
Ren FZ, Wang N, Shang YW, et al. Research progress on flagella of Campylobacter jejuni[J]. Chinese Journal of Zoonoses, 2013, 29(8): 812-816. (in Chinese) 任方哲, 王楠, 商宇伟, 等. 空肠弯曲菌鞭毛研究进展[J]. 中国人兽共患病学报, 2013, 29(8): 812-816. DOI:10.3969/cjz.j.issn.1002-2694.2013.08.016 |
| [35] |
Salah Ud-Din AIM, Roujeinikova A. Flagellin glycosylation with pseudaminic acid in Campylobacter and Helicobacter: prospects for development of novel therapeutics[J]. Cellular and Molecular Life Sciences, 2018, 75(7): 1163-1178. DOI:10.1007/s00018-017-2696-5 |
| [36] |
Latousakis D, Juge N. How sweet are our gut beneficial bacteria? A focus on protein glycosylation in Lactobacillus[J]. International Journal of Molecular Sciences, 2018, 19(1): 136. DOI:10.3390/ijms19010136 |
| [37] |
Szymanski CM, Burr DH, Guerry P. Campylobacter protein glycosylation affects host cell interactions[J]. Infection and Immunity, 2002, 70(4): 2242-2244. DOI:10.1128/IAI.70.4.2242-2244.2002 |
| [38] |
Kowarik M, Numao S, Feldman MF, et al. N-linked glycosylation of folded proteins by the bacterial oligosaccharyltransferase[J]. Science, 2006, 314(5802): 1148-1150. DOI:10.1126/science.1134351 |
| [39] |
Wacker M, Feldman MF, Callewaert N, et al. Substrate specificity of bacterial oligosaccharyltransferase suggests a common transfer mechanism for the bacterial and eukaryotic systems[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(18): 7088-7093. DOI:10.1073/pnas.0509207103 |
| [40] |
Chen MM, Glover KJ, Imperiali B. From peptide to protein: comparative analysis of the substrate specificity of N-linked glycosylation in C. jejuni[J]. Biochemistry, 2007, 46(18): 5579-5585. DOI:10.1021/bi602633n |
| [41] |
Kowarik M, Young NM, Numao S, et al. Definition of the bacterial N-glycosylation site consensus sequence[J]. The EMBO Journal, 2006, 25(9): 1957-1966. DOI:10.1038/sj.emboj.7601087 |
| [42] |
Faridmoayer A, Fentabil MA, Haurat MF, et al. Extreme substrate promiscuity of the Neisseria oligosaccharyl transferase involved in protein O-glycosylation[J]. Journal of Biological Chemistry, 2008, 283(50): 34596-34604. DOI:10.1074/jbc.M807113200 |
| [43] |
Zhai YF, Li H, Liu XW, et al. Research progress of protein O-glycosylation in prokaryotes[J]. Microbiology China, 2017, 44(4): 970-975. (in Chinese) 翟娅菲, 李红, 刘现伟, 等. 原核生物蛋白O-糖基化的研究进展[J]. 微生物学通报, 2017, 44(4): 970-975. |
| [44] |
Müller A, Del R León-Kempis M, Dodson E, et al. A bacterial virulence factor with a dual role as an adhesin and a solute-binding protein: the crystal structure at 1.5 Å resolution of the PEB1a protein from the food-borne human pathogen Campylobacter jejuni[J]. Journal of Molecular Biology, 2007, 372(1): 160-171. DOI:10.1016/j.jmb.2007.06.041 |
| [45] |
Howard SL, Jagannathan A, Soo EC, et al. Campylobacter jejuni glycosylation island important in cell charge, legionaminic acid biosynthesis, and colonization of chickens[J]. Infection and Immunity, 2009, 77(6): 2544-2556. DOI:10.1128/IAI.01425-08 |
| [46] |
Ewing CP, Andreishcheva E, Guerry P. Functional characterization of flagellin glycosylation in Campylobacter jejuni 81-176[J]. Journal of Bacteriology, 2009, 191(22): 7086-7093. DOI:10.1128/JB.00378-09 |
| [47] |
Sherlock O, Dobrindt U, Jensen JB, et al. Glycosylation of the self-recognizing Escherichia coli Ag43 autotransporter protein[J]. Journal of Bacteriology, 2006, 188(5): 1798-1807. DOI:10.1128/JB.188.5.1798-1807.2006 |
| [48] |
Lu QH, Yao Q, Xu Y, et al. An iron-containing dodecameric heptosyltransferase family modifies bacterial autotransporters in pathogenesis[J]. Cell Host & Microbe, 2014, 16(3): 351-363. |
| [49] |
Wells TJ, Tree JJ, Ulett GC, et al. Autotransporter proteins: novel targets at the bacterial cell surface[J]. FEMS Microbiology Letters, 2007, 274(2): 163-172. DOI:10.1111/j.1574-6968.2007.00833.x |
| [50] |
Sherlock O, Vejborg RM, Klemm P. The TibA adhesin/invasin from enterotoxigenic Escherichia coli is self recognizing and induces bacterial aggregation and biofilm formation[J]. Infection and Immunity, 2005, 73(4): 1954-1963. DOI:10.1128/IAI.73.4.1954-1963.2005 |
| [51] |
Côté JP, Charbonneau MÈ, Mourez M. Glycosylation of the Escherichia coli TibA self-associating autotransporter influences the conformation and the functionality of the protein[J]. PLoS One, 2013, 8(11): e80739. DOI:10.1371/journal.pone.0080739 |
| [52] |
Coyne MJ, Fletcher CM, Chatzidaki-Livanis M, et al. Phylum-wide general protein O-glycosylation system of the Bacteroidetes[J]. Molecular Microbiology, 2013, 88(4): 772-783. DOI:10.1111/mmi.12220 |
| [53] |
Fletcher CM, Coyne MJ, Villa OF, et al. A general O-glycosylation system important to the physiology of a major human intestinal symbiont[J]. Cell, 2009, 137(2): 321-331. DOI:10.1016/j.cell.2009.02.041 |
| [54] |
Fredriksen L, Moen A, Adzhubei AA, et al. Lactobacillus plantarum WCFS1 O-linked protein glycosylation: an extended spectrum of target proteins and modification sites detected by mass spectrometry[J]. Glycobiology, 2013, 23(12): 1439-1451. DOI:10.1093/glycob/cwt071 |
| [55] |
Lebeer S, Claes IJJ, Balog CIA, et al. The major secreted protein Msp1/p75 is O-glycosylated in Lactobacillus rhamnosus GG[J]. Microbial Cell Factories, 2012, 11: 15. DOI:10.1186/1475-2859-11-15 |
| [56] |
Alemka A, Nothaft H, Zheng J, et al. N-glycosylation of Campylobacter jejuni surface proteins promotes bacterial fitness[J]. Infection and Immunity, 2013, 81(5): 1674-1682. DOI:10.1128/IAI.01370-12 |
| [57] |
Zhang QJ, Meitzler JC, Huang SX, et al. Sequence polymorphism, predicted secondary structures, and surface-exposed conformational epitopes of Campylobacter major outer membrane protein[J]. Infection and Immunity, 2000, 68(10): 5679-5689. DOI:10.1128/IAI.68.10.5679-5689.2000 |
| [58] |
Guerry P, Ewing CP, Schirm M, et al. Changes in flagellin glycosylation affect Campylobacter autoagglutination and virulence[J]. Molecular Microbiology, 2006, 60(2): 299-311. DOI:10.1111/j.1365-2958.2006.05100.x |
| [59] |
Avilés-Reyes A, Miller JH, Simpson-Haidaris PJ, et al. Modification of Streptococcus mutans Cnm by PgfS contributes to adhesion, endothelial cell invasion, and virulence[J]. Journal of Bacteriology, 2014, 196(15): 2789-2797. DOI:10.1128/JB.01783-14 |
| [60] |
Yang YH, Jiang YL, Zhang J, et al. Structural insights into SraP-mediated Staphylococcus aureus adhesion to host cells[J]. PLoS Pathogens, 2014, 10(6): e1004169. DOI:10.1371/journal.ppat.1004169 |
| [61] |
Rolain T, Bernard E, Beaussart A, et al. O-glycosylation as a novel control mechanism of peptidoglycan hydrolase activity[J]. Journal of Biological Chemistry, 2013, 288(31): 22233-22247. DOI:10.1074/jbc.M113.470716 |
| [62] |
Konstantinov SR, Smidt H, de Vos WM, et al. S layer protein A of Lactobacillus acidophilus NCFM regulates immature dendritic cell and T cell functions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(49): 19474-19479. DOI:10.1073/pnas.0810305105 |
| [63] |
Zhu F, Zhang H, Wu H. Glycosyltransferase-mediated sweet modification in oral streptococci[J]. Journal of Dental Research, 2015, 94(5): 659-665. DOI:10.1177/0022034515574865 |
| [64] |
Coyne MJ, Reinap B, Lee MM, et al. Human symbionts use a host-like pathway for surface fucosylation[J]. Science, 2005, 307(5716): 1778-1781. DOI:10.1126/science.1106469 |
| [65] |
Reunanen J, von Ossowski I, Hendrickx APA, et al. Characterization of the SpaCBA pilus fibers in the probiotic Lactobacillus rhamnosus GG[J]. Applied and Environmental Microbiology, 2012, 78(7): 2337-2344. DOI:10.1128/AEM.07047-11 |
| [66] |
von Ossowski I, Pietilä TE, Rintahaka J, et al. Using recombinant Lactococci as an approach to dissect the immunomodulating capacity of surface piliation in probiotic Lactobacillus rhamnosus GG[J]. PLoS One, 2013, 8(5): e64416. DOI:10.1371/journal.pone.0064416 |
| [67] |
Tytgat HLP, van Teijlingen NH, Sullan RMA, et al. Probiotic gut microbiota isolate interacts with dendritic cells via glycosylated heterotrimeric pili[J]. PLoS One, 2016, 11(3): e0151824. DOI:10.1371/journal.pone.0151824 |
| [68] |
Howard SL, Jagannathan A, Soo EC, et al. Campylobacter jejuni glycosylation island important in cell charge, legionaminic acid biosynthesis, and colonization of chickens[J]. Infection and Immunity, 2009, 77(6): 2544-2556. DOI:10.1128/IAI.01425-08 |
| [69] |
McNally DJ, Aubry AJ, Hui JPM, et al. Targeted metabolomics analysis of Campylobacter coli VC167 reveals legionaminic acid derivatives as novel flagellar glycans[J]. Journal of Biological Chemistry, 2007, 282(19): 14463-14475. DOI:10.1074/jbc.M611027200 |
| [70] |
Stephenson HN, Mills DC, Jones H, et al. Pseudaminic acid on Campylobacter jejuni flagella modulates dendritic cell IL-10 expression via Siglec-10 receptor: a novel flagellin-host interaction[J]. The Journal of Infectious Diseases, 2014, 210(9): 1487-1498. DOI:10.1093/infdis/jiu287 |
| [71] |
Pearson JS, Giogha C, Ong SY, et al. A type Ⅲ effector antagonizes death receptor signalling during bacterial gut infection[J]. Nature, 2013, 501(7466): 247-251. DOI:10.1038/nature12524 |
| [72] |
Li S, Zhang L, Yao Q, et al. Pathogen blocks host death receptor signalling by arginine GlcNAcylation of death domains[J]. Nature, 2013, 501(7466): 242-246. DOI:10.1038/nature12436 |
| [73] |
Gao XF, Wang XG, Pham TH, et al. NleB, a bacterial effector with glycosyltransferase activity, targets GAPDH function to inhibit NF-κB activation[J]. Cell Host & Microbe, 2013, 13(1): 87-99. |
 2020, Vol. 47
2020, Vol. 47