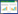扩展功能
文章信息
- 高明明, 王灿, 李璞媛, 刘慧莹, 裴广倩, 范航, 张湘莉兰, 米志强, 童贻刚, 柏长青
- GAO Ming-Ming, WANG Can, LI Pu-Yuan, LIU Hui-Ying, PEI Guang-Qian, FAN Hang, ZHANG Xiang-Li-Lan, MI Zhi-Qiang, TONG Yi-Gang, BAI Chang-Qing
- 一株新型裂解K63荚膜型肺炎克雷伯菌的噬菌体分离鉴定和生物学特性研究及全基因组分析
- Biological characteristics and complete genomic analysis of a novel virulent bacteriophage that infects Klebsiella pneumoniae capsular type K63
- 微生物学通报, 2020, 47(1): 210-221
- Microbiology China, 2020, 47(1): 210-221
- DOI: 10.13344/j.microbiol.china.190585
-
文章历史
- 收稿日期: 2019-06-25
- 接受日期: 2019-09-24
- 网络首发日期: 2019-10-10
2. 解放军总医院第五医学中心呼吸与危重症医学科 北京 100071;
3. 军事科学院军事医学研究院微生物流行病研究所 病原微生物安全国家重点实验室 北京 100071;
4. 北京化工大学生命科学与技术学院 北京 100029
2. Department of Respiratory and Critical Care Diseases, The Fifth Medical Center, Chinese General Hospital of the PLA, Beijing 100071, China;
3. State Key Laboratory of Pathogenic Microbiology Safety, Institute of Microbiological Epidemiology, Institute of Military Medicine, Academy of Military Sciences, Beijing 100071, China;
4. College of Life Science and Technology, Beijing University of Chemical Technology, Beijing 100029, China
肺炎克雷伯菌(Klebsiella pneumoniae)为革兰氏阴性杆菌,是医院和社区获得性尿路感染常见病原体,败血症和肺炎的常见原因[1-2]。近几十年来,由肺炎克雷伯菌并发转移性脑膜炎和眼内炎引起的社区获得性化脓性肝脓肿病例已在全世界范围内出现,特别是在亚洲[3-4]。由于抗生素滥用和细菌进化,抗生素耐药问题正在变得越来越严重。抗生素长期以来被认为是治疗细菌感染性疾病最有力的武器,碳青霉烯类抗生素被认为是最后的抗生素。但是随着更多碳青霉烯类抗生素的广泛使用,产超广谱β-内酰胺酶(extended spectrum β-lactamase,ESBLs)的菌株正在增加,这给临床治疗带来了困难。因此,美国疾病控制和预防中心认为耐碳青霉烯类肠杆菌科是公共卫生的直接威胁[5]。
噬菌体(bacteriophage)作为一种新兴的治疗方法,已经越来越受到人们的关注。噬菌体对细菌具有高度专一性,可在细菌中繁殖并杀死细菌却对动植物没有毒性。Cao等[6]用分离自临床患者的多药耐药肺炎克雷伯菌,使用滴鼻法构建肺炎感染的小鼠模型,随后用单剂量噬菌体(2×109 PFU/小鼠)治疗感染小鼠,结果显示,未用噬菌体处理的小鼠24 h内全部死亡,使用噬菌体治疗组,只有20%的小鼠在感染后72 h死亡,这种治疗方法显着提高了感染小鼠的存活率,提示肺炎克雷伯菌噬菌体可用于噬菌体治疗。此外,Manohar等将污水样品中分离出的噬菌体制备成噬菌体鸡尾酒,然后加入到城市污水中孵育2 h,细菌负荷从106 CFU/mL降至103 CFU/mL,这个结果表明噬菌体鸡尾酒也是一个很好的生物防治剂[7]。
我们根据Pan等的方法鉴定了宿主菌226肺炎克雷伯菌菌株属于K63荚膜型[8]。在本研究中,我们以一株K63荚膜型肺炎克雷伯菌菌株226为宿主菌,从医院废水样品中获得一株新型肺炎克雷伯菌噬菌体,命名为vB_KpnP_IME308,并研究其生物学特性,确定其基因组序列,这些结果可为进一步应用和研究不同荚膜类型的肺炎克雷伯菌噬菌体在临床治疗中作为抗生素替代品的应用奠定基础。
1 材料与方法 1.1 材料 1.1.1 菌株来源与培养条件肺炎克雷伯菌菌株226分离自中国人民解放军总医院呼吸与重症医学科一名肺炎感染患者的痰液,使用引物16S-F (5′-AGAGTTTGATCCTGGC TCAG-3′)和16S-R (5′-ACGGCTACCTTGTTACGA CTT-3′),通过16S rRNA基因的PCR扩增进行初步的肺炎克雷伯菌鉴定[9],然后使用终浓度25%的甘油保存至军事医学科学院微生物流行病研究所−80 ℃细菌库中。肺炎克雷伯菌通常在LB培养基中于37 ℃通风培养。
1.1.2 培养基及主要试剂和仪器LB液体培养基(g/L):氯化钠10.0,酵母提取物5.0,胰蛋白胨10.0,于1×105 Pa灭菌20 min。LB固体培养基(g/L):氯化钠10.0,酵母提取物5.0,胰蛋白胨10.0,琼脂15.0,1×105 Pa灭菌20 min。LB半固体培养基(g/L):氯化钠10.0,酵母提取物5.0,胰蛋白胨10.0,琼脂7.5,1×105 Pa灭菌20 min。
蛋白酶K、RNase、DNase,Thermo Fisher Scientific公司;酚:氯仿:异戊醇(25:24:1,体积比)、DNA平衡酚,北京索莱宝科技有限公司。MiSeq测序仪,Illumina公司。
1.2 噬菌体的分离鉴定噬菌体的分离纯化按照文献[10]所描述的步骤进行。具体来说,噬菌体的分离使用污水富集法:将污水12 000×g离心10 min,0.22 μm微孔滤膜(millipore膜,孔径0.22 μm)进行过滤,并将2 mL收集的液体与10 mL指数期(OD600=0.4−0.6)指示菌混合。将混合物在37 ℃温育过夜,12 000×g离心10 min,收集上清液并用0.22 μm微孔滤膜进行过滤以除去残留的细菌细胞。然后,100 μL上清液和500 μL指示菌指数生长期(OD600=0.4−0.6)混合,混合物加入约5 mL上层琼脂(LB含0.7%琼脂,50 ℃水浴)中,倒入含有下层琼脂的LB平板上(双层法)[11],待上层琼脂凝固后移至37 ℃恒温培养箱培养过夜。通过5次连续单斑分离获得纯噬菌体菌株。
1.3 噬菌体最佳感染复数(multiplicity of infection,MOI)测定参照Li等[12]的方法,略有改动。为了确定最佳MOI,根据感染比例,将噬菌体和宿主菌液分别按照MOI为10、1、0.1、0.01、0.001的拷贝数比例混合,各200 μL,加入新鲜的5 mL LB培养基中,37 ℃、220 r/min振荡培养6 h后,6 000×g离心15 min除去细胞沉淀。收集上清液,使用0.22 μm的滤膜过滤,通过双层平板法测定噬菌体的滴度。其中噬菌体滴度最高的MOI是最佳的感染复数[13]。每次独立实验重复3次。
1.4 噬菌体一步生长曲线(one-step growth)的测定参照Wang等的方法[14],略有改动。使用培养至生长指数中期肺炎克雷伯氏菌菌株226 (OD600=0.2−0.6)并与噬菌体vB_KpnP_IME308以0.001的MOI混合,将混合物置于37 ℃恒温培养箱孵育10 min,使噬菌体与细菌充分吸附,然后将混合物以10 000×g离心1 min,将含有宿主细菌的沉淀物用37 ℃新鲜LB洗涤2次,期间无需振荡混匀。然后将混合物转移到20 mL LB培养基中并在37 ℃、220 r/min振荡培养。然后在感染后0、5、10、20、30、40、50、60、70、80、90、100、110、120 min收集3个150 μL等分试样。将样品以10 000×g离心2 min,除去上清液,然后使用双层琼脂测定噬菌体滴度,稀释样品并立即铺板以确定噬菌体滴度。每次独立实验重复3次。
1.5 噬菌体温度和pH敏感性的测定使用无菌1.5 mL离心管将900 μL LB液体培养基在4、20、30、40、50、60、70和80 ℃金属浴中预热60 min,待温度稳定后,加入100 μL噬菌体(109噬斑形成单位PFU/mL),并将样品在培养箱中加热。孵育60 min后收集样品,然后通过双层琼脂方法测定噬菌体滴度。该测定重复3次[15]。对于pH敏感性,用1 mol/L HCl和1 mol/L NaOH将LB培养基调节至pH 2.0−14.0。接下来,将100 μL噬菌体原液(109噬菌斑形成单位PFU/mL)加入到900 μL分别装不同pH值LB溶液的1.5 mL EP管中,37 ℃孵育60 min,通过双层琼脂方法测定噬菌体滴度。这个实验也重复了3次[16]。
1.6 噬菌体电镜噬菌体vB_KpnP_IME308的形态进一步可视化通过透射电子显微镜(transmission electron microscope,TEM)观察[17]。吸取一滴纯化的噬菌体悬液滴在铜网上,用2%磷钨酸溶液染色1 min后立即除去过量的染色剂,在100 kV TEM 106下观察噬菌体的形态。
1.7 噬菌体基因组提取噬菌体基因组使用文献[18]描述的标准苯酚-氯仿提取方案。取600 μL已纯化过滤的噬菌体裂解液,加入DNase I (1 μg/mL)和RNase A (1 μg/mL),将混合物充分涡旋并在37 ℃下温育,消化过夜;80 ℃酶灭活20 min后加入裂解缓冲液(终浓度:0.5% SDS,20 mmol/L EDTA和50 μg/mL蛋白酶K),将混合物在金属浴中56 ℃温育1 h;加入等体积的DNA平衡酚,充分涡旋振荡1 min,然后将混合物12 000×g离心10 min。转移上层水相到一个新的1.5 mL离心管中,加入相同体积的苯酚:氯仿:异戊醇(25:24:1,体积比),充分涡旋振荡1 min,12 000×g离心10 min以去除蛋白质和多糖物质。将上层水相再次转移至新的1.5 mL离心管中,加入等体积的异丙醇,−20 ℃放置4 h沉淀基因组DNA。然后将样品在4 ℃条件下10 000×g离心20 min后收集沉淀物。用75%的乙醇(预冷)洗涤上述DNA沉淀,室温干燥后使用适量无核酸水重悬DNA沉淀,并储存在−20 ℃保存备用。
1.8 噬菌体的全基因组测序及生物信息学分析噬菌体vB_KpnP_IME308的DNA全基因组测序使用的是Illumina MiSeq测序平台。DNA文库构建使用NEBNext® Ultra™ II DNA Library Prep Kit来完成。
基因组序列组装使用的使Newbler V3.0软件[19] (Roche applied science)和CLC 3.0软件。使用RAST在线数据库[20] (http://www.rast.nmpdr.org)进行开放阅读框(open reading frame,ORF)初步确定,所有的ORF使用NCBI的蛋白质基本局部比对工具BLASTp (http://www.ncbi.nlm.nih.gov/BLAST/)手动验证。此外,进一步的基因组分析使用tRNA scan-SE 2.0版(http://lowelab.ucsc.edu/tRNAscan-SE/)[21]预测tRNA,TMHMM Server V2.0 (http://www.cbs.dtu.dk/services/TMHMM/)用于筛选所有的潜在含有跨膜结构域蛋白质的开放阅读框。序列比对分析使用Mauve 2.3.1[22],生成基因组图谱使用SnapGene® V2.6.2 (GSL Biotech;www.snapgene.com)。为了确定噬菌体vB_KpnP_IME308与其他Autographivirinae亚科之间的系统发育关系,使用MEGA 6.0[23]构建系统发育树。选择主要衣壳蛋白(ORF34)和推定的末端酶大亚基(ORF43)两种蛋白,从当前的ICTV (international committee on taxonomy of viruses)库中各下载了序列,系统发育分析使用ClustalW和Neighbor-Joining的方法构建。
2 结果与分析 2.1 噬菌体的分离与鉴定使用肺炎克雷伯菌菌株226作为宿主菌,从医院污水中分离出噬菌体vB_KpnP_IME308。噬菌体经过5次纯化之后,37 ℃下孵育12 h,在双层平板上,该噬菌体产生直径1−2 mm的斑块,其中小的透明噬菌斑周围被晕环包围(图 1A)。如Hughes等所述,晕圈的存在可能表明可溶性噬菌体酶的产生,例如多糖解聚酶[24]。
|
| 图 1 噬菌体vB_KpnP_IME308的形态 Figure 1 The morphology of phage vB_KpnP_IME308 注:A:噬菌斑;B:电镜照片. Note: A: Plaques of phage; B: Transmission electron micrograph. |
|
|
电子显微照片显示噬菌体具有典型的二十面体结构和非收缩的尾部,其头部直径约为60±1 nm,尾部长度约为10±2 nm,这是Podoviridae家族(Caudovirales目)的特征(图 1B)。根据目前的国际病毒分类委员会分类系统,噬菌体vB_KpnP_ IME308被划分为Autographivirinae亚科中的Drulisvirus属。
2.3 噬菌体最佳感染复数噬菌体与宿主菌肺炎克雷伯菌菌株226按一定比例混合,当以MOI为0.001感染时,噬菌体vB_KpnP_IME308产生最大滴度(表 1),表明0.001是噬菌体vB_KpnP_IME308的最佳MOI (表 1)。
| 序号 No. |
菌落数 Numbers of bacteria (CFU/mL) |
噬菌体数 Numbers of phages (PFU/mL) |
感染复数 MOI |
噬菌体滴度 Titer (PFU/mL) |
| 1 | 108 | 109 | 10 | 1.6×108 |
| 2 | 108 | 108 | 1 | 3.0×108 |
| 3 | 108 | 107 | 0.1 | 7.6×108 |
| 4 | 108 | 106 | 0.01 | 8.8×109 |
| 5 | 108 | 105 | 0.001 | 1.2×1010 |
进行一步生长曲线实验以确定噬菌体vB_KpnP_ IME308的潜伏期(latent period)和暴发量(burst size)。由图 2A可知,噬菌体与对数生长期的肺炎克雷伯菌混合培养20 min曲线走势无明显变化,即噬菌体潜伏期约为20 min;而后噬菌体的滴度急剧增加,在约80 min后其滴度趋于平稳,则噬菌体感染期的时间约为80 min,由此可得裂解量为3.3×1010/1×108=330 PFU/cell (裂解量=裂解末期噬菌体滴度/感染初期宿主菌浓度),即噬菌体vB_KpnP_IME308感染宿主菌的裂解量约为330 PFU/cell。

|
| 图 2 噬菌体vB_KpnP_IME308的生物学特性 Figure 2 Biological properties of phage vB_KpnP_IME308 注:A:一步生长曲线;B:温度敏感性;C:pH敏感性. Note: A: One-step growth curve; B: Thermostability; C: pH stability. |
|
|
噬菌体vB_KpnP_IME308在4−50 ℃的温度范围内稳定生长。当在60 ℃温育60 min时,噬菌体滴度减少几乎1lg;随后在70 ℃水金属浴中孵育60 min噬菌体滴度迅速下降至1×104 PFU/mL,70 ℃下孵育60 min,噬菌体滴度急剧下降至0。这些结果表明噬菌体能够耐受实验环境下的常规温度(图 2B)。噬菌体vB_KpnP_IME308在pH 5.0−10.0的范围内显示较高的活性,而pH低于5.0或者大于10.0时活性下降,由此可知噬菌体vB_KpnP_IME308的最适合pH范围为4.0–10.0 (图 2C)。这些数据表明噬菌体vB_KpnP_IME308可能具有肺炎克雷伯菌生物控制的潜在应用。
2.6 噬菌体的全基因组分析噬菌体vB_KpnP_IME308的基因组为双链线性的DNA,其核酸序列NCBI登录号为MN176572。基因组组装使用Roche Newbler 3.0汇编程序和CLC软件进行的。具体而言,基因组文库产生170 656 kb的数据,平均阅读长度为298个碱基,发现vB_KpnP_IME308的完整基因组长为43 091 bp,整个读数的96.08%与完整基因组匹配(572 472个读数中550 035个),平均覆盖率为3 960。噬菌体IME308基因组A、C、G、T碱基含量分别为24.6%、24.8%、29.1%和21.5%,(G+C)mol%含量为53.9%,(A+T)mol%含量为46.1%。
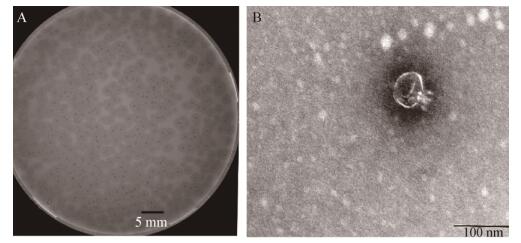
使用RAST鉴定基因组序列注释显示,噬菌体vB_KpnP_IME308含有总共49个开放阅读框,其中22个为功能编码序列(coding sequence,CDS),其余基因注释为假设蛋白质或未知功能蛋白质(表 2)。该基因组主要涉及4个功能模块,包括:噬菌体裂解系统;结构和DNA包装;代谢;复制。
| ORF | Top BLAST hita | Amino acid identity (%)b | E value | Accession No. |
| 1 | Hypothetical protein [Klebsiella phage KpV71] | 61/66 (92%) | 5.00E−35 | NC_031246.1 |
| 2 | Hypothetical protein [Dehalobacter sp. FTH1] | 15/25 (60%) | 0.092 | NZ_AQYY00000000.1 |
| 3 | Hypothetical protein [Klebsiella phage vB_KpnP_SU503] | 53/59 (90%) | 9.00E−33 | NC_028816.1 |
| 4 | Hypothetical protein [Klebsiella phage F19] | 47/67 (70%) | 3.00E−28 | NC_023567.2 |
| 5 | Hypothetical protein [Klebsiella phage vB_KpnP_KpV48] | 543/659 (82%) | 0 | KX237514.1 |
| 6 | Putative peptidase [Klebsiella phage F19] | 344/348 (99%) | 0 | NC_023567.2 |
| 7 | Hypothetical protein [Klebsiella phage F19] | 96/155 (62%) | 2.00E−57 | NC_023567.2 |
| 8 | Putative DNA primase [Klebsiella phage vB_KpnP_KpV48] | 250/260 (96%) | 0 | KX237514.1 |
| 9 | Hypothetical protein [Klebsiella phage KP34] | 63/64 (98%) | 5.00E−38 | NC_013649.2 |
| 10 | Hypothetical protein [Klebsiella phage vB_Kpn_IME260] | 23/45 (51%) | 4.00E−05 | NC_041899.1 |
| 11 | Putative DNA helicase [Klebsiella phage KpV71] | 422/426 (99%) | 0 | NC_031246.1 |
| 12 | Hypothetical protein [Klebsiella phage KpV41] | 50/51 (98%) | 5.00E−28 | NC_028670.1 |
| 13 | DNA polymerase [Klebsiella phage phiKpS2] | 801/803 (99%) | 0 | KX587949.2 |
| 14 | Hypothetical protein [Klebsiella phage KP-Rio/2015] | 73/73 (100%) | 1.00E−44 | KX856662.1 |
| 15 | Putative HNH endonuclease[Klebsiella phage NTUH-K2044-K1-1] | 32/54 (59%) | 1.00E−12 | NC_025418.1 |
| 16 | Hypothetical protein [Klebsiella phage KP34] | 311/348 (89%) | 0 | NC_013649.2 |
| 17 | Putative HNH endonuclease [Klebsiella phage KP34] | 124/130 (95%) | 5.00E−87 | NC_013649.2 |
| 18 | Hypothetical protein [Klebsiella phage vB_KpnP_KpV74] | 49/64 (77%) | 5.00E−17 | KY385423.1 |
| 19 | Hypothetical protein [Klebsiella phage F19] | 259/265 (98%) | 0 | NC_023567.2 |
| 20 | Hypothetical protein [Klebsiella phage Kp2] | 77/84 (92%) | 1.00E−44 | KT367886.1 |
| 21 | Hypothetical protein [Klebsiella phage Kp2] | 107/124 (86%) | 4.00E−69 | KT367886.1 |
| 22 | Hypothetical protein [Klebsiella phage AltoGao] | 50/53 (94%) | 5.00E−28 | MF612071.1 |
| 23 | Putative 5′-3′ exonuclease [Klebsiella phage vB_KpnP_SU552A] | 293/309 (95%) | 0 | NC_028870.1 |
| 24 | Hypothetical protein [Achromobacter phage JWAlpha] | 60/148 (41%) | 8.00E−24 | NC_023556.1 |
| 25 | Putative DNA endonuclease VII [Klebsiella phage F19] | 139/140 (99%) | 7.00E−96 | NC_023567.2 |
| 26 | Hypothetical protein [Klebsiella phage Kp2] | 100/152 (66%) | 6.00E−60 | KT367886.1 |
| 27 | Hypothetical protein [Klebsiella phage Kp2] | 36/42 (86%) | 4.00E−19 | KT367886.1 |
| 28 | RNA polymerase [Klebsiella phage Kp2] | 813/822 (99%) | 0 | KT367886.1 |
| 29 | Hypothetical protein phiKpS2_34 [Klebsiella phage phiKpS2] | 146/146 (100%) | 5.00E−104 | KX587949.2 |
| 30 | Hypothetical protein [Klebsiella phage KP34] | 86/87 (99%) | 2.00E−50 | NC_013649.2 |
| 31 | Collar protein [Klebsiella phage Kp2] | 526/531 (99%) | 0 | KT367886.1 |
| 32 | Putative scaffolding protein [Klebsiella phage KpV41] | 273/280 (98%) | 0 | NC_028670.1 |
| 33 | Putative HNH endonuclease [Klebsiella phage vB_KpnP_KpV74] | 114/153 (75%) | 5.00E−81 | KY385423.1 |
| 34 | Major capsid protein [Escherichia phage Minorna] | 325/338 (96%) | 0 | MK598851.1 |
| 35 | Hypothetical protein [Klebsiella phage KP34] | 53/59 (90%) | 7.00E−27 | NC_013649.2 |
| 36 | Tail tubular protein [Klebsiella phage myPSH1235] | 182/186 (98%) | 2.00E−133 | MG972768.1 |
| 37 | Tail tubular protein B [Klebsiella phage F19] | 737/759 (97%) | 0 | NC_023567.2 |
| 38 | Putative internal virion protein [Klebsiella phagev B_KpnP_SU552A] | 193/195 (99%) | 2.00E−135 | NC_028870.1 |
| 39 | Hypothetical protein [Klebsiella phage Kp2] | 877/894 (98%) | 0 | KT367886.1 |
| 40 | Putative internal core protein [Klebsiella phage vB_KpnP_SU503] | 1 205/1 232 (98%) | 0 | NC_028816.1 |
| 41 | Tail fibers protein [Klebsiella phage Kp2] | 503/530 (95%) | 0 | KT367886.1 |
| 42 | Putative DNA maturase A [Klebsiella phage vB_KpnP_KpV74] | 100/100 (100%) | 4.00E−63 | KY385423.1 |
| 43 | Putative large terminase subnit [Klebsiella phage phiBO1E] | 610/618 (99%) | 0 | KM576124.1 |
| 44 | Hypothetical protein [Enterobacteria phage J8-65] | 56/140 (40%) | 6.00E−11 | NC_025445.1 |
| 45 | Hypothetical protein [Klebsiella phage KPV811] | 52/60 (87%) | 6.00E−27 | KY000081.1 |
| 46 | Hypothetical protein [Klebsiella phage KpV41] | 134/134 (100%) | 6.00E−88 | NC_028670.1 |
| 47 | Hypothetical protein [Klebsiella phage Kp2] | 82/83 (99%) | 6.00E−52 | KT367886.1 |
| 48 | Endolysin [Klebsiella phage F19] | 180/184 (98%) | 4.00E−132 | NC_023567.2 |
| 49 | Tail fiber family protein [Klebsiella phage F19] | 562/579 (97%) | 0 | NC_023567.2 |
| 50 | No hit | |||
| 注:a:最相近基因组微生物物种的基因和名称;No hit:没有比对到相近的氨基酸序列;b:BLASTp搜索到比对百分比.
Note: a: The most closely related genes and names of the organisms; No hit: Indicates no significant homology detected for a particular amino acid sequence; b: Percent identity for top hits in BLASTp searches. |
||||
功能蛋白质可视化如图 3所示。其中,裂解系统中仅鉴定出一个相关基因为ORF48 (细胞内溶素),它是噬菌体编码的酶,其主要功能是在噬菌体繁殖周期的末期分解细菌肽聚糖[25]。噬菌体的尾纤(tail fiber)或尾刺(tail spikes)蛋白通常展示荚膜解聚糖的活性。在琼脂平板上的细菌菌落层上,这些酶通常会在噬菌斑块周围形成晕圈。在噬菌体vB_KpnP_IME308中预测了3个噬菌体尾部相关蛋白:ORF36和ORF37 (尾管蛋白)及ORF49 (尾丝蛋白),它们主要参与了尾部组装或在感染后穿透宿主细胞外膜的过程。注释结果还显示了ORF34 (主要衣壳蛋白)与Escherichia phage Minorna (MK598851.1)高度相似,同一性为325/338 (96%),E值为0。该衣壳蛋白编码序列高度保守,通常用于分类[26-28]。在噬菌体复制及代谢相关基因中,噬菌体vB_KpnP_ IME308编码几种核苷酸代谢基因,包括核酸外切酶(CDS15,CDS17,CDS25,CDS33)和DNA核酸内切酶(CDS23),这些酶通过水解宿主基因组DNA和RNA产生用于噬菌体DNA合成的脱氧核糖核苷酸[29]。此外,ORF8 (引物酶)和ORF11 (水解酶)在DNA复制、修复和转录中具有重要作用[30]。
|
| 图 3 噬菌体vB_KpnP_IME308功能结构图谱 Figure 3 Genomic map of phage vB_KpnP_IME308 with structural and functional annotation 注:基因从ORF1开始,橙色代表复制模块;绿色代表代谢模块;黄色代表结构和包装模块;红色代表裂解模块;暗红色代表假想蛋白. Note: The genome starts from the upstream of ORF1, the ORF involved in DNA replication is shown in orange, the ORF involved in metabolism is shown in green, and the ORF involved in structure and packing is shown in yellow, red represents the ORF for phage lytic and ORFs without phage related functions are shown in dark red. |
|
|
参与噬菌体包装的蛋白质主要鉴定了ORF32 (脚手架蛋白),其与Klebsiella phage KpV41 (NC_028670.1)具有高度的序列相似性,同一性为273/280 (98%),E值为零;ORF42 (推定的DNA成熟蛋白A)和ORF43 (推定的末端酶大亚基)分别与Klebsiella phage vB_KpnP_KpV74 (KY385423.1)和Klebsiella phage phiBO1E (KM576124.1)具有100/100 (100%)和610/618 (99%)的一致性。
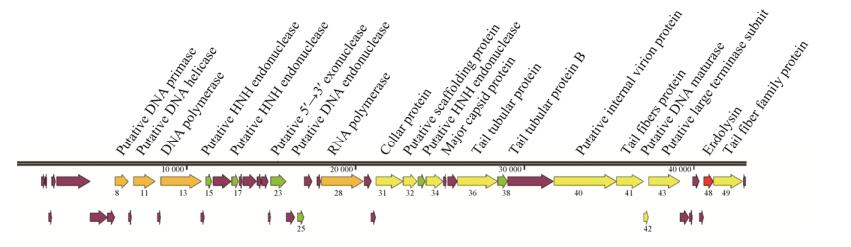
2.7 噬菌体vB_KpnP_IME308比较基因组学分析及系统进化树的构建使用NCBI的BLASTn程序进行全基因组序列分析,噬菌体vB_KpnP_IME308的基因组与vB_KpnP_ KpV74 (KY385423.1)、phage phiKpS2 (KX587949.2)、phage vB_KpnP_SU552A (NC_028870.1)、phage KP34 (NC_013649.2)最为相似。因此,使用Mauve软件首次对这4个基因组进行了比对分析。图 4显示主要在结构和代谢区域发现插入和删除点。例如,发现了CDS15和CDS17的插入,两者均编码推定的HNH核酸内切酶。由噬菌体vB_KpnP_IME308编码的ORF14 (尾纤维蛋白)与宿主特异性相关,因此,在这些噬菌体之间主要显示了较大的差异(覆盖率为48%−100%;同一性为64.6%–79.3%)。

|
| 图 4 噬菌体vB_KpnP_IME308与其近源噬菌体phage phiKpS2、vB_KpnP_KpV74和phage vB_KpnP_SU552A同源性分析 Figure 4 Comparative genome map of phage vB_KpnP_IME308, closely related phage phiKpS2, vB_KpnP_KpV74, and phage vB_KpnP_SU552A |
|
|
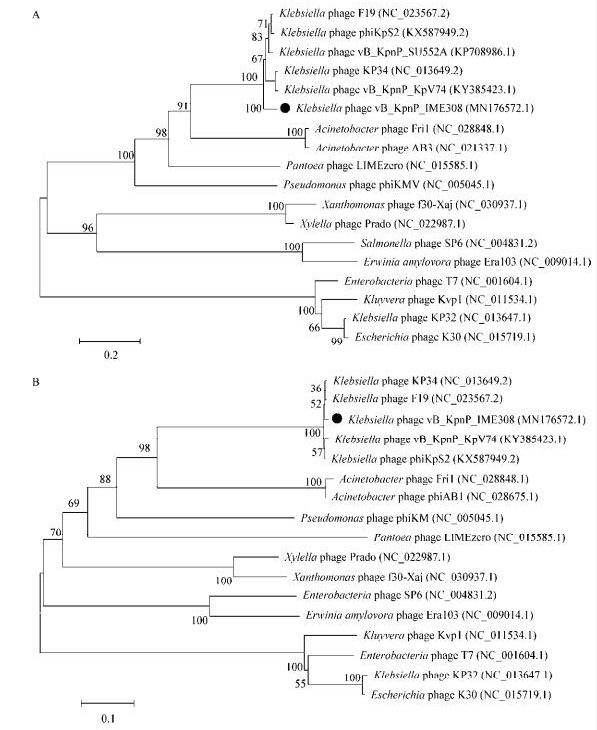
为了说明vB_KpnP_IME308与其他代表性相似噬菌体之间的进化关系,使用噬菌体vB_KpnP_ IME308中具有进化意义的主要衣壳蛋白(ORF34)和推定的末端酶大亚基(ORF43),以及从ICIV数据库获得的其他同源蛋白序列构建了系统发育树。两个进化树结果都显示噬菌体vB_KpnP_IME308与phage KP34(NC_013649.2)、phage F19 (NC_023567.2)、vB_KpnP_KpV74 (KY385423.1)、phage phiKpS2 (KX587949.2)最密切相关并且均属于Autographivirinae亚科的Drulisvirus属(图 5)。
|
| 图 5 使用噬菌体主要衣壳蛋白(A)和推定的末端酶大亚基(B)制作的进化树 Figure 5 Phylogenetics tree were formed by major capsid protein (A) and putative large terminase subnit (B) of phage 注:分支点上的数字代表可信度,数值越接近100,可信度越强;标尺:代表遗传距离,标尺越短代表亲缘关系越近;黑色圆点:vB_KpnP_IME308. Note: The number at the branch point: The confidence level, the closer to 100 the bootstrap value is, the higher confidence level would be; Scale: The genetic distance; Black ball: vB_KpnP_IME308. |
|
|
噬菌体具有特定的杀菌能力及环境多样性,对生态和细菌的进化具有重要影响[31-32]。尽管自20世纪20年代以来噬菌体被认为是潜在的治疗药物[33-35],但这种疗法的发展落后于广泛应用的抗生素[36]。然而,由于多重耐药细菌的全球出现,近年来对噬菌体治疗的研究和兴趣逐渐复兴。
肺炎克雷伯菌是一种临床上重要的机会致病菌,主要引起医院和社区获得性感染,本研究分离了一株新型的肺炎克雷伯菌噬菌体vB_KpnP_IME308。一步生长曲线是定量描述噬菌体生长规律的实验曲线,它可以反映每个噬菌体的3个最重要的特征参数:潜伏期、裂解期和平稳期。基于一步生长动力学曲线,噬菌体vB_KpnP_IME308具有较短的潜伏期和裂解周期,潜伏期和暴发期分别为20 min和80 min。噬菌体vB_KpnP_IME308感染宿主菌的裂解量约为330 PFU/cell。当宿主完全裂解时,溶液中噬菌体的滴度达到最高点或平台。通常,如果此时存在过量的噬菌体并且受感染的细菌被同一噬菌体反复攻击,则噬菌体裂解细菌的触发过程就会受到抑制,细菌就会产生耐受而继续生长。然而,如果在临床环境中重复使用单个噬菌体作为抗菌剂,则可能产生噬菌体抗性。因此,将不同的噬菌体组合成噬菌体混合物可以帮助扩大噬菌体裂解谱,并避免病原菌对单个噬菌体的抗性[37-39]。从这个角度来看,具有相似裂解能力但不同性质和特征的新噬菌体的发现可以促进噬菌体疗法的临床应用。
在本研究中,噬菌体vB_KpnP_IME308的最佳温度确定为4−50 ℃,最合适的pH值范围为5.0–10.0,与我们之前报道的噬菌体vB_SauS_ IMEP5[40]有相似的温度和pH耐受能力。将噬菌体vB_SauS_IMEP5在70 ℃孵育20 min后噬菌体全部失活并在pH 3.0–12.0下稳定。Kęsik-Szeloch等[41]也报道了肺炎克雷伯氏菌噬菌体颗粒在pH 5.0–8.0的范围内相对稳定。在pH 4.0下孵育导致KP16和KP34的噬菌体滴度降低3lg。然而,KP15显示出对高温的相对敏感性,在60 ℃温育10 min后观察到滴度降低了2lg[41]。
综上所述,本实验从解放军总医院第五医学中心污水处理站的污水中分离出一株新型肺炎克雷伯菌噬菌体,命名为vB_KpnP_IME308,通过透射电镜观察,按照噬菌体分类学中的分类标准,判断该噬菌体属于短尾噬菌体科(Podoviridae),进一步进行生物学特性研究发现该噬菌体最佳感染复数为0.001,一步生长曲线显示vB_KpnP_IME308的潜伏期为20 min,暴发期为80 min,暴发量为330 PFU/cell,裂解量大且具有一定的温度和pH稳定性。此外,噬菌体vB_KpnP_IME308裂解酶实验与动物实验也值得深入研究,可为未来使用该噬菌体治疗肺炎克雷伯菌耐药感染奠定基础。
| [1] |
Podschun R, Ullmann U. Klebsiella spp. as nosocomial pathogens:epidemiology, taxonomy, typing methods, and pathogenicity factors[J]. Clinical Microbiology Reviews, 1998, 11(4): 589-603. DOI:10.1128/CMR.11.4.589 |
| [2] |
Daszak P, Cunningham AA, Hyatt AD. Emerging infectious diseases of wildlife– threats to biodiversity and human health[J]. Science, 2000, 287(5452): 443-449. DOI:10.1126/science.287.5452.443 |
| [3] |
Chiu CT, Lin DY, Liaw YF. Metastatic septic endophthalmitis in pyogenic liver abscess[J]. Journal of Clinical Gastroenterology, 1988, 10(5): 524-527. DOI:10.1097/00004836-198810000-00009 |
| [4] |
Lin YT, Siu LK, Lin JC, et al. Seroepidemiology of Klebsiella pneumoniae colonizing the intestinal tract of healthy Chinese and overseas Chinese adults in Asian countries[J]. BMC Microbiology, 2012, 12: 13. DOI:10.1186/1471-2180-12-13 |
| [5] |
Ventola CL. The antibiotic resistance crisis:part 1:causes and threats[J]. P & T, 2015, 40(4): 277-283. |
| [6] |
Cao F, Wang XT, Wang LH, et al. Evaluation of the efficacy of a bacteriophage in the treatment of pneumonia induced by multidrug resistance Klebsiella pneumoniae in mice[J]. BioMed Research International, 2015, 2015: 752930. |
| [7] |
Manohar P, Tamhankar AJ, Lundborg CS, et al. Therapeutic characterization and efficacy of bacteriophage cocktails infecting Escherichia coli, Klebsiella pneumoniae, and Enterobacter species[J]. Frontiers in Microbiology, 2019, 10: 574. DOI:10.3389/fmicb.2019.00574 |
| [8] |
Pan YJ, Lin TL, Chen YH, et al. Capsular types of Klebsiella pneumoniae revisited by wzc sequencing[J]. PLoS One, 2013, 8(12): e80670. DOI:10.1371/journal.pone.0080670 |
| [9] |
Greisen K, Loeffelholz M, Purohit A, et al. PCR primers and probes for the 16S rRNA gene of most species of pathogenic bacteria, including bacteria found in cerebrospinal fluid[J]. Journal of Clinical Microbiology, 1994, 32(2): 335-351. |
| [10] |
Zhao FY, Sun HZ, Zhou XY, et al. Characterization and genome analysis of a novel bacteriophage vB_SpuP_Spp16 that infects Salmonella enterica serovar pullorum[J]. Virus Genes, 2019, 55(4): 532-540. DOI:10.1007/s11262-019-01664-0 |
| [11] |
Anderson TF. Bacteriophages[J]. Annual Review of Microbiology, 1949, 4: 21-34. |
| [12] |
Li F, Xing SZ, Fu KF, et al. Genomic and biological characterization of the Vibrio alginolyticus-infecting "Podoviridae" bacteriophage, vB_ValP_IME271[J]. Virus Genes, 2019, 55(2): 218-226. DOI:10.1007/s11262-018-1622-8 |
| [13] |
Delbrück M. The growth of bacteriophage and lysis of the host[J]. The Journal of General Physiology, 1940, 23(5): 643-660. DOI:10.1085/jgp.23.5.643 |
| [14] |
Wang RH, Xing SZ, Zhao FY, et al. Characterization and genome analysis of novel phage vB_EfaP_IME195 infecting Enterococcus faecalis[J]. Virus Genes, 2018, 54(6): 804-811. DOI:10.1007/s11262-018-1608-6 |
| [15] |
Xing SZ, Zhang XLL, Sun Q, et al. Complete genome sequence of a novel, virulent Ahjdlikevirus bacteriophage that infects Enterococcus faecium[J]. Archives of Virology, 2017, 162(12): 3843-3847. DOI:10.1007/s00705-017-3503-1 |
| [16] |
Chen YB, Guo GH, Sun EC, et al. Isolation of a T7-like lytic Pasteurella bacteriophage vB_PmuP_PHB01 and its potential use in therapy against Pasteurella multocida infections[J]. Viruses, 2019, 11(1): 86. DOI:10.3390/v11010086 |
| [17] |
Ackermann HW. Basic phage electron microscopy[A]//Clokie MRJ, Kropinski AM. Bacteriophages: Methods and Protocols, Volume 1: Isolation, Characterization, and Interactions. New York: Humana Press, 2009: 113-126
|
| [18] |
Lu SG, Le S, Tan YL, et al. Genomic and proteomic analyses of the terminally redundant genome of the Pseudomonas aeruginosa phage PaP1:establishment of genus PaP1-like phages[J]. PLoS One, 2013, 8(5): e62933. DOI:10.1371/journal.pone.0062933 |
| [19] |
Margulies M, Egholm M, Altman WE, et al. Genome sequencing in microfabricated high-density picolitre reactors[J]. Nature, 2005, 437(7057): 376-380. DOI:10.1038/nature03959 |
| [20] |
Aziz RK, Bartels D, Best AA, et al. The RAST Server:rapid annotations using subsystems technology[J]. BMC Genomics, 2008, 9: 75. DOI:10.1186/1471-2164-9-75 |
| [21] |
Lowe TM, Eddy SR. tRNAscan-SE:a program for improved detection of transfer RNA genes in genomic sequence[J]. Nucleic Acids Research, 1997, 25(5): 955-964. DOI:10.1093/nar/25.5.955 |
| [22] |
Darling ACE, Mau B, Blattner FR, et al. Mauve:multiple alignment of conserved genomic sequence with rearrangements[J]. Genome Research, 2004, 14(7): 1394-1403. DOI:10.1101/gr.2289704 |
| [23] |
Tamura K, Stecher G, Peterson D, et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [24] |
Hughes KA, Sutherland IW, Clark J, et al. Bacteriophage and associated polysaccharide depolymerases-novel tools for study of bacterial biofilms[J]. Journal of Applied Microbiology, 1998, 85(3): 583-590. DOI:10.1046/j.1365-2672.1998.853541.x |
| [25] |
Loessner MJ. Bacteriophage endolysins— current state of research and applications[J]. Current Opinion in Microbiology, 2005, 8(4): 480-487. DOI:10.1016/j.mib.2005.06.002 |
| [26] |
Bamford DH, Grimes JM, Stuart DI. What does structure tell us about virus evolution?[J]. Current Opinion in Structural Biology, 2005, 15(6): 655-663. DOI:10.1016/j.sbi.2005.10.012 |
| [27] |
Hambly E, Tétart F, Desplats C, et al. A conserved genetic module that encodes the major virion components in both the coliphage T4 and the marine cyanophage S-PM2[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(20): 11411-11416. DOI:10.1073/pnas.191174498 |
| [28] |
Loessner MJ, Krause IB, Henle T, et al. Structural proteins and DNA characteristics of 14 Listeria typing bacteriophages[J]. Journal of General Virology, 1994, 75(4): 701-710. DOI:10.1099/0022-1317-75-4-701 |
| [29] |
Kropinski AM, Waddell T, Meng JC, et al. The host-range, genomics and proteomics of Escherichia coli O157:H7 bacteriophage rV5[J]. Virology Journal, 2013, 10: 76. DOI:10.1186/1743-422X-10-76 |
| [30] |
Guilliam TA, Keen BA, Brissett NC, et al. Primase-polymerases are a functionally diverse superfamily of replication and repair enzymes[J]. Nucleic Acids Research, 2015, 43(14): 6651-6664. DOI:10.1093/nar/gkv625 |
| [31] |
Labrie SJ, Samson JE, Moineau S. Bacteriophage resistance mechanisms[J]. Nature Reviews Microbiology, 2010, 8(5): 317-327. DOI:10.1038/nrmicro2315 |
| [32] |
Chan BK, Sistrom M, Wertz JE, et al. Phage selection restores antibiotic sensitivity in MDR Pseudomonas aeruginosa[J]. Scientific Reports, 2016, 6: 26717. DOI:10.1038/srep26717 |
| [33] |
Gu JM, Liu XH, Li Y, et al. A method for generation phage cocktail with great therapeutic potential[J]. PLoS One, 2012, 7(3): e31698. DOI:10.1371/journal.pone.0031698 |
| [34] |
Ho K. Bacteriophage therapy for bacterial infections:rekindling a memory from the pre-antibiotics era[J]. Perspectives in Biology and Medicine, 2001, 44(1): 1-16. DOI:10.1353/pbm.2001.0006 |
| [35] |
Summers WC. Cholera and plague in India:the bacteriophage inquiry of 1927–1936[J]. Journal of the History of Medicine and Allied Sciences, 1993, 48(3): 275-301. DOI:10.1093/jhmas/48.3.275 |
| [36] |
Sulakvelidze A, Alavidze Z, Morris Jr JG. Bacteriophage therapy[J]. The British Medical Journal, 1934, 2(3858): 1110. |
| [37] |
Tomat D, Migliore L, Aquili V, et al. Phage biocontrol of enteropathogenic and Shiga toxin-producing Escherichia coli in meat products[J]. Frontiers in Cellular and Infection Microbiology, 2013, 3: 20. |
| [38] |
Chan BK, Abedon ST, Loc-Carrillo C. Phage cocktails and the future of phage therapy[J]. Future Microbiology, 2013, 8(6): 769-783. DOI:10.2217/fmb.13.47 |
| [39] |
Yu L, Wang S, Guo ZM, et al. A guard-killer phage cocktail effectively lyses the host and inhibits the development of phage-resistant strains of Escherichia coli[J]. Applied Microbiology and Biotechnology, 2018, 102(2): 971-983. DOI:10.1007/s00253-017-8591-z |
| [40] |
Zhang Q, Xing SZ, Sun Q, et al. Characterization and complete genome sequence analysis of a novel virulent Siphoviridae phage against Staphylococcus aureus isolated from bovine mastitis in Xinjiang, China[J]. Virus Genes, 2017, 53(3): 464-476. DOI:10.1007/s11262-017-1445-z |
| [41] |
Kęsik-Szeloch A, Drulis-Kawa Z, Weber-Dabrowska B, et al. Characterising the biology of novel lytic bacteriophages infecting multidrug resistant Klebsiella pneumoniae[J]. Virology Journal, 2013, 10: 100. DOI:10.1186/1743-422X-10-100 |
 2020, Vol. 47
2020, Vol. 47