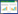扩展功能
文章信息
- 金鸣, 王威, 张雪洪
- JIN Ming, WANG Wei, ZHANG Xue-Hong
- 高产洛蒙真菌素洛蒙德链霉菌高通量筛选方法的建立与复合育种
- High throughput screening and combined breeding of Streptomyces lomondensis for high production of lomofungin
- 微生物学通报, 2020, 47(1): 200-209
- Microbiology China, 2020, 47(1): 200-209
- DOI: 10.13344/j.microbiol.china.190297
-
文章历史
- 收稿日期: 2019-07-17
- 接受日期: 2019-09-09
- 网络首发日期: 2019-06-10
洛蒙德链霉菌(Streptomyces lomondensis) S015由本实验室从植物根际土壤中分离得到,具有广谱抑菌活性并能生物合成洛蒙真菌素(Lomofungin)[1]。洛蒙真菌素的化学结构为6-醛基-4, 7, 9-三羟基-吩嗪-1-甲酸酯,是一种吩嗪类的广谱抗生素,能够抗细菌、真菌及肿瘤[2-4],因此在很多领域都有应用价值。
洛蒙德链霉菌S015生物合成洛蒙真菌素的途径与其他吩嗪类化合物的合成途径非常相似,由分支酸经过核心吩嗪合成基因簇lphzBCDEFG以及其他一系列相关修饰基因等的作用后生成洛蒙真菌素[4]。afsR基因是链霉菌中普遍存在的一个全局调控基因,可以调控很多次级代谢产物的合成,Wang等[1]的研究表明,afsR基因能通过提高吩嗪合成基因phzC和phzE的表达来促进洛蒙真菌素的生物合成。在洛蒙德链霉菌的色氨酸合成途径中,由trpE基因编码的邻氨基苯甲酸合成酶能催化分支酸与氨基反应生成邻氨基苯甲酸[5],因此该途径会消耗洛蒙真菌素合成所需的重要前体——分支酸。
目前高产突变株的诱变手段较多,如常压室温等离子体(atmospheric and room temperature plasma,ARTP)技术利用等离子体射流中富含的化学活性粒子直接作用于细胞遗传物质,可引发种类丰富的DNA损伤,同时具有无污染和操作安全简单的优点,在微生物突变育种和生物医学等领域应用广泛[6-7]。紫外(ultraviolet,UV)诱变则是最为常用的诱变手段,DNA分子经紫外辐射后,形成嘧啶二聚体(cyclobutane pyrimidine dimmer,CPD)影响碱基的正常配对,从而引起突变[8]。微波诱变育种也是常见的育种方法,通过微波辐射,造成细胞内分子间的强烈摩擦,影响DNA分子的空间结构[9]。亚硝基胍(1-methyl-3-nitro-1-nitrosoguanidine,MNNG)是一种传统的化学诱变剂,常被称作超诱变剂,它会促使DNA发生烷基化反应,导致GC和AT之间互换的突变[10]。γ射线(gamma-radiation)在微生物诱变育种中有着广泛的应用,γ射线能够直接使DNA分子中碱基、脱氧核糖等的化学键断裂来破坏DNA螺旋结构[11]。虽然目前诱变手段丰富,设备技术也都比较成熟,但提高筛选效率和建立有效的高通量筛选方法至关重要。
本研究建立了针对洛蒙真菌素的高通量筛选方法,并将其运用于ARTP和紫外复合诱变后的菌株筛选,成功获得了高产菌株;之后通过基因工程改造,阻断了由trpE基因编码的竞争前体分支酸的途径,并过表达全局调控基因afsR,使其产量进一步提升。
1 材料与方法 1.1 材料 1.1.1 菌株、质粒和引物实验所用菌株和质粒及特性见表 1,来源为本实验室保存和本研究构建。研究所用引物委托上海桑尼生物科技有限公司进行合成,序列见表 2。洛蒙德链霉菌S015为诱变出发菌株。
| 菌株和质粒 Strains and Plasmids |
描述 Description |
来源 Source |
| Escherichia coli | ||
| DH5α | For E. coli transformation | TransGen Biotech |
| ET12567(pUZ8002) | For conjugation, KmR, ChlR | [12] |
| Streptomyces lomondensis | ||
| S015 | Wild type | Lab stock |
| M6 | High production mutant | This study |
| M6ΔtrpE1 | M6 with trpE1 deleted | This study |
| M6ΔtrpE2 | M6 with trpE2 deleted | This study |
| M6ΔtrpE1ΔtrpE2 | M6 with trpE1 and trpE2 deleted | This study |
| Plasmids | ||
| pKC1139 | AprR | [13] |
| pKC1139-trpE1 | pKC1139 containing trpE1 upstream and downstream | This study |
| pKC1139-trpE2 | pKC1139 containing trpE2 upstream and downstream | This study |
| pIB139-afsR | pIB139 containing afsR gene | Lab stock |
| 引物名称Primers name | 序列Sequence (5′→3′) | 用途Purpose |
| trpE1-UF | CTAGTCTAGACGGTGCGGTAGAAGCCGTCCT | Clone of trpE1 upstream |
| trpE1-UR | CGCGAACTCGTAGGCCATCCA | |
| trpE1-DF | TGGATGGCCTACGAGTTCGCGGCCTCCGGCATCCCCAAG | Clone of trpE1 downstream |
| trpE1-DR | AAATTTAAGCTTTCCCTGGAGCGTGGCCTTG | |
| trpE2-UF | CTAGTCTAGAGGGGCAGTACAGGCCCGTGTA | Clone of trpE2 upstream |
| trpE2-UR | GTCATGCACATCGTCTCGACGG | |
| trpE2-DF | CCGTCGAGACGATGTGCATGACCTTGCGGGTGACCGGGATGA | Clone of trpE2 downstream |
| trpE2-DR | AAATTTAAGCTTTTCGAGGTGACCACCCACGG |
PrimeSTAR DNA高保真聚合酶、限制性核酸内切酶、EasyPure Genomic DNA快速纯化试剂盒,宝生物工程(大连)有限公司;质粒提取试剂盒、DNA片段纯化试剂盒、胶回收试剂盒,美基生物(中国)有限公司;EasyTaq Mix DNA聚合酶、DNA Marker,北京全式金生物技术有限公司;乙腈、2-丁酮、甲酸、甘露醇、氯化钠、甘油等试剂,国药集团化学试剂有限公司;酵母粉、蛋白胨,Oxoid公司;黄豆饼粉,上海五四有限公司;麦芽提取物,北京索莱宝有限公司。
高效液相色谱分析仪、色谱柱,安捷伦科技(中国)有限公司;常压室温等离子体诱变育种仪,北京思源清生物科技公司;全波长酶标仪,Perkin Elmer公司;恒温振荡培养箱,江苏太仓实验设备厂;96孔PP酶标板,Greiner Bio-One公司;24孔深孔板,海门雷博尔实验器材有限公司。
1.1.3 培养基及培养条件LB培养基(g/L):胰蛋白胨10.0,酵母提取物5.0,氯化钠10.0,固体培养基加入琼脂12.0,1×105 Pa灭菌20 min。所有大肠杆菌在LB培养基中37 ℃培养。
YEME培养基(g/L):葡萄糖4.0,麦芽提取物10.0,酵母提取物4.0,0.68×105 Pa灭菌30 min。洛蒙德链霉菌的发酵在YEME培养基中进行,于28 ℃培养。
MS培养基(g/L):在水中加入黄豆饼粉20.0,煮沸30 min后过滤,再加入甘露醇20.0,琼脂20.0,1×105 Pa灭菌20 min。洛蒙德链霉菌在MS固体平板上28 ℃倒置培养6−8 d可以积累足量孢子。
2×YT孢子预萌发培养基(g/L):氯化钠5.0,酵母提取物10.0,胰蛋白胨15.0,1×105 Pa灭菌20 min。
抗生素:配制母液浓度为50 mg/L的硫酸安普霉素(apramycin,Apr),50 mg/L的卡那霉素(kanamycin,Km),25 mg/L的氯霉素(chloramphenicol,Chl),40 mg/L的萘啶酮酸(nalidixic acid)。使用时按1/1 000的比例加入培养基中。
本研究中洛蒙德链霉菌的培养条件与操作均参照文献[4]。
1.2 方法 1.2.1 高通量筛选及验证方法将经诱变后在MS平板上生长出的单菌落全部接种于每孔含有3 mL YEME培养基的24孔深孔板中,贴上透气封口膜后,置于恒温振荡培养箱中于28 ℃、400 r/min培养60 h。吸取24孔板中各孔的菌液400 μL于96孔深孔板中,并加入每孔400 μL的2-丁酮和2 μL的6 mol/L盐酸,加密封膜后振荡萃取10 min,取每孔200 μL上清液于新的96孔PP酶标板中,使用酶标仪对375 nm处的吸光度值(A375)进行检测。每一轮的诱变共筛选720株突变株,从中选取A375值最大的10株进行摇瓶发酵复筛,用HPLC检测产物,并将洛蒙真菌素的产量最高的菌株作为下一轮诱变的出发菌株。
1.2.2 洛蒙真菌素产量测定发酵液的处理[14]:取培养了4 d后的发酵液2 mL于5 mL离心管中,加入适量的6 mol/L盐酸调节pH至2.0,再加入2 mL的2-丁酮,充分振荡萃取10 min后,以12 000 r/min离心3 min,吸尽上层有机相于干净的1.5 mL离心管中,在真空旋转蒸发仪中35 ℃蒸干后,用1 mL乙腈加1 mL甲酸水(0.1%)充分溶解,使用0.22 μm有机相滤头过滤。
产物的HPLC分析[15]:以0.1%甲酸水溶液(A)和乙腈(B)为流动相,1−4 min,A:B=8:2;4−20 min,A:B=6:4;20−25 min,A:B=8:2。检测波长为270 nm,流速1 mL/min,柱温30 ℃,使用Agilent Eclipse Plus C18色谱柱(250×4.6 mm,5 μm)检测洛蒙真菌素,保留时间为18 min时的产物为洛蒙真菌素。
1.2.3 ARTP和紫外复合诱变(1) ARTP诱变筛选
参考Du、江耀祖等[16-17]的ARTP诱变方法,并结合ARTP诱变育种仪针对孢子的操作要求,具体如下:在超净工作台中刮取并收集MS平板上培养好的洛蒙德链霉菌孢子,在光学显微镜下用血细胞计数板进行孢子浓度的测定,然后用无菌水稀释成106−108 cells/mL的孢子悬液。打开ARTP诱变育种仪,将功率设定为100 W,气体流量为10 SLM,取10μL制备好的孢子悬液涂抹于诱变用的金属载片上,输入相应的处理时间后开始诱变。每次对菌株进行诱变之前,先以处理时间为变量,制作不同处理时间下的诱变致死率曲线,确定该菌株的最佳ARTP诱变处理时间。致死率=(诱变前的孢子总数−诱变后存活的孢子数)/诱变前的孢子总数×100%[17-18]。确定最佳的处理时间后,将处理后的金属载片倒入含有1 mL无菌水的1.5 mL离心管中,充分涡旋后用无菌水稀释100倍,取100 μL均匀涂布与MS平板上,28 ℃培养3 d后,对长出的菌落进行高通量筛选。
(2) 紫外诱变筛选
紫外诱变采取与ARTP诱变筛选保持一致的原则。仍然取10 μL制备好的孢子悬液涂于金属载片上,将金属载片置于30 W紫外灯正下方20 cm处,紫外照射一定时间后,将处理后的金属载片倒入含有1 mL无菌水的1.5 mL离心管中,涡旋稀释100倍后涂布于MS平板上,过程中用锡箔纸将离心管和培养的平板包裹,以避免紫外诱变后的光修复现象。紫外诱变处理时间的确定和高产株的筛选步骤均与ARTP诱变筛选一致。
1.2.4 基因缺失、过表达菌株的构建(1) 构建同源重组质粒pKC1139-trpE1、pKC1139-trpE2
以菌株S015基因组为模板,使用引物trpE1-UF/trpE1-UR和trpE1-DF/trpE1-DR分别扩增trpE1基因上、下游同源臂。PCR反应体系:ddH2O 8.5 μL,DMSO 1.25 μL,上、下游引物(10 μmol/L)各0.75 μL,DNA模板1.25 μL,Primer STAR DNA高保真聚合酶12.5 μL。PCR反应条件:98 ℃ 1 min;98 ℃ 20 s,60 ℃ 45 s,72 ℃ 40 s,30个循环;72 ℃ 10 min。以纯化后的trpE1基因上、下游同源臂为模板,trpE1-UF和trpE1-DR为引物进行融合PCR,扩增trpE1基因上游同源臂与下游同源臂的融合片段。PCR反应体系:ddH2O 8.5 μL,DMSO 1.25 μL,上、下游引物(10 μmol/L)各0.75 μL,DNA模板1.25 μL,Primer STAR DNA高保真聚合酶12.5 μL。PCR反应条件:98 ℃ 1 min;98 ℃ 20 s,60 ℃ 45 s,72 ℃ 90 s,30个循环;72 ℃ 10 min。将融合片段纯化后与pKC1139质粒用Hind III和Xba I双酶切、连接,并转化到大肠杆菌DH5α感受态中。抽提质粒,测序验证后,将重组质粒pKC1139-trpE1转化到穿梭大肠杆菌ET12567(pUZ8002)中。按相同步骤构建pKC1139-trpE2载体,并转化到ET12567中。
(2) 接合转移、双交换突变株的筛选与验证
接合转移的受体菌为M6,供体菌为携带相应载体的穿梭大肠杆菌ET12567(pUZ8002),接合转移、双交换突变株的筛选与验证操作主要参照盛超兰等[19]。
2 结果与分析 2.1 高通量筛选方法的建立 2.1.1 洛蒙真菌素的浓度和A375的关系首先将适量本实验室制备的洛蒙真菌素样品溶解于2-丁酮中,并用盐酸将pH调至2.0,模拟发酵液用2-丁酮萃取后的状态,取200 μL该溶液在酶标仪中进行波长为350−420 nm的吸光度值测定(图 1A),发现375 nm处存在一个吸收峰,所以推测375 nm就是洛蒙真菌素的一个特征吸收波长,处理后的发酵液上层有机相的A375值一定程度上能反映其洛蒙真菌素的含量。
|
| 图 1 洛蒙真菌素的2-丁酮溶液的吸光度值检测 Figure 1 Absorbance detection of lomofungin in methyl ethyl ketone 注:A:配制的含有洛蒙真菌素样品的2-丁酮溶液的吸光度值检测;B:配制的9种不同洛蒙真菌素浓度的2-丁酮溶液的吸光度值检测;C:不同浓度的洛蒙真菌素溶液与375 nm处吸光度值的对应关系及线性拟合图;D:11株不同洛蒙真菌素产量的洛蒙德链霉菌的发酵液处理后上层有机相的吸光度值检测,图例中数字为各样品的实际洛蒙真菌素浓度(mg/L). Note: A: Scanning of lomofungin in methyl ethyl ketone solution; B: Scanning of 9 lomofungin solution; C: Correlation between lomofungin concentration and absorbance at 375 nm; D: Scanning of methyl ethyl ketone extract from 11 samples of fermentation liquor, and the legend shows the lomofungin production (mg/L) of each sample. |
|
|
为验证以上猜想,将洛蒙真菌素样品用2-丁酮配成9种不同浓度的溶液后,用酶标仪进行350−420 nm的吸光度值检测,结果如图 1B所示,可见各曲线在375 nm处确实都有一明显的吸收峰,并且满足洛蒙真菌素浓度越高则A375值越大的规律,说明该条件下吸光度值可以用于检测2-丁酮溶液的洛蒙真菌素的含量。将图 1B中的9种溶液的A375和相应的洛蒙真菌素样品浓度做一散点图,如图 1C所示,进行线性拟合后R=0.85。说明2-丁酮的酸性溶液中A375值的大小能反映其洛蒙真菌素浓度。
为了检验实际洛蒙德链霉菌的发酵液中是否存在其他化合物干扰或掩盖发酵液萃取后的上层有机相的A375值,取11株不同洛蒙真菌素产量洛蒙德链霉菌的4 d后发酵液,按1.2.3中的步骤处理后,再用酶标仪对吸取的上层有机相进行350−420 nm的吸光度测定,结果如图 1D所示,各曲线375 nm处都存在吸收峰,除个别本身洛蒙真菌素浓度非常接近的结果有所差异,整体均满足洛蒙真菌素浓度越高则A375值越大的规律。表明实际情况下,干扰影响可以忽略。
2.1.2 运用A375建立高通量筛选方法因洛蒙真菌素溶液中浓度和A375呈良好的正相关关系,可将洛蒙德链霉菌发酵液处理后的上层有机相的A375值作为快速判断其洛蒙真菌素含量的标志,运用于ARTP、紫外诱变筛选过程中,用24孔深孔板对突变株进行大批量发酵后,按照1.2.1中步骤对发酵液进行批量萃取,将上层有机相转移至96孔酶标板中进行A375值的测定,A375值越高,即代表该突变株的产量越高。
每一轮筛选过程利用24孔深孔板可以同时进行720株突变株的发酵,使用酶标仪能以每分钟约100个菌株样品的速度进行检测,在短时间内完成大量样品的分析,并从中快速筛选出洛蒙真菌素产量较高的突变株进入复筛。
2.2 ARTP、紫外复合诱变及高产菌株筛选 2.2.1 诱变与致死率曲线运用已建立的针对洛蒙真菌素的高通量筛选方法,按照1.2.1中的步骤及操作要求进行ARTP和紫外复合诱变及筛选。按照ARTP诱变2轮紫外诱变2轮,最后再ARTP诱变2轮的顺序进行。每一轮诱变以上一轮诱变的最高产突变株为出发菌株,筛选720株突变株,分3批进行,每一批共培养240株突变株,以24株出发菌株作参照。
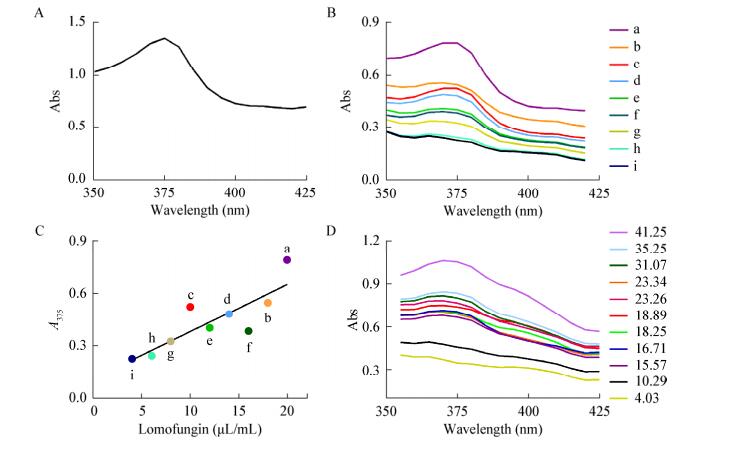
根据文献[17-18]报道及本实验室前期研究,诱变过程中将致死率保持在70%−80%可以获得较高的正突变率。由于诱变轮次较多,以第一轮ARTP诱变的致死率曲线和第一批筛选为例,如图 2A所示,选取60 s作为S015菌株的ARTP诱变处理时间,致死率为79%。确定处理时间后,将诱变后的菌株和这一轮的出发菌株S015接种于24孔深孔板中进行发酵,将发酵液处理后用酶标仪检测A375值,如图 2B所示,选取A375值最高的若干株菌进行摇瓶发酵复筛。
|
| 图 2 致死率曲线和高产菌株筛选 Figure 2 Lethality curve and screening for high-production strains 注:A:S015菌株的ARTP诱变致死率曲线;B:第1批的ARTP诱变后A375检测结果,包含24个S015菌株(灰色)和240个突变株(黑色). Note: A: Lethality curve of S015 by ARTP mutation; B: A375 of mutants in 1st round ARTP mutation breeding, including 24 S015 strains (gray) and 240 mutant strains (black). |
|
|
共经历了6轮ARTP和紫外复合诱变后,累计筛选了4 320株突变株。1−6轮诱变后筛选得到的最高产菌株分别为M1、M2、M3、M4、M5和M6。最终得到的高产株M6的洛蒙真菌素产量为61.33 mg/L,是出发菌株洛蒙德链霉菌S015的7.35倍,如图 3所示。在ARTP和紫外的交替诱变下,从野生株出发筛选获得的菌株其洛蒙真菌素产量逐步提升,说明了ARTP和紫外的复合诱变效果良好,并且也验证了本研究建立的高通量筛选高产洛蒙真菌素菌株的方法切实可行,并且快速高效,为实验节约了大量时间。

|
| 图 3 S015和M1−M6的洛蒙真菌素产量 Figure 3 Lomofungin production of S015 and strain M1−M6 Note: *: P < 0.05; **: P < 0.01. |
|
|
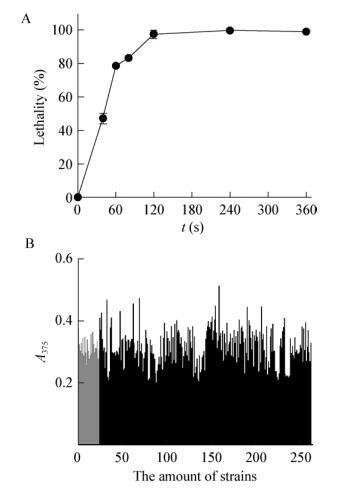
获得了高产株M6之后,不断将其在MS平板上进行划线,传代培养,每隔2 d传代一次,传代30次后对其进行发酵并通过HPLC进行洛蒙真菌素产量的检测。结果如图 4所示,连续传代30次后,M6菌株仍能保持生长的稳定性,菌落形态与S015相比无明显差异,并且洛蒙真菌素的产量仍能维持在60 mg/L左右的水平,可见M6菌株稳定性良好。
|
| 图 4 S015、M6和传代30次M6的洛蒙真菌素产量 Figure 4 Lomofungin production of S015, M6 and M6-30th Note: **: P < 0.01. |
|
|
色氨酸合成途径中,trpE基因编码的邻氨基苯甲酸合成酶能催化分支酸与氨基反应生成邻氨基苯甲酸[5],该反应会消耗洛蒙真菌素合成所需的重要前体分支酸,因此推测将trpE基因敲除可以阻断该途径,可能会使高产株M6在生长过程中积累更多的洛蒙真菌素合成前体分支酸,从而合成更多的洛蒙真菌素。在本实验的前期工作中,已经对洛蒙德链霉菌S015进行了全基因组测序[4],其结果显示,S015菌株中存在2个功能相同的trpE基因序列,为作区分,分别将其命名为trpE1和trpE2。对M6的trpE1和trpE2进行测序,发现未在诱变中发生碱基变化。
前期研究表明,在洛蒙德链霉菌S015中,全局调控基因afsR能通过提高吩嗪合成基因phzC和phzE的表达来促进洛蒙真菌素的生物合成。在突变株M6的基础上进行afsR基因的过表达,验证该调控机制在诱变后的菌株中是否依然可行。
2.3.1 高产菌株M6中trpE1、trpE2基因的敲除在trpE1基因上、下游设计了长度为1 670 bp的上游同源臂和1 544 bp的下游同源臂,同源臂融合片段长度为3 214 bp,预计敲除片段长度为759 bp。按1.2.2中步骤对M6菌株的trpE1进行敲除,以trpE1-UF和trpE1-DR为引物,对筛选到的双交换株的基因组进行PCR验证,预计M6菌株的PCR扩增条带为3 952 bp,trpE1缺失菌株的PCR扩增条带为3 214 bp,PCR验证结果如图 5A所示,与预期相符,然后将样品送公司测序,比对正确则表明trpE1基因敲除成功。
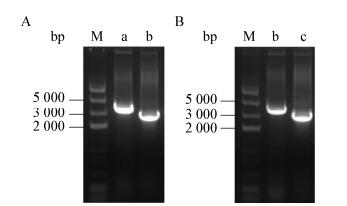
|
| 图 5 M6菌株中trpE1 (A)和trpE2 (B)基因敲除的PCR验证 Figure 5 PCR confirmation of trpE1 (A) and trpE2 (B) gene deletion in M6 strain Note: M: DNA marker; a: M6 strain; b: M6ΔtrpE1 mutant; c: M6ΔtrpE1ΔtrpE2 mutant. |
|
|
同理,在trpE2基因上、下游设计了长度为1 689 bp的上游同源臂和1 573 bp的下游同源臂,同源臂融合片段长度为3 262 bp,预计敲除片段长度为1 044 bp。从M6的trpE1基因敲除株出发,进一步敲除trpE2基因,出发菌株的PCR扩增条带为4 306 bp,trpE2缺失菌株的PCR扩增条带为3 262 bp,PCR验证结果如图 5B所示,与预期相符,然后将样品送公司测序,比对正确则表明trpE2基因敲除成功。
2.3.2 trpE基因敲除株的发酵验证对M6菌株trpE1、trpE2基因的单敲株和双敲株进行发酵,并用HPLC检测洛蒙真菌素的产量,其结果如图 6所示,M6菌株trpE1、trpE2基因单敲株的洛蒙真菌素产量分别为71.38 mg/L和66.17 mg/L,分别比M6菌株提升了18.38%和9.72%;M6菌株trpE1、trpE2基因双敲株的洛蒙真菌素产量为81.89 mg/L,比M6菌株提升了35.21%。发酵结果说明,通过对trpE1、trpE2基因的敲除,能够比较有效地阻断分支酸消耗途径——色氨酸合成途径,从而使代谢流更多地流向洛蒙真菌素合成途径。
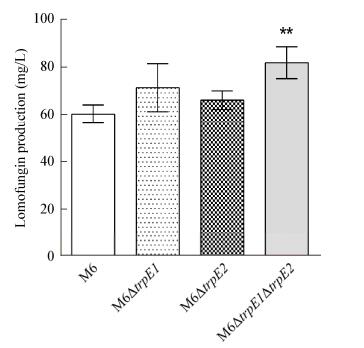
|
| 图 6 trpE1、trpE2基因对M6菌株洛蒙真菌素产量的影响 Figure 6 Effects of trpE1 and trpE2 gene on the lomofungin production of M6 strain Note: **: P < 0.01. |
|
|
前期研究表明,afsR基因能通过提高吩嗪合成基因phzC和phzE的表达来促进洛蒙真菌素的生物合成[1]。因此希望通过在M6ΔtrpE1ΔtrpE2菌株中过表达afsR基因,探究该全局调控因子的调控机制在诱变后的菌株中是否仍然有效,M6菌株是否适合基本的基因工程改造。
在M6菌株的trpE1、trpE2基因双敲株基础上,利用pIB139-afsR过表达质粒进行afsR基因的过表达,并以pIB139空载质粒为参照,发酵结果如图 7所示。在双敲株上过表达全局调控基因afsR后,最终洛蒙真菌素产量达到109.53 mg/L,比双敲株提升了33.75%,是野生型S015的13.13倍。结果表明过表达全局调控基因afsR能有效促进M6ΔtrpE1ΔtrpE2菌株中落蒙真菌素的生物合成,并且该结果与Wang等[1]报道的过表达afsR能提高S015菌株中落蒙真菌素的产量相一致,表明过表达全局调控基因afsR来获得高产菌株这一策略在高产株M6ΔtrpE1ΔtrpE2中仍然有效,M6可以通过基因工程改造不断提高产量。
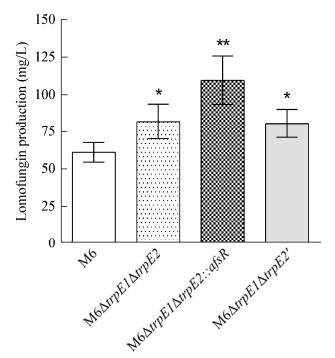
|
| 图 7 afsR基因对M6ΔtrpE1ΔtrpE突变体洛蒙真菌素产量的影响 Figure 7 Effects of afsR gene on the lomofungin production of M6ΔtrpE1ΔtrpE2 mutant 注:M6ΔtrpE1ΔtrpE2′为含有pIB139空质粒的M6ΔtrpE1ΔtrpE2的菌株. *:P < 0.05;**:P < 0.01. Note: M6ΔtrpE1ΔtrpE2′ mutant is M6ΔtrpE1ΔtrpE2 mutant containing vector pIB139. *: P < 0.05; **: P < 0.01. |
|
|
抗生素高产菌株的高通量筛选方法建立对于抗生素高产非常重要,常用的方法如荧光蛋白标记法[20],需要对出发菌株进行基因操作,插入荧光蛋白标记基因;同时,对于某些自带荧光的菌株(如:链霉菌),往往会因其荧光背景干扰而难以奏效。HPLC检测产物也是常用的筛选和检测手段,并具有较高精确性[21],但是HPLC检测单个样品通常就需要10−40 min的时间,在样品数量非常大的情况下会耗费大量的时间。
本研究根据洛蒙真菌素的2-丁酮溶液在紫外波长375 nm处的特征吸收峰,以及其洛蒙真菌素浓度和A375值的正相关关系,利用24孔深孔板的大批量发酵和酶标仪的快速检测,成功建立了高通量筛选高产洛蒙真菌素菌株的方法。每一轮筛选过程利用24孔深孔板,尽管振荡培养箱的空间有限,仍可以实现同时进行720株突变株的发酵;使用酶标仪能以每分钟约100个菌株样品的速度进行检测,在短时间内可以完成样品的快速分析,实现高通量的筛选。Bergy[3]也曾报道洛蒙真菌素的甲醇酸性溶液在375 nm处有一个吸收峰,但因洛蒙真菌素在2-丁酮中溶解度比甲醇更高,峰形清晰,所以选用2-丁酮做溶剂。而且,与荧光蛋白标记法相比,本研究建立的高通量筛选方法无需基因操作,不受菌株自身荧光干扰,过程更为简单有效。与传统的HPLC检测方法相比,本研究建立的高通量筛选方法可以在短时间内对大量样品同时进行检测,更快速高效,可以节约大量时间。
利用高通量筛选方法,通过6轮ARTP和紫外复合诱变,从4 320株突变菌株中筛选得到一株高产菌株M6,洛蒙真菌素产量为61.33 mg/L,是出发菌株S015的7.35倍,并且菌株稳定性良好。Huang等[22]通过紫外和MNNG的复合诱变,使塔宾曲霉(Aspergillus tubingensis) Rn14-88A菌株的果胶酶产量提高了161.44%;Su等[23]通过氮离子束诱变育种,使芽孢杆菌(Bacillus sp.) S65菌株的壳聚糖酶产量从4.1 mg/L提高至25 mg/L,提高了510%。从产量的提升效果和幅度来看,不论相比于传统的紫外、NTG复合诱变,还是新型的氮离子束诱变,本研究的复合诱变与筛选过程对目标产物的产量提升更为可观,具有一定优势。
本研究结合基因工程改造,在诱变高产株M6的基础上双敲除了trpE1和trpE2基因,阻断了洛蒙真菌素的前体分支酸消耗途径——色氨酸合成途径,使得其洛蒙真菌素产量达到81.89 mg/L。在M6的trpE1和trpE2基因双敲株的基础上,又过表达了全局调控基因afsR,最终洛蒙真菌素产量达到109.53 mg/L,是野生型S015的13.13倍,说明诱变菌株仍能适合基本的基因工程改造。
本研究表明将不同诱变育种和基因工程改造相结合可以有效提高目标产物的产量,可为其他抗生素及次生代谢产物的高产菌株筛选与改造提供良好的借鉴。
| [1] |
Wang W, Wang HS, Hu HB, et al. Overexpression of afsR and optimization of metal chloride to improve lomofungin production in Streptomyces lomondensis S015[J]. Journal of Microbiology and Biotechnology, 2015, 25(5): 672-680. DOI:10.4014/jmb.1409.09091 |
| [2] |
Arai J, Goto K, Tanoue Y, et al. Enzymatic inhibition of MICA sheddase ADAM17 by lomofungin in hepatocellular carcinoma cells[J]. International Journal of Cancer, 2018, 143(10): 2575-2583. DOI:10.1002/ijc.31615 |
| [3] |
Bergy ME. Lomofungin, a new broad spectrum antibiotic:isolation and characterization[J]. The Journal of Antibiotics, 1969, 22(3): 126-128. DOI:10.7164/antibiotics.22.126 |
| [4] |
Zhang CX, Sheng CL, Wang W, et al. Identification of the lomofungin biosynthesis gene cluster and associated flavin-dependent monooxygenase gene in Streptomyces lomondensis S015[J]. PLoS One, 2015, 10(8): e0136228. DOI:10.1371/journal.pone.0136228 |
| [5] |
Ashenafi M, Reddy PT, Parsons JF, et al. The fused anthranilate synthase from Streptomyces venezuelae functions as a monomer[J]. Molecular and Cellular Biochemistry, 2015, 400(1/2): 9-15. |
| [6] |
Wang Q, Feng LR, Wei L, et al. Mutation breeding of lycopene-producing strain Blakeslea trispora by a novel atmospheric and room temperature plasma (ARTP)[J]. Applied Biochemistry and Biotechnology, 2014, 174(1): 452-460. DOI:10.1007/s12010-014-0998-8 |
| [7] |
Zong H, Zhan Y, Li X, et al. A new mutation breeding method for Streptomyces albulus by an atmospheric and room temperature plasma[J]. African Journal of Microbiology Research, 2012, 6(13): 3154-3158. |
| [8] |
Ikehata H, Ono T. The mechanisms of UV mutagenesis[J]. Journal of Radiation Research, 2011, 52(2): 115-125. DOI:10.1269/jrr.10175 |
| [9] |
Doran TJ, Lu PJ, Vanier GS, et al. Microwave irradiation enhances gene and oligonucleotide delivery and induces effective exon skipping in myoblasts[J]. Gene Therapy, 2009, 16: 119-126. DOI:10.1038/gt.2008.144 |
| [10] |
Gong CJ, Liu H, Song R, et al. ATR-CHK1-E2F3 signaling transactivates human ribonucleotide reductase small subunit M2 for DNA repair induced by the chemical carcinogen MNNG[J]. Biochimica et Biophysica Acta, 2016, 1859(4): 612-626. DOI:10.1016/j.bbagrm.2016.02.012 |
| [11] |
Harrell CR, Djonov V, Fellabaum C, et al. Risks of using sterilization by gamma radiation:the other side of the coin[J]. International Journal of Medical Sciences, 2018, 15(3): 274-279. DOI:10.7150/ijms.22644 |
| [12] |
MacNeil DJ, Gewain KM, Ruby CL, et al. Analysis of Streptomyces avermitilis genes required for avermectin biosynthesis utilizing a novel integration vector[J]. Gene, 1992, 111(1): 61-68. DOI:10.1016/0378-1119(92)90603-M |
| [13] |
Bierman M, Logan R, O'Brien K, et al. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp.[J]. Gene, 1992, 116(1): 43-49. DOI:10.1016/0378-1119(92)90627-2 |
| [14] |
Li S, Ye QM, Wang W, et al. High-performance liquid chromatography analysis of lomofungin in Streptomyces lomondensis[J]. Journal of Liquid Chromatography & Related Technologies, 2013, 36(15): 2059-2068. |
| [15] |
Yan RB, Wang W, Zhang XH. Function of cutR/cutS two component system in lomofungin biosynthesis in Streptomyces lomondensis S015[J]. Microbiology China, 2019, 46(2): 261-268. (in Chinese) 严若冰, 王威, 张雪洪. 洛蒙德链霉菌S015中cutR/cutS双组分调控系统对洛蒙真菌素合成的调控[J]. 微生物学通报, 2019, 46(2): 261-268. |
| [16] |
Du ZQ, Zhang Y, Qian ZG, et al. Combination of traditional mutation and metabolic engineering to enhance ansamitocin P-3 production in Actinosynnema pretiosum[J]. Biotechnology and Bioengineering, 2017, 114(12): 2794-2806. DOI:10.1002/bit.26396 |
| [17] |
Jiang YZ, Peng HS, Zhang XH. Breeding of Pseudomonas chlororaphis GP72 based on ARTP mutagenesis and high throughput screening[J]. Microbiology China, 2017, 44(10): 2421-2427. (in Chinese) 江耀祖, 彭华松, 张雪洪. 基于ARTP诱变和高通量筛选的绿针假单胞菌GP72育种方法[J]. 微生物学通报, 2017, 44(10): 2421-2427. |
| [18] |
Tan J, Xiong X, Liang WL, et al. Breeding of a phenazine-1-carboxamid-producing strain by ARTP mutation and its optimization of fermentation[J]. Biotechnology Bulletin, 2016, 32(1): 174-179. (in Chinese) 谭剑, 熊欣, 梁万利, 等. ARTP技术选育吩嗪-1-甲酰胺高产菌株及发酵优化[J]. 生物技术通报, 2016, 32(1): 174-179. |
| [19] |
Sheng CL, Wang W, Hu HB, et al. Function of a methyltransferase gene lomo3 involved in the biosynthesis of lomofungin[J]. Microbiology China, 2016, 43(3): 575-582. (in Chinese) 盛超兰, 王威, 胡洪波, 等. 一个甲基转移酶基因lomo3在洛蒙真菌素生物合成途径中的功能[J]. 微生物学通报, 2016, 43(3): 575-582. |
| [20] |
Knapp A, Ripphahn M, Volkenborn K, et al. Activity-independent screening of secreted proteins using split GFP[J]. Journal of Biotechnology, 2017, 258: 110-116. DOI:10.1016/j.jbiotec.2017.05.024 |
| [21] |
Ganzera M, Sturm S. Recent advances on HPLC/MS in medicinal plant analysis-an update covering 2011-2016[J]. Journal of Pharmaceutical and Biomedical Analysis, 2018, 147: 211-233. DOI:10.1016/j.jpba.2017.07.038 |
| [22] |
Huang DM, Song YY, Qin LY, et al. A new strain of Aspergillus tubingensis for high-activity pectinase production[J]. Brazilian Journal of Microbiology, 2019, 50(1): 53-56. DOI:10.1007/s42770-018-0032-3 |
| [23] |
Su CX, Zhou W, Fan YH, et al. Mutation breeding of chitosanase-producing strain Bacillus sp. S65 by low-energy ion implantation[J]. Journal of Industrial Microbiology and Biotechnology, 2006, 33(12): 1037-1042. DOI:10.1007/s10295-006-0155-7 |
 2020, Vol. 47
2020, Vol. 47