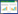扩展功能
文章信息
- 刘冲冲, 冯声宝, 吴群, 黄和强, 陈占秀, 李善文, 徐岩
- LIU Chong-Chong, FENG Sheng-Bao, WU Qun, HUANG He-Qiang, CHEN Zhan-Xiu, LI Shan-Wen, XU Yan
- 青稞酒发酵过程中的风味功能微生物及其风味代谢特征解析
- Flavor-related microbiota and their flavor metabolism during highland barley Baijiu fermentation
- 微生物学通报, 2020, 17(1): 151-161
- Microbiology China, 2020, 17(1): 151-161
- DOI: 10.13344/j.microbiol.china.190240
-
文章历史
- 收稿日期: 2019-03-26
- 接受日期: 2019-05-10
- 网络首发日期: 2019-06-20
2. 青海互助青稞酒股份有限公司 青海 互助 810500;
3. 青青稞酒国际酿酒生物技术综合研究所 江苏 无锡 214122
2. Qinghai Huzhu Barley Wine Co. Ltd., Huzhu, Qinghai 810500, China;
3. Joint International Integrated Laboratory for Biotechnology of Qinghai Qingke Jiu Making, Wuxi, Jiangsu 214122, China
多菌种固态发酵是一种由可重复的微生物菌群驱动的自然发酵过程,是生产传统发酵食品的重要技术[1],是最古老、最实惠的生产和保存食品的方式之一,对于改善食品的营养价值、口感和健康功能具有重要作用[2]。风味物质作为发酵食品的重要组成部分,主要与发酵食品生态系统中的微生物群落代谢相关。鉴定发酵食品中风味功能微生物并验证其功能,对了解发酵食品中多菌种固态发酵的机制具有重要作用。
微生物群落在白酒发酵过程中扮演着重要的角色,越来越多的研究集中于白酒微生物群落的动力学特征、组装机制以及群落代谢功能[3-4],基于多组学技术寻找发酵过程中的核心微生物菌群是一种常用的分析方法。Hu等在研究浓香型白酒窖泥的细菌群落过程中,将平均相对丰度 > 1%并且至少在多个样品中出现的17个微生物属判定为核心微生物[5]。Wang等结合微生物群落与挥发性化合物轮廓关联分析和微生物共现性网络分析判定了浓香型白酒中包含10个微生物属的核心微生物群[6]。Song等基于多组学揭示了酱香型白酒发酵过程中包含10个真菌属和11个细菌属的核心微生物群[7]。以上研究揭示了白酒发酵过程中具有功能的微生物菌群,这对于了解微生物群落在发酵食品中的角色和作用提供了重要的信息。然而,基于多组学和统计学判定的核心微生物只局限于微生物的属水平,无法准确选择核心微生物菌株以及还原原位系统的代谢功能。因此,有必要通过可培养的方法获取白酒发酵过程中的核心微生物,并通过体外发酵实验探究微生物的功能。
青稞酒作为典型的清香型白酒之一,主要分布在我国的西北部地区,由于其独特的酿造原料、发酵条件和“清蒸清烧四次清”的生产工艺,形成了有别于其他清香型白酒的独特风格[8]。白酒中的风味物质主要是由发酵过程中微生物群落代谢而成,因此,探究青稞酒发酵过程的微生物群落组成和功能有助于解析青稞酒的风味信息。目前对青稞酒的研究主要集中于酒曲微生物的组成和功能[9-10],对发酵过程中具有重要功能的优势微生物以及影响其演替的重要理化因子尚不清楚。因此本文以青稞酒为研究载体,基于扩增子测序结合风味分析,以及统计分析揭示青稞酒发酵过程中的风味功能微生物菌群并判定影响其演替的关键因子,并通过体外系统进行验证,以期为青稞酒发酵过程的风味调控提供新的视角。
1 材料与方法 1.1 酒醅样品发酵酒醅样品从青海互助青稞酒股份有限公司采集,分别从3个独立的窖池(2.6×2.4×1.9 m)发酵0、5、10、15、20和30 d取样,共计18份。取样时间为2017年5月。每份样品分别取3个平行样,采集完立即密封。一份保存在4 ℃用于微生物的筛选,另一份存在−20 ℃用于酒醅样品的理化指标检测和物质含量分析,第3份立即在液氮中冷冻,然后通过干冰保存于−80 ℃用于提取DNA。
1.2 主要试剂和仪器及培养基基因组DNA提取试剂盒(E.Z.N.A.® Soil DNA Kit),Omega公司;研究中所有试剂均为分析纯,国药(集团)上海化学试剂有限公司。核酸浓度检测仪和荧光定量PCR仪,赛默飞世尔科技有限公司;细胞破碎仪,上海玉博生物科技有限公司;液相色谱仪,Waters公司;气相色谱-质谱联仪,安捷伦公司。
WL培养基(g/L):酵母浸出粉4.0,磷酸二氢钾0.55,硫酸镁0.125,葡萄糖50.0,氯化钾0.425,氯化铁0.002 5,蛋白胨5.0,氯化钙0.125,溴甲酚绿0.022,硫酸锰0.002 5,最终pH为6.5。孟加拉红培养基(g/L):孟加拉红0.03,硫酸0.5 mL,葡萄糖10.0,蛋白胨5.0,氯霉素0.1 mL,最终pH为7.2±0.2。MRS培养基(g/L):蛋白胨10.0,酵母膏4.0,牛肉膏8.0,葡萄糖20.0,柠檬酸氢二胺2.0,吐温-80 1.0 mL,醋酸钠5.0,硫酸镁0.2,磷酸氢二钾2.0。
1.3 菌株及其筛选菌株Lactobacillus acetotolerans B1、Weissella viridescens W1、Saccharomyces cerevisiae QK1、Komagataella phaffii QK2、Aspergillus niger M1和Pichia fermentans PF均从青稞酒发酵过程的酒醅中筛选获得。准确称取5.0 g酒醅样本,加入装有50 mL 0.85%无菌生理盐水的三角瓶中,于37 ℃、200 r/min培养30 min,吸取0.1 mL样品,梯度稀释10−3、10−4、10−5、10−6四个不同的数量级,各取100 µL酒醅稀释液涂布WL培养基、孟加拉红培养基、MRS培养基,涂布后的WL平板和孟加拉红平板放入30 ℃恒温培养箱中培养1−2 d,MRS平板放入37 ℃恒温培养箱中培养1−3 d。待长出单菌落后,用灭菌的10 µL枪头挑取形态差异的菌落至对应的液体培养基中进行分离纯化。将培养好的菌液进行基因组提取,分别用真菌的26S rRNA基因通用引物(NL1:5′-GCATATCAATAAGCGGA GGAAAAG-3′;NL4:5′-GGTCCGTGTTTCAAGA CGG-3′)和细菌的16S rRNA基因通用引物(27F:5′-AGAGTTTGATCCTGGCTCA-3′;1492R:5′-GGT TACCTTGTTACGACTT-3′)进行下一步PCR和测序,测序结果进行BLAST比对,对菌株进行鉴定。
1.4 理化因子和乙醇的检测将3个温度计插入窖池约1 m深处来实时检测酒醅温度。准确称量10.0 g样品,于105 ℃下干燥4 h以确定其重量损失来测量酒醅的水分含量。采用滴定法(0.1 mol/L标准氢氧化钠溶液滴定并添加酚酞作为指示剂)来测定酒醅的酸度。酒醅样品的还原糖含量通过3, 5-二硝基水杨酸(3, 5-dinitrosalicylic acid,DNS)法测定,酒醅的乙醇含量采用液相色谱仪测定[11]。
1.5 酒醅风味成分检测称取酒醅5.0 g置于50 mL离心管中,添加20 mL去离子水,充分振荡后在0 ℃超声处理30 min,然后在4 ℃、8 000×g离心5 min。吸取8 mL上清液和20 μL薄荷醇内标(100 μg/mL)置于含有3.0 g NaCl的20 mL顶空瓶中。酒醅风味物质检测采用顶空固相微萃取结合气相色谱-质谱联仪,色谱柱为DB-Wax,检测条件参考文献[12]。
1.6 DNA提取和高通量测序称取7.0 g酒醅样品,根据E.Z.N.A.® Soil DNA Kit的操作说明来提取各样品中的DNA。对于真菌,扩增区域为ITS2区域,引物为ITS1和ITS2;对于细菌,扩增区域16S rRNA基因的V3−V4区域,引物为338F和引物806R[13]。PCR反应体系(25 μL):10×Pyrobest Buffer 2.5 μL,dNTPs (2.5 mmol/L) 2 μL,上、下游引物(10 μmol/L)各1 μL,Pyrobest DNA Polymerase 0.4 μL,模板DNA 15 ng,ddH2O补足到25 μL。真菌PCR反应条件:95 ℃ 2 min;95 ℃ 30 s,61 ℃ 30 s,72 ℃ 45 s,共32个循环;72 ℃ 10 min。细菌PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 40 s,共25个循环;72 ℃ 10 min。PCR产物的纯化参考文献[14]。纯化后的产物通过NanoDrop ND-1000 UV-Vis进行定量,然后将其与PCR产物等分子量混合,再根据Low Sample Protocol进行制备文库,最后在Illumina MiSeq测序平台上测序。将测序的原始数据提交至DDBJ (DNA Data Bank of Japan)数据库,编号为DRA008152和DRA008153。
1.7 原始序列的处理通过QIIME pipeline对原始序列进行处理。先将序列进行归类,去除标签、引物和接头序列;将长度小于110 bp、模糊碱基大于2 bp和平均质量小于30分的序列进行去除;通过UCHIME去除嵌合体得到优质序列;使用UCLUST把优质序列以97%的相似度进行可操作分类单元(operational taxonomic unit,OTU)划分,并计算Chao1和Shannon指数[15]。
1.8 模拟发酵试验及荧光定量PCR (qPCR)6株风味功能微生物Lactobacillus acetotolerans B1、Weissella viridescens W1、Saccharomyces cerevisiae QK1、Komagataella phaffii QK2、Aspergillus niger M1和Pichia fermentans PF均从青稞酒发酵过程的酒醅中筛选获得。通过特异性引物(表 1)对0 d的酒醅样本进行荧光定量PCR,PCR体系:灭菌超纯水8.2 μL,SYBR Green Supermix 10.0 μL,上、下游引物(20 μmol/L)各0.4 μL,DNA模板1.0 μL。L. acetotolerans B1和W. viridescens W1的PCR反应条件:95 ℃ 10 min;95 ℃ 5 s,60 ℃ 30 s,72 ℃ 45 s,40个循环;72 ℃梯度升温至95 ℃。S. cerevisiae QK1、K. phaffii QK2、P. fermentans PF和A. niger M1的PCR反应条件:98 ℃ 3 min;98 ℃ 30 s,55 ℃ 30 s,65 ℃ 45 s,40个循环;65 ℃梯度升温至95 ℃。得到6株菌在0 d酒醅中的生物量:S. cerevisiae QK1:4.43 lgcopies/g;K. phaffii QK2:4.97 lgcopies/g;P. fermentans PF:4.76 lgcopies/g;A. niger M1:4.66 lgcopies/g;L. acetotolerans B1:0.11 lgcopies/g;W. viridescens W1: 1.78 lgcopies/g。
| Amplicon | Primer direction | Primers sequence (5ʹ→3ʹ) | The calibration curves |
| QK1 | F | GGACTCTGGACATGCAAGAT | y=−0.288 5x+11.972 |
| R | ATACCCTTCTTAACACCTGGC | R=0.997 | |
| QK2 | F | TCGTTCATGGCAAGTTTCCG | y=−0.451 7x+14.524 |
| R | TTGCGGAACCCTCTTGCTTA | R=0.996 7 | |
| PF | F | CGGTAGACCAAGACACC | y=−0.367x+13.884 |
| R | TGTAGAAGGTGTGGTGC | R=0.998 8 | |
| M1 | F | CACTAACACCCCCATCCAGG | y=−0.433 7x+15.359 |
| R | ATGGTGTGATCGGGATGTCG | R=0.994 4 | |
| B1 | F | TTCCTCCTGTTGGCGTTT | y=−0.304 3x+11.285 |
| R | TGCTGAAATGGTGGCAATAC | R=0.999 6 | |
| W1 | F | TTGAATGACCCACAAGCGGT | y=−0.358 1x+12.004 |
| R | ATCGCACCCTGATCGTCTTC | R=0.991 9 | |
| 注:x:Ct值;y:lgcopies/g. Note: x: Ct value; y: lgcopies/g. |
|||
将6株菌活化培养,然后根据菌株在原位体系发酵0 d的生物量接种至固态培养基中(蒸煮并灭菌的青稞)。密封后在30 ℃恒温培养箱中发酵30 d,每个实验3个生物平行。分别在发酵0、5、10、15、20和30 d进行取样,并根据特异性引物对6株菌的生物量进行跟踪。模拟发酵终点的风味信息采用GC-MS检测。
1.9 数据分析使用R语言(V3.2.4)中的Vegan包,计算酒醅发酵过程中理化因子对微生物群落结构的影响,同时进行蒙特卡洛检验;为发现酒醅发酵过程中风味化合物的动态变化规律,使用R语言(V3.2.4)中的Complex heatmap包进行数据标准化和绘制热图;使用SPSS 19.0计算酒醅发酵过程中风味化合物和微生物的Sperman相关系数ρ,并用Gephi 0.9.1对ρ > 0.6且P < 0.05的数据可视化成共现网络。
2 结果与分析 2.1 理化因子分析青稞酒发酵过程中酒醅理化因子变化趋势分析如图 1所示。温度从20.0 ℃ (0 d)急剧上升至35.3 ℃ (15 d),然后缓落至32.8 ℃ (30 d);水分含量上,基本保持在40%−45%;还原糖含量呈现先上升后下降的趋势,在发酵5 d达到最高值22.36 mg/g;酸度趋于稳步上升的态势,从0.97 (0 d)上升至3.30 (30 d);乙醇含量随着发酵时间的推移不断增加,在发酵20 d达到最高值,为32.61 mg/g。

|
| 图 1 发酵过程中酒醅温度、水分(A)和还原糖、酸度、乙醇(B)的变化 Figure 1 Dynamics of temperature, moisture (A), reducing sugar, acidity and ethanol (B) during the fermentation process |
|
|
采用高通量测序技术来揭示发酵过程酒醅中的微生物群落组成。对于细菌群落,共获得高质量序列201 347条,平均每个酒醅样本含有33 558±4 075条;根据97%的序列相似度对序列进行OTU划分,共获得597个OTU,平均每个酒醅样本含有100±29个OTU。对于真菌群落,共获得高质量序列209 794条,平均每个酒醅样本含有34 966±7 202条;共获得668个OTU,平均每个酒醅样本含有111±16个OTU。总体而言,每个酒醅样品的覆盖率(coverage)均在99%以上,表明测序的深度可以准确反映青稞酒发酵过程微生物群落的真实情况。细菌群落的Chao1指数和Shannon指数均呈现先上升后下降的态势,且在10 d达到最高值,分别为152.8±6.1和4.29±0.29;在30 d达到最低值,分别为53.6±12.4和0.86±0.15,说明青稞酒发酵过程中细菌群落组成到发酵后期趋于单一。真菌群落的Chao1指数和Shannon指数在发酵过程呈现起伏的态势,但是在入池(0 d)和出池(30 d)均没有显著差异(P > 0.05),说明青稞酒发酵过程真菌群落组成相对稳定(表 2)。
| Time (d) | 16S rRNA gene | ITS2 DNA | |||||||||
| Reads | OTUs | Coverage (%) | Chao1 | Shannon | Reads | OTUs | Coverage (%) | Chao1 | Shannon | ||
| >0 | >33 160 | >98 | >99.86 | >100.2 | >2.66 | >26 482 | >119 | >99.84 | >137.2 | >2.98 | |
| >5 | >27 387 | >101 | >99.85 | >107.9 | >3.53 | >39 166 | >128 | >99.84 | >135.7 | >3.13 | |
| >10 | >31 040 | >146 | >99.84 | >152.8 | >4.29 | >23 532 | >98 | >99.86 | >113.7 | >2.88 | |
| >15 | >34 845 | >107 | >99.81 | >114.5 | >2.15 | >41 693 | >110 | >99.88 | >115.3 | >3.27 | |
| >20 | >40 847 | >99 | >99.83 | >93.5 | >1.41 | >37 850 | >85 | >99.90 | >97.0 | >2.51 | |
| >30 | >34 068 | >46 | >99.91 | >53.6 | >0.86 | >41 071 | >128 | >99.87 | >130.3 | >2.89 | |
统计分析青稞酒发酵过程中微生物群落,在微生物属水平上,共获得9个优势真菌属和8个优势细菌属(相对丰度 > 1%)。其中Pichia (24.73%−49.43%)、Hyphopichia (3.21%−29.24%)、Komagataella (0.99%−39.86%)、Aspergillus (0.32%−9.38%)、Saccharomyces (2.10%−11.47%)和Candida (0.58%−7.73%)在整个青稞酒发酵过程具有一定的优势(图 2A)。对于细菌属,在发酵0 d占优势的是Weissella (21.79%)、Pantoea (47.91%)、Acinetobacter (7.95%)和Staphylococcus (13.19%),但是从发酵5 d开始Lactobacillus迅速从25.80%增加到99.11% (30 d),取代其他微生物成为细菌群落的优势微生物(图 2B)。
|
| 图 2 发酵过程中微生物群落结构在属水平的分布变化 Figure 2 Distribution of microbial community at the genus level in fermented grains during the fermentation process 注:A:真菌;B:细菌. Note: A: Fungi; B: Bacteria. |
|
|
采用HS-SPME-GC-MS分析在青稞酒发酵过程中共检测到53种风味物质,包括2种酸类、14种醇类、4种芳香类、11种醛酮类、20种酯类以及2种其他类物质(图 3A)。基于相对含量标准化(z-score)热图可以发现,风味物质的代谢大致可以分为两个阶段(发酵前中期:0−15 d;发酵后期:20−30 d)。醇类物质在发酵前中期相对含量较高,而酸类和酯类物质在发酵后期相对含量较高,芳香类和醛酮类物质的相对含量在发酵过程则较为平均。

|
| 图 3 发酵过程中风味变化热图(A)以及微生物与风味的相关性网络图(B) Figure 3 Heatmap of change in flavors during the fermentation process (A) and correlation network between microbial genera and flavors (B) 注:黑色圆点表示微生物属,灰色圆点表示风味物质;通过z-score来标准化数据. Note: Black dots indicate microbial genera and gray dots indicate flavors. Data were processed by z-score transformation. |
|
|
为了揭示青稞酒发酵过程中的风味功能微生物菌群,选取17个优势微生物属(8个细菌属和9个真菌属,相对丰度 > 1%),计算其与53种风味物质之间的Spearman相关系数(ρ),选取ρ > 0.6且具有显著性的关联作为有效的连接来绘制相关性网络图(图 3B)。结果表明,优势微生物属中具有较大连接数的为Aspergillus (12)、Komagataella (22)、Lactobacillus (29)、Pichia (24)、Saccharomyces (16)和Weissella (17),其中Saccharomyces主要与醇类物质显著正相关,Komagataella主要与酯类物质显著正相关,Pichia主要与醇类和醛酮类物质显著正相关,Lactobacillus则主要与酸类和脂类物质显著正相关。以上结果表明,在青稞酒发酵过程中,主要的风味功能微生物菌群包含Aspergillus、Komagataella、Lactobacillus、Pichia、Saccharomyces和Weissella。
2.4 理化因子对风味功能微生物菌群的影响为探究发酵过程中的理化因子对风味功能微生物菌群的影响,使用R语言中的vegan包进行蒙特卡洛置换检验。结果表明,对风味功能微生物菌群的演替具有重要影响的理化因子是还原糖(r2=0.946 9,P=0.013 2)和酸度(r2=0.847 6,P=0.048 6),这两个因素与风味功能微生物菌群的分布具有显著的相关性(P < 0.05) (表 3)。此外,单个理化因子与风味功能微生物的Spearman相关系数的结果表明,还原糖除了与Lactobacillus具有显著(P < 0.05)负相关外,与其他风味功能微生物都具有正相关,且与Komagataella和Pichia具有显著(P < 0.05)正相关;Lactobacillus与酸度具有显著(P < 0.01)正相关,说明青稞酒发酵过程中Lactobacillus是重要的产酸微生物属;而乙醇与Saccharomyces具有显著(P < 0.05)正相关,说明Saccharomyces在青稞酒发酵过程中是重要的产乙醇微生物属(表 4)。
| Mantel test | r2 | P |
| Reducing sugar (mg/g) | 0.946 9 | 0.013 2 |
| Temperature (℃) | 0.732 5 | 0.132 6 |
| Moisture (%) | 0.821 5 | 0.080 6 |
| Acidity (0.1 mol/g) | 0.847 6 | 0.048 6 |
| Ethanol (mg/g) | 0.489 4 | 0.070 1 |
| Genus | Reducing sugar (mg/g) | Temperature (℃) | Moisture (%) | Acidity (0.1 mol/g) | Ethanol (mg/g) |
| Lactobacillus | −0.886* | 0.600 | 0.943** | 1.000** | −0.086 |
| Weissella | 0.371 | −0.429 | −0.771 | −0.886* | −0.657 |
| Pichia | 0.886* | 0.429 | 0.771 | 0.714 | 0.543 |
| Komagataella | 0.814* | 0.543 | 0.543 | 0.600 | 0.771 |
| Aspergillus | 0.429 | 0.429 | 0.257 | 0.314 | 0.314 |
| Saccharomyces | 0.629 | −0.371 | −0.200 | −0.086 | 0.886* |
| 注:**和*分别表示P < 0.01和P < 0.05. Note: ** and * indicate P < 0.01 and P < 0.05, respectively. |
|||||
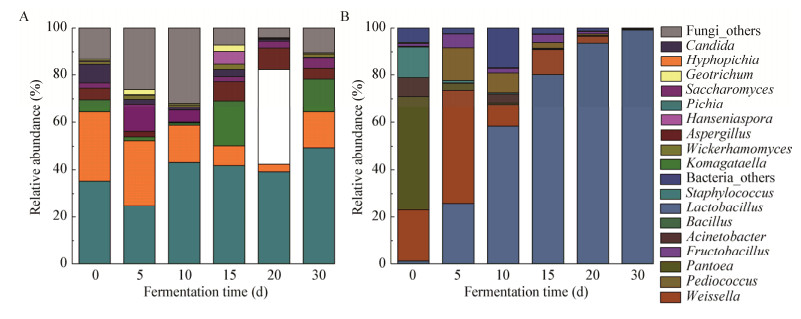
为探究风味功能微生物菌群在体外系统和原位系统中风味代谢的差异,从青稞酒发酵过程中的酒醅筛选到风味功能微生物属对应的菌株,分别为L. acetotolerans B1、W. viridescens W1、S. cerevisiae QK1、K. phaffii QK2、A. niger M1和P. fermentans PF。在实验室环境中将6株菌按照原位系统的生物量进行组合发酵实验,并基于特异性引物的荧光定量PCR进行目的菌的生物量跟踪。K. phaffii QK2、P. fermentans PF和S. cerevisiae QK1的生物量均从发酵0 d至5 d呈现增加的态势,而发酵5 d之后K. phaffii QK2的生物量从4.779 1± 0.166 8 lgcopies/g减至1.324 5±0.307 4 lgcopies/g;P. fermentans PF的生物量从6.028 3±0.514 7 lgcopies/g减至3.476 3±0.023 5 lgcopies/g;S. cerevisiae QK1的生物量从5.602 8±0.266 6 lgcopies/g减至2.646 0±0.432 7 lgcopies/g (图 4A)。A. niger M1的生物量变化分为两个阶段(0−10 d和15−30 d),两个阶段的生物量分别维持在4.772 2 lgcopies/g和4.166 8 lgcopies/g (图 4B)。W. viridescens W1的生物量则在发酵0−10 d呈现上升态势,10 d之后开始逐渐下降;而L. acetotolerans B1的生物量从发酵0 d至发酵结束一直呈现上升的趋势,最后增加至5.054 9±0.212 1 lgcopies/g (图 4C)。从6株风味功能微生物的体外发酵实验可以发现,在发酵前期占据主导地位的是K. phaffii QK2、P. fermentans PF、S. cerevisiae QK1和A. niger M1,而发酵后期占据绝对优势地位的是L. acetotolerans B1,说明体外系统与原位系统具有同样的微生物演替现象。
|
| 图 4 模拟发酵过程中各微生物的生物量变化 Figure 4 Dynamics of biomass of microbes during simulated fermentation process 注:A:酵母;B:霉菌;C:细菌. Note: A: Yeast; B: Mold; C: Bacteria. |
|
|
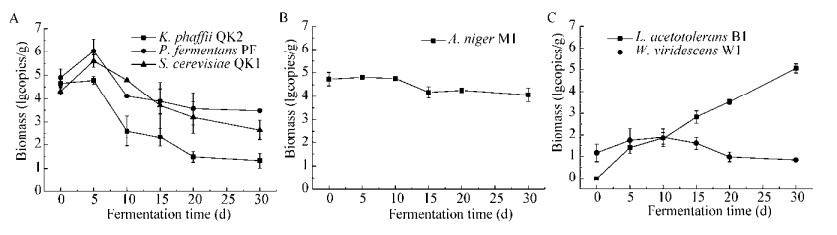
体外系统发酵终点样本中共鉴定到45种风味物质,包含4种酸类、11种醇类、2种芳香类、8种醛酮类、16种酯类以及其他类物质。其中两种酸类物质(3-甲基-丁酸和2-甲基-丙酸)以及两种醇类物质(3-甲基-1-丁醇和1-乙烯基己醇)在原位系统中未检测到,而其他类别物质均在原位系统中被检测到(图 5A)。对于风味物质含量,不同类别的风味表现出类似的比例,其中酯类物质和醇类物质的比例之和在两个系统中均超过了80%,这些结果表明被挑选出来的风味微生物在一定程度上具有重新构造原位体系风味轮廓的功能(图 5B)。
|
| 图 5 体外系统和原位系统中的风味维恩图(A)和发酵终点的风味信息比较(B) Figure 5 The venn diagram of flavors (A) and comparison of flavor information (B) between in vitro and in situ at the end of fermentation |
|
|
白酒酿造微生物群落对于白酒的质量具有重要意义。目前许多生物学方法已被用来表征白酒发酵过程中的细菌和真菌群落[16-17]。在这项研究中,采用高通量测序发现了8个优势细菌属和9个优势真菌属,与其他清香型白酒相比揭示了新的优势真菌属[18]。此外,本文揭示了青稞酒发酵过程中具有风味功能的6个微生物属,其中Lactobacillus作为丰度最高的原核微生物,为乳酸乙酯提供前体物质,提升白酒的醇厚感[12];Pichia作为丰度最高的真核微生物,可以合成多种酚类物质及其前体物质[19],此外还可以合成3-甲基丁醛、2-苯乙醇、苯乙酸乙酯和2, 3-丁二酮[20];Saccharomyces是乙醇的主要产生微生物,还可以产生大量高级醇和大量酯类物质[21];Aspergillus主要产生许多糖化酶,用以分解淀粉[22-23]。在其他酒精饮料中也存在风味功能微生物,Lactobacillus、Saccharomyces和Candida被发现在浓香型白酒发酵过程中是重要的风味物质贡献者[6];Wang等通过微生物对风味物质的贡献程度,判定Lactobacillus、Saccharomyces、Pichia、Geotrichum和Candida为汾酒发酵过程中的风味功能微生物[24];Huang等通过双向正交偏最小二乘分析判定了米酒发酵过程的风味功能微生物为Gluconacetobacter、Lactobacillus、Lactococcus、Pichia、Wickerhamomyces和Saccharomyces[25]。这些研究表明风味功能微生物在不同酒精饮料中具有相似的风味贡献能力。此外,本研究发现Komagataella在青稞酒发酵过程中与一些酯类物质显著正相关,Weissella与一些醛酮类物质显著正相关。通过体外系统的模拟组合发酵实验,初步解析了青稞酒发酵过程中风味功能微生物的风味代谢特征,下一步研究中可从单菌风味代谢和单菌强化两个方面进一步探究风味功能微生物的功能,为青稞酒发酵过程中风味调控提供理论基础。
风味功能微生物菌群的组成和丰度变化,将会造成白酒风味的变化。而白酒发酵过程中的环境因素对微生物群落的组成和演替都有重要影响[7, 26-27]。而本研究中发现还原糖对风味功能微生物菌群的分布具有重要影响,并且还原糖除了与Lactobacillus显著负相关外,与其他风味功能微生物都正相关,且与Komagataella和Pichia显著正相关。还原糖作为微生物代谢的重要碳源,对于微生物的代谢至关重要,后续研究中可以从不同种类的还原糖对风味功能微生物的影响来进一步量化还原糖的驱动作用。
本研究基于高通量测序揭示了青稞酒发酵过程中微生物群落的多样性和动态变化,然后利用关联分析判定了风味功能微生物菌群,并借助蒙特卡洛检验发现了影响风味功能微生物菌群分布的关键理化因子,最后通过模拟组合发酵验证了风味功能微生物的功能,这对于了解青稞酒发酵过程的微生物菌群以及调节青稞酒风味信息具有重要意义。
| [1] |
Giraffa G. Studying the dynamics of microbial populations during food fermentation[J]. FEMS Microbiology Reviews, 2004, 28(2): 251-260. DOI:10.1016/j.femsre.2003.10.005 |
| [2] |
Hugenholtz J. Traditional biotechnology for new foods and beverages[J]. Current Opinion in Biotechnology, 2013, 24(2): 155-159. DOI:10.1016/j.copbio.2013.01.001 |
| [3] |
Zou W, Ye GB, Zhang KZ. Diversity, function, and application of Clostridium in Chinese strong flavor Baijiu ecosystem: a review[J]. Journal of Food Science, 2018, 83(5): 1193-1199. DOI:10.1111/1750-3841.14134 |
| [4] |
Xu YQ, Sun BG, Fan GS, et al. The brewing process and microbial diversity of strong flavour Chinese spirits: a review[J]. Journal of the Institute of Brewing, 2017, 123(1): 5-12. DOI:10.1002/jib.404 |
| [5] |
Hu XL, Du H, Ren C, et al. Illuminating anaerobic microbial community and cooccurrence patterns across a quality gradient in Chinese liquor fermentation pit muds[J]. Applied and Environmental Microbiology, 2016, 82(8): 2506-2515. DOI:10.1128/AEM.03409-15 |
| [6] |
Wang P, Wu Q, Xu Y. Core microbiota in Chinese liquor fermentation and associations with environmental factors[J]. Acta Microbiologica Sinica, 2018, 58(1): 142-153. (in Chinese) 王鹏, 吴群, 徐岩. 中国白酒发酵过程中的核心微生物群及其与环境因子的关系[J]. 微生物学报, 2018, 58(1): 142-153. |
| [7] |
Song ZW, Du H, Zhang Y, et al. Unraveling core functional microbiota in traditional solid-state fermentation by high-throughput amplicons and metatranscriptomics sequencing[J]. Frontiers in Microbiology, 2017, 8: 1294. DOI:10.3389/fmicb.2017.01294 |
| [8] |
Zhao SY, Wang F. Investigation on the formation of the special styles of Huzhu highland barley wine[J]. Liquor-Making Science & Technology, 2011(7): 76-78. (in Chinese) 赵生玉, 王芳. 透视互助青稞酒独特风格的成因[J]. 酿酒科技, 2011(7): 76-78. |
| [9] |
Zhang XM, Li DM, Jin WY, et al. Analysis of microbial diversity of Tibet highland barley wine distiller's yeast based on high-throughput sequencing technology[J]. China Brewing, 2018, 37(9): 28-33. (in Chinese) 张晓蒙, 李德美, 金玮鋆, 等. 西藏青稞酒酿造小曲微生物多样性分析[J]. 中国酿造, 2018, 37(9): 28-33. |
| [10] |
Yuan YZ, Zhang WG, Xu JZ. Investigation of the microbial diversity in Highland barley Qu and optimization of the Koji-making condition with Rhizopus oryzae[J]. Food and Fermentation Industries, 2018, 44(5): 39-45. (in Chinese) 袁亦舟, 张伟国, 徐建中. 青稞酒曲微生物多样性分析及米根霉制曲条件优化[J]. 食品与发酵工业, 2018, 44(5): 39-45. |
| [11] |
Kong Y, Wu Q, Zhang Y, et al. In situ analysis of metabolic characteristics reveals the key yeast in the spontaneous and solid-state fermentation process of Chinese light-style liquor[J]. Applied and Environmental Microbiology, 2014, 80(12): 3667-3676. DOI:10.1128/AEM.04219-13 |
| [12] |
Gao WJ, Fan WL, Xu Y. Characterization of the key odorants in light aroma type Chinese liquor by gas chromatography-olfactometry, quantitative measurements, aroma recombination, and omission studies[J]. Journal of Agricultural and Food Chemistry, 2014, 62(25): 5796-5804. DOI:10.1021/jf501214c |
| [13] |
Li Z, Dong L, Huang Q, et al. Bacterial communities and volatile compounds in Doubanjiang, a Chinese traditional red pepper paste[J]. Journal of Applied Microbiology, 2016, 120(6): 1585-1594. DOI:10.1111/jam.13130 |
| [14] |
Ren GD, Ren WJ, Teng Y, et al. Evident bacterial community changes but only slight degradation when polluted with pyrene in a red soil[J]. Frontiers in Microbiology, 2015, 6: 22. |
| [15] |
Caporaso JG, Kuczynski J, Stombaugh J, et al. QⅡME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [16] |
Wang XS, Du H, Zhang Y, et al. Environmental microbiota drives microbial succession and metabolic profiles during Chinese liquor fermentation[J]. Applied and Environmental Microbiology, 2018, 84(4): e02369-17. |
| [17] |
Gou M, Wang HZ, Yuan HW, et al. Characterization of the microbial community in three types of fermentation starters used for Chinese liquor production[J]. Journal of the Institute of Brewing, 2015, 121(4): 620-627. DOI:10.1002/jib.272 |
| [18] |
Lei ZH. Primary analysis on the fermented microorganism of Fen-flavor liquor by high-throughput sequencing[J]. Food and Fermentation Industries, 2015, 41(9): 164-167. (in Chinese) 雷振河. 采用高通量测序技术分析清香型白酒酿造微生物[J]. 食品与发酵工业, 2015, 41(9): 164-167. |
| [19] |
Suezawa Y, Suzuki M. Bioconversion of ferulic acid to 4-vinylguaiacol and 4-ethylguaiacol and of 4-vinylguaiacol to 4-ethylguaiacol by halotolerant yeasts belonging to the genus Candida[J]. Bioscience, Biotechnology, and Biochemistry, 2007, 71(4): 1058-1062. DOI:10.1271/bbb.60486 |
| [20] |
Mo EK, Sung CK. Production of white pan bread leavened by Pichia anomala SKM-T[J]. Food Science and Biotechnology, 2014, 23(2): 431-437. DOI:10.1007/s10068-014-0059-7 |
| [21] |
Stribny J, Gamero A, Pérez-Torrado R, et al. Saccharomyces kudriavzevii and Saccharomyces uvarum differ from Saccharomyces cerevisiae during the production of aroma-active higher alcohols and acetate esters using their amino acidic precursors[J]. International Journal of Food Microbiology, 2015, 205: 41-46. DOI:10.1016/j.ijfoodmicro.2015.04.003 |
| [22] |
Chen B, Wu Q, Xu Y. Filamentous fungal diversity and community structure associated with the solid state fermentation of Chinese Maotai-flavor liquor[J]. International Journal of Food Microbiology, 2014, 179: 80-84. DOI:10.1016/j.ijfoodmicro.2014.03.011 |
| [23] |
Pervez S, Siddiqui NN, Ansari A, et al. Phenotypic and molecular characterization of Aspergillus species for the production of starch-saccharifying amyloglucosidase[J]. Annals of Microbiology, 2015, 65(4): 2287-2291. DOI:10.1007/s13213-015-1070-9 |
| [24] |
Wang SL, Wu Q, Nie Y, et al. Construction of synthetic microbiota for reproducible flavor metabolism in Chinese light aroma type liquor produced by solid-state fermentation[J]. Applied and Environmental Microbiology, 2019, 85(10): e03090-18. |
| [25] |
Huang ZR, Hong JL, Xu JX, et al. Exploring core functional microbiota responsible for the production of volatile flavour during the traditional brewing of Wuyi Hong Qu glutinous rice wine[J]. Food Microbiology, 2018, 76: 487-496. DOI:10.1016/j.fm.2018.07.014 |
| [26] |
Li P, Lin WF, Liu X, et al. Environmental factors affecting microbiota dynamics during traditional solid-state fermentation of Chinese Daqu starter[J]. Frontiers in Microbiology, 2016, 7: 1237. |
| [27] |
Xiao C, Lu ZM, Zhang XJ, et al. Bio-heat is a key environmental driver shaping the microbial community of medium-temperature Daqu[J]. Applied and Environmental Microbiology, 2017, 83(23): e01550-17. |
 2020, Vol. 17
2020, Vol. 17