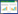扩展功能
文章信息
- 郭燕华, 安邦
- GUO Yan-Hua, AN Bang
- 基于CRISPR-Cas9技术构建橡胶树胶孢炭疽菌的基因敲除系统
- CRISPR-Cas9 ribonucleoprotein-mediated gene knock-out in Colletotrichum gloeosporioides from Hevea brasiliensis
- 微生物学通报, 2020, 47(1): 109-117
- Microbiology China, 2020, 47(1): 109-117
- DOI: 10.13344/j.microbiol.china.190263
-
文章历史
- 收稿日期: 2019-03-31
- 接受日期: 2019-05-30
- 网络首发日期: 2019-07-15
胶孢炭疽菌是引起海南省橡胶树炭疽病害的主要病原菌[1]。橡胶树胶孢炭疽菌能够侵染橡胶树嫩叶及橡胶树的多个组织,引起橡胶树嫩梢回枯及重复落叶,并严重影响胶乳产量。因此,深入研究该病原真菌致病力分子机制,能够为橡胶树炭疽病的防控提供新的思路。在前期工作中,本课题组已经成功建立了橡胶树胶孢炭疽菌的遗传转化及基因敲除系统,该基因敲除系统采用同源重组策略,能够对基因组中的单一基因进行基因敲除[2]。但是在病原真菌中,与致病力相关的基因经常呈现出多拷贝的现象,如效应蛋白编码基因等[3],因此难以采用常规的同源重组策略对这些基因进行敲除。此外,利用同源重组策略对目标基因进行定点突变、编辑等工作时,常常需要进行多次的基因扩增及载体构建工作,耗时较长。
近年来,CRISPR-Cas9基因组编辑技术的快速发展,极大地促进了遗传学的研究。与传统的基因敲除、敲入技术相比,CRISPR/Cas技术可以精确地在基因组上对靶标序列进行插入、敲除及定点突变等操作。CRISPR/Cas系统是存在于细菌和古细菌中的一种获得性免疫机制[4-5],其中属于II型的CRISPR-Cas9系统组分较为简单,应用也最为广泛[6-9]。经过研究人员的研究及改造,CRISPR/Cas系统已经成为一个有效的基因组编辑系统。CRISPR-Cas9系统的基本结构包括tracrRNA (trans-activating-crRNA)序列区、cas (CRISPR associated system)基因序列区、crRNA (CRISPR RNA)序列区[10]。其中crRNA和tracrRNA可以形成嵌合RNA分子,被称为SgRNA;该SgRNA能够进一步与Cas9蛋白形成核糖核酸-蛋白复合体(ribonucleoprotein, RNP),然后与tracrRNA序列互补的DNA双链结合,并对DNA双链进行切割,产生双链断裂(DNA double-strand breaks,DSBs);之后借助活细胞内的DNA修复机制对受损的DNA进行修复。DNA的修复过程主要以两种方式进行:非同源末端连接(non-homologous DNA end joining, NHEJ)及同源重组修复(homology directed repair, HDR)。NHEJ是指不依赖于DNA同源片段,直接将DNA断裂端连接在一起的修复过程;该修复过程保真度较低,在DNA连接的过程中易于发生碱基的随机插入或丢失,造成移码突变,从而实现基因失活或目的基因的突变[11]。HDR是指在同源片段存在的情况下,通过同源重组方式,同源片段替换受损的基因组区段,从而完成DNA的修复;该修复过程具有较高的保真度[12-13]。在CRISPR-Cas9系统的应用中,可以人为地将tracrRNA-crRNA设计为具有特定序列的sgRNA,引导Cas9与靶标DNA结合并进一步实现基因编辑。
本研究通过体外合成含有细胞核定位信号的Cas9蛋白、SgRNA,以及体外构建Cas9-SgRNA复合体,并将该复合体转入橡胶树胶孢炭疽菌原生质体等操作,完成对基因组目标基因的敲除及定点突变,建立适用于橡胶树胶孢炭疽菌的基因敲除系统,该系统可为进一步研究胶孢炭疽菌致病力分子机制打下了良好的基础。
1 材料与方法 1.1 菌株、质粒及引物橡胶树胶孢炭疽菌(Colletotrichum gloeosporioides)野生型菌株由本实验室分离保存,并在前期工作中完成该菌株的基因组测序工作。采用pQE-80L质粒作为原核表达载体。载体构建过程所使用的大肠杆菌感受态细胞Trans1-T1及原核表达所使用大肠杆菌Transetta(DE3)均购自北京全式金公司。引物合成及DNA测序由华大基因公司完成。本研究所使用的引物如表 1所示。
| Primers name | Primers sequence (5′→3′) |
| NLSCg-F | GGATCCATGCCCCCCAAGGCCGCC |
| NLSCg-R | GTCGACCTTGTAGATGTAAGAAGA |
| Cas9-F | GTCGACATGGCCCCAAAGAAGAA |
| Cas9-R | AAGCTTTTACTTTTTCTTTTTTG |
| CgU5-TF | CGATGTCCAGGAACTCTTGCG |
| CgU5-TR | TTGGCACCCTCAATGATGTCG |
| CgU5-SR | TTGGCACCCTCAATGATGTCG |
| SgCgU5-F | GAAATTAATACGACTCACTATAGGCG AGGGCGGCAACATTGTGTTTTAGAG CTAGAAATAGCAAG |
| SgRNA-UR | AAAAGCACCGACTCGGTGCCACTTTT TCAAGTTGATAACGGACTAGCCTTATT TTAACTTGCTATTTCTAGCTCTAAAAC |
| doCgU5-F | GCCAAGGACCACGGCGAGGGCGGCA ACATTTGATAACGCGCCCCTCAAGGG CAAGAAGGTCCTGAT |
| doCgU5-R | ATCAGGACCTTCTTGCCCTTGAGGGG CGCGTTATCAAATGTTGCCGCCCTCGC CGTGGTCCTTGGC |
DNA聚合酶TransStart FastPfu、PCR Mix、DNA Marker,北京全式金生物技术有限公司;限制性内切酶、T4 DNA连接酶,Fermentas公司;DNA回收试剂盒、植物基因组DNA提取试剂盒,天根生化科技有限公司;羧苄青霉素、卡那霉素、异丙基硫代半乳糖苷(IPTG)、裂解酶(lysing enzyme)、聚乙二醇(PEG-3350),Sigma-Aldrich公司;5-氟乳清酸(5-fluoroorotic acid,5-FOA)、尿嘧啶、透析袋,索莱宝公司;RNA体外转录试剂盒,New England Biolabs公司;Ni-NTA琼脂糖珠,Qiagen公司;超滤离心管,Millipore公司;不含尿嘧啶的氨基酸添加物(−URA,DO Supplement),Clontech公司;常规化学试剂均为国产分析纯,国药集团化学试剂有限公司。引物合成及DNA测序由华大基因公司完成。
PCR仪,Bio-Rad公司;生化培养箱,上海博迅实业有限公司。
土豆浸膏培养基购自北京双旋微生物培养基制品厂,称取粉状试剂62.5 g,加蒸馏水至1 L,pH自然。
改良M9基础培养基(g/L):磷酸氢二钠6.0,磷酸二氢钾3.0,不含尿嘧啶的氨基酸添加物0.77,蔗糖2.0,琼脂粉20.0,加蒸馏水至1 L,pH自然。
原生质体再生培养基(g/L):酵母浸粉2.0,酸水解酪蛋白2.0,蔗糖200.0,加蒸馏水至1 L,pH自然。
1.3 原核表达载体的构建以橡胶树胶孢炭疽菌的基因组为模板,利用引物NLSCg-F/NLSCg-R扩增组蛋白H2B的细胞核定位信号肽(nuclear localization sequence,NLS)的编码序列NLSH2B;以35S-CAS9-NOS-SK载体为模板,利用引物Cas9-F/Cas9-R扩增Cas9蛋白的编码序列。PCR反应体系参照TransStart FastPfu说明书配制:模板500 ng,缓冲液1×,正、反向引物各0.2 μmol/L,dNTPs 0.2 mmol/L,FastPfu 2.5 U,加ddH2O至50 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 30 s,52 ℃ 30 s,72 ℃ 30 s (或90 s),35个循环;72 ℃ 5 min。之后将两个PCR扩增片段分别连入测序载体并进行测序分析。待序列检测无误后,分别利用BamH I/Sal I切割NLSH2B序列、利用Sal I/Hind III切割Cas9序列,并将酶切后的DNA序列连入pQE-80L质粒的相应位点,以构建NLSH2BCas9融合蛋白的原核表达载体。
1.4 融合蛋白的原核表达及纯化将构建完成的原核表达载体转入大肠杆菌Transetta(DE3)菌株,利用引物NLSCg-F/Cas9-R对转化后的阳性菌落进行PCR鉴定。挑取生长速率较快的阳性单克隆菌落,接入含有100 μg/mL羧苄青霉素的LB培养基中,37 ℃、180 r/min振荡培养14 h。然后将菌液按照稀释100倍的比例转接入500 mL新鲜的、含有100 μg/mL羧苄青霉素的LB培养基中,继续振荡培养约4−6 h,期间测定菌液浓度。待菌液的OD600达到0.6后,加入IPTG并使其终浓度达到0.3 mmol/L,再将菌液转入室温(约26 ℃)环境中,160 r/min诱导培养5 h。诱导培养结束后,6 000×g离心10 min收集大肠杆菌菌体,并将菌体转入研钵,加液氮研磨破碎细胞,再用15 mL冰冷的裂解缓冲液(20 mmol/L NaH2PO4,300 mmol/L NaCl,1 mmol/L imidazole,1% Triton X-100,1 mmol/L PMSF,pH 7.5)重悬菌体,涡旋振荡30 s,置于冰上5 min后于4 ℃、12 000×g离心30 min,收集上清溶液,并用0.22 μm的滤器过滤除去残余细胞杂质。将诱导前的大肠杆菌菌体、诱导并破碎后的细胞菌体沉淀组分以及诱导并破碎后细胞菌体可溶组分取10 μL样品,并进行SDS-PAGE检测,分析融合蛋白的表达情况。
参照Qiagen公司提供的说明书,在上述上清液中加入300 μL平衡后的Ni-NTA琼脂糖珠,并将混合物在4 ℃下颠倒孵育3 h;然后在4 ℃、700×g离心5 min,小心吸除上清液,并加入8 mL漂洗缓冲液,4 ℃下轻柔颠倒洗涤5 min,同上离心后重复漂洗一次;最后在Ni-NTA琼脂糖珠中加入2 mL洗脱缓冲液,4 ℃下轻柔颠倒洗涤5 min,同上离心后再重复洗脱一次,并将2次的洗脱溶液合并后转入透析袋中,置于1 L冰冷的透析液(20 mmol/L NaH2PO4,300 mmol/L NaCl,20% glycerol,pH 7.5)中,在冰浴条件下透析6 h。待透析结束后,小心收集透析袋中的溶液,并将溶液转入4 mL 50 kD规格的超滤离心管中,4 ℃、2 000×g离心10−15 min,待超滤离心管上层溶液体积达到0.5 mL后,将上层溶液转入1.5 mL离心管中,−20 ℃保存备用。将诱导并破碎后细胞菌体可溶组分,以及透析后的蛋白溶液取10 μL样品进行SDS-PAGE检测,分析融合蛋白的纯化情况。
1.5 SgRNA的设计及体外转录使用SgRNA在线预测软件ChopChop[14]预测目标基因可用的SgRNA序列,在预测结果中选择切割效率较高且在目标基因序列中位置靠前的SgRNA;之后将预测到的SgRNA序列在胶孢炭疽菌基因组中进行BLAST分析,排除多位点结合的SgRNA以降低脱靶率。选择胶孢炭疽菌尿嘧啶合成途径中的乳清酸磷酸核糖转移酶的编码基因(URA5)作为验证本系统的靶标基因;根据预测所得的SgRNA序列以及Cas9所必需的SgRNA骨架序列,设计SgRNA转录所需的模板DNA的扩增引物;根据胶孢炭疽菌URA5序列及预测所得的SgRNA序列,设计引物对SgCgU5-F/SgRNA-UR通过PCR扩增该序列。PCR反应体系参照TransStart FastPfu说明书配制:缓冲液1×,正、反向引物各0.2 μmol/L,dNTPs 0.2 mmol/L,FastPfu 2.5 U,加ddH2O至50 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 30 s,50 ℃ 30 s,72 ℃ 15 s,35个循环;72 ℃ 5 min。测序分析正确后,以该DNA序列为模板,使用RNA体外转录试剂盒,参照说明书进行SgRNA的体外转录。待转录结束后,使用1%的琼脂糖凝胶电泳检测SgRNA的合成情况。
1.6 SgRNA与NLSH2BCas9复合体的切割活性检测为了验证预测所得SgRNA是否能够指导NLSH2BCas9蛋白进行目标DNA的切割,首先要进行体外切割活性检测。以橡胶树胶孢炭疽菌的基因组为模板,利用引物CgU5-TF/CgU5-TR扩增出一段1.8 kb、包含有URA5及其上下游序列的DNA片段,将该片段作为切割活性检测的切割底物DNA (DNA PCR fragment)。PCR反应体系参照TransStart FastPfu说明书配制:模板200 ng,缓冲液1×,正、反向引物各0.2 μmol/L,dNTPs 0.2 mmol/L,FastPfu 2.5 U,加ddH2O至50 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 30 s,52 ℃ 30 s,72 ℃ 60 s,35个循环;72 ℃ 5 min。然后将SgRNA、NLSH2BCas9以及10×的反应缓冲液(200 mmol/L HEPES,1 mol/L NaCl,50 mmol/L MgCl2,1 mmol/L EDTA,pH 6.5)按照表 2所示配制酶切体系并进行酶切反应。酶切检测分两步进行,首先在26 ℃条件下将SgRNA与NLSH2BCas9蛋白共同孵育10 min,使其形成复合体;然后在反应体系中加入5 μL切割底物DNA,并于37 ℃条件下酶切1 h。酶切结束后,在酶切体系中加入1 μL蛋白酶K,37 ℃继续孵育15 min后用1%的琼脂糖凝胶电泳检测酶切结果。
| Reagents | Volume |
| 10×Reaction buffer | 3 μL |
| NLSH2BCas9 | 200 ng |
| SgRNA | 40 ng |
| DEPC H2O | Up to 25 uL |
| 26 ℃, incubation for 10 min | |
| Add DNA PCR fragment | 5 μL |
| 37 ℃, incubation for 1 h | |
橡胶树胶孢炭疽菌原生质体的制备及转化参照文献[2]所述方法进行。将野生型菌株接种到PDA培养基上,28 ℃培养7 d。然后使用无菌水收集分生孢子,使用无菌滤膜除去菌丝体。再将孢悬液接种于200 mL液体培养基中,使得孢子初始浓度为105个/mL,将孢子在28 ℃、160 r/min振荡培养14−16 h。采用无菌滤膜收集菌丝体,并将菌丝体转移到含10 mg/mL裂解酶的1 mol/L山梨醇缓冲液中,28 ℃、100 r/min培养3 h以降解菌丝体细胞壁。然后在冰浴条件下,使用无菌滤膜过滤收集原生质体,在4 ℃、2 000×g条件下离心10 min收集原生质体,并用冰冷的1 mol/L山梨醇缓冲液冲洗2次除去残余酶液。最后用山梨醇缓冲液将原生质体浓度调整至108个/mL。此外,在原生质体制备结束前15 min,开始制备SgRNA与NLSH2BCas9的复合体。
采用两种DNA切割及修复方式构建目标基因的敲除突变体,原理如图 1所示。(1)非同源末端连接(NHEJ):将10倍反应缓冲液、2 μg的NLSH2BCas9与1 μg的SgRNA按比例混合,并在室温下孵育15 min,用于后续原生质体转化。(2)同源重组修复(HDR):准备用于切口修复的双链供体DNA,该DNA链序列中含有切割位点处上下游各30 bp的同源序列以及两个终止密码子。将2 μg的供体DNA、10倍反应缓冲液、2 μg的NLSH2BCas9、1 μg的SgRNA按比例混合,并在室温下孵育15 min,用于后续原生质体转化。
|
| 图 1 URA5的SgRNA结合位点及其供体DNA重组位点示意图 Figure 1 Schematic illustration of the SgRNA binding site and donor DNA for URA5 mutagenesis 注:NHEJ:非同源末端连接;HDR:同源重组修复. Note: NHEJ: Non-homologous end joining; HDR: Homology directed repair. |
|
|
待原生质体制备结束后,将上述SgRNA、NLSH2BCas9的复合体或SgRNA、NLSH2BCas9、供体DNA的复合体溶液分别加入原生质体中,轻柔混匀,并于冰上放置20 min。然后在混合物中缓慢加入1 mL含40% PEG的山梨醇缓冲液,颠倒混匀,于28 ℃放置20 min。待转化完成后,在转化混合物中加入5 mL液体再生培养基,28 ℃、100 r/min再生培养5 h。之后在培养物中加入处于熔化状态、温度在50 ℃左右、含1%琼脂的固体再生培养基,混匀后平铺于培养皿中。待培养基凝固后,再在其上平铺含2 g/L的5-FOA、20 μg/L尿嘧啶及20 μg/L尿苷的M9基础培养基。将培养皿置于28 ℃培养4−5 d,期间观察转化子的生长情况。
1.8 突变株的筛选及检测待再生培养基上长出转化子后,将每个转化子同时转接到含有5-FOA的M9基础培养基以及含有5-FOA、20 μg/L尿嘧啶、20 μg/L尿苷的M9基础培养基中进行二次筛选。能够在添加尿嘧啶和尿苷的培养基中生长而不能在不含尿嘧啶和尿苷的培养基中生长的菌株为阳性转化子。之后挑取阳性转化子的菌丝体,采用植物基因组提取试剂盒提取其基因组DNA并进行PCR扩增及测序鉴定。
利用引物对CgU5-TF/CgU5-TR扩增待测片段,再利用测序引物CgU5-SR检测片段中碱基缺失及插入情况,并依据测序结果分析突变体基因缺失情况。PCR反应体系参照PCR Mix说明书配制:模板200 ng,正、反向引物各0.2 μmol/L,PCR Mix 10 μL,加ddH2O至20 μL。PCR反应条件:94 ℃ 5 min;94 ℃ 30 s,54 ℃ 30 s,72 ℃ 90 s,35个循环;72 ℃ 5 min。
1.9 突变株的表型分析将野生型菌株及URA5的敲除突变株分别接种到M9基础培养基,含2 g/L 5-FOA的M9基础培养基,以及含2 g/L 5-FOA、20 μg/L尿嘧啶、20 μg/L尿苷的M9基础培养基中,培养4 d并观测菌落的生长情况。
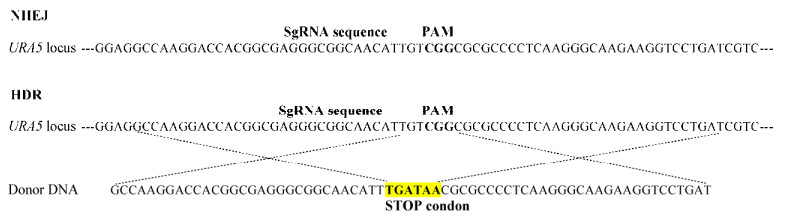
2 结果与分析 2.1 NLSH2BCas9蛋白的原核表达及纯化为了使Cas9蛋白能够顺利跨膜并进入胶孢炭疽菌的细胞核中,将组蛋白H2B的一段细胞核定位信号肽(NLSH2B)与Cas9蛋白进行融合表达。NLSH2B信号肽的DNA编码序列为162 bp,编码一段5.7 kD的肽段。以含有酿脓链球菌(Streptococcus pyogenes) Cas9基因的35S-CAS9-NOS-SK质粒为模板,扩增4 203 bp Cas9编码基因,编码一段161.6 kD的肽段。在将含有融合表达载体的大肠杆菌菌株诱导表达5 h后,NLSH2BCas9融合蛋白的表达量显著提高;此外,NLSH2BCas9融合蛋白主要分布于细胞裂解后的上清液中(图 2),说明该蛋白为可溶蛋白。在使用Ni-NTA琼脂糖纯化、透析、超滤离心管浓缩后,获得了纯度较高的NLSH2BCas9融合蛋白,可以用于下一步实验。
|
| 图 2 NLSH2BCas9融合蛋白的表达纯化 Figure 2 Protein expression and purification assay 注:A:NLSH2BCas9融合蛋白的结构图. B:融合蛋白的诱导表达情况检测:1:诱导前的细菌菌体总蛋白;2:诱导后的细菌菌体总蛋白. C:Ni-NTA纯化情况检测:1:诱导并破碎后的菌体可溶蛋白组分;2:纯化并透析后的蛋白溶液. M:蛋白Marker. Note: A: Structure chart of His-NLSH2BCas9. B: Protein induction assay: 1: Total cell protein before induction; 2: Total cell protein after induction. C: Protein expression and purification assay: 1: Soluble protein before purification; 2: Soluble protein after purification. M: Protein marker. |
|
|
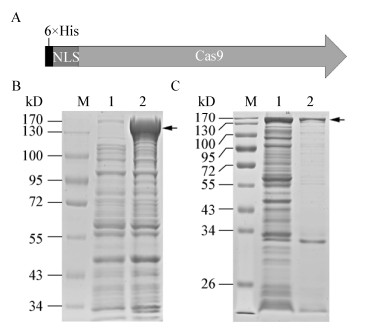
为了能够获得具有较高切割效率的SgRNA,首先采用ChopChop软件及BLAST分析对胶孢炭疽菌URA5基因序列中可能存在的SgRNA切割位点进行了预测,结果预测到了一条具有较高切割活性的SgRNA,其序列为GGCGAGGGCGGCAACATTGT;该SgRNA位于URA5编码序列的349−369 bp处,能够与URA5的反义链结合。依据Cas9蛋白所需要的骨架RNA序列,设计了该SgRNA转录模板SgDNA的引物对SgCgU5-F/SgRNA-UR,并通过PCR扩增及纯化,获得了相应的123 bp DNA片段。之后以该SgDNA为模板,采用RNA体外转录试剂盒进行SgRNA的合成。结果表明,以该DNA为模板,成功合成出一条目标大小的SgRNA链(图 3),可以用于下一步实验。
|
| 图 3 URA5的SgRNA体外转录 Figure 3 In vitro transcription of SgRNA of URA5 注:1:转录体系中不加入T7 RNA聚合酶的阴性对照;2:加入T7 RNA聚合酶后转录形成的SgRNA. Note: 1: Negative control without adding T7 RNA polymerase; 2: Transcript of SgRNA of URA5. |
|
|
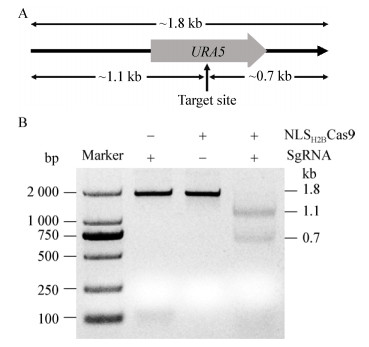
为了验证体外转录获得的SgRNA是否能与NLSH2BCas9形成复合体,并进一步完成对靶标DNA双链的切割,首先进行了体外切割活性检测分析。实验中选择了包含有URA5基因的一段1.8 kb DNA片段作为切割底物,预测的SgRNA切割位点位于该DNA的1.1 kb处,因此切割后的产物应该为两条DNA条带,分别为1.1 kb和0.7 kb。切割结果如图 4所示,在反应体系中同时加入SgRNA和NLSH2BCas9之后,能够成功切割底物DNA,通过分析条带灰度值,计算得出切割效率大于97%,而当在反应体系中只加入SgRNA或NLSH2BCas9时,则不能对底物DNA进行切割。因此,本研究中所使用的SgRNA及NLSH2BCas9具有较高的切割活性,可以用于下一步实验。
|
| 图 4 体外切割活性检测 Figure 4 In vitro cleavage activity assay 注:A:底物DNA及其切割位点示意图:Target site为理论切割位点,箭头所示为预测底物DNA切割后条带的大小;B:体外切割1 h后琼脂糖凝胶电泳检测结果:以DNA marker右边算起,泳道1为只加SgRNA而不加NLSH2BCas9蛋白的阴性对照1;泳道2为只加NLSH2BCas9蛋白而不加SgRNA的阴性对照2;泳道3为同时加入SgRNA和NLSH2BCas9蛋白的样品. Note: A: Diagram represents the template length, the sgRNA cleavage site, and the lengths of expected fragments after cleavage; B: Assessment of the cleavage efficiency by gel electrophoresis after incubation for 1 hour: Lane 1 represents negative control with SgRNA but without NLSH2BCas9; Lane 2 represents negative control with NLSH2BCas9 but without SgRNA; Lane 3 represents the assay with both NLSH2BCas9 and SgRNA. |
|
|
通过SgRNA-Cas9复合体的体外合成、原生质体转化及筛选,成功获得了一批转化子。其中通过NHEJ方式获得阳性转化子13株,通过HDR方式获得阳性转化子9株。之后在两类转化子中分别挑取3株菌落,提取其基因组,PCR扩增其切割位点附近的DNA序列并进行测序鉴定,结果证明成功获得一系列URA5的不同位点缺失突变株,并将这些突变株命名为ΔURA5。其中通过NHEJ方式获得的突变株ΔURA5-2和ΔURA5-3,其URA5的编码序列分别缺失了1个和12个碱基,而ΔURA5-1的编码序列则增加了1个碱基。通过HDR方式获得的突变株ΔURA5-14和ΔURA5-15,在URA5编码序列的目标靶标位点插入了2个终止密码子,与预期结果一致;而ΔURA5-16突变体除了在目标靶点位置插入了2个终止密码子外,还额外整合了一段同源序列(图 5)。

|
| 图 5 测序鉴定不同缺失突变体的突变位点 Figure 5 Characterization of individual mutants using PCR and sequencing analysis |
|
|
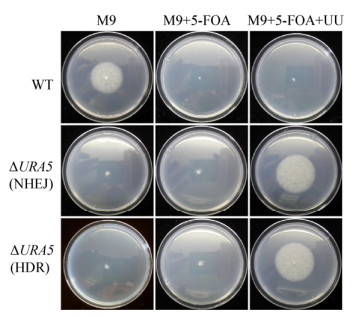
为了验证URA5的缺失是否会引起突变株生理表型的变化,分别将上述获得的两种类型的突变株接种到不同成分的培养基中,观测其生长表型。结果表明两类URA5敲除突变株均能在添加5-FOA、尿嘧啶及尿苷的培养基上生长,且突变株表现出尿嘧啶营养缺陷型(图 6),证明URA5基因被成功敲除。
|
| 图 6 突变体的生长表型分析 Figure 6 The phenotypes of mutants 注:M9表示M9基本培养基;M9+5-FOA表示添加了5-氟乳清酸的M9基本培养基;M9+5-FOA+UU表示添加了5-氟乳清酸、尿嘧啶及尿苷的M9基本培养基. Note: M9 represents M9 medium; M9+5-FOA represents M9 medium supplemented with 5-FOA; M9+5-FOA+UU represents M9 medium supplemented with 5-FOA, uracil and uridine. |
|
|
CRISPR-Cas9系统在多种动植物以及真菌中得到了广泛应用。在植物病原真菌中,研究者也对CRISPR-Cas9系统的应用进行了研究,如构巢曲霉[15]、粗糙脉孢菌[16]、稻瘟菌[17]、尖孢镰刀菌[18]、产黄青霉[19]等。目前关于CRISPR-Cas9系统的应用主要有两种策略:第一种策略是将Cas9和SgRNA序列整合到目标生物的基因组上,利用生物本身的启动子在其细胞内表达Cas9和SgRNA,并进一步完成对靶标基因的编辑[20]。然而这种策略具有一定的局限性,如启动子的强弱、Cas9密码子的选择、在细胞内的表达等,都会对基因编辑的效率及准确性产生影响。此外,细胞内过量表达Cas9会易于产生脱靶效应,产生非特异性的基因编辑。第二种策略是在体外表达Cas9和SgRNA,然后以核糖体核蛋白复合体形式导入受体细胞进行基因组编辑[21],这种策略具有特异性较高、对细胞毒性较低等优点。但是该策略的应用也具有一定的限制性:首先Cas9蛋白分子量较大,跨膜进入受体细胞的效率较低;其次Cas9需要与细胞核定位序列进行融合表达,才能准确进入细胞核中。在本研究中,选择第二种策略建立橡胶树胶孢炭疽菌基因敲除系统。为了使Cas9蛋白准确进入橡胶树胶孢炭疽菌细胞核中,本研究选择了细胞核组蛋白H2B的细胞核核定位序列用于构建融合表达蛋白。生物信息学分析表明胶孢炭疽菌组蛋白H2B的蛋白序列的N端第1–45位氨基酸为细胞核定位序列NLSH2B。因此本研究中选择扩增了H2B的DNA序列5ʹ端162 bp序列,该段序列编码一段5.7 kD的肽段。通过将该段序列与Cas9序列串联连入原核表达载体,构建相应的融合表达蛋白。为了提高原核表达的效率,本研究选择了经过密码子优化的原核表达菌株大肠杆菌Transetta(DE3)菌株,经过诱导表达,获得了较高表达量的可溶性融合蛋白NLSH2BCas9。之后再通过本课题组前期建立的原生质体转化系统,将NLSH2BCas9与SgRNA的复合体转入胶孢炭疽菌中。
为了检测所建立的系统是否能够有效地进行橡胶树胶孢炭疽菌基因的敲除,本研究中选择了URA5作为靶标基因进行基因敲除。URA5编码真菌尿嘧啶合成途径的关键酶——乳清酸磷酸核糖转移酶,该基因的缺失会导致真菌由野生型变为尿嘧啶依赖型。5-氟乳清酸(5-FOA)是尿嘧啶合成的前体乳清酸的类似物,它能够被尿嘧啶合成途径利用,生成对真菌细胞有毒的5-氟脲嘧啶,因此5-FOA能够用来筛选URA5的缺失突变型[22]。通过筛选及测序鉴定,成功获得了URA5的敲除突变株ΔURA5。通过在含有不同组分培养基中的生长表型检测,证明URA5被成功敲除,且NHEJ和HDR两种方式都能够成功获得相应的突变体。其中通过HDR方式构建的突变体中,ΔURA5-14和ΔURA5-15菌株均在目标位点插入了2个终止密码子,而ΔURA5-16突变体除了在目标靶点位置插入了2个终止密码子外,还额外整合了一段序列。经过比对发现,额外整合的序列为原生质体转化时使用的同源序列中右侧30 bp的同源序列,说明在ΔURA5-16突变体中,同源序列与基因组序列重组的过程中,只有左侧的序列发生了同源重组,从而导致右侧序列整体插入基因组中。此外,与NHEJ方式相比,HDR方式能够在基因组序列中引入特定序列,因此具有更广阔的应用场景,如基因的点突变、标签基因的插入等[23]。
本研究通过体外表达Cas9蛋白、体外转录SgRNA以及Cas9-SgRNA复合体转化真菌原生质体,成功建立了橡胶树胶孢炭疽菌的基因敲除系统,为该病原真菌致病力的分子机制研究提供了优良的工具。
| [1] |
Cai ZY, Huang GX. Research advances in anthracnose of Hevea brasiliensis[J]. Journal of Southwest Forestry College, 2011, 31(1): 89-93. (in Chinese) 蔡志英, 黄贵修. 巴西橡胶树炭疽病研究进展[J]. 西南林业大学学报, 2011, 31(1): 89-93. DOI:10.3969/j.issn.1003-7179.2011.01.020 |
| [2] |
Wang QN, An B, Hou XR, et al. Dicer-like proteins regulate the growth, conidiation, and pathogenicity of Colletotrichum gloeosporioides from Hevea brasiliensis[J]. Frontiers in Microbiology, 2018, 8: 2621. DOI:10.3389/fmicb.2017.02621 |
| [3] |
Rafiqi M, Ellis JG, Ludowici VA, et al. Challenges and progress towards understanding the role of effectors in plant-fungal interactions[J]. Current Opinion in Plant Biology, 2012, 15(4): 477-482. DOI:10.1016/j.pbi.2012.05.003 |
| [4] |
Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140 |
| [5] |
Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea[J]. Science, 2010, 327(5962): 167-170. DOI:10.1126/science.1179555 |
| [6] |
Hwang WY, Fu YF, Reyon D, et al. Efficient genome editing in zebrafish using a CRISPR-Cas system[J]. Nature Biotechnology, 2013, 31(3): 227-229. |
| [7] |
Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [8] |
Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [9] |
Cho SW, Kim S, Kim JM, et al. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease[J]. Nature Biotechnology, 2013, 31(3): 230-232. |
| [10] |
Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell, 2014, 157(6): 1262-1278. |
| [11] |
Bennardo N, Cheng A, Huang N, et al. Alternative-NHEJ is a mechanistically distinct pathway of mammalian chromosome break repair[J]. PLoS Genetics, 2008, 4(6): e1000110. DOI:10.1371/journal.pgen.1000110 |
| [12] |
Moynahan ME, Chiu JW, Koller BH, et al. Brca1 controls homology-directed DNA repair[J]. Molecular Cell, 1999, 4(4): 511-518. |
| [13] |
Ran FA, Hsu PD, Lin CY, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J]. Cell, 2013, 154(6): 1380-1389. |
| [14] |
Montague TG, Cruz JM, Gagnon JA, et al. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing[J]. Nucleic Acids Research, 2014, 42(W1): W401-W407. DOI:10.1093/nar/gku410 |
| [15] |
N dvig CS, Nielsen JB, Kogle ME, et al. A CRISPR-Cas9 system for genetic engineering of filamentous fungi[J]. PLoS One, 2015, 10(7): e0133085. DOI:10.1371/journal.pone.0133085 |
| [16] |
Matsu-Ura T, Baek M, Kwon J, et al. Efficient gene editing in Neurospora crassa with CRISPR technology[J]. Fungal Biology and Biotechnology, 2015, 2(1): 4. DOI:10.1186/s40694-015-0015-1 |
| [17] |
Foster AJ, Martin-Urdiroz M, Yan X, et al. CRISPR-Cas9 ribonucleoprotein-mediated co-editing and counterselection in the rice blast fungus[J]. Scientific Reports, 2018, 8(1): 14355. DOI:10.1038/s41598-018-32702-w |
| [18] |
Wang Q, Cobine PA, Coleman JJ. Efficient genome editing in Fusarium oxysporum based on CRISPR/Cas9 ribonucleoprotein complexes[J]. Fungal Genetics and Biology, 2018, 117: 21-29. DOI:10.1016/j.fgb.2018.05.003 |
| [19] |
Pohl C, Kiel JAKW, Driessen AJM, et al. CRISPR/Cas9 based genome editing of Penicillium chrysogenum[J]. ACS Synthetic Biology, 2016, 5(7): 754-764. DOI:10.1021/acssynbio.6b00082 |
| [20] |
Yan LH, Wei SW, Wu YR, et al. High-efficiency genome editing in Arabidopsis using YAO promoter-driven CRISPR/Cas9 system[J]. Molecular Plant, 2015, 8(12): 1820-1823. DOI:10.1016/j.molp.2015.10.004 |
| [21] |
Woo JW, Kim J, Kwon SI, et al. DNA-free genome editing in plants with preassembled CRISPR-Cas9 ribonucleoproteins[J]. Nature Biotechnology, 2015, 33(11): 1162-1164. DOI:10.1038/nbt.3389 |
| [22] |
de Montigny J, Belarbi A, Hubert JC, et al. Structure and expression of the URA5 gene of Saccharomyces cerevisiae[J]. Molecular and General Genetics, 1989, 215(3): 455-462. DOI:10.1007/BF00427043 |
| [23] |
Zhang C, Meng XH, Wei XL, et al. Highly efficient CRISPR mutagenesis by microhomology-mediated end joining in Aspergillus fumigatus[J]. Fungal Genetics and Biology, 2016, 86: 47-57. DOI:10.1016/j.fgb.2015.12.007 |
 2020, Vol. 47
2020, Vol. 47