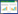扩展功能
文章信息
- 张珂珂, 周娇娇, 佘炜怡, 高云雨, 邓嘉雯, 谢宁, 田生礼
- ZHANG Ke-Ke, ZHOU Jiao-Jiao, SHE Wei-Yi, GAO Yun-Yu, DENG Jia-Wen, XIE Ning, TIAN Sheng-Li
- 敲除组蛋白去乙酰化酶基因hdac对里氏木霉纤维素酶表达的调控作用
- Effects of knocking-out histone deacetylase gene hdac on cellulase expression in Trichoderma reesei
- 微生物学通报, 2020, 47(1): 88-96
- Microbiology China, 2020, 47(1): 88-96
- DOI: 10.13344/j.microbiol.china.190260
-
文章历史
- 收稿日期: 2019-03-29
- 接受日期: 2019-05-27
- 网络首发日期: 2019-06-19
里氏木霉(Trichoderma reesei)是木霉属中产纤维素酶最具代表性的真菌之一,其基因组相较于其他丝状真菌小得多[1],该菌安全无害且繁殖速度快,具有强大的合成和分泌蛋白的能力,主要生产的酶类有纤维素酶和纤维二糖水解酶I (CBH1),纤维素酶总量每升最高可达克级以上[2],因此,里氏木霉是重组蛋白表达和生产的优良宿主。表观遗传调控是不涉及DNA序列变化的可遗传变化,具体的表现形式有DNA甲基化、组蛋白修饰(甲基化、乙酰化、去乙酰化等)、miRNA调控等。在乙酰化/脱乙酰化过程中,最为突出的是N-末端残基修饰H2A、H2B、H3和H4的高度保守赖氨酸[3-4],所以乙酰化在转录调控中可能发挥重要作用。负责脱乙酰化的是组蛋白去乙酰化酶(HDAC),研究发现人HDAC1与转录调节因子RPD3的基因序列具有较高的一致性[5]。HDAC被分为三类,第一类和第二类分别是和酵母蛋白RPD3、HDA1同源的酶类,第三类是和玉米HD2相关的蛋白,此类被证实还与小鼠体内具有ADP核糖基转移酶活性的SIR2属于同源物[6]。研究发现,C. carbonum菌株经基因工程敲除hdc1基因会导致分生孢子突变,在hdc1突变株粗提物中组蛋白去乙酰化酶活性下降约50%,与野生型菌株相比蔗糖产量稍微下降,阿拉伯糖、木糖等其他碳水化合物下降了30%−73%[7]。在C. carbonum hdac基因敲除菌株中,成熟孢子增殖被阻断[8]。蓝华辉等[9]构建了组蛋白去乙酰化酶基因ΔhosA突变体,其黄曲霉孢子的形成减少,黄曲霉的侵染能力降低。李依民等[10]获得HDACs的敲除突变体Δhda1、Δhda2、Δhda3后研究了禾谷镰刀菌突变菌株生长与致病的分子生物学机制,结果发现hda1影响糖代谢,hda2与分生孢子产孢及病原菌扩散有关。
Split-Maker技术是一种运用于丝状真菌中基因敲除的高效方法,经两轮PCR可直接将选择性抗性基因与目的基因上下游融合,利用序列同源重组从而置换目的基因。基于该技术,Fairhead等[11]研发了一种使染色体缺失和直接基因克隆的新型转化策略,并应用于XI染色体上几个开放阅读框的分析。Trushina等[12]敲除了玉米叶枯病菌和小麦赤霉病菌的致病基因。Fu等[13]与Kim等[14]改良了该技术,使得基因异位整合减少,更易于目的基因的靶向敲除。本研究利用Split-Maker技术敲除了hdac基因,筛选得到了里氏木霉敲除组蛋白去乙酰化酶的突变体T. reesei Δhdac,并探索了对纤维素酶酶活及其相关基因转录水平的影响,为里氏木霉表观遗传调控和影响纤维素酶基因表达提供了新的证据。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒里氏木霉QM9414购自美国模式菌种收藏中心(ATCC),含有遗传霉素抗性基因的表达载体pBC-GeneticinR由法国巴黎第七大学Philippe Silar惠赠。其质粒图见图 1。
|
| 图 1 质粒pBC-GeneticinR图谱 Figure 1 pBC-GeneticinR plasmid map |
|
|
土豆PDA培养基(g/L):马铃薯200.0,葡萄糖20.0,琼脂20.0。
里氏木霉液体基本培养基(g/L):参考高云雨等[15]的培养基配方。
里氏木霉产酶培养基:参考周娇娇等[16]的培养基配方。
1.1.3 主要试剂和仪器各种DNA工具酶、T4 DNA连接酶、DNA Marker、RNase Free H2O,TaKaRa公司;DNA胶回收纯化试剂盒、PCR产物回收纯化试剂盒,生工生物工程(上海)股份有限公司;真菌总RNA提取试剂盒,Omega公司;反转录试剂盒(EasyScript One-Step gDNA Removal and cDNA Synthesis SuperMix),北京全式金生物技术有限公司;qPCR试剂盒(Bestar® SybrGreen qPCR mastermix),DBI Bioscience公司;溶壁酶(lysing enzyme from Trichoderma harzianum),Sigma公司;潮霉素和氨苄青霉素,Invitrogen公司。
荧光定量PCR仪,Applied Biosystem公司;PCR仪,Bio-Rad公司;冷冻离心机,Sigma公司;正置荧光显微镜,Olympus公司;凝胶成像系统,Siemens公司。
1.2 方法 1.2.1 组蛋白去乙酰化酶基因(hdac)干扰片段的设计与合成根据GenBank公布的hdac的基因序列(登录号为18489230)寻找上、下游1 000 bp片段分别命名为hdac-1和hdac-2,并设计带有筛选抗性基因同源臂的引物,分别命名为hdacF1、hdacR2、hdacF3和hdacR4。以pBC质粒上遗传霉素序列(pBC- GenneticinR)设计上、下游引物命名为hdac-MkF和hdac-MkR,引物序列见表 1。
| 引物名称 Primers name |
序列 Sequences (5′→3′) |
目的片段Target segment |
| hdacF1 | GGGCGTTGAGTGCTTGTTTG | hdac-1 |
| hdacR2 | CTATTTAACGACCCTGCCCTGAACCGGGCTTCATTTCCAGCCATTGTG | |
| hdacF3 | CTTACCGCTGTTGAGATCCAGTTCGATGGTTCCTAACGGGGGCTGTTTTG | hdac-2 |
| hdacR4 | GGGCAACAAACGGACATTTC | |
| hdac-MkF | CACAATGGCTGGAAATGAAGCCCGGTTCAGGGCAGGGTCGTTAAATAG | GentictinR (GR) |
| hdac-MkR | CAAAACAGCCCCCGTTAGGAACCATCGAACTGGATCTCAACAGCGGTAAG |
以基因组为模板,分别以hdacF1、hdacR2和hdacF3、hdacR4为引物,单独扩增hdac基因上、下游1 000 bp片段,并命名为hdac-1、hdac-2。以质粒pBC-GeneticinR为模板,以hdac-MkF和hdac-MkR为引物扩增GR片段。PCR反应体系(25 μL):ddH2O 13.75 μL,模板DNA 1 μL,上、下游引物(10 mmol/L)各1 μL,dNTPs (10 mmol/L) 1 μL,Mg2+ (25 mmol/L) 2 μL,5×Buffer 5 μL,GoTaq® DNA Polymerase (5 U/µL) 0.25 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 2 min,58 ℃ 30 s,72 ℃ 1 min,共35个循环;72 ℃ 5 min。分别以hdac-1和GR为模板,以hdacF1和hdac-MkR为引物扩增hdac-1-GR融合片段;分别以hdac-2和GR为模板,以hdac-MkF和hdacR4为引物扩增hdac-2-GR融合片段。PCR反应体系(20 μL):ddH2O 8 μL,2个模板DNA各0.5 μL,上、下游引物(10 mmol/L)各0.5 μL,2×Prime STAR® Max DNA Polymerase 10 μL。PCR反应条件:98 ℃ 2 min;98 ℃ 10 s,58 ℃ 5 s,72 ℃ 20 s,共35个循环;72 ℃ 7 min。将融合片段送至生工生物工程(上海)股份有限公司测序验证。
1.2.3 T. reesei QM9414原生质体的转化参考Wang等[17]的方法,用聚乙二醇进行里氏木霉原生质体的转化。转化时,用hdac-1-GR、hdac-2-GR各10 µg,PEG Buffer 200 µL (60% PEG4000,50 mmol/L CaCl2,10 mmol/L Tris-HCl,pH 7.5),涂布到含100 µg/mL遗传霉素的PDA固体平板用于筛选转化子,28 ℃培养2 d。用牙签挑取转化子接种到新的筛选培养基上,并连续筛选培养观察6代。
1.2.4 基因组DNA提取及突变体T. reesei Δhdac转化子的PCR验证将转化子接种于PDA平板上,28 ℃培养5 d后,制备孢子悬液接种于液体基本培养基中,28 ℃、250 r/min培养2 d。抽滤菌液,将菌丝体液氮研磨成粉后使用E.Z.N.A® Fungal DNA Kit提取基因组DNA。用NanoDrop 2000超微量分光光度计和琼脂糖凝胶电泳检测DNA样品的浓度和质量。
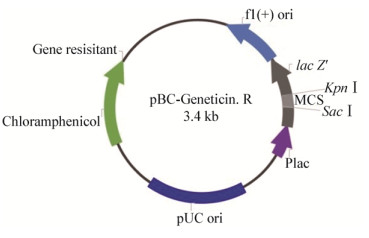
用基因组DNA为模板,以Ver-1F和Ver-1R及Ver-2F和Ver-2R为引物对分别扩增阳性转化子hdac-1、hdac-2片段上、下游约1 000 bp的验证序列Ver-1和Ver-2。PCR反应体系(25 μL):ddH2O 13.75 μL,模板DNA 1 μL,上、下游引物(10 mmol/L)各1 μL,dNTPs (10 mmol/L) 1 μL,Mg2+ (25 mmol/L) 2 μL,5×Buffer 5 μL,GoTaq® DNA Polymerase (5 U/µL) 0.25 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 2 min,58 ℃ 30 s,72 ℃ 1 min,共35个循环;72 ℃ 5 min。原理示意图见图 2,引物序列见表 2。
|
| 图 2 阳性转化子的PCR鉴定原理示意图 Figure 2 Schematic diagram for verifying positive transformant by PCR |
|
|
| 引物名称 Primers name |
序列 Sequences (5′→3′) |
目的片段 Target segment |
| hdac-Ver-1F | GGGCGTTGAGTGCTTGTTTG | Ver-1 |
| hdac-Ver-1R | TGAGAAGCACACGGTCAC | |
| hdac-Ver-2F | TCGGGGCGAAAACTCTC | Ver-2 |
| hdac-Ver-2R | CGGACTTTGCCTATTTGGCG |
以突变体Δhdac转化子DNA为模板,Ver-1F和Ver-1R及Ver-2F和Ver-2R为引物,实验组与对照组体系见表 3。PCR完成后在1%的琼脂糖凝胶电泳后切胶回收。取基因组DNA用适当的酶进行单酶切,37 ℃酶切4 h。1%琼脂糖凝胶电泳,在366 nm的长波紫外线下观察电泳结果。参考Dubeau等[18]的实验方法进行Southern blotting验证,并将结果采用图像仪扫描拍照留存。
| 反应试剂 Reaction reagent |
实验组 Test group (µL) |
对照组 Control group (µL) |
| GoTaq DNA Polymerase (5 U/µL) | 0.5 | 0.5 |
| 5×Green GoTaq Reaction Buffer | 10 | 10 |
| MgCl2 (25 mmol/L) | 4 | 4 |
| Ipmo5′ Test or Ipmo3′ Test | 2 | 2 |
| 5′ Test or 3′ Test | 2 | 2 |
| PCR DIG Mix (labeled) | 2 | 0 |
| dNTPs (unlabeled) | 0 | 2 |
| Template DNA | 1 | 2 |
| ddH2O | 28.5 | 28.5 |
| Total volume | 50 | 50 |
为分析hdac基因对纤维素酶活力的影响,将重组菌T. reesei Δhdac和出发菌株T. reesei QM9414分别接种于30 mL基本培养基中,每个菌株做3个平行样品,28 ℃、250 r/min培养48 h后,每种菌液取1.5 mL接种到30 mL含有0.1%微晶纤维素(质量分数)的产酶培养基中,28 ℃、250 r/min培养5 d,按照E.Z.N.A® Fungal RNA Kit说明书将菌丝使用液氮研磨成粉后提取总RNA。用NanoDrop 2000超微量分光光度计和琼脂糖凝胶电泳检测RNA样品浓度和质量。
1.2.7 RNA反转录和荧光定量PCR使用反转录试剂盒,以Random primer为引物,将约100 ng RNA反转录成cDNA。使用荧光定量PCR仪进行荧光定量PCR。PCR反应体系(20 µL):模板4 µL,ROX Reference Dye 0.4 µL,Bestar® SybrGreen qPCR Mastermix 10 µL,上、下游引物(20 µmol/L) (表 4)各0.5 µL,RNase Free H2O 4.6 µL。PCR反应条件:95 ℃ 2 min;95 ℃ 10 s,55 ℃ 31 s,40个循环。运行结束后得到融解曲线,用以核对PCR产物的特异性。所有PCR反应样品做3个复孔。目的基因表达量通过内参基因sar1校正。以对照菌株表达量为1,其他转化子的表达量与对照样品的比值作为数据分析。
| 引物名称 Primers name |
序列 Sequences (5′→3′) |
目的片段 Target segment |
| hdac-F | ACTTTCTGGCTGCTGTTC | hdac qPCR |
| hdac-R | ATCTCCTGTTCGTTGAATCG |
为分析hdac基因对纤维素酶活力的影响,将重组菌T. reesei Δhdac和出发菌株T. reesei QM9414分别接种于30 mL基本培养基中,每个菌株做3个平行样品,28 ℃、250 r/min培养48 h后,每种菌液取1.5 mL接种到30 mL含有0.1%微晶纤维素(质量分数)的产酶培养基中,28 ℃、250 r/min培养7 d,从第2天开始每天固定时间取发酵液1 mL,于4 ℃、12 000 r/min离心5 min,取上清液进行酶活测定。滤纸酶活力(filter paper activity,FPA)和羧甲基纤维素酶活力(carboxymethyl cellulase activity,CMCA)的测定参照Ghose[19]的方法。以CMC和Whatman No.1滤纸为底物,分别加酶液0.5 mL,50 ℃孵育30 min和1 h,加入DNS沸水浴5 min,测定OD540值,每个样品做3个重复,结果取平均值。以1 h内水解50 mg滤纸释放出2.0 mg葡萄糖或30 min内水解2%羧甲基纤维素放出0.5 mg葡萄糖的酶液稀释倍数来计算FPU和CMCase活性单位,单位为IU/mL。
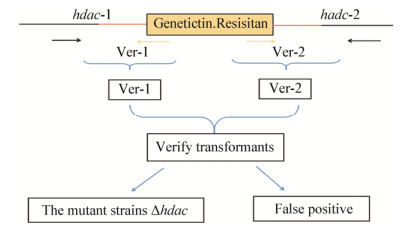
2 结果与分析 2.1 组蛋白去乙酰化酶基因敲除盒的构建以T. reesei QM9414基因组为模板,PCR扩增得到组蛋白去乙酰化酶基因上游1 000 bp左右的片段hdac-1 (1 006 bp)和下游1 000 bp左右的片段hdac-2 (1 023 bp),将PCR得到的目的片段进行1%的琼脂糖凝胶电泳,片段大小与预期相同,见图 3泳道1、3。同时,以pBC-GenetictinR质粒为模板,PCR扩增得到选择性标记抗性基因-遗传霉素抗性基因GR,大小为2 342 bp,见图 3泳道2。将3个片段送至生工生物工程(上海)股份有限公司测序,将得到的序列与测序结果经DNA assist软件比对,发现hdac-1和hdac-2与里氏木霉组蛋白去乙酰化酶基因上、下游各1 000 bp片段一致性达到100%,GR片段序列与原质粒上的序列一致性也达到100%。分别以hdac-1、GR片段和hdac-2、GR片段为模板,Overlap PCR扩增得到的融合片段hdac-1-GR和hdac-2-GR大小分别为3 348 bp和3 363 bp。将得到的融合片段进行1%凝胶电泳,片段大小与预期相同,其结果见图 3泳道4、5。
|
| 图 3 hdac基因敲除片段的琼脂糖凝胶电泳 Figure 3 Electrophoresis of fragments for knock-out of hdac gene 注:M:5 000 bp DNA分子标记;1:hdac-1片段(1 006 bp);2:GR片段(2 342 bp);3:hdac-2片段(1 023 bp);4:hdac-1-GR片段(3 348 bp);5:GR-hdac-2片段(3 363 bp). Note: M: 5 000 bp DNA ladder marker; 1: hdac-1 fragment (1 006 bp); 2: GR fragment (2 342 bp); 3: hdac-2 fragment (1 023 bp); 4: hdac-1-GR fragment (3 348 bp); 5: GR-hdac-2 fragment (3 363 bp). |
|
|
实验组平板上有大量凸起伴辐射状黄绿色菌落,对照组平板上无菌落,经筛选后共得到10株转化子,随机挑取3株转接到里氏木霉液体基本培养基中,28 ℃、250 r/min培养2 d,以转化子基因组为模板,以出发株基因为阴性对照,进行PCR验证。1%琼脂糖凝胶电泳结果见图 4。从突变株基因中扩增得到验证序列Ver-1 (1 005 bp)和Ver-2 (1 021 bp),说明目的基因整合到突变体T. reesei Δhdac的基因组中。
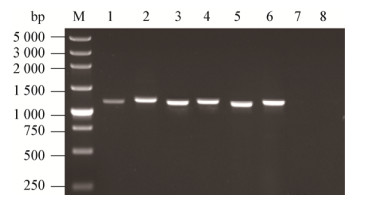
|
| 图 4 3株突变体T. reesei Δhdac转化子的Ver-1和Ver-2片段琼脂糖凝胶电泳 Figure 4 Electrophoresis of amplifiied Ver-1 and Ver-2 fragments from 3 mutant T. reesei Δhdac transformants 注:M:5 000 bp DNA分子标记;1−2,3−4,5−6:第1、2、3株T. reesei Δhdac Ver-1和Ver-2片段;7−8:T. reesei QM9414 Ver-1和Ver-2片段. Note: M: 5 000 bp DNA ladder marker; 1−2, 3−4, 5−6: The first, second, and third strain of T. reesei Δhdac Ver-1 and Ver-2 fragments; 7−8: T. reesei QM9414 Ver-1 and Ver-2 fragments. |
|
|
在遗传霉素基因内部、hdac基因内部及上下游3 000 bp和5 000 bp位置找到一个共同的酶切位点Nar I,酶切图谱见图 5A,T. reesei QM9414基因组和T. reesei Δhdac转化子敲除盒均被切成长度互不相同的两部分。T. reesei QM9414基因组两条条带大小分别为1 864 bp和6 456 bp,突变体T. reesei Δhdac转化子敲除盒中两条条带大小分别为2 293 bp和5 926 bp。Southern blotting条带图见图 5B。
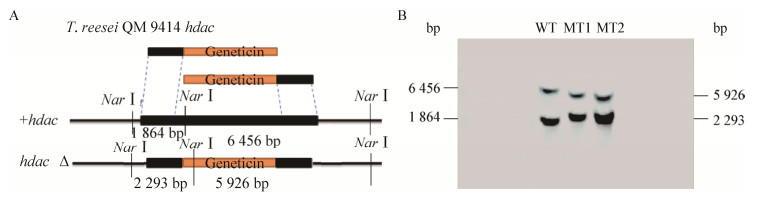
|
| 图 5 酶切位点示意图及敲除基因后Southern blotting的验证结果 Figure 5 Schematic site and southern blotting results after knocking out the gene 注:A:酶切位点示意图;B:敲除基因后Southern blotting结果. Note: A: Schematic site; B: Southern blotting results after knocking out the gene. |
|
|
取在固体培养基上的生长菌丝制成玻片在显微镜下观察,T. reesei Δhdac菌株菌丝较长,分枝较多,形态较好。表型图见图 6。
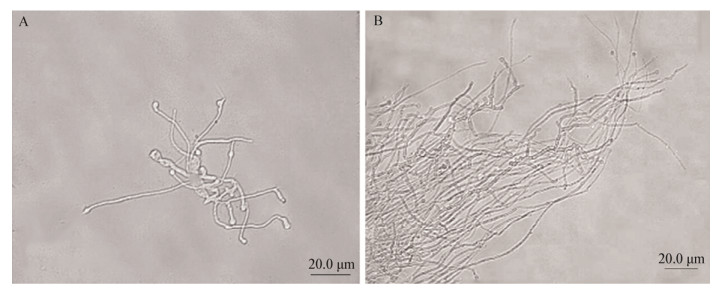
|
| 图 6 T. reesei QM9414及T. reesei Δhdac菌丝表型变化 Figure 6 Phenotypic changes in T. reesei QM9414 and T. reesei Δhdac 注:A:T. reesei QM9414菌丝表型变化;B:T. reesei Δhdac菌丝表型变化. Note: A: Phenotypic changes in T. reesei QM9414; B: Phenotypic changes in T. reesei Δhdac. |
|
|
滤纸酶活(FPA)代表的是纤维素酶的3种酶组分,即内切型葡聚糖酶、外切型葡聚糖酶、β-葡聚糖苷酶协同作用下的综合性酶活,是菌株整个纤维素酶系统活力水平的综合体现。T. reesei QM9414和T. reesei Δhdac的FPA和CMCA从第2−7天都呈先升后降的趋势,在第5天达到峰值。其中T. reesei Δhdac的两种酶活均高于出发菌株。T. reesei QM9414和T. reesei Δhdac的FPA和CMCA变化曲线见图 7。其中T. reesei Δhdac各个时段FPA和CMCA酶活力相比于T. reesei QM9414显著升高(P < 0.001),分别平均高出6.50、25.50 IU/mL,即突变菌株酶活平均分别增长了1.35、2.13倍,在峰值第5天时分别高出8.00、30.00 IU/mL,即突变菌株酶活分别增长了1.45、2.31倍。结果表明,敲除组蛋白去乙酰化酶基因可以显著提高纤维素酶的表达。
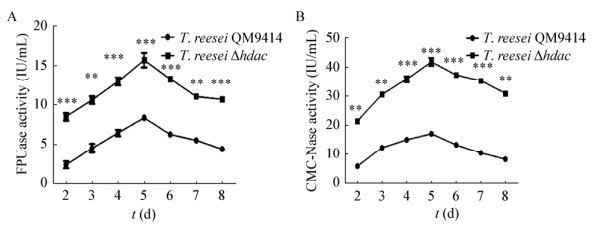
|
| 图 7 T. reesei Δhdac突变体FPA及CMCA酶活变化曲线 Figure 7 T. reesei Δhdac mutant FPA and CMCA enzyme activity curve 注:A:T. reesei Δhdac突变体FPA酶活变化曲线;B:T. reesei Δhdac突变体CMCA酶活变化曲线. Note: T. reesei Δhdac mutant FPA enzyme activity curve; B: T. reesei Δhdac mutant CMCA enzyme activity curve. **: P < 0.01; ***: P < 0.001, x±SD, n=3. |
|
|
对所有样品进行实时荧光定量PCR反应,使用qPCR Software 3.2软件对实验结果进行处理分析。实验数据经内参基因sar1定量值校正后,分别以出发菌株T. reesei QM9414中cbh1、egl1和xyr1的表达量为1,计算出培养第5天的T. reesei Δhdac中cbh1、egl1和xyr1相对表达量,结果如图 8所示,在诱导培养第5天时,T. reesei Δhdac的cbh1、egl1和xyr1的表达量与出发菌株T. reesei QM9414相比,分别约为出发菌株的6.50、6.01和4.51倍。
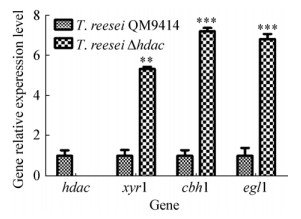
|
| 图 8 T. reesei QM9414和T. reesei Δhdac在第5天时纤维素酶基因表达量 Figure 8 Cellulase gene expression at the 5th day of T. reesei QM9414 and T. reesei Δhdac Note: **: P < 0.01; ***: P < 0.001, x, n=3. |
|
|
纤维素酶是起协同作用的多组分酶系,不仅具有内切葡聚糖酶和外切葡聚糖酶活性,还有很高活力的木聚糖酶。cbh1是纤维二糖水解酶CBH1的编码基因,egl1是内切β-1, 4-葡聚糖酶EGL1的编码基因。XYR1是主要纤维素酶和半纤维素酶基因,包括cbh1、cbh2、egl1、bgl1、xyn1和xyn2等转录所必需的激活因子。T. reesei Δhdac中cbh1、egl1和xyr1的表达量显著提高可能是由于组蛋白去乙酰化酶基因敲除后,组蛋白的乙酰化水平提高导致基因转录水平提高,激活因子xyr1转录水平提高,同时促进纤维素酶基因cbh1和egl1表达量再次提高。
3 讨论与结论里氏木霉是工业生产纤维素酶的重要菌种,为提高里氏木霉中纤维素酶的产量,需从纤维素酶表达调控系统入手进行研究。本研究就里氏木霉组蛋白去乙酰化酶基因缺失对菌体表型和纤维素酶的影响进行了探索,实现了重构基因hdac-1-GR和hdac-2-GR在T. reesei QM9414中对蛋白去乙酰化酶基因的同源替换,经酶活性测定发现突变体T. reesei Δhdac的FPA和CMCA酶活均高于出发菌株T. reesei QM9414,说明酶活性的提高与敲除组蛋白去乙酰化酶基因hdac有关,进一步对纤维素酶基因cbh1、egl1和木聚糖酶激活因子XYR1的mRNA表达水平进行了荧光定量PCR检测。结果显示,与出发菌相比,突变体T. reesei Δhdac中cbh1、egl1和xyr1的表达量均有显著提高,说明敲除组蛋白去乙酰化酶基因hdac可能使得乙酰化作用维持长久。有研究表明,cbh1基因上游的800 bp区域是转录激活因子XYRl的结合位点[20]。同时ChIP实验结果也显示,TATA盒的变化趋势与组蛋白乙酰化水平变化结果一致[21]。另一方面,激活因子xyr1转录水平提高后促进了纤维素酶基因cbh1、egl1转录。Shwab等[22]研究发现构巢曲霉的组蛋白去乙酰化酶基因主要控制青霉素和杂色曲霉素的合成,敲除该基因后其代谢产物发生明显改变,且基因簇的转录水平大大提高。另外,江艳萍[23]敲除了去乙酰化酶Tr26显著影响了菌株在逆境条件下的生长和发育状态,影响了阻遏或诱导条件下(半)纤维素降解相关酶类的表达。我们通过敲除组蛋白去乙酰化酶基因hdac后发现,表达纤维素酶及其相关基因转录水平明显提高,这与Shwab等[22]和江艳萍[23]的结果相一致,但本实验尚未检测相关代谢物产量的改变。本研究观察的T. reesei QM9414和T. reesei Δhdac均为正常生长条件,菌株的表型在去乙酰化酶基因缺失情况下生长表型未见明显变化,而对纤维素酶及相关基因的表达影响明显。综上,表观遗传修饰在激活沉默基因簇方面具有重要作用,对里氏木霉纤维素酶及相关的基因表达和调控具有重要意义。
本研究通过对纤维素酶酶活和mRNA水平等的实验结果分析,进一步确定了敲除组蛋白去乙酰化酶基因hdac可提高纤维素酶的产量,利用Split-Maker技术构建的相关基因敲除表达盒可以成功在T. reesei QM9414基因组中同源重组,得到含有遗传霉素抗性基因的突变株,为进一步研究和探讨里氏木霉纤维素酶基因的调控提供新思路和实验参考。
| [1] |
Martinez D, Berka RM, Henrissat B, et al. Genome sequencing and analysis of the biomass-degrading fungus Trichoderma reesei (syn. Hypocrea jecorina)[J]. Nature Biotechnology, 2008, 26(5): 553-560. DOI:10.1038/nbt1403 |
| [2] |
Zhang GT, Hartl L, Schuster A, et al. Gene targeting in a nonhomologous end joining deficient Hypocrea jecorina[J]. Journal of Biotechnology, 2009, 139(2): 146-151. |
| [3] |
Felsenfeld G, Boyes J, Chung J, et al. Chromatin structure and gene expression[J]. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(18): 9384-9388. DOI:10.1073/pnas.93.18.9384 |
| [4] |
Bonnaud EM, Suberbielle E, Malnou CE. Histone acetylation in neuronal (dys) function[J]. Biomolecular Concepts, 2016, 7(2): 103-116. |
| [5] |
Taunton J, Hassig CA, Schreiber SL. A mammalian histone deacetylase related to the yeast transcriptional regulator Rpd3p[J]. Science, 1996, 272(5260): 408-411. DOI:10.1126/science.272.5260.408 |
| [6] |
Lusser A, K lle D, Loidl P. Histone acetylation: lessons from the plant kingdom[J]. Trends in Plant Science, 2001, 6(2): 59-65. |
| [7] |
Baidyaroy D, Brosch G, Ahn JH, et al. A gene related to yeast HOS2 histone deacetylase affects extracellular depolymerase expression and virulence in a plant pathogenic fungus[J]. Plant Cell, 2001, 13(7): 1609-1624. DOI:10.1105/TPC.010168 |
| [8] |
Brosch G, Dangl M, Graessle S, et al. An inhibitor-resistant histone deacetylase in the plant pathogenic fungus Cochliobolus carbonum[J]. Biochemistry, 2001, 40(43): 12855-12863. DOI:10.1021/bi010508u |
| [9] |
Lan HH, Ye LQ, Ren SL, et al. Study on the function of histone deacetylase hosA gene of Aspergillus flavus[A]// 2015 Annual Meeting of Chinese Society of Fungi[C]. Shanghai: Mycological Society of China, 2015 (in Chinese) 蓝华辉, 叶柳青, 任思琳, 等.黄曲霉菌组蛋白去乙酰化酶hosA基因功能研究[A]//中国菌物学会2015年学术年会论文摘要集[C].上海: 中国菌物学会, 2015 |
| [10] |
Li YM, Wang CF, Wang GH, et al. Functional verification of Fusarium graminearum histone deacetylase gene (HDACs)[A]//Academic Conference of Chinese Society of Plant Pathology[C]. Kunming: Chinese Society for Plant Pathology, 2009 (in Chinese) 李依民, 王晨芳, 王光辉, 等.禾谷镰刀菌组蛋白去乙酰化酶基因(HDACs)功能验证[A]//中国植物病理学会2009年学术年会论文集[C].昆明: 中国植物病理学会, 2009 |
| [11] |
Fairhead C, Llorente B, Denis F, et al. New vectors for combinatorial deletions in yeast chromosomes and for gap-repair cloning using 'split-marker' recombination[J]. Yeast, 1996, 12(14): 1439-1457. DOI:10.1002/(SICI)1097-0061(199611)12:14<1439::AID-YEA37>3.0.CO;2-O |
| [12] |
Trushina N, Levin M, Mukherjee PK, et al. PacC and pH-dependent transcriptome of the mycotrophic fungus Trichoderma virens[J]. BMC Genomics, 2013, 14: 138. DOI:10.1186/1471-2164-14-138 |
| [13] |
Fu J, Hettler E, Wickes BL. Split marker transformation increases homologous integration frequency in Cryptococcus neoformans[J]. Fungal Genetics and Biology, 2006, 43(3): 200-212. |
| [14] |
Kim MS, Kim SY, Yoon JK, et al. An efficient gene- disruption method in Cryptococcus neoformans by double- joint PCR with NAT-split markers[J]. Biochemical and Biophysical Research Communications, 2009, 390(3): 983-988. DOI:10.1016/j.bbrc.2009.10.089 |
| [15] |
Gao YY, Zhong LY, Dong GY, et al. Trichoderma reseei constitutive type of expression vector siRNA interfering cre1 gene on cellulases expression regulation[J]. Journal of Microbiology, 2018, 38(1): 12-19. (in Chinese) 高云雨, 钟路遥, 董冠园, 等. 里氏木霉组成型表达siRNA干扰cre1基因对纤维素酶表达的调控作用[J]. 微生物学杂志, 2018, 38(1): 12-19. DOI:10.3969/j.issn.1005-7021.2018.01.002 |
| [16] |
Zhou JJ, She WY, Wang HR, et al. Effect of 5-Aza-2'-deoxycytidine on the expression of cellulases in Trichoderma reesei[J]. Journal of Shenzhen University (Science and Engineering), 2017, 34(2): 122-131. (in Chinese) 周娇娇, 佘炜怡, 王浩入, 等. 5-氮杂-2-脱氧胞苷对里氏木霉产纤维素酶的影响[J]. 深圳大学学报:理工版, 2017, 34(2): 122-131. |
| [17] |
Wang SW, Xing M, Liu G, et al. Improving cellulase production in Trichoderma koningii through RNA interference on ace1 gene expression[J]. Journal of Microbiology and Biotechnology, 2012, 22(8): 1133-1140. DOI:10.4014/jmb.1112.12037 |
| [18] |
Dubeau L, Chandler LA, Gralow JR, et al. Southern blot analysis of DNA extracted from formalin-fixed pathology specimens[J]. Cancer Research, 1986, 46(6): 2964-2969. |
| [19] |
Ghose TK. Measurement of cellulase activities[J]. Pure and Applied Chemistry, 1987, 59(2): 257-268. DOI:10.1351/pac198759020257 |
| [20] |
Furukawa T, Shida Y, Kitagami N, et al. Identification of specific binding sites for XYR1, a transcriptional activator of cellulolytic and xylanolytic genes in Trichoderma reesei[J]. Fungal Genetics and Biology, 2009, 46(8): 564-574. DOI:10.1016/j.fgb.2009.04.001 |
| [21] |
Seiboth B, Karimi RA, Phatale PA, et al. The putative protein methyltransferase LAE1 controls cellulase gene expression in Trichoderma reesei[J]. Molecular Microbiology, 2012, 84(6): 1150-1164. |
| [22] |
Shwab EK, Bok JW, Tribus M, et al. Histone deacetylase activity regulates chemical diversity in Aspergillus[J]. Eukaryotic Cell, 2007, 6(9): 1656-1664. DOI:10.1128/EC.00186-07 |
| [23] |
Jiang YP. Study on the mechanism of regulation proteins related to cellulase synthesis of Trichoderma[D]. Beijing: Doctoral Dissertation of University of Chinese Academy of Sciences, 2015 (in Chinese) 江艳萍.里氏木霉中纤维素酶合成相关调控蛋白的作用机制研究[D].北京: 中国科学院大学博士学位论文, 2015 |
 2020, Vol. 47
2020, Vol. 47