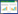扩展功能
文章信息
- 李艳鹏, 田越欣, 郝志丹, 马艳玲
- LI Yan-Peng, TIAN Yue-Xin, HAO Zhi-Dan, MA Yan-Ling
- 基于转录组分析铜绿假单胞菌DN1降解荧蒽特性
- Transcriptomic analysis of Pseudomonas aeruginosa DN1 upon degradation of fluoranthene
- 微生物学通报, 2020, 47(1): 54-65
- Microbiology China, 2020, 47(1): 54-65
- DOI: 10.13344/j.microbiol.china.190463
-
文章历史
- 收稿日期: 2019-05-28
- 接受日期: 2019-09-04
- 网络首发日期: 2019-09-27
多环芳烃(polycyclic aromatic hydrocarbons,PAHs)是一类由2个或2个以上苯环构成的稠环化合物,主要源于森林草地火灾、石油渗漏以及垃圾焚烧、石油、沥青质等不完全燃烧[1-2]。由于PAHs的毒性和“三致性”(致突变性,致畸性,致癌性),严重威胁着生态系统,尤其是人类的健康[3]。因此,多环芳烃的去除是环境科学家们共同关心的问题[4]。其中,微生物降解是去除环境中多环芳烃污染物的最主要途径,具有成本低、效率高、环境友好性等特点[5]。大量研究表明,自然界中存在许多可高效降解PAHs的菌株,如分枝杆菌属(Mycobacterium)[6]、红球菌属(Rhodococcus)[7]、不动杆菌属(Acinetobacter)[8]、假单胞菌属(Pseudomonas)[9]等,为环境保护与污染防治提供了菌种资源。假单胞菌属作为最有效的PAHs降解细菌属之一,它们能够利用碳氢化合物作为碳源生长,并在多环芳烃类污染环境中生存[9-10]。
目前,关于烃类污染物降解菌的降解机理研究大都从分子水平上对菌株的基因及基因簇进行探索,而从转录组测序与分析上揭示微生物降解机理及功能的研究工作还比较少。转录组测序反映了微生物细胞在一定条件下生理活动中基因的差异表达,直接揭示了参与特定处理过程的相关基因及其转录状态变化。Goncalves等[11]通过分析红球菌RHA1的转录组数据,发现联苯和乙苯胁迫条件下320个功能基因发生上调表达,而在苯甲酸诱导下只有65个基因上调表达,同时还发现有22个酶(含8个新酶)可能参与了联苯的降解过程。于红艳等[12]利用比较转录组分析克雷白氏肺炎杆菌Tzyx1对多环芳烃的降解特性,发现正常培养和多环芳烃胁迫培养条件下,得到254个显著性差异表达基因。因此,分析转录组可以获得RNA水平上基因的表达信息。荧蒽作为一种高分子量多环芳烃,与低分子量多环芳烃如菲或萘相比,其结构更加稳定,属于一种难降解的持久性有机污染物。本课题组前期从长庆油田石油污染土壤中分离筛选到一株具有石油烃降解功能的菌株,测序并命名为铜绿假单胞菌(Pseudomonas aeruginosa) DN1,NCBI登录号为CP017099[13-14]。全基因组测序结果显示,菌株DN1中含有100个以上可能参与降解途径的烃类降解相关基因[15]。本研究进一步对菌株DN1进行转录组测序,比较正常培养和荧蒽诱导培养这两种条件下基因的差异表达情况,寻找表型与基因型之间的相互关系,以揭示荧蒽的降解路径和特性,这些研究有助于进一步研究菌株DN1降解荧蒽的生理生化和遗传机制,有利于阐明多环芳烃的微生物降解机制,为多环芳烃生物修复技术的应用提供理论指导与科学依据。
1 材料与方法 1.1 材料 1.1.1 菌株铜绿假单胞菌(Pseudomonas aeruginosa)菌株DN1由本实验室从石油污染土壤中分离所得。
1.1.2 主要试剂和仪器及培养基蛋白胨和酵母浸粉,Oxoid公司;普通化学试剂药品,天津市科密欧化学试剂有限公司。恒温培养箱,上海智城分析仪器有限公司。LB培养基(g/L):蛋白胨10.0,酵母浸粉5.0,氯化钠10.0;MSM无机盐培养基(g/L):硫酸铵1.0,磷酸氢二钾7.0,磷酸二氢钾3.0,硫酸镁0.1,氯化钠0.5,pH 7.2。1×105 Pa灭菌30 min。
1.2 培养条件将保存的菌株DN1于LB中活化后,挑取单克隆接种于LB液体培养基中,37 ℃、200 r/min培养至对数期,4 ℃、8 000 r/min离心5 min收集菌体。无菌水洗涤菌体3次,转接入MSM无机盐培养基中。对照组及实验组分别以50 mg/L葡萄糖和荧蒽为唯一碳源,37 ℃、200 r/min培养5 d。然后分别以4 ℃、8 000 r/min离心5 min收集菌体。葡萄糖-MSM培养基培养的菌体样品命名为CK,荧蒽-MSM培养基培养的菌体样品命名为EF,每组设立3个平行。
1.3 RNA提取低温保存菌体样品,送至广州赛哲生物科技有限公司进行RNA的提取及质量检测,利用Illumina高通量测序平台HiSeq PE 150进行RNA-seq转录组测序。
1.4 转录组数据组装及注释测序得到的原始数据(raw reads),包括Reads的序列及碱基的测序质量。为保证数据质量,首先要对数据过滤以减少数据噪音,包括:去除低质量(质量值SQ≤20的碱基数占整条Read的50%以上)、测序接头和含N比例大于10%的Reads。将质量控制后的High quality clean reads利用Bowtie工具比对到核糖体数据库过滤掉核糖体的序列,之后利用TopHat2比对软件比对到参考基因组上,Cufflinks进行组装、Cuffmerge合并,得到包括已知基因和一些新基因的完整基因[16-17]。比对好的基因分别在NCBI、GO、KEGG进行比对注释[18-19]。
1.5 差异基因表达分析及富集分析根据CK、EF与参考基因组比对结果,利用FPKM (fragments per kilobase per million)方法计算基因的表达量,并以此作为每个转录本/基因在样品中各自的表达量[17, 20]。之后利用错误发生率(false discovery rate,FDR)和差异表达量倍数在2倍以上的基因(Log2FC;FC:folds change)来筛选差异基因,通过筛选FDR < 0.05且|Log2FC| > 1的差异表达基因用于后续分析[21]。经过多重检验校正后,选择Q-value≤0.05的基因进行差异表达基因富集分析,确定差异表达基因参与的最主要的生化代谢途径、信号转导途径及差异基因行使的主要生物学功能。
2 结果与分析 2.1 铜绿假单胞菌株DN1在荧蒽胁迫环境下的转录组学分析利用Illumina高通量测序平台HiSeq PE 150对荧蒽胁迫条件下菌株DN1进行转录组学测序,两个样本分别得到28 325 987条(CK、对照组、葡萄糖条件)和18 579 321条(EF、实验组、荧蒽条件)序列。将这些有效序列分别比对到核糖体和参考基因组DN1 (CP017099)上过滤之后,分别得到28 098 931条和18 434 341条序列。能够比对到基因组多个位置的被称为Multiple matched Reads,这种Reads可信度较低,一般不用作基因表达差异分析;能够比对到基因组唯一位置的被称为Unique matched reads,可信度高。因此统计Unique matched reads用于后续分析,以保证分析结果的可信度。如表 1所示,能够比对到基因组上的匹配序列分别占有效序列的99.19% (CK)和99.22% (EF),其中完全匹配的序列(mapping ratio)分别为82.34%和80.51%,而Multiple matched reads的比例较低,说明测序质量较好。
| Sample | CK | EF |
| Total reads | 28 098 931 | 18 434 341 |
| Mapped reads | 23 379 855 (82.34%) | 14 862 426 (80.51%) |
| Multiple matched reads | 635 162 (2.80%) | 476 928 (2.55%) |
| Unique matched reads | 20 825 415 (74.27%) | 14 385 498 (77.96%) |
基因表达量通常使用FPKM,即每百万测序碱基中匹配到每千个转录子测序碱基中所包含的测序片段数。FPKM只计算2个Reads能比对到同一个转录本的Fragments数量。FPKM法通过归一化处理可以消除组间差异基因如长度差异及转录组测序量差异对基因表达量的影响,并将归一化后的基因表达量直接用于分析组间样品之间的基因表达差异。利用错误发生率(FDR)和差异表达量倍数在2倍以上的基因(log2FC;FPKM-EF/FPKM-CK)的差异基因筛选方法,通过筛选FDR < 0.05且|Log2FC| > 1的差异表达基因用于后续分析。其中FPKM-EF表示荧蒽诱导下菌株DN1的基因表达量,FPKM-CK表示对照组葡萄糖诱导下菌株DN1的基因表达量。
根据铜绿假单胞菌株DN1的转录组数据分析差异表达基因(differentially expressed genes,DEGs),荧蒽(EF)和葡萄糖(CK)诱导下的RNA-Seq数据分别得到了6 189个和6 144个基因。其中,菌株在荧蒽诱导处理后共发现3 522个显著差异表达基因,其中1 919个基因(31.01%)上调表达,1 603个基因(25.90%)下调表达。转录组中显著性差异表达基因的可视化火山图如图 1所示。图 1横坐标表示2个样品的差异倍数的对数值,纵坐标表示2个样品的FDR的负lg值;红色和绿色的点分别表示基因表达量显著上调和下调,蓝色的点表示差异不显著,显著性差异的判断标准为以FDR < 0.05,差异倍数在2倍以上为准。

|
| 图 1 荧蒽与葡萄糖诱导后差异表达基因火山图 Figure 1 Volcano plot of differentially expressed genes induced by fluoranthene and glucose respectively |
|
|
Gene ontology (GO)分析是利用GO数据库对差异表达基因按照生物学过程(biological process)、分子功能(molecular function)及细胞组分(cellular component)三大功能类别进行GO功能分类。为了获取到菌株DN1在荧蒽胁迫条件下转录组学完整的基因功能信息,将实验组和对照组分析得到的差异基因进行了GO二级水平上功能富集分析,共得到2 333个显著性差异表达基因,(其中显著性上调基因有580个,下调基因有1 803个),下调基因占DEGs的77.3%,说明荧蒽对细胞有很大的毒性,会导致其代谢活性减弱。其中参与生物学过程、分子功能、细胞组分的比例分别为39.95%、28.76%、31.29% (图 2)。

|
| 图 2 差异表达基因GO富集分析 Figure 2 GO enrichment results of differentially expressed genes |
|
|
菌株DN1在降解荧蒽过程中,参与底物识别、结合转运、酶催化活性及生物降解代谢过程的相关基因发生了显著性差异表达。整体上来说,差异表达基因数量最多的两大类属于生物过程和细胞组分,分别为932个和730个,且差异表达基因主要归属于细胞过程(cellular progress)、代谢过程(metabolic process)、细胞(cell)、细胞组分(cell part)等功能分类,分别有292、278、233和223个差异表达基因。此外,分子功能类别之中变化最大的功能基因类别为催化活性(catalytic activity)、结合(binding),分别含有318、250个差异表达基因。
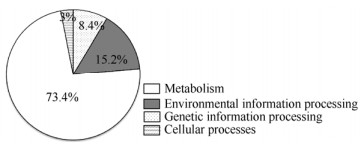
进一步分析“细胞过程”这个功能类别的相关差异表达基因,发现这些差异基因上调表达量超过8倍的基因转录本主要归类于膜转运(membrane transport)、氨基酸代谢(amino acid metabolism)、其他氨基酸代谢(metabolism of other amino acids)、能量代谢(energy metabolism)、辅助因子和维生素代谢(metabolism of cofactors and vitamins)等相关过程(表 2)。这些过程中的差异基因可能密切参与了菌株DN1降解荧蒽过程中底物识别、底物转运、信号转运以及生物降解等生理过程。
| Main class | Gene ID | Expression difference using Log2 ratio | KEGG pathway | KEGG ID |
| Cellular process | BG483_RS25255 | 5.026 776 494 | − | − |
| BG483_RS18640 | 4.674 491 400 | Membrane transport | K03227 | |
| BG483_RS12905 | 3.822 162 841 | Metabolism of other amino acids | K06163 | |
| BG483_RS30820 | 3.501 592 104 | − | − | |
| BG483_RS24070 | 3.496 614 276 | Membrane transport | K02460 | |
| BG483_RS03855 | 3.471 566 862 | − | − | |
| BG483_RS05110 | 3.460 362 137 | Amino acid metabolism | K01953 | |
| BG483_RS28200 | 3.382 398 426 | − | − | |
| BG483_RS18175 | 3.229 306 461 | Energy metabolism | K00362 | |
| BG483_RS12915 | 3.223 861 296 | Metabolism of other amino acids | K06165 | |
| BG483_RS18610 | 3.197 312 788 | Membrane transport | K04058 | |
| Metabolic process | BG483_RS03860 | 5.612 810 047 | − | − |
| BG483_RS25255 | 5.026 776 494 | − | − | |
| BG483_RS12905 | 3.822 162 841 | Metabolism of other amino acids | K06163 | |
| BG483_RS30820 | 3.501 592 104 | − | − | |
| BG483_RS03855 | 3.471 566 862 | − | − | |
| BG483_RS05110 | 3.460 362 137 | Amino acid metabolism | K01953 | |
| BG483_RS11205 | 3.422 734 74 | − | − | |
| BG483_RS28200 | 3.382 398 426 | − | − | |
| BG483_RS18175 | 3.229 306 461 | Energy metabolism | K00362 | |
| BG483_RS12915 | 3.223 861 296 | Metabolism of other amino acids | K06165 | |
| BG483_RS03870 | 3.196 715 274 | − | − | |
| BG483_RS06855 | 3.114 692 039 | Amino acid metabolism; Global and overview | K00836 | |
| Cell part | BG483_RS18590 | 5.666 837 645 | − | − |
| BG483_RS06095 | 4.087 070 697 | − | − | |
| BG483_RS30820 | 3.501 592 104 | − | − | |
| BG483_RS24070 | 3.496 614 276 | Membrane transport | K02460 | |
| BG483_RS04965 | 3.437 411 344 | Membrane transport | K13893 | |
| BG483_RS28200 | 3.382 398 426 | − | − | |
| BG483_RS18610 | 3.197 312 788 | Membrane transport | K04058 | |
| Metabolic process | BG483_RS03860 | 5.612 810 047 | − | − |
| BG483_RS25255 | 5.026 776 494 | − | − | |
| BG483_RS00650 | 4.468 132 403 | − | − | |
| BG483_RS03810 | 4.312 940 401 | Metabolism of terpenoids and polyketides | K12241 | |
| BG483_RS12905 | 3.822 162 841 | Metabolism of other amino acids | K06163 | |
| BG483_RS30820 | 3.501 592 104 | − | − | |
| BG483_RS03855 | 3.471 566 862 | − | − | |
| BG483_RS05110 | 3.460 362 137 | Amino acid metabolism | K01953 | |
| BG483_RS11205 | 3.422 734 74 | − | − | |
| BG483_RS16960 | 3.304 509 029 | − | − | |
| BG483_RS18175 | 3.229 306 461 | Energy metabolism | K00362 | |
| BG483_RS12915 | 3.223 861 296 | Metabolism of other amino acids | K06165 | |
| BG483_RS03870 | 3.196 715 274 | − | − | |
| BG483_RS06855 | 3.114 692 039 | Amino acid metabolism; Global and overview | K00836 | |
| Note: −: none. | ||||
详细分析“细胞组分”这个功能类别的相关差异表达基因,获悉这些差异表达基因的功能主要集中在膜转运(membrane transport)、能量代谢(energy metabolism)、信号转导(signal transduction)、碳水化合物代谢(carbohydrate metabolism)等相关过程。其中,在上调的40个“细胞组分”转录本中有14个转录本归类于“膜转运”,而且表达量高于8倍以上的基因主要为“膜转运”基因(表 2),这个结果表明菌株DN1在降解荧蒽的过程中,细胞组分中一些参与膜转运的基因可能会发挥其作用,参与荧蒽的识别,协助荧蒽跨膜转运,进而完成降解作用。
2.4 差异表达基因KEGG pathway富集分析对菌株DN1转录组的差异表达基因进行KEGG代谢途径分析,以进一步了解这些基因可能参与的生物学过程。分析结果显示,共有1 918个差异表达基因匹配到了112个KEGG代谢途径。主要分为四大类别:遗传信息处理(genetic information processing)、环境信息处理(environmental information processing)、细胞过程(cellular processes)、代谢(metabolism)。在KEGG注释信息中,1 408个基因注释到Metabolism pathway,约占总差异基因的73.4% (图 3所示),其中分别有269个基因属于“Global and overview”和“Amino acid metabolism”(图 4所示),在所有分类中所占比例最高,约占1 918个功能基因中的14.03%;其次,“Carbohydrate metabolism”、“Membrane transport”、“Metabolism of cofactors and vitamins”在总功能基因数中所占比例次之,分别为252个(13.14%)、190个(9.91%)、121个(6.31%)。
|
| 图 3 KEGG一级分类 Figure 3 KEGG classification in A class |
|
|
|
| 图 4 差异表达基因KEGG二级分类 Figure 4 KEGG classification of differentially expressed genes in B class |
|
|
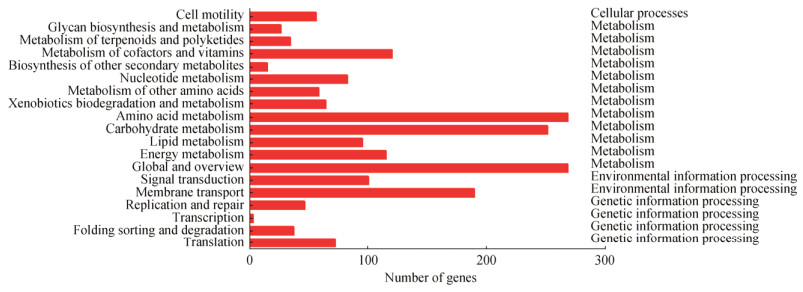
菌株DN1中含有317个基因参与了碳氢化合物代谢及含有苯环结构异源生物质的生物降解,如图 5所示,在Carbohydrate metabolism类别中,含有15条不同的代谢途径(图 5A),Xenobiotics biodegradation and metabolism类别中,含有14条不同的代谢途径(图 5B)。其中在Xenobiotics biodegradation and metabolism途径中,这些途径主要与苯环结构的代谢有关,包括:苯甲酸盐降解(benzoate degradation,ko00362)、多环芳烃降解(polycyclic aromatic hydrocarbon degradation,ko00624)、氨基苯甲酸酯降解(aminobenzoate degradation,ko00627)、氟苯甲酸降解(fluorobenzoate degradation,ko00364)、甲苯降解(toluene degradation,ko00623)、二甲苯降解(xylene degradation,ko00622)、萘降解(naphthalene degradation,ko00626)和苯乙烯降解(styrene degradation,ko00643)等。这些降解途径与我们之前的研究结果大体一致,菌株DN1可以有效地降解多环芳烃类化合物,如荧蒽、芘、萘、菲[13]。全基因组测序结果显示,菌株DN1含有100多个PAHs降解相关基因,包括多种双加氧酶及降解转运蛋白,气相色谱-质谱联用结果显示,菌株DN1降解荧蒽的第一步是通过在苯环上加氧而引发的[15]。
|
| 图 5 不同KEGG pathways基因富集分析 Figure 5 Gene enrichment analysis of different KEGG pathways 注:A:碳水化合物代谢;B:异源生物质降解及代谢. Note: A: Carbohydrate metabolism; B: Xenobiotics biodegradation and metabolism. |
|
|
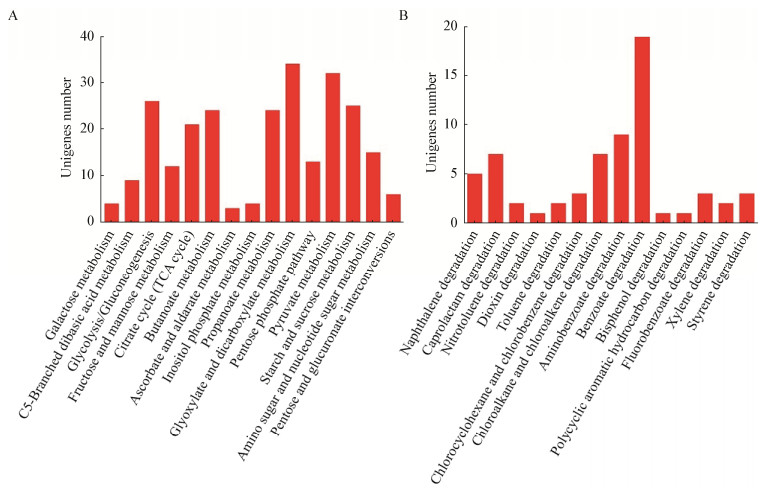
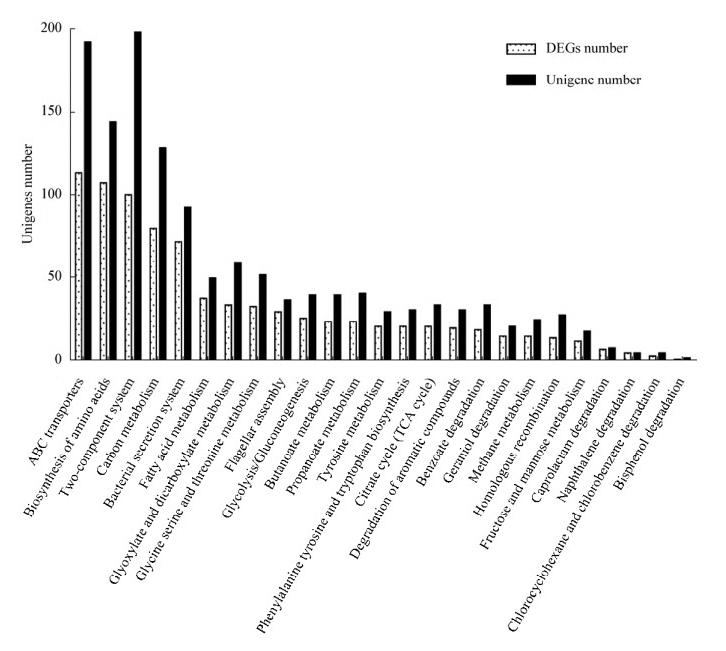
此外,菌株DN1转录组的差异表达基因主要集中在ABC转运系统(ABC transporters,ko02010)、氨基酸生物合成(biosynthesis of amino acids,ko01230)、双组分系统(two-component system,ko02020)及碳代谢(carbon metabolism,ko01200),这些途径大多数参与了底物的识别转运、信号转导及基因表达调控。如图 6所示,黑色柱形图表示差异基因在KEGG代谢通路中的所有显著性表达基因数量,带点的白色方框表示所有差异表达基因中的显著性表达上调的基因数量。同时,利用KEGG数据库比对分析到菌株DN1转录组数据中,发现含有大量差异表达基因可能与多环芳烃的生物降解有关,且其差异基因比例较高。例如:129个碳代谢基因中有80个基因发生显著性差异表达,约占总数的62.02%;31个芳烃降解基因中有20个基因发生显著性差异表达;34个苯甲酸降解基因中有19个基因显著性差异表达;除此之外,还有9个氨基苯甲酸酯降解基因,5个萘降解基因,3个氯环己烷及氯苯降解基因。这些参与芳香族化合物生物降解过程的差异表达基因,在荧蒽诱导下发生显著性差异表达,表现出对荧蒽的响应,因此预测它们可能参与了荧蒽的生物降解途径。菌株DN1转录组中还发现有11个差异表达基因匹配到细菌趋化系统(bacterial chemotaxis,ko02030),这些基因可能参与了荧蒽微生物降解过程中的底物响应。部分可能与菌株DN1降解荧蒽相关的差异表达基因如表 3所示。
|
| 图 6 主要代谢通路中的基因表达及差异表达基因情况 Figure 6 The unigene number and differently expressed genes in primary KEGG pathway |
|
|
| Gene ID | Gene length (bp) | Log2FC | Up & Down | P-value | FDR | KEGG Pathway | Annotation |
| BG483_RS00835 | 720 | 1.031 076 58 | Up | 3.43E–06 | 7.20E–06 | Benzoate degradation, Degradation of aromatic compounds | Protocatechuate 3, 4-dioxygenase subunit beta |
| BG483_RS01255 | 1 905 | 2.019 564 592 | Up | 1.98E–18 | 1.39E–17 | Ubiquinone and other terpenoid-quinone biosynthesis, Phenylalanine metabolism | 4-hydroxyphenylpyruvate dioxygenase |
| BG483_RS01265 | 855 | 2.401 643 608 | Up | 5.83E–16 | 3.19E–15 | Biosynthesis of amino acids, Phenylalanine, tyrosine and tryptophan biosynthesis | Shikimate dehydrogenase |
| BG483_RS01270 | 447 | 2.624 049 285 | Up | 3.99E–05 | 7.53E–05 | Phenylalanine, tyrosine and tryptophan biosynthesis, Biosynthesis of amino acids | 3-dehydroquinate dehydratase |
| BG483_RS01280 | 1 185 | 3.835 990 355 | Up | 2.56E–44 | 3.33E–42 | Degradation of aromatic compounds, Benzoate degradation | 4-hydroxybenzoate 3-monooxygenase |
| BG483_RS07030 | 1 257 | 1.729 866 809 | Up | 1.27E–18 | 9.05E–18 | Glycine, serine and threonine metabolism, Methane metabolism… | Serine hydroxymethyltransferase |
| BG483_RS07190 | 1 062 | 1.545 688 669 | Up | 4.79E–11 | 1.58E–10 | Tyrosine metabolism | Gentisate 1, 2-dioxygenase |
| BG483_RS07205 | 645 | 2.130 074 362 | Up | 4.53E–13 | 1.82E–12 | Styrene degradation, Tyrosine metabolism | Glutathione S-transferase |
| BG483_RS07215 | 1 335 | 1.899 689 222 | Up | 1.52E–21 | 1.43E–20 | Styrene degradation, Tyrosine metabolism | Cytochrome P450 |
| BG483_RS07375 | 933 | –1.743 486 398 | Down | 9.40E–13 | 3.66E–12 | Degradation of aromatic compounds, Benzoate degradation… | Catechol 1, 2-dioxygenase |
| BG483_RS07410 | 1 395 | –3.188 680 001 | Down | 6.35E–16 | 3.45E–15 | Aminobenzoate degradation | Anthranilate 1, 2-dioxygenase large subunit |
| BG483_RS07415 | 492 | –2.142 131 92 | Down | 1.18E–11 | 4.14E–11 | Aminobenzoate degradation | Anthranilate 1, 2-dioxygenase small subunit |
| BG483_RS07420 | 1 023 | –1.279 372 905 | Down | 1.58E–06 | 3.42E–06 | Aminobenzoate degradation | Anthranilate dioxygenase reductase |
| BG483_RS07435 | 489 | 1.785 138 515 | Up | 5.47E–07 | 1.24E–06 | Degradation of aromatic compounds, Benzoate degradation… | Benzoate 1, 2-dioxygenase small subunit |
| BG483_RS07465 | 675 | 1.181 690 512 | Up | 9.28E–07 | 2.05E–06 | Two-component system | DNA-binding response regulator |
| BG483_RS07485 | 3 111 | 1.494 048 484 | Up | 2.64E–06 | 5.61E–06 | Two-component system | Resistance-nodulation-cell division (RND) efflux transporter |
| BG483_RS07490 | 3 132 | 1.278 898 07 | Up | 3.45E–05 | 6.55E–05 | Two-component system | Resistance-nodulation-cell division (RND) efflux transporter |
| BG483_RS07520 | 1 125 | 1.319 427 761 | Up | 4.19E–11 | 1.40E–10 | Valine, leucine and isoleucine degradation, Propanoate metabolism, Carbon metabolism… | Aminotransferase |
| BG483_RS07620 | 1 230 | –3.730 142 846 | Down | 1.09E–17 | 7.07E–17 | Valine, leucine and isoleucine degradation, Propanoate metabolism, Carbon metabolism… | Acyl-CoA dehydrogenase |
| BG483_RS08230 | 1 104 | 1.359 200 945 | Up | 7.14E–11 | 2.32E–10 | ABC transporters | Spermidine/putrescine-binding protein |
| BG483_RS08245 | 957 | 2.974 392 638 | Up | 1.72E–29 | 3.55E–28 | ABC transporters, Sulfur metabolism | Hypothetical protein |
| BG483_RS08250 | 987 | 2.069 319 072 | Up | 9.90E–16 | 5.29E–15 | ABC transporters, Sulfur metabolism | Aliphatic sulfonate-binding protein |
| BG483_RS08540 | 372 | 2.874 011 605 | Up | 3.10E–05 | 5.90E–05 | Two-component system | Carbon storage regulator |
| BG483_RS00940 | 1 050 | 2.823 812 513 | Up | 1.85E–27 | 3.08E–26 | Bacterial chemotaxis, Two-component system | Chemotaxis response regulator protein-glutamate methylesterase |
| BG483_RS00950 | 843 | 2.689 517 322 | Up | 2.05E–20 | 1.70E–19 | Bacterial chemotaxis, Two-component system | Chemotaxis protein CheR |
| BG483_RS00960 | 486 | 1.140 496 008 | Up | 0.005 852 525 | 0.008 748 663 | Bacterial chemotaxis, Two-component system | Chemotaxis protein CheW |
| BG483_RS00965 | 1 920 | 1.795 457 661 | Up | 5.50E–16 | 3.02E–15 | Bacterial chemotaxis, Two-component system | Chemotaxis protein CheA |
| BG483_RS12800 | 1 158 | 1.306 371 189 | Up | 6.05E–10 | 1.79E–09 | ABC transporters | Aliphatic amidase expression-regulating protein |
| BG483_RS12930 | 795 | 2.912 435 81 | Up | 7.78E–24 | 9.23E–23 | ABC transporters | Phosphonate transporter PhnE |
| BG483_RS12935 | 1 005 | 3.100 463 528 | Up | 4.23E–36 | 1.77E–34 | ABC transporters | Phosphonate ABC transporter substrate-binding protein |
| BG483_RS12940 | 837 | 2.903 400 852 | Up | 1.06E–24 | 1.36E–23 | ABC transporters | Phosphonate ABC transporter ATP-binding protein |
荧蒽胁迫条件下,DN1菌株中双加氧酶基因及荧蒽降解过程中重要的代谢途径龙胆酸、苯甲酸、儿茶酚及原儿茶酸降解等的基因(簇)表达量普遍显著上调,与这些代谢途径基因距离较近或处于同一基因簇的ABC转运系统蛋白表达量显著提高,如:ABC转运蛋白通透酶、磺酸盐ABC转运蛋白-底物结合蛋白、ABC转运蛋白-ATP结合蛋白等。与此同时,转录组数据分析表明:在荧蒽降解过程中,趋化性蛋白及信号传导蛋白的表达量显著提高,如RS00950、RS00960及RS00965编码的趋化蛋白CheR、CheW、CheA,RS00940及RS32695编码的趋化反应调节蛋白-谷氨酸甲酯酶和混合型传感组氨酸激酶/反应调节剂调节蛋白,这些基因的显著性表达是对荧蒽胁迫条件的应答及信号传递。
3 讨论与结论目前关于铜绿假单胞菌降解烃类化合物的报道已有很多,但从转录组水平对铜绿假单胞菌降解特性研究的报道还较少[22-23]。本研究采用Illumina HiSeq PE 150高通量测序技术对正常培养组和荧蒽诱导组条件下菌株DN1进行转录组学测序,结果显示两组样品在转录组文库中的基因覆盖度均占98%以上,且能够比对到基因组上的匹配序列分别占有效序列的99.19% (葡萄糖条件)和99.22% (荧蒽条件),其中完全匹配的序列(mapping ratio)分别为82.34%和80.51%,而Multiple matched reads的比例较低,说明测序质量较好。
虽然铜绿假单胞菌株DN1已经完成了全基因组测序,但是各种烃类污染物胁迫条件下菌株DN1对其降解机制还不清楚。我们通过对葡萄糖和荧蒽处理条件下菌株DN1的转录组分析对比,发现有3 522个显著差异表达基因,其中1 919个基因上调表达,1 603个基因下调表达,说明荧蒽胁迫条件下菌株DN1体内存在着较完整的适应系统,会启动一系列基因表达变化来适应这一过程,差异表达上调基因主要表现在参与底物识别、结合转运、酶催化活性及生物降解代谢过程等功能类别中。于红艳等[12]比较了克雷白氏杆菌在正常培养和多环芳烃胁迫培养条件下2个样品的转录组数据,得到了254个差异表达基因,其中在多环芳烃作碳源时,有73个基因表达上调,181个基因表达下调。与芳烃胁迫条件下克雷白氏杆菌差异基因表达情况相比,菌株DN1在荧蒽胁迫条件下得到的差异基因个数较多,为了获得每个差异表达转录本潜在的代谢途径,进一步将这些差异表达上调基因进行KEGG注释分析[24],得到了112条KEGG代谢途径,分析发现这些代谢途径中的基因主要集中在ABC转运系统、氨基酸生物合成、双组分系统及碳代谢,大多数参与了底物的识别转运、信号转导及基因表达调控。112条KEGG代谢途径中有1 408个基因注释到“代谢途径”中,约占总差异基因的73.4%,其中有317个基因参与了碳氢化合物代谢及含有苯环结构异源生物质的生物降解。此外,在这些差异表达基因中存在大量可能与PAHs降解有关的基因,例如80个基因参与了碳代谢途径(ko01200),20个基因参与了多环芳烃化合物降解(ko00624),19个基因参与了苯甲酸降解(ko00362),5个基因参与了萘降解(ko00626)等,暗示了菌株DN1降解荧蒽可能与这些途径有密切关系。之前的研究[15]表明,菌株在降解荧蒽的过程中,主要是通过在荧蒽的C-1, 2、C-2, 3或者C-7, 8位置进行加氧反应促使苯环裂解。本研究中,菌株DN1在荧蒽降解过程中重要的双加氧酶基因及代谢途径如龙胆酸、苯甲酸、儿茶酚及原儿茶酸降解等中的基因(簇)表达量普遍显著上调,揭示了这些基因可能都参与了荧蒽的降解过程,降解酶系统的酶蛋白编码基因可能是通过相互作用来完成这个复杂的降解过程[15, 25]。此外,在转录组的差异表达基因中还有一部分是基因功能未知的假定蛋白,这些基因可能参与了菌株DN1特定的荧蒽降解机制,具体功能有待进一步探索。
| [1] |
Shen GF, Tao S, Chen YC, et al. Emission characteristics for polycyclic aromatic hydrocarbons from solid fuels burned in domestic stoves in rural China[J]. Environmental Science & Technology, 2013, 47(24): 14485-14494. |
| [2] |
Gupta S, Pathak B, Fulekar MH. Molecular approaches for biodegradation of polycyclic aromatic hydrocarbon compounds: a review[J]. Reviews in Environmental Science and Bio/Technology, 2015, 14(2): 241-269. DOI:10.1007/s11157-014-9353-3 |
| [3] |
Baird WM, Hooven LA, Mahadevan B. Carcinogenic polycyclic aromatic hydrocarbon-DNA adducts and mechanism of action[J]. Environmental and Molecular Mutagenesis, 2005, 45(2/3): 106-114. |
| [4] |
Ghosal D, Ghosh S, Dutta TK, et al. Current state of knowledge in microbial degradation of polycyclic aromatic hydrocarbons (PAHs): a review[J]. Frontiers in Microbiology, 2016, 7: 1369. |
| [5] |
Haritash AK, Kaushik CP. Biodegradation aspects of polycyclic aromatic hydrocarbons (PAHs): a review[J]. Journal of Hazardous Materials, 2009, 169(1/3): 1-15. |
| [6] |
Kim YH, Engesser KH, Cerniglia CE. Two polycyclic aromatic hydrocarbon o-quinone reductases from a pyrene-degrading Mycobacterium[J]. Archives of Biochemistry and Biophysics, 2003, 416(2): 209-217. DOI:10.1016/S0003-9861(03)00297-2 |
| [7] |
Song XH, Xu Y, Li GM, et al. Isolation, characterization of Rhodococcus sp. P14 capable of degrading high-molecular-weight polycyclic aromatic hydrocarbons and aliphatic hydrocarbons[J]. Marine Pollution Bulletin, 2011, 62(10): 2122-2128. DOI:10.1016/j.marpolbul.2011.07.013 |
| [8] |
Throne-Holst M, Wentzel A, Ellingsen TE, et al. Identification of novel genes involved in long-chain n-alkane degradation by Acinetobacter sp. strain DSM 17874[J]. Applied and Environmental Microbiology, 2007, 73(10): 3327-3332. DOI:10.1128/AEM.00064-07 |
| [9] |
Gai ZH, Zhang ZZ, Wang XY, et al. Genome sequence of Pseudomonas aeruginosa DQ8, an efficient degrader of n-alkanes and polycyclic aromatic hydrocarbons[J]. Journal of Bacteriology, 2012, 194(22): 6304-6305. DOI:10.1128/JB.01499-12 |
| [10] |
Patel PA, Kothari VV, Kothari CR, et al. Draft genome sequence of petroleum hydrocarbon-degrading Pseudomonas aeruginosa strain PK6, isolated from the Saurashtra region of Gujarat, India[J]. Genome Announcements, 2014, 2(1): e00002-14. DOI:10.1128/genomeA.00002-14 |
| [11] |
GonÇalves ER, Hara H, Miyazawa D, et al. Transcriptomic assessment of isozymes in the biphenyl pathway of Rhodococcus sp. strain RHA1[J]. Applied and Environmental Microbiology, 2006, 72(9): 6183-6193. DOI:10.1128/AEM.00947-06 |
| [12] |
Yu HY, Zhang XX, Chen HY, et al. Comparative transcriptomic analysis on Klebsiella pneumoniae Tzyx1 in the degradation of polycyclic aromatic hydrocarbons (PAHs)[J]. Acta Scientiae Circumstantiae, 2018, 38(2): 689-695. (in Chinese) 于红艳, 张昕欣, 陈红云, 等. 利用比较转录组分析克雷白氏肺炎杆菌(Klebsiella pneumoniae Tzyx1)降解多环芳烃特性[J]. 环境科学学报, 2018, 38(2): 689-695. |
| [13] |
Lu W, Luo N, Dong W, et al. Identification and characterization of a Pseudomonas aeruginosa strain DN1 in fluoranthene biodegradation[J]. Acta Scientiae Circumstantiae, 2015, 35(11): 3486-3492. (in Chinese) 路薇, 罗娜, 董文, 等. 一株降解荧蒽的铜绿假单胞菌的筛选鉴定及其特性[J]. 环境科学学报, 2015, 35(11): 3486-3492. |
| [14] |
Dong W, He CQ, Li YP, et al. Complete genome sequence of a versatile hydrocarbon degrader, Pseudomonas aeruginosa DN1 isolated from petroleum-contaminated soil[J]. Gene Reports, 2017, 7: 123-126. DOI:10.1016/j.genrep.2017.04.001 |
| [15] |
He CQ, Li YP, Huang C, et al. Genome sequence and metabolic analysis of a fluoranthene-degrading strain Pseudomonas aeruginosa DN1[J]. Frontiers in Microbiology, 2018, 9: 580-589. DOI:10.3389/fmicb.2018.00580 |
| [16] |
Kim D, Pertea G, Trapnell C, et al. TopHat2: accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions[J]. Genome Biology, 2013, 14: R36. DOI:10.1186/gb-2013-14-4-r36 |
| [17] |
Trapnell C, Roberts A, Goff L, et al. Differential gene and transcript expression analysis of RNA-Seq experiments with TopHat and Cufflinks[J]. Nature Protocols, 2012, 7(3): 562-578. DOI:10.1038/nprot.2012.016 |
| [18] |
Kanehisa M, Araki M, Goto S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Research, 2008, 36(S1): D480-D484. |
| [19] |
Young MD, Wakefield MJ, Smyth GK, et al. Gene ontology analysis for RNA-Seq: accounting for selection bias[J]. Genome Biology, 2010, 11: R14. DOI:10.1186/gb-2010-11-2-r14 |
| [20] |
Trapnell C, Pachter L, Salzberg SL. TopHat: discovering splice junctions with RNA-Seq[J]. Bioinformatics, 2009, 25(9): 1105-1111. DOI:10.1093/bioinformatics/btp120 |
| [21] |
Anders S, Huber W. Differential expression analysis for sequence count data[J]. Genome Biology, 2010, 11(10): R106. DOI:10.1186/gb-2010-11-10-r106 |
| [22] |
Shukor MY, Hassan NAA, Jusoh AZ, et al. Isolation and characterization of a Pseudomonas diesel-degrading strain from Antarctica[J]. Journal of Environmental Biology, 2009, 30(1): 1-6. |
| [23] |
Hua F, Wang H. Uptake modes of octadecane by Pseudomonas sp. DG17 and synthesis of biosurfactant[J]. Journal of Applied Microbiology, 2015, 112(1): 25-37. |
| [24] |
Shang CH, Bi GC, Yuan ZH, et al. Discovery of genes for production of biofuels through transcriptome sequencing of Dunaliella parva[J]. Algal Research, 2016, 13: 318-326. DOI:10.1016/j.algal.2015.12.012 |
| [25] |
Kweon O, Kim SJ, Jones RC, et al. A polyomic approach to elucidate the fluoranthene-degradative pathway in Mycobacterium vanbaalenii PYR-1[J]. Journal of Bacteriology, 2007, 189(13): 4635-4647. DOI:10.1128/JB.00128-07 |
 2020, Vol. 47
2020, Vol. 47