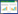扩展功能
文章信息
- 李锋, 梁念, 孙建中
- LI Feng, LIANG Nian, SUN Jian-Zhong
- 一株新型苯胺蓝降解菌MP-13的代谢特征
- Degradation characteristics of a novel aniline blue-discoloring bacterial strain MP-13
- 微生物学通报, 2020, 47(1): 43-53
- Microbiology China, 2020, 47(1): 43-53
- DOI: 10.13344/j.microbiol.china.190414
-
文章历史
- 收稿日期: 2019-05-11
- 接受日期: 2019-06-13
- 网络首发日期: 2019-07-13
三苯甲烷类染料是继偶氮类和蒽醌类染料之后使用量排位第三的染料,其中包括孔雀石绿、结晶紫、灿烂绿和苯胺蓝等,它们被广泛应用于纺织印染、造纸、医药和生物染色等[1]。许多研究结果表明此类染料在生产和使用过程中产生的有色废水不仅对环境造成污染,而且能够在环境中积累和富集后通过食物链进入到生物体内,从而对人和动物的生命健康构成巨大、潜在的威胁[2-3]。因此,如何利用环境友好型技术处理人工合成的有机染料废水,已成为国内外研究者的关注热点之一。
染料废水的脱色降解方法主要有物理法、化学法和生物法,其中生物法在处理过程中不会产生二次污染,微生物能够将染料物质矿化成低毒或者无毒的小分子化合物,因此,生物法被认为是一种环境友好和高效低成本的方法[4]。目前,三苯甲烷类染料降解的研究主要集中于孔雀石绿和结晶紫,而关于微生物降解苯胺蓝的报道较少。苯胺蓝作为一种典型的三苯甲烷类染料,不仅具有稳定、复杂的化学结构,而且还是此类染料中的高分子量化合物(分子结构见图 1),在自然条件下极难被降解。三苯甲烷类染料分子的显色基团是碳氮键(-C=N-)[5],微生物可以通过分泌特定的酶催化碳氮键断裂,从而使其脱色降解为低毒或者无毒小分子化合物。相关研究表明与三苯甲烷类染料降解相关的酶有:漆酶、锰过氧化物酶、木质素过氧化物酶、细胞色素P450单加氧酶、三苯基甲烷类染料还原酶TMR和染料脱色酶TpmD等[6]。真菌降解三苯甲烷类染料存在一定的劣势,如生长速度慢、环境适应能力差和不易于基因操作等。因此,生长速度快、环境适应性更强的细菌引起研究者的极大关注。
|
| 图 1 苯胺蓝化学结构 Figure 1 Structure of aniline blue |
|
|
迄今为止,已报道有关苯胺蓝脱色降解的细菌仅有Streptomyces AG-56、Shewanella oneidensis MR-1、Pseudomonas aeruginosa WZR-B、Acinetobacter sp. EL1、Providencia sp. EL2和Weeksella virosa EL3等[7-10]。尽管上述细菌降解苯胺蓝表现出一定效果,但细菌降解苯胺蓝可能的降解途径及分子机制尚不明确,因此发掘高效、广谱的染料脱色菌种,并进一步深入研究可能的降解途径以及参与其中的酶系等具有重要意义。
在以往的报道中利用苯胺蓝筛选木质素降解菌[11],本课题组在白蚁肠道共生菌降解木质素的筛选过程中发现多种细菌对苯胺蓝具有高效脱色功能。在此基础上,利用多种染料对细菌脱色能力进行检测,筛选出具有广谱脱色能力的细菌后,对其进行分子生物学鉴定、脱色条件和降解产物分析,并推测苯胺蓝的降解途径,进一步丰富三苯甲烷类染料废水处理中所需菌种资源并完善苯胺蓝降解分子机理研究,以期为提高该菌对三苯甲烷类染料的降解效果及实现工业化应用奠定研究基础。
1 材料与方法 1.1 材料 1.1.1 样品来源2016年7月于云南省西双版纳州中国科学院西双版纳热带植物园次生雨林中采集木食性白蚁,在被蚁蚀的腐木段中收集白蚁后,将其放于事先底部有湿润滤纸的培养皿中带回实验室,放置于避光的室温环境中培养待用。根据兵蚁形态特征及COⅡ基因序列鉴定为巴基斯坦小白蚁(Microtermes pakistanicus)。
1.1.2 主要试剂及仪器苯胺蓝、亚甲基蓝、刚果红、苯酚红、孔雀石绿和天青B,国药集团化学试剂有限公司;其余化学试剂均为国产分析纯;细菌通用PCR引物、Ezup柱式细菌基因组DNA提取试剂盒和Taq DNA聚合酶,生工生物工程(上海)股份有限公司。电泳仪,北京六一生物科技有限公司;PCR仪,伯乐公司;紫外可见分光光度计,贝克曼库尔特有限公司;傅立叶变换红外光谱仪,赛默飞世尔科技公司;气相色谱质谱联用仪(GC/MS),安捷伦科技有限公司。
1.1.3 培养基LB培养基(g/L):胰蛋白胨10.0,酵母粉5.0,NaCl 10.0,pH 7.2,1×105 Pa灭菌20 min。
分离培养基(g/L):胰蛋白胨5.0,酵母粉2.5,NaCl 5.0,琼脂15.0,pH 7.2,1×105 Pa灭菌20 min,待培养基温度降到60 ℃以下,加入100 mg经过0.22 μm滤膜过滤灭菌的苯胺蓝。
复筛培养基(g/L):胰蛋白胨5.0,酵母粉2.5,NaCl 5.0,琼脂15.0,pH 7.2,1×105 Pa灭菌20 min后,待培养基温度降到60 ℃以下,分别加入一定量经0.22 μm滤膜过滤灭菌的染料(亚甲基蓝、刚果红、苯酚红或天青B)。
液体脱色培养基(g/L):胰蛋白胨5.0,酵母粉2.5,NaCl 5.0,CaCl2 0.1,pH 7.2,1×105 Pa灭菌20 min后,加入5 mL经过0.22 μm滤膜过滤灭菌的微量元素溶液,再加入一定量过滤灭菌的染料(苯胺蓝、亚甲基蓝、天青B、刚果红或孔雀石绿)。微量元素溶液(g/L):CuSO4·5H2O 0.1,ZnSO4·7H2O 0.12,MgSO4·7H2O 0.2,FeSO4·7H2O 0.16,MnSO4·H2O 0.08。
1.2 方法 1.2.1 样品处理及菌株的分离纯化选取巴基斯坦小白蚁的工蚁在无菌水中清洗2-3次后,用75%酒精对白蚁表面消毒,然后在中性PBS缓冲液中漂洗。在无菌条件下,将解剖的白蚁肠道置于无菌的0.9%生理盐水中,然后用研磨棒将肠道研磨成匀浆状态。肠道匀浆液用无菌生理盐水经过一定梯度稀释后,取稀释103、104、105、106倍的溶液各100 μL涂布于固体分离培养基上,将培养皿放置于30 ℃的培养箱中培养2-4 d。将具有明显脱色圈的单克隆转接至LB培养基中培养后,再次用固体分离培养基进行划线分离、纯化细菌(重复2-3次划线),直至得到单克隆。纯化后的单克隆进行培养后,划线于复筛培养基平板上,检测单克隆对其他类型染料的脱色能力;挑取单菌落菌株继续分离纯化,获得纯菌株后,单独接种于不同类型染料(苯胺蓝、天青B、亚甲基蓝、刚果红和苯酚红)固体培养基、液体苯胺蓝和孔雀石绿验证细菌是否具有广谱染料脱色能力。最终筛选到一株脱色圈较大且可使多种染料脱色的细菌,然后对其进行分子生物学鉴定。
1.2.2 细菌DNA提取及鉴定将单克隆接种于LB培养基中,30 ℃、180 r/min培养过夜,取1 mL培养液置于离心管中,经12 000 r/min离心2 min后,弃上清液,收集菌体,经无菌水漂洗后的菌体,用细菌基因组试剂盒提取DNA。细菌16S rRNA基因扩增通用引物为27F (5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R (5′-GGTTACCTTGTTACGACTT-3′),PCR反应体系及条件参考文献[12]。PCR产物经1%琼脂糖凝胶电泳检测后,委托生工生物工程(上海)股份有限公司对细菌序列进行双向测序。将测序完成的16S rRNA基因序列拼接后,提交至NCBI网站的GenBank数据库,并进行BLASTn (www.ncbi.nlm.nih.gov/BLAST)相似性比对。下载BLAST比对序列相似性较高的菌株序列,利用MEGA 5.05软件计算序列相似度,同时用邻接法(neighbor-joining method)构建菌株系统发育树,确定物种的系统发育地位。
1.2.3 不同培养条件对染料脱色率的影响将菌株MP-13接种于LB培养基中,在30 ℃、180 r/min条件下培养12 h,收集一定体积培养液在12 000 r/min离心2 min,弃上清液,用无菌液体脱色培养基重悬后作为种子液。以5%接种量研究在不同温度(20、25、30、35和40 ℃)、pH (5.0、6.0、7.0、8.0和9.0)、转速(100、130、150、180、和220 r/min)条件下培养36 h后,细菌对浓度为200 mg/L苯胺蓝溶液脱色效果的影响。同时,考察菌株MP-13在接种量为5%、30 ℃和180 r/min条件下培养36 h后,不同初始浓度(300、500、800、1 000和1 500 mg/L)对苯胺蓝溶液脱色效果的影响。
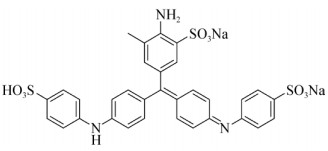
1.2.4 染料脱色率测定液体脱色培养基接菌培养一段时间后,无菌条件下吸取2 mL培养液,经12 000 r/min离心2 min,取上清液,采用分光光度法在600 nm (苯胺蓝可见光的最大吸收峰)处测定吸光度,根据公式(1)计算脱色率:
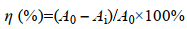
|
(1) |
式中A0表示液体脱色培养基不接细菌初始吸光度,Ai表示脱色培养基接种细菌经过一段时间脱色后的溶液吸光度。
1.2.5 紫外/可见吸收光谱分析取浓度为100 mg/L苯胺蓝溶液和降解24 h离心后的上清液,用紫外可见分光光度计在波长190-900 nm范围内进行扫描。
1.2.6 FTIR分析苯胺蓝降解前后的FTIR分析方法参考文献[13],具体步骤为:以浓度为300 mg/L苯胺蓝脱色培养基为考察对象,分别取未经细菌处理和经细菌降解36 h后的溶液各100 mL,然后经过0.45 μm孔径滤头过滤,过滤液经冷冻干燥成粉末,与KBr按照一定比例(质量比1:100)混匀研磨后,压片,进行红外光谱扫描,波数4 000-400 cm-1。
1.2.7 GC/MS分析苯胺蓝降解产物提取步骤及分析方法参考文献[13],具体步骤为:取浓度为300 mg/L苯胺蓝脱色培养基和经过36 h细菌脱色降解的培养基上清液,用等体积乙酸乙酯萃取后,取上层有机相备用,下层水相溶液经上述操作重复2次后,合并3次萃取液,加入无水亚硫酸钠干燥后,经旋转蒸发有机相至结晶,然后用色谱纯甲醇溶解结晶物,再经0.22 μm滤膜过滤后,利用GC/MS测定苯胺蓝降解后中间产物,色谱柱为HP-5MS (30 m×0.25 mm×0.25 μm),载气为He,气体流速为1 mL/min,进样体积为1 μL;升温程序:初始温度50 ℃,保持5 min,以10 ℃/min速度升温至280 ℃,保持10 min;进样口温度280 ℃,离子源温度250 ℃,在电子轰击(EI)模式70 eV条件下,质谱扫描范围30-800 (m/z)。
2 结果与分析 2.1 苯胺蓝降解细菌的筛选与鉴定 2.1.1 苯胺蓝脱色菌筛选采用平板划线法从分离固体培养基上发现多株具有明显脱色圈的细菌,其中一株编号为MP-13的细菌可以使多种类型染料脱色,结果如图 2所示。从图 2中可知,菌株MP-13在不同染料固体培养基中培养24-48 h后可以产生明显脱色圈,说明此菌株可以分泌胞外酶使除苯胺蓝以外的染料脱色,初步推断菌株MP-13具有广谱染料脱色性能。从液体染料脱色情况来看,经菌株MP-13降解后的苯胺蓝和孔雀石绿的颜色基本消失,而对照组染料颜色基本没有变化,说明此菌对苯胺蓝和孔雀石绿具有较强的脱色能力。此外,从离心后的菌体沉淀来看,苯胺蓝溶液底部菌体呈乳白色,说明此菌对苯胺蓝的脱色主要依靠胞外酶;而孔雀石绿溶液底部呈淡绿色,说明此菌对孔雀石绿的脱色能力除靠分泌胞外酶外,还有自身菌体对染料的吸附作用。
|
| 图 2 菌株MP-13在苯胺蓝(A)、天青B (B)、亚甲基蓝(C)、刚果红(D)、苯酚红(E)、液体苯胺蓝(F)和孔雀石绿(G)培养基中生长时产生脱色 Figure 2 The decolorization of aniline blue (A, F), azure B (B), methylene blue (C), congo red (D), phenol red (E) and malachite green (G) by strain MP-13 cultured on the medium |
|
|
基于16S rRNA基因序列分析的结果,确定菌株MP-13的分类地位。将双向测序获得菌株MP-13的16S rRNA基因序列,经DNAMAN序列拼接后(1 445 bp)提交至GenBank数据库,序列登录号为MF455199,同时在NCBI数据库中进行序列相似性比对,选取BLAST中相似度较高的7株细菌序列,并下载相关序列后,利用MEGA 5.05计算细菌序列相似度,同时利用其中的邻接法(neighbor- joining method)构建细菌系统发育树(图 3)。如图 3所示,菌株MP-13与Trabulsiella odontotermitis strain zjsru-4在同一个分支上,序列相似性为99.03%,因此将该菌株命名为Trabulsiella odontotermitis MP-13。

|
| 图 3 菌株MP-13基于16S rRNA基因序列的系统发育树 Figure 3 Phylogenetics tree based on 16S rRNA gene sequence of strain MP-13 注:括号中序号:菌株登录号;分支点上的数字:Bootstrap支持率;标尺刻度0.001:序列差异的分支长度. Note: Numbers in parentheses: the sequence accession number; Numbers in each branch points: the percentages supported by bootstrap; Bar=0.001: nucleotide divergence. |
|
|
紫外/可见吸收光谱分析表明细菌降解后苯胺蓝的显色基团(-C=N-)被破坏,导致苯胺蓝溶液颜色发生变化。由图 4中可知,苯胺蓝在196、312和600 nm处具有明显吸收峰,这与文献[7]报道的基本一致,600 nm处吸收峰是苯胺蓝的显色基团。然而,经过24 h细菌降解后的苯胺蓝溶液,在600 nm处的吸收峰完全消失,并且312 nm处吸收峰强度降低,而196 nm处吸光度显著增强。已有研究表明,如果染料脱色依靠菌体吸附脱色,那么3个吸收峰应该成比例降低[14],由此可以证明菌株MP-13主要通过分泌胞外酶破坏显色官能团(-C=N-)和其他类型化学键,从而导致苯胺蓝脱色降解。

|
| 图 4 脱色前后苯胺蓝的紫外/可见吸收光谱 Figure 4 UV/vis absorption spectra of aniline blue before and after decolourization |
|
|
细菌对苯胺蓝的脱色效果受多种生长条件影响,如培养温度、初始pH、转速和底物浓度等。培养基初始pH对细菌生长及代谢能力起着至关重要的作用,因此考察了培养基初始pH对菌株MP-13脱色苯胺蓝的影响。由图 5A可知,该菌的环境适应能力较强,即在广泛的pH 5.0-9.0环境下可以使苯胺蓝脱色。当pH为7.0或8.0时,苯胺蓝脱色率分别达到90.3%和96%;当pH降低或者增加时,苯胺蓝脱色率明显降低,其中pH为5.0时脱色率降到41.7%,而pH为9.0时脱色率可以达到83.6%。脱色率降低可能与细菌分泌的酶活力有关,有研究表明三苯甲烷类还原酶在pH 9.0时具有较高酶活力[15]。过碱或过酸的环境都会使细菌脱色效率降低,该菌在偏碱环境下(pH 8.0)脱色率最高,因此在处理偏碱染料废水时具有广泛的应用前景。
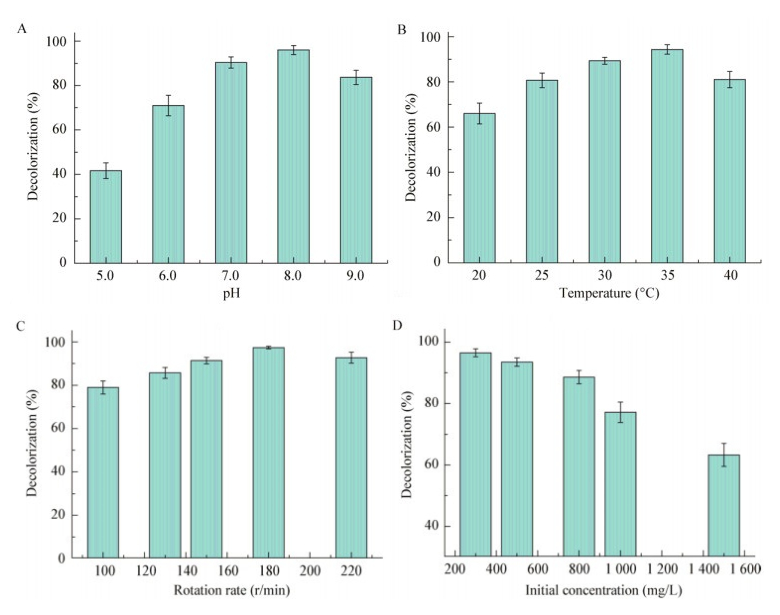
|
| 图 5 不同因素对苯胺蓝脱色率的影响 Figure 5 Effect of different environment parameters on decolorization of aniline blue dye 注:A:pH;B:温度;C:转速;D:初始浓度. Note: A: pH; B: Temperature; C: Rotation rate; D: Initial concentration. |
|
|
培养温度影响细菌生长及其所分泌胞外酶活力,从而导致苯胺蓝的脱色效率有较大差异。由图 5B可知,在温度为20-40 ℃的范围内,苯胺蓝脱色率都大于60%;当温度为35 ℃时,菌株MP-13对苯胺蓝的脱色效率达到最佳(94.3%);当温度为20 ℃时,苯胺蓝脱色效率最低(66%);当温度达到40 ℃时,脱色率也可达到81%。由此表明,温度对细菌胞内外酶活性和酶促反应影响较大,进而影响苯胺蓝脱色效率[16]。
溶氧量是影响降解有机物速率的主要因素之一。由图 5C可知,随着摇床转速的增加,脱色率也随着增加。相关研究表明三苯甲烷类染料脱色降解过中,可能要依赖NADH/NADPH加氧酶,适当增加废水中溶氧量可能促进染料脱色降解效率[17]。当摇床转速为180 r/min时,菌株MP-13对苯胺蓝的脱色率达到最大值97.3%;而当转速增加到220 r/min时,苯胺蓝脱色率降低到92.6%,这可能是由于转速达到一定值时,培养基中的溶氧基本饱和,继续增大转速反而可能造成菌体损伤,从而造成脱色率降低。因此菌株MP-13脱色降解苯胺蓝的最佳转速为180 r/min。
苯胺蓝初始浓度是影响菌株MP-13脱色效果的重要因素。由图 5D可知,随着苯胺蓝浓度的增加(浓度从300 mg/L上升到1 500 mg/L),脱色率呈现降低趋势。在中高浓度300 mg/L时,脱色率可达最高96.5%;继续增加苯胺蓝浓度到500、800和1 000 mg/L时,脱色率分别为93.5%、88.6%和77.2%;而当浓度达到1 500 mg/L时,脱色率明显降低,但仍达到63.2%。由此可以说明苯胺蓝浓度增加后,溶液中毒性随着增大,抑制菌体生长,从而导致脱色率降低。
2.4 FTIR分析红外光谱图可以直观反映苯胺蓝降解前后的化学官能团变化情况,从而推断苯胺蓝分子结构中可能断裂的化学键类型。由图 6可知,降解前苯胺蓝中存在三苯甲烷类染料的3个特征吸收峰(1 574、1 367和1 169 cm–1),并且苯胺蓝降解前后FTIR图谱在指纹区(1 700-500 cm-1)的吸收峰强度及吸收峰数量发生明显变化,比如:降解后光谱图中消失了1 496、1 367、913和731 cm–1等处的吸收峰,并有新吸收峰出现在1 660、1 449和1 401 cm–1处。经检索得知,在1 574 cm–1处的吸收峰归因于苯环上的C-H伸缩;在1 496 cm–1处的吸收峰归因于染料结构存在自由N-H基团,降解产物中消失表明苯胺蓝支链断裂;在1 367 cm–1处的吸收峰归因于C-H伸缩振动;在1 169 cm–1处的吸收峰是由芳香化合物C-N伸缩振动引起;在1 122 cm-1处的吸收峰归因于C-OH伸缩弯曲;在1 035 cm-1处的吸收峰是C-O伸缩引起的;在900 cm-1以下吸收峰都归属于芳香族化合物或苯环,其中在降解产物中731、652和593 cm-1处的吸收峰消失,同时在619 cm-1和519 cm-1处出现新吸收峰,表明芳香化合物结构因降解发生化学键断裂。降解产物中新出现的1 660 cm-1吸收峰归属于芳基酮的C=O伸缩振动,表明芳香化合物在降解过程中被羰基化,这一结果与GC/MS分析降解产物中发现芳香酮类化合物一致。在1 449 cm-1和1 400 cm-1处的吸收峰,分别归因于芳香基团和C=C伸缩引起的,表明降解产物中还有苯环类结构存在。综上所述,可以推断降解产物中存在C=O、-OH、-NH2基团及苯环结构,并由GC/MS进行了验证,本研究结果也与以往微生物降解三苯甲烷类染料的报道[18-22]基本一致。

|
| 图 6 苯胺蓝脱色前后的FTIR图谱 Figure 6 FTIR spectra of aniline blue before and after decolorization |
|
|
通过GC/MS定性分析降解过程的中间代谢产物,初步明确了菌株MP-13对苯胺蓝的主要代谢特征与途径(结果见表 1和图 7)。经细菌处理12 h的苯胺蓝溶液可以鉴定到5个新物质,保留时间分别为7.265、20.265、21.805、23.090和24.460 min,经过与NIST library中标准物质图谱比对,证实5种新物质分别为:2-苯乙醇、2-羟基-3, 5, 5-三甲基-2-环己烯-1-酮、2, 2′-亚甲基双(6-叔丁基对甲酚)、4, 4′-二氨基二苯甲酮和2, 4, 6-三羟基苯甲醛。延长细菌降解时间到36 h时,共检测到10种物质,除上述5种外,其他5种物质分别为异戊酸(3.310 min)、乙苯(3.725 min)、间二甲苯(3.815 min)、2′-氨基苯乙酮(10.025 min)和2-(4-羟苯基)乙醇(11.580 min)。其中4, 4′-二氨基二苯甲酮也在其他已报道三苯甲烷类染料(孔雀石绿、结晶紫和棉蓝)[13, 21-22]中发现,由此可以证明4, 4′-二氨基二苯甲酮是三苯甲烷类染料的主要降解中间产物,对苯胺蓝进一步降解起主要作用。
| 序号 No. |
保留时间 Retention time |
化学名称 Chemical name |
分子式 Molecular formula |
分子量 Molecular weight |
12 h | 36 h |
| 1 | 3.310 | 3-methyl-butanoic acid | C5H10O2 | 102 | - | + |
| 2 | 3.725 | Ethylbenzene | C8H10 | 106 | - | + |
| 3 | 3.815 | 1, 3-dimethyl-benzene | C8H10 | 106 | - | + |
| 4 | 7.265 | Phenylethyl alcohol | C8H10O | 122 | + | + |
| 5 | 10.025 | 1-(2-aminophenyl)-Ethanone | C8H9NO | 135 | - | + |
| 6 | 11.580 | 4-hydroxy-benzeneethanol | C8H10O2 | 138 | - | + |
| 7 | 20.265 | 2-hydroxy-3, 5, 5-trimethyl-cyclohex-2-enone | C9H14O2 | 154 | + | + |
| 8 | 21.805 | 2, 2′-methylenebis [6-(1, 1-dimethylethyl)-4-methyl-phenol | C23H32O2 | 340 | + | + |
| 9 | 23.090 | 4, 4′-diaminobenzophenone | C13H12N2O | 212 | + | + |
| 10 | 24.460 | 2, 4, 6-trihydroxybenzaldehyde | C7H6O4 | 154 | + | + |
| 注:+:检测到;–:未检测到. Note: +: Detected; –: Not detected. | ||||||

|
| 图 7 基于GC/MS结果推断菌株MP13对苯胺蓝的降解途径 Figure 7 The proposed biodegradation pathways of aniline blue by strain MP-13 based on the results of GC/MS |
|
|
根据鉴定到的代谢产物并结合已报道的三苯甲烷类染料文献[13, 21-22],推测菌株MP-13降解苯胺蓝可能的降解途径(图 7)。首先,苯胺蓝在特定酶的作用下,使显色基团碳氮键断裂,并伴随侧链断裂生成三苯甲烷为骨架的无色物质和芳香族化合物;随后,三苯甲烷类似物的中心碳原子发生羟基化反应并伴随碳碳键断裂[14],被羟基化的物质进一步发生羰基化反应生成4, 4′-二氨基二苯甲酮,而由碳碳键断裂生成物质经过去甲基反应生成2′-氨基苯乙酮;最终这些物质经过一系列酶催化,开环最终完全被矿化成二氧化碳和水。综上所述,可知菌株MP-13降解苯胺蓝代谢途径与已报道的细菌降解孔雀石绿、结晶紫和棉蓝存在相似之处。
3 讨论与结论Trabulsiella是一类广泛存在于自然环境中的革兰氏阴性细菌,具备降解多种类型有机污染物的能力。本研究从高等白蚁肠道筛选分离得到一株对苯胺蓝具有高效脱色降解能力的菌株MP-13,经过16S rRNA基因序列和系统发育分析,初步鉴定为土白蚁特拉布尔希氏菌(Trabulsiella odontotermitis)。土白蚁特拉布尔希氏菌是由Chou等首次从白蚁肠道中分离得到[23],目前关于此菌的报道也都是分离于白蚁肠道,并且该菌对芳香族化合物的降解性能还不清楚,仅在降解黄曲霉毒素B1领域有所报道[24]。迄今为止,尚未见到该菌在降解染料类有机化合物方面的报道,然而本研究发现菌株MP-13具有多种类型染料的脱色能力,并且根据报道该种菌的基因组中含有可降解双苯酚和二噁英等难降解物质的相关酶基因序列[24]。因此,进一步探索该菌对苯胺蓝降解特征及代谢途径,为将来该种菌株应用到染料废水治理中奠定基础。
菌株MP-13对三苯甲烷类的有机污染物具有广谱性、较高浓度耐受力和降解能力,表现出了潜在的产业化应用价值。与其他细菌相比,该菌可在较宽的pH (5.0-9.0)和温度(20-40 ℃)范围内,表现出对苯胺蓝较好的脱色降解能力;其中在温度为35 ℃、pH 8.0和转速180 r/min的培养条件下,该菌对200 mg/L苯胺蓝的最大脱色率可达97.3%。此外,浓度为300-1 500 mg/L的苯胺蓝溶液对菌株MP-13的生长及脱色能力没有产生较大的影响,即使染料浓度达到1 500 mg/L时,该菌对苯胺蓝脱色率也可达到63%以上,如果延长培养时间脱色率会达到更高数值。在实际环境中,印染行业废水中染料可能是多种类型共存,而菌株MP-13对亚甲基蓝、天青B、刚果红、苯酚红和孔雀石绿也具有脱色潜力,表现出了一定广谱的底物利用能力。其次,染料废水的酸碱性直接影响细菌的代谢能力,从不同pH对菌株MP-13降解苯胺蓝的降解评价可知,偏碱性环境条件下,该菌对苯胺蓝的降解能力明显优于偏酸环境,这可能是由于苯胺蓝降解相关酶的适宜pH为碱性环境。
菌株MP-13降解苯胺蓝的中间产物分析表明,中心碳原子羟基化是三苯甲烷类染料降解的关键步骤。通过GC/MS分析苯胺蓝降解中间产物,发现了主要降解产物4, 4′-二氨基二苯甲酮和2′-氨基苯乙酮等芳香族化合物,由此可推断苯胺蓝降解是经过一系列氧化还原反应,如:侧链断裂、羟基化反应、羰基化反应、去甲基化反应和芳香族化合物开环等。以上实验结果与Aspergillus flavus降解孔雀石绿[25]和Rhodococcus qingshengii JB301[1]降解结晶紫的代谢途径具有相似性,但因苯胺蓝的特殊化学结构又存在一定差异,如存在侧链C-N键断裂。
综上所述,Trabulsiella odontotermitis MP-13可高效降解苯胺蓝,且具有潜在的多种染料脱色降解能力,但该菌对三苯甲烷类染料降解转化过程中的关键酶、降解产物有无毒性和原位降解效果有待进一步深入研究和探讨。
| [1] |
Li GH, Peng L, Ding ZY, et al. Decolorization and biodegradation of triphenylmethane dyes by a novel Rhodococcus qingshengii JB301 isolated from sawdust[J]. Annals of Microbiology, 2014, 64(4): 1575-1586. DOI:10.1007/s13213-014-0801-7 |
| [2] |
Chen SH, Ting ASY. Biodecolorization and biodegradation potential of recalcitrant triphenylmethane dyes by Coriolopsis sp. isolated from compost[J]. Journal of Environmental Management, 2015, 150: 274-280. DOI:10.1016/j.jenvman.2014.09.014 |
| [3] |
Khataee AR, Dehghan G, Zarei M, et al. Neural network modeling of biotreatment of triphenylmethane dye solution by a green macroalgae[J]. Chemical Engineering Research and Design, 2011, 89(2): 172-178. DOI:10.1016/j.cherd.2010.05.009 |
| [4] |
Yang XT, Zheng JZ, Lu YM, et al. Degradation and detoxification of the triphenylmethane dye malachite green catalyzed by crude manganese peroxidase from Irpex lacteus F17[J]. Environmental Science and Pollution Research, 2016, 23(10): 9585-9597. DOI:10.1007/s11356-016-6164-9 |
| [5] |
Zhang RJ, Li HX, Zhang JH, et al. On the technical condition and the mechanism of the aniline blue decolorization under the impact of the laccase/HOBT mediator system[J]. Journal of Safety and Environment, 2015, 15(6): 233-238. (in Chinese) 张瑞景, 李慧星, 张建华, 等. 漆酶/HOBT体系对苯胺蓝的脱色条件及机理研究[J]. 安全与环境学报, 2015, 15(6): 233-238. |
| [6] |
Song JL, Mu YC, Ruan ZY, et al. Research advances on metabolic pathways and mechanisms of biodegradation of triphenylmethane dyes[J]. Chinese Fishery Quality and Standards, 2017, 7(6): 10-16. (in Chinese) 宋金龙, 穆迎春, 阮志勇, 等. 微生物降解三苯甲烷类染料的研究进展[J]. 中国渔业质量与标准, 2017, 7(6): 10-16. DOI:10.3969/j.issn.2095-1833.2017.06.002 |
| [7] |
Ma YH, Li LJ, Lu MH, et al. Isolation of aniline blue-discoloring Actinomyces and its decolorization characteristics[J]. Acta Scientiae Circumstantiae, 2016, 36(12): 4361-4366. (in Chinese) 马英辉, 李利军, 卢美欢, 等. 苯胺蓝脱色放线菌的分离及脱除特性研究[J]. 环境科学学报, 2016, 36(12): 4361-4366. |
| [8] |
Wu YM, Xiao X, Xu CC, et al. Decolorization and detoxification of a sulfonated triphenylmethane dye aniline blue by Shewanella oneidensis MR-1 under anaerobic conditions[J]. Applied Microbiology and Biotechnology, 2013, 97(16): 7439-7446. DOI:10.1007/s00253-012-4476-3 |
| [9] |
Wu C, Wang H, Zheng TL, et al. Isolation and characterization of aniline blue-degrading bacteria[J]. Journal of Huaqiao University (Natural Science), 2008, 29(1): 38-41. (in Chinese) 吴楚, 王慧, 郑天凌, 等. 一株苯胺蓝降解菌的分离鉴定及其降解特性[J]. 华侨大学学报:自然科学版, 2008, 29(1): 38-41. |
| [10] |
Shao NN, Zhang C, Gu QY, et al. Research on decolorization of aniline blue by bacteria group EL[J]. Journal of Nanjing Agricultural University, 2014, 37(1): 133-137. (in Chinese) 邵娜娜, 张充, 顾青莹, 等. 菌群EL对苯胺蓝的脱色研究[J]. 南京农业大学学报, 2014, 37(1): 133-137. |
| [11] |
Yang CX, Wang T, Gao LN, et al. Isolation, identification and characterization of lignin-degrading bacteria from Qinling, China[J]. Journal of Applied Microbiology, 2017, 123(6): 1447-1460. DOI:10.1111/jam.13562 |
| [12] |
Sun XX, Li JJ, Ning N, et al. Isolation and identification of chitin-degrading bacteria from the hindgut of Macrotermes barneyi[J]. Microbiology China, 2017, 44(7): 1649-1654. (in Chinese) 孙新新, 李净净, 宁娜, 等. 黄翅大白蚁后肠几丁质降解微生物的分离与鉴定[J]. 微生物学通报, 2017, 44(7): 1649-1654. |
| [13] |
Bharagava RN, Mani S, Mulla SI, et al. Degradation and decolourization potential of an ligninolytic enzyme producing Aeromonas hydrophila for crystal violet dye and its phytotoxicity evaluation[J]. Ecotoxicology and Environmental Safety, 2018, 156: 166-175. DOI:10.1016/j.ecoenv.2018.03.012 |
| [14] |
Wu Y, Chen M, Liu J, et al. Biodecolorization and biodegradation of malachite green by Pleurotus eryngii and degradation products analysis[J]. Acta Scientiae Circumstantiae, 2016, 36(8): 2844-2851. (in Chinese) 吴茵, 陈敏, 刘洁, 等. 刺芹侧耳对孔雀石绿的脱色降解及其产物分析[J]. 环境科学学报, 2016, 36(8): 2844-2851. |
| [15] |
Jang MS, Lee YM, Kim CH, et al. Triphenylmethane reductase from Citrobacter sp. strain KCTC 18061P: purification, characterization, gene cloning, and overexpression of a functional protein in Escherichia coli[J]. Applied and Environmental Microbiology, 2005, 71(12): 7955-7960. DOI:10.1128/AEM.71.12.7955-7960.2005 |
| [16] |
Zhao NN, Xu JF, Song XX, et al. Screening and phenol-degrading characteristics of a highly efficient phenol-degrading halophilic bacterial strain Halomonas sp. H17[J]. Acta Scientiae Circumstantiae, 2019, 39(2): 318-324. (in Chinese) 赵娜娜, 许继飞, 宋晓雪, 等. 嗜盐高效降酚菌株Halomonas sp. H17的筛选及降解苯酚特性[J]. 环境科学学报, 2019, 39(2): 318-324. |
| [17] |
Ren SZ, Guo J, Zeng GQ, et al. Decolorization of triphenylmethane, azo, and anthraquinone dyes by a newly isolated Aeromonas hydrophila strain[J]. Applied Microbiology and Biotechnology, 2006, 72(6): 1316-1321. DOI:10.1007/s00253-006-0418-2 |
| [18] |
Deivasigamani C, Das N. Biodegradation of Basic Violet 3 by Candida krusei isolated from textile wastewater[J]. Biodegradation, 2011, 22(6): 1169-1180. DOI:10.1007/s10532-011-9472-2 |
| [19] |
Chaturvedi V, Bhange K, Bhatt R, et al. Biodetoxification of high amounts of malachite green by a multifunctional strain of Pseudomonas mendocina and its ability to metabolize dye adsorbed chicken feathers[J]. Journal of Environmental Chemical Engineering, 2013, 1(4): 1205-1213. DOI:10.1016/j.jece.2013.09.009 |
| [20] |
Ayed L, Chaieb K, Cheref A, et al. Biodegradation of triphenylmethane dye Malachite Green by Sphingomonas paucimobilis[J]. World Journal of Microbiology and Biotechnology, 2009, 25(4): 705-711. DOI:10.1007/s11274-008-9941-x |
| [21] |
Shang NJ, Ding MJ, Dai MX, et al. Biodegradation of malachite green by an endophytic bacterium Klebsiella aerogenes S27 involving a novel oxidoreductase[J]. Applied Microbiology and Biotechnology, 2019, 103(5): 2141-2153. DOI:10.1007/s00253-018-09583-0 |
| [22] |
Shedbalkar U, Dhanve R, Jadhav J. Biodegradation of triphenylmethane dye cotton blue by Penicillium ochrochloron MTCC 517[J]. Journal of Hazardous Materials, 2008, 157(2/3): 472-479. DOI:10.1016/j.jhazmat.2008.01.023 |
| [23] |
Chou JH, Chen WM, Arun AB, et al. Trabulsiella odontotermitis sp. nov., isolated from the gut of the termite Odontotermes formosanus Shiraki[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(4): 696-700. DOI:10.1099/ijs.0.64632-0 |
| [24] |
Sapountzis P, Gruntjes T, Otani S, et al. The enterobacterium Trabulsiella odontotermitis presents novel adaptations related to its association with fungus-growing termites[J]. Applied and Environmental Microbiology, 2015, 81(19): 6577-6588. DOI:10.1128/AEM.01844-15 |
| [25] |
Barapatre A, Aadil KR, Jha H. Biodegradation of malachite green by the ligninolytic fungus Aspergillus flavus[J]. CLEAN–Soil, Air, Water, 2017, 45(4): 1600045. DOI:10.1002/clen.201600045 |
 2020, Vol. 47
2020, Vol. 47