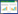扩展功能
文章信息
- 杜如冰, 吴群, 徐岩
- DU Ru-Bing, WU Qun, XU Yan
- 基于三步荧光定量PCR技术揭示不同产区白酒酿造系统中Lactobacillus sp.的分布特征
- Distribution of Lactobacillus sp. in Chinese liquor fermentation system from different producing location by three-step fluorescent quantitative PCR
- 微生物学通报, 2020, 47(1): 1-12
- Microbiology China, 2020, 47(1): 1-12
- DOI: 10.13344/j.microbiol.china.190150
-
文章历史
- 收稿日期: 2019-03-01
- 接受日期: 2019-04-29
- 网络首发日期: 2019-05-29
中国白酒酿造系统广泛分布在全国各地,根据地理概念、白酒生产及消费的集中度,全国可分为十一大产区,并且在同一产区中含有不同的产地,如川酒产区中含有宜宾、泸州等产地[1]。中国白酒属于多菌种混合酿造系统,包含细菌、酵母菌、霉菌等[2]。乳杆菌是发酵中后期的优势细菌,能够产生乳酸、乙酸等代谢产物,直接影响白酒产品的风味,被界定为白酒酿造系统中的核心微生物[3-5],有研究表明清香型白酒发酵过程中缺少Lactobacillus acetotolerans会导致发酵滞后以及重要风味物质酯类化合物含量的减少[6]。因此,有必要构建乳酸菌种的定量方法并揭示在中国白酒系统中的分布特征,推动乳酸菌种在白酒酿造系统中的功能研究。
Lactobacillus sp.是一株存在于中国白酒酿造系统中的酿造微生物,由于目前人工培养条件无法满足其生长要求,尚未得到纯培养物。邢敏钰等[4]通过高通量测序发现Lactobacillus sp.分布于山东潍坊产地芝麻香型白酒酿造系统中,在发酵5 d后相对含量急剧上升,成为中后期优势微生物,并且通过相关性分析发现该菌与其他微生物呈现负相关关系,在发酵过程中扮演着重要角色。因此,有必要构建Lactobacillus sp.的定量方法,揭示其在中国白酒酿造系统中的分布特征,为后续研究Lactobacillus sp.在酿造系统中的代谢功能提供参考。
微生物定量研究一直是微生物领域的研究热点,微生物在特定生态系统中的含量是间接反映生物代谢活性的重要指标。因此,许多方法被用来对微生物进行绝对定量,例如以磷酸脂肪酸检测(phospholipid fatty acids,PLFA)[7]、ATP检测[8-9]、生物量碳检测(microbial biomass carbon,MBC)[9-10]为代表的基于生物物质标记的检测方法;以高通量测序、共聚焦显微镜[11]、流式细胞术[9, 12]、荧光定量PCR[9, 13-15]为代表的基于生物基因标记的定量方法。其中荧光定量PCR是一种基于特异性引物定量特定物种的方法,目前广泛应用于青贮、奶酪、葡萄酒等酿造系统中[16-18],是一种高分辨率、高精准度的检测方法。有研究针对白酒酿造系统中的塔宾曲霉和链霉菌设计特异性引物,构建荧光定量PCR定量方法,用于大曲以及白酒发酵过程中微生物的定量研究[19-20],但尚无针对Lactobacillus sp.特异性引物研究的报道。另外,荧光定量PCR不仅需要特异性引物,而且要求引物自身以及所扩增的产物具有合适的长度:最优的引物长度为18−25 bp,最优的产物长度为80−150 bp[21]。目前尚未得到Lactobacillus sp.的纯培养物,仅16s rRNA基因序列可作为设计特异性引物的靶序列,限制了特异性引物的设计,使得扩增片段长度难以满足常规荧光定量PCR方法的要求。
本研究针对未培养酿造微生物Lactobacillus sp.,首次通过16s rRNA基因序列设计了一对特异性引物PLsp,扩增长度为445 bp;针对长片段扩增产物的荧光定量PCR反应体系,优化退火和延伸程序有效地提高了引物的扩增效率,构建了具有较高准确性和重复性的定量方法;跟踪检测中国10个产区的白酒酿造系统代表样本,揭示了Lactobacillus sp.在中国白酒酿造系统中的分布特征,基于特异性引物PLsp的荧光定量PCR定量方法对于其他酿造系统中未培养微生物的定量具有重要参考意义。
1 材料与方法 1.1 材料 1.1.1 菌株及其活化研究中共使用22种微生物,分别属于10个属。所有微生物均由本实验室从白酒发酵酒醅、大曲、小曲中筛选得到,以25%甘油管的形式保存于−80 ℃。筛选和培养方法为:取5 g样本加入到100 mL无菌生理盐水中,200 r/min振荡30 min,取上清进行平板涂布分离。乳酸菌的分离、活化使用MRS培养基[22],37 ℃条件下培养3 d;霉菌和酵母的分离、活化使用YPD培养基[23],培养条件为30 ℃,霉菌培养5 d,酵母培养2 d;其他细菌的分离、活化使用LB培养基[23],培养条件为37 ℃培养1 d。细菌菌株使用通用引物27F/1429R鉴定,真菌菌株使用通用引物NL1/NL4鉴定,引物序列及其扩增条件参考文献[24-25]。
1.1.2 白酒发酵过程酒醅样本采集不同产区的白酒酿造系统样本信息如表 1所示,每个产地采集3个平行,共计48个,每个样本取样量约200 g,保存于−20 ℃。发酵过程样本采集于山东潍坊产地的某芝麻香型酒厂,跟踪发酵时间取样,采集时间点为0、5、8、10、15、20、40、45 d,每个时间点取样量约200 g,保存于−20 ℃。
| 样本编号 Samples number |
采样地点 Sampling sites |
采样时间 Sampling time |
香型 Aroma style |
产区 Producing area |
| Fenyang | 山西省汾阳市 Fenyang, Shanxi province |
2018年7月 July, 2018 |
清香型 Light aroma |
华北产区 Huabei area |
| Cangzhou | 河北省沧州市 Cangzhou, Hebei province |
2018年7月 July, 2018 |
清香型 Light aroma |
华北产区 Huabei area |
| Sanmenxia | 河南省三门峡市 Sanmenxia, Henan province |
2018年7月 July, 2018 |
清香型 Light aroma |
豫酒产区 Yujiu area |
| Sanmenxia | 河南省三门峡市 Sanmenxia, Henan province |
2018年7月 July, 2018 |
浓香型 Strong aroma |
豫酒产区 Yujiu area |
| Pingdingshan | 河南省平顶山市 Pingdingshan, Henan province |
2018年7月 July, 2018 |
清香型 Light aroma |
豫酒产区 Yujiu area |
| Weifang | 山东省潍坊市 Weifang, Shandong province |
2017年12月 December, 2017 |
浓香型 Strong aroma |
鲁酒产区 Lujiu area |
| Weifang | 山东省潍坊市 Weifang, Shandong province |
2017年12月 December, 2017 |
芝麻香型 Sesame aroma |
鲁酒产区 Lujiu area |
| Fuyang | 安徽省阜阳市 Fuyang, Anhui province |
2018年7月 July, 2018 |
馥郁香型 Fuyu aroma |
皖酒产区 Wanjiu area |
| Fuyang | 安徽省阜阳市 Fuyang, Anhui province |
2018年7月 July, 2018 |
浓香型 Strong aroma |
皖酒产区 Wanjiu area |
| Xuancheng | 安徽省宣城市 Xuancheng, Anhui province |
2018年7月 July, 2018 |
浓香型 Strong aroma |
皖酒产区 Wanjiu area |
| Huangshi | 湖北省黄石市 Huangshi, Hubei province |
2017年7月 July, 2017 |
清香型 Light aroma |
两湖产区 Lianghu area |
| Suqian | 江苏省宿迁市 Suqian, Jiangsu province |
2017年12月 December, 2017 |
芝麻香型 Sesame aroma |
苏酒产区 Sujiu area |
| Luzhou | 四川省泸州市 Luzhou, Sichuan province |
2018年7月 July, 2018 |
浓香型 Strong aroma |
川酒产区 Chuanjiu area |
| Renhuai | 贵州省仁怀市 Renhuai, Guizhou province |
2018年7月 July, 2018 |
酱香型 Soy sauce aroma |
黔南产区 Qiannan area |
| Foshan | 广东省佛山市 Foshan, Guangdong province |
2017年7月 July, 2017 |
豉香型 Chi aroma |
华南产区 Huanan area |
| Xining | 青海省西宁市 Xining, Qinghai province |
2017年12月 December, 2017 |
清香型 Light aroma |
西北产区 Xibei area |
氯化钠、无水葡萄糖、琼脂粉、三氯甲烷、乙醇,国药集团化学试剂(北京)有限公司;MRS培养基、酵母浸膏、蛋白胨,Oxoid公司;细菌基因组DNA快速抽提试剂盒、饱和酚,生工生物工程(上海)股份有限公司;质粒提取试剂盒,Omega公司;AceQ Universal SYBR Green qPCR Master Mix、Green Taq Mix,中国诺唯赞生物科技有限公司。荧光定量PCR仪,Applied Biosystems公司;蛋白核酸测定分光光度计,Thermo Fisher Scientific公司;PCR仪,Eppendorf公司;凝胶成像分析系统,Bio-Rad公司。
1.2 实验方法 1.2.1 基因组DNA的提取微生物纯培养物基因组DNA的提取:各取1 mL处于稳定期的微生物纯培养菌液于1.5 mL离心管中,9 000×g离心5 min,收集菌体沉淀,采用试剂盒提取基因组DNA。酒醅样本基因组DNA提取:称取7 g酒醅于50 mL离心管中,加入15 mL PBS缓冲液,3 g玻璃珠,涡旋振荡5 min,300×g离心5 min,收集上清,重复收集3次。上清菌液中的DNA提取方法参考文献[19]。
1.2.2 引物设计由于实验目标菌株为未培养微生物,无参考基因组信息,因此特异性引物设计选取仅有的16S rRNA基因序列(GenBank登录号为KU674948.1)。选择以下两类菌株的16s rRNA基因用来筛选Lactobacillus sp.的序列差导区:与目标菌株相似度比较大的菌株,Lactobacillus caviae (90%,NR_157747.1)、Lactobacillus fructivorans (90%,NR_036789.1)、Lactobacillus homohiochii (90%,NR_042229.1)、Lactobacillus ixorae (89%,NR_153713.1)、Lactobacillus reuteri (89%,NR_025911.1)、Lactobacillus ozensis (88%,NR_113194.1);在白酒酿造体系中出现频率较高的菌株,Lactobacillus brevis (88%,NR_044704.2)、Lactobacillus acetotolerans (87%,LC202658.1)。16s rRNA基因序列差异区的筛选通过DNAMAN生物信息学软件完成。以上16S rRNA基因序列均来自于NCBI (National Center for Biotechnology Information)数据库(https://www.ncbi.nlm.nih.gov/)。
1.2.3 PCR体系和条件PCR反应体系(25 μL):Green Taq Mix 12.5 μL,上、下游引物(20 μmol/L)各0.2 μL,模板1 μL,补充ddH2O至25 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 1 min,55 ℃ 30 s,72 ℃ 25 s,30个循环。荧光定量PCR反应体系(20 μL):AceQ Universal SYBR Green qPCR Master Mix 10 μL,上、下游引物(20 μmol/L)各0.2 μL,模板1 μL,补充ddH2O至20 μL。两步荧光定量PCR反应条件:98 ℃ 1 min;98 ℃ 10 s,60 ℃ 30 s,40个循环。三步荧光定量PCR反应条件:98 ℃ 1 min;98 ℃ 10 s,55 ℃ 30 s,72 ℃ 25 s,40个循环。溶解曲线均设定为从65 ℃升温至95 ℃,每增加0.5 ℃检测一次荧光信号强度,每次检测持续5 s。每次检测均含有3个技术平行。
1.2.4 有效性和特异性选择已知含有目标微生物的酒醅基因组作为阳性对照,选择白酒酿造系统中常见的22种非目标微生物的基因组作为阴性对照,通过PCR验证引物的有效性和特异性,其中酒醅基因组选择相对含量较高的山东潍坊产区芝麻香型白酒酿造系统40 d和45 d的酒醅样本[4]。根据阳性对照组扩增产物的琼脂糖凝胶电泳结果判断引物的有效性;将PCR扩增产物送至生工生物工程(上海)股份有限公司测序,根据测序结果判断引物扩增产物的特异性,根据阴性对照组扩增产物的琼脂糖凝胶电泳结果判断对于其他微生物的特异性。
1.2.5 质粒构建利用设计的特异性引物,以山东潍坊产地芝麻香型白酒酿造系统45 d的酒醅样本基因组DNA为模板进行目标片段的扩增,取5 μL PCR产物在1.5%的琼脂糖凝胶上电泳检测扩增结果。目标片段经胶回收后与pMD19-T载体连接,具体操作方法参考文献[26]。利用质粒抽提试剂盒抽提质粒,并使用PLsp引物扩增测序验证是否转化成功。以成功构建的质粒作为标准品,使用蛋白核酸定量仪测定质粒浓度,并计算标准品质粒的拷贝数,于−20 ℃保存。
1.2.6 标准曲线构建构建好的质粒标准品以10倍比稀释梯度进行稀释,共制备7个稀释梯度。以原始浓度和经过稀释的质粒标准品作为模板进行qPCR,测定对应的Ct值,构建质粒拷贝数与Ct值之间的线性关系,绘制标准曲线。扩增效率E通过公式E=10(−1/a)−1来计算,其中a是标准曲线的斜率。
1.2.7 荧光定量PCR重复性实验以1.2.6中的质粒作为荧光定量PCR模板,测定对应的Ct值,每个稀释度5次重复,计算Ct值的变异系数,对荧光定量PCR方法的重复性进行评价。
1.2.8 荧光定量PCR方法在生产中的应用以来源不同的酒醅基因组DNA作为模板进行qPCR检测,测定目标菌株在各个样本中的生物量。每次阳性对照选择质粒标准品,阴性对照选择无菌水,保证荧光定量PCR结果的准确性。
1.2.9 数据分析荧光定量PCR实验中标准曲线的线性R2通过Origin软件中的线性拟合功能完成,同一产地不同酿造工艺之间Lactobacillus sp.生物量的差异显著性,通过SPSS软件中的独立样本t检验功能完成。
2 结果与分析 2.1 荧光定量方法的建立 2.1.1 引物设计为构建荧光定量PCR定量方法,首先通过比较Lactobacillus sp.与相似度最低的Lactobacillus acetotolerans的16S rRNA基因序列,筛选用于设计特异性引物的候选基因序列,结果如图 1所示,共筛选到8个区域(区域1−8)。通过进一步对比发现区域2、6、7与Lactobacillus ixorae相似;区域4、5与Lactobacillus caviae相似;同时区域3和区域8相差大于800 bp。因此选择区域1和区域3中的序列设计特异性引物。

|
| 图 1 16S rRNA基因序列比对 Figure 1 Sequence alignment of 16S rRNA gene sequence 注:A:Lactobacillus sp.和Lactobacillus acetotolerans 16S rRNA基因序列比对结果,红色方框代表用于设计特异性引物的候选区域;B:候选区域序列相似性比对. Note: A: 16S rRNA gene sequence alignment result between Lactobacillus sp. and Lactobacillus acetotolerans, red rectangle boxes represent the candidate sequences that were used to design species-specific primer; B: Sequence alignment of the candidate sequence. |
|
|
利用区域1和区域3的基因序列设计出一对特异性引物,命名为PLsp。其他8种微生物相对应位置的序列与PLsp的上游引物序列和比对结果如图 2所示,与上游引物序列的最小差异碱基数为10,与下游序列的最小差异碱基数为8。PLsp的基本信息:上游引物序列为5′-CGCACTCCCGTAGATGA TTTTGA-3′,长度为23 bp,(G+C)mol%为47.83%;下游引物序列为5′-TCACTACCAAGCCATTTCC TAC-3′,长度为22 bp,(G+C)mol%为45.5%。PLsp扩增产物长度为445 bp,两段在DNAMAN中均不存在自我成环结构,以上特征均满足引物的基本要求,可以应用于后续构建荧光定量PCR定量方法。

|
| 图 2 特异性引物序列比对 Figure 2 Sequence alignment of species-specific primer 注:A:上游引物序列;B:下游引物序列.红色代表差异碱基. Note: A: Sequence of upstream primer; B: Sequence of downstream primer. The different bases were marked red. |
|
|
引物适用性验证结果如图 3中泳道23、24所示。条带单一且长度在400−500 bp之间,测序结果发现片段长度为445 bp;通过序列比对,测序片段与设计引物所使用序列相似度为100%,实验结果表明PLsp适用于扩增目标片段且扩增产物单一。根据筛选鉴定结果,获得15株细菌和7株真菌(图 3)并被用来验证引物的特异性,验证结果如图 3泳道1−22所示,PLsp对22种微生物无交叉反应,结果表明PLsp在种间特异性良好,可用于区分Lactobacillus sp.与其他微生物。
|
| 图 3 PCR验证引物特异性 Figure 3 Validating primer specificity by PCR Note: M: 1 000 bp DNA Marker; 1: Lactobacillus acetotolerans; 2: Lactoabcillus brevis; 3: Lactoacillus buchneri; 4: Lactobacillus crustorum; 5: Lactobacillus plantarum; 6: Lactobacillus curvatus; 7: Lactobacillus delbrueckill; 8: Lactobacillus murinus; 9: Lactobacillus reuteri; 10: Lactobacillus johnsonii; 11: Lactobacillus casei; 12: Weissella viridescens; 13: Weissella confusa; 14: Bacillus amyloliquefaciens; 15: Aspergillus tubingensis; 16: Mucor rouxianus; 17: Bacillus licheniformis; 18: Schizosaccharomyces pombe; 19: Zygosaccharomyces bailii; 20: Issatchenkia orientalis; 21: Saccharomycopsis fibuligera; 22: Saccharomyces cerevisiae; 23: Jiupei at 40 d from sesame aroma fermentation process of Weifang region; 24: Jiupei at 45 d from sesame aroma fermentation process of Weifang region. |
|
|
通用两步荧光定量PCR扩增可能会对长片段扩增产物的扩增效率产生影响。因此,本研究通过10倍比梯度稀释山东潍坊产地芝麻香型白酒酿造系统40 d的酒醅基因组,以稀释102–106倍的样本为荧光定量PCR模板,优化退火和延伸程序,构建稳定准确的反应条件。如表 2所示,两步荧光定量PCR扩增结果显示平行之间的Ct值变异系数较大(CV=0.01%−2.65%),扩增稳定性较差;R2=0.40,线性较弱。优化扩增条件:变性98 ℃ 10 s,退火55 ℃ 30 s,延伸72 ℃ 25 s,构建三步荧光定量PCR方法。三步荧光定量PCR扩增结果显示平行之间的变异系数均小于1% (CV=0.17%−0.59%),稳定性较高;R2=0.99,线性较强。实验结果表明优化后的荧光定量PCR条件可以明显地提高检测结果的稳定性和准确性,满足在白酒酿造系统中定量Lactobacillus sp.的应用要求。
| 两步荧光定量PCR Two-step qPCR |
三步荧光定量PCR Three-step qPCR |
|||||||||
| 平行1 Test 1 |
平行2 Test 2 |
平行3 Test 3 |
CV (%) |
线性 Linearity (R2) |
平行1 Test 1 |
平行2 Test 2 |
平行3 Test 3 |
CV (%) |
线性 Linearity (R2) |
|
| 18.67 | 16.80 | 17.56 | 0.53 | 0.40 | 18.20 | 18.06 | 18.20 | 0.44 | 0.99 | |
| 18.73 | 17.25 | 17.57 | 0.44 | 21.41 | 21.66 | 21.52 | 0.59 | |||
| 26.58 | 25.30 | 15.65 | 2.65 | 25.58 | 25.32 | 25.46 | 0.51 | |||
| 30.94 | 30.89 | 30.95 | 0.01 | 28.59 | 28.67 | 28.71 | 0.21 | |||
| 38.47 | 30.85 | 30.97 | 1.31 | 32.18 | 32.28 | 32.21 | 0.17 | |||
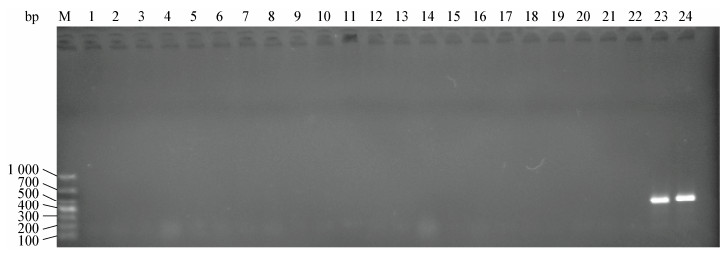
回收的标准质粒浓度为1.78×108 copies/μL,各浓度梯度质粒样本的稀释曲线均为平稳光滑的S型(图 4A);标准曲线的斜率为−3.54,截距为33.85,线性R2=0.99% (图 4B),通过斜率计算扩增效率为91.64%,满足标准曲线的要求,可应用于实验和实际生产。
|
| 图 4 引物PLsp的扩增性能 Figure 4 The amplification efficiency of PLsp primer 注:A:重组质粒梯度的10倍比梯度稀释扩增曲线;B:Lactobacillus sp. 16s rRNA基因拷贝数与Ct值之间的标准曲线. Note: A: The amplification curve of serial 10-fold dilutions of recombinant plasmid; B: The standard curve between Lactobacillus sp. 16S rRNA gene copies number and Ct value. |
|
|
以2.3.1中8个浓度的质粒为扩增模板,研究三步荧光定量PCR定量方法的可重复性。结果如表 3所示,Ct值的变异系数均小于1%,在每个稀释梯度样本的检测结果中均有良好的重复性,证明基于特异性引物PLsp构建的三步荧光定量PCR定量方法具有重复性,适用于定量含有高浓度、中浓度和低浓度Lactobacillus sp.的酒醅样本。
| 稀释度 Dilution |
Ct值 Ct value |
平均值 Mean |
CV (%) |
||||
| 1 | 2 | 3 | 4 | 5 | |||
| 0 | 5.16 | 5.20 | 5.19 | 5.18 | 5.17 | 5.18 | 0.31 |
| 101 | 9.29 | 9.25 | 9.33 | 9.28 | 9.34 | 9.30 | 0.39 |
| 102 | 12.82 | 12.80 | 12.59 | 12.86 | 12.67 | 12.75 | 0.89 |
| 103 | 16.44 | 16.28 | 16.30 | 16.22 | 16.44 | 16.34 | 0.59 |
| 104 | 19.91 | 19.67 | 19.79 | 19.58 | 19.89 | 19.77 | 0.73 |
| 105 | 23.54 | 23.32 | 23.44 | 23.45 | 23.37 | 23.42 | 0.35 |
| 106 | 26.98 | 27.16 | 27.14 | 26.95 | 26.81 | 27.01 | 0.54 |
| 107 | 30.63 | 30.95 | 30.99 | 30.89 | 30.91 | 30.87 | 0.46 |
收集全国十大产区代表产地的白酒酿造系统样本,运用基于特异性引物PLsp构建的荧光定量PCR方法统计Lactobacillus sp.在全国范围内的分布特征。结果如图 5所示,在华北、豫酒、鲁酒、皖酒、两湖、川酒、黔南和西北均检测到Lactobacillus sp.,其中在鲁酒产区酿造系统中的检测值最高(7.27±0.04 lgcopies/g),在皖酒产区酿造系统中的检测值最低(2.98±0.30 lgcopies/g)。实验结果显示Lactobacillus sp.不只是存在于山东潍坊产地的白酒酿造系统中,而是在中国白酒酿造系统中广泛分布。

|
| 图 5 Lactobacillus sp.在空间尺度下的分布 Figure 5 The distribution characteristics of Lactobacillus sp. in different regions 注:*:P < 0.05;**:P < 0.01;***:P < 0.001. A:华北产区;B:豫酒产区;C:鲁酒产区;D:皖酒产区;E:两湖产区;F:苏酒产区;G:川酒产区;H:黔南产区;I:华南产区;J:西北产区. Note: *: P < 0.05; **: P < 0.01; ***: P < 0.001. A: Huabei area; B: Yujiu area; C: Lujiu area; D: Wanjiu area; E: Lianghu area; F: Sujiu area; G: Chuanjiu area; H: Qiannan area; I: Huanan area; J: Xibei area. |
|
|
通过统计Lactobacillus sp.的分布与产区、产地和酿造工艺的关系发现:Lactobacillus sp.的分布与产区无关,虽然在豫酒、皖酒产区的各自两个不同产地酿造系统中均检测到Lactobacillus sp.,但是在华北产区中,在河北沧州产地酿造系统中检测到Lactobacillus sp.,而在山西汾阳产地中未检测到Lactobacillus sp.。Lactobacillus sp.的分布与产地有关,以产地作为分类标准统计分析,结果显示河南三门峡产地、山东潍坊产地、安徽阜阳产地的两种不同酿造工艺样本中均检测到Lactobacillus sp.,推测产地决定Lactobacillus sp.的分布。Lactobacillus sp.的分布与酿造工艺无关,统计芝麻香型、清香型、酱香型、浓香型、豉香型、馥郁香型酿造工艺样本的检测结果显示,Lactobacillus sp.并不存在于所有的清香型酿造工艺样本或者芝麻香型酿造工艺样本中,例如山西汾阳产地的清香型酿造系统和江苏宿迁产地的芝麻香型酿造系统。统计相同产地的不同酿造工艺样本中Lactobacillus sp.的分布特征,结果显示:不同的酿造工艺中均检测到Lactobacillus sp.,但含量存在显著性差异(P < 0.05),如河南三门峡产地的清香型和浓香型白酒酿造系统分别是6.38±0.37 lgcopies/g和7.19±0.25 lgcopies/g,山东潍坊产地的芝麻香型和浓香型白酒酿造系统分别是7.27±0.04 lgcopies/g和6.30±0.16 lgcopies/g,安徽阜阳产地的浓香型和馥郁香型白酒酿造系统分别是6.56±0.09 lgcopies/g和2.98±0.30 lgcopies/g。
2.4.2 Lactobacillus sp.在山东潍坊产地芝麻香型白酒发酵过程中的动态变化山东潍坊产地芝麻香型白酒酿造系统是进一步研究酿造系统中Lactobacillus sp.代谢功能的典型代表系统。邢敏钰等[4]的研究结果显示该酿造系统发酵过程中Lactobacillus sp.的相对含量较高,本研究结果也发现在该酿造系统中Lactobacillus sp.的生物量是最高的,因此以山东潍坊产地芝麻香型白酒酿造系统为研究对象,跟踪Lactobacillus sp.在发酵过程中的动态变化特征。结果如图 6所示,Lactobacillus sp.在芝麻香酿造系统中生长曲线可分为两个阶段:第一阶段为生长期,在入池0−15 d该菌的生物量从3.14±0.14 lgcopies/g增长到7.22±0.13 lgcopies/g;第二阶段(stage Ⅱ)为稳定期,在15−45 d一直维持在7.00 lgcopies/g以上。生物量在40 d时达到最大值,为7.31±0.14 lgcopies/g。Lactobacillus sp.在发酵过程中的动态特征为后续研究Lactobacillus sp.在原位系统不同生长阶段中的代谢功能提供重要参考依据。
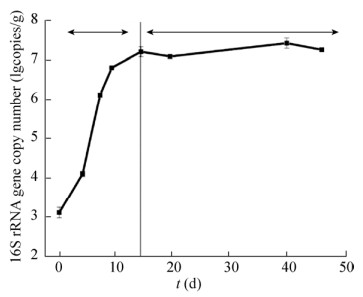
|
| 图 6 Lactobacillus sp.在芝麻香型酿造体系中的生长曲线 Figure 6 The grown curve of Lactobacillus sp. during sesame aroma fermentation process |
|
|
特异性引物和反应条件是限制研究Lactobacillus sp.在中国白酒酿造系统中分布特征的关键因素。本研究首次针对Lactobacillus sp.设计了一对特异性引物PLsp,上游引物长度为22 bp,下游引物长度为23 bp,扩增产物长度为445 bp,特异性验证结果显示PLsp可特异性区分Lactobacillus sp.,不仅适用于荧光定量PCR研究中,而且适用于鉴定Lactobacillus sp.,可应用于其快速筛选,提高筛选效率。先前研究中运用两步荧光定量PCR反应条件对小片段扩增产物的荧光定量PCR反应体系有较好的效果[20],本研究结果显示长扩增片段引物在通用的反应条件下扩增效率较低,结果重复性较差,推测原因可能与引物PLsp扩增片段较长有关,退火、延伸的温度和时间影响了引物的扩增效果。本研究优化扩增条件,构建了一种独立变性、退火、延伸过程的三步反应条件,实验结果表明该反应条件有效提高了引物的扩增效果(R2从0.40提升至0.99)和扩增结果的稳定性(各浓度下的Ct值变异系数小于1%),为今后优化荧光定量PCR反应条件提供参考。
目前,针对Lactobacillus sp.的分布特征研究较少,构建其定量方法,揭示中国白酒酿造系统中Lactobacillus sp.的分布特征,有助于揭示该菌在原位系统中的代谢功能。本研究首次运用基于特异性引物PLsp构建的荧光定量PCR方法,在全国范围内揭示Lactobacillus sp.的分布特征,研究结果表明相同产地的不同酿造工艺酿造系统均检测到Lactobacillus sp.,但不同工艺之间的生物量具有显著性差异,证明产地决定Lactobacillus sp.的分布,酿造工艺影响了其生物量。统计Lactobacillus sp.在全国10个产区的分布特征,结果表明该菌广泛存在于各白酒酿造体系中,推测Lactobacillus sp.在中国白酒酿造系统中具有重要的代谢功能。邢敏钰等[4]采用扩增子测序技术鉴定到Lactobacillus sp.,并对芝麻香型白酒中该菌的变化规律予以揭示,但是相对定量数据无法准确描述该菌的生长变化趋势,反映该菌的代谢活力,为进一步推动Lactobacillus sp.的原位代谢功能研究,本研究跟踪检测该酿造系统发酵过程中Lactobacillus sp.的生物量变化,研究表明:该目标菌株在发酵过程可分为生长期、稳定期两个阶段,并推测该菌在发酵0−15 d代谢较为旺盛,在发酵15 d之后可能开始积累次级代谢产物,但后续研究中需要获得全基因组并结合宏转录组揭示该菌在不同生长阶段中的代谢特征,明确Lactobacillus sp.的具体功能和价值,诠释与酿造品质的关系,如对风味物质代谢的贡献。本文研究结果表明,基于特异性引物的荧光定量PCR方法与基于16s rRNA基因序列的扩增子测序技术相结合,能够更精准地揭示Lactobacillus sp.在时序上的分布特征,以弥补相对定量数据的不足,并为后续利用宏组学技术研究该菌的原位代谢功能提供参考。
本研究首次设计特异性引物PLsp,构建了一种独立变性、退火、延伸过程的三步荧光定量PCR方法,揭示了Lactobacillus sp.在中国白酒酿造系统中的分布特征,同时所提供的特异性引物和定量方法可广泛应用于其他酿造系统中Lactobacillus sp.的鉴定和定量,为进一步结合基因组和宏组学数据揭示Lactobacillus sp.在整个白酒酿造系统中的功能和价值研究提供有效方法和参考。
| [1] |
Meng B, Guo WL, Wang B. Analysis of the regionalization trend of Chinese baijiu (liquor) producing areas based on the protection of geographical indications[J]. Liquor-making Science & Technology, 2014(10): 128-131, 136. (in Chinese) 孟宝, 郭五林, 王兵. 基于地理标志保护的中国白酒产区化趋势分析[J]. 酿酒科技, 2014(10): 128-131, 136. |
| [2] |
Jin GY, Zhu Y, Xu Y. Mystery behind Chinese liquor fermentation[J]. Trends in Food Science & Technology, 2017, 63: 18-28. |
| [3] |
Wang P, Wu Q, Xu Y. Core microbiota in Chinese liquor fermentation and associations with environmental factors[J]. Acta Microbiologica Sinica, 2018, 58(1): 142-153. (in Chinese) 王鹏, 吴群, 徐岩. 中国白酒发酵过程中的核心微生物群及其与环境因子的关系[J]. 微生物学报, 2018, 58(1): 142-153. |
| [4] |
Xing MY, Du H, Xu Y. Diversity and succession of lactic acid bacteria during sesame-flavor liquor fermentation[J]. Microbiology China, 2018, 45(1): 19-28. (in Chinese) 邢敏钰, 杜海, 徐岩. 芝麻香型白酒发酵过程中乳酸菌多样性及其演替规律[J]. 微生物学通报, 2018, 45(1): 19-28. |
| [5] |
Li XL, Wu Q, Xu Y. Synergism between ethanol and acidity drives microbial community succession during roasted sesame-flavor liquor solid-state fermentation[J]. Microbiology China, 2019, 46(1): 1-10. (in Chinese) 李小龙, 吴群, 徐岩. 乙醇与酸度协同作用推动芝麻香型白酒固态发酵过程的微生物群落演替[J]. 微生物学通报, 2019, 46(1): 1-10. |
| [6] |
Pang XN, Han BZ, Huang XN, et al. Effect of the environment microbiota on the flavour of light-flavour Baijiu during spontaneous fermentation[J]. Scientific Reports, 2018, 8: 3396. DOI:10.1038/s41598-018-21814-y |
| [7] |
Frostegård A, Bååth E. The use of phospholipid fatty acid analysis to estimate bacterial and fungal biomass in soil[J]. Biology and Fertility of Soils, 1996, 22(1/2): 59-65. |
| [8] |
Hammes F, Goldschmidt F, Vital M, et al. Measurement and interpretation of microbial adenosine tri-phosphate (ATP) in aquatic environments[J]. Water Research, 2010, 44(13): 3915-3923. |
| [9] |
Zhang ZJ, Qu YY, Li SZ, et al. Soil bacterial quantification approaches coupling with relative abundances reflecting the changes of taxa[J]. Scientific Reports, 2017, 7(1): 4837. |
| [10] |
Cao YF, Li Y, Li CH, et al. Relationship between presence of the desert shrub Haloxylon ammodendron and microbial communities in two soils with contrasting textures[J]. Applied Soil Ecology, 2016, 103: 93-100. |
| [11] |
Mikš-Krajnik K, Babuchowski A. 16S rRNA-targeted oligonucleotide probes for direct detection of Propionibacterium freudenreichii in presence of Lactococcus lactis with multicolour fluorescence in situ hybridization[J]. Letters in Applied Microbiology, 2014, 59(3): 320-327. |
| [12] |
Zhang YY, Chen JJ, Zheng SK. Simultaneous enumeration of yeasts and bacteria in binary samples by FISH-FCM[J]. Actae Ecologica Sinica, 2008, 28(10): 4849-4855. (in Chinese) 张燕燕, 陈进军, 郑少奎. FISH-FCM方法检测酵母-细菌二元体系中微生物数量[J]. 生态学报, 2008, 28(10): 4849-4855. |
| [13] |
Daranas N, Bonaterra A, Francés J, et al. Monitoring viable cells of the biological control agent Lactobacillus plantarum PM411 in aerial plant surfaces by means of a strain-specific viability quantitative PCR method[J]. Applied and Environmental Microbiology, 2018, 84(10): e00107-18. |
| [14] |
Kasturi KN, Drgon T. Real-time PCR method for detection of Salmonella spp. in environmental samples[J]. Applied and Environmental Microbiology, 2017, 83(14): e00644-17. |
| [15] |
Göhler A, Trung TT, Hopf V, et al. Multitarget quantitative PCR improves detection and predicts cultivability of the pathogen Burkholderia pseudomallei[J]. Applied and Environmental Microbiology, 2017, 83(8): e03212-16. |
| [16] |
Settanni L, van Sinderen D, Rossi J, et al. Rapid differentiation and in situ detection of 16 sourdough Lactobacillus species by multiplex PCR[J]. Applied and Environmental Microbiology, 2005, 71(6): 3049-3059. |
| [17] |
Achilleos C, Berthier F. Evaluation of qPCR and plate counting for quantifying thermophilic starters in cheese[J]. Food Microbiology, 2017, 65: 149-159. |
| [18] |
Soares-Santos V, Pardo I, Ferrer S. Cells-qPCR as a direct quantitative PCR method to avoid microbial DNA extractions in grape musts and wines[J]. International Journal of Food Microbiology, 2017, 261: 25-34. |
| [19] |
Lu H, Du H, Xu Y, et al. The application of real-time PCR for quantify TDMTDL producing micobes in liquor brewing process[J]. Microbiology China, 2014, 41(4): 786-792. (in Chinese) 路虎, 杜海, 徐岩, 等. 荧光定量PCR法定量白酒固态发酵过程中产土味素的链霉菌[J]. 微生物学通报, 2014, 41(4): 786-792. |
| [20] |
Chen B, Wu Q, Xu Y. Detection and quantification the biomass of Aspergillus tubingensis in Chinese liquor fermentation process by real-time qPCR[J]. Microbiology China, 2014, 41(12): 2547-2554. (in Chinese) 陈笔, 吴群, 徐岩. 荧光定量PCR方法检测白酒发酵过程中Aspergillus tubingensis生物量[J]. 微生物学通报, 2014, 41(12): 2547-2554. |
| [21] |
Chuang LY, Cheng YH, Yang CH. Specific primer design for the polymerase chain reaction[J]. Biotechnology Letters, 2013, 35(10): 1541-1549. |
| [22] |
Meng X, Wu Q, Wang L, et al. Improving flavor metabolism of Saccharomyces cerevisiae by mixed culture with Bacillus licheniformis for Chinese Maotai-flavor liquor making[J]. Journal of Industrial Microbiology & Biotechnology, 2015, 42(12): 1601-1608. |
| [23] |
Zhang Y, Du H, Wu Q, et al. Impacts of two main lactic acid bacteria on microbial communities during Chinese Maotai-flavor liquor fermentation[J]. Microbiology China, 2015, 42(11): 2087-2097. (in Chinese) 张艳, 杜海, 吴群, 等. 酱香型白酒发酵中两株主要乳酸菌对酿造微生物群体的影响[J]. 微生物学通报, 2015, 42(11): 2087-2097. |
| [24] |
Zhang LX, Gao WY, Wang HY. Identification of microorganism from traditional Chinese medicine Massa Medicata Fermentata[J]. China Journal of Chinese Materia Medica, 2012, 37(13): 1928-1931. (in Chinese) 张丽霞, 高文远, 王海洋. 六神曲中酵母菌的鉴定[J]. 中国中药杂志, 2012, 37(13): 1928-1931. |
| [25] |
Su J, Li SM. Isolation and identification of soil-borne pathogenic fungi and biocontrol becteria[J]. Grassland and Turf, 2019, 39(3): 79-84, 91. (in Chinese) 苏静, 李双明. 土传病原真菌及其生防细菌的分类鉴定[J]. 草原与草坪中国中药杂志, 2019, 39(3): 79-84, 91. |
| [26] |
Moser A, Berthoud H, Eugster E, et al. Detection and enumeration of Lactobacillus helveticus in dairy products[J]. International Dairy Journal, 2017, 68: 52-59. |
 2020, Vol. 47
2020, Vol. 47