扩展功能
文章信息
- 赵化冰, 覃琨, 谭之磊, 李华英, 李德金, 郭锦, 刘芳
- ZHAO Hua-Bin, QIN Kun, TAN Zhi-Lei, LI Hua-Ying, LI De-Jin, GUO Jin, LIU Fang
- 邻苯二酚1, 2-双加氧酶的结构、功能及同工酶现象研究进展
- Advances in study on structure, enzymatic property and isoenzymes of catechol 1, 2-dioxygenases
- 微生物学通报, 2019, 46(9): 2419-2425
- Microbiology China, 2019, 46(9): 2419-2425
- DOI: 10.13344/j.microbiol.china.180758
-
文章历史
- 收稿日期: 2018-09-29
- 接受日期: 2019-02-26
- 网络首发日期: 2019-03-21
2. 中国人民武装警察部队后勤学院天津市职业与环境危害防制重点实验室 天津 300309
2. Tianjin Key Laboratory for Prevention and Control of Occupational and Environmental Hazard, Logistic University of Chinese People's Armed Police Force, Tianjin 300309, China
多环芳烃(Polycyclic aromatic hydrocarbons,PAHs)是一类含有两个及两个以上苯环的化合物,是工业生产中一种重要的工业原料及产品,由于在环境中难以自然降解而长期存留,具有潜在的“三致效应”,一直受到研究者密切关注[1]。
微生物降解是多环芳烃降解的主要途径,目前关于微生物降解多环芳烃的相关酶类多有报道。邻苯二酚(又名儿茶酚)是芳香族化合物降解过程中一种重要的中间产物,邻苯二酚双加氧酶是一类含铁的氧化酶,是降解邻苯二酚的关键酶,它能够断裂邻苯二酚的苯环结构并在断裂的碳原子两端加上一个氧原子,使得多环芳烃类化合物转化为长碳链并最终进入三羧酸循环而降解为二氧化碳和水[2]。这一类能够使苯环结构开环的酶类在芳香族化合物降解的过程中发挥了重要作用,在多种细菌尤其是土壤菌株中广泛存在[3]。
邻苯二酚双加氧酶根据开环位置的不同,可以分为邻苯二酚1, 2-双加氧酶(Catechol 1, 2-dioxygenases,C12O)和邻苯二酚2, 3-双加氧酶(Catechol 2, 3-dioxygenases,C23O)[4],C12O催化邻苯二酚内开环生成顺, 顺-粘康酸(Cis, cis-Muconic acid),是邻位裂解途径(图 1)中的关键酶,而邻苯二酚2, 3-双加氧酶催化邻苯二酚外开环生成粘康酸半醛(2-Hydroxymuconic semialdehyde),是间位裂解途径中的关键酶,这两条降解途径是多环芳烃降解过程中最重要的分支。
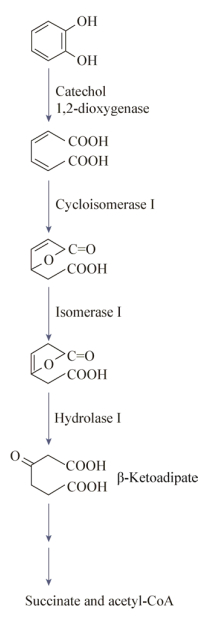
|
| 图 1 邻苯二酚的邻位降解通路 Figure 1 The intradiol-cleaving of catechol |
|
|
邻苯二酚在C12O的作用下,通过邻位裂解途径打开苯环,这一途径又叫β-酮己二酸途径。C12O是β-酮己二酸途径中的关键酶,属于内二元醇双加氧酶家族。邻苯二酚通过C12O催化生成产物顺, 顺-粘康酸,在下游粘康酸环异构酶Ⅰ、异构酶Ⅰ、水解酶的作用下继续分解,其代谢最终生成琥珀酸盐和乙酰辅酶A,将有毒性的芳香族化合物通过细菌代谢为可进入三羧酸循环的化合物[5]。C12O催化得到的中间产物顺, 顺-粘康酸也是合成新型树脂材料、药物、农药的重要原料。
根据底物特异性的不同,目前C12O可分为三类,第一类C12O对氯儿茶酚有很好的催化作用,在氯代芳香族化合物的降解途径中起到了重大作用;第二类C12O对儿茶酚、4-甲基儿茶酚具有相对高的活性;第三类C12O对3-甲基儿茶酚、4-甲基儿茶酚具有较高的活性,这类酶在革兰氏阳性菌中分布最为广泛[6]。
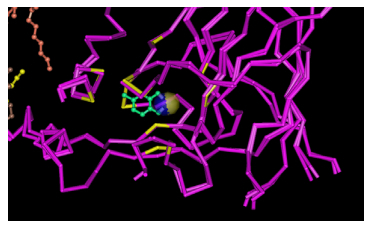
2 C12O的结构特点邻苯二酚1, 2-双加氧酶、原儿茶酸3, 4-双加氧酶(Protocatechuate 3, 4-dioxygenase,3, 4-PCD)、偏苯三酚1, 2-双加氧酶(Hydroxyquinol 1, 2-dioxygenase,1, 2-HQD)是3种能够只用非血红素铁作为辅因子催化邻位裂解途径的氧化酶类,属于非血红素铁加氧酶大家族,在结构上具有相似性[7]。1988年第一次对3, 4-PCD的晶体衍射发现,非血红色铁作为辅因子被2个组氨酸残基和2个酪氨酸残基所固定,与一个水分子或羟基基团构成3, 4-PCD的活性中心[8],之后通过对Acinetobacter sp.ADP1和Pseudomonas arvilla C-1中的C12O进行了晶体衍射,对C12O的结构也有了一定的了解[9]。
通过X射线衍射结构发现,酶和底物的结合域位于Leu73、Pro76、Ile105、Pro108、Leu109、Arg221、Phe253、Ala254所构成的袋状结构,其中Ile105、Pro108、Arg221和Phe253在C12O家族中高度保守[10],C12O与3, 4-PCD的结构相似,大多是由两个相同亚单位形成的二聚体,每个亚单位的分子量大都在30−34 kD,每个亚单位包括一个三价的铁离子,这个铁离子被两个组氨酸、两个酪氨酸和一个羟基基团固定,共同形成一个三角双锥型结构并构成了C12O催化的活性中心,与3, 4-PCD不同的是,每个C12O均包含两分子的磷脂,同时在两个亚单位的连接部分,C12O形成一个特殊的螺旋拉链式结构域[11]。扫描C12O和底物结合的结构发现,原本约束铁离子的酪氨酸和羟基不再与铁离子相连,形成一个铁离子在中心的八面体空间构象,使得底物能够与铁离子结合形成复合体[12](图 2)。
邻苯二酚经过C12O催化后生成的裂解产物顺, 顺-粘康酸在260 nm处有特异性吸收峰,因此可通过测定260 nm处反应溶液的光吸收值,来测定C12O的酶活,其吸光系数ε[L/(mol·cm)]=16 800[13]。
3.2 C12O的酶学性质 3.2.1 C12O的基本酶学性质C12O最适反应pH多在7.0−8.0之间,当pH大于9.0或小于6.0时,酶活即迅速下降。当C12O在25 ℃以邻苯二酚作为底物进行反应时,Km值大多在1−10 mmol/L之间,不同菌属间无明显差异。大多数C12O的最佳反应温度为45 ℃,研究者发现也有喜温性的C12O,嗜热土芽孢杆菌分泌的耐热C12O最适反应温度高达60 ℃[14],甚至当温度达到90 ℃时依然能保持一定的活性。大多数C12O耐热性较差,超过40 ℃ 10 min即迅速失活。
C12O的底物特异性与其菌种来源有一定的关系[15],一般而言,革兰氏阳性菌中C12O通常对邻苯二酚和甲基邻苯二酚具有较高的催化效率,对邻位和间位的甲基邻苯二酚催化效率没有差异性,而革兰氏阴性菌中C12O则仅对4-甲基邻苯二酚具有较高的催化效率,对邻苯二酚及甲基邻苯二酚的催化效率较低,其中的结构差异还需要进一步的研究[16]。
3.2.2 C12O在不同介质下的酶活为了解C12O在不同环境下酶学特性的差异,一些学者尝试在不同介质中测定C12O的酶学特性。Sanakis等为了解天然状态下C12O的性质,从Pseudomonas putida DSM437分离得到C12O,并在完全无水的有机环境中测定了C12O的酶学性质,并对水环境和有机环境中酶学性质的差异作了理论上的分析,结果发现在有机环境中酶依然具有催化活性但酶活力已大大减小[17],而Di Nardo等则尝试把纳米材料应用到酶学研究中,他们将C12O固定化到一种新型材料纳米海绵上,使得C12O的Km值从2.0±0.3 mmol/L,提高到16.6±4.8 mmol/L,最适反应温度从30 ℃提高到50 ℃,同时对环境的耐受性更好[18]。Guzik等则通过海藻酸钙凝胶将C12O进行固化,从而增加其稳定性和降解效率,结果发现通过海藻酸钙凝胶固化后的C12O具有更低的最适反应温度,更大的底物降解范围及对外界环境更强的抵抗性[19]。
4 C12O同工酶及表达调控 4.1 C12O的编码基因自1950年Hayaishi首次报道C12O以来[20],邻苯二酚1, 2-双加氧酶得到了广泛研究,已在不动杆菌属、红球菌属、鞘氨醇单胞菌属、假单胞菌属等多种菌属中克隆得到多个C12O基因[21-23]。
当前报道的大多数C12O编码基因都命名为catA,由936个核苷酸组成,多位于catRBCA基因簇上,受调控基因catR调节。大部分catA都位于细菌的基因组上,也有少数位于质粒上。例如,Pseudomonas putida ND6菌株中的质粒pND6-1上存在一个单独的C12O编码基因catA,序列分析发现其周围有转座基因,因此很有可能是基因水平转移的结果[24]。
4.2 C12O的同工酶除了在多个种属中克隆到C12O编码基因外,在多个芳香烃降解菌株中报道了C12O同工酶的存在[25-26],例如Frateuria species ANA-18[27]和Acinetobacter lwoffii K24[28]中都发现有2种不同的C12O (CatA1和CatA2),Arthrobacter BA-5-17中有4种不同的C12O[29]。我们课题组前期在天津市南排污河废水中分离得到一株可以高效降解萘的Pseudomonas putida ND6[30]。通过全基因组测序发现ND6中也存在4个不同的C12O编码基因[31],其中3个位于ND6基因组上,一个位于ND6特有的质粒pND6-1上,这4个C12O编码基因分属不同的基因簇,一致性约70%,在系统发育树上属于不同的分支。
长期进化使得微生物具有非常经济的基因组,而芳香烃降解细菌中C12O同工酶现象因何存在,是否有助于其提升降解芳香烃的能力,值得思考和研究。回顾文献可发现,同工酶往往出现在降解途径的限速步骤,例如芳香烃降解过程中编码芳环羟基化双加氧酶和芳环断裂双加氧酶的基因。邻苯二酚是多种芳香烃降解过程的共同中间产物,其开环过程是芳香烃代谢共有的限速步骤,因此邻苯二酚间位(meta-)和邻位(ortho-)两种降解途径共存在芳香烃降解菌中十分常见,例如苯甲酸降解菌Pseudomonas putida P8、萘降解菌Pseudomonas putida G7和Pseudomonas putida NCIB 9816等[32-34]。由于芳香族物质代谢过程中多有毒性产物积累,即便是能够以芳香烃为唯一碳源和能源生长的降解菌,芳香烃降解的中间代谢产物也会对其造成细胞毒性。因此,这些针对中间产物的催化步骤也往往存在同工酶,以增加酶的剂量,减轻这些中间产物对细胞的毒害。我们的研究[35]也佐证这一推论,当采用较高的萘浓度(4 g/L)胁迫萘降解菌ND6菌株后,检测其蛋白表达谱变化,发现与萘降解相关的上调的蛋白一共有4个,分别是两个C12O蛋白及其下游的粘糠酸环异构酶。其次,由于儿茶酚开环步骤的重要性,C12O的同工酶现象也可以增强微生物对于该基因突变的适应性。我们研究发现,ND6菌株中所有的C12O编码基因在萘降解过程中均发挥生物学活性,敲除其中任意一个编码基因,萘降解能力虽然略有下降,但是不会丧失(结果正在整理中)。Pseudomonas putida mt-2菌中既有儿茶酚的邻位开环途径,也有儿茶酚间位开环途径,其含有2个C12O基因(catA1,catA2),catA2对于维持mt-2菌株中TOL质粒pWW0的稳定存在具有十分重要的意义[36]。再次,我们发现ND6菌株中有4个C12O同工酶,这4个C12O在最适反应pH及底物特异性方面比较相似,但是,在最适反应温度、米氏常数等方面又有所不同,这些差异有利于ND6菌株适应不同的环境条件[37]。C12O同工酶酶学性质差异的现象在Frateuria species ANA-18[38]和Acinetobacter radioresistens S13[39]等菌株中也存在。
4.3 C12O同工酶编码基因的表达调控C12O同工酶编码基因的表达一种是菌体内组成型表达,另一种则与特定的芳香族化合物诱导有关。C12O能够被苯胺苯甲酰胺、甲苯、苯甲醇、苯甲酸盐和苯酚等多种化合物诱导产生,细菌内多个编码C12O的基因根据诱导物的不同而具有不同的调控模式。例如在Acinetobacter radioresistens S13中,如果以苯酚作为碳源培养时,菌体内只能合成一种C12O,而以甲苯作为碳源培养在同样条件下培养时,菌体内则可以产生2种不同的C12O同工酶[39]。Burkholderia sp. TH2[25]在甲苯中培养时可产生两种不同的C12O (CatA1和CatA2),但在氯苯甲酸中却只能合成一种C12O (CatA2),通过基因敲除去掉catA2后发现catA2对细菌生长并不是必需的,但可以提高在氯苯甲酸中的生长状态,在嗜盐单胞菌属G-16.1中,以苯酚作为碳源培养产生的C12O比以苯甲酸作为碳源培养产生的C12O活性高了接近一倍[40]。而我们的研究还发现,ND6菌株中染色体上的2个编码儿茶酚邻位开环的基因簇(catRⅠBⅠCⅠAⅠ和catRⅡBⅡCⅡAⅡ)可以交互调控,敲除任意一个调控基因(catRⅠ或catRⅡ),另一个基因簇仍然可以表达(结果正在整理中)。
综上所述,对降解菌株中C12O同工酶存在的生态学意义提出以下推断:
(1) C12O催化的儿茶酚开环是芳烃降解的限速步骤,因而同工酶的存在可以增加酶的剂量,减少儿茶酚积累对菌体细胞的危害。
(2) 由于儿茶酚开环步骤的重要性,C12O同工酶的存在降低了基因突变带来的风险,提高了细菌的适应性。
(3) C12O同工酶的氨基酸序列往往存在差异,因此其酶学性质也不尽相同,对增强细菌对芳烃的利用及环境适应性都是有利的。
(4) C12O同工酶的表达调控模式也往往不一样,并存在同工酶所在操纵子的交互调控现象。
因此,多种芳香烃降解菌中存在C12O同工酶不是偶然现象,而是作为细菌适应环境的一种进化优势被选择并保留了下来。
5 C12O的应用 5.1 用于污染物检测及PAHs生物降解芳香族化合物具有难以降解的特点,易在环境长期存留,对土壤微生物、植物和动物造成持续毒害,进而损害土壤功能,并可通过食物链最终富集到人体中,成为潜在的致癌物质。检测环境中PAHs具有十分重要的意义,而C12O可作为检测环境中PAHs的一种有效方式,Zucolotto等将氯儿茶酚1, 2-双加氧酶与纳米材料联用,可检测到环境中低至10−10 mol/L的邻苯二酚[41]。同时,尽管C12O对芳香烃类化合物具有催化降解能力,但由于芳香烃性质稳定,降解效率不高,限制了其工业化应用的进程,因此,研究人员试图利用计算机模拟设计、定向进化、新型材料等先进技术,对C12O编码基因进行改造,以提高C12O对芳香烃类化合物的降解能力并扩大其底物选择范围。Han等将Acinetobactersp.ADP1中C12O的76位氨基酸由脯氨酸定向突变为丙氨酸,相当于人工扩大了结构域的范围,使得C12O的催化能力提高了十倍[11],而Nardo等将C12O固定到纳米材料中,发现其催化效率、酶学特性均有大幅度提高,为降解环境中PAHs污染提供了一种好的选择[18]。
5.2 用于工业原料顺, 顺-粘康酸的生产顺, 顺-粘康酸是工业生产中一种重要的原材料,它不仅能够通过氢化作用形成脂肪酸以合成杀虫剂、杀菌剂、聚氨酯、尼龙等材料,同时也是一些新型树脂、药物的合成原料,目前通过生物合成是最有效、经济且安全的方式。
通过对C12O编码基因或表达环境进行调控,可经过多种方式提高C12O的产量或催化效能:(1)通过基因改造使得菌体内能够定向进行蛋白合成;(2)通过提高菌体内C12O产量达到提高转化效率的目的[42];(3)通过定点突变改变C12O空间构象;(4)通过加入特殊介质材料,最终达到提高C12O酶促效率,提高顺, 顺-粘康酸产量的目的,顺, 顺-粘康酸不仅具有较好的经济价值,同时对环境友好,不会有其他有毒副产物[43]。
6 总结与展望邻苯二酚双加氧酶是芳香烃降解过程中非常重要的一类酶,在芳香族化合物降解过程中起着重要的作用。C12O是邻苯二酚在β-酮己二酸降解途径中的关键酶,目前对C12O的性质和结构已做了深入的研究,对底物与酶相互作用过程也有了一定的了解,不同基因表达的C12O对不同底物具有不同的降解特性,其酶学性质也表现出不同的特点,进一步探索C12O的多样性,是目前研究C12O酶学性质的一个重要方面。
C12O在微生物体内多存在同工酶现象,它们往往出现在降解过程中的限速步骤和毒性中间产物的催化步骤,因此对同工酶的协同作用方式及分子生态学意义的研究具有十分重要的意义,不断发展的基因组学、蛋白质组学和宏基因组学等现代分子生物学技术,为研究人员开发新颖的C12O资源,寻找、发现其同工酶编码基因存在意义提供了更多更有效的手段,使C12O有了更广阔的应用前景。
| [1] |
Haritash AK, Kaushik CP. Biodegradation aspects of polycyclic aromatic hydrocarbons (PAHs): a review[J]. Journal of Hazardous Materials, 2009, 169(1/3): 1-15. |
| [2] |
Broderick JB. Catechol dioxygenases[J]. Essays in Biochemistry, 1999, 34: 173-189. DOI:10.1042/bse0340173 |
| [3] |
Wilcke W. SYNOPSIS polycyclic aromatic hydrocarbons (PAHs) in soil—a review[J]. Journal of Plant Nutrition and Soil Science, 2000, 163(3): 229-248. DOI:10.1002/1522-2624(200006)163:3<229::AID-JPLN229>3.0.CO;2-6 |
| [4] |
Murakami S, Kodama N, Shinke R, et al. Classification of catechol 1, 2-dioxygenase family: sequence analysis of a gene for the catechol 1, 2-dioxygenase showing high specificity for methylcatechols from Gram+ aniline-assimilating Rhodococcus erythropolis AN-13[J]. Gene, 1997, 185(1): 49-54. |
| [5] |
Harayama S, Rekik M. Bacterial aromatic ring-cleavage enzymes are classified into two different gene families[J]. The Journal of Biological Chemistry, 1989, 264(26): 15328-15333. |
| [6] |
Xu B, Xiong CY, Deng M, et al. Genetic diversity of catechol 1, 2-dioxygenase in the fecal microbial metagenome[J]. Journal of Basic Microbiology, 2017, 57(10): 883-895. DOI:10.1002/jobm.201700106 |
| [7] |
Matera I, Ferraroni M, Kolomytseva M, et al. Catechol 1, 2-dioxygenase from the Gram-positive Rhodococcus opacus 1CP: Quantitative structure/activity relationship and the crystal structures of native enzyme and catechols adducts[J]. Journal of Structural Biology, 2010, 170(3): 548-564. DOI:10.1016/j.jsb.2009.12.023 |
| [8] |
Micalella C, Martignon S, Bruno S, et al. X-ray crystallography, mass spectrometry and single crystal microspectrophotometry: a multidisciplinary characterization of catechol 1, 2 dioxygenase[J]. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics, 2011, 1814(6): 817-823. DOI:10.1016/j.bbapap.2010.09.008 |
| [9] |
Earhart CA, Vetting MW, Gosu R, et al. Structure of catechol 1, 2-dioxygenase from Pseudomonas arvilla[J]. Biochemical and Biophysical Research Communications, 2005, 338(1): 198-205. DOI:10.1016/j.bbrc.2005.08.221 |
| [10] |
Di Nardo G, Tilli S, Pessione E, et al. Structural roles of the active site iron(Ⅲ) ions in catechol 1, 2-dioxygenases and differential secondary structure changes in isoenzymes A and B from Acinetobacter radioresistens S13[J]. Archives of Biochemistry and Biophysics, 2004, 431(1): 79-87. DOI:10.1016/j.abb.2004.07.024 |
| [11] |
Han L, Liu P, Sun JX, et al. Engineering catechol 1, 2-dioxygenase by design for improving the performance of the cis, cis-muconic acid synthetic pathway in Escherichia coli[J]. Scientific Reports, 2015, 5: 13435. DOI:10.1038/srep13435 |
| [12] |
Váradi T, Pap JS, Giorgi M, et al. Iron(Ⅲ) complexes with meridional ligands as functional models of intradiol-cleaving catechol dioxygenases[J]. Inorganic Chemistry, 2013, 52(3): 1559-1569. DOI:10.1021/ic302378r |
| [13] |
Dorn E, Knackmuss HJ. Chemical structure and biodegradability of halogenated aromatic compounds. Substituent effects on 1, 2-dioxygenation of catechol[J]. Biochemical Journal, 1978, 174(1): 85-94. DOI:10.1042/bj1740085 |
| [14] |
Giedraityte G, Kalėdienė L. Catechol 1, 2-dioxygenase from α-naphthol degrading thermophilic Geobacillus sp. strain: purification and properties[J]. Central European Journal of Biology, 2009, 4(1): 68-73. |
| [15] |
Broderick JB, O'Halloran TV. Overproduction, purification, and characterization of chlorocatechol dioxygenase, a non-heme iron dioxygenase with broad substrate tolerance[J]. Biochemistry, 1991, 30(29): 7349-7358. DOI:10.1021/bi00243a040 |
| [16] |
Vaillancourt FH, Bolin JT, Eltis LD. The ins and outs of ring-cleaving dioxygenases[J]. Critical Reviews in Biochemistry and Molecular Biology, 2006, 41(4): 241-267. DOI:10.1080/10409230600817422 |
| [17] |
Sanakis Y, Mamma D, Christakopoulos P, et al. Catechol 1, 2-dioxygenase from Pseudomonas putida in organic media—an electron paramagnetic resonance study[J]. International Journal of Biological Macromolecules, 2003, 33(1/3): 101-106. |
| [18] |
Di Nardo G, Roggero C, Campolongo S, et al. Catalytic properties of catechol 1, 2-dioxygenase from Acinetobacter radioresistens S13 immobilized on nanosponges[J]. Dalton Transactions, 2009(33): 6507-6512. DOI:10.1039/b903105g |
| [19] |
Guzik U, Hupert-Kocurek K, Marchlewicz A, et al. Enhancement of biodegradation potential of catechol 1, 2-dioxygenase through its immobilization in calcium alginate gel[J]. Electronic Journal of Biotechnology, 2014, 17(2): 83-88. DOI:10.1016/j.ejbt.2014.02.001 |
| [20] |
Hayaishi O, Stanier RY. The bacterial oxidation of tryptophan.Ⅲ Enzymatic activities of cell-free extracts from bacteria employing the aromatic pathway[J]. Journal of Bacteriology, 1951, 62(6): 691-709. |
| [21] |
Pandeeti EVP, Siddavattam D. Purification and characterization of catechol 1, 2-dioxygenase from Acinetobacter sp. DS002 and cloning, sequencing of partial catA gene[J]. Indian Journal of Microbiology, 2011, 51(3): 312-318. DOI:10.1007/s12088-011-0123-4 |
| [22] |
Sun JQ, Huang X, Chen QL, et al. Isolation and characterization of three Sphingobium sp. strains capable of degrading isoproturon and cloning of the catechol 1, 2-dioxygenase gene from these strains[J]. World Journal of Microbiology and Biotechnology, 2009, 25(2): 259-268. DOI:10.1007/s11274-008-9888-y |
| [23] |
Guzik U, Greń I, Hupert-Kocurek K, et al. Catechol 1, 2-dioxygenase from the new aromatic compounds-degrading Pseudomonas putida strain N6[J]. International Biodeterioration & Biodegradation, 2011, 65(3): 504-512. |
| [24] |
Zhao HB, Chen W, Cai BL. Cloning and expression of catA gene from Pseudomonas putida ND6 and study on the catechol cleavage pathway[J]. Acta Microbiologica Sinica, 2007, 47(3): 387-391. (in Chinese) 赵化冰, 陈威, 蔡宝立. 恶臭假单胞菌ND6菌株catA基因的克隆和表达及其儿茶酚裂解途径探讨[J]. 微生物学报, 2007, 47(3): 387-391. DOI:10.3321/j.issn:0001-6209.2007.03.003 |
| [25] |
Suzuki K, Ichimura A, Ogawa N, et al. Differential expression of two catechol 1, 2-dioxygenases in Burkholderia sp. strain TH2[J]. Journal of Bacteriology, 2002, 184(20): 5714-5722. DOI:10.1128/JB.184.20.5714-5722.2002 |
| [26] |
Briganti F, Pessione E, Giunta C, et al. Purification and catalytic properties of two catechol 1, 2-dioxygenase isozymes from benzoate-grown cells of Acinetobacter radioresistens[J]. Journal of Protein Chemistry, 2000, 19(8): 709-716. DOI:10.1023/A:1007116703991 |
| [27] |
Murakami S, Takashima A, Takemoto J, et al. Cloning and sequence analysis of two catechol-degrading gene clusters from the aniline-assimilating bacterium Frateuria species ANA-18[J]. Gene, 1999, 226(2): 189-198. DOI:10.1016/S0378-1119(98)00560-5 |
| [28] |
Kim SI, Leem SH, Choi JS, et al. Cloning and characterization of two catA genes in Acinetobacter lwoffii K24[J]. Journal of Bacteriology, 1997, 179(16): 5226-5231. DOI:10.1128/jb.179.16.5226-5231.1997 |
| [29] |
Muakami S, Wang LC, Naito A, et al. Purification and characterization of four catechol 1, 2-dioxygenase isozymes from the benzamide-assimilating bacterium Arthrobacter species BA-5-17[J]. Microbiological Research, 1998, 153(2): 163-171. DOI:10.1016/S0944-5013(98)80036-0 |
| [30] |
Zhang XP, Yue XH, Huang JY, et al. Isolation and identification of naphthalene-degrading plasmid pND6[J]. Chinese Journal of Applied and Environmental Biology, 2000, 6(2): 187-190. (in Chinese) 张心平, 岳晓含, 黄今勇, 等. 萘降解质粒pND6的分离和鉴定[J]. 应用与环境生物学报, 2000, 6(2): 187-190. DOI:10.3321/j.issn:1006-687X.2000.02.018 |
| [31] |
Li SS, Zhao HB, Li YX, et al. Complete genome sequence of the naphthalene-degrading Pseudomonas putida strain ND6[J]. Journal of Bacteriology, 2012, 194(18): 5154-5155. DOI:10.1128/JB.01190-12 |
| [32] |
Cao B, Loh KC. Catabolic pathways and cellular responses of Pseudomonas putida P8 during growth on benzoate with a proteomics approach[J]. Biotechnology and Bioengineering, 2008, 101(6): 1297-1312. DOI:10.1002/bit.21997 |
| [33] |
Barnsley EA. Role andregulation of the ortho and meta pathways of catechol metabolism in pseudomonads metabolizing naphthalene and salicylate[J]. Journal of Bacteriology, 1976, 125(2): 404-408. |
| [34] |
Williams PA, Catterall FA, Murray K. Metabolism of naphthalene, 2-methylnaphthalene, salicylate, and benzoate by Pseudomonas PG: regulation of tangential pathways[J]. Journal of Bacteriology, 1975, 124(2): 679-685. |
| [35] |
Li SS, Hu X, Zhao H, et al. Quantitative analysis of cellular proteome alterations of Pseudomonas putida to naphthalene-induced stress[J]. Biotechnology Letters, 2015, 37(8): 1645-1654. DOI:10.1007/s10529-015-1828-y |
| [36] |
Jiménez JI, Pérez-Pantoja D, Chavarría M, et al. A second chromosomal copy of the catA gene endows Pseudomonas putida mt-2 with an enzymatic safety valve for excess of catechol[J]. Environmental Microbiology, 2014, 16(6): 1767-1778. DOI:10.1111/1462-2920.12361 |
| [37] |
Li SS, Qin K, Li HY, et al. Cloning and characterisation of four catA genes located on the chromosome and large plasmid of Pseudomonas putida ND6[J]. Electronic Journal of Biotechnology, 2018, 34: 83-90. DOI:10.1016/j.ejbt.2018.06.001 |
| [38] |
Aoki K, Konohana T, Shinke R, et al. Two catechol 1, 2-dioxygenases from an aniline-assimilating bacterium, Frateuria species ANA-18[J]. Agricultural and Biological Chemistry, 1984, 48(8): 2097-2104. |
| [39] |
Caposio P, Pessione E, Giuffrida G, et al. Cloning and characterization of two catechol 1, 2-dioxygenase genes from Acinetobacter radioresistens S13[J]. Research in Microbiology, 2002, 153(2): 69-74. DOI:10.1016/S0923-2508(01)01290-6 |
| [40] |
de Lourdes MM, Sánchez-PC, Piubeli F, et al. Cloning, characterization and analysis of cat and ben genes from the phenol degrading halophilic bacterium Halomonas organivorans[J]. PLoS One, 2011, 6(6): 21049. DOI:10.1371/journal.pone.0021049 |
| [41] |
Zucolotto V, Pinto APA, Tumolo T, et al. Catechol biosensing using a nanostructured layer-by-layer film containing Cl-catechol 1, 2-dioxygenase[J]. Biosensors and Bioelectronics, 2006, 21(7): 1320-1326. DOI:10.1016/j.bios.2005.06.001 |
| [42] |
Aya K, Yoshitaka I, Kohtaro K. High-yield production of cis, cis-muconic acid from catechol in aqueous solution by Biocatalyst[J]. Chemistry Letters, 2011, 40(4): 381-383. DOI:10.1246/cl.2011.381 |
| [43] |
Guzik U, Hupert-Kocurek K, Sitnik M, et al. High activity catechol 1, 2-dioxygenase from Stenotrophomonas maltophilia strain KB2 as a useful tool in cis, cis-muconic acid production[J]. Antonie van Leeuwenhoek, 2013, 103(6): 1297-1307. DOI:10.1007/s10482-013-9910-8 |
 2019, Vol. 46
2019, Vol. 46