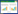扩展功能
文章信息
- 张彤, 张杰, 钟瑾
- ZHANG Tong, ZHANG Jie, ZHONG Jin
- 前导肽切割位点对Ⅱ类羊毛硫细菌素切割酶活性的影响
- Effects of cleavage sites of leader peptides on the activities of class Ⅱ lantibiotic peptidases
- 微生物学通报, 2019, 46(9): 2198-2206
- Microbiology China, 2019, 46(9): 2198-2206
- DOI: 10.13344/j.microbiol.china.190120
-
文章历史
- 收稿日期: 2019-02-21
- 接受日期: 2019-03-28
- 网络首发日期: 2019-05-15
2. 中国科学院大学 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
羊毛硫细菌素(Lantibiotics)是一类主要由革兰氏阳性菌的核糖体合成,经过翻译后修饰产生的天然产物,它具有丰富的结构多样性和抑菌活性多样性[1-2]。羊毛硫细菌素最显著的特点是分子内含有形成硫醚环的特殊氨基酸——羊毛硫氨酸(Lan)和β-甲基羊毛硫氨酸(MeLan)[3],它们赋予了羊毛硫细菌素一系列优良的性质,例如热稳定性、蛋白酶抗性、耐酸性以及抑制耐药性菌株(如MRSA和VRE等)的特性[4]。近年来,抗生素的滥用导致细菌耐药性增强,而新型抗感染类药物的缺乏已经对全球公共卫生安全造成了严重的影响。羊毛硫细菌素因其独特的抑菌机制[5-7]而不易引起细菌产生耐药性,并且它作为一种小肽,容易被消化吸收而不产生残留,不会对人体产生副作用[8],因此对羊毛硫细菌素的研究和应用已引起了科研者的广泛关注。
羊毛硫细菌素的生物合成是一个相对复杂的过程,需要生物合成基因簇上的多个基因共同发挥作用,以基因座名称lan统一命名该基因簇。根据修饰酶LanM的不同,羊毛硫细菌素可分为4类(Ⅰ、Ⅱ、Ⅲ、Ⅳ类) [9],其中Ⅱ类Lantibiotics由双功能酶LanM完成修饰且其成员众多,具有较优良的抑菌活性[10]。Ⅱ类Lantibiotics基因簇包括前体基因lanA (经过翻译后修饰的前体为LanAm)、修饰酶基因lanM和转运基因lanT等。但是某一个特定Lantibiotic有其专属的命名,例如Lacticin 481生物合成相关基因采用lac命名,而Bovicin HJ50生物合成相关基因采用bov命名。Ⅱ类Lantibiotics生物合成过程如下:首先在核糖体中合成前体蛋白LanA (包括前导肽和核心肽两部分);LanA经修饰酶LanM的作用,形成修饰后的前体蛋白(LanAm),然后LanAm被转运蛋白LanT转运到胞外并切除前导肽,形成具有活性的羊毛硫细菌素[11]。
转运蛋白LanT在Ⅱ类羊毛硫细菌素的生物合成过程中发挥着重要的功能,它属于ABC转运子成熟与分泌蛋白(ABC transporter maturation and secretion,AMS)家族,由三部分组成:N端的肽酶结构域PEP (Peptidase domain)、C端的核苷酸结合结构域NBD (Nucleotide-binding domain)和中间的跨膜结构域TMD (Transmembrane domain) [12]。其中N端肽酶结构域的主要作用是对底物前导肽C末端的GG或GA位点进行切割,从而切除前导肽。研究表明LanT N端150个氨基酸(LanT150)可能具备体外切割前导肽的功能,而目前仅有LctT150 (Lacticin 481的切割酶)和NukT150 (Nukacin ISK-1的切割酶)被证明具备该功能[13-14],其中Furgerson Ihnken等发现前导肽中的GG位点对LctT150肽酶水解活性很重要[13]。
本工作是对两个Ⅱ类羊毛硫细菌素Bovicin HJ50和Salivaricin B进行研究,它们分别对巨大芽孢杆菌(Bacillus megatherium)和酿脓链球菌(Streptococcus pyogenes)具有很好的抑制作用[15-16]。Bovicin HJ50是由牛链球菌(S. bovis) HJ50产生的,其前体的前导肽是由切割酶BovT150切掉,而Salivaricin B是由唾液链球菌(S. salivarius) K12产生的,由切割酶SboT150将其前体的前导肽切掉。从分子结构可以看出(图 1A),Bovicin HJ50和Salivaricin B分别具有典型和非典型(含有一个二硫键形成的环结构) II类Lantibiotics的特征,然而它们前导肽切割位点对切割效率影响的研究还未见报道。本研究以这两种Lantibiotics为研究对象,考察不同的前导肽切割位点分别对切割酶BovT150和SboT150活性的影响,为开发高效的羊毛硫细菌素切割酶奠定理论基础。
|
| 图 1 Ⅱ类羊毛硫细菌素前体序列比对(A)及其一级结构(B) Figure 1 Sequence alignment of class Ⅱ lantibiotics precursors (A) and primary structure of the lantibiotics (B) 注:红色:序列相似性为75%;蓝色:序列相似性为100%;−1:前导肽C端的最后一位氨基酸;1:核心肽N端的第一位氨基酸;箭头:切割酶切割前导肽的位置. Note: Red: Sequence similarity level is 75%; Blue: Sequence similarity level is 100%; −1: The last amino acid of C-terminal region of the leader peptide; 1: The first amino acid of N-terminal region of the core peptide; Arrow: The position of leader peptide cleaved by peptidase. |
|
|
E. coli DH5α、BL21(DE3)和Rosetta(DE3)感受态细胞购于北京博迈德基因公司。质粒pET28a-bovAM (Bovicin HJ50的前体蛋白BovAm表达载体)、pET28a-bovT150、pETDuet-sboAM (Salivaricin B的前体蛋白SboAm表达载体)和pET28a-sboT150由本实验室保存。
1.1.2 主要试剂和仪器质粒提取试剂盒、DNA琼脂糖凝胶回收试剂盒、PCR产物回收试剂盒和DNA Marker均购于博迈德公司;DpnⅠ限制性内切酶购于Thermo Scientific公司;IPTG购于VWR Chemicals公司;Protein marker购于Genstar公司;High-Fidelity Master Mix和DNA Loading Buffer购于北京擎科公司;一步法快速克隆试剂盒购于翊圣公司;Ni-NTA His结合树脂购于GE公司;色谱纯乙腈购于MREDA公司;色谱纯三氟乙酸(TFA)购于DIKMA公司。
PCR仪购于Bio-Gener公司;DNA琼脂糖凝胶电泳仪购于君意公司;蛋白电泳仪购于Bio-Rad公司;凝胶成像系统购于天能公司;高效液相色谱(HPLC)购于岛津公司;5800蛋白质组分析器质谱仪购于Applied Biosystems公司。引物由北京天一辉远公司合成,测序由北京睿博兴科公司完成。
1.1.3 培养基培养E. coli使用LB培养基[17],需要时向其中加入终浓度50 μg/mL卡那霉素、100 μg/mL氨苄青霉素或20 μg/mL氯霉素。
培养指示菌Micrococcus luteus NCIB 8166使用S1培养基[18]。
1.2 方法 1.2.1 前体序列比对分析将Ⅱ类羊毛硫细菌素的前体LctA、NukA、MutA、SboA和BovA氨基酸序列以FASTA格式导入ClustalW网站(https://www.genome.jp/tools-bin/clustalw)进行序列比对,随后用DNAMAN软件对结果进行标注分析。
1.2.2 前导肽序列突变载体构建根据Chiu等所述的定点突变方法(Site-directed Ligase-Independent Mutagenesis,SLIM)[19]设计引物(表 1),将Bovicin HJ50的前导肽切割位点氨基酸残基由Gly(ggt)突变成Ala(gct),突变体前体基因命名为bovA(G-1A),而将Salivaricin B的Ala(gct)突变成Gly(ggt),突变体前体基因命名为sboA(A-1G)。以pET28a-bovAM为模板进行PCR扩增,反应体系(50 μL):2×High-Fidelity Master Mix 25 μL,引物BovG-1A-L1和BovG-1A-S1 (10 μmol/L)各2 μL,质粒pET28a-bovAM (150 ng/μL) 1 μL,ddH2O 20 μL。PCR反应条件:98 ℃ 30 s;98 ℃ 10 s,56 ℃ 20 s,72 ℃ 2 min,30个循环;72 ℃ 10 min;16 ℃保存。PCR回收产物经DpnⅠ酶切后进行退火反应,然后将产物转化至E. coli DH5α感受态细胞,待克隆长出后挑取转化子培养,提取质粒测序鉴定,得到突变载体pET28a-bovAM(G-1A)。按照同样的方法构建突变载体pETDuet-sboAM(A-1G)。
| Primers name | Primers sequence (5′→3′) | Size (bp) | Purpose |
| BovG-1A-L1 | GAAATGTTAATTGGTGCTGCAGATCGTGGATGG | 8 085 | Constructing pET28a-bovAM(G-1A) |
| BovG-1A-S1 | TAATTCTTGGTCACTCACAGTCTC | ||
| BovG-1A-L2 | CCATCCACGATCTGCAGCACCAATTAACATTTC | ||
| BovG-1A-S2 | ATTAAGACTTTAACAAAAGATTGTC | ||
| SboA-1G-L1 | GATTACTCCACCACCACCACCAAGAACGTTATCAAG | 8 324 | Constructing pETDuet-sboAM(A-1G) |
| SboA-1G-S1 | CAAACCATTTCACACGAATG | ||
| SboA-1G-L2 | CTTGATAACGTTCTTGGTGGTGGTGGTGGAGTAATC | ||
| SboA-1G-S2 | TTCTTCAAGAGTCAATTCTTG |
将质粒pET28a-bovAM、pET28a-bovAM(G-1A)和pET28a-bovT150转化至E. coli BL21(DE3)感受态细胞,pETDuet-sboAM、pETDuet-sboAM(A-1G)和pET28a-sboT150转化至E. coli Rosetta(DE3)感受态细胞。E. coli BL21(DE3)和Rosetta(DE3)于37 ℃、220 r/min培养,待菌液浓度OD600为0.6−0.8 (对数生长期)时,加入IPTG至终浓度为0.5 mmol/L,18 ℃、180 r/min振荡培养20 h。4 ℃、5 000 r/min离心30 min收集菌体,重悬菌体以400 W功率超声破碎25 min,切割酶蛋白BovT150和SboT150利用钴柱纯化,其余前体蛋白利用镍柱纯化,具体操作见说明书。
1.2.4 体外酶切反应两种切割酶BovT150和SboT150经超滤浓缩后,交换至Assay buffer (50 mmol/L Na2HPO4,50 mmol/L Na2SO4,pH 7.4)。根据Tricine-SDS-PAGE胶上对应的蛋白条带宽度估算底物BovAm、SboAm及其突变体和切割酶BovT150、SboT150的浓度,并将底物和切割酶分别稀释成相同浓度。按照终浓度为BovT150≈2×BovAm建立体外酶切反应体系。体系总体积为400 μL,25 ℃分别反应3 h和8 h后取样用于后续分析。
1.2.5 HPLC分析酶切产物反应时间为3 h的反应液以13 000 r/min离心5 min后,0.22 μm水系滤膜过滤,得到的样品利用HPLC进行检测。使用岛津公司的InertSustain系列C18分析柱,采用梯度洗脱的方法,流速为1.0 mL/min。流动相A相为含0.1% TFA的超纯水,B相为含0.1% TFA的色谱纯乙腈。该C18柱的平衡浓度为5% B,洗脱浓度范围为30%−70% B,25 min。
1.2.6 抑菌活性测定反应液的抑菌活性采用琼脂打孔扩散法测定。挑取M. luteus NCIB 8166种子平板的菌,悬浮混匀于5 mL生理盐水,加入融化的S1固体培养基混匀,倒入无菌培养皿中。待培养基凝固后,用直径7 mm打孔器在指示菌平板上打孔,每孔加入50 μL样品,30 ℃培养24−36 h观察抑菌圈。
1.2.7 质谱鉴定取200 μL反应8 h的酶切产物由Zip-tip (Millipore)除盐,最后溶于60%的乙腈水溶液中制备成样品。质谱采用MALDI-TOF MS 5800蛋白质组分析器进行样品分子量检测。基质CHCA水溶液(5 mg CHCA溶解于1 mL 50%乙腈水溶液,含0.1% TFA)上样量为1 μL,样品上样量为1.2 μL。
2 结果与分析 2.1 前体序列分析为了比较前导肽序列的保守性,利用ClustalW对5种Ⅱ类的羊毛硫细菌素前体(LctA、NukA、MutA、SboA和BovA,前3种的前体序列在Ⅱ类羊毛硫细菌素中具有代表性)氨基酸序列进行比对(图 1B),从图 1B可以看出,这5种羊毛硫细菌素的前导肽在−3至−8区间的序列相似性较高,由于该区域被认为与Ⅱ类羊毛硫细菌素的成熟加工密切相关,推测BovT150和SboT150具有与其他Ⅱ类切割酶相似的切割机制。BovA和MutA的前导肽切割位点是GG,而SboA等其他3种均为GA,可以看出切割位点残基的保守性较高,因此考察GG和GA位点的变换对切割酶活性的影响。
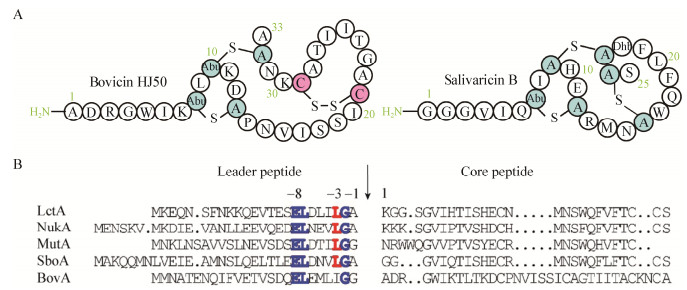
2.2 异源表达前体蛋白和切割酶蛋白运用本实验室建立的羊毛硫细菌素半体外生物合成系统(Semi-in vitro biosynthesis,SIVB)[20],在E. coli中共表达前体基因lanA和修饰酶基因lanM,获得修饰后的前体蛋白LanAm,将LanAm与单独表达获得的切割酶LanT150在体外进行酶切反应,从而得到有活性的羊毛硫细菌素,该系统结合定点突变技术可方便快捷地获得突变体前体蛋白。本研究针对两个羊毛硫细菌素前导肽切割位点的氨基酸残基进行突变,将bovA前导肽−1位氨基酸Gly突变为Ala,命名为bovA(G-1A);将sboA前导肽−1位氨基酸Ala突变Gly,命名为sboA(A-1G)。在E. coli BL21(DE3)中将野生型前体基因bovA和突变型前体基因bovA(G-1A)分别与bovM共表达,得到经过修饰的野生型前体蛋白His6-BovAm和突变型前体蛋白His6-BovAm(G-1A)。计算得到它们的分子量分别为8 277 Da和8 291 Da,经Tricine-SDS-PAGE检测可以看到清晰的条带(图 2A)。由于E. coli BL21(DE3)不含稀有密码子的tRNA,因此在E. coli Rosetta(DE3)中运用同样的共表达方法获得含稀有密码子的野生型前体His6-SboAm (分子量大小为7 740 Da)和突变型前体His6-SboAm(A-1G) (分子量大小为7 726 Da) (图 2A)。另外两种切割酶BovT150-His6和SboT150-His6的大小分别为18.6 kD和18.5 kD (图 2B)。
|
| 图 2 四种前体蛋白(A)和两种切割酶(B)的Tricine-SDS-PAGE电泳图 Figure 2 Tricine-SDS-PAGE of four precursor peptides (A) and two peptidases (B) 注:A:1:蛋白Marker;2:纯化的His6-BovAm蛋白;3:纯化的His6-BovAm(G-1A)蛋白;4:纯化的His6-SboAm蛋白;5:纯化的His6-SboAm(A-1G)蛋白. B:1:蛋白Marker;2:纯化的BovT150-His6蛋白;3:纯化的SboT150-His6蛋白. Note: A: 1: Protein marker; 2: Purified His6-BovAm; 3: Purified His6-BovAm(G-1A); 4: Purified His6-SboAm; 5: Purified His6-SboAm(A-1G). B: 1: Protein marker; 2: Purified BovT150-His6; 3: Purified SboT150-His6. |
|
|
利用切割酶在体外酶切修饰后的前体可以得到成熟肽。由于成熟肽和前体的疏水性不同,经过C18反相柱分离后会在不同保留时间出现色谱峰。以His6-BovAm为底物,分别利用BovT150和SboT150进行酶切。从图 3A中可以看到,BovT150酶切对应前体BovAm (色谱保留时间为19.5 min),得到了成熟肽Bovicin HJ50的色谱峰(保留时间为17.5 min)。然而SboT150酶切BovAm后未出现成熟肽的色谱峰,表明SboT150不能切掉BovAm的GG位点。当将Bovicin HJ50前导肽的切割位点突变为GA时,SboT150仍然不能切掉Bovicin HJ50的前导肽。此时虽然BovT150也能切割GA位点,但产生的成熟肽浓度明显低于GG位点切割产生的,表明BovT150的切割效率降低(图 3B和表 2)。

|
| 图 3 酶切产物的HPLC谱图和抑菌活性 Figure 3 The HPLC spectra and antimicrobial activities of the enzymatic products 注:A:BovAm;B:BovAm(G-1A);C:SboAm;D:SboAm(A-1G). 1:反应Buffer;2:SboT150;3:BovT150;箭头:成熟肽吸收峰. Note: A: BovAm; B: BovAm(G-1A); C: SboAm; D: SboAm(A-1G). 1: Assay buffer; 2: SboT150; 3: BovT150; Arrow: The peak of mature peptides. |
|
|
| 前体 Precursor |
切割酶 Peptidase |
峰面积 Peak area |
| BovAm | BovT150 | 1 199 422 |
| BovAm | SboT150 | 0 |
| BovAm(G-1A) | BovT150 | 125 946 |
| BovAm(G-1A) | SboT150 | 0 |
| SboAm | BovT150 | 258 560 |
| SboAm | SboT150 | 384 887 |
| SboAm(A-1G) | BovT150 | 677 391 |
| SboAm(A-1G) | SboT150 | 207 476 |
当以His6-SboAm为底物时,SboT150酶切得到了成熟肽Salivaricin B (其前体和成熟肽的保留时间分别为24.5 min和21 min,图 3C)。此时BovT150竟也能切割SboAm的前导肽,得到成熟肽的色谱峰。将Salivaricin B前导肽切割位点由GA突变为GG后,SboT150和BovT150都能将其切掉,但从成熟肽浓度可以看出,SboT150的相对切割效率降低,而BovT150的相对效率升高(图 3D和表 2)。
2.4 酶切产物的抑菌活性检测由于前体的前导肽被切除后才能显示出抑菌活性,通过对酶切产物抑菌活性的分析也能考察BovT150和SboT150对野生型和突变型前体的酶切活性。从图 3右侧的抑菌活性图可以看出,出现成熟肽色谱峰(图 3A和3B的保留时间为17.5 min,图 3C和3D的保留时间为21 min)的酶切产物均显示抑菌活性,未在对应时间出现吸收峰的酶切产物均无抑菌活性。以上结果再次表明,BovT150可以切割BovAm和SboAm前导肽的GG和GA位点,而SboT150仅能切割SboAm的GG和GA位点。
2.5 质谱鉴定酶切产物为了验证切割酶对前体切割后是否产生了成熟肽以及切割位点是否和预期一致,对上述酶切产物进行了质谱分析。从Bovicin HJ50和Salivaricin B前导肽的–1位氨基酸残基进行切割,产生的成熟肽分子量分别为3 428 Da和2 732 Da[16, 21]。质谱结果显示,当以BovAm作为底物时,BovT150酶切后的反应产物检测到了分子量为3 428.0 Da的质谱峰(图 4A),而在SboT150酶切产物中没有检测到(图 4B),表明BovT150能切割BovAm的GG位点,而SboT150不能。当以BovAm(G-1A)作为底物时,经SboT150酶切后未检测到成熟肽的分子量,表明SboT150不能切割底物的GA位点;该底物经BovT150酶切也未出现相应的分子量,推测可能是由于成熟肽的产量较低(从HPLC成熟肽的色谱峰可以看出)。当分别以SboAm和SboAm(A-1G)作为底物时,BovT150和SboT150的酶切产物都检测到了分子量为2 731.6 Da的质谱峰(图 4C和4D),表明BovT150和SboT150均能切割SboAm的GG和GA位点。详细的酶切产物质谱检测结果见表 3。
|
| 图 4 酶切产物的质谱图 Figure 4 Mass spectra of the enzymatic products 注:A:BovT150切割BovAm;B:SboT150切割BovAm;C:BovT150切割SboAm;D:SboT150切割SboAm. Note: A: BovAm cleaved by BovT150; B: BovAm cleaved by SboT150; C: SboAm cleaved by BovT150; D: SboAm cleaved by SboT150. |
|
|
| 前体 Precursor |
切割酶 Peptidase |
切割位点 Cleavage site |
分子量 Molecular weights (Da) |
| BovAm | BovT150 | GG | 3 428.0 |
| BovAm | SboT150 | GG | − |
| BovAm(G-1A) | BovT150 | GA | − |
| BovAm(G-1A) | SboT150 | GA | − |
| SboAm | BovT150 | GA | 3 731.6 |
| SboAm | SboT150 | GA | 2 731.6 |
| SboAm(A-1G) | BovT150 | GG | 2 731.6 |
| SboAm(A-1G) | SboT150 | GG | 2 731.6 |
| Note: −: No product was detected. | |||
羊毛硫细菌素的生物合成是一个十分复杂却又精准的过程,而前导肽在这个过程中发挥的作用一直是研究的热点。一般认为,前导肽可以保护合成的前体不被产生菌的蛋白酶降解,且保护产生菌不被自身产生的物质杀伤(一般未去除前导肽的前体不具有生物活性)。羊毛硫细菌素的前导肽中还存在修饰酶的识别基序和转运蛋白的识别序列及切割位点,以指导其进行翻译后修饰和成熟过程[22]。目前Ⅱ类羊毛硫细菌素中,有关前导肽切割位点的报道较少,值得深入研究。例如对Mutacin II的研究发现,前导肽G-1A的突变引起Mutacin II成熟肽的生物合成受阻,推测是由于切割位点的突变导致前体不能被切割酶切掉[23]。将Lacticin 481的前导肽切割位点Ala-1突变为残基Asp、Lys或Ile,完全破坏了His10-LctT150的蛋白酶活性,而A-1G突变体前体却能够被His10-LctT150切割,并且与野生型的成熟肽产量相差不大[13]。在本研究中,Bovicin HJ50和Salivaricin B的分子结构分别是非典型和典型II类羊毛硫细菌素的代表,对其前导肽切割位点的研究有助于全面了解该类前导肽的切割机制。虽然两者前导肽序列相似度很低,但都具有保守的−3至−8区,以及切割位点GG或GA (图 1A)。结合液相色谱、抑菌活性以及质谱分析结果发现,BovT150对前导肽切割位点为Gly的切割效率较高,而SboT150对前导肽切割位点为Ala的切割效率较高。这表明BovT150和SboT150对酶切位点的选择性都不同于LctT150 (其对Gly和Ala的作用效率几乎一致)。
BovT150和SboT150对不同切割位点的选择性可能与其空间结构或底物序列相关。由于迄今II类羊毛硫细菌素切割酶的空间结构尚未得到解析,从与其属于同一家族(半胱氨酸蛋白酶)的ComA的PEP空间结构可以得到一些信息[24]。该PEP的晶体结构数据显示,其结构中存在一个底物通道,适合底物前导肽的双甘氨酸位点(GG)进入。当将GG位点突变为GA后,该PEP的催化效率下降约800倍,推测是由于突变后残基较大,使GA位点较难进入底物通道[24]。本研究中发现BovT150对切割位点GG的催化活性明显高于GA,推测也可能是与BovT150的底物结合区域有关。然而SboT150对切割位点GA的催化活性高于GG,这与底物通道理论的预测不一致,推测除氨基酸残基的大小外,该位点氨基酸残基的性质(例如疏水性)或空间构象也影响切割酶的效率。这些推论还有待进一步的研究确认。
目前针对羊毛硫细菌素的PEP研究不多,主要集中于PEP对同源前导肽的切割活性研究。本研究中BovT150能够切除异源羊毛硫细菌素前体SboAm及其突变体SboAm(A-1G)的前导肽,而SboT150对BovAm及其突变体都无酶切活性,表明BovT150蛋白具有一定的底物宽泛性,有潜力作为工具酶去进行外源蛋白的特异性切割,这将有助于对一些生物合成基因簇中缺乏切割酶的羊毛硫细菌素进行体外生物合成。这些对羊毛硫细菌素切割酶与切割位点相互作用的研究将极大促进羊毛硫细菌素的开发与应用。
| [1] |
Jung G. Lantibiotics-ribosomally synthesized biologically active polypeptides containing sulfide bridges and α, β-didehydroamino acids[J]. Angewandte Chemie-International Edition, 1991, 30(9): 1051-1068. DOI:10.1002/anie.199110513 |
| [2] |
Chatterjee C, Paul M, Xie LL, et al. Biosynthesis and mode of action of lantibiotics[J]. Chemical Reviews, 2005, 105(2): 633-684. DOI:10.1021/cr030105v |
| [3] |
Willey JM, van der Donk WA. Lantibiotics: peptides of diverse structure and function[J]. Annual Review of Microbiology, 2007, 61: 477-501. DOI:10.1146/annurev.micro.61.080706.093501 |
| [4] |
Piper C, Cotter PD, Ross RP, et al. Discovery of medically significant lantibiotics[J]. Current Drug Discovery Technologies, 2009, 6(1): 1-18. |
| [5] |
Hasper HE, de Kruijff B, Breukink E. Assembly and stability of nisin-lipid II pores[J]. Biochemistry, 2004, 43(36): 11567-11575. DOI:10.1021/bi049476b |
| [6] |
Hasper HE, Kramer NE, Smith JL, et al. An alternative bactericidal mechanism of action for lantibiotic peptides that target lipid II[J]. Science, 2006, 313(5793): 1636-1637. DOI:10.1126/science.1129818 |
| [7] |
Mcauliffe O, Ross RP, Hill C. Lantibiotics: structure, biosynthesis and mode of action[J]. FEMS Microbiology Reviews, 2001, 25(3): 285-308. DOI:10.1111/j.1574-6976.2001.tb00579.x |
| [8] |
Dischinger J, Chipalu SB, Bierbaum G. Lantibiotics: promising candidates for future applications in health care[J]. International Journal of Medical Microbiology, 2014, 304(1): 51-62. DOI:10.1016/j.ijmm.2013.09.003 |
| [9] |
Zhang Q, Yu Y, Vélasquez JE, et al. Evolution of lanthipeptide synthetases[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(45): 18361-18366. DOI:10.1073/pnas.1210393109 |
| [10] |
Knerr PJ, van der Donk WA. Discovery, biosynthesis, and engineering of lantipeptides[J]. Annual Review of Biochemistry, 2012, 81: 479-505. DOI:10.1146/annurev-biochem-060110-113521 |
| [11] |
Xie LL. Lacticin 481: in vitro reconstitution of lantibiotic synthetase activity[J]. Science, 2004, 303(5658): 679-681. DOI:10.1126/science.1092600 |
| [12] |
Havarstein LS, Diep DB, Nes IF. A family of bacteriocin ABC transporters carry out proteolytic processing of their substrates concomitant with export[J]. Molecular Microbiology, 1995, 16(2): 229-240. DOI:10.1111/j.1365-2958.1995.tb02295.x |
| [13] |
Furgerson Ihnken LA, Chatterjee C, van der Donk WA. In vitro reconstitution and substrate specificity of a lantibiotic protease[J]. Biochemistry, 2008, 47(28): 7352-7363. DOI:10.1021/bi800278n |
| [14] |
Nishie M, Shioya K, Nagao JI, et al. ATP-dependent leader peptide cleavage by NukT, a bifunctional ABC transporter, during lantibiotic biosynthesis[J]. Journal of Bioscience and Bioengineering, 2009, 108(6): 460-464. DOI:10.1016/j.jbiosc.2009.06.002 |
| [15] |
Xiao HJ, Chen XZ, Chen ML, et al. Bovicin HJ50, a novel lantibiotic produced by Streptococcus bovis HJ50[J]. Microbiology, 2004, 150(1): 103-108. DOI:10.1099/mic.0.26437-0 |
| [16] |
Barbour A, Tagg J, Abou-Zied OK, et al. New insights into the mode of action of the lantibiotic salivaricin B[J]. Scientific Reports, 2016(6): 31749. |
| [17] |
Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual[M]. 3rd edition. New York: Cold Spring Harbor, 2001: 1595.
|
| [18] |
Ma HC, Gao Y, Zhao FY, et al. Individual catalytic activity of two functional domains of bovicin HJ50 synthase BovM[J]. Acta Microbiologica Sinica, 2015, 55(1): 50-58. (in Chinese) 马宏初, 高涌, 赵方圆, 等. 羊毛硫细菌素bovicin HJ50修饰酶BovM双功能域单独催化活性鉴定[J]. 微生物学报, 2015, 55(1): 50-58. |
| [19] |
Chiu J, March PE, Lee R, et al. Site-directed, ligase-independent mutagenesis (SLIM): a single-tube methodology approaching 100% efficiency in 4 h[J]. Nucleic Acids Research, 2004, 32(21): e174. DOI:10.1093/nar/gnh172 |
| [20] |
Lin YH, Teng KL, Huan LD, et al. Dissection of the bridging pattern of bovicin HJ50, a lantibiotic containing a characteristic disulfide bridge[J]. Microbiological Research, 2011, 166(3): 146-154. DOI:10.1016/j.micres.2010.05.001 |
| [21] |
Zhang J, Feng YG, Teng KL, et al. Type AII lantibiotic bovicin HJ50 with a rare disulfide bond: structure, structure-activity relationships and mode of action[J]. Biochemical Journal, 2014, 461(3): 497-508. DOI:10.1042/BJ20131524 |
| [22] |
Oman TJ, van der Donk WA. Follow the leader: the use of leader peptides to guide natural product biosynthesis[J]. Nature Chemical Biology, 2010, 6(1): 9-18. DOI:10.1038/nchembio.286 |
| [23] |
Chen P, Qi FX, Novak J, et al. Effect of amino acid substitutions in conserved residues in the leader peptide on biosynthesis of the lantibiotic mutacin II[J]. FEMS Microbiology Letters, 2001, 195(2): 139-144. DOI:10.1111/j.1574-6968.2001.tb10511.x |
| [24] |
Ishii S, Yano T, Ebihara A, et al. Crystal structure of the peptidase domain of Streptococcus ComA, a bifunctional ATP-binding cassette transporter involved in the quorum-sensing pathway[J]. Journal of Biological Chemistry, 2010, 285(14): 10777-10785. DOI:10.1074/jbc.M109.093781 |
 2019, Vol. 46
2019, Vol. 46