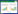扩展功能
文章信息
- 王晶, 高娜, 刘双元, 戴景程, 刘亚琦, 邱东茹
- WANG Jing, GAO Na, LIU Shuang-Yuan, DAI Jing-Cheng, LIU Ya-Qi, QIU Dong-Ru
- 活性污泥微生物解壳聚糖松江菌中菌胶团形成相关大型基因簇的鉴定和分析
- Identification and analysis of a large gene cluster involved in floc formation of activated sludge bacterium Mitsuaria chitosanitabida
- 微生物学通报, 2019, 46(8): 1946-1953
- Microbiology China, 2019, 46(8): 1946-1953
- DOI: 10.13344/j.microbiol.china.190329
-
文章历史
- 收稿日期: 2019-04-15
- 接受日期: 2019-06-11
- 网络首发日期: 2019-06-13
2. 中国科学院大学 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
活性污泥法这一百年工艺是市政污水和工业废水处理的主流技术,活性污泥菌胶团形成是该技术成功的关键,可以实现泥水重力分离和污泥回用,工艺简单、适应性强。活性污泥微生物所分泌的胶质状胞外多聚物(Extracellular polymeric substance,EPS)是污泥菌胶团形成的“黏合剂”,这种由胞外多糖为主组成的高吸水性、难降解的高分子物质又是造成剩余污泥含水量高和难以处置利用的主要原因。研究发现市政污水处理厂活性污泥通常以枝状动胶菌(Zoogloea ramigera)等菌胶团形成菌为优势种类,而工业废水处理厂污泥中则以陶厄氏菌(Thauera)占优势[1]。近年来基于16S rRNA基因高通量测序的活性污泥宏基因组学分析揭示出极高的生物多样性,已经约有700余属的微生物在活性污泥中存在。我们对活性污泥中的菌胶团形成菌进行了分离、纯化和鉴定,并对微生物菌胶团形成机制进行了研究[2-4]。除了动胶菌和陶厄氏菌,我们还分离纯化了一批其他菌胶团形成菌,包括从西安北石桥污水处理厂活性污泥中分离到的解壳聚糖松江菌(Mitsuaria chitosanitabida) XHY-A6,该菌株是一种淡黄色的菌胶团形成菌[4]。
解壳聚糖松江菌模式株3001 (IAM 14711)最早在1998年从日本岛根县松江市的土壤中被分离纯化并鉴定为新属新种,属名来自地名,种加词是根据其降解壳聚糖(Chitosan)的特性而来[5-6]。该菌株可以分泌胞外甲壳素酶[5]。日本学者从该菌中克隆和鉴定了壳聚糖酶A (Chitosanase A,ChoA)基因并对该酶的酶学性质和催化活性相关氨基酸残基进行研究和解析[7-8]。还通过定向突变改善该酶的分泌效率和热稳定性[9]。我国学者对某些氨基酸残基的重要性也进行了分析并在巴氏毕赤酵母中进行异源表达[10]。该酶的晶体结构也已经解析出来,进一步揭示了降解壳聚糖的机制[11]。我们还从武汉二郎庙污水处理厂(处于武昌中心城区)、汤逊湖污水处理厂(城郊)和深圳南山污水处理厂活性污泥中分离纯化出松江菌,说明该菌是我国市政污水处理厂活性污泥中的常见种类。此外,一些具有生物防治作用的松江菌属根际细菌和植物内生菌也陆续被分离纯化,其抗真菌活性和植物保护作用也被认为与其几丁质和壳聚糖降解活性有关,可以降解病原真菌细胞壁中的几丁质[12-13]。松江菌也是农作物根际常见的几丁质降解菌和植物内生菌[14-15]。松江菌在湖泊中型围隔系统中也因特殊浮游植物群体的存在而得到富集[16]。最近我国学者还从豆科植物刺槐(Robinia pseudoacacia L.)的根瘤中分离纯化和命名了一个植物内生菌新种——根瘤松江菌(Mitsuaria noduli)[17]。在处理养猪场废水的膜生物反应器(Membrane Bio-Reactor,MBR)中,在稳定条件下适应后的污泥微生物组中发现降解多糖的解壳聚糖松江菌和降解蛋白质的马赛莱朗河菌(Reyranella massiliensis)[18]。在我国西南地区的传统淡水养殖池塘底泥中也检测出解壳聚糖松江菌[19]。添加稻谷壳的填充床硫酸盐还原生物反应器在运行870多天处理酸性矿井废水的效果变差,宏基因组分析结果表明因为底物耗尽填充床下层硫酸盐还原菌减少,而反应器上层好养微生物以Alishewanella和松江菌相关微生物占优势[20]。这些结果说明松江菌极有可能具有较广的应用价值,而且其形成菌胶团的能力可以在相关工艺技术加以利用。为了进一步揭示活性污泥微生物菌胶团形成机制并与在喜树脂动胶菌(Zoogloea resiniphila)中所取得的研究结果相互印证,我们结合分子遗传学和基因组学分析手段对松江菌胞外多聚物合成和菌胶团形成机制进行了研究。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒本实验所使用的菌株和质粒见表 1。
| 菌株/质粒 Strains/Plasmids |
类型 Description |
引用或来源 Source |
| Escherichia coli WM3064 |
细胞壁营养缺陷型 Cell wall defective strain |
Provided by W. Metcalf |
| Mitsuaria chitosanitabida XHY-A6 |
活性污泥细菌,能够形成菌胶团 Activated sludge bacterium, floc formation |
This study |
| Ymc7 | 转座子插入破坏gt3基因Mariner transposon insertion in gt3 gene | This study |
| Ymc6 | 转座子插入破坏wzz基因Mariner transposon insertion in wzz gene | This study |
| Ymc10 | 转座子插入破坏wzz基因Mariner transposon insertion in wzz gene | This study |
| pBBR1-MCS-5 | 广泛宿主范围克隆载体Broad-host-range cloning vector | [21] |
| pminiHmarRB1 | pMyk6K的衍生产物含有R6Kγ复制起点,卡那霉素抗性和转座酶原件 A derivative of pMyk6K, with oriT-Plac, inverted repeats (IR), replication (R6K) and kanamycin (Km) resistance gene |
[22] |
解壳聚糖松江菌XHY-A6野生型及突变株使用R2A液体和固体培养基[(g/L):胰蛋白胨0.5,酵母提取液0.5,淀粉0.5,酶水解酪蛋白0.5,葡糖糖0.5,丙酮酸钠0.3,磷酸氢二钾0.3,硫酸镁0.024],培养温度为28 ℃。大肠杆菌使用LB液体或固体培养基,培养温度为37 ℃。培养所需抗生素浓度:卡那霉素50 μg/mL,庆大霉素15 μg/mL,E. coli WM3064培养时需加入终浓度50 μg/mL的二氨基庚二酸。
1.1.3 主要试剂和仪器质粒提取试剂盒、琼脂糖凝胶DNA回收试剂盒、基因组提取试剂盒,Omega公司;限制性内切酶(Kpn Ⅰ,Hind Ⅲ),高保真酶PrimeSTAR Max Premix及抗生素(卡那霉素,庆大霉素),TaKaRa公司;MonCloneTM Hi-Fusion Cloning Mix,莫纳生物科技公司。PCR仪及凝胶成像仪,Bio-Rad公司。
1.1.4 引物设计与合成所有引物均由武汉天一辉远生物科技有限公司合成(表 2)。
| 引物 Primers |
序列 Sequence (5′→3′) |
| LAD1-1 | ACGATGGACTCCAGAGCGGCCGCVNVNNNGGAA |
| LAD1-2 | ACGATGGACTCCAGAGCGGCCGCBNBNNNGGTT |
| LAD1-3 | ACGATGGACTCCAGAGCGGCCGCVVNVNNNCCAA |
| LAD1-4 | ACGATGGACTCCAGAGCGGCCGCBDNBNNNCGGT |
| AC-1 | ACGATGGACTCCAGAG |
| SP-1 | GACCGAGATAGGGTTGAGTGTTGTTCC |
| SP-2 | ATCTGGGAATCATTTGAAGGTTGGTAC |
| Ymc7-F | GGTCGACGGTATCGATAAGCTTCTTCGTCGAGGATGTGCGCA |
| Ymc7-R | GCCGCTCTAGAACTAGTGGATCCGGAGGGCAGCATCAGGAGAG |
| M13(-47) | CGCCAGGGTTTTCCCAGTCACGAC |
| M13(-48) | GAGCGGATAACAATTTCACACAGG |
| Ymc6-F | GGTCGACGGTATCGATAAGCTTACATCCTCATCATTCCCCAG |
| Ymc6-R1 | GCCGCTCTAGAACTAGTGGATCCCGCTTGTTCAATCAGACTG |
| Ymc6-R2 | GCCGCTCTAGAACTAGTGGATCCCTGGTGTAGCCCTGGGAGAC |
将用于接合转移实验的供体菌大肠杆菌WM3064 (携带pMiniHmar RB1)和受体菌菌株XHY-A6培养至一定浓度后,4 ℃、10 000r/min离心5 min收集菌体,用无抗性的培养基洗去培养液中残留抗性,重悬并混合菌体,然后滴至含有二氨基庚二酸的LB固体培养基上于28 ℃培养5-8 h后,用1 mL ddH2O从培养平板上将菌体洗下收集。将收集的菌体按照1:100的比例进行稀释后均匀涂布于含有卡那霉素的R2A固体培养平板上,28 ℃培养箱中培养至长出明显克隆。逐一挑取单克隆,用含有卡那霉素的R2A培养基于28 ℃、220 r/min振荡培养,观察其表型,选择菌胶团形成缺陷的菌株进行后续研究。
1.2.2 转座子插入突变株的鉴定采用TAIL-PCR法即热不对称交错PCR法对上文筛选到的突变株进行鉴定[23],第一轮以突变株的基因组为模板,从随机简并引物LAD1-1、LAD1-2、LAD1-3、LAD1-4中选取2-3个作为上游引物,下游引物为特异引物SP-1,PCR反应体系(20 μL):模板0.5 μL,2×PCR Master Mix 10 μL,20 μmol/L引物各1 μL,灭菌ddH2O 7.5 μL。PCR反应条件:93 ℃ 2 min;95 ℃ 1 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 80 s,11个循环;94 ℃ 30 s;25 ℃ 2 min;72 ℃ 3 min;94 ℃ 20 s,58 ℃ 30 s,72 ℃ 80 s,26个循环;72 ℃ 5 min。第二轮以稀释20倍的第一轮产物为模板,AC-1和SP-2为引物进行PCR扩增,PCR反应体系(20 μL):模板0.5 μL,2×PCR Master Mix 10 μL,20 μmol/L引物各1 μL,灭菌ddH2O 7.5 μL。PCR反应条件:94 ℃ 2 min,65 ℃ 30 s,72 ℃ 40 s,2个循环;94 ℃ 1 min,68 ℃ 30 s,72 ℃ 40 s,94 ℃ 1 min,68 ℃ 30 s,72 ℃ 40 s,94 ℃ 1 min,50 ℃ 30 s,72 ℃ 40 s,14个循环;72 ℃ 5 min。扩增产物经琼脂糖凝胶电泳检测,特异性条带按试剂盒方法进行回收后送公司测序,利用NCBI数据库结合菌株XHY-A6基因组测序结果,确定突变株被破坏的基因。
1.2.3 突变株的遗传互补分析以菌株XHY-A6野生型基因组DNA为模板,PCR扩增目的基因,回收PCR扩增产物后,利用一步克隆试剂盒将目的基因片段连接至经过(Kpn Ⅰ和Hind Ⅲ)双酶切的pBBR-1MCS-5载体上,连接液转化大肠杆菌WM3064,筛选阳性克隆子,提质粒送公司测序,验证互补载体是否构建成功。将构建好的互补载体通过接合转移的方式转入相应突变株中,获得互补菌株,同时将pBBR-1MCS-5空载体转入菌株XHY-A6野生型和突变株中作为对照,通过培养和显微镜观察它们的表型变化。
2 结果与讨论 2.1 菌胶团形成缺陷的转座子插入突变株的筛选与鉴定因为Mariner转座酶可识别宿主DNA上的TA二核苷酸序列来介导转座子随机进行插入,我们利用自杀质粒pMiniHimar RB1构建细菌转座子插入突变株[2-4]。已经成功在松江菌中构建转座子插入突变株库。从中我们初步筛选到3株菌胶团形成缺陷的菌株,经过Tail-PCR鉴定,这3个突变株中转座子插入破坏了参与胞外多糖合成与分泌的两个相关基因,如表 3所示。
| 基因 Genes |
突变株编号 Number of mutants |
转座子插入位点 Transposon insertion site |
| 糖基转移酶基因 Glycosyltransferase 3 (gt3) |
Ymc7 | ACGCCCACTACTACTACCC |
| 多糖链长决定蛋白基因 wzz | Ymc6 | GAACAGTTATTCTCTCAG |
| Polysaccharide chain length determinant |
Ymc10 | AAGAAGAATTACGAAGA |
| 注:具有下划线的TA二核苷酸是转座子插入位点. Note: TA dinucleotide with underline is the transposon insertion site. |
||
大部分细菌胞外多糖、荚膜多糖和脂多糖生物合成的机制非常保守,即先在细胞质中由糖基转移酶将一个活化的单糖先转移到聚异戊二烯脂-十一碳烯醇二磷酸受体分子上,然后由其他糖基转移酶依次将其他活化单糖转移形成寡糖单位。寡糖单位通过翻转酶的催化翻转越过质膜进入周质空间,再由聚合酶Wzy将寡糖单位连成多糖长链,多糖链再通过外膜孔蛋白所形成的孔分泌出胞外,这就是所谓的Wzy依赖途径(Wzy-dependent pathway)。我们初步筛选到的糖基转移酶3可能在细胞质中参与寡糖单位的合成;多糖链长决定蛋白Wzz则可能参与决定由寡糖单位组成的多糖链长度。这两个基因在喜树脂动胶菌MMB株和解叔丁醇水居菌RN12株的胞外多糖合成基因簇中也存在[2-3]。
2.2 糖基转移酶基因的遗传互补分析通过遗传互补分析可进一步确认基因型-表现型之间的相关性,因此我们将糖基转移酶3的基因克隆到穿梭质粒pBBR1MCS-5中,得到pBBR1MCS-5-gt3质粒。通过接合作用将所构建的pBBR1MCS-5-gt3质粒转化Ymc7突变株,同时将pBBR1MCS-5空载体分别转化Ymc7突变株和菌株XHY-A6野生型菌株(图 1)。克隆到穿梭质粒上的糖基转移酶基因gt3可以完全恢复Ymc7突变株的菌胶团形成表型。说明该基因的确参与菌胶团形成。从显微照片上看背景清晰,并无染色后的絮状物质(图 1B),说明几乎没有胞外多糖的合成和分泌。

|
| 图 1 菌胶团形成相关基因gt3的筛选与遗传互补分析 Figure 1 Floc-deficient transposon insertional mutant screening and genetic complementary analysis of the requirement of the gt3 gene for floc formation in Mitsuaria sp. XHY-A6 注:A:菌株XHY-A6野生型(携带pBBR1MCS-5空质粒)液体培养物的显微镜观察,可观察到菌胶团形成(亚甲基美蓝染色);B:Ymc7转座子插入突变株(携带空质粒)的显微镜图片,无菌胶团形成,单个细胞均匀分散分布在培养液中,呈浑浊状态(D中);C:Ymc7互补株(携带克隆有gt3基因的质粒pBBR1MCS-5-gt3)的显微镜图片,菌胶团形成表型恢复;D:振荡后细菌菌胶团(A和C)可悬浮在培养基中(模拟曝气池曝气时形成混合液);突变株培养物(B)则与不能形成菌胶团的假单胞菌和大肠杆菌等细菌的浑浊均匀的培养物相似,单细胞均匀分布;E:培养管静置后,形成菌胶团的动胶菌菌株(A和C)沉淀到培养管底(模拟活性污泥在二次沉淀池进行重力沉淀,可实现固液分离,进而实现污泥回用);突变株(B)依然呈浑浊状态,不能沉淀.标尺:10 μm. Note: A: The floc-forming wild-type strain carrying the empty pBBR1MCS-5 vector under microscope (stained by methylene blue); B: The floc-deficient Ymc7 mutant carrying the empty vector; C: The Ymc7 mutant carrying the complementary pBBR1MCS-5-gt3 construct; D: Agitated cell culture of A, B and C (simulating aeration tank); E: Settled cell culture of A, B and C (simulating settlement tank). The turbid cell culture of the Ymc7 mutant (B) could not settle down and the scattered single cells were observed under microscope. Bar: 10 μm. |
|
|
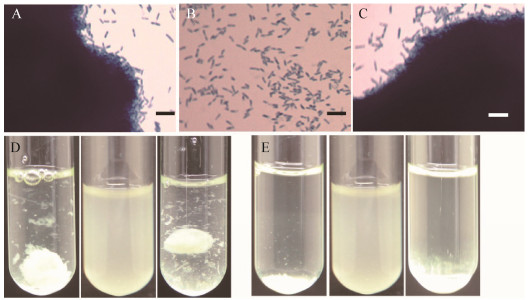
我们将编码多糖链长决定蛋白的基因wzz克隆到穿梭质粒pBBR1MCS-5中,得到pBBR1MCS-5-wzz重组质粒。通过接合作用将所构建的pBBR1MCS- 5-wzz质粒转化Ymc6、Ymc10突变株,发现突变株的表型没有回复至野生型水平。由于转座子插入突变可能会产生极性效应,于是我们将多糖链长决定蛋白基因wzz与其下游酪氨酸自激酶基因wzc一起克隆至穿梭质粒pBBR1MCS-5中,通过接合作用将构建的重组质粒转化Ymc6、Ymc10突变株,同时将空载体pBBR1MCS-5分别转化相应的突变株和菌株XHY-A6野生型(图 2)。wzz和wzc两个基因一起克隆至穿梭质粒上才能恢复Ymc6突变株菌胶团形成的表型,说明突变株中确实存在极性效应,wzz和wzc这两个基因都参与菌胶团的形成。
|
| 图 2 菌胶团形成相关基因wzz和wzc的筛选与遗传互补分析 Figure 2 Floc-deficient transposon insertional mutant screening and genetic complementary analysis of the requirement of the wzz and wzc genes for floc formation in Mitsuaria sp. XHY-A6 注:A:携带pBBR1MCS-5空质粒的XHY-A6野生型菌株显微镜观察;B:携带pBBR1MCS-5空质粒的Ymc6突变株显微镜观察;C:Ymc6互补菌株(携带克隆有wzz和wzc基因的重组质粒pBBR1MCS-5-wzzc)的显微镜观察;D:从左至右分别是振荡培养状态的A至C;E:从左至右分别是静置培养状态的A至C.标尺:10 μm. Note: A: The floc-forming wild-type strain carrying the empty pBBR1MCS-5 vector under microscope (stained by methylene blue); B: The floc-deficient Ymc6 mutant carrying the empty vector; C: The Ymc6 mutant carrying the complementary pBBR1MCS-5-wzzc construct; D: Agitated cell culture of A, B and C (simulating aeration tank); E: Settled cell culture of A, B and C (simulating settlement tank). Bar: 10 μm. |
|
|
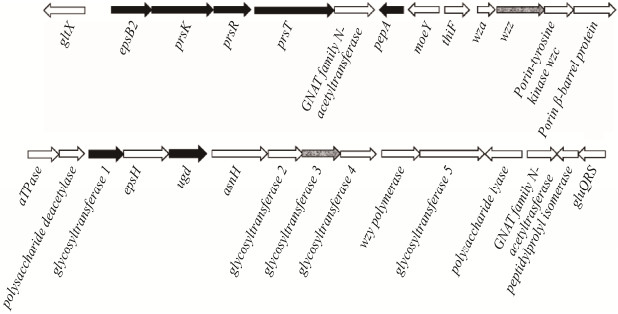
根据上述两个基因在松江菌染色体上的位置,可以发现位于谷氨酸-tRNA连接酶基因gltX和tRNA谷氨酰胺(Q34)合成酶基因gluQRS之间的一个大型基因簇,共有26个基因(图 3),长度约38 kb。通过基因组注释和BLAST分析发现,该基因簇编码糖基转移酶EpsB2,该酶的直系同源物在大肠杆菌和其他细菌中催化一个活化的单糖转移到聚异戊二烯脂-十一碳烯醇二磷酸受体分子上[24]。其下游是prsK-prsR-prsT基因[25]。PrsK和PrsR分别为双组分系统(Two-component system)的感受器组氨酸激酶(Sensor histidine kinase)和响应转录调节因子(Response regulator),最近我们发现在喜树脂动胶菌中PrsK-PrsR二组分系统调控RpoN依赖的PEP-CTERM基因的转录[4]。PrsK感受的信号依然未知。PrsT是一个脂蛋白,可能参与多糖的分泌[26]。其下游的GNAT家族N-乙酰基转移酶可能参与多糖的修饰。菌株XHY-A6染色体DNA互补链上编码的PEP-CTERM蛋白质PepA的表达也可能像在喜树脂动胶菌MMB中的直系同源基因一样受到PrsK-PrsR双组分系统和RpoN (sigma54) sigma因子的调控[4]。除MoeY和ThiF同源蛋白的基因功能还无从推测之外,该基因簇的其他基因多参与胞外多糖的生物合成、修饰和分泌[4, 23, 27],包括转座子插入突变造成菌胶团形成表型缺陷的糖基转移酶基因gt3和wzz基因。AsnH可能是一个天冬酰胺合成酶,催化天冬酰胺的合成,而细胞质中较高含量的天冬酰胺可能保证富含天冬酰胺残基的PEP-CTERM家族蛋白质的表达。这些推测需要进一步的实验验证。
|
| 图 3 解壳聚糖松江菌基因组中的胞外多糖合成基因簇 Figure 3 The extracellular polysaccharide biosynthesis gene cluster in the genome of Mitsuaria chitosanitabida XHY-A6 注:阴影所示的已筛选出菌胶团形成功能缺失的突变株中转座子插入所破坏的基因wzz和gt3. Note: The shaded genes indicate the transposon-disrupted locus, wzz or glycosyltransferase gene 3 (termed gt3), identified in the specific floc-forming deficient mutants that we obtained. |
|
|
综上所述,我们对解壳聚糖松江菌的初步分析揭示活性污泥微生物的菌胶团形成机制与优势种动胶菌非常相似,在不同细菌中取得的研究结果可以相互印证和补充。菌株XHY-A6合成和分泌的胞外多糖化学结构和性质尚待进一步研究。从土壤中分离解壳聚糖松江菌KMBL 5781菌株大量分泌高保水性的胞外多糖,该多糖可能由葡萄糖、甘露糖和半乳糖组成,其摩尔比大致为18:6:1[28]。微生物菌胶团可以称之为一类特殊的生物被膜(Biofilm),即一类不需要附着在生物和非生物表面上的生物被膜,除抵抗原生动物摄食和吸水保湿以外,菌胶团其他生理功能还不清楚。活性污泥微生物胞外多聚物合成和菌胶团形成和调控机制需要进一步研究,例如是什么信号触发菌胶团形成?这些研究有助于活性污泥膨胀的控制、污泥减量和剩余污泥的处置和资源化利用。
| [1] |
An WX, Gao N, Xia M, et al. Physiological characteristics and systematic classification of the Zoogloea species and their role in the activated sludge[J]. Chinese Journal of Applied and Environmental Biology, 2016, 22(6): 1167-1174. (in Chinese) 安卫星, 高娜, 夏明, 等. 动胶菌属系统分类、生理特征及其在活性污泥中的作用[J]. 应用与环境生物学报, 2016, 22(6): 1167-1174. |
| [2] |
An WX, Guo F, Song YL, et al. Comparative genomics analyses on EPS biosynthesis genes required for floc formation of Zoogloea resiniphila and other activated sludge bacteria[J]. Water Research, 2016, 102: 494-504. DOI:10.1016/j.watres.2016.06.058 |
| [3] |
Yu DZ, Xia M, Zhang LP, et al. RpoN (σ54) is required for floc formation but not for extracellular polysaccharide biosynthesis in a floc-forming Aquincola tertiaricarbonis strain[J]. Applied and Environmental Microbiology, 2017, 83(14): e00709-17. |
| [4] |
Gao N, Xia M, Dai JC, et al. Both widespread PEP-CTERM proteins and exopolysaccharides are required for floc formation of Zoogloea resiniphila and other activated sludge bacteria[J]. Environmental Microbiology, 2018, 20(5): 1677-1692. DOI:10.1111/emi.2018.20.issue-5 |
| [5] |
Park JK, Shimono K, Ochiai N, et al. Purification, characterization, and gene analysis of a chitosanase (ChoA) from Matsuebacter chitosanotabidus 3001[J]. Journal of Bacteriology, 1999, 181(21): 6642-6649. |
| [6] |
Amakata D, Matsuo Y, Shimono K, et al. Mitsuaria chitosanitabida gen. nov., sp. nov., an aerobic, chitosanase-producing member of the 'Betaproteobacteria'[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(5): 1927-1932. DOI:10.1099/ijs.0.63629-0 |
| [7] |
Shimono K, Shigeru K, Tsuchiya A, et al. Two glutamic acids in chitosanase A from Matsuebacter chitosanotabidus 3001 are the catalytically important residues[J]. The Journal of Biochemistry, 2002, 131(1): 87-96. |
| [8] |
Yun CS, Amakata D, Matsuo Y, et al. New chitosan-degrading strains that produce chitosanases similar to ChoA of Mitsuaria chitosanitabida[J]. Applied and Environmental Microbiology, 2005, 71(9): 5138-5144. DOI:10.1128/AEM.71.9.5138-5144.2005 |
| [9] |
Yun CS, Matsuda H, Kawamukai M, et al. Directed evolution to enhance secretion efficiency and thermostability of chitosanase from Mitsuaria chitosanitabida 3001[J]. Bioscience, Biotechnology, and Biochemistry, 2006, 70(2): 559-563. DOI:10.1271/bbb.70.559 |
| [10] |
Peng N, Xu WL, Wang F, et al. Mitsuaria chitosanase with unrevealed important amino acid residues: characterization and enhanced production in Pichia pastoris[J]. Applied Microbiology and Biotechnology, 2013, 97(1): 171-179. DOI:10.1007/s00253-012-3901-y |
| [11] |
Yorinaga Y, Kumasaka T, Yamamoto M, et al. Crystal structure of a family 80 chitosanase from Mitsuaria chitosanitabida[J]. FEBS Letters, 2017, 591(3): 540-547. DOI:10.1002/feb2.2017.591.issue-3 |
| [12] |
Rong XQ, Gurel FB, Meulia T, et al. Draft genome sequences of the biocontrol bacterium Mitsuaria sp. strain H24L5A[J]. Journal of Bacteriology, 2012, 194(3): 734-735. DOI:10.1128/JB.06537-11 |
| [13] |
Marian M, Nishioka T, Koyama H, et al. Biocontrol potential of Ralstonia sp. TCR112 and Mitsuaria sp. TWR114 against tomato bacterial wilt[J]. Applied Soil Ecology, 2018, 128: 71-80. DOI:10.1016/j.apsoil.2018.04.005 |
| [14] |
Someya N, Ikeda S, Morohoshi T, et al. Diversity of culturable chitinolytic bacteria from rhizospheres of agronomic plants in Japan[J]. Microbes and Environments, 2011, 26(1): 7-14. DOI:10.1264/jsme2.ME10149 |
| [15] |
Nascimento SB, Lima AM, Borges BN, et al. Endophytic bacteria from Piper tuberculatum Jacq.: isolation, molecular characterization, and in vitro screening for the control of Fusarium solani f. sp. piperis, the causal agent of root rot disease in black pepper (Piper nigrum L.)[J]. Genetics and Molecular Research, 2015, 14(3): 7567-7577. DOI:10.4238/2015.July.3.32 |
| [16] |
Paver SF, Hayek KR, Gano KA, et al. Interactions between specific phytoplankton and bacteria affect lake bacterial community succession[J]. Environmental Microbiology, 2013, 15(9): 2489-2504. DOI:10.1111/emi.2013.15.issue-9 |
| [17] |
Fan MC, Nan LJ, Zhu YM, et al. Mitsuaria noduli sp. nov., isolated from the root nodules of Robinia pseudoacacia in a lead-zinc mine[J]. International Journal of Systematic and Evolutionary Microbiology, 2018, 68(1): 87-92. DOI:10.1099/ijsem.0.002459 |
| [18] |
Inaba T, Hori T, Navarro RR, et al. Revealing sludge and biofilm microbiomes in membrane bioreactor treating piggery wastewater by non-destructive microscopy and 16S rRNA gene sequencing[J]. Chemical Engineering Journal, 2018, 331: 75-83. DOI:10.1016/j.cej.2017.08.095 |
| [19] |
Zhang Q, Tan X, Lin K, et al. Temporal heterogeneity of prokaryotic micro-organism communities in sediment of traditional freshwater cultured fish ponds in Southwest China[J]. Biotechnology & Biotechnological Equipment, 2018, 32(1): 102-108. |
| [20] |
Aoyagi T, Morishita F, Sugiyama Y, et al. Identification of active and taxonomically diverse 1, 4-dioxane degraders in a full-scale activated sludge system by high-sensitivity stable isotope probing[J]. The ISME Journal, 2018, 12(10): 2376-2388. DOI:10.1038/s41396-018-0201-2 |
| [21] |
Kovach ME, Elzer PH, Hill DS, et al. Four new derivatives of the broad-host-range cloning vector pBBR1MCS, carrying different antibiotic-resistance cassettes[J]. Gene, 1995, 166(1): 175-176. DOI:10.1016/0378-1119(95)00584-1 |
| [22] |
Bouhenni R, Gehrke A, Saffarini D. Identification of genes involved in cytochrome c biogenesis in Shewanella oneidensis, using a modified mariner transposon[J]. Applied and Environmental Microbiology, 2005, 71(8): 4935-4937. DOI:10.1128/AEM.71.8.4935-4937.2005 |
| [23] |
Liu YG, Chen YL. High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences[J]. BioTechniques, 2007, 43(5): 649-650, 652. DOI:10.2144/000112601 |
| [24] |
Whitfield C. Biosynthesis and assembly of capsular polysaccharides in Escherichia coli[J]. Annual Review of Biochemistry, 2006, 75: 39-68. DOI:10.1146/annurev.biochem.75.103004.142545 |
| [25] |
Haft DH, Paulsen IT, Ward N, et al. Exopolysaccharide-associated protein sorting in environmental organisms: the PEP-CTERM/EpsH system. Application of a novel phylogenetic profiling heuristic[J]. BMC Biology, 2006, 4: 29. DOI:10.1186/1741-7007-4-29 |
| [26] |
Whitfield C, Mainprize IL. TPR motifs: hallmarks of a new polysaccharide export scaffold[J]. Structure, 2010, 18(2): 151-153. DOI:10.1016/j.str.2010.01.006 |
| [27] |
Cuthbertson L, Mainprize IL, Naismith JH, et al. Pivotal roles of the outer membrane polysaccharide export and polysaccharide copolymerase protein families in export of extracellular polysaccharides in gram-negative bacteria[J]. Microbiology and Molecular Biology Reviews, 2009, 73(1): 155-177. DOI:10.1128/MMBR.00024-08 |
| [28] |
Charchoghlyan H, Park HD. Characteristics of a novel bacterial polysaccharide consisted of glucose and mannose as major components[J]. Food Hydrocolloids, 2013, 30(2): 512-518. DOI:10.1016/j.foodhyd.2012.07.011 |
 2019, Vol. 46
2019, Vol. 46