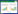扩展功能
文章信息
- 李华华, 梁庆, 崔寒, 邢德峰
- LI Hua-Hua, LIANG Qing, CUI Han, XING De-Feng
- 厌氧产氢颗粒污泥蛋白质组分析样品的高效制备方法
- Efficient preparation method of samples for proteomic analysis of anaerobic hydrogen-producing granular sludge
- 微生物学通报, 2019, 46(8): 1867-1875
- Microbiology China, 2019, 46(8): 1867-1875
- DOI: 10.13344/j.microbiol.china.190221
-
文章历史
- 收稿日期: 2019-03-20
- 接受日期: 2019-05-07
- 网络首发日期: 2019-05-16
2. 城市水资源与水环境国家重点实验室 黑龙江 哈尔滨 150090
2. State Key Laboratory of Urban Water Resources and Environment, Harbin, Heilongjiang 150090, China
蛋白质是生理功能的执行者,蛋白质组(Proteome)研究为解析生命体中复杂的生理功能机制提供了重要的技术手段。研究表明,mRNA水平的变化并不足以诠释蛋白质水平的丰度变化,在细菌以及真核生物中发现,细胞内蛋白质丰度与其对应的mRNA丰度的相关性只有40%左右[1]。蛋白质组学(Proteomics)以蛋白质组为研究对象,基于质谱的蛋白质组学技术可同时检测和定量细胞内外蛋白质丰度变化,也可以检测蛋白翻译后修饰的变化,是研究微生物细胞内复杂的代谢变化与功能机制的有力工具之一[2-3]。宏蛋白质组学是研究微生物群落的一种新策略,通过质谱技术对微生物群落的蛋白质信息进行采集与鉴定,有助于特定功能微生物以及代谢方式的筛查与鉴定,可揭示群落中的微生物之间以及微生物与外界环境之间的关系,目前宏蛋白质组学被广泛应用于污水处理、极端环境微生物群落、肠道微生物群落等领域的研究[4-5]。蛋白质组学数据的质量很大程度上依赖于蛋白质样品的制备质量,然而这一重要步骤在很多蛋白质组学研究中均未给予足够重视。由于不同类型生物样品的成分存在差异,目前没有通用的蛋白样品制备方法,特定类型的生物样品需要特有的蛋白样品制备方案[6]。
颗粒污泥是一种由微生物聚集形成的特殊形式的生物膜,直径为0.1 mm-5.0 mm,在高效厌氧技术中,颗粒污泥可提高反应器的处理能力,利于增加反应器中的微生物浓度以及固液分离等[7-8]。胞外聚合物(Extracellular polymeric substances,EPS)是多糖、蛋白质、脂类等大分子的复杂混合物,不仅是颗粒污泥的保护层,也作为黏结剂将微生物群聚在一起形成颗粒,是颗粒污泥形成的主要条件[9-11]。利用颗粒污泥产氢是具有广泛应用前景的制氢技术之一,相比传统絮状产氢污泥,产氢颗粒污泥具有更好的稳定性与产氢效率[12-14]。产氢颗粒污泥由于细菌含量高通常呈乳白色,平均粒径为1.6 mm,形成过程复杂,凝胶包埋、合成材料封装、静电力吸附等固定化技术都可以促进产氢颗粒污泥的形成[14-15]。目前关于产氢颗粒污泥的相关研究主要集中在其特性分析、形成条件的考察与优化、利用产氢颗粒污泥对产氢条件进行优化等方面[14, 16-18]。产氢颗粒污泥的蛋白质组学研究尚未见文献报道。哈尔滨产乙醇杆菌(Ethanoligenens harbinense)是乙醇型发酵法制氢的主要功能菌群[19]。Ethanoligenens harbinense YUAN-3是哈尔滨产乙醇杆菌属的模式菌株,有较高的发酵产氢能力,该菌株不需要固定化技术处理即可形成直径为0.5 mm-10.0 mm的自凝集颗粒,具有良好的工程应用价值,同时由于单一菌株形成的活性颗粒污泥蛋白质组大小已知,便于后续数据的量化分析,因此选择该菌株形成的活性颗粒污泥作为本研究中的模式实验材料[20]。
对生物样品进行有效破碎是提取总蛋白非常关键的一步,可使细胞内的蛋白质得到有效释放,最大限度地增加提取所得蛋白质的种类。细菌菌体破碎的难易程度主要取决于细胞壁中肽聚糖的含量;另外,肽聚糖含量高也增加了蛋白提取纯化的难度[21]。与革兰氏阴性菌相比,革兰氏阳性菌的细胞壁更厚,肽聚糖含量也更高,例如革兰氏阴性菌铜绿假单胞菌(Pseudomonas aeruginosa)与大肠杆菌(Escherichia coli)的肽聚糖层厚度分别为2.14±0.54 nm与6.35±0.53 nm[22]。革兰氏阳性菌枯草芽孢杆菌(Bacillus subtilis)与金黄色葡萄球菌(Staphylococcus aureus)的肽聚糖层厚度为33.6±4.0 nm与33.8±5.4 nm[23-24]。哈尔滨产乙醇杆菌属于革兰氏阳性菌,因此本研究也可为其它革兰氏阳性菌的蛋白质组学研究提供方法参考。
1 材料与方法 1.1 主要试剂和仪器及培养基胰蛋白酶,Promega公司;TMT 6-plex试剂,ThermoFisher公司;iTRAQ 4-plex试剂,AB SCIEX公司;2D Clean-Up试剂盒,GE Healthcare公司。超滤管,Merck Millipore公司;双向电泳仪,GE Healthcare公司;细胞破碎仪,MP FastPrep公司;超声破碎仪,宁波新芝生物科技股份有限公司;纳升液相色谱、质谱仪,Thermo Scientific公司。PYG厌氧液体培养基成分参照文献[25]配制。
1.2 培养条件及菌体收集E. harbinense YUAN-3接种至PYG厌氧液体培养基中(pH 7.0),35 ℃、120 r/min条件下培养至对数期。取对数期的菌液,10 000 r/min离心5 min收集菌体,除尽上清,PBS缓冲液(pH 7.4)清洗菌体,离心除尽上清,重复清洗一次,液氮冻存于-80 ℃冰箱中。
1.3 蛋白提取与双向电泳总蛋白提取分别使用苯酚抽提法与TCA-丙酮沉淀法,实验步骤参照文献[26-27]。蛋白浓度测定使用2D Clean-Up试剂盒,双向电泳实验步骤参照文献[28]。
1.4 总蛋白的TMT标记与iTRAQ标记 1.4.1 TMT 6-plex标记0.75 mg蛋白中加入100 μL 6 mol/L的盐酸胍进行重溶,用浓度为1 mol/L的Tris-HCl (pH 8.1)将pH调整到7.5以上,加入终浓度为20 mmol/L的DTT,60 ℃处理30 min进行还原。冷却至室温后加入终浓度为40 mmol/L的碘乙酰胺,室温避光1 h进行烷基化。再将样品转移至透析盒(10 kD,0.5-3.0 mL),在含有2 mol/L尿素与50 mmol/L碳酸氢铵的溶液中透析2 h,然后在含有50 mmol/L碳酸氢铵的溶液中透析2次,每次2 h。透析结束后,样品中加入质量比为1:50 (胰蛋白酶:样品)的胰蛋白酶,37 ℃进行酶切过夜。酶切后的肽段用Amicon超滤管过滤,然后真空干燥去除残留的胺。用TMT 6-plex试剂将肽段进行TMT标记。
1.4.2 iTRAQ 4-plex标记0.75 mg蛋白中加入150 μL含有50 mmol/L DTT的1×LDS上样缓冲液,90 ℃水浴10 min。取10%的样品进行SDS-PAGE (10%)凝胶电泳,待上样蛋白全部进入凝胶后(1-2 cm)停止电泳。将蛋白凝胶切下,加入终浓度为10 mmol/L的DTT,60 ℃处理30 min进行还原。冷却至室温后,加入终浓度为20 mmol/L的碘乙酰胺,室温避光1 h进行烷基化。样品中加入质量比为1:50 (胰蛋白酶:样品)的胰蛋白酶,37 ℃进行酶切过夜。用含有60%乙腈与5%甲酸的溶液将酶切后的肽段从凝胶上洗脱下来,然后真空干燥去除残留的胺。用iTRAQ 4-plex试剂将肽段进行iTRAQ标记。
1.5 质谱与生物信息学分析应用纳升液相色谱与串联质谱联用(Nano-LC-MS/MS)的方法对标记后的肽段样品进行分析鉴定。将标记的肽段样品溶于0.1%的三氟乙酸中,上样至Trap柱上,以10 μL/min的流速用0.2%的甲酸洗5 min,样品进入与Trap柱相连的分析柱中以300 nL/min的流速用溶液B (0.16%甲酸,80%乙腈)进行线性梯度洗脱:4%-15%的溶液B洗脱25 min,15%-25%的溶液B洗脱65 min,25%-50%的溶液B洗脱55 min。质谱仪采用数据依赖模式采集数据,MS全扫描的分辨率为120 000,选取离子强度排名前20的进行MS/MS扫描。MS/MS扫描的分辨率为30 000,扫描的相对碰撞能量设置为30%,动态排除持续时间为30 s。Nano-LC-MS/MS分析完成后,在MUDPIT模式下对串联质谱图通过X!Tandem (Sledgehammer版)搜索引擎进行数据库检索,数据库为Ethanoligenens harbinense YUAN-3数据库(Bacteria.ensembl.org;genome assembly:ASM17811v2)。固定修饰为半胱氨酸的脲甲基化以及赖氨酸的TMT/iTRAQ标记,可变修饰为蛋白N端的TMT/iTRAQ标记与甲硫氨酸的氧化。母离子与子离子误差分别设置为+/-7×10-6与2×10-5,总离子强度 > 20 000,肽段log(e) ≤-2.0。提取报告离子丰度,根据每种报告离子比例的中位数值进行均一化统计分析确定报告离子强度百分比。蛋白分子量与等电点的计算利用Compute pI/Mw (https://web.expasy.org/compute_pi/),蛋白亚细胞定位数据通过PSORTb 3.0.2预测。
2 结果与分析 2.1 不同破碎方法对蛋白质提取量的影响综合考虑破碎效果、实验操作时间以及后续实验对蛋白样品的要求,选择超声破碎法、细胞匀浆破碎法、液氮研磨法进行比较,以未经任何处理的样品为对照组,用苯酚抽提法提取样品总蛋白,比较以上几种破碎方法对总蛋白提取量的影响。
2.1.1 液氮研磨法在研钵中加入液氮,将菌体充分研磨至滑腻状粉末。结果如图 1所示,未经处理的1 g菌体样品提取的蛋白量为4.42±0.24 mg;经过液氮研磨的样品提取的蛋白量为8.88±0.45 mg,是对照样品的2.0倍。
|
| 图 1 液氮研磨法对蛋白提取量的影响 Figure 1 The protein yields by liquid nitrogen grinding method |
|
|
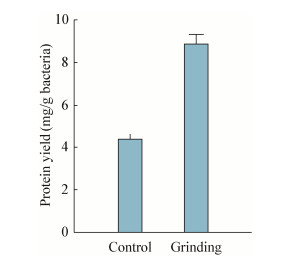
除对照样品外,其他样品的超声条件为功率550 W,振幅20%,超声1 s,间歇8 s,分别超声100、200、400个循环,最后一个样品超声条件为550 W,振幅30%,超声1 s,间歇8 s,进行400个循环。结果如图 2所示,随着超声循环数的增加,提取的蛋白量也在增加。其中,对照样品提取的蛋白量最少,而振幅20%并进行400个循环处理的样品提取的蛋白量最多,为17.35±0.27 mg,是对照样品的3.9倍。
|
| 图 2 超声时间及功率对蛋白提取量的影响 Figure 2 The protein yields by sonication method |
|
|
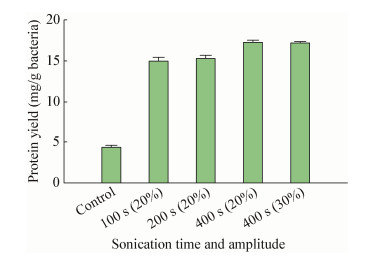
在细胞破碎仪上进行破碎处理,破碎总时间分别为20、40、60、80、100 s,仪器运行速度为4.5 m/s,每破碎20 s后,迅速将样品取下置于冰上降温3 min。结果如图 3所示,对照样品提取的总蛋白量最少,而破碎总时间为80 s的样品提取的总蛋白量最多,为23.14±0.42 mg,是对照样品的5.2倍。
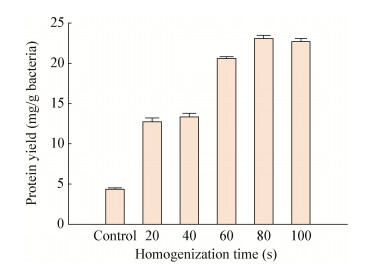
|
| 图 3 匀浆破碎时间对蛋白提取量的影响 Figure 3 The protein yields by homogenization method |
|
|
以上结果表明,与其它两种方法相比,匀浆破碎法操作简单、破碎效果最好,而且所需时间最短,大大降低了样品中蛋白降解的风险。超声破碎法虽然操作方法简单,破碎效果较为理想,但所需时间较长,超声过程中会产生热量,增加了蛋白降解的风险。所以细胞匀浆破碎法是一种有效的厌氧产氢颗粒污泥与革兰氏阳性菌破碎方法。
2.2 苯酚抽提法与TCA-丙酮沉淀法的比较TCA-丙酮沉淀法与苯酚抽提法是蛋白质组学中最常用的两种总蛋白提取方法,因此本研究选取这两种方法进行比较[29]。TCA-丙酮沉淀法中,TCA使提取液呈酸性,可以有效抑制蛋白水解酶的活性;盐离子、多糖和脂类等物质溶于丙酮相中,使样品得以纯化。该方法优点是操作步骤简单,蛋白提取得率高;缺点是提取所得蛋白再重新溶解比较困难,从而影响后续实验分析[21, 30]。苯酚抽提法中,苯酚是一种良好的蛋白溶剂,蛋白能溶于苯酚层,核酸、脂类、多糖以及一些小分子杂质可溶于水相而被去除。该方法中使用醋酸铵作为蛋白沉淀剂,使蛋白质的重新溶解性更好;使用甲醇反复清洗蛋白沉淀可去除醋酸铵和一些杂质离子,同时残留的醋酸铵和甲醇可在低温下完全挥发,不影响后续实验[31]。苯酚抽提法优点是提取蛋白纯度高,重新溶解性好;缺点是操作步骤繁琐,易造成蛋白损失[32]。
本研究通过双向电泳技术考察了TCA-丙酮沉淀法与苯酚抽提法对产氢颗粒中蛋白提取质量的影响,结果如图 4所示,在蛋白上样量(0.5 mg)相同的情况下,苯酚抽提法样品在双向电泳图谱上的蛋白质点明显增多,分布均匀,同时其在碱性蛋白端与小分子量蛋白端的蛋白质点也明显增多。因此,苯酚抽提法的蛋白提取效率更高,提取的蛋白种类也更丰富,是一种更适用于产氢颗粒污泥总蛋白提取的方法。
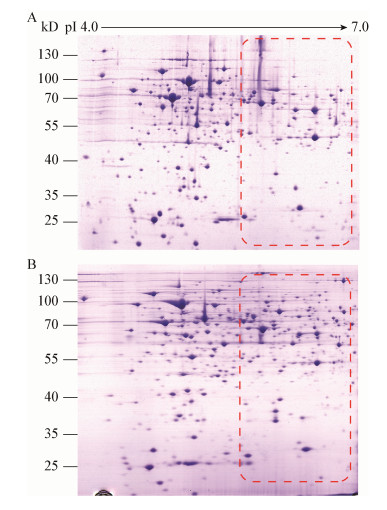
|
| 图 4 TCA-丙酮提取法(A)与苯酚抽提法(B)提取的蛋白双向电泳图谱 Figure 4 Two-dimensional electrophoresis map of total proteins which were extracted using TCA-acetone extraction method (A) and phenol extraction method (B) respectively |
|
|
为了进一步考察蛋白质组分析样品的制备效果,通过质谱技术分析了TMT 6-plex标记的总蛋白样品与iTRAQ 4-plex标记的总蛋白样品。在YUAN-3基因组注释中共有2 701个预测蛋白,iTRAQ 4-plex标记的菌株YUAN-3总蛋白样品中共鉴定到1 797个蛋白,占基因组中预测蛋白的66.5%。而TMT 6-plex标记的YUAN-3总蛋白样品中共鉴定到1 644个蛋白,占基因组预测蛋白的60.9%。蛋白质的分子量、等电点以及亚细胞定位是反映其功能的重要理化指标,将质谱鉴定的蛋白与YUAN-3基因组中预测蛋白的分子量、等电点、亚细胞定位分布进行了比较。与菌株YUAN-3基因组中预测蛋白相比,iTRAQ/TMT标记样品中鉴定到的蛋白有以下3个特点:
(1) 小分子量蛋白比例减少,尤其是0-10 kD的蛋白(表 1)。理论上,蛋白分子量越小,蛋白分子上的胰蛋白酶酶切位点的数量就越少,因此小分子量蛋白在酶切后得到的相应肽段数目越少,从而降低了小分子量蛋白在质谱分析中的鉴定几率[33]。这是导致iTRAQ/TMT标记样品中鉴定到小分子量蛋白比例减少的原因。
| Molecular weight (kD) | Genome (%) | iTRAQ labeling (%) | TMT labeling (%) |
| 0-10 | 8.81 | 3.78 | 4.56 |
| 10-20 | 21.66 | 17.97 | 18.37 |
| 20-30 | 20.99 | 22.48 | 22.63 |
| 30-40 | 18.36 | 20.26 | 20.26 |
| 40-50 | 12.51 | 15.14 | 14.42 |
| 50-60 | 6.92 | 7.23 | 7.42 |
| 60-70 | 3.41 | 4.23 | 3.83 |
| 70-80 | 2.30 | 2.78 | 2.49 |
| 80-90 | 1.70 | 2.06 | 1.95 |
| 90-100 | 1.04 | 1.34 | 1.34 |
| 100-110 | 0.59 | 0.67 | 0.73 |
| 110-120 | 0.44 | 0.67 | 0.55 |
| ≥120 | 1.26 | 1.39 | 1.46 |
(2) 等电点3.0-5.0的酸性蛋白与等电点9.0-13.0的碱性蛋白比例减少(表 2)。原核生物中蛋白分子量与等电点之间存在正相关的关系,小分子量蛋白通常是一些极碱性或极酸性蛋白,因为小分子量蛋白的氨基酸数量较少,氨基酸种类的变化造成蛋白等电点的波动相对更大[34]。因此,iTRAQ/TMT标记样品中鉴定到等电点3.0-5.0的酸性蛋白与等电点9.0-13.0的碱性蛋白比例减少也与小分子量蛋白比例减少有关。
| Isoelectric point | Genome (%) | iTRAQ labeling (%) | TMT labeling (%) |
| 3-4 | 0.6 | 0.3 | 0.4 |
| 4-5 | 11.0 | 9.2 | 10.0 |
| 5-6 | 30.5 | 36.4 | 38.1 |
| 6-7 | 17.7 | 18.6 | 18.9 |
| 7-8 | 6.1 | 6.1 | 6.0 |
| 8-9 | 11.1 | 11.1 | 10.8 |
| 9-10 | 17.9 | 14.6 | 12.7 |
| 10-11 | 4.1 | 2.9 | 2.5 |
| 11-12 | 1.0 | 0.7 | 0.7 |
| 12-13 | 0.1 | 0.0 | 0.1 |
(3) 定位至细胞膜与细胞外的蛋白比例减少,即膜蛋白与细胞表面蛋白比例减少(表 3)。由于膜蛋白与细胞表面蛋白存在跨膜疏水结构域,胰蛋白酶很难对跨膜疏水结构域进行有效酶切,使得膜蛋白与细胞外蛋白在酶切后的肽段长度过大,降低了iTRAQ/TMT的标记效率[35]。另外,膜蛋白与细胞表面蛋白固有的疏水特性造成这部分蛋白在总蛋白提取的沉淀步骤中也容易损失[36]。这些都是导致iTRAQ/TMT标记样品中鉴定到的膜蛋白与细胞表面蛋白比例减少的原因。
| Subcellular localization | Genome (%) | iTRAQ labeling (%) | TMT labeling (%) |
| Cytoplasmic | 55.2 | 64.2 | 66.8 |
| Cytoplasmic membrane | 26.0 | 22.3 | 19.6 |
| Extracellular | 2.0 | 1.2 | 1.2 |
| Cell wall | 0.4 | 0.4 | 0.2 |
| Unknown | 16.3 | 12.0 | 12.1 |
除了以上三点,在分子量、等电点、亚细胞定位的各个分布范围内,iTRAQ/TMT标记样品中鉴定到的蛋白占比都与菌株YUAN-3基因组中预测蛋白占比相一致,或高于菌株YUAN-3基因组中预测蛋白占比,进一步表明研究中采用的总蛋白样品制备方法能够良好地覆盖菌株YUAN-3中各个类型的蛋白,从而有利于获取可靠的蛋白质组数据。
3 讨论与结论本研究选择液氮研磨法、超声破碎法、细胞匀浆破碎法对产氢颗粒进行破碎,目前关于上述破碎方法进行比较的相关研究还未见报道。以未经任何处理的实验样品为对照组,3种破碎方法下总蛋白的提取量分别是对照样品的2.0、3.9与5.2倍,因此匀浆破碎法(80 s)更适用于产氢颗粒污泥及革兰氏阳性菌的破碎。
通过双向电泳技术考察TCA-丙酮沉淀法与苯酚抽提法对产氢颗粒中蛋白提取质量的影响,结果表明,使用苯酚抽提法所得的总蛋白中蛋白种类更多,样品在双向电泳图谱上的蛋白质点明显增多,分布均匀,同时其在碱性蛋白端与小分子量蛋白端的蛋白质点也明显增多。TCA-丙酮沉淀法提取的水稻纹枯病菌总蛋白在双向电泳图谱上也存在碱性端蛋白明显缺失的情况,与之相比,苯酚抽提法所得蛋白的种类也更丰富[31]。研究表明,与丙酮沉淀法相比,运用苯酚抽提法提取的野油菜黄单胞杆菌(Xanthomonas campestris)蛋白种类更多,更适合基于双向电泳技术的蛋白质组分析[29]。通过对两株富含脂多糖细菌的总蛋白双向电泳分析也发现,运用苯酚抽提法提取的总蛋白质量优于TCA-丙酮沉淀法,更适合双向电泳分析[27]。
通过质谱分析,iTRAQ标记与TMT标记的蛋白样品中分别鉴定到1 797与1 644个蛋白,以往的研究表明,iTRAQ 4-plex比TMT 6-plex具有更高的标记效率,iTRAQ 4-plex标记的样品中鉴定到的蛋白数量也更多[37]。本研究结果与上述发现相一致,与TMT 6-plex标记相比,iTRAQ 4-plex标记的YUAN-3总蛋白样品中鉴定到的蛋白数量增加了5.6%。在iTRAQ标记的蓝细菌总蛋白样品中,质谱鉴定到1 452个蛋白,占其基因组预测蛋白数量的40%左右[38]。在梭菌(Clostridium sp. strain BOH3)的iTRAQ标记蛋白质组学研究中,通过质谱技术鉴定到838个蛋白,数量可占其基因组预测蛋白的20%左右[39]。在脑膜炎奈瑟菌(Neisseria meningitidis)的TMT标记蛋白质组分析中,通过质谱技术鉴定到609个蛋白,占其基因组预测蛋白数量的28.2%[40]。在大肠杆菌(E. coli K-12 MG1655) TMT标记蛋白质组分析中,质谱鉴定到3 123个蛋白,占其基因组注释蛋白数量的63%[41]。在iTRAQ 4-plex标记或TMT 6-plex标记的YUAN-3总蛋白样品中,质谱鉴定到的蛋白数量都超过了E. harbinense YUAN-3基因组预测蛋白数量的60%。将质谱鉴定的蛋白与YUAN-3基因组中预测蛋白的分子量、等电点、亚细胞定位分布进行了比较。结果表明,在分子量、等电点、亚细胞定位的各个分布范围内,这些蛋白良好地覆盖了E. harbinense YUAN-3中各个类型的蛋白。
综上所述,匀浆破碎法(80 s)与苯酚抽提法联用的蛋白制备方法更适用于厌氧产氢颗粒污泥与革兰氏阳性细菌,该方法有利于获取可靠的蛋白质组数据,在后续的蛋白质双向电泳和定量蛋白质组质谱分析中,可作为产氢颗粒污泥以及革兰氏阳性菌总蛋白制备的方法参考。
| [1] |
Vogel C, Marcotte EM. Insights into the regulation of protein abundance from proteomic and transcriptomic analyses[J]. Nature Reviews Genetics, 2012, 13(4): 227. DOI:10.1038/nrg3185 |
| [2] |
Aebersold R, Mann M. Mass-spectrometric exploration of proteome structure and function[J]. Nature, 2016, 537(7620): 347-355. DOI:10.1038/nature19949 |
| [3] |
van Oudenhove L, Devreese B. A review on recent developments in mass spectrometry instrumentation and quantitative tools advancing bacterial proteomics[J]. Applied Microbiology and Biotechnology, 2013, 97(11): 4749-4762. DOI:10.1007/s00253-013-4897-7 |
| [4] |
Xu HK, Yan KQ, He YB, et al. The strategies and challenges in metaproteomics bioinformatics[J]. Progress in Biochemistry and Biophysics, 2018, 45(1): 23-35. (in Chinese) 徐洪凯, 闫克强, 何燕斌, 等. 宏蛋白质组学信息分析的基本策略及其挑战[J]. 生物化学与生物物理进展, 2018, 45(1): 23-35. |
| [5] |
Herbst FA, Lünsmann V, Kjeldal H, et al. Enhancing metaproteomics — the value of models and defined environmental microbial systems[J]. Proteomics, 2016, 16(5): 783-798. DOI:10.1002/pmic.v16.5 |
| [6] |
Rabilloud T, Lelong C. Two-dimensional gel electrophoresis in proteomics: a tutorial[J]. Journal of Proteomics, 2011, 74(10): 1829-1841. DOI:10.1016/j.jprot.2011.05.040 |
| [7] |
Li JJ, Huang Y, Li DP. Advance of characteristics, cultivation and applications of anaerobic granular sludge[J]. Environmental Science and Technology, 2011, 24(3): 59-63. (in Chinese) 李建金, 黄勇, 李大鹏. 厌氧颗粒污泥的特性、培养及应用研究进展[J]. 环境科技, 2011, 24(3): 59-63. |
| [8] |
Song YX, Liao Q, Yu C, et al. Physicochemical and microbial properties of settled and floating anammox granules in upflow reactor[J]. Biochemical Engineering Journal, 2017, 123: 75-85. DOI:10.1016/j.bej.2017.04.002 |
| [9] |
Sheng GP, Yu HQ, Li XY. Extracellular polymeric substances (EPS) of microbial aggregates in biological wastewater treatment systems: a review[J]. Biotechnology Advances, 2010, 28(6): 882-894. DOI:10.1016/j.biotechadv.2010.08.001 |
| [10] |
Lim SJ, Kim TH. Applicability and trends of anaerobic granular sludge treatment processes[J]. Biomass and Bioenergy, 2014, 60: 189-202. DOI:10.1016/j.biombioe.2013.11.011 |
| [11] |
Ding ZJ, Bourven I, Guibaud G, et al. Role of extracellular polymeric substances (EPS) production in bioaggregation: application to wastewater treatment[J]. Applied Microbiology and Biotechnology, 2015, 99(23): 9883-9905. DOI:10.1007/s00253-015-6964-8 |
| [12] |
Liu M, Zhou XJ, Zhou Z, et al. Effects of heat preconditioning in cultured hydrogen-producing granular sludge[J]. Journal of Sichuan University (Engineering Science Edition), 2013, 45(4): 171-175. (in Chinese) 刘敏, 周湘杰, 周筝, 等. 种泥热预处理对培养产氢颗粒污泥的影响[J]. 四川大学学报:工程科学版, 2013, 45(4): 171-175. |
| [13] |
Qu YY, Guo WQ, Ding J, et al. Influence of seed sludge on granulation of hydrogen-production sludge[J]. Journal of Harbin Institute of Technology, 2010, 42(6): 876-880. (in Chinese) 曲媛媛, 郭婉茜, 丁杰, 等. 接种污泥对产氢颗粒污泥形成的影响[J]. 哈尔滨工业大学学报, 2010, 42(6): 876-880. |
| [14] |
Banu JR, Kannah RY, Kumar MD, et al. Recent advances on biogranules formation in dark hydrogen fermentation system: mechanism of formation and microbial characteristics[J]. Bioresource Technology, 2018, 268: 787-796. DOI:10.1016/j.biortech.2018.07.034 |
| [15] |
Salem AH, Mietzel T, Brunstermann R, et al. Effect of cell immobilization, hematite nanoparticles and formation of hydrogen-producing granules on biohydrogen production from sucrose wastewater[J]. International Journal of Hydrogen Energy, 2017, 42(40): 25225-25233. DOI:10.1016/j.ijhydene.2017.08.060 |
| [16] |
Fang HH, Liu H, Zhang T. Characterization of a hydrogen-producing granular sludge[J]. Biotechnology and Bioengineering, 2002, 78(1): 44-52. DOI:10.1002/(ISSN)1097-0290 |
| [17] |
Zhang ZP, Show KY, Tay JH, et al. Rapid formation of hydrogen-producing granules in an anaerobic continuous stirred tank reactor induced by acid incubation[J]. Biotechnology and Bioengineering, 2007, 96(6): 1040-1050. DOI:10.1002/(ISSN)1097-0290 |
| [18] |
Zhao BH, Yue ZB, Zhao QB, et al. Optimization of hydrogen production in a granule-based UASB reactor[J]. International Journal of Hydrogen Energy, 2008, 33(10): 2454-2461. DOI:10.1016/j.ijhydene.2008.03.008 |
| [19] |
Xing DF, Ren NQ, Wang AJ, et al. Continuous hydrogen production of auto-aggregative Ethanoligenens harbinense YUAN-3 under non-sterile condition[J]. International Journal of Hydrogen Energy, 2008, 33(5): 1489-1495. DOI:10.1016/j.ijhydene.2007.09.038 |
| [20] |
Xing DF, Ren NQ, Li QB, et al. Ethanoligenens harbinense gen. nov., sp. nov., isolated from molasses wastewater[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(4): 755-760. DOI:10.1099/ijs.0.63926-0 |
| [21] |
Shah HN, Gharbia SE. Mass Spectrometry for Microbial Proteomics[M]. London: John Wiley & Sons, 2010.
|
| [22] |
Matias VRF, Al-Amoudi A, Dubochet J, et al. Cryo-transmission electron microscopy of frozen-hydrated sections of Escherichia coli and Pseudomonas aeruginosa[J]. Journal of Bacteriology, 2003, 185(20): 6112-6118. DOI:10.1128/JB.185.20.6112-6118.2003 |
| [23] |
Matias VR, Beveridge TJ. Cryo-electron microscopy reveals native polymeric cell wall structure in Bacillus subtilis 168 and the existence of a periplasmic space[J]. Molecular Microbiology, 2005, 56(1): 240-251. DOI:10.1111/j.1365-2958.2005.04535.x |
| [24] |
Matias VRF, Beveridge TJ. Native cell wall organization shown by cryo-electron microscopy confirms the existence of a periplasmic space in Staphylococcus aureus[J]. Journal of Bacteriology, 2006, 188(3): 1011-1021. DOI:10.1128/JB.188.3.1011-1021.2006 |
| [25] |
Zhao X, Xing DF, Liu BF, et al. The effects of metal ions and L-cysteine on hydA gene expression and hydrogen production by Clostridium beijerinckii RZF-1108[J]. International Journal of Hydrogen Energy, 2012, 37(18): 13711-13717. DOI:10.1016/j.ijhydene.2012.02.144 |
| [26] |
Hurkman WJ, Tanaka CK. Solubilization of plant membrane proteins for analysis by two-dimensional gel electrophoresis[J]. Plant Physiology, 1986, 81(3): 802-806. DOI:10.1104/pp.81.3.802 |
| [27] |
Alam M, Ghosh W. Optimization of a phenol extraction-based protein preparation method amenable to downstream 2DE and MALDI-MS based analysis of bacterial proteomes[J]. Proteomics, 2014, 14(2/3): 216-221. |
| [28] |
Li QS, Chen J, Lin SS, et al. Optimization of two-dimensional electrophoresis condition for rice grain protein and comparison of relevant proteomic methods[J]. Acta Agronomica Sinica, 2012, 38(5): 921-927. (in Chinese) 李奇松, 陈军, 林世圣, 等. 水稻籽粒蛋白双向电泳条件的优化及其蛋白组学方法的比较[J]. 作物学报, 2012, 38(5): 921-927. |
| [29] |
Schulte F, Hardt M, Niehaus K. A robust protocol for the isolation of cellular proteins from Xanthomonas campestris to analyze the methionine effect in 2D-gel experiments[J]. Electrophoresis, 2017, 38(20): 2603-2609. DOI:10.1002/elps.v38.20 |
| [30] |
Cañas B, Piñeiro C, Calvo E, et al. Trends in sample preparation for classical and second generation proteomics[J]. Journal of Chromatography A, 2007, 1153(1/2): 235-258. |
| [31] |
Yu GM, Hu CJ, Wei YW, et al. Efficient protein extraction method from Rhizoctonia solani for proteomic analysis using two-dimensional electrophoresis analysis[J]. Southwest China Journal of Agricultural Sciences, 2011, 24(6): 2160-2163. (in Chinese) 余功明, 胡春锦, 魏源文, 等. 适于双向电泳分析的水稻纹枯病菌菌体蛋白提取方法的比较[J]. 西南农业学报, 2011, 24(6): 2160-2163. DOI:10.3969/j.issn.1001-4829.2011.06.028 |
| [32] |
Faurobert M, Pelpoir E, Chaïb J. Phenol extraction of proteins for proteomic studies of recalcitrant plant tissues[A]//Thiellement H, Zivy M, Damerval C, et al. Plant Proteomics: Methods and Protocols[M]. Totowa, NJ, USA: Humana Press, 2007
|
| [33] |
Aggarwal K, Choe LH, Lee KH. Shotgun proteomics using the iTRAQ isobaric tags[J]. Briefings in Functional Genomics and Proteomics, 2006, 5(2): 112-120. DOI:10.1093/bfgp/ell018 |
| [34] |
Kiraga J, Mackiewicz P, Mackiewicz D, et al. The relationships between the isoelectric point and: length of proteins, taxonomy and ecology of organisms[J]. BMC Genomics, 2007, 8(1): 163. DOI:10.1186/1471-2164-8-163 |
| [35] |
Pham TK, Sierocinski P, Van Der Oost J, et al. Quantitative proteomic analysis of Sulfolobus solfataricus membrane proteins[J]. Journal of Proteome Research, 2010, 9(2): 1165-1172. DOI:10.1021/pr9007688 |
| [36] |
Dunn MJ, Cordwell SJ, Thingholm TE. Technologies for plasma membrane proteomics[J]. Proteomics, 2010, 10(4): 611-627. DOI:10.1002/pmic.v10:4 |
| [37] |
Pichler P, Köcher T, Holzmann J, et al. Peptide labeling with isobaric tags yields higher identification rates using iTRAQ 4-plex compared to TMT 6-plex and iTRAQ 8-plex on LTQ Orbitrap[J]. Analytical Chemistry, 2010, 82(15): 6549-6558. DOI:10.1021/ac100890k |
| [38] |
Tian XX, Chen L, Wang JX, et al. Quantitative proteomics reveals dynamic responses of Synechocystis sp. PCC 6803 to next-generation biofuel butanol[J]. Journal of Proteomics, 2013, 78: 326-345. DOI:10.1016/j.jprot.2012.10.002 |
| [39] |
Basu A, Xin FX, Lim TK, et al. Quantitative proteome profiles help reveal efficient xylose utilization mechanisms in solventogenic Clostridium sp. strain BOH3[J]. Biotechnology and Bioengineering, 2017, 114(9): 1959-1969. DOI:10.1002/bit.v114.9 |
| [40] |
van Ulsen P, Kuhn K, Prinz T, et al. Identification of proteins of Neisseria meningitidis induced under iron-limiting conditions using the isobaric tandem mass tag (TMT) labeling approach[J]. Proteomics, 2009, 9(7): 1771-1781. DOI:10.1002/pmic.v9:7 |
| [41] |
Treitz C, Enjalbert B, Portais JC, et al. Differential quantitative proteome analysis of Escherichia coli grown on acetate versus glucose[J]. Proteomics, 2016, 16(21): 2742-2746. DOI:10.1002/pmic.v16.21 |
 2019, Vol. 46
2019, Vol. 46