扩展功能
文章信息
- 肖冬来, 马璐, 杨驰, 林衍铨
- XIAO Dong-Lai, MA Lu, YANG Chi, LIN Yan-Quan
- 不同碳源条件下广叶绣球菌转录组分析
- Transcriptome analysis of Sparassis latifolia cultivated with different carbon sources
- 微生物学通报, 2019, 46(7): 1654-1661
- Microbiology China, 2019, 46(7): 1654-1661
- DOI: 10.13344/j.microbiol.china.180637
-
文章历史
- 收稿日期: 2018-08-15
- 接受日期: 2018-10-25
- 网络首发日期: 2018-11-08
木质纤维素包括木质素、纤维素和半纤维素。白腐菌可通过其分泌的酶破坏植物细胞壁,从而完全降解木质纤维素,使其分解为小分子糖类。褐腐菌则与白腐菌不同,其仅能利用半纤维素和纤维素,而不能降解木质素。纤维素和半纤维素降解的酶属于碳水化合物活性酶(Carbohydrate-active enzymes,CAZymes),CAZymes可根据氨基酸序列特征及催化机制的不同,可以分为四个功能类别:糖苷水解酶(Glycoside hydrolases,GHs)、糖基转移酶(Glycosyltransferases,GTs)、多糖裂解酶(Polysaccharide lyases,PLs)和碳水化合物酯酶(Carbohydrate esterases,CEs)[1]。某些纤维素和半纤维素酶中的非催化活性结构域还包含碳水化合物结合模块(Carbohydrate-binding modules,CBMs),能够促进酶与底物的结合,提高酶的效率[2]。
纤维素酶系主要包括内切葡聚糖酶(Endoglucanase)、外切葡聚糖酶(Exoglucanase)和葡萄糖苷酶(Glucosidase)。基因组测序技术的发展使得在全基因组水平研究真菌木质纤维素利用的过程成为可能。通过比较基因组学研究表明褐腐菌不含有攻击结晶纤维素的糖苷水解酶家族GH6、GH7[3]。不同碳源可以诱导不同木质纤维素降解酶基因的表达[4],表明碳源是影响真菌降解木质纤维素关键酶表达及活性的重要的因素。
广叶绣球菌(Sparassis latifolia)属于多孔菌目、绣球菌科,是木腐真菌中的褐腐菌。绵腐卧孔菌(Postia placenta)是研究褐腐菌木质纤维素降解的模式真菌,其在多种不同的碳源诱导下,共同上调的木质纤维素降解相关基因包括GH1、GH3、GH5、GH12、GH10、GH27、GH31、GH35、GH47等糖苷水解酶家族,同时,芬顿反应相关基因和细胞色素P450也参与了细胞壁组分的解聚和芳香族化合物的代谢[5]。目前,广叶绣球菌木质纤维素降解机制的研究还未见报道。本实验通过转录组测序技术对分别以葡萄糖、纤维素+木质素、纤维素及松木屑为碳源的广叶绣球菌转录组进行分析,开展广叶绣球菌木质纤维素降解关键酶系的研究,为深入了解其木质纤维素降解的分子机理和相关功能基因奠定基础。
1 材料与方法 1.1 材料 1.1.1 菌株广叶绣球菌“闽绣1号”由福建省农业科学院食用菌研究所保藏。
1.1.2 培养基PDA斜面培养基(g/L):马铃薯200.0,葡萄糖20.0,琼脂20.0,pH自然。基础液体培养基(g/L):葡萄糖20.0,蛋白胨2.0,pH自然。3种诱导培养基则是将基础培养基中的葡萄糖分别替换为纤维素20.0 g/L、松木屑20.0 g/L (松木屑干燥、粉碎后过1 mm筛网)及同时添加纤维素20.0 g/L和木质素1.0 g/L。
1.1.3 主要试剂和仪器纤维素和木质素,Sigma公司;Trizol total RNA extraction kit,Invitrogen公司;Illumina TruSeqTM RNA Sample Prep Kit,Illumina公司;RNA Nano 6000 Assay Kit,Agilent公司;其余化学试剂均为分析纯,生工生物工程(上海)股份有限公司。超离心粉碎仪,Retsch公司;分光光度计,北京凯奥科技发展有限公司;2100 Bioanalyzer,Agilent公司。
1.2 方法 1.2.1 样品处理将保藏的绣球菌经PDA斜面培养基活化后,分别接种于含100 mL基础液体培养基的三角瓶中,25 ℃、150 r/min黑暗培养25 d。分别收集菌丝体,无菌水洗涤3次后分别转入含纤维素、松木屑、同时添加纤维素和木质素的诱导培养基及用于对照试验的基础液体培养基。25 ℃黑暗静置培养4 d后收集菌丝体,液氮速冻后利用干冰运输至安诺优达基因科技(北京)有限公司利用Illumina平台进行测序,测序策略为PE150。以葡萄糖为碳源的对照样品及以纤维素、松木屑、纤维素+木质素为碳源的诱导样品分别编号为:Glucose、Cellulose、Pine sawdust和Cellulose/lignin。
1.2.2 转录组测序数据处理测序得到的原始数据(Raw reads)会含有测序接头序列以及低质量序列,通过去除接头污染的Reads、低质量的Reads以及含N比例大于5%的Reads后得到高质量的Clean reads。采用TopHat V2.0.12软件[6],选用默认参数将Clean reads比对到广叶绣球菌基因组和基因序列(https://genome.jgi.doe.gov/Spalat1/Spalat1.home.html)。
1.2.3 表达量分析及差异基因筛选基因表达水平利用FPKM (fragments per kilobase of exon per million fragments mapped)[7]方法计算。以基础液体培养基培养的样品为对照,采用DEGSeq[8]进行基因差异表达分析,比较处理组与对照组,并选取|log2 Ratio|≥1且Q-value≤0.05的基因作为差异表达基因,计算出上下调基因数量。
1.2.4 GO功能分析将筛选获得的差异基因利用BLAST2GO进行GO功能注释,得到差异基因对应的GO条目。利用在线软件WEGO (http://wego.genomics.org.cn)[9]将差异基因的GO功能进行分类。根据每个GO条目的基因数,应用超几何检验,找出与整个基因组相比差异基因显著富集的GO条目。
1.2.5 碳水化合物活性酶和转录因子注释利用在线注释工具dbCAN2[10]对差异表达基因进行碳水化合物活性酶功能注释(默认参数)。转录因子注释按Todd等[11]的方法进行。
2 结果与分析 2.1 差异基因的筛选以葡萄糖为单一碳源的菌丝为对照,纤维素作为碳源的处理组可获得差异基因529个,其中上调基因402个,下调基因127个;纤维素加木质素作为碳源的处理组可获得差异基因1 336个,其中上调基因1 153个,下调基因183个;松木屑作为碳源的处理组可获得差异基因1 345个,其中上调基因974个,下调基因371个(图 1、2)。3个处理组共有的差异基因数为242个,其中共同上调有209个,共同下调的有5个,其余28个基因变化规律不一致。
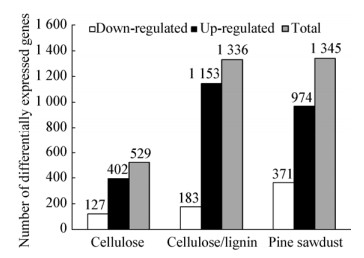
|
| 图 1 差异基因统计图 Figure 1 Statistical diagram of differentially expressed genes |
|
|

|
| 图 2 差异基因韦恩图 Figure 2 Venn diagram of differentially expressed genes |
|
|
对获得的差异基因进行GO注释分析,结果显示(图 3,以松木屑样本为例):在分别以纤维素、纤维素/木质素、松木屑为碳源的条件下,相对于以葡萄糖为单一碳源的对照,差异基因属于生物过程(Biological process)分类的主要为代谢过程(Metabolic process)、细胞过程(Cellular process)和单组织过程(Single-organism process);属于细胞组分(Cellular component)分类的主要为细胞组分(Cell part)、细胞器(Organelle)和细胞器组分(Organelle part);属于分子功能(Molecular function)分类的主要为催化(Catalytic)、结合(Binding)和转运子(Transporter)。通过差异基因GO功能显著性富集分析可确定差异基因参与的主要生物学功能。本实验中3个不同碳源诱导样品通过富集分析可以看出,差异基因在生物过程分类中主要富集在碳水化合物利用的过程(图 4),如多糖催化过程(Polysaccharide catabolic process)、碳水化合物催化过程(Carbohydrate catabolic process)、碳水化合物代谢过程(Carbohydrate metabolic process)及多糖代谢过程(Polysaccharide metabolic process)等。在分子功能分类中,富集的基因参与的功能主要为水解酶活性(Hydrolase activity)和甘露糖苷酶活性(Mannosidase activity)等。三者共有差异基因GO富集分析结果表明,差异基因主要富集的生物学过程和分子功能分别为碳水化合物代谢过程和水解酶活性。

|
| 图 3 差异表达基因GO注释分类图(松木屑) Figure 3 GO annotation classification diagram of differentially expressed genes (Pine sawdust) |
|
|

|
| 图 4 GO富集条目Q值分布图 Figure 4 Q-value distribution diagram of GO enrichment items |
|
|
通过在线软件dbCAN2 meta server对差异基因进行碳水化合物活性酶功能注释。在纤维素为碳源的处理中,差异表达显著的碳水化合物活性酶基因共有25个(上调基因23个,下调基因2个;其中13个基因含有信号肽),包含21个糖苷水解酶、2个糖基转移酶和2个碳水化合物酯酶;在纤维素和木质素为碳源的处理中,差异表达显著的碳水化合物活性酶基因共有40个(上调基因36个,下调基因4个;其中19个基因含有信号肽),包含28个糖苷水解酶、8个糖基转移酶和4个碳水化合物酯酶;在松木屑为碳源的处理中,差异表达显著的碳水化合物活性酶基因共有47个(上调基因43个,下调基因4个;其中22个基因含有信号肽),包含35个糖苷水解酶、7个糖基转移酶和5个碳水化合物酯酶(图 5)。从差异基因数量上可以看出,随着所利用碳源复杂程度的增加,碳水化合物活性酶基因的表达受诱导表达的数量也在增加,更多的基因参与了复杂碳源的利用过程。根据各样品组中差异表达的碳水化合物活性酶基因表达量(FPKM值)构建热图并进行聚类分析,从图 6聚类分析中可以看出,A和B分支基因表达量丰度在3个处理组中较高,其中差异基因在任一组中上调超过4倍的有15个(表 1)。从表 1中可以看出涉及半纤维素降解的相关基因上调幅度最大,如内切1, 4-β-甘露糖苷酶(Endo-1, 4-β- mannosidase)、α-半乳糖苷酶(α-galactosidase)等。同时纤维素降解相关的内切β-1, 4-葡聚糖酶(Endo-β-1, 4-glucanase)、β-葡萄糖苷酶(β-glucosidase)在3个诱导处理组中也表现了上调。
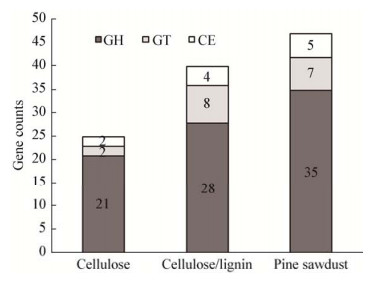
|
| 图 5 差异显著碳水化合物活性酶基因统计图 Figure 5 Statistical diagram of differentially expressed CAZyme genes |
|
|

|
| 图 6 差异显著碳水化合物活性酶基因表达量聚类热图 Figure 6 Heat map showing clustering of differentially expressed CAZyme genes with transcript accumulation |
|
|
| 基因编号 Gene ID |
变化倍数 Fold change |
CAZyme 分类 CAZyme class |
功能注释 Function annotation |
EC编号 EC number |
||
| 纤维素 Cellulose |
纤维素/木质素 Cellulose/lignin |
松木屑 Pine sawdust |
||||
| Spalat1|814297 | 256.9 | 289.8 | 737.0 | GH5 | Endo-1, 4-beta-mannosidase | EC:3.2.1.78 |
| Spalat1|771993 | 48.8 | 124.0 | 311.9 | GH27 | Alpha-galactosidase | EC:3.2.1.22 |
| Spalat1|743345 | 34.2 | 79.0 | 131.4 | GH1 | Beta-glucosidase | EC:3.2.1.21 |
| Spalat1|449309 | 26.0 | 42.8 | 35.5 | GH12 | Xyloglucan-specific endo-beta-1, 4-glucanase | EC:3.2.1.151 |
| Spalat1|814527 | 7.1 | 15.5 | 18.2 | GH5 | Endo-beta-1, 4-glucanase | EC:3.2.1.4 |
| Spalat1|758753 | 5.3 | 6.7 | 16.4 | GH2 | Beta-mannosidase | EC:3.2.1.25 |
| Spalat1|815356 | 6.0 | 8.8 | 11.7 | GH3 | Beta-glucosidase | EC:3.2.1.21 |
| Spalat1|177241 | 6.3 | 9.9 | 11.3 | GH12 | Xyloglucan-specific endo-beta-1, 4-glucanase | EC:3.2.1.151 |
| Spalat1|724470 | 4.7 | 4.7 | 10.4 | GH3 | Beta-glucosidase | EC:3.2.1.21 |
| Spalat1|741529 | 2.1 | 2.4 | 9.9 | GH3 | Beta-glucosidase | EC:3.2.1.21 |
| Spalat1|826704 | 3.7 | 4.1 | 8.4 | GH2 | Beta-mannosidase | EC:3.2.1.25 |
| Spalat1|733349 | 3. 4 | 4.3 | 7.0 | GH2 | Beta-mannosidase | EC:3.2.1.25 |
| Spalat1|901357 | 4.9 | 4.4 | 4.9 | GH37 | Alpha-trehalase | EC:3.2.1.28 |
| Spalat1|391061 | 2.2 | 2.4 | 4.7 | CE2 | Acetyl xylan esterase | EC:3.1.1.72 |
| Spalat1|520923 | 1.0 | 1.1 | 4.4 | GH31 | Alpha-glucosidase | EC:3.2.1.20 |
在以纤维素加木质素、松木屑为碳源的培养条件下,分别有9和10个转录因子相对于以葡萄糖为碳源时表达量显著上调。而以纤维素为碳源的条件下仅有1个转录因子表达量表现为上调,该基因(Spalat1|45187)在3个处理组中均表现为上调。以基因表达量(FPKM值)构建热图并进行聚类分析(图 7),从图 7中可以看出,3个不同碳源诱导组中上调表达的转录因子主要为Zn(2)-C6和Homeobox类型。其中Homebox型转录因子(Spalat1|186697)上调幅度最大,在纤维素加木质素、松木屑为碳源的诱导组中分别上调5.0和10.1倍。

|
| 图 7 上调表达转录因子表达量聚类热图 Figure 7 Heat map showing clustering of up-regulated transcription factor genes with transcript accumulation 注:热图上数字为相对于葡萄糖的变化倍数. Note: The number on the heat map is the fold change value compared to glucose. |
|
|
本研究结果表明碳源的复杂程度直接影响着广叶绣球菌的基因表达谱,碳源越复杂所获得差异基因数量越多。碳水化合物活性酶基因中半纤维素降解相关的基因变化最明显,其中与甘露聚糖代谢内切1, 4-β-甘露糖苷酶在木屑诱导下,上调达737倍,远高于纤维素代谢的关键酶内切β-1, 4-葡聚糖酶(上调18.2倍)。同时半乳糖和木葡聚糖代谢的相关的α-半乳糖苷酶和木葡聚糖特异的内切β-1, 4-葡聚糖酶(Xyloglucan-specific endo-β-1, 4-glucanase)在木屑诱导下上调幅度也较大。褐腐菌茯苓(Wolfiporia cocos)在以松树木屑为碳源时,相对于以葡萄糖为单一碳源也可诱导半纤维素代谢相关的基因显著上调[12]。褐腐菌半纤维素代谢相关基因在种类和数量上均少于白腐菌[13],相关基因的显著上调暗示其在褐腐菌木质纤维素降解过程中的重要作用[14]。绣球菌的这种基因诱导表达模式可能与绣球菌在利用松木屑时首先利用纤维素,以及松木半纤维素的主要单糖组分为甘露糖和木聚糖有关[15]。另外,某些基因在分别以葡萄糖、纤维素、纤维素/木质素和松木屑为碳源时基因表达量均很高,但组间差异未达到上、下调大于2倍(Q-value≤0.05)的差异基因筛选标准,这类基因在绣球菌基质利用过程中同样可能发挥着重要作用[16]。例如GH16家族的基因(Spalat1|740831)在分别以葡萄糖、纤维素、纤维素/木质素和松木屑为碳源时的基因表达量FPKM值分别为465、454、370和522,几丁质脱乙酰酶(Chitin deacetylase) (Spalat1|830233)在基因表达量FPKM值分别为830、881、959和1 191。
本实验中,3个Homeobox家族的转录因子在纤维素+木质素、松木屑诱导组中均显著上调,可能参与了木质纤维素降解相关酶的调控过程。关于Homeobox家族的转录因子研究较多的主要集中在其调控真菌形态发育方面[17-18],在木质纤维素降解调控方面的研究较少,白腐菌黄孢原毛平革菌(Phanerochaete chrysosporium)在杉木屑中诱导40 h和96 h后2个Homeobox家族的转录因子表达量变化显著(上、下调各一个)[19]。在转录因子中调控木质纤维素酶活性的转录因子主要为Zn2-C(6)和zf-C2H2类型,调控着真菌对纤维素、半纤维素、淀粉及果胶利用相关酶基因的表达[20]。本实验中也检测出数个可能参与木质纤维素降解调控的Zn2-C(6)家族转录因子。
本实验通过转录组测序分析了广叶绣球菌在不同碳源诱导下的基因表达差异,相关碳水化合活性酶和转录因子基因可能在广叶绣球菌木质纤维素降解及降解调控方面发挥重要作用。本研究为深入了解广叶绣球菌木质纤维素降解的分子机理和相关基因功能提供了一些参考。
| [1] |
Lombard V, Golaconda Ramulu H, Drula E, et al. The carbohydrate-active enzymes database (CAZy) in 2013[J]. Nucleic Acids Research, 2014, 42(D1): D490-D495. DOI:10.1093/nar/gkt1178 |
| [2] |
Black GW, Rixon JE, Clarke JH, et al. Evidence that linker sequences and cellulose-binding domains enhance the activity of hemicellulases against complex substrates[J]. Biochemical Journal, 1996, 319(2): 515-520. DOI:10.1042/bj3190515 |
| [3] |
Floudas D, Binder M, Riley R, et al. The Paleozoic origin of enzymatic lignin decomposition reconstructed from 31 fungal genomes[J]. Science, 2012, 336(6089): 1715-1719. DOI:10.1126/science.1221748 |
| [4] |
Rytioja J, Hildén K, Hatakka A, et al. Transcriptional analysis of selected cellulose-acting enzymes encoding genes of the white-rot fungus Dichomitus squalens on spruce wood and microcrystalline cellulose[J]. Fungal Genetics and Biology, 2014, 72: 91-98. DOI:10.1016/j.fgb.2013.12.008 |
| [5] |
Kameshwar AKS, Qin WS. Molecular networks of Postia placenta involved in degradation of lignocellulosic biomass revealed from metadata analysis of open access gene expression data[J]. International Journal of Biological Sciences, 2018, 14(3): 237-252. DOI:10.7150/ijbs.22868 |
| [6] |
Trapnell C, Pachter L, Salzberg SL. TopHat: discovering splice junctions with RNA-Seq[J]. Bioinformatics, 2009, 25(9): 1105-1111. DOI:10.1093/bioinformatics/btp120 |
| [7] |
Trapnell C, Williams BA, Pertea G, et al. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology, 2010, 28(5): 511-515. DOI:10.1038/nbt.1621 |
| [8] |
Wang LK, Feng ZX, Wang X, et al. DEGseq: an R package for identifying differentially expressed genes from RNA-seq data[J]. Bioinformatics, 2010, 26(1): 136-138. DOI:10.1093/bioinformatics/btp612 |
| [9] |
Ye J, Fang L, Zheng HK, et al. WEGO: a web tool for plotting GO annotations[J]. Nucleic Acids Research, 2006, 34(S2): W293-W297. |
| [10] |
Yin YB, Mao XZ, Yang JC, et al. dbCAN: a web resource for automated carbohydrate-active enzyme annotation[J]. Nucleic Acids Research, 2012, 40(W1): W445-W451. DOI:10.1093/nar/gks479 |
| [11] |
Todd RB, Zhou MM, Ohm RA, et al. Prevalence of transcription factors in ascomycete and basidiomycete fungi[J]. BMC Genomics, 2014, 15: 214. DOI:10.1186/1471-2164-15-214 |
| [12] |
Gaskell J, Blanchette RA, Stewart PE, et al. Transcriptome and secretome analyses of the wood decay fungus Wolfiporia cocos support alternative mechanisms of lignocellulose conversion[J]. Applied and Environmental Microbiology, 2016, 82(13): 3979-3987. DOI:10.1128/AEM.00639-16 |
| [13] |
Hori C, Gaskell J, Igarashi K, et al. Genomewide analysis of polysaccharides degrading enzymes in 11 white- and brown-rot Polyporales provides insight into mechanisms of wood decay[J]. Mycologia, 2013, 105(6): 1412-1427. DOI:10.3852/13-072 |
| [14] |
Zhang JW, Presley GN, Hammel KE, et al. Localizing gene regulation reveals a staggered wood decay mechanism for the brown rot fungus Postia placenta[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(39): 10968-10973. DOI:10.1073/pnas.1608454113 |
| [15] |
Li PP, Lei YC, Chen C, et al. Effect of hot water prehydrolysis on the major components of the pine and the hydrolysate[J]. China Pulp & Paper, 2012, 31(11): 1-6. (in Chinese) 李萍萍, 雷以超, 陈灿, 等. 热水预水解对松木及其水解液主要化学成分的影响[J]. 中国造纸, 2012, 31(11): 1-6. |
| [16] |
Vanden Wymelenberg A, Gaskell J, Mozuch M, et al. Significant alteration of gene expression in wood decay fungi Postia placenta and Phanerochaete chrysosporium by plant species[J]. Applied and Environmental Microbiology, 2011, 77(13): 4499-4507. DOI:10.1128/AEM.00508-11 |
| [17] |
Pelkmans JF, Patil MB, Gehrmann T, et al. Transcription factors of Schizophyllum commune involved in mushroom formation and modulation of vegetative growth[J]. Scientific Reports, 2017, 7: 310. DOI:10.1038/s41598-017-00483-3 |
| [18] |
Vonk PJ, Ohm RA. The role of homeodomain transcription factors in fungal development[J]. Fungal Biology Reviews, 2018, 32(4): 219-230. DOI:10.1016/j.fbr.2018.04.002 |
| [19] |
Kameshwar AKS, Qin WS. Analyzing Phanerochaete chrysosporium gene expression patterns controlling the molecular fate of lignocellulose degrading enzymes[J]. Process Biochemistry, 2018, 64: 51-62. DOI:10.1016/j.procbio.2017.10.007 |
| [20] |
Benocci T, Aguilar-Pontes MV, Zhou MM, et al. Regulators of plant biomass degradation in ascomycetous fungi[J]. Biotechnology for Biofuels, 2017, 10: 152. DOI:10.1186/s13068-017-0841-x |
 2019, Vol. 46
2019, Vol. 46




