扩展功能
文章信息
- 陈程, 王曈, 常佳伟, 万佳宏, 王桂琴
- CHEN Cheng, WANG Tong, CHANG Jia-Wei, WAN Jia-Hong, WANG Gui-Qin
- 宁夏地区一株奶牛源耐甲氧西林金黄色葡萄球菌流行株的高通量测序分析
- High-throughput sequencing analysis of a methicillin-resistant Staphylococcus aureus strain in dairy cattle in Ningxia province
- 微生物学通报, 2019, 46(7): 1636-1644
- Microbiology China, 2019, 46(7): 1636-1644
- DOI: 10.13344/j.microbiol.china.180601
-
文章历史
- 收稿日期: 2018-07-31
- 接受日期: 2019-02-13
- 网络首发日期: 2019-04-16
金黄色葡萄球菌(Staphylococcus aureus,SA)是一类重要的人畜共患病原菌,在全球范围内引起了较高的发病率和死亡率[1]。在20世纪40年代青霉素推广使用之前,由SA感染导致的败血病的死亡率极高,青霉素在一段时间内有效遏制了SA的肆虐,但是不久后便出现了对青霉素耐药的菌株[2-3]。甲氧西林在1959年开始用于治疗SA感染性疾病,两年后就在英国报道了第一例耐甲氧西林SA (Methicillin-resistant SA,MRSA)[4]。MRSA的耐药性强,人感染后病死率高,与艾滋病、乙型肝炎并列为世界三大医学难题[5]。在随后的几十年里,MRSA在世界各地被报道,MRSA的耐药性以及流行病学特征也越来越复杂。20世纪80年代末期,万古霉素对于治疗MRSA感染性疾病显示出很好的疗效,但是在选择压力作用下,1996年在日本发现了第一例对万古霉素中介耐药SA (vancomycin-intermediate SA,VISA)[6],2002年在美国发现首例耐万古霉素SA (vancomycin-resistant SA,VRSA)[7]。
MRSA主要分为医院获得性MRSA (Hospital- acquired MRSA,HA-MRSA)和社区获得性MRSA (community-acquired MRSA,CA-MRSA)[8]。随着人们对动物食品的需求增加,养殖业迅速发展,动物源MRSA逐渐被人们关注,动物源MRSA又称为家畜相关性耐甲氧西林SA (Livestock-associated MRSA,LA-MRSA)[9]。早在1972年,就有牛源MRSA的相关报道[10],目前在各类养殖动物及伴侣宠物身上分离到MRSA的报道比比皆是[11-13]。流行病学调查显示,LA-MRSA不仅可以在家畜中繁殖,而且可以直接传播给人[14],甚至能感染与畜禽没有接触的社区人员[15]。
奶牛养殖业是宁夏地区的重要产业,每年由SA导致的奶牛乳腺炎都会给奶牛养殖户带来重大的经济损失。本研究基于本实验室前期分型研究结果,选取宁夏地区一株奶牛源MRSA流行株进行全基因组测序分析,旨在发现宁夏地区MRSA流行株特有的序列特征,为研究MRSA菌株之间的序列差异以及MRSA感染的防控提供理论依据。
1 材料与方法 1.1 材料 1.1.1 样品来源试验菌株ld11由本实验室于2015年12月从宁夏地区规模化奶牛养殖场临床型乳房炎乳样中分离鉴定并收集,通过科玛嘉SA显色培养基检测和多重PCR检测16S rRNA、nuc和mecA基因鉴定为MRSA菌株,运用spa和MLST分型鉴定为宁夏地区MRSA流行株t037-ST239,20%甘油BHI肉汤−80 ℃冻存。质控菌株SA ATCC 29213,由中国农业大学动物医学院提供。
1.1.2 主要试剂和仪器BHI肉汤培养基、BHI琼脂培养基,北京陆桥技术有限公司;细菌基因组提取试剂盒、溶菌酶、溶葡萄球菌素,北京博迈德基因技术有限公司;荧光定量试剂盒Qubit dsDNA HS Assay Kit,Life Tech公司;建库试剂盒KAPA HyperPlus DNA Library Preparation Kit for Illumina,KAPA Biosystems公司;苯唑西林、多西环素、氨苄西林、头孢噻呋、环丙沙星、左氧氟沙星、四环素、磺胺异恶唑、红霉素、庆大霉素、克林霉素、阿米卡星、利奈唑胺、万古霉素,中国兽医药品监察所和中国药品与生物制品检定所。
超声破碎仪,Covaris公司;生物分析仪,安捷伦科技有限公司;磁力架,Invitrogen公司;涡旋振荡器,广州仪科实验室技术有限公司。
1.2 方法 1.2.1 抗菌药物敏感性测定根据美国临床实验室标准委员会(Clinical and laboratory standards institute,CLSI)推荐的琼脂稀释法[16]进行,测定苯唑西林、多西环素、氨苄西林、头孢噻呋、环丙沙星、左氧氟沙星、四环素、磺胺异恶唑、红霉素、庆大霉素、克林霉素、阿米卡星、利奈唑胺和万古霉素等14种抗菌药物对试验菌株的最小抑菌浓度值,结果判定参照CLSI推荐标准。
1.2.2 菌株基因组的提取根据细菌基因组DNA提取试剂盒说明书进行提取。
1.2.3 测序前样品的处理根据参考文献[17]对提取的基因组进行质量检测和文库构建。
1.2.4 文库质检和测序DNA文库送北京安诺优达基因科技有限公司进行高通量测序。
1.2.5 测序结果的处理运用分析软件CLC genomic workbench 9.0对测序结果进行拼接组装,得到所测菌株的全基因组序列。
1.2.6 基因组基本特征将组装好的菌株ld11全基因组序列上传到cge网络数据库(https://cge.cbs.dtu.dk/services/cge/),对基因组序列中的耐药基因进行查找统计,同时从Genbank下载人源MRSA (Mu50、MW2、N315和MRSA252)基因组作为参考基因组,对参考基因组的耐药基因进行查找统计并将其与菌株ld11耐药基因的统计结果进行比较分析。通过Harvest软件包内的Parsnp模块对分离株ld11和4株人源参考菌株的基因组进行基于核心基因组的单核苷酸位点多态性(Single nucleotide polymorphism,SNP)系统进化树的构建。运用Glimmer 3.02 (http://www.cbcb.umd.edu/software/glimmer/)软件进行菌株ld11基因组序列信息的预测。
1.2.7 基因功能注释主要通过网络数据库对菌株基因组序列信息进行COG (Clusters of orthologous groups of proteins)功能分析(http://www.ncbi.nlm.nih.gov/COG/)、KEGG (Kyoto encyclopedia of genes and genomes)通路分析(http://www.genome.jp/kegg/;http://www.genome.jp/kegg/document/help_pathway.html)和GO (Gene ontology)注释统计(http://www.geneontology.org/)。
1.2.8 基因岛预测利用已获得的基因组gbk文件,使用Islander- viewer、SIGI-HMM等软件对测序菌株中包含的基因岛序列进行预测。
1.2.9 CRISPR分析使用CRISPR Finder在线工具(http://crispr.i2bc.paris-saclay.fr/)对菌株基因组进行CRISPR (Clustered regularly interspaced short palindromic repeats)预测,找出基因组上所有潜在的CRISPR序列,展示CRISPR序列在基因组上的位置、repeats和Spacer的碱基组成等。
1.2.10 前噬菌体序列分析通过在线软件PHAST (http://phast.wishartlab.com/index.html)预测菌株基因组上的前噬菌体(Prophage),并对结果进行统计分析。
2 结果与分析 2.1 药敏试验结果药敏试验结果显示分离株ld11耐药情况严重,对头孢噻呋、磺胺异恶唑、氨苄西林、红霉素、庆大霉素、苯唑西林、克林霉素、四环素和多西环素9种抗菌药物耐药,对环丙沙星、左氧氟沙星、阿米卡星、利奈唑胺和万古霉素5种抗菌药物敏感。
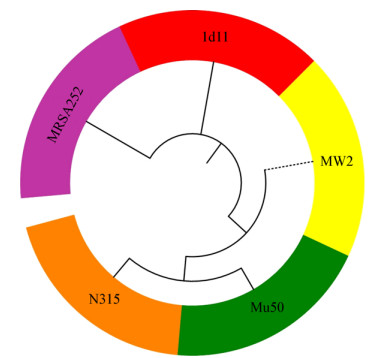
2.2 基因组基本特征概述将通过Illumina高通量测序技术得到的序列进行拼接组装,通过cge网络数据库(https://cge.cbs.dtu.dk/services/cge/)和Glimmer 3.02软件分析得出试验菌株ld11的基因数量为2 699个,基因总长度为2 408 730 bp,基因平均长度为892 bp,基因区域(G+C)mol%含量为33.4%,基因占基因组百分比为83%,遗传区域长度为494 727 bp,遗传区域的(G+C)mol%含量为28.8%,分离株ld11基因组和4株MRSA参考菌株基因组均携带多种不同的耐药基因(表 1),分离株ld11携带有氨基糖苷类耐药基因aadD、spc和str,β-内酰胺类耐药基因blaZ和mecA,酰胺醇类耐药基因cat(pC194),大环内酯类耐药基因erm(A),氟喹诺酮类耐药基因norA,以及四环素类耐药基因tet(k)和tet(M),分离株ld11携带耐药基因数较多于4种参考菌株(表 1)。对分离株ld11和4株人源MRSA参考基因组基于核心基因组的进化树构建结果如图 1所示,结果表明分离株ld11和参考菌株MRSA252亲缘关系较近,与参考菌株MW2、N315及Mu50亲缘关系相对较远。
| 菌株 Strains |
耐药基因 Drug resistance genes |
| ld11 | aadD, spc, str, blaZ, mecA, cat(pC194), erm(A), norA, tet(k), tet(M) |
| Mu50 | mecA, tet(M), norA, aadD, spc, erm(A) |
| MW2 | mecA, norA |
| N315 | mecA, norA, spc, aadD, erm(A) |
| MRSA252 | mecA, norA, blaZ, spc, aadD, erm(A) |
|
| 图 1 分离株ld11和4株人源MRSA参考菌株的核心基因组系统发育树 Figure 1 Phylogenetic tree of core genome of isolate ld11 and 4 human MRSA reference strains |
|
|
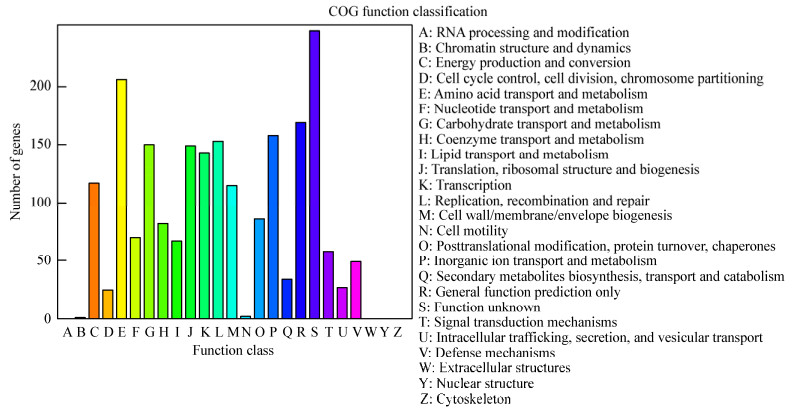
对试验菌株ld11的基因组进行COG功能分析、KEGG通路分析以及GO注释统计。COG功能分类显示,所有注释蛋白分布于21个条目中(图 2),其中具有较多功能未知的基因(S),其次碳水化合物的运输和代谢(G)、氨基酸转运和代谢(E)、无机离子转运和代谢(P)等维持菌体基本功能的相关基因占优势。KEGG通路分析结果显示,共计1 497条注释序列归类为157个通路分类条目中,最常见的6个通路依次是代谢通路(450,30.06%)、次生代谢物的生物合成通路(227,15.2%)、不同环境中的微生物代谢通路(133,8.9%)、氨基酸生物合成通路(109,7.3%)、ABC转运蛋白通路(97,6.7%)和碳代谢通路(86,5.7%)。GO注释结果显示,该基因组的所有注释序列被映射到3大类45个子功能条目中,总计10 801条注释序列分别归类到分子功能(2 614,24.2%)、细胞成分(2 723,25.2%)及生物学过程(5 464,50.6%)三大类中(图 3)。在GO分子功能分类中,与催化活性以及结合相关的注释序列较多,分别为1 252条序列和933条序列;在细胞成分分类中,细胞和细胞组分所包含的注释序列较多,均为727条序列;在生物学过程分类中,与代谢进程以及细胞进程相关的注释序列较多,分别为1 503条和1 337条。
|
| 图 2 ld11基因组注释序列COG功能分类图 Figure 2 The COG function classification of the genome annotation sequence of MRSA ld11 strain |
|
|

|
| 图 3 ld11基因组注释序列GO功能分类图 Figure 3 The GO function classification of the genome annotation sequence of MRSA ld11 strain |
|
|
通过分析软件对试验菌株基因组序列上的基因岛进行预测分析,结果显示从ld11基因组序列上共预测到4个基因岛(表 2),基因岛1的基因编码产物多是假定蛋白;基因岛2的基因编码产物多是假定蛋白和肠毒素;基因岛3的基因编码产物多是蛋白酶和调节因子;基因岛4的基因编码产物多是假定蛋白和转座酶。
| 基因岛编号 Gene island number |
序列长度 Sequence length (bp) |
基因数 Number of genes |
| 1 | 9 209 | 14 |
| 2 | 5 657 | 9 |
| 3 | 10 176 | 10 |
| 4 | 3 587 | 7 |
通过分析软件对试验菌株基因组序列上可能存在的CRISPR序列进行预测分析,分析结果显示,ld11基因组序列上存在9个疑似CRISPR序列,其中6个CRISPR在同一条序列上,该序列长度为882 317 bp,剩余的3个CRISPR在不同的序列上,序列长度分别为614 960、401 863、109 482 bp (表 3)。对试验菌株基因组序列上的前噬菌体序列进行预测分析,结果显示,ld11基因组序列含有一个完整的前噬菌体序列,序列长度为45.5 kb,位置在83 731–129 253 bp。
| 序列号 Sequence number |
序列长度 Sequence length (bp) |
CRISPR编号 CRISPR number |
重复序列 Repeat sequence |
间隔序列 Spacer sequence |
| NODE_1 | 882 317 | 1 | CTAAAGGTGAAGAGGAACGC AGTTGGATGCTA |
AGGCACAACTGCATAAAAGCC TCTAATCG |
| NODE_1 | 882 317 | 2 | CCAATTTCTACAGACAATGCA AGTTGG |
CGGGGCCCCAACATAGAGAAA TTGGAACG |
| NODE_1 | 882 317 | 3 | CCAACTTGCACATTATTGTAAG CTGACTTT |
CCGCCAGCTTCTGTGTTGGGG CCCCG |
| NODE_1 | 882 317 | 4 | CCCCAACTTGCATTGTCTGTAG AA |
ATTGGGAATCCAATTTCTCTTT GTTGGGGCCCAT |
| NODE_1 | 882 317 | 5 | CCCCTAACAACTAAAAGTTGT AAGG |
AAGGAGAGGGATACATGCGCC GAGCACATGCATAAAAT |
| NODE_1 | 882 317 | 6 | AAAGTCAGCTTACAATAATGT GCAAGTTGG |
CGGGGCCCCAACATAGAAGCT GGCGG |
| NODE_2 | 614 960 | 7 | GAAATTCTACAGGCAATGCGA GTTAGGGTG |
TGGGTCCCAACACAGAGAATT TCGAAAT |
| NODE_3 | 401 863 | 8 | TTTATGAATTGAGTATTAAATTT G |
AAAGTAAATTACAGTTCTATAG GTTTATAAAAATCAGTTTTTAA ATCAAAGCTA |
| NODE_6 | 109 482 | 9 | TGCATAAGAGCCCCTAACAAC TAAAGTT |
GAAAAATAGAGGAACACAGTT GGATGCTACGCATCAAC |
MRSA是一类重要的人畜共患病原菌,由MRSA感染导致的奶牛乳腺炎疾病给奶牛养殖业带来了巨大的经济损失[18]。MRSA的研究和防控一直是各国研究人员关注的重点,从MRSA的外部形态以及生化特征等表型鉴定,到后来基因组测序技术的应用等,研究人员对MRSA菌株的致病机理、流行病学特征等研究一直在逐步深入。本研究通过全基因组测序技术及数据库比对分析,对宁夏地区奶牛源MRSA流行株ld11的基因组基本特征进行了详细的概述。同时对分离株ld11进行了药敏试验,结果显示分离株ld11对9种抗菌药物均耐药(表 1),运用网络数据库对ld11基因组携带的耐药基因进行筛查,发现分离株ld11携带多种耐药基因,二者之间呈现出了很好的对应性。将分离株ld11和4株人源MRSA参考基因组进行对比分析,结果表明分离株ld11携带的耐药基因多于参考菌株携带的耐药基因,体现出了分离株ld11的耐药性较为严重。分离株ld11和参考菌株的核心基因组进化树结果显示分离株ld11和参考菌株MRSA252亲缘关系较近,值得关注。
COG数据库是通过对测序完的编码蛋白序列进行相互比较而构建的,通过COG数据库比对,可以对预测蛋白进行归类以及蛋白进化分析。本研究中菌株ld11的COG功能分析结果如图 2所示,除了功能未知的基因外,碳水化合物的运输和代谢(G)、氨基酸转运和代谢(E)、无机离子转运和代谢(P)等维持菌体基本功能的相关基因占优势,使菌体对周围环境具有较强的适应性,这与Kuroda等报道的人源MRSA基因组的基因功能预测结果[19]类似,值得注意的是其报道的人源MRSA基因功能预测结果中的RNA合成和修饰功能基因占有一定优势,而本研究中动物源MRSA ld11的RNA加工和修饰的功能基因占比较低,不同MRSA基因组功能基因之间的区别与联系有待进一步探讨和研究。KEGG是用于基因组分析的数据库,KEGG数据库中丰富的通路信息有助于从分子水平了解生物系统的高级功能和效用。本研究中菌株ld11基因组序列的KEGG通路分析结果显示属于代谢通路的基因占比最多,这可能与菌株自身的产毒性有关,菌体通过代谢可产生多种毒素和侵袭性酶,对细菌的致病性具有重大的作用。KEGG通路分析结果与之前的一株人源MRSA报道相似,略有不同的是本研究中KEGG通路分析注释序列较多[20]。GO注释更便于理解基因背后所代表的生物学意义,GO注释一般包括细胞成分、分子功能和生物学过程3个方面的内容。本研究通过对菌株基因组序列进行GO注释,注释结果显示与催化活性、细胞、细胞组分、结合、代谢进程以及细胞进程相关的注释序列较多,而这些优势序列通常与菌体的生长增殖以及各项生理活动密切相关,表明MRSA菌株ld11具有发达的新陈代谢能力和很强的传播能力,给人类带来巨大的危害和经济损失[21]。GO注释在个体的基因产物和本体论术语之间起到了一种连接作用,并在定量和定性信息之间扮演了桥梁的作用[22]。目前国内关于MRSA基因组序列信息GO注释分析的报道较少,需要加强补充分析。
基因岛通常包含不同的基因,可以编码多种功能蛋白,涉及多种共生关系,还可以提高生物的适应性。本研究从ld11基因组序列中预测到4个基因岛,基因岛内包含的基因产物大部分是假定蛋白,但是从其中的一个基因岛上发现了肠毒素基因的存在,肠毒素会给动物和人类脏器带来重大的损害,同时携带肠毒素的基因岛还能通过水平转移的方式传播到其他菌株,需加以重视。基因岛由于携带耐药基因或毒力基因通常又被称为“耐药岛”或“毒力岛”,MRSA菌株的产生主要就是因为其基因组序列中的葡萄球菌染色体mec基因岛(Staphylocossal cassestte chromosome mec,SCCmec)获得了外源性mecA基因,从而较大程度地加强了菌株的耐药性。对MRSA菌株基因组序列中基因岛的持续关注和研究,使我们能更好地在分子水平了解MRSA的多重耐药性和毒力性产生的原因,做好防控措施,减少MRSA感染带来的损失。
CRISPR是一串包含多个短而重复的碱基序列,重复序列之间是一些约30 bp左右的间隔序列,CRISPR序列在原核生物中起免疫保护的作用。本研究CRISPR分析结果显示,ld11基因组序列上存在9个疑似CRISPR序列,其中6个CRISPR在同一条序列上,剩余的3个CRISPR在不同的序列上。CRISPR序列的发现及CRISPR位点间间隔序列的变化可能对MRSA的毒力、耐药性传递和生物膜形成等生理学特性有影响,同时可以反映该菌株的进化历程,为研究该地区MRSA菌株间的亲缘关系提供参考依据[23-24]。前噬菌体是指病毒在宿主体内真正形成之前,以核酸的形式插入宿主基因组的阶段。本研究对ld11基因组序列上的前噬菌体序列进行预测分析,结果显示ld11基因组序列含有1个完整的前噬菌体序列。前噬菌体序列的存在可能会允许一些细菌获取抗生素抗性,增强对环境的适应性,提高粘附力或使细菌成为致病菌。近年来,国内外关于MRSA基因组中前噬菌体的报道也逐渐增多,不同地区报道的MRSA基因组携带的前噬菌体的分布种类可能有所不同,多个研究表明,前噬菌体种类分布与其所属克隆谱系均有密切关系[17, 25]。
目前,随着基因组测序技术的不断进步,第二、三代测序成本在不断降低,SA基因组的测序、数据分析已经成为当前研究的热点。对MRSA菌株进行高通量测序,利用高性能计算平台和生物信息学方法,为MRSA菌株基因组信息的解析、功能基因的挖掘、致病因子的预测等都提供了可靠的技术和方法的支持[26],同时还可以对包括基因组岛、CRISPR-Cas系统、前噬菌体在内的具有水平转移功能的可移动元件进行预测,这些可移动元件携带的基因可能赋予菌株特殊的抗性和代谢途径,对它们进行预测分析,有利于更全面地认识MRSA菌株基因组的特征,为MRSA的感染治疗和中长期防控提供理论依据。
| [1] |
Adnan SN, Ibrahim N, Yaacob WA. Disruption of methicillin-resistant Staphylococcus aureus protein synthesis by tannins[J]. Germs, 2017, 7(4): 186-192. DOI:10.18683/germs.2017.1125 |
| [2] |
Kim YK, Kim JS, Kim HS, et al. Molecular typing of Staphylococcus aureus isolated from blood on the basis of coagulase gene polymorphism and toxin genes[J]. The Korean Journal of Laboratory Medicine, 2008, 28(4): 286-292. DOI:10.3343/kjlm.2008.28.4.286 |
| [3] |
Montesinos I, Salido E, Delgado T, et al. Epidemiologic genotyping of methicillin-resistant Staphylococcus aureus by pulsed-field gel electrophoresis at a university hospital and comparison with antibiotyping and protein a and coagulase gene polymorphisms[J]. Journal of Clinical Microbiology, 2002, 40(6): 2119-2125. DOI:10.1128/JCM.40.6.2119-2125.2002 |
| [4] |
Vázquez-sánchez D, López-cabo M, Saá-Ibusquiza P, et al. Incidence and characterization of Staphylococcus aureus in fishery products marketed in Galicia (Northwest Spain)[J]. International Journal of Food Microbiology, 2012, 157(2): 286-296. |
| [5] |
Boucher HW, Corey GR. Epidemiology of methicillin-resistant Staphylococcus aureus[J]. Clinical Infectious Diseases, 2008, 45(2): S344-S349. |
| [6] |
Kérouanton A, Hennekinne JA, Letertre C, et al. Characterization of Staphylococcus aureus strains associated with food poisoning outbreaks in France[J]. International Journal of Food Microbiology, 2007, 115(3): 369-375. |
| [7] |
Tenover FC, Weigel LM, Appelbaum PC, et al. Vancomycin-resistant Staphylococcus aureus isolate from a patient in Pennsylvania[J]. Antimicrobial Agents and Chemotherapy, 2004, 48(1): 275-280. DOI:10.1128/AAC.48.1.275-280.2004 |
| [8] |
Harada D, Nakaminami H, Miyajima E, et al. Change in genotype of methicillin-resistant Staphylococcus aureus (MRSA) affects the antibiogram of hospital-acquired MRSA[J]. Journal of Infection and Chemotherapy, 2018, 24(7): 563-569. DOI:10.1016/j.jiac.2018.03.004 |
| [9] |
Kizerwetter-Świda M, Pławińska-Czarnak J. Livestock-associated strains of methicillin resistant Staphylococcus aureus (LA-MRSA)-the current state of knowledge[J]. Medycyna Weterynaryjna, 2017, 73(2): 92-98. DOI:10.21521/mw.5639 |
| [10] |
Devriese LA, Hommez J. Epidemiology of methicillin-resistant Staphylococcus aureus in dairy herds[J]. Research in Veterinary Science, 1975, 19(1): 23-27. |
| [11] |
Dhup V, Kearns AM, Pichon B, et al. First report of identification of livestock-associated MRSA ST9 in retail meat in England[J]. Epidemiology and Infection, 2015, 143(14): 2989-2992. DOI:10.1017/S0950268815000126 |
| [12] |
Peeters LEJ, Argudín MA, Azadikhah S, et al. Antimicrobial resistance and population structure of Staphylococcus aureus recovered from pigs farms[J]. Veterinary Microbiology, 2015, 180(1/2): 151-156. |
| [13] |
Fox A, Pichon B, Wilkinson H, et al. Detection and molecular characterization of Livestock-Associated MRSA in raw meat on retail sale in North West England[J]. Letters in Applied Microbiology, 2017, 64(3): 239-245. DOI:10.1111/lam.2017.64.issue-3 |
| [14] |
Liu YD, Wang H, Du N, et al. Molecular evidence for spread of two major methicillin-resistant Staphylococcus aureus clones with a unique geographic distribution in Chinese hospitals[J]. Antimicrobial Agents and Chemotherapy, 2009, 53(2): 512-518. DOI:10.1128/AAC.00804-08 |
| [15] |
Van Rijen MML, Bosch T, Verkade EJ, et al. Livestock-associated MRSA carriage in patients without direct contact with livestock[J]. PLoS One, 2014, 9(6): e100294. DOI:10.1371/journal.pone.0100294 |
| [16] |
Clinical and Laboratory Strandards Institute (CLSI). Performance Standards for Antimicrobial Disk and Dilution Susceptibility Tests for Bacteria Isolated from Animals[M]. 3rd ed. Wayne, PA: Clinical and Laboratory Strandards Institute, 2015
|
| [17] |
Li J. Epidemiological characteristics and phylogenetic analysis of pig associated methicillin-resistant Staphylococcus aureus[D]. Beijing: Doctoral Dissertation of China Agricultural University, 2017 (in Chinese) 李君.猪源耐甲氧西林金黄色葡萄球菌流行病学特征及遗传进化分析[D].北京: 中国农业大学博士学位论文, 2017 http://cdmd.cnki.com.cn/Article/CDMD-10019-1017164571.htm |
| [18] |
Neeraj S, Varsha S, Anju N, et al. Prevalence and characterization of methicillin-resistant Staphylococcus aureus (MRSA) mastitis in dairy cattle in Jabalpur, Madhya Pradesh[J]. Journal of Animal Research, 2017, 7(1): 77-84. DOI:10.5958/2277-940X.2017.00011.0 |
| [19] |
Kuroda M, Ohta T, Uchiyama I, et al. Whole genome sequencing of meticillin-resistant Staphylococcus aureus[J]. The Lancet, 2001, 357(9264): 1225-1240. DOI:10.1016/S0140-6736(00)04403-2 |
| [20] |
Yao WM, Chen Z, Wang HY, et al. Analysis of complete genome sequence of methicillin resistant Staphylococcus Aureus-MS4[J]. Shenzhen Journal of Integrated Traditional Chinese and Western Medicine, 2017, 27(15): 6-9. (in Chinese) 姚伟明, 陈重, 王红燕, 等. 一株耐甲氧西林金黄色葡萄球菌MS4的全基因组测序结果分析[J]. 深圳中西医结合杂志, 2017, 27(15): 6-9. |
| [21] |
Wang C. Primary screening of virulence genes of B. melitensis base on whole-genome sequencing[D]. Yinchuan: Master's Thesis of Ningxia Medical University, 2014 (in Chinese) 汪超.基于全基因组测序技术对B. melitensis毒力基因的初筛[D].银川: 宁夏医科大学硕士学位论文, 2014 http://cdmd.cnki.com.cn/Article/CDMD-10752-1015087802.htm |
| [22] |
Broadbent JA, Sampson DL, Broszczak DA, et al. Choose wisely: Network, ontology and annotation resources for the analysis of Staphylococcus aureus omics data[J]. International Journal of Medical Microbiology, 2015, 305(3): 339-347. DOI:10.1016/j.ijmm.2015.02.001 |
| [23] |
Zhang K, Chen JC, Li Y, et al. Application progress of CRISPR/Cas9 technology in microbiological research[J]. Microbiology China, 2018, 45(2): 451-464. (in Chinese) 张昆, 陈景超, 李祎, 等. CRISPR/Cas9技术在微生物研究中的应用进展[J]. 微生物学通报, 2018, 45(2): 451-464. |
| [24] |
Ye LN, Chen L, Qu D. CRISPR/Cas systems of Staphylococcus[J]. Journal of Microbes and Infections, 2017, 12(4): 254-262. (in Chinese) 叶立娜, 陈力, 瞿涤. 葡萄球菌CRISPR/Cas系统[J]. 微生物与感染, 2017, 12(4): 254-262. DOI:10.3969/j.issn.1673-6184.2017.04.013 |
| [25] |
Rahimi F, Bouzari M, Katouli M, et al. Prophage and antibiotic resistance profiles of methicillin-resistant Staphylococcus aureus strains in Iran[J]. Archives of Virology, 2012, 157(9): 1807-1811. DOI:10.1007/s00705-012-1361-4 |
| [26] |
Zhang R, Zhang CT. The impact of comparative genomics on infectious disease research[J]. Microbes and Infection, 2006, 8(6): 1613-1622. DOI:10.1016/j.micinf.2005.11.019 |
 2019, Vol. 46
2019, Vol. 46




