扩展功能
文章信息
- 吴欣欣, 吴晓刚, 马怡茗, 李冀, 吴巧玉, 隋维康, 张晓君
- WU Xin-Xin, WU Xiao-Gang, MA Yi-Ming, LI Ji, WU Qiao-Yu, SUI Wei-Kang, ZHANG Xiao-Jun
- 反应器进水管生物膜中喹啉降解菌的分离鉴定及降解特性
- Identification and characterization of quinoline-degrading bacterial strains isolated from the influent pipeline of a bioreactor
- 微生物学通报, 2019, 46(7): 1590-1596
- Microbiology China, 2019, 46(7): 1590-1596
- DOI: 10.13344/j.microbiol.china.181003
-
文章历史
- 收稿日期: 2018-12-07
- 接受日期: 2019-03-07
- 网络首发日期: 2019-04-24
喹啉(C9H7N)是一类无色吸湿高毒性液体,其化学结构由一个吡啶环与苯环组成。喹啉及其衍生物重要的人为途径来自于煤焦油加工,常在焦化废水中以污染物的形成存在,此外也常被用作溶剂、染料、杀虫剂及药物生产中间体等。据统计,全球每年加工煤焦油的量可达15×106 t,而其中喹啉含量占到0.2%-0.3%[1]。由于在环境中广泛存在,喹啉作为典型的难降解有机污染物引起了重要关注[2]。
微生物降解喹啉由于高效、经济且不易引起二次污染的特点而作为一种重要手段被应用。目前已经分离的喹啉降解菌以假单胞菌属(Pseudomonas)[3-4]为主,其它分布于芽孢杆菌属(Bacillus)[5]、红球菌属(Rhodococcus)[6]、黄杆菌属(Flavobacterium)、节细菌属(Arthrobacter)、丛毛单胞菌属(Comamonas)[7]、伯克氏菌属(Burkholderia)[8]、苍白杆菌属(Ochrobactrum)[9]、无色杆菌属(Achromobacter)[10]、食酸菌属(Acidovorax)和脱硫杆菌属(Desulfobacterium)[11-12],这些喹啉降解菌大多分离自石油污染的土壤及废水处理系统的活性污泥中。由于微生物不同种类具有不同的酶系,因此其降解途径也各不相同。通过对不同降解菌株的培养研究,目前发现的好氧降解途径有4种,分别称为5, 6-二羟基-2(1H)喹诺酮途径、7, 8-二羟基-2(1H)喹诺酮途径、邻氨基苯甲酸途径和8-羟基香豆素途径。降解途径虽然有差异,但初始反应都要先生成2-羟基喹啉,随后再发生不同的代谢过程[1]。
本实验室建立了一个喹啉反硝化降解生物反应器,体积为2 L,鞣性材料作为填料,种子污泥采集于上海某焦化厂废水处理系统的二沉池,人工合成废水成分为100 mg/L喹啉和240 mg/L NaNO3及140 mg/L K2HPO4·3H2O。反应器运行到稳定阶段,硝酸盐去除率达到90%,化学需氧量(Chemical oxygen demand,COD)、喹啉去除率均达到80%。经过长时间的监测发现,在管理不善的情况下,人工合成废水流经反应器进水管的过程中(约80 cm长,全程约15 min)喹啉降解率可达到60%。这导致了直接进入反应器内部的碳源浓度降低,影响到反应器微生物群落的发育;但另一方面,这时管壁生物膜的喹啉降解效率很高,其微生物资源值得研究。
本文从反应器进水管内附着的生物膜中筛选喹啉降解菌,获得了4株在好氧条件下以喹啉为唯一碳源的菌株,通过核酸序列同源性分析对其进行了系统发育地位鉴定,并比较了这些菌株在喹啉代谢特性上的差异及pH、温度等环境因素对底物降解的影响。
1 材料与方法 1.1 主要试剂、仪器及培养基喹啉(质量分数98%)和2-羟基喹啉(质量分数97%)均为色谱纯,国药集团化学试剂有限公司;甲醇为色谱纯,上海安普科技股份有限公司;其它试剂均为国产分析纯。超纯水的电阻率=18 MΩ/cm。
液相色谱仪、色谱柱,安捷伦科技有限公司;微孔板检测系统,Molecular Devices公司;PCR仪,Applied Biosystems公司。
喹啉无机盐培养基(MMQ,mg/L):K2HPO4·3H2O 570,KH2PO4 233,NH4Cl 26.75,NaCl 500,微量元素NaHCO3 168,MgSO4 120,CaCl2 54.4,Disodium EDTA 25,H3BO3 3.6,FeSO4·7H2O 1.5,CoCl2 1.2,Ni(NH4)2(SO4)2 1.2,Na2MoO4 0.94,Na2SeO4 0.26,MnSO4 0.2,ZnSO4 0.16,CuSO4 0.032。固体培养基加入15 g/L琼脂,调节pH为7.0,1×105 Pa灭菌30 min。待冷却至50 ℃左右后每100 mL培养基加入5 μL过滤除菌的喹啉母液,使喹啉终浓度为50 mg/L。
营养肉汤培养基(NB)配制方法见文献[13]。
1.2 细菌菌株分离以喹啉降解反应器进水管内壁收集的生物膜样品为菌源,接种在含50 mg/L的喹啉无机盐培养液中,30 ℃、150 r/min条件下培养24 h,得到富集的菌液。吸取1 mL富集菌液至9 mL无菌培养基中,得到10-1稀释度菌悬液,依次稀释至10-3。分别吸取10-1、10-2、10-3稀释度菌悬液100 μL至喹啉无机盐培养基固体平板,涂布均匀。于30 ℃恒温培养箱中静置培养2-5 d,直至长出明显的单菌落。挑取不同形态的菌落再进行多轮划线纯化,获得4株喹啉降解菌,分别命名为Q1、Q3、Q7和Q8。
1.3 16S rRNA基因测序及系统发育分析采用酚氯仿法[14]提取菌株基因组DNA。16S rRNA基因的扩增采用细菌通用引物27F (5′-AGAG TTTGATCCTGGCTCAG-3′)和1492R (5′-GGYTAC CTTGTTACGACTT-3′)。PCR反应体系(25 μL):10×Buffer 2.5 μL,MgCl2 (25 mmol/L) 2.0 μL,dNTPs (2.5 mmol/L) 2.0 μL,27F/1492R (12.5 μmol/L)各0.5 μL,Taq酶(5 U/μL) 0.2 μL,模板DNA (100 ng/μL) 1.0 μL,dd H2O补足25 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,56 ℃ 30 s,72 ℃ 1 min,20个循环;72 ℃ 8 min。PCR产物经琼脂糖凝胶电泳检测后送至英潍捷基(上海)有限公司测序。将测序结果通过NCBI网站(http://www.ncbi.nlm.nih.gov)进行同源序列搜索(BLAST),匹配出同源序列。选取代表性菌株序列,利用MEGA 5.0软件,采用邻接(Neighbor-Joining)法构建系统发育树。分离到的4株菌的16S rRNA基因序列提交至GenBank数据库,获得登录号分别为MK231748(Q1)、MK231749(Q3)、MK231750(Q7)和MK231751(Q8)。
1.4 喹啉降解实验 1.4.1 降解曲线供试菌株在NB液体培养基中扩大培养24 h后,4 000×g离心5 min,去上清,菌体沉淀用MMQ洗涤后离心去上清。所得菌体悬浮于300 μL MMQ中,按2% (体积分数)接种量接种至5 mL MMQ中,每组设置3个重复。30 ℃、150 r/min避光振荡培养,每隔6、12、24、48、72 h取样。14 000 r/min离心5 min收集上清,测定喹啉及其中间产物的浓度,利用微孔板检测系统测定细菌菌液的OD600值。
1.4.2 不同pH及温度对喹啉降解的影响以HCl或NaOH溶液分别调节喹啉无机盐培养基初始pH值为5.0、6.0、7.0、8.0和9.0,接种菌液后置于30 ℃、150 r/min振荡培养48 h;在pH为7.0的培养基中接种菌液,分别在20、25、30、35和40 ℃条件下150 r/min振荡培养48 h,测定喹啉浓度并计算降解率。
1.5 喹啉及其中间产物的定量分析高效液相色谱法(HPLC)测定喹啉及其中间产物,色谱柱:ZORBAX SB-C18 (4.6 mm×150 mm,5 μm);流动相为甲醇:水=60:40 (体积比);流速1 mL/min;检测波长225 nm;进样体积20 μL;柱温30 ℃。喹啉及2-羟基喹啉标准品的保留时间分别为4.28 min及2.57 min,以此作为产物判断依据。
2 结果与分析 2.1 分离菌株的分类地位对以喹啉为唯一碳源的4株分离菌株进行基因组DNA的提取,并完成16S rRNA基因测序,将得到的序列与GenBank中已知序列进行比对,构建的系统发育树如图 1所示。分离株Q1与鞘氨醇杆菌属(Sphingobium)中Sphingobium hydrophobicum C1的序列相似性为99.9%;分离株Q3与马赛菌属(Massilia)中Massilia oculi CCUG 43427A的序列相似性为99.6%;分离株Q7与红球菌属(Rhodococcus)中Rhodococcus pyridinivorans WZ026的序列相似性为99.9%;分离株Q8与发酵成对杆菌属(Dyadobacter)中Dyadobacter fermentans SW1-11的序列相似性为99.9%。

|
| 图 1 基于菌株16S rRNA基因序列构建的系统发育树 Figure 1 Phylogenetic tree based on 16S rRNA gene of isolates and reference sequences 注:圆括号表示序列号;每一结点表示分类学单元;分支长度表示该分支进化过程中的变化程度;距离标尺表示序列之间差异的数字. Note: GenBank accession numbers were given in parentheses; Each node represents the taxonomic unit; The branch length shows the extent of variation in the branch evolution; Distance figures indicate the difference between sequences. |
|
|
图 2显示了以喹啉为唯一碳源的无机盐培养液中,在特定条件下(喹啉浓度50 mg/L、pH 7.0、30 ℃、有氧),Q1等4株菌分别对喹啉(Quinoline)的降解曲线及其检测到的中间产物2-羟基喹啉(2-OH-Q)的积累情况。其中,菌株Q7对底物的降解速率最快,在6 h内完全消耗;而菌株Q1在前6 h内将喹啉全部转化为第一步产物2-羟基喹啉,直至24 h时中间产物被逐渐去除;Q3和Q8则在24 h后才快速代谢底物,并在48 h内完全去除,过程伴有少量的2-羟基喹啉积累。因此,除了菌株Q7以外,Q1、Q3和Q8都产生了2-羟基喹啉的积累,其中Q1的中间产物积累量最大,且最早产生积累现象。同时,随着底物的消耗,菌体生物量逐渐积累,当底物消耗完全时,菌体生长达到平台期。
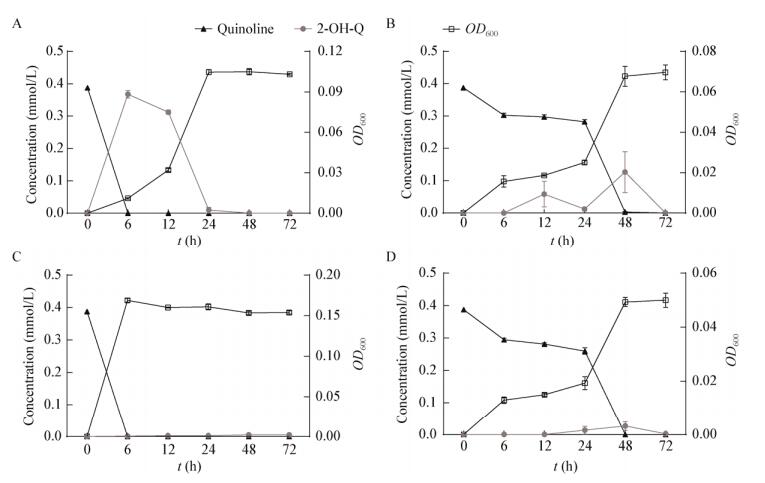
|
| 图 2 不同供试菌株对喹啉的降解动力学曲线 Figure 2 Dynamics of quinoline degradation by four isolates Note: A–D: Strain Q1, Q3, Q7 and Q8. |
|
|
4株菌降解喹啉的最适pH值范围均在7.0-8.0,且发现菌株Q3和Q8在pH低于6.0时降解能力受到抑制,菌株Q7在pH 5.0时底物利用受到抑制(图 3);值得注意的是,菌株Q1尽管在pH 5.0时仍有较高的喹啉去除率,但全部喹啉均转化成2-羟基喹啉积累于培养液中(结果未显示)。

|
| 图 3 不同pH条件下喹啉的去除率 Figure 3 Quinoline removal efficiency under different pH conditions |
|
|
所有4株菌对底物利用的最适温度为30 ℃,其中菌株Q7发挥降解作用的适宜温度范围较广,在20-40 ℃均有较高的降解率;菌株Q1适宜温度为20-35 ℃,不耐受高温;菌株Q8适宜温度为25-40 ℃,低温抑制了其降解能力;菌株Q3降解喹啉的适宜温度范围较为受限(25-35 ℃),低温和高温均不适宜其发挥作用(图 4)。综合比较,菌株Q7对逆境的耐受能力较其它3株喹啉降解菌略强。
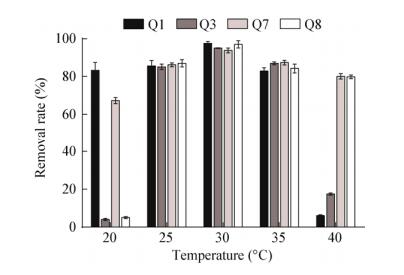
|
| 图 4 不同温度条件下喹啉的去除率 Figure 4 Quinoline removal efficiency under different temperature conditions |
|
|
本文发现缺氧喹啉降解生物反应器进水管内壁的生物膜形成会影响反应器的正常运行,该生物膜可发挥高效去除喹啉的作用。因此本研究采集了进水管内壁附着的生物膜,通过分离培养从中共获得了4株纯培养菌株,这些菌株在有氧条件下被证明具有稳定的喹啉利用能力,说明进水管内壁确实存在降解微生物,贡献了合成废水中部分喹啉的降解作用。分离喹啉降解菌的原始样本一般来源于污染土壤、废水及活性污泥中[2],而本研究从一个长年运行的反应器进水管累积的生物膜中尝试获取降解菌株,为纯培养重要价值菌株提供了新的来源。
本研究从反应器进水管生物膜筛选了一些新的喹啉降解菌类型。目前已知的喹啉降解菌如Pseudomonas aeruginosa KDQ4可在24 h内完全降解400 mg/L喹啉[15],Pseudomonas sp. BW003可在3-8 h内几乎完全降解192-911 mg/L喹啉[16],Rhodococcus sp. QL2可在8 h内去除150 mg/L喹啉[17],Brevundimonas sp. K4对100 mg/L的喹啉具有94.8%的去除率[18]。这些菌株对100 mg/L以上浓度的喹啉均有较好的处理效果,而本文测试了几株分离菌对50 mg/L喹啉的去除能力,因为在实际生产页岩油排放的废水中,喹啉的浓度一般为2-50 mg/L[1]。此外,洪璇、仲汇慧等此前从同一反应器中也分离出多株具有喹啉降解能力的菌株,主要属于Ochrobactrum、Bacillus、Pseudomonas和Rhodococcus等属[9, 19]。本文筛选的Sphingobium、Massilia和Dyadobacter属菌株从未被报道过具有喹啉降解功能。菌株Q1属于Sphingobium属,其中与Q1相似性最高的Sphingobium hydrophobicum是2016年于电子废弃物污泥中鉴定的一个种,具有电子垃圾降解能力且有很强的疏水性,可能与外层细胞膜上含有鞘糖脂(Sphingoglycolipid,SGL)有关[20]。尽管对于该物种的功能研究并不多,但Sphingobium属已被报道具有降解多种疏水性芳香族化合物的能力[21]。值得注意的是,Massilia和Dyadobacter属都不属于典型的污染物降解菌,比如Dyadobacter属的特征是在老化的培养物中产生细胞丝、色素及发酵葡萄糖的能力[22],但在本文中却发现其具有喹啉降解能力。Massilia属分离自眼炎病人体内,关于病原菌具有潜在污染物降解能力也曾被报道,如Burkholderia属作为能够引起马鼻疽的病原菌,同时也被发现具有高效的喹啉降解能力[23]。以上从反应器进水管生物膜中分离的菌株类型新颖,对完善喹啉生物降解机理研究具有实际意义。
本文筛选的4株菌表现出了不同的喹啉降解特性。喹啉生物转化过程中的一个主要特征是有色衍生物的生成[2],如Rhodococcus sp. QL2在降解喹啉过程中会出现培养液先变为粉红色,然后逐渐加深为棕红色[15],而在本实验对4株菌的培养全过程中未观察到任何有色产物的积累。其中Rhodococcus pyridinivorans Q7对喹啉的降解速率最快,且通过HPLC未检测到任何中间产物的积累,这与在Rhodococcus sp. QL2中发现有中间产物2-羟基喹啉、2, 6-二羟基喹啉和8-羟基香豆素的现象不一致[6, 24]。推测可能是由于菌株Q7对喹啉具有高效利用能力,中间产物被迅速完全消耗。而其它3株菌都检测到了不同程度的2-羟基喹啉化合物的积累,研究证实,尽管喹啉的生物降解途径多样,而2-羟基喹啉总是喹啉的第一步中间产物[2],而且2-羟基喹啉是喹啉降解过程产生的主要中间产物[15]。Sphingobium sp. Q1先将喹啉全部转化为2-羟基喹啉,再逐渐消耗积累的2-羟基喹啉,这一降解特性与复杂菌群降解喹啉采取的策略较为相似[25],只有当全部喹啉转化成2-羟基喹啉后,后者才会被进一步降解。也就是说,在喹啉和2-羟基喹啉同时存在的情况下,菌株Q1总是优先利用喹啉。Massilia oculi Q3和Dyadobacter fermentans Q8两株菌不同于一般的污染物降解菌,它们对喹啉的降解均存在较长的延滞期(约24 h),且检测到少量2-羟基喹啉积累。这一现象背后的具体原因尚不清楚,需进一步研究。此外,影响喹啉生物降解过程的因素有很多,其中温度和pH作为最重要的影响因素被广泛研究。从废水处理厂分离的一株Pseudomonas aeruginosa KDQ4降解喹啉的最适温度为33-38 ℃,最佳pH值范围为8.0-9.0[13];Pseudomonas sp. BW003降解喹啉的最适条件为30 ℃和pH 8.0[14];Brevundimonas sp. K4对喹啉降解的最适温度和pH值分别为30 ℃和9.0[16];一株Acidovorax sp.利用喹啉的最适温度为35 ℃,pH值范围为8.0-10.0[26]。本文分离的4株菌发挥喹啉降解作用的最适温度范围为25-35 ℃,pH范围为7.0-9.0,与已有研究结果基本一致。但值得注意的是,4株菌在25 ℃常温条件下仍具备80%以上的喹啉去除率,尤其是菌株Sphingobium sp. Q1和Rhodococcus sp. Q7在20℃也能发挥60%以上的降解率,有应用于常温降解喹啉的潜力,可降低污染物处理的成本。
综上所述,本文对分离自反硝化喹啉降解生物反应器进水管道生物膜的4株菌进行了鉴定,并初步探究了降解过程中的重要影响因素。但工业排放废水中可能存在多种污染物并存、盐度不同等复杂情况,因此在实际应用时仍需考虑具体情况,对菌种特性进行深入研究。总之,本文获得的新的喹啉降解菌种资源各具特性,具有潜在的应用价值。
| [1] |
Fetzner S. Bacterial degradation of pyridine, indole, quinoline, and their derivatives under different redox conditions[J]. Applied Microbiology and Biotechnology, 1998, 49(3): 237-250. DOI:10.1007/s002530051164 |
| [2] |
Felczak A, Zawadzka K, Lisowska K. Efficient biodegradation of quinolone – Factors determining the process[J]. International Biodeterioration & Biodegradation, 2014, 96: 127-134. |
| [3] |
Qiao L, Wang JL. Biodegradation characteristics of quinoline by Pseudomonas putida[J]. Bioresource Technology, 2010, 101(19): 7683-7686. DOI:10.1016/j.biortech.2010.05.026 |
| [4] |
Bai YH, Sun QH, Zhao C, et al. Quinoline biodegradation and its nitrogen transformation pathway by a Pseudomonas sp. strain[J]. Biodegradation, 2010, 21(3): 335-344. DOI:10.1007/s10532-009-9304-9 |
| [5] |
Tuo BH, Yan JB, Fan BA, et al. Biodegradation characteristics and bioaugmentation potential of a novel quinoline-degrading strain of Bacillus sp. isolated from petroleum-contaminated soil[J]. Bioresource Technology, 2012, 107: 55-60. DOI:10.1016/j.biortech.2011.12.114 |
| [6] |
Zhu SN, Liu DQ, Fan L, et al. Degradation of quinoline by Rhodococcus sp. QL2 isolated from activated sludge[J]. Journal of Hazardous Materials, 2008, 160(2/3): 289-294. |
| [7] |
Cui MC, Chen FZ, Fu JM, et al. Microbial metabolism of quinoline by Comamonas sp.[J]. World Journal of Microbiology and Biotechnology, 2004, 20(6): 539-543. DOI:10.1023/B:WIBI.0000043149.61562.3f |
| [8] |
Wang JL, Wu WZ, Zhao X. Microbial degradation of quinoline: kinetics study with Burkholderia picekttii[J]. Biomedical and Environmental Sciences, 2004, 17(1): 21-26. |
| [9] |
Hong X, Zhang XJ, Meng ZQ, et al. Isolation and characteristics of quinoline degrading bacteria from wastewater denitrifying bioreactor[J]. Chinese Journal of Applied & Environmental Biology, 2008, 14(6): 803-808. (in Chinese) 洪璇, 张晓君, 孟智奇, 等. 废水反硝化生物反应器中喹啉降解细菌的分离与特性[J]. 应用与环境生物学报, 2008, 14(6): 803-808. |
| [10] |
Deng XQ, Ren Y, Wei CH, et al. Isolation and characterization of Achromobacter sp. DN-06 for degradation of pyridine and quinoline[A]//Proceedings of the 2009 Annual Conference of the Chinese Society of Environmental Sciences (Volume IV)[C] Wuhan: Chinese Society for Environmental Sciences, 2009 (in Chinese) 邓秀琼, 任源, 韦朝海, 等.降解吡啶与喹啉的Achromobacter sp. DN-06筛选、鉴定与降解特性研究[A]//中国环境科学学会2009年学术年会论文集(第4卷)[C].武汉: 中国环境科学学会, 2009 |
| [11] |
Johansen SS, Licht D, Arvin E, et al. Metabolic pathways of quinoline, indole and their methylated analogs by Desulfobacterium indolicum (DSM 3383)[J]. Applied Microbiology and Biotechnology, 1997, 47(3): 292-300. DOI:10.1007/s002530050929 |
| [12] |
Licht D, Johansen SS, Arvin E, et al. Transformation of indole and quinoline by Desulfobacterium indolicum (DSM 3383)[J]. Applied Microbiology and Biotechnology, 1997, 47(2): 167-172. DOI:10.1007/s002530050907 |
| [13] |
Mao YJ, Zhang XJ, Zhang BR, et al. Specific-PCR and denaturing gradient gel electrophoresis assistant isolation of Thauera spp. from a coking wastewater treatment plant[J]. Acta Microbiologica Sinica, 2008, 48(12): 1634-1641. (in Chinese) 毛跃建, 张晓君, 张宝让, 等. 专一性PCR和变性梯度胶电泳协助从焦化废水处理装置中分离优势功能菌Thauera属菌株[J]. 微生物学报, 2008, 48(12): 1634-1641. DOI:10.3321/j.issn:0001-6209.2008.12.011 |
| [14] |
Renshaw MA, Olds BP, Jerde CL, et al. The room temperature preservation of filtered environmental DNA samples and assimilation into a phenol-chloroform-isoamyl alcohol DNA extraction[J]. Molecular Ecology Resources, 2015, 15(1): 168-176. DOI:10.1111/men.2014.15.issue-1 |
| [15] |
Zhang PH, Jia R, Zhang YX, et al. Quinoline-degrading strain Pseudomonas aeruginosa KDQ4 isolated from coking activated sludge is capable of the simultaneous removal of phenol in a dual substrate system[J]. Journal of Environmental Science and Health, Part A, 2016, 51(13): 1139-1148. DOI:10.1080/10934529.2016.1206377 |
| [16] |
Sun QH, Bai YH, Zhao C, et al. Aerobic biodegradation characteristics and metabolic products of quinoline by a Pseudomonas strain[J]. Bioresource Technology, 2009, 100(21): 5030-5036. DOI:10.1016/j.biortech.2009.05.044 |
| [17] |
Zhu SN, Liu DQ, Fan L, et al. Isolation, identification and degradation characteristics of a quinoline-degrading bacterium Rhodococcus sp. QL2[J]. Environmental Science, 2008, 29(2): 488-493. (in Chinese) 朱顺妮, 刘冬启, 樊丽, 等. 喹啉降解菌Rhodococcus sp. QL2的分离鉴定及降解特性[J]. 环境科学, 2008, 29(2): 488-493. |
| [18] |
Wang CR, Zhang MR, Cheng FL, et al. Biodegradation characterization and immobilized strains' potential for quinoline degradation by Brevundimonas sp. K4 isolated from activated sludge of coking wastewater[J]. Bioscience, Biotechnology, and Biochemistry, 2015, 79(1): 164-170. DOI:10.1080/09168451.2014.952615 |
| [19] |
Zhong HH, Zhang XJ, Yue SQ, et al. Analysis for metabolic activity of predominant bacterial isolates in a lab-scale denitrifying bioreactor for quinoline-degradation[J]. Microbiology China, 2010, 37(5): 645-650. (in Chinese) 仲汇慧, 张晓君, 岳思青, 等. 喹啉废水反硝化反应器中优势菌的代谢功能分析[J]. 微生物学通报, 2010, 37(5): 645-650. |
| [20] |
Chen XJ, Wang HJ, Xu JJ, et al. Sphingobium hydrophobicum sp. nov., a hydrophobic bacterium isolated from electronic-waste- contaminated sediment[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(10): 3912-3916. DOI:10.1099/ijsem.0.001287 |
| [21] |
Zhang XY, Peng X, Masai E. Recent advances in Sphingobium sp. SYK-6 for lignin aromatic compounds degradation—a review[J]. Acta Microbiologica Sinica, 2014, 54(8): 854-867. (in Chinese) 张晓琰, 彭学, 政井英司. 木质素芳香族化合物降解菌Sphingobium sp. SYK-6的研究进展[J]. 微生物学报, 2014, 54(8): 854-867. |
| [22] |
Lang E, Lapidus A, Chertkov O, et al. Complete genome sequence of Dyadobacter fermentans type strain (NS114T)[J]. Standards in Genomic Sciences, 2009, 1(2): 133-140. DOI:10.4056/sigs.19262 |
| [23] |
Wang JL, Han LP, Shi HC, et al. Biodegradation of quinoline by gel immobilized Burkholderia sp.[J]. Chemosphere, 2001, 44(5): 1041-1046. DOI:10.1016/S0045-6535(00)00469-0 |
| [24] |
Zhu SN, Fan L, Ni JR. Metabolic pathway analysis of two quinoline-degrading bacterial strains[J]. China Environmental Science, 2008, 28(5): 456-460. (in Chinese) 朱顺妮, 樊丽, 倪晋仁. 两株喹啉降解菌代谢途径的分析[J]. 中国环境科学, 2008, 28(5): 456-460. DOI:10.3321/j.issn:1000-6923.2008.05.014 |
| [25] |
Bai Q, Yang LH, Li RJ, et al. Accelerating quinoline biodegradation and oxidation with endogenous electron donors[J]. Environmental Science & Technology, 2015, 49(19): 11536-11542. |
| [26] |
Li J, Li WY. Screening of a highly efficient quinoline-degrading strain and its enhanced biotreatment on coking waste water[J]. Environmental Science, 2015, 36(4): 1385-1391. (in Chinese) 李静, 李文英. 喹啉降解菌筛选及其对焦化废水强化处理[J]. 环境科学, 2015, 36(4): 1385-1391. |
 2019, Vol. 46
2019, Vol. 46




