扩展功能
文章信息
- 田晓荣, 于敏, 李蓓, 杜瑞, 张静静, 张晓华
- TIAN Xiao-Rong, YU Min, LI Bei, DU Rui, ZHANG Jing-Jing, ZHANG Xiao-Hua
- 青岛近海沉积物生物被膜中密度感应及密度感应淬灭细菌多样性
- Diversity of quorum sensing and quorum quenching bacteria in sediment biofilm of Qingdao offshore
- 微生物学通报, 2019, 46(6): 1272-1281
- Microbiology China, 2019, 46(6): 1272-1281
- DOI: 10.13344/j.microbiol.china.180543
-
文章历史
- 收稿日期: 2018-07-14
- 接受日期: 2018-10-11
- 网络首发日期: 2018-12-14
2. 青岛海洋科学与技术国家实验室 海洋生态与环境科学功能实验室 山东 青岛 266237
2. Laboratory for Marine Ecology and Environmental Science, Qingdao National Laboratory for Marine Science and Technology, Qingdao, Shandong 266237, China
密度感应(Quorum sensing,QS)是广泛存在于细菌中的一种交流方式[1],细菌在生长过程中能够不断产生一些化学信号分子并分泌到周围环境中,通过感应这些化学信号分子的浓度,细菌可以监测菌群的数量,调控某些相关基因的表达[2]。在众多的QS系统中,酰基高丝氨酸内酯(Acyl-homoserine lactone,AHL)类的QS调控机理是目前研究最为透彻的。典型的AHL类信号分子由一个高丝氨酸内酯环和酰基侧链组成[3]。近年来,随着环境中QS信号分子的发现,密度感应现象已经开始从单个细菌延伸到了复杂多变的生态环境中。在海洋环境中,QS信号分子最容易存在于细菌密度很高的微生境中,例如动物的发光器官、微生物席、生物被膜以及颗粒有机碳(Particulate organic carbon,POC)等[4]。目前从海雪[5-6]、海绵[7]、刺细胞动物[8]以及海洋潮汐生物被膜[9]等环境中都成功分离得到了具有AHL合成能力的菌株。
密度感应淬灭(Quorum quenching,QQ)是指通过干扰信号分子的产生、释放、积累或应答,从而达到阻抑密度感应通路的作用。QQ现象的检测与QS正好相反,通过检测AHL类信号分子浓度的降低来检测是否具有QQ现象。相比QS,目前对海洋环境中QQ的作用还未开展广泛的研究。一些研究也表明QQ现象在海洋环境中是非常丰富的,例如Hmelo等[4]发现AHL信号分子在天然海水中降解的速度比人工海水快很多,其原因主要在于QQ的作用。
QS和QQ现象与细菌生物被膜关系非常密切。尽管近年来在海洋环境中QS和QQ现象相继被发现,然而对于海洋环境中天然生物被膜的QS和QQ现象却鲜有报道。我们对海洋沉积物表层生物被膜这个微生物密集热点区域的QS和QQ现象更是知之甚少。在海洋环境中浮游植物碎屑、浮游动物尸体残骸以及海雪等有机质通过沉降为沉积物表层输送了大量的有机质[10-11]。在这个特殊的区域,微生物群落系统具有非常高的密度和多样性,并且与海水的物质交换尤为密切[11]。探究该区域QS和QQ菌株的多样性可为了解QS现象在海洋环境中的生态学意义提供参考意见。
本研究中,我们以青岛近海表层沉积物生物被膜为研究对象,利用海水琼脂培养基2216E从生物被膜中分离获得可培养细菌,采用平板交互划线法和高通量QQ菌株筛选法分别筛选具有AHL类信号分子合成和降解能力的菌株,并利用细菌16S rRNA基因构建系统发育树,从而揭示该生境中QS和QQ细菌的多样性和丰度。
1 材料与方法 1.1 材料 1.1.1 主要试剂和仪器 EDTA和抗生素购自上海索莱宝生物科技有限公司;X-Gal购自上海源叶生物科技有限公司;C12-HSL和C6-HSL购自Sigma公司;16S rRNA基因扩增引物由铂尚生物技术有限公司合成;PBS缓冲液:Na2HPO4∙12H2O 2.9 g,NaH2PO4 0.3 g,NaCl 8.5 g,ddH2O定容至1 L,用NaOH调pH至7.2−7.4,1×105 Pa灭菌30 min,4 ℃保存。PCR仪,Applied Biosystems公司。 1.1.2 培养基 LB培养基的配制参照文献[12];海水营养培养基2216E参照《海洋微生物学》(第2版)[13]配制;AT培养基的配制参照文献[12];抗生素的终浓度为壮观霉素50 mg/L、四环素4.5 mg/L。 1.1.3 菌株和培养条件 从青岛近海沉积物表层生物被膜所分离到的所有菌株,均采用海水营养培养基2216E进行培养和分离纯化;用于高通量筛选具有AHL类信号分子降解能力的菌株采用AT培养基培养筛选。报告菌株Agrobacterium tumefaciens A136 (pCF218/pCF372)[14-15]可检测酰基侧链长度为C4−C14的AHL信号分子,其常规培养条件为LB培养基(壮观霉素:50 mg/L;四环素:4.5 mg/L),28 ℃。该菌由美国德克萨斯州立大学Robert JC. McLean教授惠赠,其工作原理为外源质粒pCF218过量表达AHL受体蛋白TraR,AHL-TraR复合体与质粒pCF372的启动子结合,诱导质粒下游lacZ基因过量表达β-半乳糖苷酶。因此,A136可通过检测β-半乳糖苷酶的活性,指示其他菌株合成分泌AHLs的能力。
1.2 样品采集2017年7月中旬在青岛近海海滩(120°33′E,36°07′N),选取未被人为破坏的潮间带泥质沉积物,用高温灭菌棉签蘸取沉积物表层天然生物被膜(图 1),立即带回实验室用无菌生理盐水冲洗下棉签上的样品,将得到的细菌悬液按照100−105倍稀释涂布在2216E平板上,28 ℃培养箱中培养2 d。

|
| 图 1 青岛近海栈桥泥质表层沉积物生物被膜 Figure 1 Biofilm of surface sediment in Qingdao offshore |
|
|
选取菌落数目合适的2216E平板,采取三区划线的方式进行细菌的分离纯化,28 ℃培养。连续纯化3次,当菌落形态单一时,挑取单克隆在斜面上进行划线培养,待菌落长出后,使用含有15%甘油的生理盐水重悬后保存于−80 ℃超低温冰箱中。
1.4 DNA的提取和16S rRNA基因鉴定采用煮沸法[16]提取细菌基因组DNA。采用标准的PCR[17]方法,使用通用引物B8F (5′-AGAGT TTGATCCTGGCTCAG-3′)和B1510R (5′-GGTTAC CTTGTTACGACTT-3′)[18]对细菌的16S rRNA基因进行扩增。将扩增成功的16S rRNA基因用限制性核酸内切酶BamHⅠ和Hae Ⅲ进行双酶切,选取酶切图谱不同的PCR产物送至青岛华大基因有限公司测序,将测序结果提交至微生物标准数据库EzBioCloud (https://www.ezbiocloud.net/)[19]以及NCBI中进行BLAST同源性序列比对,以16S rRNA基因序列相似性≥98.65%作为同种菌株的划分标准确定细菌的分类地位[20]。
1.5 具有密度感应(QS)活性菌株的筛选以根癌农杆菌A136 (pCF218/pCF372)菌株为报告菌,采用平板交互划线法[21]检测待测菌株是否具有AHL类信号分子的合成能力,如图 2A所示。
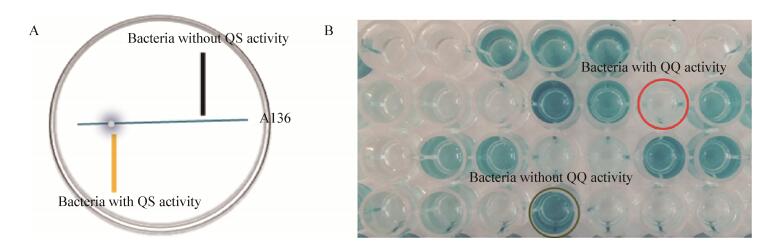
|
| 图 2 QS (A)和QQ (B)活性菌株的筛选方法 Figure 2 The screening methods of bacteria with QS (A) and QQ (B) activities |
|
|
将活化好的待测菌与A136在固体培养基上画线共培养,二者呈T字形,相近但不接触。在28 ℃培养24 h,随后覆盖一层含终浓度为100 μmol/L X-Gal的水琼脂(10 g琼脂,1 L蒸馏水),室温静置12 h。阳性细菌所产生的AHL类信号分子可以通过琼脂扩散被AHL报告菌A136所感应,通过观察两菌交互点附近A136的颜色变化推知AHL类信号分子的存在。报告菌显蓝色即为AHL合成阳性。阳性对照菌为已知具有AHL合成能力的哈维氏弧菌BB120[22]。
1.6 具有密度感应淬灭(QQ)活性菌株的筛选采用Tang等[12]建立的密度感应淬灭细菌高通量筛选方法进行QQ活性菌株的筛选。如图 2B所示,培养基为AT培养基,温度为28 ℃。
将分离得到的83株待测菌株接种于海水2216E液体培养基中,28 ℃、170 r/min培养24 h。178 μL菌液与2 μL C12-HSL或C6-HSL (1 mmol/L)、20 μL PIPES (1 mol/L,pH 6.7)混匀,28 ℃孵育24 h后4 ℃、12 000 r/min离心5 min,取无菌上清10 μL,加入到含有190 μL AT-A136检测液的96孔板中充分混匀。在28 ℃显色24 h后用酶标仪检测492 nm和630 nm的吸光度,按照公式计算β-半乳糖苷酶活性。其中阳性对照菌为具有较强AHL降解能力的菌株Th120[13],阴性对照为PBS缓冲液。
标准化的β-半乳糖苷酶活性= 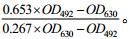
分别将所有具有AHL类信号分子降解能力和合成能力的菌株与其近缘菌株的16S rRNA基因序列利用软件MEGA 7.0以邻接法(Neighbor-Joining)构建系统发育进化树,1 000次自展(Bootstrap)来评估进化树的准确性。
2 结果与分析 2.1 细菌分离及菌种鉴定从青岛近海沉积物表层生物被膜中共分离得到83株54种可培养细菌,结果如图 3所示。这些细菌可以分为4个门类,包括变形菌门(63株,36种),拟杆菌门(14株,10种),厚壁菌门(5株,5种),放线菌门(1株,1种)。63株变形菌门细菌主要分属于γ-变形菌纲(29株,21种)和α-变形菌纲(34株,15种)。29株γ-变形菌纲的细菌包括弧菌目(12株,7种);假单胞菌目(8株,7种);交替单胞菌目(8株,7种)。34株α-变形菌纲细菌全部属于红杆菌目。14株拟杆菌门细菌分属于黄杆菌纲和鞘脂杆菌纲,黄杆菌纲细菌共13株,分属于7个种;鞘脂杆菌纲1株,该菌具有较强的纤维素降解能力。厚壁菌门的5株细菌都属于芽孢杆菌。值得一提的是,弧菌属和鲁杰氏菌属细菌数量最多,分别有12株和29株。此外共分离获得2株潜在细菌新种,菌株TXR405的16S rRNA基因序列与最相似菌株Seonamhaeicola algicola的相似性为97.1%;TXR437的16S rRNA基因序列与最相似菌株Maribacter polysiphoniae的相似性为95.8%。它们是否为细菌新种,仍需进一步生理生化实验进行分类鉴定。
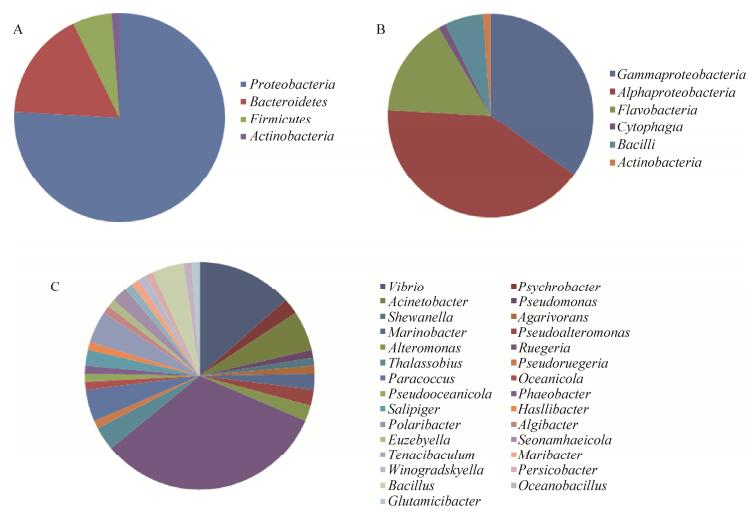
|
| 图 3 表层沉积物生物被膜细菌在门(A)、纲(B)、属(C)水平上的微生物群落组成 Figure 3 Microbial community composition of surface sediment biofilm at phylum (A), class (B) and genus (C) level |
|
|
利用AHL报告菌A136采用平板交互划线法对具有AHL合成能力的细菌进行筛选,得到具有AHL类信号分子合成能力的菌株38株,占所有菌株的45.8%。从门/纲的水平分析,它们主要分属于α-变形菌纲和γ-变形菌纲,只有一株TXR5161属于拟杆菌门的黄杆菌纲。从属水平来看,它们分别属于弧菌属、鲁杰氏菌属、嗜琼胶菌属、不动杆菌属、浮球菌属、海洋生菌属以及Thalassobius、Hasllibacter和Winogradskyella,并且以弧菌属和鲁杰氏菌属的细菌居多,分别有9株和19株。其中嗜琼胶菌属、Hasllibacter、Winogradskyella菌株的AHL信号分子合成能力为首次发现。选取本研究筛选到的所有能够合成AHL类信号分子的菌株与参考菌株利用软件MEGA 7.0构建系统发育进化树,如图 4所示。
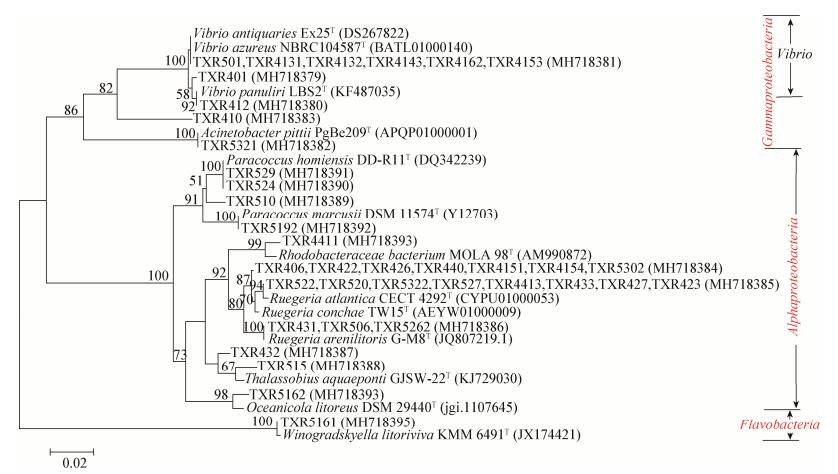
|
| 图 4 具有QS活性的菌株与参考菌株的系统进化树 Figure 4 Phylogenetic tree of bacteria with QS activity and their reference taxa 注:括号中的序号表示菌株的GenBank登录号;标尺:核苷酸的替代率为0.02;各分支点显示的数字代表通过1 000次重复运算得到的置信值. Note: The serial number in brackets denotes the GenBank accession number of the strain; Scale bar: The nucleotide substitution rate was 0.02; Bootstrap values (expressed as percentages of 1 000 replications) are shown at branch points. |
|
|
根据Tang等[12]建立的高通量筛选QQ细菌的方法及公式计算β-半乳糖苷酶活性。与阴性对照比较,β-半乳糖苷酶活性降低即为具有AHL降解能力的菌株。本研究以两种不同长度酰基侧链的信号分子为底物(C6-HSL,C12-HSL),发现在所筛选菌株中,能够降解长链信号分子(C12-HSL)并使β-半乳糖苷酶活性降低程度达到40%及以上的菌株有57株,占所有菌株的68.7%。它们在变形菌门、拟杆菌门、厚壁菌门以及放线菌门中均有分布。同时具有降解短链(C6-HSL)和长链信号分子(C12-HSL)能力的细菌有5株,它们是Alteromonas TXR425,鲁杰氏菌属TXR442和TXR503,黄杆菌属TXR5231,以及Maribacter的TXR437,并且TXR437为一株潜在海洋新菌。既能合成AHLs又能降解AHLs的菌株有24株,它们主要分布在变形菌门中,分属于鲁杰氏菌属、浮球菌属、弧菌属、不动杆菌属以及拟杆菌门中的Winogradskyella。此外,发现Glutamicibacter和微球菌科的菌株具有AHL类信号分子的降解能力。选取本研究中筛选到的所有具有AHL降解能力的菌株与参考菌株,利用软件MEGA 7.0构建系统发育进化树,如图 5所示。

|
| 图 5 具有QQ活性的菌株与参考菌株的系统进化树 Figure 5 Phylogenetic tree of bacteria with QQ activity and their reference taxa 注:●表示既具有QQ活性,又能够合成AHL类信号分子的菌株;括号中的序号表示菌株的GenBank登录号;标尺:核苷酸的替代率为0.05;各分支点显示出来的数字代表通过1 000次重复运算得到的置信值. Note: AHLs were identified from the isolates marked by ●; The number in brackets denotes the GenBank accession number of the strain; Scale bar: The nucleotide substitution rate was 0.05; Bootstrap values (expressed as percentages of 1 000 replications) are shown at branch points. |
|
|
本研究共从青岛近海表层沉积物生物被膜中分离到83株海洋细菌,主要分为4个门类:变形菌门、黄杆菌门、厚壁菌门和放线菌门,而且大部分为典型的海洋细菌,这一点符合海洋环境中细菌多样性的分布特征[5, 7]。变形菌门细菌(63株)占据绝对优势,比例高达75.9%,分属于γ-变形菌纲(29株)和α-变形菌纲(34株)。在γ-变形菌纲细菌中,弧菌属占有很高的比例,共有12株。在α-变形菌纲细菌中,鲁杰氏菌占据绝对优势,共有20株。该结果与香港渔场潮汐生物膜中分离菌株的结果相似[9],证明了弧菌和鲁杰氏菌属细菌为该环境中的优势菌属。丰度仅次于变形菌门的是拟杆菌门细菌,它们大部分属于黄杆菌科,其中有一株属于鞘脂杆菌纲,在分离过程中发现其具有极强的纤维素降解能力。除了这些优势种属,我们还分离得到了一些厚壁菌门和放线菌门细菌以及2株潜在新菌。海洋天然生物被膜细菌的分离纯化结果展示了一种高度多样且非常有特点的微型生态系统。
海洋天然生物被膜内如此高的细菌多样性,为筛选具有AHL合成及降解能力的细菌提供了很好的材料。通过使用A136平板交互划线的方法,我们共筛选到38株具有AHL类信号分子合成能力的菌株,占所有菌株的45.8%,这个比例比前人的研究结果[5-9]更高,究其原因,我们认为沉积物表层生物被膜这个特殊的生境中较高的微生物丰度和多样性使得细菌之间的相互交流更为密切,QS在该环境中发挥的作用更大。该38株菌主要分布在两个门:变形菌门(37株)和拟杆菌门(1株)。其中γ-变形菌纲11株,5种;α-变形菌纲26株,10种。变形菌门的嗜琼胶菌属和Hasllibacter的AHL类信号分子合成能力为首次报道。在γ-变形菌纲中占很高比例的弧菌属(9株)以及α-变形菌纲中的鲁杰氏菌属(19株)也组成了QS菌株的主要来源,这两个属的菌株在QS菌株中所占的比例高达74%。这两个属的细菌,也是目前在海洋环境中报道较多的AHL类信号分子产生菌[23]。变形菌门细菌是QS信号分子的主要来源[24],弧菌是研究最早的QS信号分子产生菌。而鲁杰氏菌属于MRC细菌(Marine Roseobacter clade)的类群,是近年在QS和QQ领域研究较多的细菌[25]。截至目前,已报道的拟杆菌门QS菌株非常少。本文报道了一株拟杆菌门中Winogradskyella TXR5161的AHL类信号分子合成能力。实验室培养条件下QS与生物被膜关系的研究非常多,而在这种天然生物被膜环境中QS的作用机制尚不明确[26]。有研究表明,QS是微生物表面定殖的重要调节机制,还可以参与调节生物被膜中微生物的相互作用[27]。在环境中很多重要的机制都与QS有关,例如氨氧化、硝化氧化、反硝化、硫氧化作用等[28-31]。目前有报道称超过80%的MRC细菌都拥有QS调节系统[32-33],并且有研究者猜测被海水淹没的表层沉积物可能会作为一种有机营养物质来源,从而吸引MRC细菌并支持其生长到产生QS的阶段[34],这也解释了为何本研究中从表层沉积物生物被膜中分离到的MRC细菌——鲁杰氏菌如此之多。在天然生物被膜中,QS菌株占比很高,远远超过了海水、海雪、海绵等地,暗示了细菌可能利用QS作为一种生存竞争武器,使自己在如此高密度的环境中占据一定的优势。
通过高通量筛选方法对具有AHL降解能力菌株的筛选结果来看,QQ在天然生物被膜中所占比例非常高。此前Saurav等[35-36]从红海和地中海地区收集的海绵提取物进行了细菌分离及QQ活性菌株的筛选,结果显示20%的菌株表现出了QQ活性。在大堡礁的一项研究表明,在284个海洋生物的提取物中,有23%的菌株表现出降解QS信号分子的能力。Romero等[37]曾从166株海洋微生物中分离得到24株具有短链AHL降解能力的细菌。而在本研究中,我们发现具有长链AHL信号分子降解能力的菌株更为普遍,其中β半乳糖苷酶活性降低程度达到40%及以上的有57株,占所有菌株的68.7%;具有短链信号分子C6-HSL降解能力的菌株有5株。这个结果与Muras等[38]对海水中604株自由生活细菌的筛选结果类似。Huang等[9]通过对生物被膜形成过程中信号分子的检测,发现在成熟生物被膜中,以长链信号分子居多。此外也有研究表明,短链AHL信号分子的内酯环更容易被具有较高pH的海水打开。因此推断,在生物被膜中具有降解长链AHL信号分子菌株的大量存在,与被膜中长链信号分子的积累有很大的关系,细菌为了在生物被膜的群体中维持自身的生存,抵抗优势种群如弧菌、鲁杰氏菌的QS武器,不得不进化出降解信号分子的机制。另外生物被膜等高密度环境下信号分子的积累很有可能使得AHL成为细菌的一种碳源[39-40]。此外,从分离结果来看,有18株鲁杰氏菌分别属于Ruegeria atlantica、Ruegeria conchae和Ruegeria arenilitoris,它们非常特殊,不仅在所分离到的可培养细菌中占据了较高的比例,而且同时具有合成和降解AHL类信号分子的能力,该属细菌在海洋环境中提供的生态意义,以及是否可以进一步开发利用有待进一步研究。
从海洋沉积物表层生物被膜分离得到的QS与QQ活性菌株具有较高的丰度和多样性,丰富了除海雪、海绵、刺细胞动物、POC等微生物密集区的密度感应现象研究,为密度感应现象在海洋环境中的生态学意义提供了一些基础数据。本研究分离得到的优势属——弧菌属和鲁杰氏菌属的生态学意义也有待进一步研究。此外,QS与细菌毒力因子之间的关系研究已经十分明确,通过QQ干扰QS系统,能够在不杀死细菌的情况下特异性阻断致病菌毒力基因的表达,从而有效控制病原菌致病性,为新型抗生素的开发提供了新的思路。从本研究在青岛近岸沉积物表层生物被膜分离得到的高丰度的QQ活性菌株有待作为新的海洋药物进行开发利用。
| [1] |
Nealson KH, Hastings JW. Bacterial bioluminescence: its control and ecological significance[J]. Microbiological Reviews, 1979, 43(4): 496-518. |
| [2] |
Winzer K, Williams P. Quorum sensing and the regulation of virulence gene expression in pathogenic bacteria[J]. International Journal of Medical Microbiology, 2001, 291(2): 131-143. DOI:10.1078/1438-4221-00110 |
| [3] |
Ng WL, Bassler BL. Bacterial quorum-sensing network architectures[J]. Annual Review of Genetics, 2009, 43: 197-222. DOI:10.1146/annurev-genet-102108-134304 |
| [4] |
Hmelo L, van Mooy BAS. Kinetic constraints on acylated homoserine lactone-based quorum sensing in marine environments[J]. Aquatic Microbial Ecology, 2009, 54(2): 127-133. |
| [5] |
Gram L, Grossart HP, Schlingloff A, et al. Possible quorum sensing in marine snow bacteria: production of acylated homoserine lactones by Roseobacter strains isolated from marine snow[J]. Applied and Environmental Microbiology, 2002, 68(8): 4111-4116. DOI:10.1128/AEM.68.8.4111-4116.2002 |
| [6] |
Jatt AN, Tang K, Liu JW, et al. Quorum sensing in marine snow and its possible influence on production of extracellular hydrolytic enzymes in marine snow bacterium Pantoea ananatis B9[J]. FEMS Microbiology Ecology, 2015, 91(2): 1-13. |
| [7] |
Mohamed NM, Cicirelli EM, Kan JJ, et al. Diversity and quorum-sensing signal production of Proteobacteria associated with marine sponges[J]. Environmental Microbiology, 2008, 10(1): 75-86. |
| [8] |
Ransome E, Munn CB, Halliday N, et al. Diverse profiles of N-acyl-homoserine lactone molecules found in cnidarians[J]. FEMS Microbiology Ecology, 2014, 87(2): 315-329. DOI:10.1111/1574-6941.12226 |
| [9] |
Huang YL, Ki JS, Case RJ, et al. Diversity and acyl-homoserine lactone production among subtidal biofilm-forming bacteria[J]. Aquatic Microbial Ecology, 2008, 52(2): 185-193. |
| [10] |
Hall-Stoodley L, Costerton JW, Stoodley P. Bacterial biofilms: from the natural environment to infectious diseases[J]. Nature Reviews Microbiology, 2004, 2(2): 95-108. DOI:10.1038/nrmicro821 |
| [11] |
Dang HY, Lovell CR. Bacterial primary colonization and early succession on surfaces in marine waters as determined by amplified rRNA gene restriction analysis and sequence analysis of 16S rRNA genes[J]. Applied and Environmental Microbiology, 2000, 66(2): 467-475. DOI:10.1128/AEM.66.2.467-475.2000 |
| [12] |
Tang KH, Su Y, Brackman G, et al. MomL, a novel marine-derived N-acyl homoserine lactonase from Muricauda olearia[J]. Applied and Environmental Microbiology, 2015, 81(2): 774-782. DOI:10.1128/AEM.02805-14 |
| [13] |
Zhang XH. Marine Microbiology[M]. 2nd ed. Beijing: Science Press, 2016: 368-369. (in Chinese) 张晓华. 海洋微生物学[M]. 2版. 北京: 科学出版社, 2016: 368-369. |
| [14] |
McLean RJ, Whiteley M, Stickler DJ, et al. Evidence of autoinducer activity in naturally occurring biofilms[J]. FEMS Microbiology Letters, 1997, 154(2): 259-263. DOI:10.1111/j.1574-6968.1997.tb12653.x |
| [15] |
Zhu J, Beaber JW, Moré MI, et al. Analogs of the autoinducer 3-oxooctanoyl-homoserine lactone strongly inhibit activity of the TraR protein of Agrobacterium tumefaciens[J]. Journal of Bacteriology, 1998, 180(20): 5398-5405. |
| [16] |
Margassery LM, Kennedy J, O'Gara F, et al. Diversity and antibacterial activity of bacteria isolated from the coastal marine sponges Amphilectus fucorum and Eurypon major[J]. Letters in Applied Microbiology, 2012, 55(1): 2-8. |
| [17] |
Romero M, Martin-Cuadrado AB, Roca-Rivada A, et al. Quorum quenching in cultivable bacteria from dense marine coastal microbial communities[J]. FEMS Microbiology Ecology, 2011, 75(2): 205-217. DOI:10.1111/fem.2010.75.issue-2 |
| [18] |
Weisburg WG, Barns SM, Pelletier DA, et al. 16S ribosomal DNA amplification for phylogenetic study[J]. Journal of Bacteriology, 1991, 173(2): 697-703. DOI:10.1128/jb.173.2.697-703.1991 |
| [19] |
Kim OS, Cho YJ, Lee K, et al. Introducing EzTaxon-e: a prokaryotic 16S rRNA gene sequence database with phylotypes that represent uncultured species[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62: 716-721. DOI:10.1099/ijs.0.038075-0 |
| [20] |
Kim M, Oh HS, Park SC, et al. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(2): 346-351. |
| [21] |
Chu WH, Vattem DA, Maitin V, et al. Bioassays of quorum sensing compounds using Agrobacterium tumefaciens and Chromobacterium violaceum[A]//Rumbaugh KP. Quorum Sensing[M]. New York: Humana Press, 2011, 692: 3-19
|
| [22] |
Mok KC, Wingreen NS, Bassler BL. Vibrio harveyi quorum sensing: a coincidence detector for two autoinducers controls gene expression[J]. EMBO Journal, 2003, 22(4): 870-881. DOI:10.1093/emboj/cdg085 |
| [23] |
Milton DL. Quorum sensing in vibrios: complexity for diversification[J]. International Journal of Medical Microbiology, 2006, 296(2/3): 61-71. |
| [24] |
Boyer M, Wisniewski-Dyé F. Cell-cell signalling in bacteria: not simply a matter of quorum[J]. FEMS Microbiology Ecology, 2009, 70(1): 1-19. |
| [25] |
Zan JD, Liu Y, Fuqua C, et al. Acyl-homoserine lactone quorum sensing in the Roseobacter clade[J]. International Journal of Molecular Sciences, 2014, 15(1): 654-669. DOI:10.3390/ijms15010654 |
| [26] |
Parsek MR, Greenberg EP. Sociomicrobiology: the connections between quorum sensing and biofilms[J]. Trends in Microbiology, 2005, 13(1): 27-33. DOI:10.1016/j.tim.2004.11.007 |
| [27] |
Shrout JD, Nerenberg R. Monitoring bacterial twitter: does quorum sensing determine the behavior of water and wastewater treatment biofilms?[J]. Environmental Science & Technology, 2012, 46(4): 1995-2005. |
| [28] |
González JE, Marketon MM. Quorum sensing in nitrogen-fixing rhizobia[J]. Microbiology and Molecular Biology Reviews, 2003, 67(4): 574-592. DOI:10.1128/MMBR.67.4.574-592.2003 |
| [29] |
Toyofuku M, Nomura N, Kuno E, et al. Influence of the Pseudomonas quinolone signal on denitrification in Pseudomonas aeruginosa[J]. Journal of Bacteriology, 2008, 190(24): 7947-7956. DOI:10.1128/JB.00968-08 |
| [30] |
De Clippeleir H, Defoirdt T, Vanhaecke L, et al. Long-chain acylhomoserine lactones increase the anoxic ammonium oxidation rate in an OLAND biofilm[J]. Applied Microbiology and Biotechnology, 2011, 90(4): 1511-1519. DOI:10.1007/s00253-011-3177-7 |
| [31] |
Mellbye BL, Bottomley PJ, Sayavedra-Soto LA. Nitrite-oxidizing bacterium Nitrobacter winogradskyi produces N-acyl-homoserine lactone autoinducers[J]. Applied and Environmental Microbiology, 2015, 81(17): 5917-5926. DOI:10.1128/AEM.01103-15 |
| [32] |
Slightom RN, Buchan A. Surface colonization by marine Roseobacters: integrating genotype and phenotype[J]. Applied and Environmental Microbiology, 2009, 75(19): 6027-6037. DOI:10.1128/AEM.01508-09 |
| [33] |
Wagner-D bler I, Biebl H. Environmental biology of the marine Roseobacter lineage[J]. Annual Review of Microbiology, 2005, 60: 255-280. |
| [34] |
Buchan A, Mitchell A, Cude WN, et al. Acyl-homoserine lactone-based quorum sensing in members of the marine bacterial Roseobacter clade: complex cell-to-cell communication controls multiple physiologies[A]//de Bruijn FJ. Stress and Environmental Regulation of Gene Expression and Adaptation in Bacteria, I & II[M]. New York: John Wiley & Sons, Inc, 2016: 225-233
|
| [35] |
Saurav K, Costantino V, Venturi V, et al. Quorum sensing inhibitors from the sea discovered using bacterial N-acyl-homoserine lactone-based biosensors[J]. Marine Drugs, 2017, 15(3): 53. DOI:10.3390/md15030053 |
| [36] |
Chen CL, Maki JS, Rittschof D, et al. Early marine bacterial biofilm on a copper-based antifouling paint[J]. International Biodeterioration & Biodegradation, 2013, 83: 71-76. |
| [37] |
Romero M, Martin-Cuadrado A, Otero A. Determination of whether quorum quenching is a common activity in marine bacteria by analysis of cultivable bacteria and metagenomic sequences[J]. Applied and Environmental Microbiology, 2012, 78(17): 6345-6348. DOI:10.1128/AEM.01266-12 |
| [38] |
Muras A, López-Pérez M, Mayer C, et al. High prevalence of quorum-sensing and quorum-quenching activity among cultivable bacteria and metagenomic sequences in the Mediterranean sea[J]. Genes (Basel), 2018, 9(2): 100. DOI:10.3390/genes9020100 |
| [39] |
Torres M, Rubio-Portillo E, Antón J, et al. Selection of the N-acylhomoserine lactone-degrading bacterium Alteromonas stellipolaris PQQ-42 and of its potential for biocontrol in aquaculture[J]. Frontiers in Microbiology, 2016, 7: 646. |
| [40] |
Torres M, Romero M, Prado S, et al. N-acylhomoserine lactone-degrading bacteria isolated from hatchery bivalve larval cultures[J]. Microbiological Research, 2013, 168(9): 547-554. DOI:10.1016/j.micres.2013.04.011 |
 2019, Vol. 46
2019, Vol. 46




