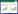扩展功能
文章信息
- 朱渊, 戴云山, 王俊虎, 王萍, 吕凤霞, 顾秋琴
- ZHU Yuan, DAI Yun-Shan, WANG Jun-Hu, WANG Ping, LÜ Feng-Xia, GU Qiu-Qin
- 基于聚合酶螺旋反应技术快速检测铜绿假单胞菌
- Rapid detection of Pseudomonasaeruginosa by polymerase spiral reaction (PSR)
- 微生物学通报, 2019, 46(5): 1246-1256
- Microbiology China, 2019, 46(5): 1246-1256
- DOI: 10.13344/j.microbiol.china.180775
-
文章历史
- 收稿日期: 2018-10-09
- 接受日期: 2018-12-29
- 网络首发日期: 2018-01-29
2. 泰州市食品药品监督管理局 江苏 泰州 225300;
3. 南京农业大学食品学院 江苏 南京 210095
2. 2 Taizhou Food and Drug Administration, Taizhou, Jiangsu 225300, China;
3. College of Food Science and Technology, Nanjing Agricultural University, Nanjing, Jiangsu 210095, China
铜绿假单胞菌(Pseudomonas aeruginosa)是一种重要的水源性致病菌,广泛存在于各种水源中,对消毒剂、干燥、紫外线等理化因素具有很强的抵抗力,可引起急性肠道炎、脑膜炎、败血症和皮肤炎症等疾病[1]。桶(瓶)装水中有机物质含量极低,且刚出厂的产品中铜绿假单胞菌数量较少,但桶(瓶)装水消费周期较长,保存期至少半个月以上,兼性化能自养代谢而对有机营养要求低的铜绿假单胞菌可生长繁殖达到104 CFU/mL[2]。水质卫生管理方面,美国、加拿大、巴西、日本、欧洲各国及世界卫生组织/粮农组织等,均限定瓶装饮用水(包括天然矿泉水水源)中铜绿假单胞菌最大可能数(Most probable number,MPN) < 3/L或每250 mL中不得检出[3]。我国实施的《GB 19298-2014食品安全国家标准包装饮用水》和《GB 8537-2008饮用天然矿泉水》标准中也明确规定每250 mL包装饮用水和饮用天然矿泉水中不得检出铜绿假单胞菌[3-4]。近年来,随着国内桶装饮用水消费量的不断上升,有关铜绿假单胞菌污染桶装水的报道逐渐增多,引起了诸多学者和消费者的普遍关注[5-9]。
目前我国规定的包装饮用水铜绿假单胞菌检测方法为传统国标法,其检验程序费时费力、繁琐复杂,需要2-7 d才能得到检验结果[3]。近年来,普通PCR、荧光定量PCR和环介导等温扩增技术(Loop-mediated isothermal amplification,LAMP)等分子检测方法逐步建立起来,应用于水样快速检测[3, 10]。但普通PCR和荧光定量PCR都需要复杂的具有变温功能的仪器,荧光定量PCR还需要特异性探针,价格昂贵。LAMP法是在PCR的基础上建立起来的一种常温核酸扩增技术[11],目前已被应用于各种致病菌的快速检测[12-16]。但是LAMP法需要针对靶序列上6-8个片段设计引物,一般需要6条引物,引物设计较复杂,这6条引物都要规避突变点区域,因此对靶序列的要求高,并且日本的公司特别注重对其专利产权的保护,我国在转化应用上有很大的局限性[17-18]。
聚合酶螺旋反应(Polymerase spiral reaction,PSR)是一种新型核酸等温扩增法,利用具有链转移活性的DNA聚合酶(Bst DNA聚合酶)在等温条件下进行扩增[17-19]。该法从LAMP引物设计中得到启示,并融合PCR引物设计简便的优点,仅在PCR引物的基础上从目标序列上再取一段序列,将这段序列加到PCR引物的5′端,构成正反向复合引物,满足了简便快速、高灵敏度和特异性的要求[17]。目前已有研究人员应用PSR技术进行细菌、真菌和病毒的临床现场检测工作,其中包括以toxA为靶基因PSR检测铜绿假单胞菌的研究[17-22]。本研究以铜绿假单胞菌外毒素A调控基因——ETA基因(toxA)为靶基因,基于PSR技术建立铜绿假单胞菌的快速检测法,从而为铜绿假单胞菌的快速检测提供有力手段。
1 材料与方法 1.1 材料 1.1.1 菌种各种标准菌株铜绿假单胞菌(P. aeruginosa) CGMCC1.10274、志贺氏菌(Shigella flexneri) CGMCC1.10599、大肠埃希菌(Escherichia coli) CGMCC1.4245、沙门氏菌(Salmonella enterica) CGMCC1.10603、阪崎杆菌(Cronobacter sakazakii) CGMCC1.6765、副溶血弧菌(Vibrio parahaemolyticus) CGMCC1.1997、粪肠球菌(Enterococcus faecium) CGMCC 1.101、马红球菌(Rhodococcus equi) CGMCC1.4262、蜡状芽孢杆菌(Bacillus cereus) CGMCC 1.10559、金黄色葡萄球菌(Staphylococcus aureus subsp. aureus) CGMCC1.2465购自中国普通微生物菌种保藏管理中心,恶臭假单胞菌(P. putida)、荧光假单胞菌(P. fluorescens)、施氏假单胞菌(P. stutzeri)[23]由本实验室提供,铜绿假单胞菌(P. aeruginosa)分离株(16株)从包装饮用水中分离纯化,按照国家食品安全风险监测工作手册(GB/T 8538-2008)检验方法检测,并使用VITECK2 Compact微生物鉴定仪进行结果确认。
1.1.2 主要试剂和仪器营养肉汤、CN琼脂平板,北京陆桥技术有限责任公司;Bst DNA聚合酶,NEB公司;甜菜碱、吐温-20、钙黄绿素、氯化锰、羟基萘酚蓝、二甲基亚砜,Sigma公司;MiniBEST Bacteria Genomic DNA Extraction Kit、ExTaq® DNA Polymerase,TaKaRa公司;石蜡油,百奥莱博公司;氢氧化钾、Tris-HCl、氯化钾、硫酸铵、硫酸镁,国药集团化学试剂有限公司;Nuclease-Free Water、dNTP Mixture,生工生物工程(上海)股份有限公司;QX Alignment Marker 15 kb/3kb、QX Size Marker 0.1-2.5 kb,Qiagen公司。
低温高速离心机,Beckman公司;PCR仪,ABI公司;紫外分光光度计,Shimadzu公司;恒温水浴锅,杭州博日科技有限;全自动核酸蛋白分析仪,Qiagen公司;DensiCHEKTM比浊仪,Bio Merieux公司;NanoDrop One超微量分光光度计,Thermo Fisher Scientific公司。
1.2 方法 1.2.1 引物设计根据GenBank中公布的铜绿假单胞菌toxA基因序列(登录号为NC_002516.2),在NCBI上进行BLAST比对分析后,基于toxA基因的特异性区域,利用软件Primer premier 5设计PSR反应的4条特异性引物,分别为引物Ft/Bt,加速引物IF/IB。同时设计PCR反应的引物toxA-1f/toxA-1f,见表 1。引物交由生工生物工程(上海)股份有限公司合成。
| 引物名称Primers | 检测方法Detection methods | 引物序列Primer sequences (5′→3′) | 基因位置Gene locations |
| Ft* | PSR | gtcgatggccagcttgagcgATCGCCGACACCAACGG | 258-238, 175-192 |
| Bt* | gcgagttcgaccggtagctgGCCAGTTCAGCGACCAACT | 238-258, 360-379 | |
| IF | GTAGTGCAGCACGCCCTG | 210-192 | |
| IB | CGCTACAGCTACACGCG | 331-348 | |
| toxA-1f | PCR | AGATGGGCGACGAGTTGCTG | 482-501 |
| toxA-1r | GACTGATGACCGTGGGCTTGAT | 811-789 | |
| 注:*:Ft与Bt分别由Nr (小写字母)和F (大写字母)、N (小写字母)和B (大写字母)组成. Note: *: Ft and Bt consist of Nr (lowercase) and F (uppercase), N (lowercase) and B (uppercase), respectively. |
|||
在CN琼脂平板上取细菌单菌落接入营养肉汤[9]中,培养12 h到对数期。取1.0 mL OD600为0.15的菌悬液,12 000×g离心2 min,轻轻倒掉上清。向革兰氏阳性细菌沉淀中加入500 μL BS缓冲液和50 μL溶菌酶(20 mg/mL),充分混匀沉淀,37 ℃水浴60 min,12 000×g离心2 min (革兰氏阴性细菌省去此步骤)。向细菌沉淀中加入20 μL蛋白酶K (20 mg/mL)和10 μL RNase A (10 mg/mL),180 μL GL裂解液,充分混匀沉淀,56 ℃水浴10 min,之后严格按照TaKaRa MiniBEST Bacteria Genomic DNA Extraction Kit操作步骤提取细菌基因组DNA。
基因组DNA的质量浓度和纯度检测:取DNA溶液1 μL,用NanoDrop One超微量分光光度计测定以上提取的DNA在波长260 nm和280 nm处的紫外吸收值,通过OD260/OD280和浓度值判断DNA的纯度和浓度。
1.2.3 PSR反应体系的建立及优化扩增反应体系参考Liu等[19]的方法并进行一定优化。RM储备液(20×):KCl 1.49 g,(NH4)2SO4 2.64 g,MgSO4·7H2O 3.95 g,吐温-20 2.0 mL,加水溶解并定容至100 mL。RM反应液(2×):RM储备液(20×) 200 μL,Tris-HCl (1.5 mol/L,pH 8.8) 53.33 μL,dNTPs (10 mmol/L,Each) 560 μL,甜菜碱溶液(0.375 g/mL) 1.0 mL,ddH2O补齐至2 mL。25 μL反应体系:RM反应液(2×) 12.5 μL,引物Ft/Bt (50 μmol/L,Each) 1.6 μL,模板DNA 2 μL,Bst DNA聚合酶(8 U/μL) 1 μL,ddH2O 7.9 μL。再加入石蜡油10 μL后的反应混合液放入水浴锅中65 ℃反应90 min,毛细管凝胶电泳检测扩增结果。
为使PSR反应更加迅速,25 μL反应体系中引入加速引物IF/IB,上述25 μL反应体系中加入加速引物IF/IB (50 μmol/L,Each) 0.8 μL。同时对DNA双链解链的关键物质——甜菜碱的浓度进行优化。PSR反应体系中其他物质浓度不变,设置甜菜碱浓度梯度0.2、0.5、0.8、1.1、1.3 mol/L,摸索最佳浓度。同时,为避免气溶胶污染,确保实验准确性,PSR反应前在反应液液面上加入10 μL石蜡油,上层形成的石蜡油密封层能有效阻止反应过程中产生的气溶胶污染。
1.2.4 PSR反应结果的可视化研究为符合等温扩增法简便、快捷的特点,进行PSR反应结果的可视化研究,即在PSR反应体系中加入颜色指示剂,通过阴、阳性反应的颜色差异快速判断实验结果。
钙黄绿素(Calcein)作指示剂:设立钙黄绿素(6.5 mmol/L,DMSO配制)和氯化锰(130 mmol/L)不同组别见表 2,在25 μL PSR反应体系中加入不同组别的钙黄绿素指示剂1 μL,分别设立阳性组与阴性组,65 ℃反应后取出于黑背景下观察结果。PSR反应阳性,溶液由橙色变成绿色;PSR反应阴性,溶液保持橙色不变。当肉眼观察无法清晰分辨绿色和橙色时,可在紫外光下观察,可见阳性样品管发绿色荧光,阴性样品管无荧光发出。
| 组别 Group |
钙黄绿素体积 Calcein (μL/mL) |
氯化锰体积 MnCl2 (μL/mL) |
钙黄绿素终浓度 Final concentration of calcein (μmol/L) |
氯化锰终浓度 Final concentration of MnCl2 (mmol/L) |
钙黄绿素与氯化锰摩尔比 Calcein:MnCl2 (M/M) |
| 1 | 100 | 100 | 25 | 0.5 | 1:20 |
| 2 | 100 | 80 | 25 | 0.4 | 1:16 |
| 3 | 100 | 60 | 25 | 0.3 | 1:12 |
| 4 | 100 | 40 | 25 | 0.2 | 1:8 |
| 5 | 200 | 200 | 50 | 1.0 | 1:20 |
| 6 | 200 | 160 | 50 | 0.8 | 1:16 |
| 7 | 200 | 120 | 50 | 0.6 | 1:12 |
| 8 | 200 | 80 | 50 | 0.4 | 1:8 |
羟基萘酚蓝(HNB)作指示剂:参考Goto等[24]方法,采用0.2% HNB指示剂溶液,在25 μL PSR反应体系中加1.0 μL HNB溶液,65 ℃反应后白色背景下观察溶液的变化。PSR反应阳性,溶液由紫色变成天蓝色;PSR反应阴性,溶液保持紫色不变。
1.2.5 PSR反应特异性和灵敏度检测提取铜绿假单胞菌和12株非铜绿假单胞菌基因组DNA,以此为模板进行PSR反应,验证方法特异性。
将提取的铜绿假单胞菌(P. aeruginosa)原菌液总DNA测定浓度后,对DNA进行10倍比梯度稀释,最后选取10.115 fg、101.15 fg、1.011 5 pg、10.115 pg、101.15 pg、1.011 5 ng、10.115 ng/μL共7个浓度梯度,取2 μL各浓度梯度的DNA为模板进行PSR反应检测纯培养铜绿假单胞菌灵敏度。
1.2.6 PCR和PSR反应在包装饮用水中的灵敏度检测为保证检测结果的准确性,经国标GB/T 8538-2008方法验证试验所用包装饮用水不含铜绿假单胞菌。取新鲜培养12 h的铜绿假单胞菌(CGMCC1.10274),用生理盐水充分洗涤培养基,10倍比梯度系列稀释。取各稀释度菌液1 mL加入到100 mL包装饮用水样品中,使包装饮用水中菌液浓度达到2×100、2×101、2×102、2×103、2×104、2×105、2×106 CFU/mL,充分混合,作为人工污染包装饮用水样品。
取各菌液浓度的人工污染包装饮用水样品1 mL,提取样品DNA,同时取不加铜绿假单胞菌的包装饮用水样品1 mL作为阴性对照。分别进行PCR和PSR反应,比较两种方法的检测灵敏度。按照ExTaq® DNA Polymerase说明书进行PCR扩增反应。
1.2.7 PSR反应在实际样本检测中的应用将实际检验工作中分离纯化的包装饮用水来源的16株铜绿假单胞菌菌株提取核酸分别进行PSR反应,同时以无菌水和铜绿假单胞菌阴性的包装饮用水样本为对照,观察检测结果。
2 结果与分析 2.1 PSR反应体系的建立及优化以铜绿假单胞菌基因组DNA为模板,按PSR反应体系配制反应液,以无菌水为阴性对照。毛细管凝胶电泳检测结果显示,65 ℃水浴反应90 min后呈现出特异性梯状条带,且肉眼观察可见反应溶液处于浑浊状态,阴性对照溶液澄清且无梯状条带(图 1)。结果表明PSR能且只能在有特定核酸及能与特定核酸序列结合的引物时发生反应,验证了PSR反应的可行性。
|
| 图 1 铜绿假单胞菌PSR反应毛细管凝胶电泳结果 Figure 1 The capillary gel electrophoresis results of PSR 注:M:Alignment marker 15 kb/3 kb;N:阴性对照;1:引物Ft/Bt;2:引物Ft/Bt+IF/IB. Note: M: Alignment marker 15 kb/3 kb; N: Negative control; 1: Primer Ft/Bt; 2: Primer Ft/Bt+IF/IB. |
|
|
PSR反应体系中使用Ft/Bt一对引物时,反应时间要在60 min之后,不能满足快速检测的要求,因此引入加速引物IF/IB。为使Ft/Bt首先与靶序列结合,加速引物IF/IB使用浓度(终浓度0.8 μmol/L)是引物Ft/Bt使用浓度(终浓度1.6 μmol/L)的一半。结果显示引入加速引物后,65 ℃水浴反应40 min即可观察到溶液呈现浑浊状态,毛细管凝胶电泳检测结果显示出特异性梯状条带(图 1)。
甜菜碱是恒温下使DNA双链解链的关键物质,RM反应液中其他物质浓度不变,甜菜碱浓度分别为0.2、0.5、0.8、1.1、1.3 mol/L时,65 ℃水浴反应40 min均能观察到溶液浑浊状态。毛细管凝胶电泳检测结果显示,同等时间下,甜菜碱浓度为0.8 mol/L时梯状条带更加清晰(图 2),表示其反应效率更高,因此0.8 mol/L可作为PSR反应下一步试验的甜菜碱浓度。

|
| 图 2 甜菜碱浓度筛选结果 Figure 2 The betaine concentration of PSR 注:M:Alignment marker 15 kb/3 kb;N:阴性对照;1:甜菜碱0.2 mol/L;2:甜菜碱0.5 mol/L;3:甜菜碱0.8 mol/L;4:甜菜碱1.1 mol/L;5:甜菜碱1.3 mol/L. Note: M: Alignment marker 15 kb/3 kb; N: Negative control; 1: Betaine 0.2 mol/L; 2: Betaine 0.5 mol/L; 3: Betaine 0.8 mol/L; 4: Betaine 1.1 mol/L; 5: Betaine 1.3 mol/L. |
|
|
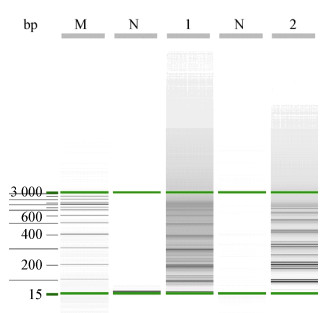
按PSR反应的可视化方法(表 2),使用不同浓度组合的钙黄绿素指示剂(组别1-8)进行PSR反应,观察其产物在可见光下(图 3A)和紫外光激发下的荧光情况(图 3B)。钙黄绿素指示剂低浓度组(组别2、3、4)和高浓度组(组别8)的阳性组溶液呈现绿色,阴性组保持橙色不变,肉眼即可分辨(图 3A),且在紫外灯下可见绿色荧光(图 3B)。为确保检测结果的准确性,进行3次重复实验,数据统计如表 3所示。结果表明氯化锰终浓度低于0.5 mmol/L (组别4、3、2、8)时,建立的PSR反应正常进行。但氯化锰终浓度高于0.4 mmol/L (组别1、7、6、5)时,过量的锰离子可能对PSR反应有抑制作用。同时组别8阴、阳性颜色对比与组别2、3、4相比,视觉差异更加明显,而且显色时间更短。因此,组别8 (钙黄绿素50 μmol/L、氯化锰0.4 mmol/L)可作为钙黄绿素指示剂的使用浓度。
|
| 图 3 钙黄绿素指示剂显色(A)和荧光素(B)结果 Figure 3 Indicating results of Calcein indicators by coloration (A) and fluorescence (B) 注:+:阳性组;-:阴性组;1-8:组别1-8. Note: +: Positive group; -: Negative group; 1-8: Group 1-8. |
|
|
| Indicating method | Group 1 | Group 2 | Group 3 | Group 4 | Group 5 | Group 6 | Group 7 | Group 8 |
| Coloration | - | + | + | + | - | - | - | + |
| - | + | + | + | - | - | - | + | |
| - | + | + | + | - | - | - | + | |
| Fluorescence | - | + | + | + | - | - | - | + |
| - | + | + | + | - | - | - | + | |
| - | + | + | + | - | - | - | + | |
| 注:+:阳性组;-:阴性组. Note: +: Positive group; -: Negative group. |
||||||||
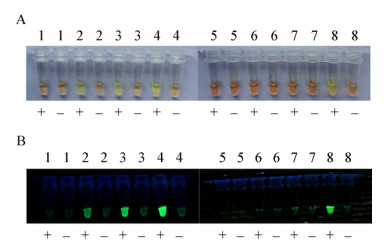
同时羟基萘酚蓝指示剂(HNB)结果显示,阳性组溶液反应40 min后由紫色变成天蓝色,而阴性组溶液保持紫色不变(图 4),因此0.2% HNB可作为HNB指示剂的使用浓度。
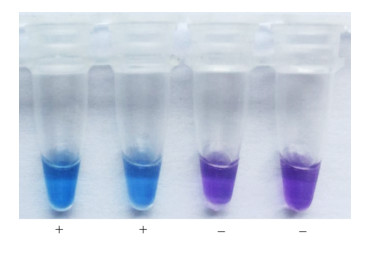
|
| 图 4 羟基萘酚蓝指示剂显色结果 Figure 4 Indicating results of HNB indicator 注:+:阳性组;-:阴性组. Note: +: Positive group; -: Negative group. |
|
|
根据建立的PSR反应体系,对包括铜绿假单胞菌在内的13株常见细菌进行PSR特异性检测。只有P. aeruginosa的基因组DNA呈现阳性结果(钙黄绿素指示剂显绿色,HNB指示剂显蓝色且电泳呈特异性梯状条带)。其他12个对照样品,分别为Shigella flexneri、Escherichia coli、Salmonella enterica、Cronobacter sakazakii、Vibrio parahaemolyticus、Enterococcus faecium、Rhodococcus equi、Bacillus cereus、Staphylococcus aureus subsp. aureus、P. putida、P. fluorescens、P. stutzeri以及阴性对照(添加无菌水)呈现阴性结果(图 5)。实验结果表明,基于toxA建立的PSR检测法对P. aeruginosa具有较高的特异性。
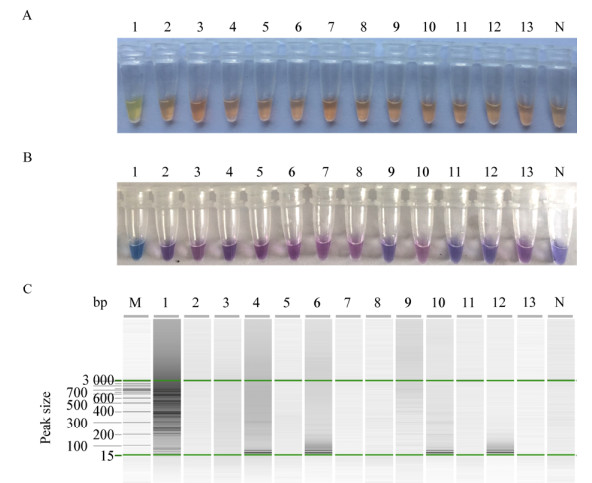
|
| 图 5 基于铜绿假单胞菌toxA基因PSR检测对其他不同病原菌的特异性 Figure 5 The product specificity of toxA targeted PSR for different pathogens 注:A:钙黄绿素显色结果;B:羟基萘酚蓝显色结果;C:毛细管凝胶电泳结果. M:Alignment marker 15 kb/3 kb;1:铜绿假单胞菌;2:志贺氏菌;3:大肠埃希菌;4:沙门氏菌;5:阪崎杆菌;6:副溶血弧菌;7:粪肠球菌;8:马红球菌;9:蜡状芽孢杆菌;10:金黄色葡萄球菌;11:恶臭假单胞菌;12:荧光假单胞菌;13:施氏假单胞菌;N:阴性对照. Note: A: Visualized with Calcein; B: HNB; C: Capillary gel electrophoresis. M: Alignment marker 15 kb/3 kb; 1: P. aeruginosa; 2: Shigella flexneri; 3: Escherichia coli; 4: Salmonella enterica; 5: Cronobacter sakazakii; 6: Vibrio parahaemolyticus; 7: Enterococcus faecium; 8: Rhodococcus equi; 9: Bacillus cereus; 10: Staphylococcus aureus subsp. aureus; 11: P. putida; 12: P. fluorescens; 13: P. stutzeri; N: Negative control. |
|
|
提取纯培养铜绿假单胞菌基因组DNA作为阳性模板,对模板进行10倍比梯度稀释,检测PSR反应的灵敏度。由图 6可知,PSR检测toxA的灵敏度为1.011 5 pg/μL。

|
| 图 6 PSR灵敏性检测钙黄绿素显色结果(A)、羟基萘酚蓝显色结果(B)和毛细管凝胶电泳结果(C) Figure 6 The detection sensitivity of PSR visualized with Calcein (A), HNB (B) and capillary gel electrophoresis (C) Note: M: Alignment marker 15 kb/3 kb; 1: 10.115 ng/μL; 2: 1.011 5 ng/μL; 3: 101.15 pg/μL; 4: 10.115 pg/μL; 5: 1.011 5 pg/μL; 6: 101.15 fg/μL; 7: 10.115 fg/μL; N: Negative control. |
|
|
对人工污染包装饮用水样品提取DNA进行PSR和PCR灵敏度检测。由图 7可知,PSR反应灵敏度达到20 CFU/mL (图 7,A6、B6、C6),PCR反应灵敏度达到200 CFU/mL (图 7,D5)。结果表明建立的PSR方法灵敏度较高,是PCR反应灵敏度的10倍。
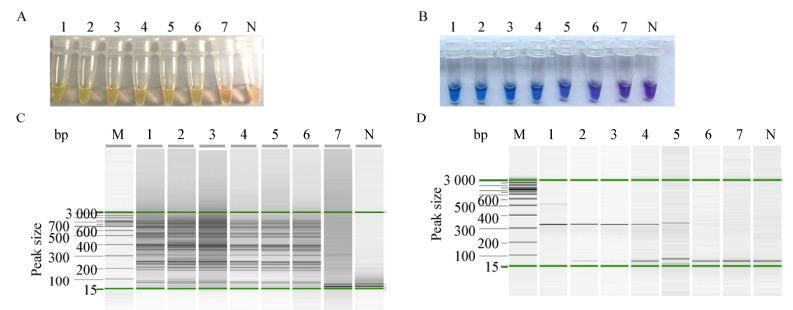
|
| 图 7 人工接种铜绿假单胞菌污染的包装饮用水PSR (A、B、C)和PCR (D)反应灵敏度对比 Figure 7 The toxA detection sensitivity by PSR (A, B, C) and PCR (D) for artificial contamination of P. aeruginosa in packaged drinking water 注:A:PSR钙黄绿素显色结果;B:PSR羟基萘酚蓝显色结果;C:PSR毛细管电泳结果;D:PCR毛细管凝胶电泳结果. M:Alignment marker 15 kb/3 kb;1:2×106 CFU/mL;2:2×105 CFU/mL;3:2×104 CFU/mL;4:2×103 CFU/mL;5:2×102 CFU/mL;6:2×101 CFU/mL;7:2×100 CFU/mL;N:阴性对照. Note: A: Calcein; B: HNB; C: Capillary electrophoresis; D: PCR capillary gel electrophoresis. M: Alignment marker 15 kb/3 kb; 1: 2×106 CFU/mL; 2: 2×105 CFU/mL; 3: 2×104 CFU/mL; 4: 2×103 CFU/mL; 5: 2×102 CFU/mL; 6: 2×101 CFU/mL; 7: 2×100 CFU/mL; N: Negative control. |
|
|
对16株包装饮用水来源的铜绿假单胞菌分离株提取基因组DNA,以无菌水和铜绿假单胞菌阴性的包装饮用水样本为对照,进行PSR检测。由图 8可知,从包装饮用水中分离纯化到的16株铜绿假单胞菌菌株都被PSR反应检测出来,检出率100%。实验结果表明PSR反应可以用来检测实际样本可能存在的铜绿假单胞菌。

|
| 图 8 PSR反应检测样本分离结果 Figure 8 PSR test results of packaged drinking water isolates 注:A:钙黄绿素显色结果;B:羟基萘酚蓝显色结果;C:毛细管凝胶电泳结果. M:Alignment marker 15 kb/3 kb;1-16:样本分离株1-16;H:H2O;N:阴性对照. Note: A: Calcein; B: HNB; C: Capillary electrophoresis. M: Alignment marker 15 kb/3 kb; 1-16: Isolate 1-16; H: H2O; N: Negative control. |
|
|
绿脓杆菌外毒素A (ETA)是P. aeruginosa最具毒性的胞外产物,95%以上的临床分离株都会分泌该种毒素[25]。外毒素A调控基因——ETA基因(toxA)在P. aeruginosa中高度保守[26],已被应用于PCR和RT-PCR法检测P. aeruginosa的工作中[10, 27-30]。本研究中利用生物信息学技术对toxA基因进行分析,选取175-379 bp的基因片段作为PSR扩增的靶基因片段(表 1),BLAST结果显示选取的基因片段高度保守。针对该toxA片段设计了一对特异性扩增引物(Ft/Bt),该引物不同于其他研究引物[17],扩增结果证明了PSR反应的可行性(图 1),且灵敏度得到提升。同时为满足快速检测的要求,我们又设计了一对特异性的加速引物(IF/IB),将PSR反应时间由90 min缩短至40 min (图 1)。在toxA基因、扩增引物、加速引物的高度特异性的三重保证下,检测更为准确,特异性实验结果显示基于toxA基因的PSR检测具有很好的特异性(图 5)。
我们建立的PSR方法是利用具有链置换活性的高效Bst DNA聚合酶,使反应在等温(65 ℃)条件下快速进行,因此等温条件下使DNA双链解链的关键物质甜菜碱的浓度变化至关重要。甜菜碱浓度梯度实验结果显示浓度为0.8 mol/L时,PSR反应效率最高(图 2)。气溶胶污染是困扰整个核酸扩增领域的难题,PSR反应扩增效率高,反应剧烈,也不免会形成带有大量核酸的气溶胶。为防止气溶胶污染,确保实验准确性,反应前将10 μL石蜡油加入到反应管中,反应过程中有效地阻止了气溶胶的挥发,并且对扩增反应无任何影响。
PSR反应过程中,大量DNA和不溶性的焦磷酸镁被合成,导致溶液浊度增加,浊度达到一定浓度时,可通过凝胶电泳观察形成特异性“梯形条带”[19]。因此PSR扩增产物的检测法主要有浊度仪检测和凝胶电泳检测。为符合快速简单的检测要求,我们采用钙黄绿素和羟基萘酚蓝为指示剂。钙黄绿素是一种螯合剂,在Mn2+存在时被淬灭不发光。随着PSR反应的进行,阳性反应形成的焦磷酸根与钙黄绿素竞争Mn2+,当钙黄绿素游离出来便可自发荧光,由原本的橘黄色变成亮绿色。本实验中,我们研究了不同浓度比的钙黄绿素与氯化锰(表 2)的显色效果,实验结果显示钙黄绿素50 μmol/L、氯化锰0.4 mmol/L时显色效果最佳,显色时间最短(图 3)。羟基萘酚蓝(HNB)是一种金属离子指示剂,高浓度金属离子环境下溶液呈紫色,低浓度金属离子环境下溶液呈天蓝色。在PSR反应开始前,游离的Mg2+浓度较高(8 mmol/L),加入HNB后溶液呈紫色,而当发生PSR反应后,游离的Mg2+不断消耗生成焦磷酸镁沉淀,导致溶液由紫色变为天蓝色。我们参考Goto等[24]的方法,采用0.2% HNB,能有效地显示PSR反应结果(图 4)。两种颜色指示剂均在反应前添加,使得反应从开始至结束均不需要打开反应管,可避免产生假阳性,检测时间短,直接观察颜色变化,肉眼即可判读结果。
通过灵敏度检测发现,PSR灵敏性高,最低检测限度可达到1.011 5 pg/μL (图 6)。同时,通过人工污染包装饮用水样品检测发现,PSR对包装饮用水样品中铜绿假单胞菌的检测限度可达20 CFU/mL,是常规PCR反应灵敏度的10倍(图 7)。通过建立的PSR方法检测包装饮用水来源的铜绿假单胞菌分离株,检出率为100% (图 8),表明该法可应用于实际的检测工作中。据我们所知,本研究首次实现对包装饮用水样品实现高灵敏度快速可视化检测。综上所述,本研究所建立的PSR检测技术,引物设计简单,只需一种酶,无需反复加样,能在恒温条件下完成反应,具有良好的特异性、敏感性、易操作性。同时使用适用于该技术的扩增结果快速判读方法,在反应基础上不再借助于浊度仪、电泳槽等任何检测仪器,直接通过肉眼观察即可判读实验结果,具有方便、快速的优点。
| [1] |
Micek ST, Lloyd AE, Ritchie DJ, et al. Pseudomonas aeruginosa bloodstream infection: importance of appropriate initial antimicrobial treatment[J]. Antimicrobial Agents and Chemotherapy, 2005, 49(4): 1306-1311. DOI:10.1128/AAC.49.4.1306-1311.2005 |
| [2] |
Niu ST. Microbiological quality of bottled water on the market and related regulations[J]. Foreign Medical Sciences (Section of Hygiene), 1994, 21(1): 31-34. (in Chinese) 牛胜田. 市售瓶装水的微生物学质量及有关条例[J]. 国外医学卫生学分册, 1994, 21(1): 31-34. |
| [3] |
Zhang SH, Wu QP, Xu XK, et al. Comparison of detection of Pseudomonas aeruginosa in bottled water[J]. Modern Food Science and Technology, 2011, 27(11): 1403-1405, 1335. (in Chinese) 张淑红, 吴清平, 徐晓可, 等. 桶装水中铜绿假单胞菌检测方法的比较[J]. 现代食品科技, 2011, 27(11): 1403-1405, 1335. |
| [4] |
Zhang XD. Interpretation of National Food Safety Standard Packaged drinking water (GB 19298-2014)[J]. The Beverage Industry, 2016, 19(2): 7-9. (in Chinese) 张旭东. 《食品安全国家标准包装饮用水》(GB 19298-2014)标准解读[J]. 饮料工业, 2016, 19(2): 7-9. |
| [5] |
Guo LP, Ren YH, Niu SW. Pollution investigation of Pseudomonas aeruginosa in bottled drinking water of Luohe City between 2013 and 2014[J]. Chinese Journal of Public Health Engineering, 2017, 16(1): 33-35. (in Chinese) 郭辽朴, 仁蕴慧, 牛世文. 2013-2014年漯河市桶装饮用水中铜绿假单胞菌污染状况调查[J]. 中国卫生工程学, 2017, 16(1): 33-35. |
| [6] |
Wang WJ, Yan Y, Luo YB, et al. Analysis of the contamination status of Pseudomonas aeruginosa in mineral water and packed drinking water in Jiangxi province[J]. Chinese Journal of Health Laboratory Technology, 2017, 27(3): 419-421. (in Chinese) 王文娟, 颜瑛, 罗玉彬, 等. 江西省矿泉水和包装饮用水中铜绿假单胞菌污染情况分析[J]. 中国卫生检验杂志, 2017, 27(3): 419-421. |
| [7] |
Chen S, Li H, Li DH, et al. Analysis of the contamination of barreled drinking water with P. aeruginosa[J]. Modern Preventive Medicine, 2015, 42(6): 1129-1130, 1135. (in Chinese) 陈松, 李红, 李德华, 等. 桶装饮用水中铜绿假单胞菌污染情况分析[J]. 现代预防医学, 2015, 42(6): 1129-1130, 1135. |
| [8] |
Wang YM, Tang Z, Qiao X, et al. Survey of Pseudomonas aeruginosa contamination in barreled water in Jiangsu province[J]. Chinese Journal of Health Laboratory Technology, 2015, 25(12): 2019-2020. (in Chinese) 王燕梅, 唐震, 乔昕, 等. 江苏省桶装饮用水中铜绿假单胞菌污染情况调查[J]. 中国卫生检验杂志, 2015, 25(12): 2019-2020. |
| [9] |
Luo YQ. Analysis of the microbial contaminations of packaged drinking purified water[J]. Journal of Food Safety and Quality, 2017, 8(1): 360-363. (in Chinese) 骆业巧. 包装饮用纯净水微生物污染情况分析[J]. 食品安全质量检测学报, 2017, 8(1): 360-363. |
| [10] |
Xiao XL, Zhang JW, Gong J, et al. Rapid detection of Pseudomonas aernginosa by the fluorescence quantitative TaqMan PCR assay targetting ETA gene[J]. Chinese Journal of Biotechnology, 2008, 24(4): 581-585. (in Chinese) 肖性龙, 张经纬, 龚俊, 等. ETA基因作为荧光定量PCR靶基因设计TaqMan探针快速检测铜绿假单胞菌的研究[J]. 生物工程学报, 2008, 24(4): 581-585. DOI:10.3321/j.issn:1000-3061.2008.04.009 |
| [11] |
Tomita N, Mori Y, Kanda H, et al. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products[J]. Nature Protocols, 2008, 3(5): 877-882. DOI:10.1038/nprot.2008.57 |
| [12] |
Wang H, Huang Q, Huang JF, et al. Rapid detections of OprD2 gene in Pseudomoas aeruginosa using loop-mediated isothermal amplification[J]. Chinese Journal of Laboratory Medicine, 2013, 36(6): 543-547. (in Chinese) 王欢, 黄庆, 黄君富, 等. 铜绿假单胞菌OprD2基因环介导等温扩增快速检测技术的研究[J]. 中华检验医学杂志, 2013, 36(6): 543-547. DOI:10.3760/cma.j.issn.1009-9158.2013.06.015 |
| [13] |
Xu YM, Zou DY, Tang F, et al. Design and analysis of LAMP primers used in differentially detecting Yersinia enterocolitica and Brucella spp.[J]. Chinese Journal of Laboratory Diagnosis, 2013, 17(1): 19-22. (in Chinese) 徐云明, 邹德颖, 唐峰, 等. 区别检测小肠结肠炎耶尔森氏菌与布鲁氏菌LAMP引物的设计与分析[J]. 中国实验诊断学, 2013, 17(1): 19-22. DOI:10.3969/j.issn.1007-4287.2013.01.006 |
| [14] |
Xiao LP. Application and study of LAMP technique in the detection of Salmonella in infectious diarrhea[J]. Chinese Journal of Health Laboratory Technology, 2015, 25(24): 4240-4242. (in Chinese) 肖丽萍. 环介导技术在感染性腹泻沙门菌检测中的应用与研究[J]. 中国卫生检验杂志, 2015, 25(24): 4240-4242. |
| [15] |
Zhou Y, Wan Q, Cai ZH, et al. Loop-mediated isothermal amplification-based rapid detection for Shigella dysenteriae in food samples[J]. Microbiology China, 2017, 44(9): 2247-2254. (in Chinese) 周杨, 万强, 蔡芷荷, 等. 环介导等温扩增技术在食品中痢疾志贺氏菌快速检测[J]. 微生物学通报, 2017, 44(9): 2247-2254. |
| [16] |
Ding WC, Hu JR, Shi YH, et al. Establishment of loop-mediated isothermal amplification for detection of Vibrio alginolyticus[J]. Journal of Molecular Cell Biology, 2009, 42(1): 70-76. (in Chinese) 丁文超, 胡健饶, 史雨红, 等. 环介导恒温扩增技术快速检测溶藻弧菌[J]. 分子细胞生物学报, 2009, 42(1): 70-76. |
| [17] |
Dong DR, Zou DY, Liu H, et al. Rapid detection of Pseudomonas aeruginosa targeting the toxA gene in intensive care unit patients from Beijing, China[J]. Frontiers in Microbiology, 2015, 6: 1100. |
| [18] |
Dong DR. The study of a new nucleic acid amplification technology termed Polymerase Spiral Reaction (PSR) and application in POCT detection[D]. Beijing: Doctoral Dissertation of Academy of Military Medical Sciences, 2016 (in Chinese) 董德荣.一种新型核酸恒温扩增方法的研究及其在现场检测中的应用[D].北京: 中国人民解放军军事医学科学院博士学位论文, 2016 http://cdmd.cnki.com.cn/Article/CDMD-90106-1016211528.htm |
| [19] |
Liu W, Dong DR, Yang Z, et al. Polymerase Spiral Reaction (PSR): a novel isothermal nucleic acid amplification method[J]. Scientific Reports, 2015, 5: 12723. DOI:10.1038/srep12723 |
| [20] |
Dong DR, Liu W, Li H, et al. Survey and rapid detection of Klebsiella pneumoniae in clinical samples targeting the rcsA gene in Beijing, China[J]. Frontiers in Microbiology, 2015, 6: 519. |
| [21] |
Liu W, Xu YH, Dong DR, et al. Survey and rapid detection of Bordetella pertussis in clinical samples targeting the BP485 in China[J]. Frontiers in Public Health, 2015, 3: 39. |
| [22] |
Kidd TJ, Grimwood K, Ramsay KA, et al. Comparison of three molecular techniques for typing Pseudomonas aeruginosa isolates in sputum samples from patients with cystic fibrosis[J]. Journal of Clinical Microbiology, 2011, 49(1): 263-268. DOI:10.1128/JCM.01421-10 |
| [23] |
Wang P, Zhang ZL, Qiao YS. Differentiation and identification of Pseudomonas aeruginosa and its interference strains by Fourier transform-infrared spectroscopy[J]. Food Science and Technology, 2017, 42(4): 302-305. (in Chinese) 王萍, 张占林, 乔勇升. 傅立叶变换红外光谱技术对铜绿假单胞菌及其干扰菌的分类鉴定[J]. 食品科技, 2017, 42(4): 302-305. DOI:10.3969/j.issn.1672-979X.2017.04.019 |
| [24] |
Goto M, Honda E, Ogura A, et al. Colorimetric detection of loop-mediated isothermal amplification reaction by using hydroxy naphthol blue[J]. Biotechniques, 2009, 46(3): 167-172. |
| [25] |
Iglewski BH, Kabat D. NAD-dependent inhibition of protein synthesis by Pseudomonas aeruginosa toxin[J]. Proceedings of the National Academy of Sciences of the United States of America, 1975, 72(6): 2284-2288. DOI:10.1073/pnas.72.6.2284 |
| [26] |
Gray GL, Smith DH, Baldridge JS, et al. Cloning, nucleotide sequence, and expression in Escherichia coli of the exotoxin A structural gene of Pseudomonas aeruginosa[J]. Proceedings of the National Academy of Sciences of the United States of America, 1984, 81(9): 2645-2649. DOI:10.1073/pnas.81.9.2645 |
| [27] |
Zhang W, Li W, Zhang WW, et al. The methodological study on rapid identification of Pseudomonas aeruginosa based on PCR technique[J]. Chinese Journal of Health Laboratory Technology, 2005, 15(9): 1065-1067. (in Chinese) 张伟, 李闻, 张伟尉, 等. 基于PCR技术的绿脓杆菌快速检测方法研究[J]. 中国卫生检验杂志, 2005, 15(9): 1065-1067. DOI:10.3969/j.issn.1004-8685.2005.09.016 |
| [28] |
Khan AA, Cerniglia CE. Detection of Pseudomonas aeruginosa from clinical and environmental samples by amplification of the exotoxin A gene using PCR[J]. Applied and Environmental Microbiology, 1994, 60(10): 3739-3745. |
| [29] |
Qin X, Emerson J, Stapp J, et al. Use of real-time PCR with multiple targets to identify Pseudomonas aeruginosa and other nonfermenting gram-negative bacilli from patients with cystic fibrosis[J]. Journal of Clinical Microbiology, 2003, 41(9): 4312-4317. DOI:10.1128/JCM.41.9.4312-4317.2003 |
| [30] |
Xu JR, Moore JE, Murphy PG, et al. Early detection of Pseudomonas aeruginosa—comparison of conventional versus molecular (PCR) detection directly from adult patients with cystic fibrosis (CF)[J]. Annals of Clinical Microbiology and Antimicrobials, 2004, 3: 21. DOI:10.1186/1476-0711-3-21 |
 2019, Vol. 46
2019, Vol. 46