扩展功能
文章信息
- 赵美琳, 诸葛斌, 陆信曜, 宗红, 施丁昌
- ZHAO Mei-Lin, ZHUGE Bin, LU Xin-Yao, ZONG Hong, SHI Ding-Chang
- 工业酵母抗逆机理研究进展
- Research progress in stress tolerance of industrial yeasts
- 微生物学通报, 2019, 46(5): 1155-1164
- Microbiology China, 2019, 46(5): 1155-1164
- DOI: 10.13344/j.microbiol.china.180446
-
文章历史
- 收稿日期: 2018-06-05
- 接受日期: 2018-12-17
- 网络首发日期: 2019-01-28
2. 江南大学生物工程学院 工业微生物研究中心 江苏 无锡 214122
2. Research Center of Industrial Microbiology, School of Biotechnology, Jiangnan University, Wuxi, Jiangsu 214122, China
随着社会和经济的不断发展,日益增长的能源需求与传统化石燃料使用带来的环境污染问题成为世界能源发展的主要矛盾[1],这一矛盾促使政府和能源部门着手寻求及开发对环境友好、可持续的液体能源。美国能源信息局预测,到2019年全球对液体燃料的需求将达到1亿桶[2]。作为潜在的原油替代能源,生物乙醇、丁醇等不仅能够减轻原油消耗带来的负担,也能减少原油使用造成的环境污染。产甘油假丝酵母、粟酒裂殖酵母和马克斯克鲁维酵母等工业酵母能够利用各种不同的原料发酵生产乙醇和甘油等化合物[3-5]。木质纤维素与蔗糖和淀粉等原料相比,存在更广泛、更容易获取并且不与人类食物竞争,近年来被作为乙醇等化合物生产的主要原料[6]。然而,木质纤维素水解过程中产生乙酸、糠醛和羟甲基糠醛(HMF)等抑制物,其种类及浓度主要取决于生物质原料结构及水解类型、强度。此外,浓醪发酵高浓度底物糖引起的高渗透压、发酵过程中温度的升高以及产物大量积累等均会抑制细胞生长[7]。
提高酵母的发酵抗逆性是解决以上问题的关键途径。已有研究发现产甘油假丝酵母等工业酵母对多种抑制因素具有高抗逆性[5, 8]。掌握酵母多压力抗逆机制,有助于科学设计基因改造路线,从基因水平定向改造酵母细胞,提高酵母细胞的多压力抗逆性,同时从根本上解决发酵过程中抑制因素影响细胞生长等问题。本文着重总结了工业酵母对渗透压、高温、木质纤维素水解液抑制物等因素的抗逆机制。
1 工业酵母渗透压抗逆机制节能高效的浓醪发酵具有重要的经济价值[9],但浓醪发酵高浓度底物引起的高渗透压会导致细胞内水分流失,引起细胞萎缩,影响细胞正常生长。酵母的高渗甘油应答途径(HOG途径)能够快速调控细胞参与外部渗透压的响应。
产甘油假丝酵母能够在含55%葡萄糖或15% NaCl的培养基上正常生长[3],具有耐高渗、抗逆性强等特点。已有研究发现,Hog1 MAP激酶(CgHog1p)是产甘油假丝酵母HOG途径的核心分子,与来源于酿酒酵母的ScHog1p具有类似的保守功能[8]。产甘油假丝酵母基因Cghog1缺失会降低细胞对渗透压等外界压力的抗逆性。Cghog1缺失株在含0.2 mol/L NaCl的培养基中几乎不能生长,而酿酒酵母Schog1缺失株在含0.5 mol/L NaCl的培养基中才表现出显著的生长抑制。因此,与酿酒酵母相比,产甘油假丝酵母对渗透压的压力响应更依赖于由Hog1p介导的HOG途径[8]。
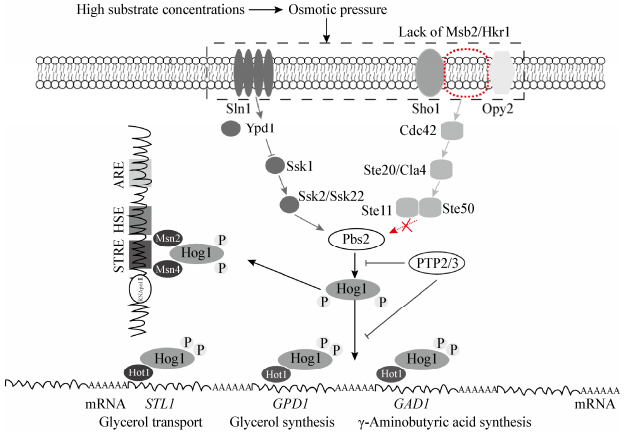
酿酒酵母受到渗透压刺激后,上游分支途径Sho1和Sln1激活Hog1p使其双重磷酸化,双重磷酸化的Hog1p快速进入细胞核并激活多个下游转录因子(Hot1p、Msn2/4p等)控制基因的表达[10]。转录因子Hot1p通过调控参与甘油合成的3-磷酸甘油脱氢酶基因(gpd)和3-磷酸甘油磷酸酶基因(gpp),以及参与甘油转运的甘油-质子同向转运蛋白基因(stl)和甘油-水通道蛋白基因(fps1)的表达,增加胞内甘油积累来抵御外界渗透压力。转录因子Msn2/4p则主要是通过结合位于启动子上的压力响应元件(STRE)来诱导基因的表达[11]。通过生物信息学比对发现,产甘油假丝酵母在Sho1分支途径上缺乏两个重要的膜上感受器Msb2和Hkr1,以及Ste11 MAPKKK上的SAM结构域,推测产甘油假丝酵母HOG途径可能仅依赖于Sln1分支途径,这一推测仍有待证实(图 1)。与酿酒酵母不同,产甘油假丝酵母只存在一个gpd基因[12],并且未发现gpp的同源基因。产甘油假丝酵母gpd缺失株在高浓度葡萄糖(350 g/L)条件下生长,最终生物量不受影响;而阻断甘油合成的酿酒酵母在高于150 g/L葡萄糖的培养基中几乎不能生长[13]。因此,与酿酒酵母相比,产甘油假丝酵母可能还存在其他的渗透压保护机制。此外,与只存在一个Stl1蛋白的酿酒酵母不同,产甘油假丝酵母含有Cgstl1和Cgstl2两个甘油转运蛋白,值得注意的是,两个Stl受不同的转录调节。其中,Hog1p通过调控Cgstl1的表达均衡胞内的甘油,保护细胞正常生长,而Cgstl2呈组成型方式表达,其表达基本不受渗透压影响[14]。
酵母在渗透压胁迫下,还可通过调节细胞内氨基酸的含量来增加细胞对外界渗透压的响应。在产甘油假丝酵母中,细胞受到渗透压刺激后胞内γ-氨基丁酸含量有所提高。外源添加γ-氨基丁酸或过表达γ-氨基丁酸关键合成酶谷氨酸脱羧酶基因(Cggad1),能够促进细胞在高渗条件下的生长,增强酵母的耐高渗能力[15]。有研究发现在酿酒酵母中γ-氨基丁酸通过减少活性氧的产生来增强细胞的热抗逆性[16],但尚未见报道证实渗透压诱导了γ-氨基丁酸的积累过程。以上这些差异也许是产甘油假丝酵母比酿酒酵母具有更好的渗透压抗逆能力的原因。
目前,酵母的渗透压抗逆机制主要集中于酿酒酵母,对产甘油假丝酵母等具有高渗透压抗逆性的工业酵母研究尚存不足,仍需从基因水平、转录水平、蛋白水平等多个方面来探究工业酵母的高渗抗逆机制。
2 工业酵母热抗逆机制在工业酵母发酵过程中,温度的升高会破坏酵母细胞膜,造成细胞损伤,影响细胞生长和代谢。提高工业酵母的热抗逆性是解决发酵过程中温度影响的重要途径。已有研究发现,马克斯克鲁维酵母细胞膜中高比例的饱和脂肪酸能够减少热对细胞的损伤[17]。细胞膜中麦角固醇含量的增加,也能够进一步提高酵母的热抗逆性[18]。此外,高温能够激活酵母细胞壁合成途径(CWI途径),诱导该合成途径相关的基因大量表达,通过增强细胞壁的稳定性能有效的改善酵母的耐热能力[19]。参与酵母热压力响应的关键基因及其作用机制见表 1。
| 菌株 Strains |
基因 Genes |
作用机制 Functional mechanisms |
参考文献 References |
| 热抗逆机制 Tolerance mechanisms in response to heat |
|||
| 酿酒酵母 Saccharomyces cerevisiae |
dfg5 | DFG5缺失增加细胞热稳定 Deletion of DFG5 enhancing the thermal stability of cells |
[20] |
| 马克斯克鲁维酵母 Kluyveromyces marxianus |
msn2 | 调控脂质代谢相关基因的表达 Regulating expression of lipid metabolism-related genes |
[21] |
| 酿酒酵母 S. cerevisiae |
cyr1 | 介导营养信号的感知和传导 Mediating of sensing and transmitting nutrient signals |
[22] |
| 酿酒酵母 S. cerevisiae |
fmp21 | 参与细胞热压力响应 Involving in response to heat stress |
[23] |
| 产甘油假丝酵母 C. glycerinogenes |
dip5 | 参与胞内相溶性物质积累 Involving in accumulation of compatible solutes |
[24] |
| 产甘油假丝酵母 C. glycerinogenes |
gpd | 参与胞内甘油积累 Involving in glycerol accumulation |
[24] |
| 产甘油假丝酵母 C. glycerinogenes |
ino4 | 参与膜脂质代谢 Involving in membrane lipid metabolism |
[24] |
| 产甘油假丝酵母 C. glycerinogenes |
rpa190 | 核糖体功能相关基因 Ribosomal function related gene |
[24] |
| 乙酸抗逆机制 Tolerance mechanisms in response to acetic acid |
|||
| 酿酒酵母 S. cerevisiae |
pep3 | 增加细胞液泡表面积和液泡V-ATP酶的活性 Increasing vacuolar surface area and V-ATPase activity |
[25] |
| 酿酒酵母 S. cerevisiae |
cox20 | 促进细胞色素C释放 Facilitating cytochrome C release |
[26] |
| 酿酒酵母 S. cerevisiae |
acs2 | 减少胞内乙酸根的积累 Decreasing accumulation of acetic acid |
[27] |
| 酿酒酵母 S. cerevisiae |
pdr12 | 减少胞内乙酸根的积累 Decreasing accumulation of acetic acid |
[28] |
| 酿酒酵母 S. cerevisiae |
ady2 | ADY2缺失减少细胞内乙酸的积累 Deletion of ADY2 decreasing accumulation of acetic acid |
[29] |
| 酿酒酵母 S. cerevisiae |
haa1 | 弱酸转录因子参与80%乙酸应答基因的转录 Involving in the activation of 80% of the acetic acid-responsive genes |
[30] |
| 酿酒酵母 S. cerevisiae |
yro2 | Haa1p的靶基因 Target gene of Haa1p |
[31-32] |
| 酿酒酵母 S. cerevisiae |
hrk1 | 参与转运蛋白磷酸化来调控细胞质膜转运 Involving in phosphorylation of transport-related proteins to regulate membrane transport |
[33] |
| 酿酒酵母 S. cerevisiae |
whi2 | 结合Psr1组成Whi2-Psr1功能复合体 Forming a functional complex Whi2-Psr in combination with Psr1 |
[34] |
| 产甘油假丝酵母 C. glycerinogenes |
haa1 | 参与细胞乙酸压力响应 Involving in response to acetic acid stress |
[35] |
| 呋喃醛类抑制物抗逆机制 Tolerance mechanisms in response to furan aldehyde inhibitors |
|||
| 酿酒酵母 S. cerevisiae |
idh1 | 参与糠醛降解 Involving in furfural degradation |
[36] |
| 酿酒酵母 S. cerevisiae |
dic1 | 调节细胞生长极限通量,加速糠醛降解 Regulating limiting flux for cell growth and facilitating furfural degradation |
[36] |
| 酿酒酵母 S. cerevisiae |
adh7 | 参与糠醛、HMF降解 Involving in furfural and HMF degradation |
[37] |
| 酿酒酵母 S. cerevisiae |
gre2 | 参与糠醛降解 Involving in furfural degradation |
[38] |
| 酿酒酵母 S. cerevisiae |
YKL071W | 编码SDRs醛还原酶,参与糠醛降解 Encoding a short-chain dehydrogenase/reductase and involving in furfural degradation |
[39] |
| 酿酒酵母 S. cerevisiae |
YNL134C | 编码NADH-依赖的醛还原酶,参与糠醛降解 Encoding a NADH-dependent aldehyde reductase and involving in furfural degradation |
[40] |
| 酿酒酵母 S. cerevisiae |
YLL056C | 编码SDRs醛还原酶,参与糠醛、HMF降解 Encoding a short-chain dehydrogenase/reductase and involving in furfural and HMF degradation |
[41] |
| 酿酒酵母 S. cerevisiae |
yap1 | 参与多种醛还原酶的转录调控,并激活相关转运蛋白的表达 Involving in transcriptional regulation of various aldehyde reductases and activating expression of related transporters |
[42] |
酿酒酵母编码GPI-锚定质膜蛋白的基因dfg5缺失,使得胞内活性氧含量积累减少,细胞膜通透性降低,显著提高了细胞的热抗逆性;此外,dfg5缺失株在受到热刺激后,轻微激活HOG途径的Hog1p及CWI途径的Slt2,进而增加细胞对外界压力的抗逆性[20]。转录因子Msn2p通过调控脂质代谢相关基因的表达,改变细胞膜的流动性,增加细胞热稳定性;在酿酒酵母中过表达来自马克斯克鲁维酵母的基因Kmmsn2,重组酿酒酵母在高温条件下生长明显改善,乙醇产量提高了50%[21]。
热激蛋白在酵母热抗逆机制中起关键作用,通过激活热激转录因子(HSF),引起编码热激蛋白的基因(hsp40、hsp70、hsp104等)转录上调,增加热激蛋白的表达,进一步提高细胞内可溶性蛋白、超氧化物歧化酶(SOD)、膜ATP酶和质子泵的热稳定性,保护细胞免受热损伤,使细胞正常生长[43]。有研究发现环腺苷酸(cAMP)介导营养信号的感知和传导,参与细胞对热的压力响应;通过对编码腺苷酸环化酶基因(cyr1)的启动子修饰,调控cyr1的表达改变胞内cAMP的含量,最终细胞的耐热能力及乙醇发酵性能均得到提高[22]。Nakamura等研究发现位于线粒体的蛋白Fmp21也与细胞热响应相关;基因fmp21的缺失,增加了细胞的热敏感性,致使细胞在高温条件下的生长速率降低[23]。此外,外源添加钙离子(Ca2+),通过增加细胞的抗氧化酶活性、降低细胞内活性氧(ROS)积累、减少蛋白的氧化损伤等方面改善细胞的热稳定性[44]。酿酒酵母这些热抗逆机制的研究为产甘油假丝酵母等工业酵母提供了借鉴。
Yang等通过qRT-PCR考察产甘油假丝酵母与酿酒酵母在高温、联合高温高糖条件下基因表达水平变化,研究发现编码二羧酸氨基酸通透酶(Dip5)、Gpd和磷脂生物合成的积极调节因子(Ino4)等参与胞内相溶性物质积累以及膜脂质代谢相关酶的基因是产甘油假丝酵母耐高温高糖的关键基因[24]。与酿酒酵母不同,酵母RNA聚合酶A大亚基基因(rpa190)、60S核糖体亚基生物起源蛋白基因(nop53)、核糖体成熟因子基因(mrt4)等核糖体功能相关基因也参与了产甘油假丝酵母对高温高糖的压力应答响应[24]。核糖体功能相关基因的激活表达可能是联合高温高糖条件下产甘油假丝酵母较酿酒酵母具有优良抗逆能力的重要因素。
3 工业酵母乙酸抗逆机制在木质纤维素预处理过程中,乙酰化的半纤维素完全水解释放乙酸。在低pH条件下,乙酸改变细胞膜蛋白构象及脂质组成,破坏细胞膜稳定性。未解离的乙酸通过简单扩散或甘油-水通道蛋白进入酵母细胞,解离成质子(H+)和乙酸根(CH3COO-),造成胞内过量质子的积累和细胞酸化。高浓度的乙酸诱导胞内ROS的积累,破坏DNA及蛋白的结构和功能,造成细胞营养和能量缺乏,从而影响细胞生长和代谢,导致细胞程序性死亡[45]。
Dong等通过转录组学分析发现,酿酒酵母受乙酸压力刺激后,有多达295个基因发生了不同程度的转录上调,这些基因主要参与胞内稳态平衡、转录调控以及蛋白折叠与稳定等[45]。参与酵母乙酸压力响应的关键基因及其作用机制见表 1。维持细胞内pH稳定是酿酒酵母乙酸抗逆机制的关键[25, 46]。酿酒酵母通过质膜H+-ATP酶和液泡V-ATP酶排出细胞内过量积累的质子来维持胞内pH稳定,以维持细胞正常生长[46],这一点在Ding等的研究中得到了进一步的证实,过表达参与液泡合成的基因(pep3),增加细胞液泡表面积和液泡V-ATP酶的活性,减轻乙酸对细胞的损害[25]。此外,分子伴侣Cox20也参与酿酒酵母乙酸压力响应;Cox20能够促进线粒体内膜细胞色素C氧化酶复合体前体Cox2p水解,释放细胞色素C,从而响应乙酸引起的氧化应激,并可作为线粒体外膜的一部分参与ATP/ADP的转运[26]。通过增强参与维持胞内pH稳定的相关基因及cox20的表达可以作为进一步提高酿酒酵母乙酸抗逆性的手段。除胞内过量积累的质子外,乙酸根的累积也影响细胞生长[27]。细胞内的乙酸根可经乙酰-CoA合成酶或过氧化物酶进一步转化为乙酰-CoA,随后进入TCA循环或乙醛酸循环进行代谢。过表达编码乙酰-CoA合成酶基因(acs2),减少胞内乙酸根的积累,能够显著改善细胞的乙酸抗逆能力[27]。乙酸根也可通过ABC转运蛋白(Pdr12p等)排出胞外[28]。此外,Zhang等通过研究发现,敲除乙酸转运蛋白基因(ady2)能够减少酿酒酵母胞内乙酸的积累;在3.6 g/L乙酸存在的条件下,ady2缺失株乙醇产量提高了14.7%[29]。
在乙酸胁迫下,酿酒酵母中参与转录调控、蛋白折叠与稳定的基因发生了不同程度的转录变化[27]。研究发现弱酸转录因子Haa1p参与酿酒酵母中80%乙酸应答基因的转录[30]。基因yro2编码质膜-结合热休克蛋白,是转录因子Haa1p的靶基因之一,Haa1p通过Msn2/4p等转录因子间接激活Yro2的转录[31]。其中,Yro2及其同源蛋白Mrh1并不直接参与酵母细胞在细胞质中对乙酸的代谢,而是通过参与细胞膜活动,调节H+-ATP酶和Fps1阻止乙酸进入细胞,减缓乙酸对细胞的损伤[32]。编码来自Npr1-激酶家族的丝氨酸/苏氨酸激酶基因(hrk1),是转录因子Haa1p的另一靶基因;Hrk1通过参与转运蛋白磷酸化来调控细胞质膜转运,促进细胞对乙酸的响应[33]。Swinnen等应用全转录调控机制对转录因子Haa1p进行改造,将氨基酸序列第135位丝氨酸突变为苯丙氨酸,这一改变进一步增强了菌株的乙酸抗逆性[30]。细胞质球状支架蛋白Whi2与其同源的磷酸酶Psr1结合组成Whi2-Psr1功能复合体,作用于转录因子Msn2/4p去磷酸化,诱导相关乙酸应答元件表达;过表达基因whi2能显著提高酿酒酵母对乙酸的抗逆性[34]。此外,Kawazoe等研究发现乙酸压力条件下内质网未折叠蛋白聚集水平升高,激活Ire1p-Hac1p途径,诱导非折叠蛋白反应,从而避免内质网压力,维持或者提高蛋白的折叠能力,增强酿酒酵母对乙酸的抗逆能力[28]。酿酒酵母这些乙酸抗逆机制的研究为产甘油假丝酵母等工业酵母提供了借鉴。
已有研究发现产甘油假丝酵母具有优于酿酒酵母的乙酸抗逆性;过表达来自产甘油假丝酵母的基因Cghaa1,与酿酒酵母自身的Schaa1相比,能显著改善重组酿酒酵母的乙酸抗逆性;转录因子CgHaa1p与ScHaa1p具有不同的DNA结合域,分别为Met1-K60与Met1-Ser40,推测前者能更有效地与下游靶基因结合,从而起到更强的调控作用[35]。产甘油假丝酵母与酿酒酵母的转录因子Haa1p影响细胞抗乙酸能力的差异,表明产甘油假丝酵母可能具有更强的抗逆元件参与细胞对乙酸压力的响应。然而,目前对产甘油假丝酵母等工业酵母的乙酸抗逆机制研究尚不足,借鉴酿酒酵母对工业酵母乙酸胁迫下参与胞内pH稳定、乙酸转运、内质网应激反应等相关基因进行深入解析。
4 酵母呋喃醛类抑制物抗逆机制木质纤维素水解液中戊糖和己糖脱水分别产生糠醛和HMF等呋喃醛类物质。在酵母细胞中,糠醛和HMF作为一种硫醇反应亲电试剂,激活转录因子Yap1p,减少细胞内谷胱甘肽含量,引起胞内ROS积累,造成DNA损伤,抑制蛋白和RNA的合成。高的硫醇活性使得糠醛与HMF相比具有更强的毒性[42, 47]。此外,糠醛和HMF也会影响细胞糖酵解及乙醇合成途径关键酶的活性,破坏细胞膜组分和通透性,从而抑制细胞生长。
参与酵母呋喃醛类抑制物压力响应的关键基因及其作用机制见表 1。Unrean等通过研究发现在酿酒酵母中过量表达控制乙醇合成速率的异柠檬酸脱氢酶(Idh1),菌株的糠醛脱毒速率和乙醇产量分别提高了2.0倍和1.2倍;此外,二羧酸转运蛋白(Dic1)调节细胞生长极限通量,增加酿酒酵母中dic1的表达,细胞生长速率和糠醛脱毒速率分别提高了1.3倍和1.4倍[36]。过表达乙醇脱氢酶(Adh7),使得重组酿酒酵母的糠醛和HMF抗逆能力增强[37]。与Adh7相似,有研究发现醛还原酶(Gre2)也参与糠醛和HMF的压力响应,通过氨基酸序列突变等手段提高Gre2的酶活性是改善细胞糠醛等压力抗逆性的有效策略[38]。酿酒酵母可以在NAD(P)H脱氢酶/还原酶作用下降解糠醛或HMF,减少糠醛或HMF对细胞的毒害作用,维持细胞正常生长。通过增强酵母对糠醛的降解提高工业酵母的糠醛抗逆性是解除发酵过程中糠醛影响的主要途径。除Idh1、Adh7和Gre2外,酿酒酵母细胞中还存在其他脱氢酶/还原酶及其糠醛抗逆机制[39-41, 47]。
酵母细胞中脱氢酶/还原酶超家族主要分为3种:长链(LDRs)、中链(MDRs)和短链脱氢酶/还原酶超家族(SDRs)。研究发现多数中链、短链脱氢酶/还原酶能够降解糠醛和HMF,不同来源的脱氢酶/还原酶遵循同一种机制来响应压力胁迫[39]。Li等在酿酒酵母中发现一种来源于MDRs的醛还原酶,受转录因子Yap1p、Msn2/4p及Hsf1p调控参与糠醛降解[48]。基因YNL134C编码NADH依赖的醛还原酶,也参与酿酒酵母对糠醛的压力响应,其中转录因子Yap1p、Msn2/4p参与该基因的转录调控[40]。随后,由基因YLL056C编码的醛还原酶被证实属于SDRs,在高浓度糠醛或HMF作用下,Pdr1/3p、Yrr1p和Stb5p等转录因子调控该基因上调表达[41]。同样来源于SDRs的醛还原酶YKL071W对糠醛和乙醇醛具有较强的降解作用,其中参与调控的转录因子分别为Yap1p、Cad1p、Pdr3p和Stb5p[39]。除参与糠醛、HMF降解的脱氢酶/还原酶外,有研究发现转运蛋白MDR受转录因子Yap1p和Stb5p调控,通过参与细胞氧化应激响应作用于糠醛和HMF[49]。Yap1p除参与多种醛还原酶的转录调控外,也激活抗氧化酶基因(cat1、gsh1、glr1)与转运蛋白基因(flr1、snq2)的表达[42]。目前,对酵母细胞糠醛抗逆机制的研究主要集中于NAD(P)H脱氢酶/还原酶作用下糠醛或HMF降解。研究人员获得许多参与糠醛降解的脱氢酶/还原酶基因,以及可能调控相关基因转录的转录因子,但除Yap1p外,缺乏对其他转录因子的进一步研究,阻碍了糠醛响应机制的深入讨论及在发酵领域中的应用,基于转录水平的酵母细胞糠醛抗逆机制仍需进一步研究。
通过基因组学和转录组学分析发现酿酒酵母NRRLY-12632存在3种MAPK信号途径参与细胞对HMF的压力响应,其中CWI途径和PI信号途径起主要作用[45]。Jung等通过代谢组学研究发现,酵母细胞通过调节甘油、二糖等应激-保护分子的合成,改善糖酵解活性,提高亚精胺和甲硫氨酸的合成,维持辅因子NAD(P)H平衡来响应糠醛胁迫[50]。目前,酵母的抗逆机制主要集中于酿酒酵母,其糠醛抗逆机制研究为产甘油假丝酵母等工业酵母提供了借鉴,后续可从糠醛降解、转运以及MAPK-信号途径等多个方面来进行深入研究。
5 总结如何提高发酵过程中抑制环境压力下酵母的发酵性能,是工业酵母利用木质纤维素为原料以及环保节能型浓醪发酵生产亟待解决的首要问题之一。提高酵母的发酵抗逆性,获得在各种环境压力刺激下仍能维持高效代谢活动的酵母菌株,是解决以上问题的关键。研究者主要通过:(1)应用适应性进化,随机突变等技术筛选具有优良压力抗逆性的工业酵母菌株;(2)基于酵母抗逆机制相关研究,获得相关压力抗逆基因,通过同源、异源表达或敲除相关基因,提高菌株的环境压力抗逆性,改善菌株发酵性能。
增强工业酵母抗逆性、提高工业酵母利用木质纤维素为原料以及环保节能型浓醪发酵性能,在过去的几年中取得了重要突破,但目前对工业酵母抗逆机制的研究主要着重于酿酒酵母,对其他工业酵母研究不足。与酿酒酵母相比,产甘油假丝酵母、马克斯克鲁维酵母等对渗透压、温度、乙酸等环境压力具有较强的抗逆性。基于转录组学和代谢组学等技术,深入研究相关抗逆机制,挖掘工业酵母中压力应答基因及相关转录因子,通过基因工程技术等进一步改善菌株的环境压力抗逆性,提高菌株发酵性能。此外,可将这些具有优良特性的工业酵母作为底盘细胞进行发酵获得包括乙醇在内的各种化合物。
| [1] |
Wu YM, Ge JP. A literature review on biological fuel ethanol industry and outlook for in-depth study[J]. Journal of Anhui Agricultural Sciences, 2013, 41(9): 4008-4012. (in Chinese) 吴永民, 葛建平. 生物燃料乙醇产业研究综述与展望[J]. 安徽农业科学, 2013, 41(9): 4008-4012. DOI:10.3969/j.issn.0517-6611.2013.09.088 |
| [2] |
Kadhum HJ, Rajendran K, Murthy GS. Effect of solids loading on ethanol production: Experimental, Economic and Environmental analysis[J]. Bioresource Technology, 2017, 244: 108-116. DOI:10.1016/j.biortech.2017.07.047 |
| [3] |
Zhuge J, Fang HY, Wang ZX, et al. Glycerol production by a novel osmotolerant yeast Candida glycerinogenes[J]. Applied Microbiology and Biotechnology, 2001, 55(6): 686-692. DOI:10.1007/s002530100596 |
| [4] |
Wang LJ, Dong Y. Ethanol production from jerusalem artichoke using Schizosaccharomyces pombe[J]. Transactions of the Chinese Society for Agricultural Machinery, 2010, 41(1): 106-110, 116. (in Chinese) 汪伦记, 董英. 粟酒裂殖酵母发酵菊芋生产燃料乙醇试验[J]. 农业机械学报, 2010, 41(1): 106-110, 116. DOI:10.3969/j.issn.1000-1298.2010.01.021 |
| [5] |
Pilap W, Thanonkeo S, Klanrit P, et al. The potential of the newly isolated thermotolerant Kluyveromyces marxianus for high-temperature ethanol production using sweet sorghum juice[J]. Biotech, 2018, 8: 126. |
| [6] |
Khoo HH. Review of bio-conversion pathways of lignocellulose-to-ethanol: sustainability assessment based on land footprint projections[J]. Renewable and Sustainable Energy Reviews, 2015, 46: 100-119. DOI:10.1016/j.rser.2015.02.027 |
| [7] |
Almeida JRM, Modig T, Petersson A, et al. Increased tolerance and conversion of inhibitors in lignocellulosic hydrolysates by Saccharomyces cerevisiae[J]. Journal of Chemical Technology and Biotechnology, 2007, 82(4): 340-349. DOI:10.1002/(ISSN)1097-4660 |
| [8] |
Ji H, Zhuge B, Zong H, et al. Role of CgHOG1 in stress responses and glycerol overproduction of Candida glycerinogenes[J]. Current Microbiology, 2016, 73(6): 827-833. DOI:10.1007/s00284-016-1132-7 |
| [9] |
Zhang Q, Han DM, Li MT. Research progress of high-concentration mash ethanol fermentation techniques[J]. Chemical Industry and Engineering Progress, 2014, 33(3): 724-729. (in Chinese) 张强, 韩德明, 李明堂. 乙醇浓醪发酵技术研究进展[J]. 化工进展, 2014, 33(3): 724-729. |
| [10] |
de Naal E, Alepuz PM, Posas F. Dealing with osmostress through MAP kinase activation[J]. EMBO Reports, 2002, 3(8): 735-740. DOI:10.1093/embo-reports/kvf158 |
| [11] |
Petelenz-Kurdziel E, Kuehn C, Nordlander B, et al. Quantitative analysis of glycerol accumulation, glycolysis and growth under hyper osmotic stress[J]. PLoS Computational Biology, 2014, 10(5): e1003663. DOI:10.1371/journal.pcbi.1003663 |
| [12] |
Chen XZ, Fang HY, Rao ZM, et al. Cloning and characterization of a NAD+-dependent glycerol-3-phosphate dehydrogenase gene from Candida glycerinogenes, an industrial glycerol producer[J]. FEMS Yeast Research, 2008, 8(5): 725-734. DOI:10.1111/fyr.2008.8.issue-5 |
| [13] |
Ding WT, Zhang GC, Liu JJ. 3' Truncation of the GPD1 promoter in Saccharomyces cerevisiae for improved ethanol yield and productivity[J]. Applied and Environmental Microbiology, 2013, 79(10): 3273-3281. DOI:10.1128/AEM.03319-12 |
| [14] |
Ji H, Lu XY, Zong H, et al. Functional and expression studies of two novel STL1 genes of the osmotolerant and glycerol utilization yeast Candida glycerinogenes[J]. The Journal of General and Applied Microbiology, 2018, 64(3): 121-126. DOI:10.2323/jgam.2017.10.001 |
| [15] |
Ji H, Lu XY, Zong H, et al. γ-aminobutyric acid accumulation enhances the cell growth of Candida glycerinogenes under hyperosmotic conditions[J]. The Journal of General and Applied Microbiology, 2018, 64(2): 84-89. DOI:10.2323/jgam.2017.08.002 |
| [16] |
Cao JX, Barbosa JM, Singh NK, et al. Gaba shunt mediates thermotolerance in Saccharomyces cerevisiae by reducing reactive oxygen production[J]. Yeast, 2013, 30(4): 129-144. DOI:10.1002/yea.v30.4 |
| [17] |
Mejía-Barajas J, Montoya-Pérez R, Manzo-Avalos S, et al. Fatty acid addition and thermotolerance of Kluyveromyces marxianus[J]. FEMS Microbiology Letters, 2018, 365(7). DOI:10.1093/femsle/fny043 |
| [18] |
Caspeta L, Chen Y, Ghiaci P, et al. Altered sterol composition renders yeast thermotolerant[J]. Science, 2014, 346(6205): 75-78. DOI:10.1126/science.1258137 |
| [19] |
Heilmann CJ, Sorgo AG, Mohammadi S, et al. Surface stress induces a conserved cell wall stress response in the pathogenic fungus Candida albicans[J]. Eukaryotic Cell, 2013, 12(2): 254-264. DOI:10.1128/EC.00278-12 |
| [20] |
Nasution O, Lee J, Srinivasa K, et al. Loss of Dfg5 glycosylphosphatidylinositol-anchored membrane protein confers enhanced heat tolerance in Saccharomyces cerevisiae[J]. Environmental Microbiology, 2015, 17(8): 2721-2734. DOI:10.1111/1462-2920.12649 |
| [21] |
Li PS, Fu XF, Zhang L, et al. The transcription factors Hsf1 and Msn2 of thermotolerant Kluyveromyces marxianus promote cell growth and ethanol fermentation of Saccharomyces cerevisiae at high temperatures[J]. Biotechnology for Biofuels, 2017, 10: 289. DOI:10.1186/s13068-017-0984-9 |
| [22] |
Hou XY, Hong KQ, Hao AL, et al. Truncation of CYR1 promoter in industrial ethanol yeasts for improved ethanol yield in high temperature condition[J]. Process Biochemistry, 2017, 65: 37-45. |
| [23] |
Nakamura T, Yamamoto M, Saito K, et al. Identification of a gene, FMP21, whose expression levels are involved in thermotolerance in Saccharomyces cerevisiae[J]. AMB Express, 2014, 4: 67. DOI:10.1186/s13568-014-0067-2 |
| [24] |
Yang F, Lu XY, Zong H, et al. Gene expression profiles of Candida glycerinogenes under combined heat and high-glucose stresses[J]. Journal of Bioscience and Bioengineering, 2018, 126(4): 464-469. DOI:10.1016/j.jbiosc.2018.04.006 |
| [25] |
Ding J, Holzwarth G, Bradford CS, et al. PEP3 overexpression shortens lag phase but does not alter growth rate in Saccharomyces cerevisiae exposed to acetic acid stress[J]. Applied Microbiology and Biotechnology, 2015, 99(20): 8667-8680. DOI:10.1007/s00253-015-6708-9 |
| [26] |
Kumar V, Hart AJ, Keerthiraju ER, et al. Expression of mitochondrial cytochrome C oxidase chaperone gene (COX20) improves tolerance to weak acid and oxidative stress during yeast fermentation[J]. PLoS One, 2015, 10(10): e0139129. DOI:10.1371/journal.pone.0139129 |
| [27] |
Ding J, Holzwarth G, Penner MH, et al. Overexpression of acetyl-CoA synthetase in Saccharomyces cerevisiae increases acetic acid tolerance[J]. FEMS Microbiology Letters, 2015, 362(3): 1-7. |
| [28] |
Kawazoe N, Kimata Y, Izawa S. Acetic acid causes endoplasmic reticulum stress and induces the unfolded protein response in Saccharomyces cerevisiae[J]. Frontiers in Microbiology, 2017, 8: 1192. DOI:10.3389/fmicb.2017.01192 |
| [29] |
Zhang MM, Zhang KY, Mehmood MA, et al. Deletion of acetate transporter gene ADY2 improved tolerance of Saccharomyces cerevisiae against multiple stresses and enhanced ethanol production in the presence of acetic acid[J]. Bioresource Technology, 2017, 245: 1461-1468. DOI:10.1016/j.biortech.2017.05.191 |
| [30] |
Swinnen S, Henriques SF, Shrestha R, et al. Improvement of yeast tolerance to acetic acid through Haa1 transcription factor engineering: towards the underlying mechanisms[J]. Microbial Cell Factories, 2017, 16: 7. DOI:10.1186/s12934-016-0621-5 |
| [31] |
Mira NP, Becker JD, Sá-Correia I. Genomic expression program involving the Haa1p-regulon in Saccharomyces cerevisiae response to acetic acid[J]. OMICS: A Journal of Integrative Biology, 2010, 14(5): 587-601. DOI:10.1089/omi.2010.0048 |
| [32] |
Takabatake A, Kawazoe N, Izawa S. Plasma membrane proteins Yro2 and Mrh1 are required for acetic acid tolerance in Saccharomyces cerevisiae[J]. Applied Microbiology and Biotechnology, 2015, 99(6): 2805-2814. DOI:10.1007/s00253-014-6278-2 |
| [33] |
Guerreiro JF, Mira NP, Santos A, et al. Membrane phosphoproteomics of yeast early response to acetic acid: role of Hrk1 kinase and lipid biosynthetic pathways, in particular sphingolipids[J]. Frontiers in Microbiology, 2017, 8: 1302. DOI:10.3389/fmicb.2017.01302 |
| [34] |
Chen YY, Stabryla L, Wei N. Improved acetic acid resistance in Saccharomyces cerevisiae by overexpression of the WHI2 gene identified through inverse metabolic engineering[J]. Applied and Environmental Microbiology, 2016, 82(7): 2156-2166. DOI:10.1128/AEM.03718-15 |
| [35] |
Li QQ, Wang Y, Zhuge B, et al. Effect of overexpressing transcription factors of Candida glycerinogenes on acid tolerance of Saccharomyces cerevisiae[J]. Chinese Journal of Applied and Environmental Biology, 2017, 23(6): 1006-1010. (in Chinese) 李倩倩, 王燕, 诸葛斌, 等. 产甘油假丝酵母抗逆转录因子的过表达对酿酒酵母耐酸胁迫性的影响[J]. 应用与环境生物学报, 2017, 23(6): 1006-1010. |
| [36] |
Unrean P. Flux control-based design of furfural-resistance strains of Saccharomyces cerevisiae for lignocellulosic biorefinery[J]. Bioprocess and Biosystems Engineering, 2017, 40(4): 611-623. DOI:10.1007/s00449-016-1725-3 |
| [37] |
Ishida Y, Nguyen TTM, Izawa S. The yeast ADH7 promoter enables gene expression under pronounced translation repression caused by the combined stress of vanillin, furfural, and 5-hydroxymethylfurfural[J]. Journal of Biotechnology, 2017, 252: 65-72. DOI:10.1016/j.jbiotec.2017.04.024 |
| [38] |
Moon J, Liu ZL. Engineered NADH-dependent GRE2 from Saccharomyces cerevisiae by directed enzyme evolution enhances HMF reduction using additional cofactor NADPH[J]. Enzyme and Microbial Technology, 2012, 50(2): 115-120. DOI:10.1016/j.enzmictec.2011.10.007 |
| [39] |
Wang HY, Ouyang YD, Zhou C, et al. YKL071W from Saccharomyces cerevisiae encodes a novel aldehyde reductase for detoxification of glycolaldehyde and furfural derived from lignocellulose[J]. Applied Microbiology and Biotechnology, 2017, 101(23/24): 8405-8418. |
| [40] |
Zhao XX, Tang J, Wang X, et al. YNL134C from Saccharomyces cerevisiae encodes a novel protein with aldehyde reductase activity for detoxification of furfural derived from lignocellulosic biomass[J]. Yeast, 2015, 32(5): 409-422. DOI:10.1002/yea.3068 |
| [41] |
Wang HY, Xiao DF, Zhou C, et al. YLL056C from Saccharomyces cerevisiae encodes a novel protein with aldehyde reductase activity[J]. Applied Microbiology and Biotechnology, 2017, 101(11): 4507-4520. DOI:10.1007/s00253-017-8209-5 |
| [42] |
Kim D, Hahn JS. Roles of the Yap1 transcription factor and antioxidants in Saccharomyces cerevisiae's tolerance to furfural and 5-hydroxymethylfurfural, which function as thiol-reactive electrophiles generating oxidative stress[J]. Applied Microbiology and Biotechnology, 2013, 79(16): 5069-5077. |
| [43] |
Kitichantaropas Y, Boonchird C, Sugiyama M, et al. Cellular mechanisms contributing to multiple stress tolerance in Saccharomyces cerevisiae strains with potential use in high-temperature ethanol fermentation[J]. AMB Express, 2016, 6: 107. DOI:10.1186/s13568-016-0285-x |
| [44] |
An B, Li BQ, Qin GZ, et al. Exogenous calcium improves viability of biocontrol yeasts under heat stress by reducing ROS accumulation and oxidative damage of cellular protein[J]. Current Microbiology, 2012, 65(2): 122-127. DOI:10.1007/s00284-012-0133-4 |
| [45] |
Dong YC, Hu JJ, Fan LL, et al. RNA-Seq-based transcriptomic and metabolomic analysis reveal stress responses and programmed cell death induced by acetic acid in Saccharomyces cerevisiae[J]. Scientific Reports, 2017, 7: 42659. DOI:10.1038/srep42659 |
| [46] |
Stratford M, Nebe-von-Caron G, Steels H, et al. Weak-acid preservatives: pH and proton movements in the yeast Saccharomyces cerevisiae[J]. International Journal of Food Microbiology, 2013, 161(3): 164-171. DOI:10.1016/j.ijfoodmicro.2012.12.013 |
| [47] |
Zhang XH, Zhang Y, Wang Q, et al. Research and development on the microbial furfural-tolerance mechanism[J]. Advances in New and Renewable Enengy, 2017, 5(3): 189-196. (in Chinese) 张小欢, 张宇, 王琼, 等. 微生物耐受糠醛机制的研究进展[J]. 新能源进展, 2017, 5(3): 189-196. DOI:10.3969/j.issn.2095-560X.2017.03.005 |
| [48] |
Li X, Yang RH, Ma MG, et al. A novel aldehyde reductase encoded by YML131W from Saccharomyces cerevisiae confers tolerance to furfural derived from lignocellulosic biomass conversion[J]. Bioenergy Research, 2015, 8(1): 119-129. DOI:10.1007/s12155-014-9506-9 |
| [49] |
Wu GC, Xu ZX, Jönsson LJ. Profiling of Saccharomyces cerevisiae transcription factors for engineering the resistance of yeast to lignocellulose-derived inhibitors in biomass conversion[J]. Microbial Cell Factories, 2017, 16: 199. DOI:10.1186/s12934-017-0811-9 |
| [50] |
Jung YH, Kim S, Yang JW, et al. Intracellular metabolite profiling of Saccharomyces cerevisiae evolved under furfural[J]. Microbial Biotechnology, 2017, 10(2): 395-404. DOI:10.1111/1751-7915.12465 |
 2019, Vol. 46
2019, Vol. 46