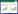扩展功能
文章信息
- 王亚鸽, 闫鹤
- WANG Ya-Ge, YAN He
- 一株ST477型单增李斯特菌的全基因组测序及inlA基因遗传多样性
- Whole-genome sequencing of a Listeria monocytogenes ST477 and genetic diversity of inlA gene
- 微生物学通报, 2019, 46(5): 1100-1107
- Microbiology China, 2019, 46(5): 1100-1107
- DOI: 10.13344/j.microbiol.china.180881
-
文章历史
- 收稿日期: 2018-11-07
- 接受日期: 2019-03-07
- 网络首发日期: 2019-03-13
单增李斯特菌(Listeria monocytogenes)是一类重要的食源性条件致病菌,可引起人和动物李斯特菌病,主要感染免疫力低下(老年人、孕妇及新生儿)和免疫缺陷人群,临床症状包括败血症、脑膜炎、流产和死胎等[1]。近几年,通过食物传播引起的李斯特菌病的暴发被频繁报道[2-3],中国虽然关于李斯特菌病的报道较少[4-5],但多位学者从各地多种食品中均分离出单增李斯特菌[6-8],其风险不容忽视。
多位点序列分型(Mutilocus sequence typing,MLST)方法是一种基于7个管家基因(abcZ,bglA,cat,dapE,dat,ldh,lhkA)的核苷酸序列确定菌株之间的高度差异性,进而揭示流行病学及分子进化的分析方法[9]。细菌在长期进化过程中基因组信息存在差异,随着全基因组测序水平的发展,单核苷酸多态性(Single nucleotide polymorphism,SNP)已经成为确定细菌关联性的分析方法[10]。单增李斯特菌的一些毒素及相关基因对于细菌的侵染及宿主细胞内的生存具有重要作用。其中表面蛋白内化素可促进该菌入侵宿主上皮细胞,基因inlA编码的内化素A (InlA)通过与上皮细胞的钙粘蛋白受体相互作用,调节菌体被上皮细胞摄入的过程。研究表明,基因inlA的有效突变会导致翻译提前终止(Premature stop codon,PMSC),进而降低细菌入侵宿主上皮细胞的能力[11]。
复合克隆系(Clonal complex,CC) 9是在中国流行度最高的三大克隆系之一[6, 12-13]。但目前对于单增李斯特菌CC9克隆系的进化关系及基因特点少有报道,更无针对序列型(Sequence type,ST)为477菌株的研究。本研究以分离自中国食品的ST477型别的单增李斯特菌为研究对象,进行比较基因组学分析并研究基因inlA的遗传多样性,旨在为单增李斯特菌的传播及毒力检测提供理论依据。
1 材料与方法 1.1 菌株单增李斯特菌NH1分离自河北省某大型超市的冷冻食品[14]。
1.2 主要试剂和仪器脑心浸出液肉汤(Brain heart infusion broth,BHI),广州环凯微生物科技有限公司;细菌基因组DNA快速提取试剂盒,北京博迈德生物技术有限公司。Pacbio RS Ⅱ测序仪,Pacific Biosciences公司。
1.3 基因组测序与注释将菌株NH1划线接种至BHI琼脂培养基,37 ℃培养过夜,再挑取单菌落至BHI中,37 ℃培养12–16 h。DNA提取按照细菌基因组DNA快速提取试剂盒说明书进行。将得到的总DNA通过Pacbio RS Ⅱ测序仪进行全基因组测序,得到937 153条测序片段,利用HGAP (Hierarchical genome assembly process)进行拼接组装,将完整的基因组序列提交至NCBI GenBank数据库,获得该菌株GenBank登录号为CP021325,并通过PGAP (Prokaryotic genome annotation pipeline)进行基因预测与功能注释。采用Glimmer 3.02软件进行开放阅读框(Open reading frame,ORF)预测,将所有预测蛋白序列与非冗余蛋白数据库NR、Swiss-Prot数据库、eggNOG (Evolutionary genealogy of genes:Non-supervised Orthologous Groups)数据库进行BLASTp (E < 1e−10)比对,完成蛋白序列功能注释。
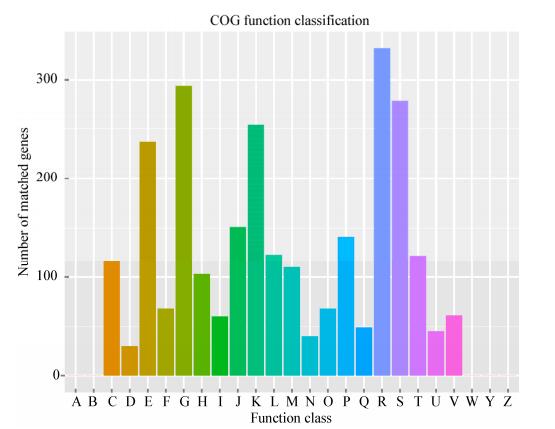
1.4 MLST进化分析PasteurMLST数据库中已登记的ST477型别单增李斯特菌有2株,分别为SLCC1798和Lm_0080。SLCC1798于1964年分离自菲律宾病人,Lm_0080于1988年分离自日本病人。将单增李斯特菌NH1的管家基因序列与8株CC9菌株的管家基因序列进行MLST分析,包括2株ST477型别的SLCC1798和Lm_0080,5株ST9型别的10-5025 (NZ_CP007194)、10-5026 (NZ_CP007195)、10-5027 (NZ_CP007196)、SHL006 (NZ_APIF00000000)、SLCC2479 (NC_018589)及1株ST122型别的SLCC2372 (NC_018588)。根据PasteurMLST数据库(http://bigsdb.pasteur.fr/listeria/listeria.html),确定7个管家基因(abcZ,bglA,cat,dapE,dat,ldh,lhkA)的等位基因号。归纳9株菌的7个等位基因的代号和ST型别,根据每株菌的7个管家基因的序列,按照abcZ-bglA-cat-dapE-dat-ldh-lhkA的顺序拼接成9条序列,利用软件MEGA 5.0中的Neighbor-Joining法构建管家基因的系统发育树(1 000 Bootstrap),分析9株菌的进化关系。
1.5 SNP分析PasteurMLST数据库中登记的2株ST477型别菌株无全基因组序列,因此,对其余7株CC9克隆系菌株进行全基因组水平上的差异分析。以NH1为参考菌株,通过软件GATK和SAMtools确定每个菌株的SNPs。用VarScan软件进行SNP信息的检测,并过滤掉测序深度和比对质量值较低的位点。将每个基因组的SNPs拼接为一个序列,利用软件MEGA 5.0构建进化树(1 000 Bootstrap)。
1.6 内化素基因序列比对分析PasteurMLST数据库中无2株ST477型别菌株的inlA基因序列,因此,通过PasteurMLST数据库提取出其他7个CC9克隆系菌株基因组中10个内化素基因的核苷酸序列,以NH1为参考菌株,进行基因同源性比较。进一步从每个分离株的inlA基因序列中获得翻译的蛋白序列,确定该基因PMSC的突变类型[11],并根据inlA基因的核苷酸序列,利用软件MEGA 5.0构建进化树(1 000 Bootstrap)。
2 结果与分析 2.1 NH1基因组的基本特征单增李斯特菌NH1的基因组全长为3 002 491 bp,(G+C)mol%含量为37.92%,共有2 995个编码基因。将其编码的对应蛋白序列与eggNOG数据库比对,完成同源基因注释分类(图 1)。结果表明,共有2 370个蛋白获得COG功能注释,占全部基因的79.13%,分布于20个COG的条目中。这些基因大多与细菌的代谢有关。其中参与碳水化合物转运和代谢的基因有294个,占比9.82%;与转录相关的基因有254个,占比8.48%;参与氨基酸转运和代谢的基因有237个,占比7.91%。噬菌体作为微生物基因重组及获得外源基因的重要因素之一,研究表明单增李斯特菌的噬菌体相关基因可调节宿主的毒性,而且可协助细菌在细胞内生存[15-17]。NH1基因组中检测到13个与噬菌体相关的基因,分布于转录、一般功能预测、未知功能及信号转导机制,即K、R、S及T四个分类单元。
|
| 图 1 单增李斯特菌NH1蛋白质eggNOG聚类分析 Figure 1 Gene distribution based on eggNOG classification of L. monocytogenes NH1 注:A:RNA加工和修饰;B:染色质结构和动力学;C:能量生产和转换;D:细胞周期调控、细胞分裂和染色体分配;E:氨基酸转运和代谢;F:核苷酸转运和代谢;G:碳水化合物转运和代谢;H:辅酶转运和代谢;I:脂质转运和代谢;J:翻译、核糖体结构和生物合成;K:转录;L:复制、重组和修复;M:细胞壁/细胞膜/胞外被膜生物合成;N:细胞运动;O:翻译后修饰、蛋白质转换、伴侣;P:机离子转运和代谢;Q:次级代谢产物的生物合成、转运和代谢;R:一般功能预测;S:功能未知;T:信号转导机制;U:胞内运输、分泌和囊泡运输;V:防御机制;W:胞外结构;Y:细胞核结构;Z:细胞骨架. Note: A: Processing and modification; B: Chromatin structure and dynamics; C: Energy production and conversion; D: Cell cycle control, cell division, chromosome partitioning; E: Amino acid transport and metabolism; F: Nucleotide transport and metabolism; G: Carbohydrate transport and metabolism; H: Coenzyme transport and metabolism; I: Lipid transport and metabolism; J: Translation, ribosomal structure and biogenesis; K: Transcription; L: Replication, recombination and repair; M: Cell wall/membrane/envelope biogensi; N: Cell motility; O: Posttranslational modification, protein turnover, chaperones; P: Inorganic ion transport and metabolism; Q: Secondary netabolites biosynthesis, transport and catabolism; R: General function prediction only; S: Function unknown; T: Signal transduction mechanisms; U: Intracellular trafficking, secretion, and vesicular transport; V: Defense mechanisms; W: Extracellular structures; Y: Nuclear structure; Z: Cytoskeleton. |
|
|
根据PasteurMLST数据库比对7个管家基因,确定单增李斯特菌NH1的分型为ST477 (abcZ-6,bglA-5,cat-6,dapE-4,dat-1,ldh-183,lhkA-1),属于CC9克隆复合体。该克隆系菌株在全球范围内广泛分布,且在食品中的分离率较高[18]。研究表明,分离自中国的CC9克隆系的代表ST型为ST9[12-13],而对于ST477型别菌株少有报道。因此,本研究对我们已完成测序的ST477型别菌株与2株分离自临床的ST477型别,以及5株ST9型别和1株ST122型单增李斯特菌进行MLST进化分析(图 2)。进化分析结果表明,3种不同STs型别菌株分为4个进化谱系。其中,3株ST477型别菌株,1个食品菌株,2个临床菌株位于同一个分支;4株ST9型别菌株,包括3个食品菌株,1个临床菌株位于同一个分支;1株未知来源的ST9和临床ST122型别菌株分别单独为一个进化谱系。同时,分离自不同国家的菌株位于同一个分支,存在较近的亲缘关系。

|
| 图 2 单增李斯特菌MLST聚类分析图 Figure 2 MLST genetic profile of L. monocytogenes isolates 注:括号内为GenBank登录号;分支点上的数字表示构建系统树时1 000次计算形成该节点的百分比;标尺长度代表进化距离. Note: Numbers in parentheses are GenBank accession numbers; The bootstrap values (%) presented at the branches were calculated from 1 000 replications; The scale bar represents the evolutionary distance. |
|
|
Chenal-Francisque等[18]研究表明单增李斯特菌大部分复合克隆系菌株存在全球分布的情况,而CC9克隆系菌株在欧洲和西半球的流行率较高。Angelo等[2]通过PFGE和wgMLST对2014–2015年美国暴发的李斯特菌病的相关单增李斯特菌进行分析,结果表明本次暴发的污染菌株是以苹果为介质进行全国范围的传播。刘二龙等[19]对我国出入境口岸动物源性食品中分离的单增李斯特菌进行MLST分析,结果表明存在潜在传播和暴发的李斯特菌病分子亚型。而且,越来越多研究表明,随着食品贸易的国际化,单增李斯特菌可通过贸易食品进行洲际间传播[20-21]。
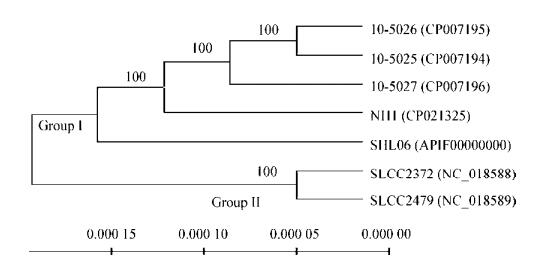
2.3 SNP分析以NH1为参考菌株,对5株ST9型别和1株ST122型别菌株进行全基因组水平的SNP分析,共发现291个非同义突变位点。其中,3株来自加拿大的食品菌株10-5026、10-5025和10-5027中检测到的SNPs数量较少(分别为91、93和93个),来自中国和英国的临床菌株(分别为SHL006和SLCC2372)的SNPs为113个和127个,而未知来源菌株SLCC2479含有最多SNPs (136个)。为直观展示物种进化地位,基于SNP系统应用软件MEGA 5.0构建进化树,结果见图 3。4株分离自不同国家食品的菌株聚为一个分支,1株分离自中国临床的菌株单独为一个分支。与MLST聚类结果不同,ST477型别菌株与4个ST9型菌株聚为一大类群(Group Ⅰ),一株ST9型与ST122型菌株聚为另一大类群(Group Ⅱ)。
|
| 图 3 基于7株单增李斯特菌全基因组SNPs进化分析 Figure 3 Phylogenetic analysis of 7 L. monocytogenes whole genome SNPs 注:括号内为GenBank登录号;分支点上的数字表示构建系统树时1 000次计算形成该节点的百分比;标尺长度代表进化距离. Note: Numbers in parentheses are GenBank accession numbers; The bootstrap values (%) presented at the branches were calculated from 1 000 replications; The scale bar represents the evolutionary distance. |
|
|
虽然目前没有明确的SNP标准规定菌株进化关系,但ST477菌株与3株来自加拿大食品菌株的SNP差异数最少,说明其亲缘关系较近。该结果与Zhang等[22]对分离自上海的单增李斯特菌基因组多样性进行研究的结果相似:来自中国的2株菌与来自加拿大的2株暴发菌株仅存在11–97个SNP差异,具有较近亲缘关系;进一步分析表明,这些菌株拥有共同的祖先,在不同环境下进化成为不同的分支。由图 3可知,SNP聚类结果与型别及地域无显著关系,但与宿主有一定联系。该结果与Halbedel等[23]对分离自德国病人的413株单增李斯特菌的SNP进化分析结果相似。同时,Toledo等[24]对分离自智利不同来源的菌株进行SNP进化分析的结果表明,临床和非临床菌株的分布具有显著差异。
2.4 内化素基因序列比对分析以NH1为参考菌株,对6株CC9克隆系菌株的10个内化素基因进行同源性比较。结果表明,除基因inlA外,其余9个内化素基因inlB、inlC、inlE、inlF、inlG、inlH、inlJ、inlK及inlP均在核苷酸水平上高度保守,相似度达100%。来自临床的菌株SHL006和SLCC2372及一株未知来源的菌株SLCC2479的inlA基因存在差异,相似度为99%。内化素基因inlA的突变已被广泛报道,且有效突变会致使该基因翻译提前终止,进而导致细菌毒力减弱[12]。根据突变位点确定该基因PMSC的突变类型。目前已知的基因inlA的突变有19种类型[25-26],深入研究表明,与临床和动物来源菌株相比,PMSC突变情况在谱系Ⅱ和食品及其环境的分离株中存在的概率显著较高[11, 26-27]。
提取7株CC9型单增李斯特菌的inlA基因序列,长度约为2 400 bp,(G+C)mol%含量为37.4%。其中,ST122型菌株(SLCC2372)中基因inlA的序列完整,3株来自加拿大的ST9型菌株与ST477菌株中基因inlA突变类型相同(PMSC type 4),另外2株ST9型菌株(SHL006和SLCC2479)中基因inlA突变类型相同(PMSC type 7) (表 1)。Manuel等[27]对191株不同来源(动物、食品病人及相关环境)单增李斯特菌的inlA基因序列进行分析,结果表明,不同来源菌株缩短序列的类型存在3种情况(PMSC type 4、PMSC type 7和PMSC type 12),其中90%为PMSC type 4。本研究结果表明,突变类型PMSC type 4和PMSC type 7的基因序列与完整序列的相似度分别为29.8%和77.3%;PMSC type 4与PMSC type 7的序列长度相同但相似度为52.6%。
| 菌株 Strain |
来源 Source |
长度 Length (bp) |
类型 Subtype |
相似度 Identity (%) |
||||||
| NH1 | 10-5025 | 10-5026 | 10-5027 | SHL006 | SLCC2479 | SLCC2372 | ||||
| NH1 | Frozen food | 2 402 | 4 | 100.0 | ||||||
| 10-5025 | Bacon flake | 2 402 | 4 | 100.0 | 100.0 | |||||
| 10-5026 | Food processing | 2 402 | 4 | 100.0 | 100.0 | 100.0 | ||||
| 10-5027 | Food processing | 2 402 | 4 | 100.0 | 100.0 | 100.0 | 100.0 | |||
| SHL006 | Human | 2 402 | 7 | 52.6 | 52.6 | 52.6 | 52.6 | 100.0 | ||
| SLCC2479 | N.A. | 2 402 | 7 | 52.6 | 52.6 | 52.6 | 52.6 | 100.0 | 100.0 | |
| SLCC2372 | Human | 2 403 | / | 29.8 | 29.8 | 29.8 | 29.8 | 77.3 | 77.3 | 100.0 |
| 注:/:inlA基因为全长. Note: /: Fult-length inlA. |
||||||||||
根据7株CC9克隆系菌株携带的inlA的核苷酸序列构建系统进化树,该进化树含有3个簇,同一突变类型的菌株序列聚在一个分支:具有完整inlA基因序列的ST122型菌株单独为一个分支;4株PMSC type 4型菌株聚为一个分支(ST477和ST9);2株PMSC type 7型菌株聚为一个分支(ST9)。图 4表明,PMSC type 4和PMSC type 7均由完整序列菌株进化而来,且PMSC type 7与原始菌株inlA基因的进化关系更近。该分析结果与SNP进化分析结果不同。Palma等[28]对56株ST14和ST121的单增李斯特菌进行分析,结果表明ST14型别菌株的inlA基因均为完整序列,所有的ST121均为缩短序列。Wu等[13]对分离自中国即食食品的35株存在突变的单增李斯特菌的inlA基因进行进化分析,结果表明,inlA进化结果与MLST进化结果一致。因此,推测inlA与菌株的管家基因有相似的基因特征。

|
| 图 4 基于7个单增李斯特菌的inlA序列的最大似然树 Figure 4 Maximum likelihood tree of 7 L. monocytogenes isolates based on inlA sequence 注:括号内为GenBank登录号;分支点上的数字表示构建系统树时1 000次计算形成该节点的百分比;标尺长度代表进化距离. Note: Numbers in parentheses are GenBank accession numbers; The bootstrap values (%) presented at the branches were calculated from 1 000 replications; The scale bar represents the evolutionary distance. |
|
|
本研究在基因组层面揭示了单增李斯特菌CC9克隆系不同ST型别菌株的关系,同时分析了内化素基因inlA的遗传多样性。MLST进化分析表明,分离自中国食品的ST477型别单增李斯特菌与2株来自菲律宾和日本临床的ST477型别菌株具有较近亲缘关系。通过SNP分析发现,分离自中国食品的ST477型别菌株与3株来自加拿大食品的ST9型别菌株的SNP差异数最少,而且具有较近亲缘关系。基因inlA的核苷酸序列分析结果表明,突变类型PMSC type 4和PMSC type 7的基因序列与完整序列的相似度分别为29.8%和77.3%;PMSC type 4与PMSC type 7的序列长度相同但相似度为52.6%。进化分析表明,PMSC type 4和PMSC type 7均由完整序列菌株进化而来,且PMSC type 7与原始菌株inlA基因的进化关系更近。本研究在食源性单增李斯特菌的毒力及进化研究中具有一定的意义。
| [1] |
Shetty A, McLauchlin J, Grant K, et al. Outbreak of Listeria monocytogenes in an oncology unit associated with sandwiches consumed in hospital[J]. Journal of Hospital Infection, 2009, 72(4): 332-336. DOI:10.1016/j.jhin.2009.01.012 |
| [2] |
Angelo KM, Conrad AR, Saupe A, et al. Multistate outbreak of Listeria monocytogenes infections linked to whole apples used in commercially produced, prepackaged caramel apples: United States, 2014-2015[J]. Epidemiology & Infection, 2017, 145(5): 848-856. |
| [3] |
Jackson BR, Tarr C, Strain E, et al. Implementation of nationwide real-time whole-genome sequencing to enhance listeriosis outbreak detection and investigation[J]. Clinical Infectious Diseases, 2016, 63(3): 380-386. DOI:10.1093/cid/ciw242 |
| [4] |
Jiao Y, Zhang W, Ma JR, et al. Early onset of neonatal listeriosis[J]. Pediatrics International, 2011, 53(6): 1034-1037. DOI:10.1111/ped.2011.53.issue-6 |
| [5] |
Wang H, Wang Y, Zhang ZD, et al. Clinical and epidemiological characteristics of three cases of Listeria monocytogenes infection[J]. Chinese Journal of Zoonoses, 2015, 31(7): 678-680. (in Chinese) 王红, 王艳, 张正东, 等. 3例单增李斯特菌感染的病原学、临床及流行病学特征分析[J]. 中国人兽共患病学报, 2015, 31(7): 678-680. DOI:10.3969/cjz.j.issn.1002-2694.2015.07.018 |
| [6] |
Wu S, Wu QP, Zhang JM, et al. Listeria monocytogenes prevalence and characteristics in retail raw foods in China[J]. PLoS One, 2015, 10(8): e0136682. DOI:10.1371/journal.pone.0136682 |
| [7] |
Ma AJ, Wang Y, Wang Y, et al. Molecular epidemiological characteristics of Listeria monocytogenes isolated from raw meat samples in some regions of Beijing, China[J]. Chinese Journal of Zoonoses, 2015, 31(5): 403-407. (in Chinese) 马爱静, 王艳, 王毅, 等. 北京市一些地区生肉标本中单增李斯特菌的分离及其分子流行病学特征分析[J]. 中国人兽共患病学报, 2015, 31(5): 403-407. DOI:10.3969/cjz.j.issn.1002-2694.2015.05.003 |
| [8] |
Li ZY, Jia JT, Zeng J, et al. Identification of Listeria monocytogenes from imported Trachurus japonicas and prokaryotic expression of flagellin[J]. Microbiology China, 2014, 41(6): 1160-1167. (in Chinese) 李正义, 贾俊涛, 曾静, 等. 进口竹荚鱼中单核细胞增生李斯特菌的鉴定及其鞭毛蛋白的原核表达[J]. 微生物学通报, 2014, 41(6): 1160-1167. |
| [9] |
Ragon M, Wirth T, Hollandt F, et al. A new perspective on Listeria monocytogenes evolution[J]. PLoS Pathogen, 2008, 4(9): e1000146. DOI:10.1371/journal.ppat.1000146 |
| [10] |
Ward TJ, Ducey TF, Usgaard T, et al. Multilocus genotyping assays for single nucleotide polymorphism-based subtyping of Listeria monocytogenes isolates[J]. Applied and Environmental Microbiology, 2008, 74(24): 7629-7642. DOI:10.1128/AEM.01127-08 |
| [11] |
Nightingale KK, Windham K, Martin KE, et al. Select Listeria monocytogenes subtypes commonly found in foods carry distinct nonsense mutations in inlA, leading to expression of truncated and secreted internalin A, and are associated with a reduced invasion phenotype for human intestinal epithelial cells[J]. Applied and Environmental Microbiology, 2005, 71(12): 8764-8772. DOI:10.1128/AEM.71.12.8764-8772.2005 |
| [12] |
Wang Y, Zhao AL, Zhu RF, et al. Genetic diversity and molecular typing of Listeria monocytogenes in China[J]. BMC Microbiology, 2012, 12: 119. DOI:10.1186/1471-2180-12-119 |
| [13] |
Wu S, Wu QP, Zhang JM, et al. Analysis of multilocus sequence typing and virulence characterization of Listeria monocytogenes isolates from Chinese retail ready-to-eat food[J]. Frontiers in Microbiology, 2016, 7: 168. |
| [14] |
Yan H, Neogi SB, Mo ZY, et al. Prevalence and characterization of antimicrobial resistance of foodborne Listeria monocytogenes isolates in Hebei province of Northern China, 2005-2007[J]. International Journal of Food Microbiology, 2010, 144(2): 310-316. DOI:10.1016/j.ijfoodmicro.2010.10.015 |
| [15] |
Rabinovich L, Sigal N, Borovok I, et al. Prophage excision activates Listeria competence genes that promote phagosomal escape and virulence[J]. Cell, 2012, 150(4): 792-802. DOI:10.1016/j.cell.2012.06.036 |
| [16] |
Wang X, Li J, Zhang ZH, et al. Comparative genomic analysis of one Listeria monocytogenes containing five prophages[J]. Genomics and Applied Biology, 2017, 36(2): 670-685. (in Chinese) 王旭, 李骏, 张昭寰, 等. 一株含有5个原噬菌体的单增李斯特菌比较基因组分析[J]. 基因组学与应用生物学, 2017, 36(2): 670-685. |
| [17] |
Kuenne C, Billion A, Mraheil MA, et al. Reassessment of the Listeria monocytogenes pan-genome reveals dynamic integration hotspots and mobile genetic elements as major components of the accessory genome[J]. BMC Genomics, 2013, 14: 47. DOI:10.1186/1471-2164-14-47 |
| [18] |
Chenal-Francisque V, Lopez J, Cantinelli T, et al. Worldwide distribution of major clones of Listeria monocytogenes[J]. Emerging Infectious Diseases, 2011, 17(6): 1110-1112. DOI:10.3201/eid/1706.101778 |
| [19] |
Liu EL, Yuan MY, Deng JY, et al. Multilocus sequence typing of Listeria monocytogenes isolated from imported and exported animal-derived foods[J]. Food Science, 2016, 37(12): 212-216. (in Chinese) 刘二龙, 袁慕云, 邓建英, 等. 出入境动物源性食品中单增李斯特菌的多位点序列分型分析[J]. 食品科学, 2016, 37(12): 212-216. DOI:10.7506/spkx1002-6630-201612038 |
| [20] |
Moura A, Criscuolo A, Pouseele H, et al. Whole genome-based population biology and epidemiological surveillance of Listeria monocytogenes[J]. Nature Microbiology, 2016, 2(2): 16185. |
| [21] |
Fretz R, Pichler J, Sagel U, et al. Update: multinational listeriosis outbreak due to 'Quargel', a sour milk curd cheese, caused by two different L. monocytogenes serotype 1/2a strains, 2009-2010[J]. EuroSurveillance, 2010, 15(16): 19543. |
| [22] |
Zhang JM, Cao GJ, Xu XB, et al. Evolution and diversity of Listeria monocytogenes from clinical and food samples in Shanghai, China[J]. Frontiers in Microbiology, 2016, 7: 1138. |
| [23] |
Halbedel S, Prager R, Fuchs S, et al. Whole-genome sequencing of recent Listeria monocytogenes isolates from Germany reveals population structure and disease clusters[J]. Journal of Clinical Microbiology, 2018, 56(6): e00119-18. |
| [24] |
Toledo V, den Bakker HC, Hormazábal JC, et al. Genomic diversity of Listeria monocytogenes isolated from clinical and non-clinical samples in Chile[J]. Genes, 2018, 9(8): 396. DOI:10.3390/genes9080396 |
| [25] |
Gelbíčová T, Koláčková I, Pantůček R, et al. A novel mutation leading to a premature stop codon in inlA of Listeria monocytogenes isolated from neonatal listeriosis[J]. New Microbiologica, 2015, 38(2): 293-296. |
| [26] |
van Stelten A, Simpson JM, Ward TJ, et al. Revelation by single-nucleotide polymorphism genotyping that mutations leading to a premature stop codon in inlA are common among Listeria monocytogenes isolates from ready-to-eat foods but not human listeriosis cases[J]. Applied and Environmental Microbiology, 2010, 76(9): 2783-2790. DOI:10.1128/AEM.02651-09 |
| [27] |
Manuel CS, van Stelten A, Wiedmann M, et al. Prevalence and distribution of Listeria monocytogenes inlA alleles prone to phase variation and inlA alleles with premature stop codon mutations among human, food, animal, and environmental isolates[J]. Applied and Environmental Microbiology, 2015, 81(24): 8339-8345. DOI:10.1128/AEM.02752-15 |
| [28] |
Palma F, Pasquali F, Lucchi A, et al. Whole genome sequencing for typing and characterisation of Listeria monocytogenes isolated in a rabbit meat processing plant[J]. Italian Journal of Food Safety, 2017, 6(3): 6879. |
 2019, Vol. 46
2019, Vol. 46