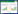扩展功能
文章信息
- 邓益琴, 陈偿, 苏友禄, 程长洪, 马红玲, 刘松林, 冯娟
- DENG Yi-Qin, CHEN Chang, SU You-Lu, CHENG Chang-Hong, MA Hong-Ling, LIU Song-Lin, FENG Juan
- 溶藻弧菌ZJ-T小RNA srvg23535基因突变株的构建及其功能初探
- Construction and characterization of the sRNA srvg23535 knock-out mutant of Vibrio alginolyticus ZJ-T
- 微生物学通报, 2019, 46(4): 829-841
- Microbiology China, 2019, 46(4): 829-841
- DOI: 10.13344/j.microbiol.china.180217
-
文章历史
- 收稿日期: 2018-03-20
- 接受日期: 2018-06-24
- 网络首发日期: 2018-07-30
2. 中国科学院南海海洋研究所 西沙、南沙深海海洋环境观测研究站 广东 广州 510301;
3. 中国科学院南海海洋研究所 热带海洋生物资源与生态重点实验室 广东 广州 510301
2. Xisha and Nansha Ocean Observation and Research Station, South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou, Guangdong 510301, China;
3. Key Laboratory of Tropical Marine Bio-resources and Ecology, South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou, Guangdong 510301, China
溶藻弧菌(Vibrio alginolyticus)是一种常见的兼性厌氧的革兰氏阴性嗜盐嗜温短杆菌,同时是一种机会致病菌,对海洋动物和人类造成潜在威胁[1-3]。在美国,溶藻弧菌引起的疾病发病率在1996–2010年间显著增加,与副溶血性弧菌和创伤弧菌一起被列为最常见的病原体之一[4]。此外,溶藻弧菌常引起人体严重腹泻、发热并脱水,甚至通过严重感染引起中耳炎及败血症,是人类重要的致病菌[5-6]。
溶藻弧菌广泛分布在河口与海洋,为了适应不断变化的环境,演化出多种环境适应机制,如菌落相变、生物膜、群感效应等[7-8]。这些机制都处于严格且精准的基因表达调控体系中。基因表达调控发生在蛋白表达的各个水平上,转录、转录后、翻译及翻译后修饰等过程都可以作为基因表达的调控节点[9-10],除了极少数组成型表达的基因外,绝大多数基因的表达都在其中一个或多个节点接收环境信号的控制,从而组成复杂的调控网络以应对不断变化的环境条件。
越来越多的研究表明一类位于基因间区,转录但不翻译的反式编码sRNAs (Trans-encoded sRNAs,sRNAs)通过调控多个靶标基因,从而形成响应环境的调节活性网络[11]。此类sRNA主要通过与其靶标mRNA不完全配对影响mRNA的稳定性,从而激活或者抑制靶mRNA的表达;还可通过模拟靶蛋白底物,修饰或抑制靶蛋白的生物学活性,例如CsrB sRNA通过隔离的方式调节蛋白活性[12];此外,也有少数sRNAs与转录因子等蛋白相互作用,从而抑制相关蛋白活性[13]。小RNA在真核生物基因表达调控中的作用已被广泛研究,但是直到2000年以后,原核生物小RNA及其功能才逐渐被发现,越来越多的研究表明小RNA对细菌遗传变异、生长繁殖、致病性等具有重要的调控作用,其在细菌环境适应和流行分布方面的重要性已经得到广泛认可[13-14]。然而,大多数研究在模式生物体中进行,如大肠杆菌(Escherichia coli)和沙门氏菌(Salmonella)。随着高通量测序技术的发展,科学家开始探索非模式生物体,特别是人类病原体,如金黄色葡萄球菌(Staphylococcus aureus)或幽门螺杆菌(Helicobacter pylori)中的调控RNA[15-16],但对于大多数原核生物小RNA调控功能和作用机制依然所知有限。
在弧菌科中,大多数研究集中在霍乱弧菌和哈氏弧菌。溶藻弧菌中关于小RNA的研究仅Huang等[17-18]发现了3个与粘附、趋化及运动相关的小RNA,从小RNA角度揭示溶藻弧菌致病性、环境适应及流行特点的研究仍未见报道。本实验室前期通过转录组预测、Northern Blot鉴定得到溶藻弧菌ZJ-T的小RNA srvg23535,深入分析发现srvg23535是一个新的小RNA,并且仅存在于弧菌中,序列相对保守(未发表数据),但是其功能未知。本研究通过同源重组技术构建小RNA srvg23535的缺失突变株,并对突变株环境适应相关的生物学特性进行研究。该研究为深入了解srvg23535的功能,并获取其调控靶标基因,进而阐明其与靶标基因的相互作用机制奠定基础,研究成果将揭示小RNA在溶藻弧菌,甚至海洋病原弧菌环境适应和疾病暴发中的作用,也为控制海洋弧菌病害的暴发提供新的思路和防治靶标。
1 材料与方法 1.1 材料 1.1.1 菌株及质粒溶藻弧菌野生株ZJ-T[19]保存于本实验室;自杀质粒pSW7848[20]、自杀质粒中间宿主大肠杆菌Π3813[21]以及接合作用供体宿主大肠杆菌GEB883[22]来源于Didier Mazel和Annik Jacq等;小RNA突变重组自杀质粒pSW7848-∆srvg23535及小RNA突变株ZJ-T-Δsrvg23535构建于本研究。详细信息见表 1。
| 菌株或质粒 Strain or plasmid |
相关特征 Relevant characteristics |
来源 Source |
| V. alginolyticus | ||
| ZJ-T | Apr (ampicillin resistant), translucent/smooth variant of wild strain ZJ-51[19]; isolated from diseased Epinephelus coioides off the Southern China coast | [19] |
| ZJ-T-∆srvg23535 | Apr; ZJ-T carrying an deletion of srvg23535 | This study |
| E. coli | ||
| Ⅱ3813 | laclQ, thi1, supE44, endA1, recA1, hsdR17, gyrA462, zei298::Tn10[Tc], ΔthyA::erm-pir116; suicide vector pSW7848’s intermediate host | [21] |
| GEB883 | WT E. coli K12 ΔdapA::erm pir RP4-2 ΔrecA gyrA462, zei298::Tn10; donor strain for conjugation | [22] |
| Plasmids | ||
| pSW7848 | Cmr; suicide vector with an R6K origin, requiring the Pir protein for its replication, and the ccdB toxin gene | [20] |
| pSW7848-∆srvg23535 | Cmr; pSW7848 containing the mutant allele of ∆srvg23535 | This study |
| 注:Cmr和Apr分别表示氯霉素和氨苄青霉素抗性. Note: Cmr and Apr indicate chloramphenicol and ampicillin resistance, respectively. |
||
高保真酶PrimeSTAR® Max DNA polymerase、限制性内切酶Dpn I购自宝日医生物技术(北京)有限公司;用于营养缺陷型大肠杆菌Ⅱ3813生长的2ʹ-Deoxythymidine (Thy)购自生工生物工程(上海)股份有限公司;用于营养缺陷型大肠杆菌GEB883生长的2, 6-Diaminopimelic acid (DAP)购自西格玛奥德里奇(上海)贸易有限公司公司;用于等温组装的试剂盒ClonExpress MultiS One Step Cloning Kit购自南京诺唯赞生物科技有限公司公司。用于PM (Phenotype MicroArray)板细菌代谢实验测定OD590的MultiDisk Ascent读板仪购自Thermo Fisher Scientific公司。
1.1.3 培养基LB培养基、TSB (Tryptic soy broth)培养基购自BD公司。LB培养基(g/L):胰蛋白胨10.0,酵母提取物5.0,NaCl 10.0。TSB培养基(g/L):胰酪蛋白胨17.0,大豆蛋白胨3.0,D-葡萄糖2.5,NaCl 5.0,K2HPO4 2.5。固体培养基含琼脂15.0。在LB培养基基础上将NaCl浓度提高至3%即配制得到LBS培养基。
1.2 srvg23535突变株的构建参考Deng等[23]的方法通过同源重组技术构建srvg23535缺失突变株,srvg23535的序列信息见图 1,所用引物见表 2,但在重组质粒的构建以及反向筛选过程略有修改。将线性化载体与上、下游同源臂片段通过等温组装得到重组自杀质粒pSW7848-∆srvg23535。接合作用之后,由于自杀质粒pSW7848携带氯霉素抗性,且不能在溶藻弧菌中复制,并在受阿拉伯糖诱导后毒性基因ccdB表达。因此,通过氯霉素以及阿拉伯糖先后两次筛选后,自杀质粒上的srvg23535上下游同源臂片段与溶藻弧菌基因组的srvg23535上下游同源臂片段先后发生两次同源交换,从而获得溶藻弧菌srvg23535缺失株,命名为ZJ-T-∆srvg23535。

|
| 图 1 srvg23535的序列信息 Figure 1 The sequence information of srvg23535 注:加粗、斜体部分为srvg23535的序列,下划线部分依次为上、下游同源臂扩增的引物对. Note: The italic and bold part is the sequence of srvg23535 and the underlined parts are the primer pairs for the amplification of two flanking fragments. |
|
|
| 引物 Primers |
序列 Sequence (5′→3′) |
大小 Size (bp) |
| pSW7848-F | GTCTGATTCGTTACCAATTATGACAAC | 3 310 |
| pSW7848-R | GAATTCGATATCAAGCTTATCGATAC | |
| srvg23535-UP-F | AAGCTTGATATCGAATTCGAGGCACCTGTGGAGGGA | 1 084 |
| srvg23535-UP-R | GAATTTTAGGTAAGAGTTAAGTCTGTTTTTTATCATCGAAAG | |
| srvg23535-DOWN_F | TTAACTCTTACCTAAAATTCCGCTAATTAAG | 1 105 |
| srvg23535-DOWN-R | TTGGTAACGAATCAGACCCATCACCTAACACGTCC | |
| Del-check-pSW7848-F | TCACTGTCCCTTATTCGCACC | 2 474 |
| Del-check-pSW7848-R | CTGCTTTTGAGCACTACCCG | |
| ∆srvg23535-check-F | GGCACTCCTATGCCTCAATCAAAG | 618 (ZJ-T)/463 |
| ∆srvg23535-check-R | CATGTTGCGTTGATGCGTTCATTAC | (ZJ-T-∆srvg23535) |
参考邓益琴等[24]的方法进行菌落表型测试:将单个克隆的溶藻弧菌野生株ZJ-T及小RNA突变株ZJ-T-Δsrvg23535分别接种至LBS液体培养基,30 ℃、200 r/min培养过夜(下同);调整培养液浓度至相同吸光度(OD600=1.0),取5 μL平行3次点样到LBS固体平板上,30 ℃静置培养24 h,观察菌落形态。实验至少重复3次。
1.4 0.3%琼脂LB平板上Swimming运动性测试参考邓益琴等[24]的方法进行运动性测试:同1.3培养过夜菌液,调整培养液浓度至相同吸光度(OD600=1.0),取5 μL平行3次点样到0.3%琼脂半固体LB平板上,30 ℃静置培养16 h,测量菌斑直径。实验至少重复3次。
1.5 1%脱脂奶粉LBS平板上的胞外蛋白酶分泌测试参考邓益琴等[24]的方法进行胞外蛋白酶分泌测试:同1.3培养过夜菌液,调整培养液浓度至相同吸光度(OD600=5.0),取5 μL平行3次点样到LBS添加1%脱脂奶粉平板上,30 ℃静置培养24 h,测量蛋白分解圈直径以及克隆直径。实验至少重复3次。
1.6 TSB平板上对H2O2、Cu2+抗性以及对铁离子的吸收测试参考邓益琴等[24-25]的方法进行对H2O2、Cu2+抗性以及对铁离子的吸收测试:同1.3培养过夜菌液,调整培养液浓度至相同吸光度(OD600=5.0),同时10倍比稀释各菌株菌液至最高稀释倍数为106,然后将倍比稀释菌液依次取5 μL平行3次点样到TSB平板,以及TSB添加0.001 5% H2O2或5 mmol/L CuSO4或120 µmol/L DIP平板上,30 ℃静置培养24 h,观察细菌生长情况。实验至少重复3次。
1.7 LBS平板上对不同抗生素的敏感性测试参考Deng等[23]的方法进行对不同抗生素的敏感性测试:同1.3培养过夜菌液,将200 μL过夜菌液与10 mL新鲜TSB液体培养基混匀后倒入TSB平板(12 cm,正方形),待平板被稀释菌液浸湿后将剩余菌液倒掉,平板晾干后将各抗生素纸片置于平板上,30 ℃静置培养24 h后观察并测量抑菌圈直径。重复测试3次,使用IBM SPSS Statistics 19软件进行Student’s t检验,P < 0.05作为统计学显着性的阈值。
1.8 不同培养条件下的菌株生长情况测试参考Deng等[23]的方法进行不同培养条件下的菌株生长情况测试:同1.3培养过夜菌液。(1)为测试细菌在富营养培养基中的生长:菌株用新鲜LBS培养液稀释至OD600为1.0后再分别稀释1 000倍培养。(2)为测试细菌在寡营养培养基中生长,12 000×g离心10 min收集过夜细菌,并用不含D-葡萄糖和(NH4)2SO4的M63培养基(M63-C-N)洗涤2次,然后重悬并稀释至OD600为0.1于以下修饰的M63:测试细菌对不同碳源的利用,则M63寡营养培养基中D-葡萄糖被替换成其他碳水化合物(D-葡萄糖,D-蔗糖,D-麦芽糖,D-葡萄糖,D-果糖,D-海藻糖)作为单一碳源;测试细菌以葡萄糖作为单一碳源,以不同氨基酸作为单一氮源的生长情况,则(NH4)2SO4被不同氨基酸代替(150 mmol/L L-丙氨酸,50 mmol/L L-苏氨酸,10 mmol/L L-天冬氨酸,10 mmol/L L-谷氨酸,50 mmol/L L-丝氨酸,50 mmol/L L-精氨酸,50 mmol/L L-组氨酸盐酸,10 mmol/L L-谷氨酰胺盐,20 mmol/L L-亮氨酸,20 mmol/L L-缬氨酸,20 mmol/L L-异亮氨酸以及3种支链氨基酸混合物各20 mmol/L)。然后将稀释好的菌液100 μL/孔吸取至96孔板中,30 ℃、200 r/min连续振荡温育。使用MultiDisk Ascent读板仪相同时间间隔测定OD600,指示细菌的代谢活性。重复实验3次,使用IBM SPSS Statistics 19软件进行单因素协方差分析(ANCOVA) (以野生株和突变株为固定因子,以时间为协变量,以OD600值为因变量),并对显著差异的因子各时间点进行Student’s t检验,P < 0.05作为统计学显着性的阈值。
1.9 PM板测试细菌代谢参考Bochner等[26]的方法,利用PM02和PM08板分别测试细菌对95种不同碳源以及95种不同多肽氮源的利用。细菌在LBS固体平板上划线复活,30 ℃静置培养过夜。首先,510 μL的染料A与7 990 μL含14% NaCl的水溶液一起加入1.2×IF-0得到51 mL的1.0×IF-0;其次,对于每个菌株,用灭菌棉签刮取固体平板上的克隆,重悬在25 mL的1.0×IF-0中,得到浊度为85%的孵育液;然后,对于PM02板,每孔吸取浊度为85%的孵育液100 μL,对于PM08板,向浊度为85%的孵育液中加入适当体积的2 mol/L的琥珀酸钠,使得最终浓度为20 mmol/L,并每孔吸取100 μL孵育液;最后,将准备好的孵育板于30 ℃静置培养5 d,并在不同的时间点测定OD590,指示细菌的代谢活性。重复实验3次,使用IBM SPSS Statistics 19软件进行单因素协方差分析(ANCOVA) (以野生株和突变株为固定因子,以时间为协变量,以OD590值为因变量),并对显著差异的因子各时间点进行Student’s t检验,P < 0.05作为统计学显著性的阈值。
2 结果与分析 2.1 突变株的构建扩增得到的质粒线性化片段以及上游同源臂片段,电泳结果与预期大小相符(图 2A、B);等温组装后,重组自杀质粒PCR扩增检测电泳结果与预期大小相符(图 2C),证实重组自杀质粒pSW7848-∆srvg23535构建成功。接合作用后经过两次同源交换,可能使溶藻弧菌染色体上的srvg23535缺失,经PCR检测,溶藻弧菌野生株可扩增出大小为618 bp的片段,而srvg23535缺失株则扩增出大小为463 bp的片段(图 2D),从而获得溶藻弧菌srvg23535缺失株。图 2所有扩增结果与预期大小相符,测序结果也与预期序列一致,说明构建得到srvg23535缺失株。
|
| 图 2 pSW7848 PCR线性化片段(A)、srvg23535上下游同源臂片段(B)、重组自杀质粒的PCR鉴定扩增片段(C)以及srvg23535缺失株的PCR鉴定(D) Figure 2 Linearized segment of pSW7848 (A), upstream and downsteam homologous segments of srvg23535 (B), the segment of identification of the recombinant suicide vector by PCR (C) and identification of srvg23535 deletion mutant by PCR (D) 注:M1、M3:DL10000 DNA marker;M2、M4:DL2000 DNA marker;1:pSW7848线性化片段;2/3:srvg23535上/下游片段;4:重组子pSW7848-srvg23535检测片段;5:野生株ZJ-T;6:srvg23535候选突变株. Note: M1, M3: DL10000 DNA marker; M2, M4: DL2000 DNA marker; 1: Linearized segment of pSW7848; 2/3: UP/DOWN segments of srvg23535; 4: The segment of identification of the recombinant suicide vector by PCR; 5: Wild type ZJ-T; 6: The deletion candidate of srvg23535. |
|
|
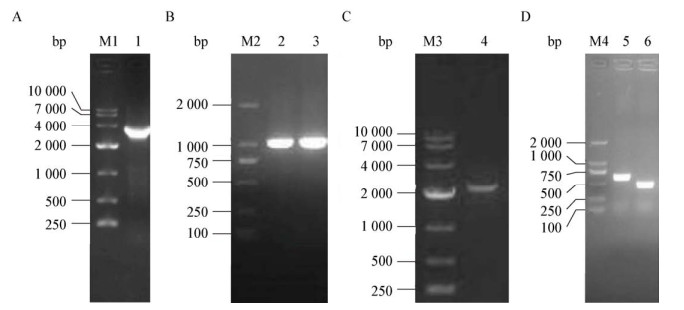
srvg23535缺失后,在LBS平板上菌落表型相对野生株均无变化,菌落表现为透明光滑,并且存在涌动性(Swarming),这说明srvg23535不参与调控溶藻弧菌的菌落形态(图 3)。
|
| 图 3 野生株和srvg23535突变株在LBS平板上的菌落形态 Figure 3 Colony morphology of the wild type and the srvg23535 mutant on LBS plates |
|
|
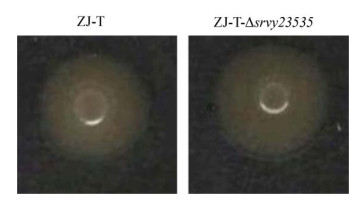
srvg23535缺失后,在0.3%琼脂LB平板上的运动性相对野生株无显著变化,这说明srvg23535不参与调控溶藻弧菌的运动性(图 4)。
|
| 图 4 野生株和srvg23535突变株在0.3%琼脂LB平板上的运动性 Figure 4 Swimming of the wild type and the srvg23535 mutant on 0.3% LB agar plates |
|
|
srvg23535缺失后,在1%脱脂奶粉LBS平板上,菌株胞外蛋白酶的分泌相对野生株没有显著差异,该结果表明srvg23535不参与调控溶藻弧菌胞外蛋白酶的分泌(图 5)。

|
| 图 5 野生株和srvg23535突变株在1%脱脂奶粉LBS平板上的胞外蛋白酶分泌圈 Figure 5 Extracellular protease secretion loop of the wild type and the srvg23535 mutant on 1% skimmed milk LBS plates |
|
|
过量的铜离子催化大量羟基自由基的形成,如同过氧化氢产生强的氧化压力,从而对细胞产生毒性;此外,摄铁系统是溶藻弧菌重要致病因子之一[24-26]。srvg23535缺失后,突变株相对野生株对H2O2、Cu2SO4抗性以及铁离子的获取无显著差异,这说明srvg23535不参与调控溶藻弧菌对H2O2、Cu2+的抗性以及对铁离子的吸收(图 6)。

|
| 图 6 野生株和srvg23535突变株对H2O2、CuSO4以及铁离子螯合剂DIP的敏感性 Figure 6 Sensitivity to H2O2, CuSO4 and DIP of the wild type and the srvg23535 mutant on LBS plate 注:1:ZJ-T;2:ZJ-T-Δsrvg23535. Note: 1: ZJ-T; 2: ZJ-T-Δsrvg23535. |
|
|
srvg23535缺失后,突变株相对野生株对所测试的30种抗生素敏抗性无显著差异;野生株和srvg23535突变株对包括林可霉素、万古霉素、克林霉素在内的12种抗生素具有抗性,对其他18种抗生素表现出不同程度的敏感,但是野生株和突变株之间无明显的差异(P < 0.05) (表 3)。
| 抗生素Antibiotics | 浓度 Concentration |
抑菌圈直径Size of inhibition zone (mm) | |
| (µg per disk) | ZJ-T | ZJ-T-Δsrvg23535 | |
| Lincomycin | 2 | − | − |
| Vancomycin | 30 | − | − |
| Clindamycin | 2 | − | − |
| Chloramphenicol | 300 | 21.00±1.41 | 22.00±2.83 |
| Ampicillin | 10 | − | − |
| Oxacillin | 1 | − | − |
| Piperacillin | 100 | − | − |
| Kanamycin | 30 | − | − |
| Penicillin-G | 10 | − | − |
| Cefazolin | 30 | 11.00±1.41 | 11.00±1.41 |
| Tobramycin | 10 | 12.50±0.71 | 11.00±0.00 |
| Neomycin | 30 | 13.67±2.52 | 15.67±2.08 |
| Novobiocin | 30 | 21.00±5.66 | 20.00±4.24 |
| Tetracycline | 30 | 15.67±6.03 | 19.00±4.24 |
| Erythromycin | 15 | 12.00±2.65 | 14.67±0.58 |
| Medemycin | 30 | 11.50±0.71 | 13.00±1.41 |
| Polymyxin B | 300 | 12.33±2.31 | 13.50±2.12 |
| Doxycycline | 30 | 19.50±3.54 | 20.50±3.54 |
| Acetylspiramycin | 30 | − | − |
| Spectinomycin | 100 | 10.33±1.53 | 10.50±2.12 |
| Minocycline | 30 | 24.00±5.66 | 25.50±6.36 |
| Cefixime | 5 | 10.67±1.53 | 11.00±1.41 |
| Amoxicillin | 10 | − | − |
| Azithromycin | 15 | − | − |
| Clarithromycin | 15 | 12.33±0.58 | 13.50±0.71 |
| Roxithromycin | 15 | 11.00±1.41 | 11.50±2.12 |
| Teicoplanin | 30 | − | − |
| Fosfomycin | 200 | 17.33±2.31 | 19.00±2.83 |
| Streptomycin | 300 | 11.33±1.53 | 10.33±0.58 |
| Gentamicin | 120 | 18.00±4.36 | 18.67±3.21 |
(1) 在富营养培养基中,突变株的生长与野生型相同(P > 0.05);在不同单一碳源和(NH4)2SO4作为氮源的培养基中,仅当以D-海藻糖为单一碳源时,srvg23535缺失株稳定期相比野生株生长量更小(One-way ANCOVA,F=81.194,P < 0.001;Student’s t检验,6.0–24.0 h之间,各时间点P < 0.05) (图 7F),说明srvg23535正调控溶藻弧菌对D-海藻糖的利用。
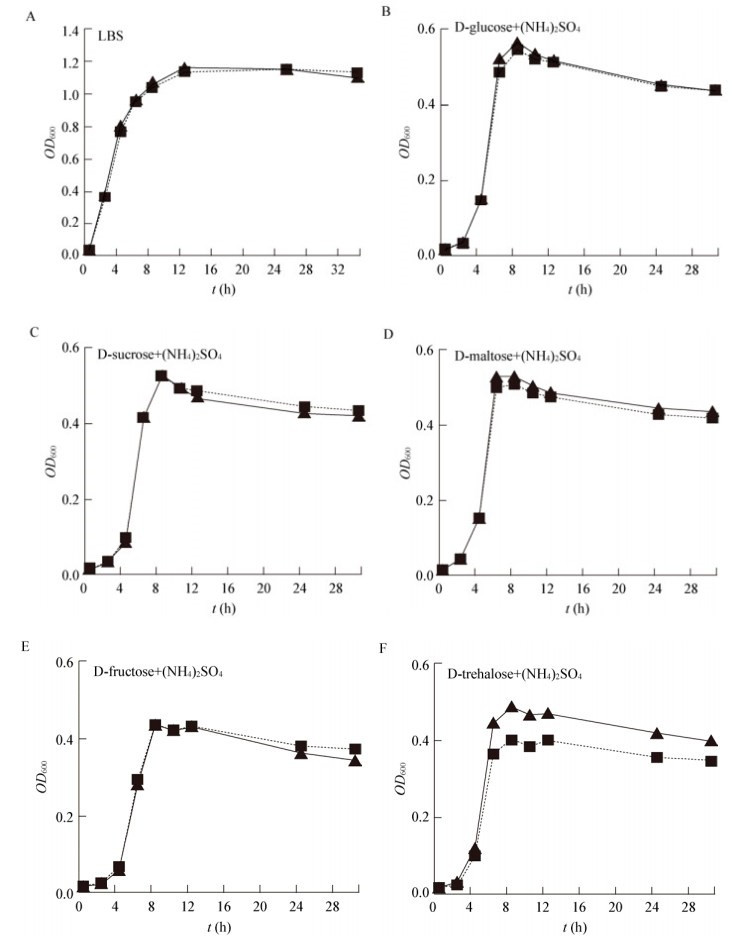
|
图 7 srvg23535缺失对富培养基中生长和各种碳源利用的影响
Figure 7 Effect of srvg23535 deletion on the growth in rich medium and the utilization of various carbon sources
注: : ZJ-T; : ZJ-T;
 :ZJ-T-Δsrvg23535;误差线对应于标准偏差.
Note: :ZJ-T-Δsrvg23535;误差线对应于标准偏差.
Note:  : ZJ-T;
: ZJ-T;  : ZJ-T-Δsrvg23535; Error bars correspond to standard deviations.
: ZJ-T-Δsrvg23535; Error bars correspond to standard deviations.
|
|
|
(2) M63中以葡萄糖作为单一碳源,以不同氨基酸作为单一氮源时,ZJ-T-Δsrvg23535与野生株生长无明显差异(P > 0.05) (图 8)。
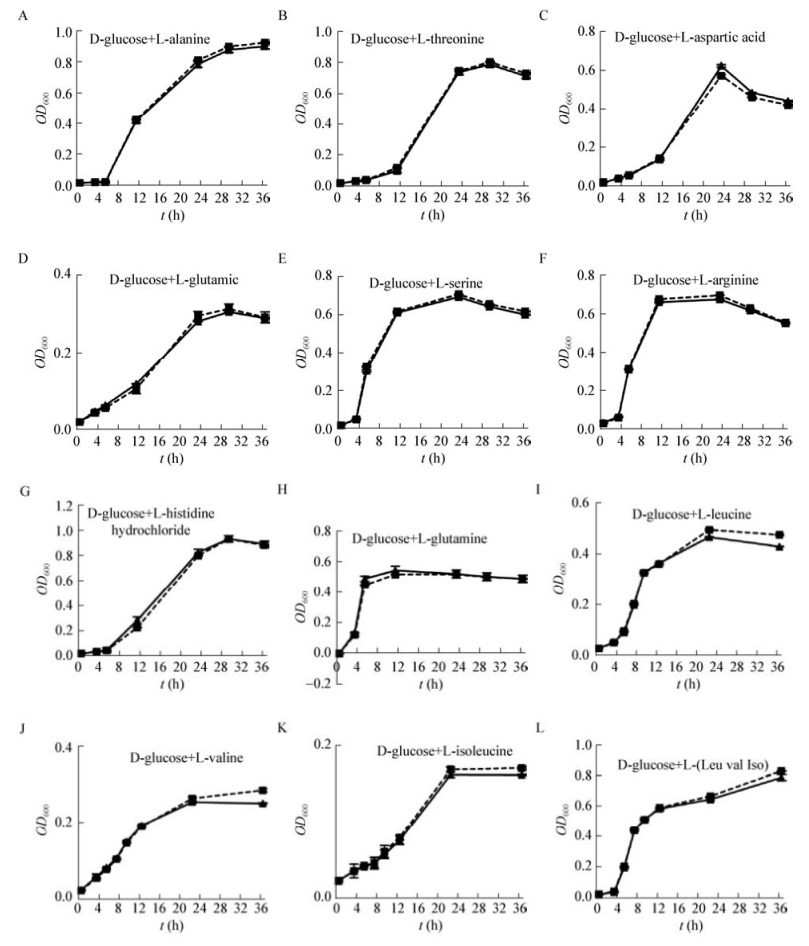
|
图 8 srvg23535缺失对各种氨基酸作为单一氮源利用的影响
Figure 8 Effect of srvg23535 deletion on the utilization of various amino acids as sole nitrogen source
注: :ZJ-T; :ZJ-T; :ZJ-T-Δsrvg23535;误差线对应于标准偏差.
Note: :ZJ-T-Δsrvg23535;误差线对应于标准偏差.
Note:  : ZJ-T;
: ZJ-T;  : ZJ-T-Δsrvg23535; Error bars correspond to standard deviations.
: ZJ-T-Δsrvg23535; Error bars correspond to standard deviations.
|
|
|
(3) PM02测试了细菌对95种不同碳源的利用,发现srvg23535缺失后,细菌以果胶为碳源时,在稳定期相对野生株生长量更大(One-way ANCOVA,F=23.024,P < 0.001;Student’s t检验,19.5 h之后,各时间点P < 0.05) (图 9A),说明srvg23535负调控溶藻弧菌对果胶的代谢。
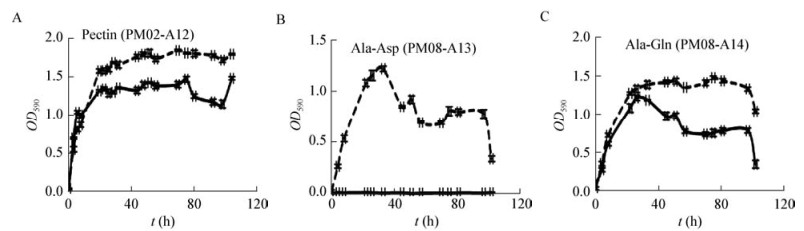
|
图 9 srvg23535缺失对果胶为单一碳源、丙氨酸-天冬氨酸和丙氨酸-谷氨酰胺为单一氮源利用的影响
Figure 9 Effect of srvg23535 deletion on the utilization of pectin as sole carbon source, and the utilization of Ala-Asp or Ala-Gln as sole nitrogen sourc
注: :ZJ-T, :ZJ-T, :ZJ-T-Δsrvg23535;误差线对应于标准偏差.
Note: :ZJ-T-Δsrvg23535;误差线对应于标准偏差.
Note:  : ZJ-T,
: ZJ-T,
 : ZJ-T-Δsrvg23535; Error bars correspond to standard deviations. : ZJ-T-Δsrvg23535; Error bars correspond to standard deviations.
|
|
|
(4) PM08测试了细菌对95种不同多肽的利用,发现srvg23535缺失后,可利用丙氨酸-天冬氨酸为氮源,而野生株不能利用其为氮源(One-way ANCOVA,F=212.914,P < 0.001;Student’s t检验,0.0 h之后,各时间点P < 0.05) (图 9B),此外,当以丙氨酸-谷氨酰胺为氮源时,突变株在潜伏期生长更快,并且稳定期相对野生株生长量更大(One-way ANCOVA,F=22.089,P < 0.001;Student’s t检验,4.0 h之后,各时间点P < 0.05) (图 9C),说明srvg23535负调控溶藻弧菌对丙氨酸-天冬氨酸和丙氨酸-谷氨酰胺的代谢。
3 讨论与结论菌落形态、运动性、胞外蛋白酶分泌、对H2O2和Cu2+的压力感应、铁离子摄取、抗生素抗性以及对不同营养物质利用程度等均被报道与细菌环境适应相关[23, 27]。近年来,原核小RNA的基因调控作用越来越受到重视[28-29]。小RNA通过与mRNA碱基配对,调控mRNA的稳定性,从而达到转录后调控蛋白表达的目的[29]。小RNA已被报道参与调控多种生物过程,如Spot42和SgrS调控糖代谢[29],GcvB和SR1调控氨基酸代谢[30-32],Qrrs和CsrBs调控群感效应[12, 33],RybB和MicA调控外膜蛋白合成[34],以及Fur调控铁平衡[35]。很多小RNA还可同时参与调控多种生物过程,如CsrB既调控群感效应,又调控碳代谢[36]。本研究中,我们以pSW7848自杀质粒为载体,通过等温组装和同源重组筛选技术,成功构建溶藻弧菌小RNA srvg23535缺失突变株,并对野生株和突变株的生物学特性进行了比较研究。
通过生物学特性比较,发现srvg23535缺失后并不影响溶藻弧菌的菌落形态、运动性、胞外蛋白酶分泌、对H2O2和Cu2SO4的压力感应、对铁的吸收利用、对所测试的30种抗生素抗性以及对所测试的大多数单一碳源、单一氮源的利用。但srvg23535缺失后,对D-海藻糖的代谢减弱,表现为稳定期生长量更小;对果胶的代谢增强,表现为稳定期生长量更大;对丙氨酸-谷氨酰胺的代谢增强,表现为潜伏期生长更快,并且稳定期生长量更大;并变得可利用丙氨酸-天冬氨酸作为氮源。截至目前仅发现SR1一个既参与氨基酸代谢又参与碳代谢的小RNA,并在细菌中高度保守[31-32]。SR1是枯草芽孢杆菌(Bacillus subtilis)中发现的第一个双功能小RNA,既能以RNA调控子形式调控精氨酸代谢,同时也能编码多肽调控gapA操纵子,从而参与调控糖分解[31-32]。因此,srvg23535很可能是第二个被发现的既参与碳代谢又参与氮代谢的小RNA,但是否都以小RNA形式在RNA水平进行调控,还是类似SR1既能以小RNA形式在RNA水平进行调控,又能编码成小肽进行蛋白水平的调控,仍需进一步研究揭示。
海藻糖作为一种碳源被储存在生物体内,必要的时候被分解代谢提供能量[37];同时,海藻糖作为许多生物的应急代谢物,对生物在逆境中的生存起着重要作用,如酿酒酵母在干燥脱水、热休克和高氧化等环境中其胞内海藻糖含量会显著升高[38-39],低温环境下大肠杆菌体内海藻糖含量会显著增加[40]。本研究发现srvg23535缺失后,以D-海藻糖为单一碳源生长时,稳定期的生长量降低,但是前期的生长速度与野生株无显著差异。我们推测,稳定期细菌处于营养饥饿以及缺氧等胁迫条件下,srvg23535缺失后,一方面影响稳定期D-海藻糖从体外转运到体内储存,另一方面影响D-海藻糖的分解代谢,从而导致其稳定期生长量降低。对野生株基因组[41]序列信息分析发现,其基因组上存在一个海藻糖转运代谢的操纵子treRBC,因此,srvg23535可能通过调控treRBC的表达而调控海藻糖的转运和代谢,具体调控机制需要进一步研究。果胶是一类杂多糖,由以D-半乳糖醛酸为主,少量D-半乳糖、D-阿拉伯糖、D-甘露糖、L-岩藻糖等多达12种的单糖组成[42-43],因此对果胶的代谢也涉及多种酶的共同作用。本研究发现srvg23535缺失后,以果胶为碳源生长时,稳定期的生长量增加,因此srvg23535可能调控了其中一种或者多种单糖的代谢,但是具体调控作用需要进一步以各单糖作为单一碳源进行代谢测试鉴定。
氨基酸作为蛋白质的基本骨架,酶的主要成分参与能量供给、酶催化、碳氮代谢等几乎所有生命活动,其精细调控对细菌生命活动具有重要意义。能量供给过程,氨基酸主要通过脱氨或者转氨作用,进而进入三羧酸循环,氧化供能。本研究发现,srvg23535缺失后,缺失株对丙氨酸-谷氨酰胺的代谢增强,并可利用丙氨酸-天冬氨酸为氮源;但当以L-丙氨酸、L-谷氨酰胺盐和L-天冬氨酸单独作为氮源时,srvg23535缺失前后生长并无差异,因此srvg23535很有可能通过调控丙氨酸-谷氨酰胺和丙氨酸-天冬氨酸的转运以及分解成单一氨基酸过程,从而影响细菌生长,但不直接调控二肽的降解代谢。基因组[41]序列信息分析发现,溶藻弧菌ZJ-T基因组上存在多个寡肽转运蛋白,如BAU10_09580、BAU10_15980和BAU10_15990,因此srvg23535可能通过调控这些基因的表达从而调控溶藻弧菌氮代谢,具体调控机制需要进一步深入研究。
本研究中,我们对溶藻弧菌小RNA srvg23535的环境适应调控功能进行了初探,发现srvg23535与溶藻弧菌的D-海藻糖、果胶、丙氨酸-谷氨酰胺和丙氨酸-天冬氨酸代谢相关。小RNA的调控方式主要是与mRNA配对,影响其稳定性,从而达到调控基因表达的目的。因此,下一步将对野生株和突变株的基因表达谱进行分析,通过比较野生株和突变株基因表达的差异,并结合本研究发现的调控表型,找出srvg23535调控的可能靶标基因,并通过凝胶迁移结合(EMSA)实验、双质粒荧光报告基因相互作用检测系统[44]等鉴定srvg23535与靶标基因的相互作用机制,从而阐明srvg23535对溶藻弧菌碳氮代谢的基因调控机制。
| [1] |
Siddiqui R, Alam MM, Naser MN, et al. Prevalence of Vibrio alginolyticus in sediment samples of river and coastal areas of Bangladesh[J]. Bangladesh Journal of Microbiology, 2016, 29(1): 1-6. DOI:10.3329/bjm.v29i1.28421 |
| [2] |
Citil BE, Derin S, Sankur F, et al. Vibrio alginolyticus associated chronic myringitis acquired in Mediterranean waters of Turkey[J]. Case Reports in Infectious Diseases, 2015, 2015(42): 187212-187214. |
| [3] |
Zheng TL, Wang GL, Jin S, et al. Studies on pathogen of ulcer disease of cultured large yellow croakers, Pseudosciaena crocea and its control medicines[J]. South China Fisheries Science, 2006, 2(1): 5-10. (in Chinese) 郑天伦, 王国良, 金珊, 等. 养殖大黄鱼溃疡病的病原菌及其防治药物研究[J]. 南方水产科学, 2006, 2(1): 5-10. DOI:10.3969/j.issn.2095-0780.2006.01.002 |
| [4] |
Newton A, Mahon BE. Increasing rates of Vibriosis in the United States, 1996-2010: review of surveillance data from 2 systems[J]. Clinical Infectious Diseases, 2012, 54(s5): S391-395. |
| [5] |
Baran I, Acar A, Gen Y, et al. Case report: A case of otitis externa due to Vibrio alginolyticus[J]. Türk Hijiyen Ve Deneysel Biyoloji Dergisi Turkish Bulletin of Hygiene & Experimental Biology, 2016, 73(1): 49-54. |
| [6] |
Gaüzère BA, Chanareille P, Vandroux D. Post nearly drowning Vibrio alginolyticus septicemia acquired in reunion (Indian ocean)][J]. Bulletin De La Societe De Pathologie Exotique, 2016, 109(3): 151-154. DOI:10.1007/s13149-016-0505-2 |
| [7] |
Chen C, Xie J, Hu C. Phenotypic and genetic differences between opaque and translucent colonies of Vibrio alginolyticus[J]. Biofouling, 2009, 25(6): 525-531. DOI:10.1080/08927010902964578 |
| [8] |
Solano C, Echeverz M, Lasa I. Biofilm dispersion and quorum sensing[J]. Current Opinion in Microbiology, 2014, 18(4): 96-104. |
| [9] |
Alonso CR. Post-transcriptional gene regulation via RNA control[M]. Humana Press, 2008.
|
| [10] |
Wang Z, Sun X, Zhao Y, et al. Evolution of gene regulation during transcription and translation[J]. Genome Biology & Evolution, 2015, 7(4): 1155-1167. |
| [11] |
Storz G, Vogel J, Wassarman KM. Regulation by small RNAs in bacteria: expanding frontiers[J]. Molecular Cell, 2011, 43(6): 880-891. DOI:10.1016/j.molcel.2011.08.022 |
| [12] |
Lenz DH, Miller M B, Zhu J, et al. CsrA and three redundant small RNAs regulate quorum sensing in Vibrio cholerae[J]. Molecular Microbiology, 2005, 58(4): 1186-1202. DOI:10.1111/j.1365-2958.2005.04902.x |
| [13] |
Liu JM, Camilli A. A broadening world of bacterial small RNAs[J]. Current Opinion in Microbiology, 2010, 13(1): 18-23. DOI:10.1016/j.mib.2009.11.004 |
| [14] |
Toledo-Arana A, Repoila F, Cossart P. Small noncoding RNAs controlling pathogenesis[J]. Current Opinion in Microbiology, 2007, 10(2): 182-188. DOI:10.1016/j.mib.2007.03.004 |
| [15] |
Bohn C, Rigoulay C, Chabelskaya S, et al. Experimental discovery of small RNAs in Staphylococcus aureus reveals a riboregulator of central metabolism[J]. Nucleic Acids Research, 2010, 38(19): 6620-6636. DOI:10.1093/nar/gkq462 |
| [16] |
Sharma CM, Hoffmann S, Darfeuille F, et al. The primary transcriptome of the major human pathogen Helicobacter pylori[J]. Nature, 2010, 464(7286): 250-255. DOI:10.1038/nature08756 |
| [17] |
Huang L, Hu J, Su Y, et al. Identification and characterization of three Vibrio alginolyticus non-coding RNAs involved in adhesion, chemotaxis, and motility processes[J]. Frontiers in Cellular & Infection Microbiology, 2015, 5: 56-68. |
| [18] |
Huang L, Hu J, Su Y, et al. Genome-wide detection of predicted non-coding RNAs related to the adhesion process in Vibrio alginolyticus using high-throughput sequencing[J]. Frontiers in Microbiology, 2016, 7: 619-628. |
| [19] |
Huang X, Chen C, Ren C, et al. Identification and characterization of a locus putatively involved in colanic acid biosynthesis in Vibrio alginolyticus ZJ-51[J]. Biofouling, 2017, 34(1): 1-14. |
| [20] |
Val ME, Skovgaard O, Ducos-galand M, et al. Genome engineering in Vibrio cholerae: a feasible approach to address biological issues[J]. PLoS Genetics, 2012, 8(1): e1002472. DOI:10.1371/journal.pgen.1002472 |
| [21] |
Le RF, Binesse J, Saulnier D, et al. Construction of a Vibrio splendidus mutant lacking the metalloprotease gene vsm by use of a novel counterselectable suicide vector[J]. Applied and Environmental Microbiology, 2007, 73(3): 777-784. DOI:10.1128/AEM.02147-06 |
| [22] |
Nguyen AN, Disconzi E, Charrière GM, et al. csrB gene duplication drives the evolution of redundant regulatory pathways controlling expression of the major toxic secreted metalloproteases in Vibrio tasmaniensis LGP32[J]. mSphere, 2018, 3(6): e00582-18. |
| [23] |
Deng YQ, Chen C, Zhao Z, et al. The RNA chaperone Hfq is involved in colony morphology, nutrient utilization and oxidative and envelope stress response in Vibrio alginolyticus[J]. PLoS One, 2016, 11(9): e0163689. DOI:10.1371/journal.pone.0163689 |
| [24] |
Deng YQ, Zhao Z, Liu SL, et al. Preliminary research of effect of different carbon sources on adhesion-related phenotypes in Vibrio alginolyticus[J]. Marine Sciences, 2016, 40(11): 99-107. (in Chinese) 邓益琴, 赵哲, 刘松林, 等. 不同碳源对溶藻弧菌黏附相关表型的影响初探及Hfq对其的调控[J]. 海洋科学, 2016, 40(11): 99-107. DOI:10.11759/hykx20160225001 |
| [25] |
Deng YQ, Zhao JJ, Liu SL, et al. Role of hfq in virulence regulation of Vibrio alginolyticus[J]. Oceanologia Et Limnologia Sinica, 2016, 47(3): 604-611. (in Chinese) 邓益琴, 赵晶晶, 刘松林, 等. Hfq对溶藻弧菌(Vibrio alginolyticus)毒力的调控分析[J]. 海洋与湖沼, 2016, 47(3): 604-611. |
| [26] |
Bochner BR, Gadzinski P, Panomitros E. Phenotype microarrays for high-throughput phenotypic testing and assay of gene function[J]. Genome Research, 2001, 11(7): 1246-1255. DOI:10.1101/gr.186501 |
| [27] |
Ahmad S, Ahmad M, Khan S, et al. An overview on phase variation, mechanisms and roles in bacterial adaptation[J]. Jpma the Journal of the Pakistan Medical Association, 2017, 67(2): 285-291. |
| [28] |
Vogel J. A rough guide to the non‐coding RNA world of Salmonella[J]. Molecular Microbiology, 2009, 71(1): 1-11. DOI:10.1111/mmi.2009.71.issue-1 |
| [29] |
Wagner EGH, Romby P. Small RNAs in bacteria and archaea: who they are, what they do, and how they do it[J]. Advances in Genetics, 2015, 90: 133-208. DOI:10.1016/bs.adgen.2015.05.001 |
| [30] |
Urbanowski ML, Stauffer LT, Stauffer GV. The gcvB gene encodes a small untranslated RNA involved in expression of the dipeptide and oligopeptide transport systems in Escherichia coli[J]. Molecular Microbiology, 2000, 37(4): 856-868. DOI:10.1046/j.1365-2958.2000.02051.x |
| [31] |
Heidrich N, Moll I, Brantl S. In vitro analysis of the interaction between the small RNA SR1 and its primary target ahrC mRNA[J]. Nucleic Acids Research, 2007, 35(13): 4331-4346. DOI:10.1093/nar/gkm439 |
| [32] |
Gimpel M, Heidrich N, Mäder U, et al. A dual-function sRNA from Bacillus subtilis: SR1 acts as a peptide encoding mRNA on the gapA operon[J]. Molecular Microbiology, 2010, 76(4): 990-1009. DOI:10.1111/mmi.2010.76.issue-4 |
| [33] |
Nguyen AN, Jacq A. Small RNAs in the Vibrionaceae: an ocean still to be explored[J]. Wiley Interdisciplinary Reviews Rna, 2014, 5(3): 381-392. DOI:10.1002/wrna.1218 |
| [34] |
Kai P, Pfeiffer V, Mika F, et al. σE‐dependent small RNAs of Salmonella respond to membrane stress by accelerating global omp mRNA decay[J]. Molecular Microbiology, 2006, 62(6): 1674-1688. DOI:10.1111/mmi.2006.62.issue-6 |
| [35] |
Massé E, Gottesman S. A small RNA regulates the expression of genes involved in iron metabolism in Escherichia coli[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(7): 4620-4625. DOI:10.1073/pnas.032066599 |
| [36] |
Revelles O, Millard P, Nougayrède JP, et al. The carbon storage regulator (Csr) system exerts a nutrient-specific control over central metabolism in Escherichia coli strain Nissle 1917[J]. PLoS One, 2013, 8(6): e66386. DOI:10.1371/journal.pone.0066386 |
| [37] |
Becker A, Schlöder P, Steele JE, et al. The regulation of trehalose metabolism in insects[J]. Experientia, 1996, 52(5): 433-439. DOI:10.1007/BF01919312 |
| [38] |
Attfield PV. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response[J]. FEBS Letters, 1987, 225(1-2): 259-263. DOI:10.1016/0014-5793(87)81170-5 |
| [39] |
Benaroudj N, Lee DH, Goldberg AL. Trehalose accumulation during cellular stress protects cells and cellular proteins from damage by oxygen radicals[J]. Journal of Biological Chemistry, 2001, 276(26): 24261-24267. DOI:10.1074/jbc.M101487200 |
| [40] |
Kandror O, Deleon A, Goldberg AL. Trehalose synthesis is induced upon exposure of Escherichia coli to cold and is essential for viability at low temperatures[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(15): 9727-9732. DOI:10.1073/pnas.142314099 |
| [41] |
Deng Y, Chen C, Zhao Z, et al. Complete Genome Sequence of Vibrio alginolyticus ZJ-T[J]. Genome Announcements, 2016, 4(5): e00912-16. |
| [42] |
Pérez S, Mazeau K, Penhoat CHD. The three-dimensional structures of the pectic polysaccharides[J]. Plant Physiology & Biochemistry, 2000, 38(1–2): 37-55. |
| [43] |
Mohnen D. Pectin structure and biosynthesis[J]. Current Opinion in Plant Biology, 2008, 11(3): 266-277. DOI:10.1016/j.pbi.2008.03.006 |
| [44] |
Urban JH, Vogel J. Translational control and target recognition by Escherichia coli small RNAs in vivo[J]. Nucleic Acids Research, 2007, 35(3): 1018-1037. DOI:10.1093/nar/gkl1040 |
 2019, Vol. 46
2019, Vol. 46