扩展功能
文章信息
- 张清霞, 孔祥伟, 张迎, 吉艳艳, 童蕴慧, 纪兆林
- ZHANG Qing-Xia, KONG Xiang-Wei, ZHANG Ying, JI Yan-Yan, TONG Yun-Hui, JI Zhao-Lin
- 土壤中群体感应淬灭细菌的原位分离与筛选
- Isolation and screen of quorum quenching bacteria in situ
- 微生物学通报, 2019, 46(3): 504-511
- Microbiology China, 2019, 46(3): 504-511
- DOI: 10.13344/j.microbiol.china.180128
-
文章历史
- 收稿日期: 2018-02-19
- 接受日期: 2018-05-07
- 网络首发日期: 2018-06-15
群体感应(Quorum sensing,QS)是细菌通过感知环境中信号分子的浓度判断细菌群体密度的大小,从而启动或关闭相关基因的表达来适应环境的变化,是细菌群体间的一种调控方式[1]。许多植物病原细菌通过群体感应系统来调控毒性基因的表达,因此群体感应系统是潜在的植物病害防治靶标。这种对细菌QS调控机制的干扰和破坏称为群体感应淬灭(Quorum quenching,QQ)。阻断这些信号通路的酶被称为群体淬灭酶[2]。已报道的N-酰基高丝氨酸内酯(N-acylhomoserine laetones,AHLs)淬灭酶主要有3种类型:内酯酶(Lactonase),可降解高丝氨酸内酯环;酰基转移酶(Acylase),可将AHL分解为高丝氨酸内酯和游离的脂肪酸;氧化还原酶(Oxidoreductase),可将酰胺侧链上位点5和6的羰基还原为羟基[3]。自2000年报道第一个AHL淬灭酶AiiA以来,约有40个AHL淬灭酶基因得到克隆和解析,其中大部分为AHL内酯酶,尚未见到关于AHL脱羧酶和AHL脱氨酶的报道[4]。由于细菌分离培养技术和AHL淬灭酶筛选报告菌体系的局限性,已报道的AHL降解菌的种类范围相似,而且多数淬灭酶基因已经被克隆并完成酶活机制解析。因此,通过传统方法进一步挖掘AHL淬灭酶基因资源将会更加困难。
近年已有学者通过不同手段,如使用营养少或不添加营养的培养基,相对长的培养时间,保护细胞不受外源氧化物的伤害,添加群体感应信号分子、短肽、腐殖酸及腐殖酸类似物等从土壤中原位分离细菌的报道[5]。Kaeberlein等[6]最早设计一个扩散池(Diffusion chamber)在模拟自然环境中分离获得了一些新型微生物,Nichols等[7]在扩散池基础上又研发出一个隔离芯片(Ichip),Ichip可实现高通量土壤原位培养细菌,通过这种方法可以使得从土壤中分离获得的细菌占土壤微生物总量的50%。本研究在Ichip基础上拟通过垫圈法从不同地区土样中分离细菌,采用琼脂条法、报告菌平板法、β-半乳糖苷酶的活性测定法进行复筛,构建细菌系统发育进化树,并检测对不同信号分子的降解能力,旨在为群体感应淬灭提供新型菌种资源。
1 材料与方法 1.1 细菌菌株信号分子产生菌荧光假单胞菌Pseudomonas fluorescens 2P24[8]、报告菌根癌土壤杆菌Agrobacterium tumefaciens NTL4 (pZLR4)[9] (Gmr,使用浓度20 μg/mL)和群体感应信号分子降解酶产生菌苍白杆菌Ochrobactrum sp. T63[10]由中国农业大学张力群教授馈赠。报告菌和待检测细菌均用ABM培养基在28 ℃、180 r/min振荡培养48 h测定降解信号分子的能力。N-(3-氧代己酰)高丝氨酸内酯(3OC6-HSL)、N-3-羰基十二酰基高丝氨酸内酯(3OC12-HSL)和N-3-羰基辛酰基高丝氨酸内酯(3OC8-HSL)购自Sigma公司。
1.2 培养基ABM培养基(g/L):将880 mL水琼脂加热融化并冷却至50 ℃,分别向其中加已灭菌的50 mL 20×盐溶液(NH4Cl 10.00,MgSO4·7H2O 3.00,KCl 1.50,CaCl2·2H2O 0.10和FeSO4·7H2O 0.03,蒸馏水500 mL,pH 7.2)、50 mL 20×缓冲液(K2HPO4 30.00和KH2PO4 11.50,蒸馏水500 mL,pH 7.0)和20 mL 10%甘露醇,制备成固体ABM培养基[11]。
LB培养基(g/L):胰蛋白胨10.0,酵母提取物5.0,NaCl 10.0,pH 7.0-7.2。
CN培养基(g/L):酸水解酪蛋白1.0;牛肉膏蛋白胨培养基1.0[12]。
Z buffer(g/L):Na2HPO4·12H2O 21.4,NaH2PO4·2H2O 6.2,MgSO4·7H2O 0.2,KCl 0.4,β-巯基乙醇7.0 mL[11]。
1.3 主要试剂和仪器硝化纤维素膜(直径0.22 μm和0.45 μm),英潍捷基贸易有限公司;制霉菌素(50 μg/mL)和放线菌酮(100 μg/mL),INALCO公司;维生素复合液(稀释1 000倍),ATCC公司。恒温培养箱,宁波江南仪器厂;旋转蒸发仪,安捷伦公司;分光光度计和PCR仪,东胜兴业科学仪器有限公司;离心机,Thermo Scientific公司。
1.4 P. fluorescens 2P24所产信号的粗提液制备P. fluorescens 2P24用900 mL液体LB培养基在28 ℃、180 r/min振荡培养24 h,12 000 r/min离心15 min去沉淀,等体积乙酸乙酯萃取,32 ℃旋转蒸发有机相后用2 mL甲醇溶解,粗提液保存于–20 ℃备用。信号分子粗提液系列稀释4个倍数浓度梯度:2、2×10–1、2×10–2、2×10–3倍。
1.5 垫圈法分离细菌参照Nichols等[7]方法,略作修改。将从不同地区采集的土样平铺于培养皿底部,将0.45 μm的微孔滤膜置于土样表面(土壤中细菌能够通过这个膜),上面放置内径为3 cm的垫圈,将含有维生素和制霉菌素的水琼脂培养基加入垫圈中,待培养基凝固后再将0.22 μm的微孔滤膜放置在水琼脂表面(图 1),封口膜密封后,放置28 ℃培养10 d后取出,将琼脂片切成4块,分别放于5 mL的CN培养基中,28℃、180 r/min振荡培养1 d取出试管,将培养液在LB平板上划线,28 ℃培养3 d后选取不同形态的单菌落保存。

|
| 图 1 垫圈法原位培养土壤中微生物 Figure 1 "Washer method" for in situ cultivation of soil microorganisms 注:1:直径0.22 µm微孔滤膜;2:金属垫圈;3:直径0.45 µm微孔滤膜;4:琼脂、维生素复合液和制霉菌素混合物. Note: 1: Top membrane filter (0.22 µm diameter); 2: Metal washer; 3: Bottom membrane filter (0.45 µm diameter); 4: Mixture (agar+ vitamins+nystatin). |
|
|
AHLs作为信号物质能诱导报告菌NTL4中lacZ基因启动子的表达,产生β-半乳糖苷酶。其活性大小与信号分子的多少呈正相关性,所以通过测定报告菌产生酶的活性可以检测待测细菌干扰信号分子生物功能的能力。本研究采用琼脂条法[8]初筛、报告菌平板法[10]复筛,筛选AHLs降解酶产生菌,选取平板上没有出现蓝色的细菌进一步研究。
1.7 待测细菌对模式QS系统干扰能力的测定 1.7.1 待测细菌外泌物对QS系统的干扰能力将600 μL待测细菌上清液和等体积报告菌培养液混合;28 ℃培养30 min后加20 μL信号分子粗提液,28 ℃反应6 h后12 000 r/min离心5 min;测定沉淀细胞中β-半乳糖苷酶的活性。酶活力的定义:在以上条件下每分钟分解1 nmol ONPG所需的酶量为1个酶活单位(U)。1 U=(1 000×OD420)/ (T×V×OD600)。T表示β-半乳糖苷酶与底物反应的时间(min);V表示与Z buffer混合的报告菌细胞悬浮液的体积(mL)。
1.7.2 待测细菌菌体对QS系统的干扰能力将菌体用无菌双蒸水清洗2次,配成细胞浓度为1×108-9×108 CFU/mL细胞悬浮液后取400 μL与20 μL信号粗提液混匀,28 ℃、130 r/min培养4-5 h;12 000 r/min离心1 min后将上清液与报告菌培养液按2:1 (体积比)混匀后28℃、180 r/min振荡培养3 h,测定OD600,12 000 r/min离心5 min后测定沉淀细胞中β-半乳糖苷酶的活性[10]。
1.8 16S rRNA基因的克隆及序列分析采用CTAB/NaCl法提取细菌基因组DNA作为模板,以扩增细菌16S rRNA基因的保守引物63F (5′-CAGGCCTAACACATGCAAGTC-3′)和1387R (5′-GGGCGGTGATGTACAAGGC-3′)进行PCR扩增[13]。PCR扩增产物送交生工生物工程(上海)股份有限公司测序。将所得序列登录NCBI网站进行BLAST比对,选取相似性96%以上的序列,再结合其模式菌株序列,采用MEGA 5.05软件进行遗传距离分析,基于邻位相连法(Neighbor-Joining,NJ)构建细菌16S rRNA基因的系统发育进化树,1 000次自举(Bootstrap)进行进化树的可信度评估[14]。
2 结果与分析 2.1 群体感应信号降解酶产生菌的分离与筛选通过垫圈法共分离得到502株细菌。从假单胞菌2P24中提取的信号分子梯度稀释后,用琼脂条法确定最佳浓度,其中稀释2 000倍报告菌颜色浅蓝,因此后续筛选时使用稀释200倍的信号分子溶液。A. tumefaciens NTL4 (pZLR4)作为报告菌受到外源AHLs类型的信号分子刺激后,激发NTL4中lacZ基因表达,产生β-半乳糖苷酶,分解培养基中X-Gal呈现蓝色,并且蓝斑的大小间接反映降解能力的强弱。通过琼脂条初筛和报告菌平板复筛最终得到21株细菌。
为进一步明确待测细菌对模式QS系统干扰能力的方式,分别检测待测细菌培养液上清和菌体对QS系统的干扰能力。已知苍白杆菌T63可产生降解AHLs的内酯酶,并且该酶是在细胞内发挥降解作用[10]。通过与对照菌株T63比较,筛选到11株具有较强降解酶活性的细菌,其中菌株120、129、151上清液中β-半乳糖苷酶活力较低,明显低于阳性对照(图 2C),说明这3株细菌可以产生AHLs降解酶分泌到培养液中;菌株3-49、3-58、3-59、61、66等菌株菌体β-半乳糖苷酶活性较低,均显著低于阳性对照(图 2B、D),说明这5株细菌产生的AHLs降解酶存在于胞内;菌株2-34、2-63、3-41胞内外的β-半乳糖苷酶活性均较低,说明这3株细菌可能产生不止一种AHLs降解酶(图 2A、B)。

|
| 图 2 群体感应淬灭细菌上清和细胞对QS系统的干扰能力 Figure 2 Quorum quenching activity in culture supernatant and culture cells on QS system 注:图A和C表示细菌外泌物中β-半乳糖苷酶活性;图B和D表示细菌菌体中β-半乳糖苷酶活性;C和NC分别表示阳性对照和阴性对照. Note: A and C: The activity of β-galactosidase in culture supernatant; B and D: The activity of β-galactosidase in culture cells; "C" and "NC" indicated positive control and negative control, respectively. |
|
|
将分离得到的11株群体感应淬灭细菌的16S rRNA基因序列上传到NCBI与已知标准菌株16S rRNA基因序列进行比对,结果表明菌株3-59和151均与标准菌株A. pittii CIP 70.29相似性达99%,菌株2-34和3-41分别与标准菌株A. baumannii ATCC 19606和A. radioresistens DSM 6976相似度为99%;菌株3-49与P. gessardii DSM 17152相似度为96%;菌株61、66和120与P. granadensis F-278770相似度均为99%;菌株3-58与P. taiwanensis BCRC 17751相似度为99%;菌株129与标准菌株Rheinheimera tangshanensis JA3-B52相似度为98%;菌株2-63与标准菌株Proteus penneri NCTC 12737和Proteus vulgaris ATCC 29905相似度均为99%以上。而且11株细菌与各自相似性最高的标准菌株在系统进化树上均处在同一分支上(图 3)。综上,分离得到的11株群体感应淬灭细菌经鉴定可分为4个属,不动杆菌属(4株)、假单胞菌属(5株)、变形杆菌属(1株)、莱茵海默氏菌属(1株)。
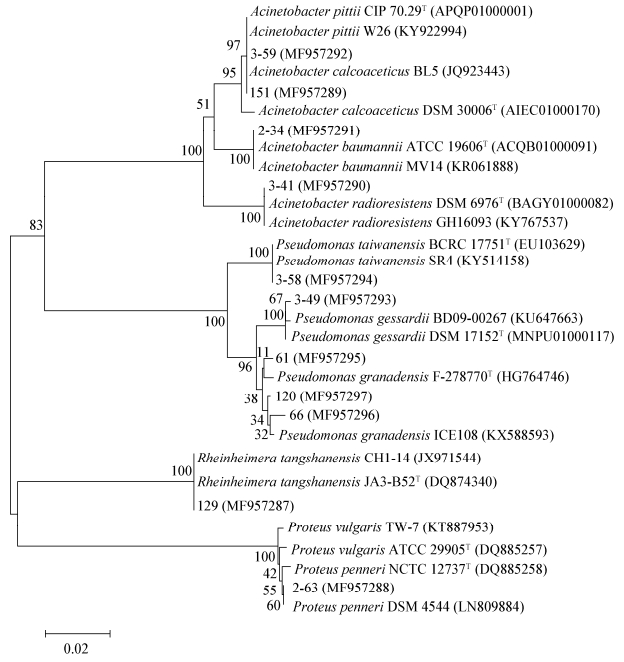
|
| 图 3 基于16S rRNA基因序列的系统进化树 Figure 3 Phylogenetic tree based on 16S rRNA gene sequences 注:分支点上的数字为自展值,标尺表示0.02核苷酸置换率,序列登录号列于括号中. Note: Numbers at the nodes represented the bootstrap values. The scale bar 0.02 means the nucleotide substitution rate of 0.02. The sequence accession numbers are shown in the brackets. |
|
|
通过报告菌平板法,以3种标准信号分子3OC6-HSL、3OC8-HSL和3OC12-HSL做底物,检测供试细菌对不同信号分子的降解能力。苍白杆菌T63产生的AidH内酯酶可降解这3种信号分子[15],作为本文参照菌株。结果表明大部分待测细菌可完全降解3OC12-HSL,报告菌不变蓝,部分细菌可降解3OC6-HSL和3OC8-HSL,报告菌蓝斑明显小于阳性对照(表 1)。
| 分离物 Isolate |
3OC6- HSL |
3OC8- HSL |
3OC12- HSL |
分离地点或参考文献 Isolation location or reference |
| Ochrobactrum sp. T63 | ++ | ++ | ++ | [10] |
| Proteus sp. 2-63 | + | + | ++ | 内蒙赤峰Chifeng, Inner Mongolia |
| Rheineimera sp. 129 | + | – | ++ | 吉林长春Changchun, Jilin province |
| Acinetobacter sp. 2-34 | – | – | + | 内蒙赤峰Chifeng, Inner Mongolia |
| Acinetobacter sp. 3-41 | + | + | ++ | 江苏盐城Yancheng, Jiangsu province |
| Acinetobacter sp. 3-59 | + | + | ++ | 江苏盐城Yancheng, Jiangsu province |
| Acinetobacter sp. 151 | – | – | + | 吉林长春Changchun, Jilin province |
| Pseudomonas sp. 3-49 | ++ | ++ | ++ | 江苏盐城Yancheng, Jiangsu province |
| Pseudomonas sp. 3-58 | ++ | ++ | ++ | 江苏盐城Yancheng, Jiangsu province |
| Pseudomonas sp. 61 | + | – | ++ | 吉林长春Changchun, Jilin province |
| Pseudomonas sp. 66 | + | – | ++ | 吉林长春Changchun, Jilin province |
| Pseudomonas sp. 120 | ++ | + | ++ | 吉林长春Changchun, Jilin province |
| 注:++:降解能力强(报告菌不变蓝);+:具有一定降解能力(报告菌部分变蓝);–:无降解能力.所有标准信号分子使用浓度为0.001 mmol/L. Note: ++: Strong degradation ability (Reporter strain does not turn blue); +: Degradation ability (Reporter strain partially turn blue); –: No degradation ability. The concentration of all AHLs is 0.001 mmol/L. |
||||
有报道指出环境中存在的微生物估计达到105-106种,但在当前的实验条件下可分离鉴定的种类数量较少,自然界中约有99%的微生物在常规试验条件下难以分离获得,这使微生物资源的开发利用受到限制[16]。宏基因组技术通过不培养微生物而直接对环境中总DNA进行克隆筛选来突破这个瓶颈。近年已有利用宏基因组学方法筛选AHL淬灭酶的报道,如内酯酶BpiB07、BpiB04、BpiB01、BpiB05、QlcA,AHL酰基转移酶QsdB,AHL氧化还原酶BpiB09,然而从土壤宏基因组文库中筛选QS淬灭酶得到的阳性克隆概率仍然较低[4]。
本研究从垫圈法模拟土壤环境分离到的细菌中筛选得到11株AHL淬灭细菌,经16S rRNA基因序列分析,结果显示3-49、3-58、61、66、120为假单胞菌属细菌,3-59、151、3-41、2-34为不动杆菌属细菌,2-63为变形杆菌,129为莱茵海默氏菌。本论文中垫圈法原位分离到的假单胞菌和不动杆菌也曾经有文献报道过。如假单胞菌P. aeruginosa和P. syringae均可产生2种以上AHLs酰基转移酶,而且主要降解长链信号分子[17-18]。不动杆菌中也可产生AHL酰基转移酶和AHL内酯酶[19-20]。Sun等[21]从海洋细菌Rheineimera aquimaris QS102的乙酸乙酯提取物中分离获得二酮哌嗪物质,该物质可干扰许多革兰氏阴性细菌的群体感应活性,但尚未有关于变形杆菌和莱茵海默氏菌通过产生淬灭酶降解酰基高丝氨酸内酯信号分子的报道。可见,使用营养少或不添加营养的培养基、相对长的培养时间可分离到新型AHLs降解细菌。采用常规丁内酯平板法主要分离得到节杆菌、假单胞菌和芽孢杆菌,这3种类型的细菌在土壤中较常见,容易分离获得[22]。垫圈法分离得到的大部分细菌仍然是假单胞菌,推测可能因为未进行富集培养,使得分离物中假单胞菌成为优势种群,因此今后可采用γ-己内酯处理土壤1个月后再用垫圈法进行分离,以提高分离物的种群丰度[23]。
不同类型信号分子降解酶的作用底物有差异。通常内酯酶可降解多种AHLs,而酰基转移酶可有效降解长链的信号分子,具有较强的底物特异性且大部分属于胞内酶。假单胞菌产生的信号降解酶均属于酰基转移酶[4],本文分离到的假单胞菌3-49、3-58、61、66和120可完全降解3OC12-HSL,而对3OC6-HSL和3OC8-HSL有较弱的降解能力,是否属于酰基转移酶仍需进一步研究。同样地Proteus sp. 2-63和Rheineimera sp. 129也对长链信号分子3OC12-HSL具有较强的降解活性,可部分降解3OC6-HSL。这2种细菌能降解信号分子尚未见文献报道,今后将进一步挖掘新的降解酶基因并解析其作用机制,为预防和控制依赖于群体感应系统表达致病性的细菌病害提供新型微生物资源。
| [1] |
Fuqua C, Parsek MR, Greenberg EP. Regulation of gene expression by cell-to-cell communication: acyl-homoserine lactone quorum sensing[J]. Annual Review of Genetics, 2001, 35: 439-468. DOI:10.1146/annurev.genet.35.102401.090913 |
| [2] |
Zhang LQ, Tian T, Mei GY. Quorum quenching, a new strategy for controlling plant bacterial diseases[J]. Chinese Journal of Biological Control, 2010, 26(3): 241-247. (in Chinese) 张力群, 田涛, 梅桂英. 群体感应淬灭—防治植物细菌病害的新策略[J]. 中国生物防治, 2010, 26(3): 241-247. |
| [3] |
Chen F, Gao YX, Chen XY, et al. Quorum quenching enzymes and their application in degrading signal molecules to block quorum sensing-dependent infection[J]. International Journal of Molecular Sciences, 2013, 14(9): 17477-17500. DOI:10.3390/ijms140917477 |
| [4] |
Fetzner S. Quorum quenching enzymes[J]. Journal of Biotechnology, 2015, 201: 2-14. DOI:10.1016/j.jbiotec.2014.09.001 |
| [5] |
Stevenson BS, Eichorst SA, Wertz JT, et al. New strategies for cultivation and detection of previously uncultured microbes[J]. Applied and Environmental Microbiology, 2004, 70(8): 4748-4755. DOI:10.1128/AEM.70.8.4748-4755.2004 |
| [6] |
Kaeberlein T, Lewis K, Epstein SS. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment[J]. Science, 2002, 296(5570): 1127-1129. DOI:10.1126/science.1070633 |
| [7] |
Nichols D, Cahoon N, Trakhtenberg EM, et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species[J]. Applied and Environmental Microbiology, 2010, 76(8): 2445-2450. DOI:10.1128/AEM.01754-09 |
| [8] |
Wei HL, Zhang LQ. Quorum-sensing system influences root colonization and biological control ability in Pseudomonas fluorescens2P24[J]. Antonie Van Leeuwenhoek, 2006, 89(2): 267-280. DOI:10.1007/s10482-005-9028-8 |
| [9] |
Cha C, Gao P, Chen YC, et al. Production of acyl-homoserine lactone quorum-sensing signals by gram-negative plant-associated bacteria[J]. Molecular Plant-Microbe Interactions, 1998, 11(11): 1119-1129. DOI:10.1094/MPMI.1998.11.11.1119 |
| [10] |
Mei GY, Yan XX, Turak A, et al. AidH, an alpha/beta-hydrolase fold family member from an Ochrobactrum sp. strain, is a novel N-acylhomoserine lactonase[J]. Applied and Environmental Microbiology, 2010, 76(15): 4933-4942. DOI:10.1128/AEM.00477-10 |
| [11] |
Chilton MD, Currier TC, Farrand SK, et al. Agrobacterium tumefaciens DNA and PS8 bacteriophage DNA not detected in crown gall tumors[J]. Proceedings of the National Academy of Sciences of the United States of America, 1974, 71(9): 3672-3676. DOI:10.1073/pnas.71.9.3672 |
| [12] |
Gavrish E, Bollmann A, Epstein S, et al. A trap for in situ cultivation of filamentous actinobacteria[J]. Journal of Microbiological Methods, 2008, 72(3): 257-262. DOI:10.1016/j.mimet.2007.12.009 |
| [13] |
Marchesi JR, Sato T, Weightman AJ, et al. Design and evaluation of useful bacterium-specific PCR primers that amplify genes coding for bacterial 16s rRNA[J]. Applied and Environmental Microbiology, 1998, 64(2): 759-799. |
| [14] |
Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology Evolution, 2011, 28(10): 2731-2739. DOI:10.1093/molbev/msr121 |
| [15] |
Gao A, Mei G, Liu S, et al. High-resolution structures of AidH complexes provide insights into a novel catalytic mechanism for N-acyl homoserine lactonase[J]. Acta Crystallographica Section D, Biological Crystallography, 2013, 69(1): 82-91. DOI:10.1107/S0907444912042369 |
| [16] |
Lewis K. Platforms for antibiotic discovery[J]. Nature Reviews Drug Discovery, 2013, 12: 371-387. DOI:10.1038/nrd3975 |
| [17] |
Wahjudi M, Papaioannou E, Hendrawati O, et al. PA0305 of Pseudomonas aeruginosa is a quorum quenching acylhomoserine lactone acylase belonging to the Ntn hydrolase superfamily[J]. Microbiology, 2011, 157: 2042-2055. DOI:10.1099/mic.0.043935-0 |
| [18] |
Shepherd RW, Lindow SE. Two dissimilar N-acyl-homoserine lactone acylases of Pseudomonas syringae influence colony and biofilm morphology[J]. Applied and Environmental Microbiology, 2009, 75(1): 45-53. DOI:10.1128/AEM.01723-08 |
| [19] |
Ochiai S, Yasumoto S, Morohoshi T, et al. AmiE, a novel N-acylhomoserine lactone acylase belonging to the amidase family, from the activated-sludge isolate Acinetobacter sp. strain Ooi24[J]. Applied and Environmental Microbiology, 2014, 80(22): 6919-6925. DOI:10.1128/AEM.02190-14 |
| [20] |
Chan KG, Atkinson S, Mathee K, et al. Characterization of N-acylhomoserine lactone-degrading bacteria associated with the Zingiber officinale (ginger) rhizosphere: co-existence of quorum quenching and quorum sensing in Acinetobacter and Burkholderia[J]. BMC Microbiology, 2011, 11: 51. DOI:10.1186/1471-2180-11-51 |
| [21] |
Sun SW, Dai XY, Sun J, et al. A diketopiperazine factor from Rheinheimera aquimaris QS102 exhibits anti-quorum sensing activity[J]. Scientific Reports, 2016, 6: 39637. DOI:10.1038/srep39637 |
| [22] |
Zhang QX, Zhang Y, Ji YY, et al. Isolation and characterization of quorum-quenching genes from bacterium Pseudomonas sp. WX14[J]. Chinese Journal of Biological Control, 2018, 341(1): 109-116. (in Chinese) 张清霞, 张迎, 吉艳艳, 等. 假单胞菌WX14群体感应淬灭酶基因的克隆及其功能研究[J]. 中国生物防治学报, 2018, 341(1): 109-116. |
| [23] |
Tannières M, Beury-Cirou A, Vigouroux A, et al. A metagenomics study highlights phylogenetic proximity of quorum-quenching and xenobiotic-degrading amidases of the AS-family[J]. PLoS One, 2013, 8(6): e65473. DOI:10.1371/journal.pone.0065473 |
 2019, Vol. 46
2019, Vol. 46




