扩展功能
文章信息
- 陈伟, 季秀玲, 张琦, 张东芳, 袁飞, 魏云林
- CHEN Wei, JI Xiu-Ling, ZHANG Qi, ZHANG Dong-Fang, YUAN Fei, WEI Yun-Lin
- 纳帕海高原湿地真菌群落多样性和组成的分布
- Diversity and composition of fungal community in Napahai plateau wetlands
- 微生物学通报, 2019, 46(3): 494-503
- Microbiology China, 2019, 46(3): 494-503
- DOI: 10.13344/j.microbiol.china.180128
-
文章历史
- 收稿日期: 2018-02-10
- 接受日期: 2018-04-23
- 网络首发日期: 2018-05-09
2. 昆明理工大学医学院 云南 昆明 650500
2. Medical School, Kunming University of Science and Technology, Kunming, Yunnan 650500, China
在湿地生态系统中,植被种类多样性对维持湿地生态系统功能和稳定的重要性已得到普遍认识,但作为对环境变化更为敏感的微生物群落,事实上,远比植被种类多样性能更好地反映环境的变化过程。湿地土壤微生物群落多样性和组成能更为灵敏和全面地反映湿地生态条件,是决定湿地土壤环境质量和生态特征的敏感指标,也是开展湿地生态修复过程中不可忽视的因素,其研究对于维持湿地生态系统平衡、修复受损湿地和开展环境综合治理均具有重要意义[1]。
在真核微生物中,真菌种类繁多且分布广泛,土壤中真菌几乎包括了所有的真菌群落类型,在土壤表层0-10 cm处真菌数量最多。真菌作为土壤中主要的分解者,可分解纤维素、淀粉和木质素等,在腐殖质形成和营养循环中起着重要作用,且有利于土壤结构和理化性质的稳定及肥力的提高[2]。此外,真菌中的共生型真菌可与植物形成菌根,进而影响植物群落的生产力和多样性,而寄生于植物的病原型真菌则降低植物的竞争力,从而间接改变植物的群落结构[3]。土壤中真菌参与湿地生态系统的维持和稳定,并发挥着特殊的作用和功能。土壤真菌参与有机质分解,改变土壤结构和理化性质,影响植物生长,同时,植物-土壤的反馈作用又改变土壤真菌的数量和群落组成,影响生态系统的功能与稳定。因此,以真菌多样性和组成作为退化的纳帕海高原湿地生态环境监测的微生物学指标更为直观。研究发现,在真菌rRNA编码基因中存在一段特殊非转录区——ITS (Internal transcribed spacer),Liu等[4]利用454高通量测序对高盐地区土壤中真核微生物的ITS基因多样性进行了研究,结果表明Dunaliella、Alternaria和Chlamydomonas为真核微生物优势属,真核微生物群落多样性极高;Yang等[5]利用Illumina MiSeq高通量测序对黄土高原土壤中真菌的ITS2基因多样性进行了研究,结果表明,Ascomycota、Basidiomycota和Zygomycota占主导地位(纲分级水平)。由此可见,ITS适合于真菌群落的分子鉴定及差异显著菌群间的系统发育关系分析[6]。土壤真菌多样性对维持生态系统的平衡具有重要的作用。
纳帕海高原湿地是云南高原沼泽面积最大和最具代表性的湿地类型,有着若尔盖高原湿地和我国北方湿地所不具备的特点,是我国独有的低纬度高海拔季节性沼泽湿地。年均温度低,旱雨两季分明,11月至翌年5月降水少为旱季,6月至10月为明显雨季[7]。由于纳帕海高原湿地海拔高,半封闭,且土壤有机质丰富,因此,其特殊的自然环境选择压力使得纳帕海高原湿地成为研究微生物群落组成与环境关系以及多样性形成和进化的理想场所。在微生物群落中,哪些真菌群落更具有优势?哪些种群进化出了更好的响应环境变化的机制?探索纳帕海高原湿地真菌群落多样性和组成以及与时空变化和环境因子之间的关系,对保护和恢复湿地生态系统具有重要意义。
本研究分别在雨季(RS)和旱季(DS)采集纳帕海高原湿地沼泽土(YN)、沼泽化草甸土(SD)和泥炭土(NT) 3种不同类型的土壤样品,基于荧光定量PCR (qPCR)和Illumina MiSeq高通量测序技术,分析纳帕海高原湿地真菌数量的分布,阐明真菌群落多样性和组成的时空变化规律。同时,进一步探索真菌群落多样性与土壤理化因子关系,揭示该区域微生物资源和湿地环境保护的重要意义。
1 材料与方法 1.1 土壤样品的采集及保存根据典型性和可行性原则,2013年11月(DS)和2014年6月(RS)到纳帕海高原湿地E99°37′22″- 99°38′16″和N27°50′01″-N27°53′35″样区进行采样。
由于土壤的不均匀性,每种土壤类型在采样单元内选择8个样点,样点呈“S”分布,对每个样点均选取景观植被相似或土壤类型条件相对接近的区域,土层深度为10 cm,记录其经纬度和海拔。8个样点采集的土壤样品等量(250 g)混合从而构成每种土壤类型内的一份样品。
用无菌手套采完土样后,立即装入已灭菌的自封袋中,迅速放入冰盒保存,于当天送回实验室。部分样品4 ℃保存,用于测定土壤样品理化因子;部分样品-20 ℃冷冻保存,用于提取土壤样品基因组DNA,分析真菌数量及群落多样性;剩余样品-80 ℃长期保存。
1.2 土壤样品理化因子的测定YN、SD和NT采样区土壤样品理化因子测定参照国家标准,由农业部农产品质量监督检验测试中心(昆明)协助完成。每个土样重复3次,数值以平均值计。
1.3 土壤样品DNA的提取土壤样品运至实验室后,对每个土样去除树根、树叶等杂质,放入无菌研钵内研磨。由于YN采样区土壤样品含水量大,因此放入烘箱(50℃)烘干后待用。根据PowerSoil® DNA Isolation Kit (MoBio公司)说明书,分别提取6个土壤样品的基因组DNA。
1.4 真菌数量荧光定量PCR (qPCR)检测合成真菌rRNA ITS区特异性PCR扩增引物:NSIF (5′-GATTGAATGGCTWAGTGAGG-3′)和58A2R (5′-CTGCGTTCTTCATCGAT-3′[8])用于本研究中YN、SD和NT采样区土壤样品中真菌微生物数量的qPCR分析。以红冬孢酵母YM25235为标准菌株,构建真菌qPCR标准品质粒。
采用SYBR® Premix ExTaqTM试剂盒(TaKaRa公司),在荧光定量PCR仪(Applied Biosystems)上进行检测。qPCR反应体系:Premix ExTaq 10 μL,10 μmol/L F引物0.4 μL,10 μmol/L R引物0.4 μL。ROX Dye 0.08 μL,DNA模板1 μL,ddH2O 8.12 μL;反应条件:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 34 s,40个循环。在60 ℃延伸时检测荧光信号,以标准质粒模板拷贝数为横坐标,荧光信号达设定阈值是所经历的循环数Ct值为纵坐标,生成标准曲线。标准曲线每个浓度梯度做3次平行重复,同时设置阴性对照。真菌的扩增效率为99.1%,R2为0.996。
参照标准曲线构建方法,对6个土壤样品真菌数量进行qPCR分析,每个土壤样品设3个平行,阴性对照模板以超纯水替换。将检测到的Ct值平均后代入相应的标准曲线推算初始拷贝数,最后以基因拷贝数每克土壤样品为单位进行分析。
1.5 真菌基因组DNA PCR扩增以YN、SD和NT采样区土壤样品中提取的基因组DNA为模板,对真菌rRNA ITS2区进行PCR扩增。真菌rRNA ITS2引物序列为[9]:ITSF (5′-GCATCGATGAAGAACGCAGC-3′)和ITSR (5′-TCCTCCGCTTATTGATAGC-3′)。PCR反应体系:2×MightyAmp Buffer 25 μL,10 μmol/L ITSF引物2 μL,10 μmol/L ITSR引物2 μL,MightyAmp DNA Polymerase 1 μL,DNA模板(100-200 ng) 2 μL,ddH2O补足到50 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 1 min,50 ℃ 1 min,72 ℃ 1 min,25个循环;72 ℃ 5 min。
PCR扩增产物用琼脂糖凝胶电泳进行检测。每个样品重复进行3次PCR扩增,将同一样品PCR产物进行混样,用GeneJET试剂盒回收产物,按照Illumina公司提供的标准流程进行建库,测序部分由北京计算机中心协助完成。
1.6 高通量测序及数据处理对测序所获得的序列,根据样品的INDEX序列,使用软件FLASH V1.2.7 (http://ccb.jhu.edu/software/FLASH/)分别将每条序列的READ1和READ2的Overlap进行拼接,去除接头杂序列,得到拼接原始序列,随后使用Chimera进行序列过滤查找嵌合体,最终得到优化序列。
使用Mothur V1.34.4将优化后的序列在基于97%序列相似性的水平上进行各样品的OTU生成[10],并根据OTU数目绘制OTU等级曲线(Rank abundance curve)[11],从而比较土壤样品中真菌群落的丰富度和均匀度。
根据数据库的参考序列对划分的OTU进行种属鉴定,使用R V3.0.2进行图展示,阐明土壤样品微生物群落多样性和组成的时空差异[12]。在不同的分类水平上,将土壤样品中微生物群落结构不能分类到目前已知微生物分类单元的定义为“Unknow”,将微生物群落组成所占比例小于1%的定义为“Others”,将微生物群落信息虽然能够与数据库中的序列比对上,但数据尚未有明确的注释信息定义为“Unclassified”。
1.7 数据分析采用MJ Opticon Monitor TM 3.1实时荧光定量分析软件分析qPCR数据,利用Canoco V4.5.1对土壤样品中真菌群落多样性和土壤理化因子进行CCA (Canonical correlation analysis)相关性分析。
2 结果与分析 2.1 土壤样品采样区分布情况在纳帕海高原湿地沼泽土(YN)、沼泽化草甸土(SD)和泥炭土(NT)中旱季(DS)采集的DS.YN、DS.SD、DS.NT和雨季(RS)采集的RS.YN、RS.SD、RS.NT的6个土壤样品,其采样记录见表 1,分布见图 1。从现场实际的采样环境来看,YN采样区在腊浪附近,土壤样品明显湿润,含水量最大,且人为活动干扰几乎没有,在本研究中可视为环境本底区;NT采样区耗牛和马等畜牧过度放牧现象严重,成为了天然牧场,且属于旅游热点地,游客多,在本研究中可视为受人为活动干扰严重区。
| 土壤样品Soil samples | 采样点Sampling points | 经度Longitude | 纬度Latitude | 海拔Average altitude (m) |
| YN | NPH-YN1-YN8 | E99°37′43.00″–41.26″ | N27°54′26.00″–24.38″ | 3 290 |
| SD | NPH-SD1-SD8 | E99°38′6.46″–0.67″ | N27°51′36.33″–40.22″ | 3 277 |
| NT | NPH-NT1-NT8 | E99°38′7.00″–3.63″ | N27°50′1.00″–49′58.92″ | 3 273 |

|
| 图 1 纳帕海高原湿地采样区分布图 Figure 1 The distribution of Napahai plateau wetland sampling sites 注:A:采样区总图;B:YN、SD和NT采样区具体分布图. Note: A: General of sampling areas; B: The sampling areas distribution of YN, SD and NT. |
|
|
分别对旱季和雨季YN、SD和NT采样区采集的6个土壤样品测定pH、有机质含量(SOM)、全氮(TN)、水解性氮(N)、全磷(TP)、速效磷(P)、全钾(TK)、速效钾(K)、速效锌(Zn)、速效锰(Mn)、速效铁(Fe)和速效铜(Cu)理化因子,结果见表 2。
| Samples/factors | DS.YN | DS.SD | DS.NT | RS.YN | RS.SD | RS.NT |
| SOM (g/kg) | 105.800±13.430 | 31.200±3.630 | 43.700±3.150 | 145.800±11.210 | 22.300±2.130 | 59.700±3.190 |
| TN (%) | 0.568±0.060 | 0.260±0.020 | 0.445±0.020 | 0.659±0.030 | 0.180±0.000 | 0.435±0.050 |
| N (mg/kg) | 393.000±20.100 | 121.000±14.500 | 246.300±18.700 | 381.000±30.100 | 106.000±9.800 | 259.000±16.800 |
| TP (%) | 0.032±0.000 | 0.102±0.000 | 0.279±0.000 | 0.088±0.000 | 0.090±0.000 | 0.120±0.000 |
| P (mg/kg) | 9.140±0.680 | 13.800±0.130 | 35.200±1.210 | 12.800±1.050 | 30.500±2.960 | 42.600±3.690 |
| TK (%) | 0.416±0.021 | 0.664±0.015 | 0.795±0.020 | 0.647±0.013 | 0.732±0.023 | 0.950±0.031 |
| K (mg/kg) | 97.200±5.700 | 185.600±10.800 | 385.900±22.000 | 85.600±4.600 | 156.700±8.600 | 372.400±21.900 |
| Zn (mg/kg) | 17.200±2.300 | 5.340±0.230 | 3.520±0.100 | 10.400±1.120 | 3.950±0.160 | 2.240±0.070 |
| Mn (mg/kg) | 74.820±8.370 | 8.630±0.210 | 2.110±0.050 | 140.000±9.310 | 64.380±5.100 | 23.410±1.310 |
| Fe (mg/kg) | 666.000±38.700 | 206.000±8.300 | 122.000±10.900 | 664.000±40.200 | 412.000±30.800 | 248.000±21.300 |
| Cu (mg/kg) | 21.100±6.120 | 7.700±0.060 | 17.800±1.530 | 28.200±2.310 | 19.600±1.590 | 11.000±0.950 |
利用qPCR技术,采用SYBR GREEN法对旱季和雨季采集的YN、SD和NT采样区的6个土壤样品真菌数量进行定量分析,结果见表 3。从表 3中可以看出,在NT采样区土壤样品中,真菌数量在雨季约为旱季的2倍;在YN和SD采样区土壤样品中,真菌数量在雨季和旱季基本无差异。
| 土壤样品 Soil samples |
真菌数量 Fungal numbers (copies/g) |
| DS.YN | 1.07×104 |
| DS.SD | 3.15×104 |
| DS.NT | 3.28×104 |
| RS.YN | 1.36×104 |
| RS.SD | 3.76×104 |
| RS.NT | 6.32×104 |
对纳帕海高原湿地6个土壤样品(DS.YN、DS.SD、DS.NT、RS.YN、RS.SD和RS.NT)扩增ITS2基因。PCR产物通过MiSeq高通量测序,共获得2 303 134条初始序列。对每个样品序列相似性聚类进行标准化后,基于97%序列相似性,计算OTU。旱季共得到2 669 OTU,其中DS.NT 944 OTU、DS.SD 877 OTU和DS.YN 848 OTU;雨季共得到2 941 OTU,其中RS.NT 756 OTU、RS.SD 1203 OTU和RS.YN 982 OTU。两季真菌群落均表现出较高的多样性,雨季土壤样品中的分类学信息比旱季土壤样品中的分类学信息丰富。
通过统计土壤真菌群落的OTU所含序列数,绘制真菌群落Rank abundance曲线(图 2)。结果表明,随着OTU所含序列数的增多,土壤样品中真菌群落丰度变高且分布均匀。雨季较旱季相比,曲线平缓下降,表明雨季土壤样品中真菌群落多样性更高。
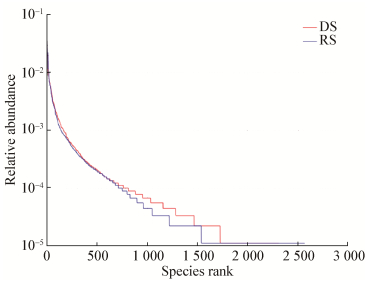
|
| 图 2 基于97%序列相似性真菌群落测序等级曲线 Figure 2 Rank abundance curves of fungal community for soil samples at the 97% similarity level in dry and rain seasons |
|
|
在采集的纳帕海高原湿地土壤样品中,共检测到有确定分类信息的真菌群落有6个门,主要包括Ascomycota、Basidiomycota和Chytridiomycota;17个纲,主要包括Sordariomycetes、Leotiomycetes、Agaricomycetes、Dothideomycetes和Chytridiomycetes;37个目,主要包括Hypocreales、Helotiales、Pleosporales、Rhizophydiales、Agaricales和Xylariales;53个科,主要包括Nectriaceae、Plectosphaerellaceae、Pleosporaceae、Clavicipitaceae和Helotiaceae;63个属,主要包括Gibberella、Phoma、Plectosphaerella和Myrothecium。在分类单元的门到属的变化过程中,真菌群落未确定分类信息始终在60%以上(其门和属水平的分布见图 3)。由于在真菌群落中有正式描述的只有不到10%,绝大多数都属于未知的,因此未确定的分类信息高。
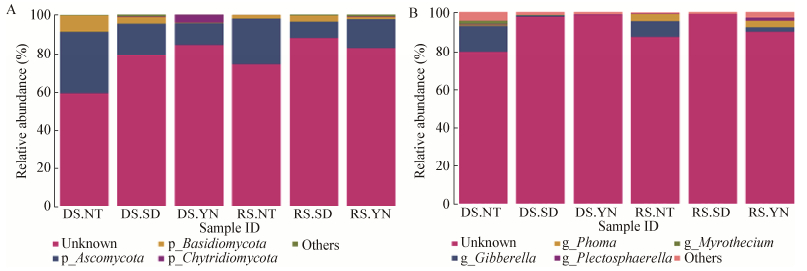
|
| 图 3 真菌群落组成及其相对丰度 Figure 3 Composition and relative abundance of fungal community in soil samples 注:A:真菌群落组成及相对丰度门水平;B:真菌群落相对丰度属水平. Note: A: Composition and relative abundance of fungal at phyla level; B: Composition and relative abundance of fungal at family level. |
|
|
对土壤样品中真菌群落所含序列的前50 OTU进行双向二维聚类,根据聚类后土壤样品中不同的OTU所包含序列的丰度,绘制Heatmap图(图 4),从而直观反映纳帕海高原湿地中高丰度真菌群落组成的相似性和差异性。从图 4可看出,YN、SD和NT采样区土壤样品中真菌群落组成无论旱季还是雨季,均各自聚为一簇,NT与SD聚为一大簇,表明真菌群落组成的变化受时间影响较小,具有空间性。

|
| 图 4 真菌群落组成的Heatmap图 Figure 4 The Heatmap diagram of fungal community of soil samples in dry and rain seasons |
|
|
利用R语言Venn Diagram程序包,根据旱季和雨季YN、SD和NT采样区土壤样品中真菌群落的OTU数目,绘制Venn图(图 5),从而间接反映纳帕海高原湿地中真菌群落组成的相似性及重叠情况。从图 5可以看出,在旱季,YN、SD和NT采样区土壤样品中共有59个OTU相同,YN和SD有61个OTU相同,YN和NT有43个OTU相同,SD和NT有148个OTU相同;在雨季,YN、SD和NT采样区土壤样品共有61个OTU相同,YN和SD有79个OTU相同,YN和NT有54个OTU相同,SD和NT有110个OTU相同。结果表明,SD和NT采样区土壤样品中真菌群落组成差异性较小,与相对高丰度(序列前50的OTU) Heatmap图表征具有一致性趋势。
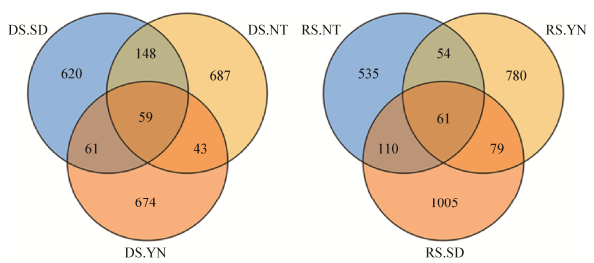
|
| 图 5 真菌群落组成的Venn图 Figure 5 The venn diagram of fungal community of soil samples in dry and rain seasons |
|
|
利用Weightes_UniFrac距离,进行PCoA分析,从而进一步评估对于纳帕海高原湿地时间和空间整体真菌群落组成的相似性和差异性(图 6)。由图 6可看出,在SD和NT采样区土壤样品中,无论旱季和雨季均独立处于同一象限,且真菌群落组成在SD和NT中与YN中相比组成差异性较小。
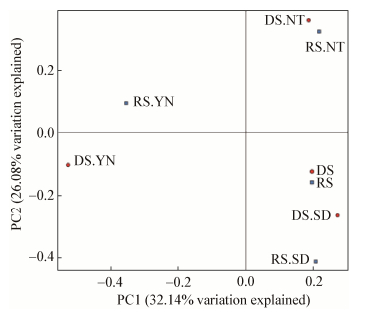
|
| 图 6 真菌群落组成的PCoA分析 Figure 6 The PCoA analysis of fungal community of soil samples in dry and rain seasons |
|
|
通过分析SOM、TN、TP、TK、N、P、K、Zn、Mn、Fe、Mg和Cu等环境因子对真菌群落多样性影响的结果表明(图 7),土壤样品中真菌群落多样性基本不受TN和N的影响。SD聚为同一个象限,基本不受环境因子的影响;YN聚为同一个象限,只受TP元素的影响;NT聚为同一个象限,受SOM和微量元素的影响。
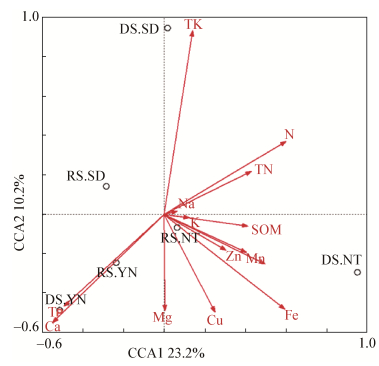
|
| 图 7 真菌群落多样性与环境因子的关系 Figure 7 The relationship between the fungal community diversity and environmental factors of soil samples in dry and rain seasons |
|
|
本研究中利用qPCR检测了旱季(DS)和雨季(RS)中YN、SD和NT采样区中真菌的数量。就不同土壤类型中而言,真菌数量在NT采样区最高,在YN采样区最低。通过对采样区的现场观察和对土壤理化因子的分析可以看出,从YN到SD到NT采样区土壤类型的变化,可视为纳帕海高原湿地人为活动干扰下逐渐退化的原生沼泽土、沼泽草甸土和草甸土3种湿地土壤类型[13]。YN采样区处于半厌氧条件下,土壤样品中SOM和TN含量最高,但由于厌氧条件对好氧真菌生存繁殖不利,因此好氧菌的数量偏低,从而导致真菌总数量略低;SD采样区处于沼泽化草甸和草甸过渡中,人为活动干扰加剧,有机养分分解加速,土壤样品中SOM和TN含量最低,但由于土壤pH适宜、通透性增加等,真菌数量反而变高;NT采样区处于畜牧和施肥等农业生产活动区,人为活动干扰最为严重,然而由于其承接了随地表径流汇集的养分和农业生产的有机肥,土壤样品中SOM和TN含量增多,对真菌生长最为有利,因此数量最高。由此可以看出,真菌的数量对于人为干扰下的湿地土壤退化是个更灵敏的响应指标。通过对不同土壤类型下真菌数量的变化规律的总结,初步阐明在纳帕海湿地中真菌数量变化与土壤退化程度直接相关。
在真菌群落的研究中,我们发现Ascomyeotat为纳帕海高原湿地的优势门,与前期人们发现Ascomyeotat为淡水中的优势门的结论一致[14]。纳帕海高原湿地水量补给主要依靠降雨、河流和冰雪融水等,属于典型的人工调控的高原淡水季节性湖泊湿地,因此Ascomyeotat成为优势菌门。由于植物的根际效应对真菌群落的多样性变化有主导作用,因此即使相同的生态环境下,由于植被种类的不同,从而造成优势种属的差别[15]。Yang等发现,在黄土高原土壤中由于植被的恢复,从以Basidiomycota为主导的群落逐渐过渡到Ascomycota[15]。在青藏高原对土壤真菌多样性的研究中同样发现,真菌多样性与植被多样性具有密切关系[16]。在本研究中,虽然同样检测到了上述的优势菌群,但纳帕海高原湿地真菌群落的相对优势属为Gibberella。在纳帕海高原湿地人为活动干扰最多的NT采样区、人为活动干扰较少的SD采样区和几乎没有人为活动干扰的YN采样区,Gibberella含量分别为13%、0.4%和0.04%。由此可以看出,Gibberella在纳帕海高原湿地不同采样区的含量与人为活动干扰有直接相关性,很可能为外来侵入物种。然而Gibberella大多为具有破坏性的植物病原真菌,其感染可导致部分植物出现生长缓慢或者死亡,而对该病原微生物不敏感的植物将大量生长,逐步取代原有的优势种,导致植物物种多样性降低,引起植物群落改变[17]。Gibberella在纳帕海高原湿地的出现会对该地区植物群落带来哪些影响?又如何防止Gibberella可能导致的湿地植物多样性减少?已成为摆在我们面前一个亟待解决的问题。
通过对真菌群落从分类水平、OTU水平及β多样性分析的结果均表明,在纳帕海高原湿地整体的真菌群落多样性和组成随时间(季节)变化不显著,而随空间(不同土壤类型)的变化真菌群落组成呈现出差异性,与我国三江湿地土壤真菌的多样性受季节的显著影响不同[18]。三江平原湿地季节性变化主要是从植被生长期和凋落期为依据划分的,由于植物的根际效应和种类对真菌群落的多样性变化有主导作用,因此真菌群落多样性和组成在不同的生态环境中季节性变化不同[19]。而纳帕海高原湿地季节划分按照降水量的不同而划分,旱季和雨季温差较小,对于同样土壤类型而言,植被种类及数量变化情况不明显,生长趋于平稳,凋落物及根系分泌物维持在一个相应的水平上,因此真菌群落多样性季节差异不显著。
| [1] |
Masto RE, Ahirwar R, George J, et al. Soil biological and biochemical responses to Cd exposure[J]. Open Journal of Soil Science, 2011, 1(1): 8-15. |
| [2] |
Rillig MC, Mummey DL. Mycorrhizas and soil structure[J]. The New Phytologist, 2006, 171(1): 41-53. DOI:10.1111/nph.2006.171.issue-1 |
| [3] |
Finlay RD. Ecological aspects of mycorrhizal symbiosis: with special emphasis on the functional diversity of interactions involving the extraradical mycelium[J]. Journal of Experimental Botany, 2008, 59(5): 1115-1126. DOI:10.1093/jxb/ern059 |
| [4] |
Liu KH, Ding XW, Wang HF, et al. Eukaryotic microbial communities in hypersaline soils and sediments from the alkaline hypersaline Huama Lake as revealed by 454 pyrosequencing[J]. Antonie van Leeuwenhoek, 2014, 105(5): 871-880. DOI:10.1007/s10482-014-0141-4 |
| [5] |
Yang Y, Dou YX, Huang YM, et al. Links between soil fungal diversity and plant and soil properties on the Loess Plateau[J]. Frontiers in Microbiology, 2017, 8: 2198. DOI:10.3389/fmicb.2017.02198 |
| [6] |
Leblond-Bourget N, Philippe H, Mangin I, et al. 16S rRNA and 16S to 23S internal transcribed spacer sequence analyses reveal inter- and intraspecific Bifidobacterium phylogeny[J]. International Journal of Systematic Bacteriology, 1996, 46(1): 102-111. DOI:10.1099/00207713-46-1-102 |
| [7] |
Tian K, Guo HJ, Yang YM, et al. Ecological Structure Characteristics of Plateau Wetland Nature Reserve and Functional Zoning Research and Practice[M]. Beijing: Science Press, 2009. (in Chinese) 田昆, 郭辉军, 杨宇明, 等. 高原湿地保护区生态结构特征及功能分区研究与实践[M]. 北京: 科学出版社, 2009. |
| [8] |
Martin KJ, Rygiewicz PT. Fungal-specific PCR primers developed for analysis of the ITS region of environmental DNA extracts[J]. BMC Microbiology, 2005, 5: 28. DOI:10.1186/1471-2180-5-28 |
| [9] |
Gade L, Scheel CM, Pham CD, et al. Detection of fungal DNA in human body fluids and tissues during a multistate outbreak of fungal meningitis and other infections[J]. Eukaryotic Cell, 2013, 12(5): 677-683. DOI:10.1128/EC.00046-13 |
| [10] |
Schloss PD, Westcott SL, Ryabin T, et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [11] |
Reeder J, Knight R. Rapidly denoising pyrosequencing amplicon reads by exploiting rank-abundance distributions[J]. Nature Methods, 2010, 7(9): 668-669. |
| [12] |
Oksanen J. Multivariate analysis of ecological communities in R: vegan tutorial[Z]. R Package Version, 2007, 1: 1-39
|
| [13] |
Tian K, Lu M, Chang FL, et al. The ecological environment degradation and degradation mechanism of Napahai karst wetland in southwestern Yunnan plateau[J]. Journal of Lake Sciences, 2004, 16(1): 35-42. (in Chinese) 田昆, 陆梅, 常凤来, 等. 云南纳帕海岩溶湿地生态环境变化及驱动机制[J]. 湖泊科学, 2004, 16(1): 35-42. DOI:10.3321/j.issn:1003-5427.2004.01.005 |
| [14] |
Vijaykrishna D, Jeewon R, Hyde KD. Molecular taxonomy, origins and evolution of freshwater ascomycetes[J]. Fungal Diversity, 2006, 23: 351-390. |
| [15] |
Costa R, Götz M, Mrotzek N, et al. Effects of site and plant species on rhizosphere community structure as revealed by molecular analysis of microbial guilds[J]. FEMS Microbiology Ecology, 2006, 56(2): 236-249. DOI:10.1111/fem.2006.56.issue-2 |
| [16] |
Yang T, Adams JM, Shi Y, et al. Soil fungal diversity in natural grasslands of the Tibetan Plateau: associations with plant diversity and productivity[J]. New Phytologist, 2017, 215(2): 756-765. DOI:10.1111/nph.2017.215.issue-2 |
| [17] |
Jurgenson JE, Zeller KA, Leslie JF. Expanded genetic map of Gibberella moniliformis (Fusarium verticillioides)[J]. Applied and Environmental Microbiology, 2002, 68(4): 1972-1979. DOI:10.1128/AEM.68.4.1972-1979.2002 |
| [18] |
Chen HH, Xu XH, Li HT. Diversity analysis of soil fungal community structure in Sanjiang wetland by PCR-DGGE[J]. Research of Environmental Sciences, 2012, 25(11): 1272-1278. (in Chinese) 陈会海, 许修宏, 李洪涛. 应用PCR-DGGE分析三江湿地土壤真菌群落结构的多样性[J]. 环境科学研究, 2012, 25(11): 1272-1278. |
| [19] |
Pace NR. A molecular view of microbial diversity and the biosphere[J]. Science, 1997, 276(5313): 734-740. DOI:10.1126/science.276.5313.734 |
 2019, Vol. 46
2019, Vol. 46




