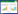扩展功能
文章信息
- 刘涛, 王晓婕, 李英俊, 彭楠
- LIU Tao, WANG Xiao-Jie, LI Ying-Jun, PENG Nan
- 冰岛硫化叶菌遗传操作体系的研究进展
- Systems for the genetic manipulation of Sulfolobus islandicus
- 微生物学通报, 2019, 46(2): 398-406
- Microbiology China, 2019, 46(2): 398-406
- DOI: 10.13344/j.microbiol.china.180957
-
文章历史
- 收稿日期: 2018-11-26
- 接受日期: 2019-01-09
- 网络首发日期: 2019-01-09
冰岛硫化叶菌(Sulfolobus islandicus)是古菌界泉古菌门的一种嗜酸嗜热微生物[1-2],因其易于培养且富含病毒和质粒等遗传因子而受到广泛的研究[3-5]。该菌最先发现和分离的株系来自冰岛的热泉[6],随后人们对冰岛硫化叶菌多个株系进行了全基因组测序[7-8],并鉴定到很多病毒和质粒等遗传元件[9-10]。研究人员在此基础上成功构建了适用于冰岛硫化叶菌的载体和高效的转化方法,实现了多种策略的基因敲除。近年来冰岛硫化叶菌CRISPR-Cas系统被开发成一种高效的基因编辑工具[11],使其遗传操作体系得到了补充和完善。本文详细介绍了冰岛硫化叶菌遗传操作体系的基本元素,分析了现有基因编辑方法的优缺点,为选择合适的方法对冰岛硫化叶菌进行遗传改造提供一定的指导。
1 冰岛硫化叶菌遗传操作体系的基本元素 1.1 筛选标记冰岛硫化叶菌中常用的筛选标记包括pyrEF和lacS基因。特殊的细胞结构和极端高温酸性培养条件使大部分抗生素基因都不适用于冰岛硫化叶菌,虽然有研究报道潮霉素抗生素基因hph可以在硫化叶菌中使用[12],但是因其难以重复而没有被推广使用。在没有合适抗生素基因的前提下,研究者们尝试使用营养缺陷型作为筛选标记。
pyrEF基因是硫化叶菌中运用最广泛的一个营养缺陷型筛选标记,它包含pyrE (Orotate phosphoribosyl-transferase)和pyrF (Orotidine 5′-phosphate decarboxylase)两个基因。两个基因共同参与UMP合成途径[13],可以作为一种正筛选标记使细胞在缺少尿嘧啶的培养基中生长。当培养基中添加5-氟乳清酸(5-FOA)时,pyrEF基因编码的酶也能将无毒的5-FOA变成毒性物质5-氟-dUMP从而杀死细胞,因此又是一种负筛选标记。Deng等在冰岛硫化叶菌Rey15A菌株中筛选5-FOA抗性菌株,从而获得了pyrEF基因缺失突变体E233。然后通过PCR扩增硫磺矿硫化叶菌P2 (Sulfolobus solfataricus P2)的pyrEF基因预测序列(启动子、ORF和终止子)作为筛选标记,为pyrEF筛选标记在冰岛硫化叶菌遗传体系中的应用开创了道路[14]。值得注意的是,使用pyrEF基因作为筛选标记时会因为培养基中含有的微量尿嘧啶而产生非常小的背景菌落[15-16],所以使用该筛选标记时需要控制菌体量及培养时间。
pyrEF基因并不能完全满足遗传操作的需求,所以研究人员尝试使用lacS基因作为另一种筛选标记。在冰岛硫化叶菌中lacS基因编码β-糖苷酶(β-glycosidase),能使菌株在以乳糖为唯一碳源和能源的培养基中生长[17],也可以将X-gal催化生成深蓝色物质。因此可以利用lacS基因在乳糖限制性培养基中进行筛选,或者使用X-gal染色进行筛选。Deng等在E233菌株中以pyrEF基因作为筛选标记成功敲除lacS基因,获得pyrEF和lacS双基因缺失突变体E233S,并将来源于S. solfataricus P2的lacS基因克隆到pHZ2质粒进行互补验证,确定lacS基因可以作为一种有效的筛选标记[14]。由于冰岛硫化叶菌培养周期相对较长,所以使用lacS基因作为筛选标记并进行X-gal染色时,需要固体培养基上形成可分辨的转化子后再进行染色。
1.2 穿梭质粒最初的冰岛硫化叶菌-大肠杆菌穿梭质粒是基于从S. islandicus REN1H1中分离得到的pRN1和pRN2质粒[18-19]。这两个质粒都含有repA、copG和plrA三个保守基因,repA编码复制蛋白[20-21],copG编码可能参与调节质粒拷贝数的DNA结合蛋白[22],而plrA编码一类序列特异性DNA结合蛋白[23]。Berkner等以pRN1质粒为基础构建了含有pyrEF筛选标记的穿梭质粒,可以成功进行基因互补[24]。但是该质粒转化困难且并不稳定,所以并没有被广泛使用。pRN2比pRN1的拷贝数高,所以更适合改造为穿梭质粒。Deng等将pRN2质粒插入到pHZ1质粒(含有pyrEF基因的pGEM-3Z质粒)中得到pHZ2[14],Peng等基于对pRN2质粒的遗传分析,去除了pHZ2的一些非必需序列并加上pyrEF筛选标记得到pZC1质粒[25],再去除pZC1质粒上的Pvu Ⅱ、Nde Ⅰ和Nde Ⅰ三个多余的酶切位点得到pDL1,在pDL1上插入阿拉伯糖启动子、两个His标签和多克隆位点得到pEXA,最终使用含有SD序列的阿拉伯糖启动子替换原始的阿拉伯糖启动子得到pSeSD质粒[26]。现在pSeSD是冰岛硫化叶菌中使用最广泛的一个冰岛硫化叶菌-大肠杆菌穿梭质粒(图 1),该质粒转化效率高达104-106 CFU/μg DNA且具有较好的稳定性,利用该质粒可以进行启动子分析、蛋白表达、遗传互补和基因编辑等一系列研究。
|
| 图 1 冰岛硫化叶菌-大肠杆菌穿梭质粒pSeSD Figure 1 Sulfolobus islandicus-Escherichia coli shuttle vector pSeSD 注:pSeSD质粒图谱,包含来源于pRN2的3个保守基因repA、copG和plrA,阿拉伯糖启动子ParaS,多克隆位点MCS,冰岛硫化叶菌筛选标记pyrEF,大肠杆菌筛选标记Ampr. Note: Plasmid map of pSeSD, which contains three conserved genes of repA, copG and plrA from pRN2, an arabinose-inducible promoter, multiple cloning site, pyrEF selection marker of Sulfolobus islandicus and Ampr selection marker of Escherichia coli. |
|
|
在冰岛硫化叶菌中普遍使用的唯一高效的转化方法是电转化法(Electroporation)。Schleper等在硫化叶菌中最早使用电转化法成功将硫化叶菌病毒SSV1的DNA导入S. solfataricus P1中[27],随后该方法在多种硫化叶菌中推广使用并被不断改进。目前在冰岛硫化叶菌中使用的电转参数为:1 200 V,600 Ω,25 μF,1 mm电转杯,电击后需加入预热至75 ℃的孵育培养基,并于75 ℃条件下静置孵育2-3 h。电转参数的设置并不是绝对的,最早的电转参数为1 500 V和800 Ω,Albers等采用2 mm电转杯进行转化也得到了很高的转化效率[28],Aucelli等在转化时连续电击两次也能取得较好的转化结果[29],因此在一个较广的范围内改变电转参数都可以成功转化,可以针对不同的DNA进行一定的参数优化以取得理想的转化效率,同时孵育时间的长短也会影响最终的转化效率。
2 冰岛硫化叶菌遗传操作体系的应用 2.1 基于常规同源重组的基因敲除体系基因敲除策略是遗传操作体系的重要组成部分,冰岛硫化叶菌最初的基因敲除策略是基于同源重组的原理构建的。
“质粒整合分离”(Plasmid integration and segregation,PIS)是在很多古菌中已经成功运用的最简单的一种敲除策略。Deng等最先在冰岛硫化叶菌中尝试使用该方法敲除lacS基因[14],通过在以pyrEF基因作为筛选标记的pHZ1质粒上克隆目的基因的左臂和右臂得到pPIS敲除质粒。该质粒以环状的形式转入冰岛硫化叶菌后,质粒上的左臂或右臂与基因组上对应位置发生一次同源单交换将pyrEF、左臂和右臂插入基因组。然后使用5-FOA进行负筛选促使基因组上两个左臂或两个右臂发生同源重组,从而使pyrEF单独环出或者pyrEF和目的基因同时环出,理论上两种环出方式的概率相同,因此最终得到的转化子中敲除菌株和野生型各占50%。敲除质粒的构建及策略如图 2所示,因为此方法转化效率较低,只能得到很少的几个转化子,所以现在很少使用该方法进行基因敲除。

|
| 图 2 质粒整合分离法 Figure 2 The method of plasmid integration and segregation 注:质粒pPIS含有筛选标记pyrEF和用于单交换的左臂和右臂,第一次单交换将pyrEF,左臂和右臂插入基因组,在筛选压力下发生第二次单交换得到突变体和野生型. Note: The plasmid pPIS contains pyrEF selection marker and the left-arm and right-arm for single cross over, pyrEF, left-arm and right-arm are inserted into genome by the first single cross over, and then the second single cross over results in both mutant and wild type under the pressure of selection. |
|
|
“标记替换及环出法”(Marker replacement and looping out,MRL)是对PIS策略进行优化而发展出的一种敲除策略。Deng等最先利用该策略在冰岛硫化叶菌中敲除lacS基因。pMRL敲除质粒经过线性化后转入细胞,左臂和右臂与基因组发生一次同源双交换将目的基因替换为pyrEF筛选标记和一个左臂,再使用5-FOA进行负筛选促使2个左臂发生同源重组将pyrEF和一个左臂环出。敲除质粒的构建及策略如图 3所示,该方法得到的转化子理论上都是敲除菌株,并且转化效率可以达到102 CFU/μg DNA[14]。但是因为第一步同源双交换会破坏基因组上目的基因,因此使用此方法需注意敲除片段不影响相邻基因的转录与表达。

|
| 图 3 标记替换及环出法 Figure 3 The method of marker replacement and looping out 注:在pPIS上增加一个左臂得到pMRL质粒,质粒线性化后经同源双交换将pyrEF和左臂与目标基因进行替换,在筛选压力下两个左臂单交换得到突变体. Note: The plasmid pMRL is the pPIS with another added left-arm and is linearized, then pyrEF selection marker and the left-arm replace the target gene by double cross over, and the single cross over with two left-arm results in mutant under the pressure of selection. |
|
|
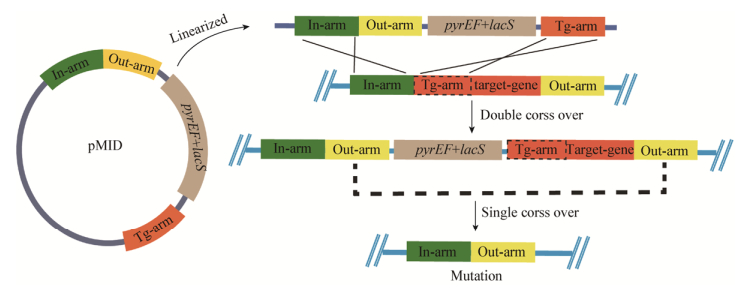
然而Zhang等用以上两种敲除策略进行基因敲除时遇到了一些问题,使用质粒整合分离的方法敲除增殖细胞核抗原基因(Proliferating cell nuclear antigen,pcna)时,多次重复转化都没有得到任何转化子,而使用标记替换及环出法可以得到转化子,但最终验证发现都是野生型。因此他推测在敲除某些与DNA复制、修复和细胞生长相关基因时,这两种方法都不适用。因此Zhang等对标记替换及环出法进行改进,建立了一种新的敲除策略“选择标记插入/无选择标记的基因缺失法”(Marker insertion and unmarkerd target gene deletion,MID),该方法将标记替换及环出法中的一个同源臂变为基因臂Tg-arm,两个同源臂也变为将标记基因交换到基因组上的In-arm和将标记基因Marker及目的基因环出的Out-arm。In-arm和Tg-arm与基因组发生同源双交换将标记基因和Out-arm插入基因组,用不含尿嘧啶的培养基和X-gal染色两种方法筛选正确转化子。然后再用含有尿嘧啶和5-FOA的平板进行负筛选,基因组上两个Out-arm发生单交换将筛选标记和目的基因环出从而得到缺失突变菌株(图 4)。该方法在第一步双交换时不会破坏目的基因,第二步筛选时如果目的基因是必需基因则得不到正确转化子,即使有少量转化子也是发生回复突变或筛选标记自发突变的转化子,所以可以使用该方法鉴定基因的必需性[30-31]。使用此方法需要注意的是要确保第一步同源双交换以后目的基因及其两侧基因可以正常表达,同时要尽可能减少In-arm和Tg-arm之间的重复序列以避免其发生重组。
|
| 图 4 选择标记插入/无选择标记的基因缺失法 Figure 4 The method of marker insertion and unmarkerd target gene deletion 注:pMID质粒包含pyrEF和lacS双筛选标记,In-arm,Out-arm和Tg-arm,线性化后通过同源双交换将筛选标记和Out-arm插入In-arm与目标基因之间,然后在筛选压力下两个Out-arm单交换得到突变体. Note: The plasmid pMID contains pyrEF and lacS double selection marker, In-arm, Out-arm and Tg-arm. After it is linearized, selection marker and Out-arm insert between In-arm and the target gene in the genome by double cross over, and the single cross over with two Out-arms results in mutant under the pressure of selection. |
|
|
CRISPR-Cas系统是原核生物的一种获得性免疫系统[32],近年来被开发为一种新的基因编辑方法,在动植物基因编辑等方面的研究中展现了独特的科学魅力[33-35]。冰岛硫化叶菌含有一个Ⅰ-A型和两个Ⅲ-B型CRISPR-Cas系统,迄今为止关于其核酸干涉机制的研究已经较为清楚。Gudbergsdottir等使用含有CRISPR簇中的间隔序列的质粒进行转化实验证实冰岛硫化叶菌Ⅰ-A型系统是依赖于识别“CCN”的PAM (Protospacer adjacent motif)序列发挥DNA干涉活性[36]。Deng等将含有不能被Ⅰ-A型系统干涉的间隔序列的质粒转化冰岛硫化叶菌,证实Ⅲ-B型Cmr-α具有依赖于转录的DNA干涉活性[37]。Peng等分别敲除Ⅲ-B型系统中Cmr-α和Cmr-β的相关基因,再用干涉质粒和报告基因两种方法证实Cmr-α和Cmr-β都具有RNA干涉活性[38]。
冰岛硫化叶菌Ⅲ-B型系统的RNA干涉活性最先被开发应用为一种基因沉默的工具,为研究必需基因的功能开创了新的思路和方法。Peng等在pSeSD质粒的多克隆位点插入被2个BspM Ⅰ内切酶隔开的2个CRISPR簇重复序列(Repeat),得到原始基因沉默质粒pSe-Rp。然后针对需要沉默的基因在其编码链上选取5′-GAAAG-3′或5′-CAGAG-3′后面40 bp作为Spacer,该Spacer转录加工得到的crRNA的5′端可以与编码链的5′-GAAAG-3′或5′-CAGAG-3′形成互补配对以避免Ⅲ-B型系统依赖于转录的DNA干涉活性,又因缺少有活性的PAM序列可以避免Ⅰ-A型系统依赖于PAM的DNA干涉活性,但该Spacer仍具有RNA干涉活性。设计引物时在编码链的引物5′端加上5′-TAGC-3′,在非编码链的引物5′端加上5′-AAAG-3′,2条引物直接经过退火形成带5′端突出的双链短片段,短片段与BspM Ⅰ酶切以后的质粒通过酶连插入两个Repeat之间形成mini-CRISPR,最终得到人工干涉质粒pAC。pAC上mini-CRISPR转录的crRNA可以介导Ⅲ型核酸干涉符合物特异性切割目的基因转录的mRNA,从而降低目的基因的表达水平(图 5)。Spacer在基因上的位置会影响基因沉默的效率,同时Spacer上的25–28位和29–32位碱基突变也会大大降低RNA干涉活性,mRNA水平与蛋白质表达水平也不一定呈正相关,因此还需要通过RT-qPCR和Western blot对基因沉默的水平进行验证和定量[38]。

|
| 图 5 基于CRISPR-Cas系统的基因沉默方法 Figure 5 Method of gene silencing based on CRISPR-Cas system |
|
|
基于以上的研究结果,Li等[11]开发了一种基于冰岛硫化叶菌Ⅰ-A型和Ⅲ-B型CRISPR-Cas系统的基因编辑技术。该方法在基因沉默质粒pSe-Rp基础上用相同方法克隆具有DNA干涉活性的Spacer到2个Repeat之间组成mini-CRISPR,再将经SOE得到的供体DNA (Donor DNA)用Sal Ⅰ和Not Ⅰ两个酶切位点克隆到该质粒上,最终得到目的基因编辑质粒pGE。pGE上mini-CRISPR转录的crRNA可以介导CRISPR-Cas核酸干涉复合物特异性切割目的基因,供体DNA上可以设计缺失、插入和点突变,用于与基因组上编辑位点发生同源重组从而使细胞存活(图 6)。该方法质粒构建十分简便,而且转化效率可以高达102-103 CFU/μg DNA。与基因沉默不同的是Spacer需要“CCN”或“TCN”的PAM序列才能发挥Ⅰ-A型系统DNA干涉活性,而Ⅲ-B型系统需要Spacer位于编码链,且与Spacer互补的非编码链的mRNA的3′端序列与mini-CRISPR转录的crRNA的5′端序列错配。在cmr2α上开展的点突变实验证实在10 bp左右的DNA片段间可以发生同源重组,所以尤其需要注意的是,设计点突变时突变位点应尽量选取在Spacer上或者靠近Spacer的位置以确保同源重组时突变位点可以重组到基因组上,另外利用Ⅰ-A型DNA干涉活性需要将PAM进行同义突变,利用Ⅲ-B型DNA干涉活性需要将Spacer的3′端序列进行同义突变,以保护突变以后的序列不会再次被编辑质粒干涉[11]。

|
| 图 6 基于CRISPR-Cas系统的基因编辑方法 Figure 6 Method of gene editing based on CRISPR-Cas system |
|
|
冰岛硫化叶菌作为嗜酸热古菌的一种模式菌株,研究者们越来越关注其DNA复制及修复[39]、CRISPR-Cas系统[40-46]和蛋白修饰[47]等机制的研究,方便成熟的遗传操作体系将对这些研究起巨大推动作用。得益于对冰岛硫化叶菌分子机制的研究,其遗传操作系统日趋完善和便捷,但是也待进一步改进和优化。
本文介绍了目前在冰岛硫化叶菌中取得成功应用的几种基因操作策略。PIS法实验设计最简单,质粒构建也最简便,但是此方法转化效率最低,负筛选得到含有野生型和敲除菌株的混合转化子,因此具有很大的不确定性。MRL法在PIS的基础上进行优化,一定程度提高了转化效率,且最终负筛选得到的都是敲除菌株,但是因为第一步同源双交换破坏了原始基因,所以当得不到任何转化子时无法确定该基因是否为必需基因还是实验设计及操作存在问题。MID法是MRL的改进方法,克服了之前方法的所有缺点,但是质粒构建较复杂,也存在质粒自身发生环化整合到宿主染色体上,进而在负筛选时环出得到野生型转化子的问题。基于CRISPR-Cas系统的基因编辑方法是目前效率最高、使用最多且操作最简便的策略,可以满足突变、缺失和插入等多种研究需求,但会出现编辑质粒成功转入但目的基因并未敲除的现象,出现此现象的原因可能是目的基因是必需基因或者CRISPR-Cas系统功能异常。此方法不适用于在CRISPR-Cas系统crRNA加工和干涉相关功能缺失突变菌株中进行基因编辑,也不适用于编辑基因组上的重复序列。另外,可能存在类似于Cas9系统的脱靶现象。综上所述,不同敲除策略有各自的特点与适用性,研究者需要根据实际情况和目的进行选择。
本文所述的遗传操作系统也同样适用于某些与冰岛硫化叶菌生存环境十分相似的,如:硫磺矿硫化叶菌(S. solfataricus)和噬酸热硫化叶菌(S. acidocaldarius)。研究者可以使用相同的筛选标记、质粒载体和转化方法,通过相同的基因敲除策略对这两种硫化叶菌进行遗传改造。但是对于东工大硫化叶菌(S. tokodaii),人们目前只对其进行了全基因组测序和分析,并没有建立十分稳定有效的遗传操作体系。
鉴于现有冰岛硫化叶菌遗传操作体系存在的一些问题,未来可以从以下几个方向对其进行补充和完善:(1)开发更多适用于冰岛硫化叶菌的筛选标记,如氨基酸营养缺陷型标记。(2)开发多位点编辑方法,目前基于CRISPR-Cas系统的基因编辑方法只含有一个mini-CRISPR,所以只能对单一位点进行编辑,可以构建含有多个Spacer的mini-CRISPR并在质粒上引入多个供体DNA,从而同时对多个基因进行编辑。(3)开发全基因组范围的基因调控技术。Konermann等将CRISPR-Cas9系统开发成一种全基因组水平转录激活的新方法[48],对于这方面工作冰岛硫化叶菌内源CRISPR-Cas系统具有独特的优势,只需将核酸干涉蛋白进行一定改造,就可以利用其CRISPR-Cas系统免疫适应获取的间隔序列完成全基因组水平的基因表达调控。基于内源CRISPR-Cas系统的冰岛硫化叶菌遗传操作体系已经成为当下主流的基因编辑方法,在该方法基础上进行不断优化将大大提高对冰岛硫化叶菌遗传改造的效率和准确性。
| [1] |
Woese CR, Kandler O, Wheelis ML. Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya[J]. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(12): 4576-4579. DOI:10.1073/pnas.87.12.4576 |
| [2] |
Brock TD, Brock KM, Belly RT, et al. Sulfolobus: a new genus of sulfur-oxidizing bacteria living at low pH and high temperature[J]. Archiv Fur Mikrobiologie, 1972, 84(1): 54-68. DOI:10.1007/BF00408082 |
| [3] |
Zillig W, Arnold HP, Holz I, et al. Genetic elements in the extremely thermophilic archaeon Sulfolobus[J]. Extremophiles, 1998, 2(3): 131-140. DOI:10.1007/s007920050052 |
| [4] |
Lipps G. Plasmids and viruses of the thermoacidophilic crenarchaeote Sulfolobus[J]. Extremophiles, 2006, 10(1): 17-28. DOI:10.1007/s00792-005-0492-x |
| [5] |
Prangishvili D, Stedman K, Zillig W. Viruses of the extremely thermophilic archaeon Sulfolobus[J]. Trends in Microbiology, 2001, 9(1): 39-43. |
| [6] |
Whitaker RJ, Grogan DW, Taylor JW. Geographic barriers isolate endemic populations of hyperthermophilic archaea[J]. Science, 2003, 301(5635): 976-978. DOI:10.1126/science.1086909 |
| [7] |
Guo L, Brügger K, Liu C, et al. Genome analyses of icelandic strains of Sulfolobus islandicus, model organisms for genetic and virus-host interaction studies[J]. Journal of Bacteriology, 2011, 193(7): 1672-1680. DOI:10.1128/JB.01487-10 |
| [8] |
Reno ML, Held NL, Fields CJ, et al. Biogeography of the Sulfolobus islandicus pan-genome[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(21): 8605-8610. DOI:10.1073/pnas.0808945106 |
| [9] |
Redder P, Peng X, Brügger K, et al. Four newly isolated fuselloviruses from extreme geothermal environments reveal unusual morphologies and a possible interviral recombination mechanism[J]. Environmental Microbiology, 2009, 11(11): 2849-2862. |
| [10] |
Contursi P, Jensen S, Aucelli T, et al. Characterization of the Sulfolobus host-SSV2 virus interaction[J]. Extremophiles, 2006, 10(6): 615-627. DOI:10.1007/s00792-006-0017-2 |
| [11] |
Li YJ, Pan SF, Zhang Y, et al. Harnessing Type Ⅰ and Type Ⅲ CRISPR-Cas systems for genome editing[J]. Nucleic Acids Research, 2016, 44(4): e34. DOI:10.1093/nar/gkv1044 |
| [12] |
Cannio R, Contursi P, Rossi M, et al. An autonomously replicating transforming vector for Sulfolobus solfataricus[J]. Journal of Bacteriology, 1998, 180(12): 3237-3240. |
| [13] |
Thia-Toong TL, Roovers M, Durbecq V, et al. Genes of de novo pyrimidine biosynthesis from the hyperthermoacidophilic crenarchaeote Sulfolobus acidocaldarius: novel organization in a bipolar operon[J]. Journal of Bacteriology, 2002, 184(16): 4430-4441. DOI:10.1128/JB.184.16.4430-4441.2002 |
| [14] |
Deng L, Zhu HJ, Chen ZJ, et al. Unmarked gene deletion and host-vector system for the hyperthermophilic crenarchaeon Sulfolobus islandicus[J]. Extremophiles, 2009, 13(4): 735-746. DOI:10.1007/s00792-009-0254-2 |
| [15] |
Berkner S, Lipps G. Mutation and reversion frequencies of different Sulfolobus species and strains[J]. Extremophiles, 2008, 12(2): 263-270. DOI:10.1007/s00792-007-0125-7 |
| [16] |
Jonuscheit M, Martusewitsch E, Stedman KM, et al. A reporter gene system for the hyperthermophilic archaeon Sulfolobus solfataricus based on a selectable and integrative shuttle vector[J]. Molecular Microbiology, 2003, 48(5): 1241-1252. |
| [17] |
Grogan DW. Evidence that β-galactosidase of Sulfolobus solfataricus is only one of several activities of a thermostable β-D-glycosidase[J]. Applied and Environmental Microbiology, 1991, 57(6): 1644-1649. |
| [18] |
Keeling PJ, Klenk HP, Singh RK, et al. Sulfolobus islandicus plasmids pRN1 and pRN2 share distant but common evolutionary ancestry[J]. Extremophiles, 1998, 2(4): 391-393. DOI:10.1007/s007920050083 |
| [19] |
Purschke WG, Schäfer G. Independent replication of the plasmids pRN1 and pRN2 in the archaeon Sulfolobus islandicus[J]. FEMS Microbiology Letters, 2001, 200(1): 97-102. DOI:10.1111/fml.2001.200.issue-1 |
| [20] |
Lipps G. The replication protein of the Sulfolobus islandicus plasmid pRN1[J]. Biochemical Society Transactions, 2004, 32: 240-244. DOI:10.1042/bst0320240 |
| [21] |
Lipps G, Röther S, Hart C, et al. A novel type of replicative enzyme harbouring ATPase, primase and DNA polymerase activity[J]. The EMBO Journal, 2003, 22(10): 2516-2525. DOI:10.1093/emboj/cdg246 |
| [22] |
Lipps G, Stegert M, Krauss G. Thermostable and site-specific DNA binding of the gene product ORF56 from the Sulfolobus islandicus plasmid pRN1, a putative archael plasmid copy control protein[J]. Nucleic Acids Research, 2001, 29(4): 904-913. DOI:10.1093/nar/29.4.904 |
| [23] |
Lipps G, Ibanez P, Stroessenreuther T, et al. The protein ORF80 from the acidophilic and thermophilic archaeon Sulfolobus islandicus binds highly site-specifically to double-stranded DNA and represents a novel type of basic leucine zipper protein[J]. Nucleic Acids Research, 2001, 29(24): 4973-4982. DOI:10.1093/nar/29.24.4973 |
| [24] |
Berkner S, Grogan D, Albers SV, et al. Small multicopy, non-integrative shuttle vectors based on the plasmid pRN1 for Sulfolobus acidocaldarius and Sulfolobus solfataricus, model organisms of the (cren-)archaea[J]. Nucleic Acids Research, 2007, 35(12): e88. DOI:10.1093/nar/gkm449 |
| [25] |
Peng N, Xia Q, Chen ZJ, et al. An upstream activation element exerting differential transcriptional activation on an archaeal promoter[J]. Molecular Microbiology, 2009, 74(4): 928-939. DOI:10.1111/j.1365-2958.2009.06908.x |
| [26] |
Peng N, Deng L, Mei YX, et al. A synthetic arabinose-inducible promoter confers high levels of recombinant protein expression in hyperthermophilic archaeon Sulfolobus islandicus[J]. Applied and Environmental Microbiology, 2012, 78(16): 5630-5637. DOI:10.1128/AEM.00855-12 |
| [27] |
Schleper C, Kubo K, Zillig W. The particle SSV1 from the extremely thermophilic archaeon Sulfolobus is a virus: demonstration of infectivity and of transfection with viral DNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(16): 7645-7649. DOI:10.1073/pnas.89.16.7645 |
| [28] |
Albers SV, Driessen AJM. Conditions for gene disruption by homologous recombination of exogenous DNA into the Sulfolobus solfataricus genome[J]. Archaea, 2008, 2(3): 145-149. |
| [29] |
Aucelli T, Contursi P, Girfoglio M, et al. A spreadable, non-integrative and high copy number shuttle vector for Sulfolobus solfataricus based on the genetic element pSSVx from Sulfolobus islandicus[J]. Nucleic Acids Research, 2006, 34(17): e114. DOI:10.1093/nar/gkl615 |
| [30] |
Zhang CY, Tian B, Li SM, et al. Genetic manipulation in Sulfolobus islandicus and functional analysis of DNA repair genes[J]. Biochemical Society Transactions, 2013, 41(1): 405-410. |
| [31] |
Zhang CY, Guo L, Deng L, et al. Revealing the essentiality of multiple archaeal pcna genes using a mutant propagation assay based on an improved knockout method[J]. Microbiology, 2010, 156: 3386-3397. DOI:10.1099/mic.0.042523-0 |
| [32] |
Deveau H, Garneau JE, Moineau S. CRISPR/Cas system and its role in phage-bacteria interactions[J]. Annual Review of Microbiology, 2010, 64: 475-493. DOI:10.1146/annurev.micro.112408.134123 |
| [33] |
Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [34] |
Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [35] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [36] |
Gudbergsdottir S, Deng L, Chen ZJ, et al. Dynamic properties of the Sulfolobus CRISPR/Cas and CRISPR/Cmr systems when challenged with vector-borne viral and plasmid genes and protospacers[J]. Molecular Microbiology, 2011, 79(1): 35-49. |
| [37] |
Deng L, Garrett RA, Shah SA, et al. A novel interference mechanism by a type ⅢB CRISPR-Cmr module in Sulfolobus[J]. Molecular Microbiology, 2013, 87(5): 1088-1099. |
| [38] |
Peng WF, Feng MX, Feng X, et al. An archaeal CRISPR type Ⅲ-B system exhibiting distinctive RNA targeting features and mediating dual RNA and DNA interference[J]. Nucleic Acids Research, 2015, 43(1): 406-417. DOI:10.1093/nar/gku1302 |
| [39] |
Liu JF, Gao RX, Li CT, et al. Functional assignment of multiple ESCRT-Ⅲ homologs in cell division and budding in Sulfolobus islandicus[J]. Molecular Microbiology, 2017, 105(4): 540-553. |
| [40] |
Liu T, Liu ZZ, Ye Q, et al. Coupling transcriptional activation of CRISPR-Cas system and DNA repair genes by Csa3a in Sulfolobus islandicus[J]. Nucleic Acids Research, 2017, 45(15): 8978-8992. DOI:10.1093/nar/gkx612 |
| [41] |
Li YJ, Zhang Y, Lin JZ, et al. Cmr1 enables efficient RNA and DNA interference of a Ⅲ-B CRISPR-Cas system by binding to target RNA and crRNA[J]. Nucleic Acids Research, 2017, 45(19): 11305-11314. |
| [42] |
He F, Vestergaard G, Peng WF, et al. CRISPR-Cas type Ⅰ-A Cascade complex couples viral infection surveillance to host transcriptional regulation in the dependence of Csa3b[J]. Nucleic Acids Research, 2017, 45(4): 1902-1913. |
| [43] |
Han WY, Li YJ, Deng L, et al. A type Ⅲ-B CRISPR-Cas effector complex mediating massive target DNA destruction[J]. Nucleic Acids Research, 2017, 45(4): 1983-1993. |
| [44] |
León-Sobrino C, Kot WP, Garrett RA. Transcriptome changes in STSV2-infected Sulfolobus islandicus REY15A undergoing continuous CRISPR spacer acquisition[J]. Molecular Microbiology, 2016, 99(4): 719-728. DOI:10.1111/mmi.2016.99.issue-4 |
| [45] |
Liu T, Li YJ, Wang XD, et al. Transcriptional regulator-mediated activation of adaptation genes triggers CRISPR de novo spacer acquisition[J]. Nucleic Acids Research, 2015, 43(2): 1044-1055. |
| [46] |
Peng WF, Li H, Hallstrom S, et al. Genetic determinants of PAM-dependent DNA targeting and pre-crRNA processing in Sulfolobus islandicus[J]. RNA Biology, 2013, 10(5): 738-748. DOI:10.4161/rna.23798 |
| [47] |
Cao JJ, Wang Q, Liu T, et al. Insights into the post-translational modifications of archaeal Sis10b (Alba): lysine-16 is methylated, not acetylated, and this does not regulate transcription or growth[J]. Molecular Microbiology, 2018, 109(2): 192-208. |
| [48] |
Konermann S, Brigham MD, Trevino AE, et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex[J]. Nature, 2015, 517(7536): 583-588. |
 2019, Vol. 46
2019, Vol. 46