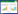扩展功能
文章信息
- 贾德臣, 姜卫红, 顾阳
- JIA De-Chen, JIANG Wei-Hong, GU Yang
- 食气梭菌的研究进展
- Research progresses in gas-fermenting clostridia
- 微生物学通报, 2019, 46(2): 374-387
- Microbiology China, 2019, 46(2): 374-387
- DOI: 10.13344/j.microbiol.china.180841
-
文章历史
- 收稿日期: 2018-10-27
- 接受日期: 2018-12-12
- 网络首发日期: 2018-12-24
2. 中国科学院大学 北京 100039
2. University of Chinese Academy of Sciences, Beijing 100039, China
一碳气体(如CO2、CO)储量巨大,是重要的游离性碳资源,主要来源于劣质天然气、石化和冶炼企业排放的尾气,以及生物质加工处理后产生的合成气等。近年来,一碳气体的转化利用作为前沿研究领域引起了国内外研究者的广泛兴趣。生物转化路线为一碳气体的资源化利用提供了除物理化学催化以外的另一种选择,不仅可有效降低现有生物制造产业的原料成本,增强市场竞争力,而且有助于缓解企业一碳废气排放所引发的环境问题,对工业可持续发展具有重要意义。
固碳微生物可通过生命活动将游离态的一碳气体直接转变为含碳有机物加以利用,在地球生物圈的碳循环中扮演着重要角色,也是目前一碳气体生物固定和转化利用的主要研究对象。迄今已知的固碳微生物按能量需求类型的不同主要可分为化学能、光能以及电能自养微生物[1]。化能型固碳微生物以氢气或其他还原性化合物作为还原力和能量供体用于固定一碳气体,其气体发酵过程与传统工业发酵设备的匹配性较好,因而具有良好的工业应用前景。
食气梭菌是一类主要的化能固碳菌,属于革兰氏阳性的厌氧梭状芽胞杆菌。已报道的食气梭菌主要包括扬氏梭菌(又有译名为永达尔梭菌) (Clostridium ljungdahlii)、自产醇梭菌(Clostridium autoethanogenum)、食一氧化碳梭菌(Clostridium carboxidivorans)等(表 1)。需要指出的是,“自产醇梭菌”和“食一氧化碳梭菌”的表述是根据其学名意译而来,“永达尔梭菌”的表述是其学名的音译,而“扬氏梭菌”的表述则来自该菌在中国申请的相关专利。食气梭菌通过Wood-Ljungdahl途径固定CO/CO2气体[25],经一系列催化反应后转化为重要的代谢中间体——乙酰辅酶A,此后再通过各分支代谢途径合成各种产物,如乙醇、丁醇、己醇等。因此,食气梭菌也是构建一碳气体细胞工厂的理想宿主之一。
| 菌种 Strains |
原料 Substrate |
最适温度 Topt (℃) |
最适pH pHopt |
产物 Products |
基因组 Genome sequence |
分子工具 Genetic tools |
引文 References |
| Clostridium aceticum | H2/CO2, CO | 30 | 8.3 | Acetate | Available | Available | [2-3] |
| Clostridium autoethanogenum | H2/CO2, CO | 37 | 5.8-6.0 | Acetate, ethanol, 2, 3-butanediol, lactate | Available | Available | [4-6] |
| Clostridium carboxidivorans | H2/CO2, CO | 38 | 6.2 | Acetate, ethanol, butyrate, butanol, lactate | Available | [7-8] | |
| Clostridium coskatii | H2/CO2, CO | 37 | 5.8-6.5 | Acetate, ethanol | Available | [9] | |
| Clostridium difficile | H2/CO2, CO | 35-40 | 6.5-7.0 | Acetate, ethanol, butyrate | Available | Available | [10-11] |
| Clostridium drakei | H2/CO2, CO | 30-37 | 5.4-7.5 | Acetate, ethanol, butyrate | Available | [12-13] | |
| Clostridium formicoaceticum | CO | 37 | 8.1 | Acetate, formate | Available | [14-15] | |
| Clostridium glycolicum | H2/CO2 | 37-40 | 7.0-7.5 | Acetate | [16] | ||
| Clostridium ljungdahlii | H2/CO2, CO | 37 | 6.0 | Acetate, ethanol, 2, 3-butanediol, lactate | Available | Available | [17-18] |
| Clostridium magnum | H2/CO2 | 30-32 | 7.2 | Acetate | Available | [19-20] | |
| Clostridium methoxybenzovorans | H2/CO2 | 37 | 7.4 | Acetate, formate | Available | [21] | |
| Clostridium ragsdalei | H2/CO2, CO | 37 | 6.3 | Acetate, ethanol, 2, 3-butanediol, lactate | Available | [4, 9] | |
| Clostridium scatologenes | H2/CO2, CO | 37-40 | 5.4-7.5 | Acetate, ethanol, butyrate | Available | [22-23] | |
| Moorella thermoacetica (Clostridium thermoaceticum) |
H2/CO2, CO | 55-60 | 6.5-6.8 | Acetate | Available | Available | [24] |
| Note: pHopt: Optimum pH; Topt: Optimum temperature. | |||||||
近年来,国内外研究者在优化和提升食气梭菌发酵性能方面做了诸多尝试。此外,得益于近几年分子遗传操作工具的发展,食气梭菌的代谢工程设计、改造以及合成生物学研究也有了一定的进展和发现。基于此,本文对这方面的内容进行了比较系统的梳理和总结,并展望了该研究领域的发展前景。
1 食气梭菌的生理和代谢特点食气梭菌利用Wood-Ljungdahl (WL)途径吸收和固定CO2及CO气体。WL途径被称为还原性乙酰辅酶A途径,是定义产乙酸菌的主要依据。此外,食气梭菌具有独特的能量分配和利用途径,在不同生长条件下具有不同的能量代谢和产物合成途径(图 1),而且不同食气梭菌的产物种类也呈现差异[4, 26-27]。除了最主要的乙酸和乙醇外,一些食气梭菌可在一碳气体生长条件下合成丁醇、2, 3-丁二醇、己醇等高值化合物,因而具有良好的工业应用前景。

|
| 图 1 食气梭菌中心代谢网络 Figure 1 Metabolic pathways of gas-fermenting clostridia 注:2, 3 Bdh:2, 3-丁二醇脱氢酶;Ack:乙酸激酶;Acs:乙酰辅酶A合酶;AdhE:醇醛脱氢酶;Adi:精氨酸脱亚胺酶;Ald:乙醛脱氢酶;Als:乙酰乳酸合酶;Aor:醛氧化还原酶;ArcD:精氨酸/鸟氨酸转运蛋白;ATPase:ATP合酶;BudA:乙偶姻脱羧酶;Cbk:氨基甲酸激酶;Codh:一氧化碳脱氢酶;Eno:磷酸丙酮酸水合酶;Fda:果糖二磷酸醛缩酶;Fdh:甲酸脱氢酶;Fhs:甲酰-四氢叶酸合成酶;FolD:亚甲基四氢叶酸环化酶/脱氢酶;FruK:果糖-1, 6-二磷酸酶;GapdH:甘油醛-3-磷酸脱氢酶;HytA-D:HytABCD氢化酶;HytA-E:HytCBDE1AE2氢化酶;Ldh:乳酸脱氢酶;MetF:亚甲基四氢叶酸还原酶;MetTr:甲基转移酶;Nfn:铁氧还蛋白依赖的电子歧化转氢酶;Otc:鸟氨酸转氨甲酰酶;PEP:磷酸烯醇式丙酮酸;Pfk:磷酸果糖激酶;Pfor:丙酮酸铁氧还蛋白氧化还原酶;Pgk:磷酸甘油酸激酶;Pgm:磷酸甘油酸异构酶;PRPP:5-磷酸核糖焦磷酸;Prs:磷酸核糖焦磷酸激酶;Pta:磷酸乙酰转移酶;Pts:磷酸转移酶;Pyk:丙酮酸激酶;Rnf:铁氧还蛋白:NAD+氧化还原酶;Rpe:磷酸核酮糖异构酶;Rpia:5-磷酸核糖异构酶;Tal:转醛酶;Tkt:转酮酶. Note: 2, 3 Bdh: 2, 3-Butanediol dehydrogenase; Ack: Acetate kinase; Acs: Acetyl-CoA synthase; AdhE: Acetaldehyde/alcohol dehydrogenase; Adi: Arginine deiminase; Ald: Acetaldehyde dehydrogenase; Als: Acetolactate synthase; Aor: Aldehyde: ferredoxin oxidoreductase; ArcD: L-arginine/L-ornithine transporter; ATPase: ATPsynthase; BudA: Acetoin decarboxylase; Cbk: Carbamate kinase; Codh: CO dehydrogenase; Eno: Phosphopyruvate hydratase; Fda: Fructose-1, 6-bisphosphate aldolase; Fdh: Formate dehydrogenase; Fhs: Formyl-THF synthetase; FolD: Methenyl-THF cyclohydrolase; FruK: Fructose-1, 6-bisphosphatase; GapdH: Glyceraldehyde-3-phosphate dehydrogenase; HytA-D: HytABCD hydrogenase; HytA-E: HytCBDE1AE2 hydrogenase; Ldh: Lactate dehydrogenase; MetF: Methylene-THF reductase; MetTr: Methyltransferase; Nfn: Electron-bifurcating and ferredoxin-dependent transhydrogenase; Otc: Ornithine transcarbamylase; PEP: Phosphoenolpyruvate; Pfk: Phosphofructokinase; Pfor: Pyruvate:ferredoxin oxidoreductase; Pgk: Phosphoglycerate kinase; Pgm: Phosphoglycerate mutase; PRPP: Phosphoribosyl pyrophosphate; Prs: Phosphoribosylpyrophosphate synthetase; Pta: Phosphotransacetylase; Pts: Phosphotransferase; Pyk: pyruvate kinase; Rnf: Ferredoxin:NAD+ oxidoreductase; Rpe: Phosphoribulose epimerase; Rpia: Ribose5-phosphate isomerase; Tal: Transadolase; Tkt: Transketolase. |
|
|
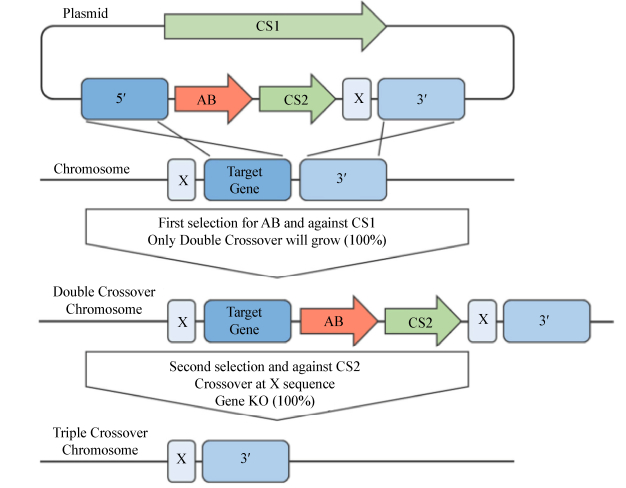
WL途径是目前已知的反应步骤最短且能量消耗最少的一种生物固碳途径[28]。该途径由甲基分支反应和羰基分支反应两个通路组成[2, 29](图 2)。在甲基分支反应中,CO2在甲酸脱氢酶、甲酰-四氢叶酸合成酶、亚甲基四氢叶酸环化酶/脱氢酶、亚甲基-四氢叶酸脱氢酶、亚甲基-四氢叶酸还原酶、甲基转移酶和一氧化碳脱氢酶/乙酰辅酶A合成酶(CODH/ACS)复合物等酶的催化下,先后形成甲酸、甲酰四氢叶酸、亚甲基四氢叶酸、甲基四氢叶酸等,最终与羰基分支反应中的羰基形成乙酰辅酶A。在羰基分支反应中,CO和CO2在一氧化碳脱氢酶(CODH)的催化下先形成羰基,随后羰基与辅酶A和甲基在一氧化碳脱氢酶/乙酰辅酶A合成酶(CODH/ACS)的催化下形成乙酰辅酶A。CODH/ACS复合体是一个α2β2的四聚体,可以催化羰基、辅酶A和甲基生成乙酰辅酶A[2, 29]。在催化过程中,α亚基催化乙酰辅酶A的形成,而β亚基则将二氧化碳还原为一氧化碳。此外,该复合体还可将CO氧化为CO2,从而产生电子。此后,乙酰辅酶A在磷酸乙酰转移酶和乙酸激酶的催化下形成乙酸,乙酸在醛氧化还原酶(AOR)的作用下消耗Fdred形成乙醛,最后在醇醛脱氢酶的作用下形成乙醇[30-31]。
|
| 图 2 Wood-Ljungdahl通路 Figure 2 Wood-Ljungdahl pathway 注:Ack:乙酸激酶;Acs:乙酰辅酶A合酶;AdhE:醇醛脱氢酶;Ald:醛脱氢酶;Codh:一氧化碳脱氢酶;Fdh:甲酸脱氢酶;Fhs:甲酰-四氢叶酸合成酶;FolD:亚甲基四氢叶酸环化酶/脱氢酶;MetF:亚甲基四氢叶酸还原酶;MetTr:甲基转移酶;Pta:磷酸乙酰转移酶. Note: Ack: Acetate kinase; Acs: Acetyl-CoA synthase; AdhE: Acetaldehyde/alcohol dehydrogenase; Ald: Acetaldehyde dehydrogenase; Codh: CO dehydrogenase; Fdh: Formate dehydrogenase; Fhs: Formyl-THF synthetase; FolD: Methenyl-THF cyclohydrolase; MetF: Methylene-THF reductase; MetTr: Methyltransferase; Pta: Phosphotransacetylase. |
|
|
Wood-Ljungdahl途径生成的乙酰辅酶A能够被代谢生成乙酸、丁酸,以及多种有机醇(乙醇、丁醇、2, 3-丁二醇)。乙酸合成过程中可产生ATP作为能量,因而在食气梭菌代谢一碳气体这类“能量贫瘠”的碳源时是重要的能量供应途径。因此,食气梭菌发酵一碳气体时会产生大量的乙酸。此外,在WL途径的甲基分支反应中,类咕啉铁硫蛋白(CH3-CoFeS-P)在一氧化碳脱氢酶/乙酰辅酶A合成酶(CODH/ACS)的作用下与等量的CO聚合形成乙酰辅酶A,该过程需要大量的能量和CO,因此,该步反应也被认为是整个WL途径的限速步骤[30-31]。
1.2 乙酸、丁酸合成及回用目前已被鉴定的食气梭菌在气体发酵过程中均可以催化乙酰辅酶A形成乙酸,并产生1分子的ATP[2]。此外,乙酸可在醛氧化还原酶(AOR)的作用下消耗Fdred形成乙醛,最后在醇醛脱氢酶的作用下形成乙醇[2]。此外,丁酸也是食气梭菌的主要产物之一。在C. carboxidivorans[9]、C. drakei[14]、C. magnum[32]、C. scatologenes[24]、C. methoxybenzovorans[22]的气体发酵产物中均可检测到丁酸的存在。丁酸的合成主要包括以下步骤:乙酰辅酶A在硫酯酶(Thl)、3-羟基丁酸辅酶A脱氢酶(Hbd)、巴豆酸酶(Crt)、丁酰辅酶A脱氢酶(Bcd)、电子转运黄素蛋白(EtfA, EtfB)、磷酸转丁酰酶(Ptb)和丁酸激酶(Buk)的作用下形成丁酸。其中,电子转运黄素蛋白(EtfA,EtfB)对丁酰辅酶A脱氢酶(Bcd)的活力起着至关重要的作用[33]。已知在解糖型梭菌丙酮丁醇梭菌(Clostridium acetobutylicum)中,丁酸合成相关基因(crt,bcd,etfA/B,hbd)是成簇分布的,构成了一个操纵子结构(bcs operon),由crt基因上游的启动子驱动表达[34]。尽管在多数食气梭菌中,上述丁酸合成途径的基因也是成簇分布的,但有些基因间存在较长的非编码序列,因此不能确认这些基因是否也构成一个操纵子结构[2]。此外,通过TBLASTx分析发现,在C. carboxidivorans 和C. scatologenes的基因组上存在两个bcs基因簇,其中一个基因簇中存在一个水合酶基因,该基因被认为可能编码一个R型的3-羟基丁酸辅酶A脱氢酶,从而催化3-羟基丁酸辅酶A进一步生成R型3-羟基丁酸[2]。
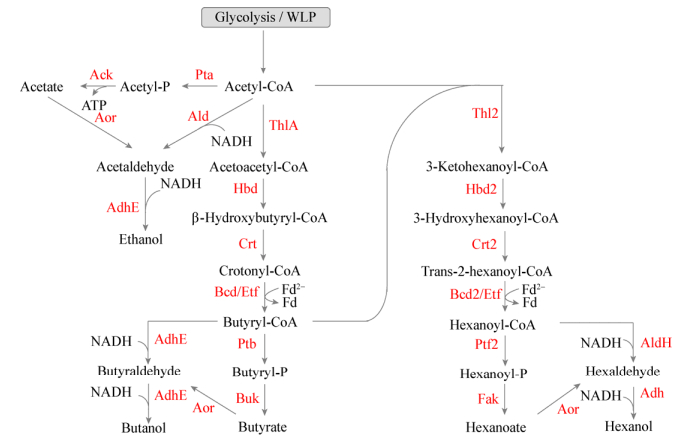
1.3 其他产物合成许多食气梭菌的产物谱较为丰富。例如,C. carboxidivorans和C. drakei不仅能够产生乙酸和丁酸,还可以合成乙醇、丁醇、己酸和己醇等,因此,C. carboxidivorans的发酵又可被称为HBE (Hexanol, Butanol, Ethanol)发酵(图 3)[9, 35-36]。在C. carboxidivorans己酸和己醇代谢通路中,乙酰辅酶A和丁酰辅酶A在硫解酶(Thl2)的催化下形成3-酮基己酰辅酶A,随后3-酮基己酰辅酶A经三步反应形成己酰辅酶A。己酰辅酶A在脱氢酶(AldH,Adh)或磷酸转乙酰酶(Ptf2)和激酶(Fak)的作用下生成己醇或己酸。在以上反应中,催化3-羟基己酰辅酶A形成己酰辅酶A的酶尚未被鉴定,但推测丁酰辅酶A合成途径所用酶也可在此发挥功能[36]。
|
| 图 3 C. carboxidivorans溶剂合成途径 Figure 3 Solvent synthesis pathways of C. carboxidivorans 注:Ack:乙酸激酶;Adh:醇脱氢酶;AdhE:醇醛脱氢酶;Ald:乙醛脱氢酶;AldH:己醛脱氢酶;Aor:醛氧化还原酶;Bcd(Bcd2):丁酰辅酶A脱氢酶;Buk:丁酸激酶;Crt(Crt2):巴豆酸酶;Etf:电子转移黄素蛋白;Fak:脂肪酸激酶;Hbd(Hbd2):3-羟基丁酸辅酶A脱氢酶;Pta:磷酸乙酰转移酶;Ptb(Ptf2):磷酸乙酰转移酶;ThlA(Thl2):硫解酶. Note: Ack: Acetate kinase; Adh: Alcohol dehydrogenase; AdhE: Acetaldehyde/alcohol dehydrogenase; Ald: Acetaldehyde dehydrogenase; AldH: Aldehyde dehydrogenasel; Aor: Aldehyde:ferredoxin oxidoreductase; Bcd(Bcd2): Butyryl-CoA dehydrogenase; Buk: Butyrate kinase; Crt(Crt2): Crotonase; Etf: Electron transfer flavoprotein; Fak: Fatty acid kinase; Hbd(Hbd2): 3-Hydroxybutyryl-CoA dehydrogenase; Pta: Phosphotransacetylase; Ptb(Ptf2): Phosphotransferase; ThlA(Thl2): Thiolase. |
|
|
除以上代谢产物外,C. ljungdahlii、C. autoethanogenum和C. ragsdalei还可以合成乙偶姻、2, 3-丁二醇和乳酸(图 1)。
1.4 能量代谢在以气体为碳源的食气梭菌培养过程中,Wood-Ljungdahl途径中甲酸至甲酰四氢叶酸的催化过程需要消耗1分子ATP,而在后续的乙酰辅酶A至乙酸的催化过程中,则会产生1分子ATP,因此,整个代谢过程理论上没有多余的能量产生。此外,Wood-Ljungdahl途径产生的乙酰辅酶A并不能全部转化为乙酸。因此,为维持菌体生长和代谢,食气梭菌还需要额外的能量合成机制。
研究表明,食气梭菌中存在一个参与能量合成调控的Rnf酶复合物[37](图 1)。该复合物可以将Fd2-转换为Fd,同时伴随着能量和NADH的产生。此外,该复合物构建了一个跨膜的离子转运通道,运用Fd2-转换为Fd产生的能量将胞内的质子如H+、Na+等转运至胞外,使细胞膜内外形成质子浓度梯度差。此后,膜外的质子可再次回到胞内,从而产生能量,此部分能量被膜上的ATP合酶转化为ATP储存起来,供细胞生长使用。该机制被认为是胞内NADH的主要来源[38-40]。在C. ljungdahlii和C. autoethanogenum中,Rnf酶复合物将胞内H+转运至胞外,使得细胞内外形成化学渗透离子梯度。此后,H+依赖型的ATPase将H+转运入胞内并伴随ATP的合成[37]。Rnf酶复合物在C. ljungdahlii[19, 37, 40]、C. autoethanogenum[41]和C. aceticum[4]中都已被发现,而且被证实与长链化合物的合成有关[42]。此外,在C. kluyveri中还发现了一种新的基于四氢酮醇的电子转运供能模式,该供能模式在多种产乙酸菌包括食气梭菌中具有广泛的分布[43-46]。
精氨酸脱亚胺酶途径(精氨酸水解途径)是食气梭菌中存在的另外一条能量合成途径。该途径转换精氨酸为鸟氨酸、CO2和氨,并伴随着1分子ATP的产生[47]。精氨酸通过精氨酸脱亚胺酶(Adi,由arcA编码)转化为瓜氨酸和氨。瓜氨酸在鸟氨酸转氨酶(Otc,由arcB编码)的催化下与磷酸结合,产生鸟氨酸和氨基甲酸乙酯。此后,氨基甲酸乙酯在氨基甲酸激酶(Cbk,由arcC编码)的作用下,产生ATP、二氧化碳和氨(图 1)。此外,该途径的关键酶为精氨酸/鸟氨酸转运蛋白(arcD或arcE编码),该酶在精氨酸和鸟氨酸的等量转换中发挥着重要的功能[48]。已有文献报道,基因arcA、arcB、arcC、arcD(arcE)在许多食气梭菌中是成簇分布的[2]。有研究者发现,在培养基中添加精氨酸能够促进自产醇梭菌(C. autoethanogenum)的生长,并提升菌株的乙醇合成能力[47]。
除ATP合成途径外,铁氧还原蛋白以及NADH或NADPH对维持胞内生理代谢发挥着重要的作用。已有研究表明,当以CO或合成气作为碳源时,食气梭菌氧化CO为CO2并产生还原性铁氧还原蛋白[49]。而当以H2和CO2作为能源和碳源时,食气梭菌则依赖双功能的电子传递氢化酶催化氢气产生还原性铁氧还原蛋白以及NADH或NADPH[44, 50]。已知在C. autoethanogenum中,[FeFe]氢化酶包括6个蛋白亚基(HytCBDE1AE2)且与NADPH依赖的甲酸脱氢酶(Fdh)构成一个大的复合体,氧化氢气并还原NAD+合成NADH[43]。而对C. ljungdahlii全基因组的分析发现,该菌株中存在4种不同类型的氢化酶,包括:(1) HytCBDE1AE2 (CLJU_c07030-CLJU_c07080);(2) HydABCD (CLJU_c14700-CLJU_c14720);(3)镍铁依赖的氢化酶(CLJU_c28660-CLJU_c28670);(4) 3个同源的铁离子氢化酶(CLJU_c17280,CLJU_c37220,CLJU_c20290)。然而,C. ljungdahlii蛋白质组数据表明,可能仅有HytCBDE1AE2发挥了氢化酶的功能[51]。HytCBDE1AE2氢化酶和甲酸脱氢酶复合体的形成使得菌株能够直接利用氢气还原CO2为甲酸,并产生还原性铁氧化还原蛋白,这一机制显著提高了Wood-Ljungdahli途径固碳效率[43]。同时,该过程产生的还原性铁氧化还原蛋白对Rnf复合体离子转运、维持胞内外离子浓度差并引导ATPase合成ATP也至关重要[43]。
2 食气梭菌的分子操作工具近年来,随着多株食气梭菌的全基因组序列陆续被报道,从分子水平对这类厌氧微生物进行深入研究已成为可能。过去几年中,研究者们对食气梭菌分子遗传操作工具进行了优化和改进,并开发出一些新的基因编辑、基因表达技术和方法。基于此,本章对食气梭菌遗传操作工具的发展进行了梳理和总结,主要包括外源DNA的导入、基因编辑、分子元件的发掘和构建三个方面。
2.1 外源DNA导入对于梭菌属和大多数革兰氏阳性菌株而言,其细胞外的肽聚糖细胞壁以及体内的限制修饰系统是制约外源DNA进入细胞的最大障碍[52]。丙酮丁醇梭菌(C. acetobutylicum)是第一个成功实现电穿孔转化的梭菌。近年来,该方法也在食气梭菌中得到应用,如C. ljungdahlii[53]、C. autoethanogenum[47]、C. aceticum[54]。
与此同时,新一代测序技术如单分子实时测序(Single molecule real-time sequencing)的出现,为鉴定细菌的限制性修饰位点提供了可靠的帮助。2014年,Utturkar等使用该技术成功鉴定了C. autoethanogenum基因组中的甲基化修饰位点,寻找到相关的甲基化酶并成功在大肠杆菌中表达,提高了C. autoethanogenum的电转化效率[55]。此外,通过细胞间的直接接触,供体菌DNA转入受体菌的接合转移方法也在食气梭菌中进行了尝试[41]。
此外,选用合适的复制子和筛选标记是实现携带外源DNA质粒进入宿主微生物的关键,从而保证外源质粒能够在新宿主体内正常复制和保留。2009年,Heap等构建了一系列大肠杆菌-梭菌穿梭质粒[56],并在食气梭菌中成功应用[53, 57]。
2.2 基因编辑 2.2.1 基于二类内含子的基因插入失活二类内含子是一类由具备自剪切功能的内含子RNA和反转录酶活性的IEP (Intron-encoding protein)蛋白构成的逆转录因子。内含子RNA在IEP蛋白的协助下可特异性地识别和插入到目标基因的DNA位点,并由IEP蛋白协助反转录成cDNA,随后通过宿主的修复机制修补DNA链上的缺口,最终在该位置植入一段新的DNA片段。2007年,科研人员利用来自乳酸球菌的二类内含子分别在丙酮丁醇梭菌和拜氏梭菌中成功实现了基因敲除,该方法又被称为“ClosTron”[58-59]。目前这一方法在食气梭菌C. autoethanogenum和C. ljungdahlii等菌株中也得到了成功应用[7, 41, 57]。
2.2.2 基于同源重组的基因删除基于同源重组的基因中断技术已被广泛应用,但梭菌的低重组效率制约了该方法的应用。近年来,研究者通过技术改良已经开发了适用于梭菌的此类技术。除直接将质粒导入宿主菌进行同源重组的方法外,反向筛选标记的使用也是重要的辅助手段,有助于快速筛选到目标突变株。例如,Koepke等在C. autoethanogenum中开发了两个反向筛选标记pheS*和thiK,成功实现了基因的敲除[60]。此外,Ueki等将筛选标记和Cre-lox系统组合使用,在C. ljungdahlii中实现了基因pta的敲除[57]。2016年,Walker等在C. autoethanogenum中开发出了一种“Triple Cross”基因中断工具,该工具同样应用了同源重组的原理,但与传统的双交换同源重组不同,该工具运用了一个阳性筛选标记和两个阴性筛选标记,通过两步同源重组反应来实现基因的敲除[61] (图 4)。
2.2.3 CRISPR/Cas近年来,CRISPR/Cas系统被广泛应用于基因组编辑[63-64]。我们研究组在国际上率先建立了重要食气梭菌C. ljungdahlii的CRISPR-Cas9基因组编辑技术,实现对目标基因的快速删除,为基因功能研究和代谢工程改造奠定了基础[65]。随后,该基因编辑技术也在另一种主要食气梭菌C. autoethanogenum中得到建立[66]。此外,dCas9蛋白介导的CRISPR interference (CRISPRi)技术也于近期被研究者开发用于抑制C. ljungdahlii中的乙酸合成途径[67],实现对副产物乙酸合成量的调节,并显著提升了目标产物3-羟基丁酸的产量。
2.3 分子元件的发掘和构建分子元件的发掘和构建对于食气梭菌的合成生物学研究具有重要意义。研究者将来源于拜氏梭菌中的乳糖诱导性启动子bgaR-PbgaL进行优化,并应用于食气梭菌C. ljungdahlii的丙酮合成中[68]。此外,四环素诱导的启动子Ptet也于近期在C. ljungdahlii中得以应用[67]。
为了有效拓展食气梭菌的启动子资源并应用于代谢工程及合成生物学研究,我们课题组开发了一套适用于梭菌的双报告基因系统以及快速构建启动子突变体文库的技术路线,用于高效筛选、获取不同强度的人工启动子并构建启动子文库。借助该技术体系,我们建立了梭菌通用性的人工启动子文库,文库中的启动子强度覆盖两个数量级,而且包含多个强启动子[69]。该启动子文库在梭菌中具有较好的普适性且适用于食气梭菌,有效地扩充和丰富了现有食气梭菌的基因表达元件[69]。
3 食气梭菌的遗传改造近年来,食气梭菌的研究获得广泛关注,研究者们已先后完成了C. autoethanogenum[6, 55, 70]、C. ljungdahlii[19]、C. carboxidivorans P7[8]、C. aceticum[4]等菌株的全基因组测序,发现和鉴定了食气梭菌中多个关键功能基因[7, 41, 43, 71-73]。
基于近年来食气梭菌分子遗传操作工具的发展,相关的菌株遗传改造已经得以进行。目前已经实现了丁醇[5, 19]、2, 3-丁二醇[5]、丙酮[54, 68, 74]、异丙醇[10]、3-羟基丁酸[74]、3-羟基丙二酸[75]、甲羟戊酸、异戊二烯[76]、丁酸丁酯[77]等产物合成途径在食气梭菌中的构建和表达。例如,将来源于丙酮丁醇梭菌C. acetobutylicum的丁醇合成基因簇导入C. ljungdahlii和C. autoethanogenum中,并强化电子转运蛋白编码基因etfAB的表达,实现了这两株食气梭菌中的丁醇合成[19, 60];而将C. acetobutylicum的丙酮合成基因簇在C. aceticum和C. ljungdahlii中表达,也实现了发酵一碳气体合成丙酮[54, 68]。此外,研究人员在C. ljungdahlii中以来源于C. acetobutylicum的丁酸合成基因簇替代其自身乙酸合成途径中的磷酸乙酰基转移酶基因pta,实现了丁酸的合成[78]。
抑制或敲除旁路途径是提高代谢途径中目标产物产量的方法之一。研究者利用同源重组和二类内含子介导的基因失活方法抑制了C. autoethanogenum丁二醇合成途径中乙酰乳酸脱羧酶基因(budA)的表达,提高了乙醇产量[60]。另一项研究表明,在C. autoethanogenum中抑制乳酸脱氢酶基因(ldhA)同样使乙醇的产量得到了提高[60]。
除旁路途径的影响外,菌株对产物的耐受性同样制约着目标产物产量的提高。为减弱高浓度乙醇对于菌株的抑制作用,研究者们在C. autoethanogenum中过表达了两个与蛋白折叠相关的辅因子GroES和GroEL,提高了菌株对乙醇的耐受能力[60]。
2016年,Jones等在C. ljungdahlii中引入丙酮丁醇梭菌来源的丙酮合成途径,采用混合碳源(糖/一碳气体)发酵策略,使得糖酵解途径产生的CO2被WL途径重新利用,减少了糖酵解途径的碳流失,提升了菌株的碳固定能力。此外,通过对菌株的富集,实现细胞的高密度连续发酵,最终使得丙酮合成量达到10 g/L[74]。据文献报道,通过过表达来源于Bacillus subtilis的硫酯酶(teII)和Haemophilus influenzae的乙酰辅酶硫酯酶(ybgC)均可提高C. ljungdahlii合成气发酵时的丙酮产量[54, 79]。另一项研究发现,在C. ljungdahlii和C. autoethanogenum中存在一个依赖于NADPH的次级醇醛脱氢酶基因(sadh),能够将丙酮转化为异丙醇[80-82],进一步拓展了食气梭菌的产物谱。此外,通过在C. autoethanogenum和C. ljungdahlii中表达MVA途径和异戊二烯代谢途径,证实了食气梭菌生产倍半萜类化合物如甲羟戊酸、异戊二烯等的可能性[60, 76, 83-85]。
新西兰朗泽公司近年来开发了多个食气梭菌工程菌株,转化一碳气体合成高附加值产物,如甲基乙基酮(MEK)[60]、3-羟基丙酸(3-HP)[86]、生物柴油、1, 3-丁二烯[87]等。然而,基于气体转化效率和生产成本等多方面因素的考虑,目前这些工程菌株还远未达到工业化生产水平。因此,对食气梭菌进行代谢工程改造,提升菌株气体转化效率,提高产物合成气发酵产率和产量,是目前研究的重要方向。
4 食气梭菌的发酵优化气液传质效率是一碳气体发酵的主要限速步骤之一[88]。高效的气液传质效率是气体生物反应器设计的关键,给予更多的能量供体、加快气体流通速度或增加发酵罐内压力等均可以提升气液传质效率。
连续搅拌釜式反应器是目前实验室使用较多的气体反应器,可通过机械搅拌将通入的气体变成微小气泡,从而增大气液接触面积,达到较好的气液传递效果。与连续搅拌釜式反应器相比,鼓泡塔反应器通过喷射方式将气体注入培养基,气体分散效果较好,无需机械搅拌,具有良好的传热和传质效率。除增大气液接触面积外,提高反应器中气体压力或在反应液中补加碳酸盐等,也是提高发酵液中气体溶解度、加速菌株气体利用效率的有效方法。据文献报道,在以果糖作为碳源时,在C. ljungdahlii的培养基中补加CaCO3等化合物,能显著提升乙醇和乙酸的合成产量[89];增加碳源中CO的分压也能够显著提升菌株的生长和产物合成量[90]。此外,为了解决食气梭菌发酵中的低生物量问题,研究者在气体发酵装置中加入一些吸附菌体的填料和介质来提升细胞密度。例如,采用中空纤维膜富集细胞,可有效提高发酵气体时的丙酮产量[74]。
多细胞体系利用不同微生物间的共生关系来提高底物利用率,进而提高产物产量,因此是一种拓展发酵产物种类以及提升发酵效率的有效策略。解糖型梭菌,如C. acetobutylicum和C. beijerinckii与食气梭菌具有良好的互补关系。前者在糖酵解过程中释放的CO2和H2,恰恰是后者的碳源和能源。因此,如将两者按一定的比例进行共培养,在糖基原料的发酵中可避免过多的碳损失(以CO2的形式释放),从而极大地提升碳利用率[91]。近期,已有研究者尝试了上述发酵策略,通过将C. acetobutylicum和C. ljungdahlii的共培养,显著提高了产物得率且检测到新产物的生成[92]。
培养基成分的优化对于气体发酵同样重要。虽然食气梭菌以CO、CO2、H2为碳源和能源,但仍需要其他成分如维生素、微量元素、矿物质元素和还原剂等保持菌种的代谢活性[9, 93]。例如,通过降低C. ljungdahlii培养基中B族维生素和酵母提取物的浓度可提升乙醇产量[93]。Kundiyana等在C. carboxidivorans P7的培养基中使用棉花籽提取物替代酵母提取物等成分,使得合成气发酵的乙醇产量得到明显提高[94]。此外,调整培养基中的微量金属离子浓度能够提高某些金属离子依赖的氧化还原酶的活力,可提升食气梭菌中乙醇的产量[95];优化发酵培养基中各矿质元素的浓度,同时采用低温培养的方式能提高食气梭菌的产物合成能力[9];而在培养基中加入还原剂,如甲基紫晶、Na2S等也能显著提升菌株的气体发酵水平[96-97]。
pH值和温度等对食气梭菌的生长也有重要的影响。研究发现,在pH 6.8时,C. ljungdahlii的细胞密度和乙醇产量均高于pH 5.5时的水平[98]。食气梭菌最适生长温度在30-40 ℃之间,培养温度不仅影响底物利用率、生长率和膜脂组成,而且影响气体碳源在培养中的溶解度。已经发现,适当降低培养温度虽然会造成食气梭菌生长速率的一定延迟,但最终的产物合成量相对于常规培养温度有显著提高[9],这一现象背后的分子机制尚待解析。
5 展望以一碳气体为原料经化学催化合成氨、含氧化合物和烃类等化学品的生产技术已投入商业运行,但仍存在局限性。相比于化学催化路线,生物转化方式有其特色和优点,可为一碳气体的资源化利用提供另一种路线选择,是一种创新性的绿色可持续生产模式。
在现有的三类主要生物固碳形式中(化学能、光能和电能),化能自养菌的气体发酵过程与现有工业发酵技术及体系的相容性较好,因而具有良好的工业应用前景。食气梭菌作为化能自养菌的主要成员,是构建一碳气体生物转化平台的理想宿主微生物之一。近年来,研究者们已在优化食气梭菌气体转化利用和发酵技术方面做了诸多尝试,但主要集中于外部条件。受限于对食气梭菌生理代谢相关分子机制的有限认识以及分子技术手段的低效,目前通过理性设计和改造提高菌株气体发酵性能的研究进展比较缓慢。因此,食气梭菌菌株优化方面的研究未来需在以下几方面进行重点突破:(1)进一步优化现有分子技术平台,尤其是功能更为强大的分子操作工具。例如,高效的外源大片段DNA在染色体上的整合技术,用于快速构建和稳定表达新产物途径,高效创建一碳细胞工厂合成高值大宗化学品。(2)拓展和完善适用于食气梭菌的各类生物元件和功能模块,从而实现更为精细的分子操作,为合成生物学研究奠定基础。(3)加强食气梭菌的功能基因组学研究,系统发掘与重要表型相关的功能基因,解析分子机制,从而更全面、深入地认识和理解食气梭菌,为菌株改造提供有效靶点和思路。
此外,相对于传统有机质原料发酵而言,气体发酵的装置设计和过程控制有一定的特殊性,这一点对于工业规模的气体发酵而言尤为重要。但目前,已报道的工业尾气发酵中试运行装置的关键技术均由国外公司掌握,国内相关科研机构尚未在这方面进行有效布局和系统研究,其中的技术短板极有可能成为未来国内气体发酵技术大规模推广时的主要限制因素,这无疑是未来值得重点关注的一个研究方向。
| [1] |
Liao JC, Mi L, Pontrelli S, et al. Fuelling the future: Microbial engineering for the production of sustainable biofuels[J]. Nature Reviews Microbiology, 2016, 14(5): 288-304. DOI:10.1038/nrmicro.2016.32 |
| [2] |
Lee S, Song Y, Choe D, et al. Reconstruction of acetogenesis pathway using short-read sequencing of Clostridium aceticum genome[J]. Journal of Nanoscience and Nanotechnology, 2015, 15(5): 3852-3861. DOI:10.1166/jnn.2015.9537 |
| [3] |
Poehlein A, Cebulla M, Ilg MM, et al. The complete genome sequence of Clostridium aceticum: a missing link between Rnf- and cytochrome-containing autotrophic acetogens[J]. mBio, 2015, 6(5): e01168-15. |
| [4] |
Köpke M, Mihalcea C, Liew F, et al. 2, 3-butanediol production by acetogenic bacteria, an alternative route to chemical synthesis, using industrial waste gas[J]. Applied and Environmental Microbiology, 2011, 77(15): 5467-5475. DOI:10.1128/AEM.00355-11 |
| [5] |
Brown SD, Nagaraju S, Utturkar S, et al. Comparison of single-molecule sequencing and hybrid approaches for finishing the genome of Clostridium autoethanogenum and analysis of CRISPR systems in industrial relevant Clostridia[J]. Biotechnology for Biofuels, 2014, 7(1): 40. |
| [6] |
Marcellin E, Behrendorff JB, Nagaraju S, et al. Low carbon fuels and commodity chemicals from waste gases – systematic approach to understand energy metabolism in a model acetogen[J]. Green Chemistry, 2016, 18(10): 3020-3028. DOI:10.1039/C5GC02708J |
| [7] |
Li N, Yang JJ, Chai CS, et al. Complete genome sequence of Clostridium carboxidivorans P7T, a syngas-fermenting bacterium capable of producing long-chain alcohols[J]. Journal of Biotechnology, 2015, 211: 44-45. DOI:10.1016/j.jbiotec.2015.06.430 |
| [8] |
Shen SH, Gu Y, Chai CS, et al. Enhanced alcohol titre and ratio in carbon monoxide-rich off-gas fermentation of Clostridium carboxidivorans through combination of trace metals optimization with variable-temperature cultivation[J]. Bioresource Technology, 2017, 239: 236-243. DOI:10.1016/j.biortech.2017.04.099 |
| [9] |
Bengelsdorf FR, Poehlein A, Linder S, et al. Industrial acetogenic biocatalysts: A comparative metabolic and genomic analysis[J]. Frontiers in Microbiology, 2016, 7: 1036. |
| [10] |
Köpke M, Straub M, Dürre P. Clostridium difficile is an autotrophic bacterial pathogen[J]. PLoS One, 2013, 8(4): e0062157. |
| [11] |
Riedel T, Bunk B, Thürmer A, et al. Genome resequencing of the virulent and multidrug-resistant reference strain Clostridium difficile 630[J]. Genome Announcements, 2015, 3(2): e00276-15. |
| [12] |
Gößner AS, Picardal F, Tanner RS, et al. Carbon metabolism of the moderately acid-tolerant acetogen Clostridium drakei isolated from peat[J]. FEMS Microbiology Letters, 2010, 287(2): 236-242. |
| [13] |
Jeong Y, Song Y, Shin HS, et al. Draft genome sequence of acid-tolerant Clostridium drakei SL1T, a potential chemical producer through syngas fermentation[J]. Genome Announcements, 2013, 2(3): e00387-14. |
| [14] |
Andreesen JR, Gottschalk G, Schlegel HG. Clostridium formicoaceticum nov. spec. isolation, description and distinction from C. aceticum and C. thermoaceticum[J]. Archiv Fur Mikrobiologie, 1970, 72(2): 154-174. DOI:10.1007/BF00409521 |
| [15] |
Karl MM, Poehlein A, Bengelsdorf FR, et al. Complete genome sequence of the autotrophic acetogen Clostridium formicaceticum DSM 92T using nanopore and illumina sequencing data[J]. Genome Announcements, 2017, 5(21): e00423-17. |
| [16] |
Cai DH, Sorokin V, Lutwick L, et al. C. glycolicum as the sole cause of bacteremia in a patient with acute cholecystitis[J]. Annals of Clinical & Laboratory Science, 2012, 42(2): 162-164. |
| [17] |
Tanner RS, Miller LM, Yang DC. Clostridium ljungdahlii sp. nov., an acetogenic species in clostridial rRNA homology group Ⅰ[J]. International Journal of Systematic Bacteriology, 1993, 43(2): 232-236. DOI:10.1099/00207713-43-2-232 |
| [18] |
Köpke M, Held C, Hujer S, et al. Clostridium ljungdahlii represents a microbial production platform based on syngas[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(29): 13087-13092. DOI:10.1073/pnas.1004716107 |
| [19] |
Bomar M, Hippe H, Schink B. Lithotrophic growth and hydrogen metabolism by Clostridium magnum[J]. FEMS Microbiology Letters, 1991, 83(3): 347-349. DOI:10.1111/fml.1991.83.issue-3 |
| [20] |
Uhlig R, Poehlein A, Fischer RJ, et al. Genome sequence of the autotrophic acetogen Clostridium magnum DSM 2767[J]. Genome Announcements, 2016, 4(3): e00464-16. |
| [21] |
Mechichi T, Labat M, Patel BKC, et al. Clostridium methoxybenzovorans sp. nov., a new aromatic o-demethylating homoacetogen from an olive mill wastewater treatment digester[J]. International Journal of Systematic Bacteriology, 1999, 49(3): 1201-1209. DOI:10.1099/00207713-49-3-1201 |
| [22] |
Song Y, Jeong Y, Shin HS, et al. Draft genome sequence of Clostridium scatologenes ATCC 25775, a chemolithoautotrophic acetogenic bacterium producing 3-methylindole and 4-methylphenol[J]. Genome Announcements, 2014, 2(3): e00459-14. |
| [23] |
Zhu ZG, Guo T, Zheng HJ, et al. Complete genome sequence of a malodorant-producing acetogen, Clostridium scatologenes ATCC 25775T[J]. Journal of Biotechnology, 2015, 212: 19-20. DOI:10.1016/j.jbiotec.2015.07.013 |
| [24] |
Pierce E, Xie G, Barabote RD, et al. The complete genome sequence of Moorella thermoacetica (f. Clostridium thermoaceticum)[J]. Environmental Microbiology, 2010, 10(10): 2550-2573. |
| [25] |
Bengelsdorf FR, Beck MH, Erz C, et al. Bacterial anaerobic synthesis gas (syngas) and CO2+H2 fermentation[J]. Advances in Applied Microbiology, 2018, 103: 143-221. DOI:10.1016/bs.aambs.2018.01.002 |
| [26] |
Schuchmann K, Müller V. Autotrophy at the thermodynamic limit of life: A model for energy conservation in acetogenic bacteria[J]. Nature Reviews Microbiology, 2014, 12(12): 809-821. DOI:10.1038/nrmicro3365 |
| [27] |
Bertsch J, Müller V. Bioenergetic constraints for conversion of syngas to biofuels in acetogenic bacteria[J]. Biotechnology for Biofuels, 2015, 8: 210. DOI:10.1186/s13068-015-0393-x |
| [28] |
Fast AG, Papoutsakis ET. Stoichiometric and energetic analyses of non-photosynthetic CO2-fixation pathways to support synthetic biology strategies for production of fuels and chemicals[J]. Current Opinion in Chemical Engineering, 2012, 1(4): 380-395. DOI:10.1016/j.coche.2012.07.005 |
| [29] |
Latif H, Zeidan AA, Nielsen AT, et al. Trash to treasure: Production of biofuels and commodity chemicals via syngas fermenting microorganisms[J]. Current Opinion in Biotechnology, 2014, 27: 79-87. DOI:10.1016/j.copbio.2013.12.001 |
| [30] |
Bengelsdorf FR, Dürre P. Gas fermentation for commodity chemicals and fuels[J]. Microbial Biotechnology, 2017, 10(5): 1167-1170. |
| [31] |
Daniell J, Nagaraju S, Burton F, et al. Low-carbon fuel and chemical production by anaerobic gas fermentation[A]//Hatti-Kaul R, Mamo G, Mattiasson B. Anaerobes in Biotechnology. Boston, MA: Springer, 2016: 293-321
|
| [32] |
Groher A, Weuster-Botz D. Comparative reaction engineering analysis of different acetogenic bacteria for gas fermentation[J]. Journal of Biotechnology, 2016, 228: 82-94. DOI:10.1016/j.jbiotec.2016.04.032 |
| [33] |
Yang YP, Lang NN, Yang GH, et al. Improving the performance of solventogenic clostridia by reinforcing the biotin synthetic pathway[J]. Metabolic Engineering, 2016, 35: 121-128. DOI:10.1016/j.ymben.2016.02.006 |
| [34] |
Boynton ZL, Bennet GN, Rudolph FB. Cloning, sequencing, and expression of clustered genes encoding beta-hydroxybutyryl-coenzyme A (CoA) dehydrogenase, crotonase, and butyryl-CoA dehydrogenase from Clostridium acetobutylicum ATCC 824[J]. Journal of Bacteriology, 1996, 178(11): 3015-3024. DOI:10.1128/jb.178.11.3015-3024.1996 |
| [35] |
Fernández-Naveira Á, Veiga MC, Kennes C. Effect of pH control on the anaerobic H-B-E fermentation of syngas in bioreactors[J]. Journal of Chemical Technology and Biotechnology, 2017, 92(6): 1178-1185. DOI:10.1002/jctb.5232 |
| [36] |
Fernández-Naveira Á, Veiga MC, Kennes C. H-B-E (hexanol-butanol-ethanol) fermentation for the production of higher alcohols from syngas/waste gas[J]. Journal of Chemical Technology and Biotechnology, 2017, 92(4): 712-731. DOI:10.1002/jctb.2017.92.issue-4 |
| [37] |
Tremblay PL, Zhang T, Dar SA, et al. The Rnf complex of Clostridium ljungdahlii is a proton-translocating ferredoxin: NAD+ oxidoreductase essential for autotrophic growth[J]. mBio, 2012, 4(1): e00406-12. |
| [38] |
Biegel E, Müller V. Bacterial Na+-translocating ferredoxin: NAD+ oxidoreductase[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(42): 18138-18142. DOI:10.1073/pnas.1010318107 |
| [39] |
Biegel E, Schmidt S, González JM, et al. Biochemistry, evolution and physiological function of the Rnf complex, a novel ion-motive electron transport complex in prokaryotes[J]. Cellular and Molecular Life Sciences, 2011, 68(4): 613-634. |
| [40] |
Hess V, Gallegos R, Jones JA, et al. Occurrence of ferredoxin: NAD+ oxidoreductase activity and its ion specificity in several gram-positive and gram-negative bacteria[J]. PeerJ, 2016, 4: e1515. DOI:10.7717/peerj.1515 |
| [41] |
Mock J, Zheng YN, Mueller AP, et al. Energy conservation associated with ethanol formation from H2 and CO2 in Clostridium autoethanogenum involving electron bifurcation[J]. Journal of Bacteriology, 2015, 197(18): 2965-2980. DOI:10.1128/JB.00399-15 |
| [42] |
Angenent LT, Richter H, Buckel W, et al. Chain elongation with reactor microbiomes: Open-culture biotechnology to produce biochemicals[J]. Environmental Science and Technology, 2016, 50(6): 2796-2810. DOI:10.1021/acs.est.5b04847 |
| [43] |
Wang SN, Huang HY, Kahnt J, et al. NADP-specific electron-bifurcating[J]. Journal of Bacteriology, 2013, 195(19): 4373-4386. DOI:10.1128/JB.00678-13 |
| [44] |
Wang SN, Huang HY, Kahnt J, et al. A reversible electron-bifurcating ferredoxin- and NAD-dependent[FeFe]-hydrogenase (HydABC) in Moorella thermoacetica[J]. Journal of Bacteriology, 2013, 195(6): 1267-1275. DOI:10.1128/JB.02158-12 |
| [45] |
Bertsch J, Parthasarathy A, Buckel W, et al. An electron-bifurcating caffeyl-CoA reductase[J]. Journal of Biological Chemistry, 2013, 288(16): 11304-11311. DOI:10.1074/jbc.M112.444919 |
| [46] |
Weghoff MC, Bertsch J, Müller V. A novel mode of lactate metabolism in strictly anaerobic bacteria[J]. Environmental Microbiology, 2015, 17(3): 670-677. DOI:10.1111/emi.2015.17.issue-3 |
| [47] |
Valgepea K, Loi KQ, Behrendorff JB, et al. Arginine deiminase pathway provides ATP and boosts growth of the gas-fermenting acetogen Clostridium autoethanogenum[J]. Metabolic Engineering, 2017, 41: 202-211. DOI:10.1016/j.ymben.2017.04.007 |
| [48] |
Noens EE, Kaczmarek MB, Monika Ż, et al. ArcD1 and ArcD2 arginine/ornithine exchangers encoded in the arginine deiminase pathway gene cluster of Lactococcus lactis[J]. Journal of Bacteriology, 2015, 197(22): 3545-3553. DOI:10.1128/JB.00526-15 |
| [49] |
Shanmugasundaram T, Wood HG. Interaction of ferredoxin with carbon monoxide dehydrogenase from Clostridium thermoaceticum[J]. Journal of Biological Chemistry, 1992, 267(2): 897-900. |
| [50] |
Schuchmann K, Müller V. A bacterial electron-bifurcating hydrogenase[J]. Journal of Biological Chemistry, 2012, 287(37): 31165-31171. DOI:10.1074/jbc.M112.395038 |
| [51] |
Richter H, Molitor B, Wei H, et al. Ethanol production in syngas-fermenting Clostridium ljungdahlii is controlled by thermodynamics rather than by enzyme expression[J]. Energy and Environmental Science, 2016, 9(7): 2392-2399. DOI:10.1039/C6EE01108J |
| [52] |
Pyne ME, Bruder M, Moo-Young M, et al. Technical guide for genetic advancement of underdeveloped and intractable Clostridium[J]. Biotechnology Advances, 2014, 32(3): 623-641. |
| [53] |
Leang C, Ueki T, Nevin KP, et al. A genetic system for Clostridium ljungdahlii: a chassis for autotrophic production of biocommodities and a model homoacetogen[J]. Applied and Environmental Microbiology, 2013, 79(4): 1102-1109. |
| [54] |
Schiel-Bengelsdorf B, Dürre P. Pathway engineering and synthetic biology using acetogens[J]. FEBS Letters, 2012, 586(15): 2191-2198. DOI:10.1016/j.febslet.2012.04.043 |
| [55] |
Utturkar SM, Klingeman DM, Bruno-Barcena JM, et al. Sequence data for Clostridium autoethanogenum using three generations of sequencing technologies[J]. Scientific Data, 2014, 2: 150014. |
| [56] |
Heap JT, Pennington OJ, Cartman ST, et al. A modular system for Clostridium shuttle plasmids[J]. Journal of Microbiological Methods, 2009, 78(1): 79-85. DOI:10.1016/j.mimet.2009.05.004 |
| [57] |
Ueki T, Nevin KP, Woodard TL, et al. Converting carbon dioxide to butyrate with an engineered strain of Clostridium ljungdahlii[J]. mBio, 2014, 5(5): e01636-14. |
| [58] |
Heap JT, Pennington OJ, Cartman ST, et al. The ClosTron: A universal gene knock-out system for the genus Clostridium[J]. Journal of Microbiological Methods, 2007, 70(3): 452-464. DOI:10.1016/j.mimet.2007.05.021 |
| [59] |
Shao LJ, Hu SY, Yang Y, et al. Targeted gene disruption by use of a group Ⅱ intron (targetron) vector in Clostridium acetobutylicum[J]. Cell Research, 2007, 17(11): 963-965. |
| [60] |
Koepke M, Liew F. Recombinant microorganism and methods of production thereof: US, 9359611[P]. 2016-06-07
|
| [61] |
Walker DJF, Koepke M. Method of producing a recombinant microorganism: US, 9315830[P]. 2016-04-19
|
| [62] |
Liew F, Martin ME, Tappel RC, et al. Gas fermentation-a flexible platform for commercial scale production of low-carbon-fuels and chemicals from waste and renewable feedstocks[J]. Frontiers in Microbiology, 2016, 7: 694. |
| [63] |
Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell, 2014, 157(6): 1262-1278. |
| [64] |
Lone BA, Karna SKL, Ahmad F, et al. CRISPR/Cas9 system: a bacterial tailor for genomic engineering[J]. Genetics Research International, 2018, 2018: 3797214. |
| [65] |
Huang H, Chai CS, Li N, et al. CRISPR/Cas9-based efficient genome editing in Clostridium ljungdahlii, an autotrophic gas-fermenting bacterium[J]. ACS Synthetic Biology, 2016, 5(12): 1355-1361. DOI:10.1021/acssynbio.6b00044 |
| [66] |
Nagaraju S, Davies NK, Walker DJF, et al. Genome editing of Clostridium autoethanogenum using CRISPR/Cas9[J]. Biotechnology for Biofuels, 2016, 9: 219. DOI:10.1186/s13068-016-0638-3 |
| [67] |
Woolston BM, Emerson DF, Currie DH, et al. Rediverting carbon flux in Clostridium ljungdahlii using CRISPR interference (CRISPRi)[J]. Metabolic Engineering, 2018, 48: 243-253. DOI:10.1016/j.ymben.2018.06.006 |
| [68] |
Banerjee A, Leang C, Ueki T, et al. Lactose-inducible system for metabolic engineering of Clostridium ljungdahlii[J]. Applied and Environmental Microbiology, 2014, 80(8): 2410-2416. DOI:10.1128/AEM.03666-13 |
| [69] |
Yang GH, Jia DC, Jin L, et al. Rapid generation of universal synthetic promoters for controlled gene expression in both gas-fermenting and saccharolytic Clostridium species[J]. ACS Synthetic Biology, 2017, 6(9): 1672-1678. |
| [70] |
Humphreys CM, Mclean S, Schatschneider S, et al. Whole genome sequence and manual annotation of Clostridium autoethanogenum, an industrially relevant bacterium[J]. BMC Genomics, 2016, 16: 1085. |
| [71] |
Nagarajan H, Sahin M, Nogales J, et al. Characterizing acetogenic metabolism using a genome-scale metabolic reconstruction of Clostridium ljungdahlii[J]. Microbial Cell Factories, 2013, 12: 118. DOI:10.1186/1475-2859-12-118 |
| [72] |
Tan Y, Liu JJ, Chen XH, et al. RNA-seq-based comparative transcriptome analysis of the syngas-utilizing bacterium Clostridium ljungdahlii DSM 13528 grown autotrophically and heterotrophically[J]. Molecular Biosystems, 2013, 9(11): 2775-2784. DOI:10.1039/c3mb70232d |
| [73] |
Whitham JM, Tirado-Acevedo O, Chinn MS, et al. Metabolic response of Clostridium ljungdahlii to oxygen exposure[J]. Applied and Environmental Microbiology, 2015, 81(24): 8379-8391. DOI:10.1128/AEM.02491-15 |
| [74] |
Jones SW, Fast AG, Carlson ED, et al. CO2 fixation by anaerobic non-photosynthetic mixotrophy for improved carbon conversion[J]. Nature Communications, 2016, 7: 12800. DOI:10.1038/ncomms12800 |
| [75] |
Koepke M, Simpson S, Liew F, et al. Recombinant microorganisms and uses therefor: US, 9994878[P]. 2018-06-12
|
| [76] |
Diner BA, Fan J, Scotcher MC, et al. Synthesis of heterologous mevalonic acid pathway enzymes in Clostridium ljungdahlii for the conversion of fructose and of syngas to mevalonate and isoprene[J]. Applied and Environmental Microbiology, 2018, 84(1): e01723-17. |
| [77] |
Liew F, Koepke M. Recombinant microorganisms that make biodiesel: US, 9347076[P]. 2016-05-24
|
| [78] |
Sreekumar S, Baer ZC, Gross E, et al. Chemocatalytic upgrading of tailored fermentation products toward biodiesel[J]. Chemsuschem, 2014, 7(9): 2445-2448. DOI:10.1002/cssc.201402244 |
| [79] |
Ulrich B, Gerda G, Matthias O, et al. Cells and method for producing acetone: US, 9453210[P]. 2016-09-27
|
| [80] |
Koepke M, Simpson S, Liew F, et al. Fermentation process for producing isopropanol using a recombinant microorganism: US, 9365868[P]. 2016-06-14
|
| [81] |
Köpke M, Gerth ML, Maddock DJ, et al. Reconstruction of an acetogenic 2, 3-butanediol pathway involving a novel NADPH-dependent primary-secondary alcohol dehydrogenase[J]. Applied and Environmental Microbiology, 2014, 80(11): 3394-3403. DOI:10.1128/AEM.00301-14 |
| [82] |
Mueller AP, Koepke M, Al-Sinawi B, et al. Microorganisms and methods for the production of ketones: US, US20150152445A1[P]. 2015-06-04
|
| [83] |
Beck ZQ, Cervin MA, Chotani GK, et al. Compositions and methods of producing isoprene and/or industrrial bio-products using anaerobic microorganisms: WO, WO2013181647A2[P]. 2013-12-05
|
| [84] |
Beck ZQ, Cervin MA, Chotani GK, et al. Recombinant anaerobic acetogenic bacteria for production of isoprene and/or industrial bio-products using synthesis gas: US, WO2014193473A1[P]. 2014-12-04
|
| [85] |
Furutani M, Uenishi A, Iwasa K. Recombinant cell and method for producing isoprene: EP, EP2940123[P]. 2018-04-25
|
| [86] |
Chen W, Liew F, Koepke M. Recombinant microorganisms and uses therefor: EP, EP2855662[P]. 2018-03-21
|
| [87] |
Kopke M, Havill A. Lanzatech's route to bio-butadiene[J]. Catalysis Reviews, 2014, 27: 7-12. |
| [88] |
Yasin M, Jeong Y, Park S, et al. Microbial synthesis gas utilization and ways to resolve kinetic and mass-transfer limitations[J]. Bioresource Technology, 2015, 177: 361-374. DOI:10.1016/j.biortech.2014.11.022 |
| [89] |
Xie BT, Liu ZY, Tian L, et al. Physiological response of Clostridium ljungdahlii DSM 13528 of ethanol production under different fermentation conditions[J]. Bioresource Technology, 2015, 177: 302-307. DOI:10.1016/j.biortech.2014.11.101 |
| [90] |
Al-Sinawi B, Chan G, Collet C, et al. Microbial alcohol production process: US, 8119378[P]. 2012-02-21
|
| [91] |
Fast AG, Schmidt ED, Jones SW, et al. Acetogenic mixotrophy: Novel options for yield improvement in biofuels and biochemicals production[J]. Current Opinion in Biotechnology, 2015, 33: 60-72. DOI:10.1016/j.copbio.2014.11.014 |
| [92] |
Charubin K, Papoutsakis ET. Direct cell-to-cell exchange of matter in a synthetic Clostridium syntrophy enables CO2 fixation, superior metabolite yields, and an expanded metabolic space[J]. Metabolic Engineering, 2019, 52: 9-19. DOI:10.1016/j.ymben.2018.10.006 |
| [93] |
Phillips JR, Klasson KT, Clausen EC, et al. Biological production of ethanol from coal synthesis gas: medium development studies[J]. Applied Biochemistry and Biotechnology, 1993, 39(1): 559-571. |
| [94] |
Kundiyana DK, Huhnke RL, Maddipati P, et al. Feasibility of incorporating cotton seed extract in Clostridium strain P11 fermentation medium during synthesis gas fermentation[J]. Bioresource Technology, 2010, 101(24): 9673-9680. DOI:10.1016/j.biortech.2010.07.054 |
| [95] |
Saxena J, Tanner RS. Effect of trace metals on ethanol production from synthesis gas by the ethanologenic acetogen, Clostridium ragsdalei[J]. Journal of Industrial Microbiology & Biotechnology, 2011, 38(4): 513-521. |
| [96] |
Simpson SD, Forster RLS, Tran PL, et al. Processes of producing alcohols: WO, WO2009022925A1[P]. 2009-02-19
|
| [97] |
Al-Sinawi B, Fung JMY, Mihalcea CD, et al. Optimised fermentation media: WO, WO2010064933A1[P]. 2010-06-10
|
| [98] |
Cotter JL, Chinn MS, Grunden AM. Influence of process parameters on growth of Costridium ljungdahlii and Clostridium autoethanogenum on synthesis gas[J]. Enzyme and Microbial Technology, 2009, 44(5): 281-288. |
 2019, Vol. 46
2019, Vol. 46