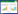扩展功能
文章信息
- 赵亚伟, 姜卫红, 邓子新, 汪志军, 芦银华
- ZHAO Ya-Wei, JIANG Wei-Hong, DENG Zi-Xin, WANG Zhi-Jun, LU Yin-Hua
- 碱基编辑器的开发及其在细菌基因组编辑中的应用
- Development and application of base editors in bacterial genome editing
- 微生物学通报, 2019, 46(2): 319-331
- Microbiology China, 2019, 46(2): 319-331
- DOI: 10.13344/j.microbiol.china.180879
-
文章历史
- 收稿日期: 2018-11-07
- 接受日期: 2018-12-13
- 网络首发日期: 2018-12-29
2. 上海交通大学生命科学技术学院 微生物代谢国家重点实验室 上海 200030;
3. 中国科学院分子植物科学卓越创新中心/植物生理生态研究所 合成生物学重点实验室 上海 200032
2. State Key Laboratory of Microbial Metabolism, School of Life Sciences and Biotechnology, Shanghai Jiao Tong University, Shanghai 200030, China;
3. CAS Key Laboratory of Synthetic Biology, Centre for Excellence in Molecular Plant Sciences/Institute of Plant Physiology and Ecology, Chinese Academy of Sciences, Shanghai 200032, China
CRISPR/Cas是原核生物的一种适应性免疫系统,用于宿主抵御外源DNA或RNA的入侵,分布于45%的细菌和85%的古细菌基因组中[1]。CRISPR/Cas发挥免疫作用主要包括3个阶段:适应、表达和干扰。在适应阶段,外源DNA或RNA序列被Cas蛋白(核酸内切酶)加工,并整合到基因组的CRISPR array序列中;接着是表达阶段,CRISPR array转录成crRNA,与相应的Cas蛋白形成复合物;最后是发挥免疫功能的干扰阶段,当外源DNA或RNA再次入侵宿主时,在crRNA的引导下,crRNA/Cas蛋白复合物对外源DNA或RNA进行干扰和免疫[2]。CRISPR/Cas系统根据是否存在PAM (Protospacer adjacent motif)序列区分宿主和外源DNA/RNA,PAM序列是Cas蛋白特异识别的一段DNA/RNA短序列,与crRNA识别的DNA/RNA序列邻近[3]。根据CRISPR/Cas的作用机制,可以将CRISPR/Cas系统分为两类:第一类,需由多个Cas蛋白组成效应复合体(Effector),在crRNA引导下,结合并降解外源入侵DNA/RNA;第二类,仅需单一的Cas蛋白就能在crRNA的引导下发挥免疫功能[4-5]。
目前研究和应用最广泛的CRISPR/Cas系统是来源于酿脓链球菌的Cas9,它属于第二类CRISPR/Cas系统,其作用机制已被研究得非常透彻,并已广泛应用于动物、植物与微生物细胞的基因组编辑,极大地促进了生命科学的发展[6]。Cas9蛋白发挥作用不仅需要crRNA引导,还需要与tracrRNA结合,三者形成一个三元复合物,对靶标DNA进行高效切割[7]。2012年,Jinek等将crRNA和tracrRNA进行融合,形成单一的sgRNA (Single guide RNA),Cas9在其引导下呈现出良好的DNA切割活性[8]。利用Cas9进行基因编辑有两种方式:(1)不精确的基因编辑,其依赖于宿主自身的非同源末端修复(NHEJ)途径,当Cas9切割基因组形成双链断裂(DSB),宿主利用NHEJ途径进行DNA修复,会引入DNA插入和缺失(Indels),Indels的引入往往使基因发生移码突变而失活;(2)精确的基因编辑,依赖于宿主自身的同源重组修复(HDR)途径,需要在Cas9切割基因组的同时引入外源同源序列。宿主利用外源同源序列与基因组进行重组,实现靶基因的精确编辑[6]。虽然HDR可以实现基因的缺失、插入、点突变等精确改造,但HDR主要发生在细胞分裂的G2期和S期,在一些分裂不活跃的细胞中,HDR效率往往不高。此外,NHEJ和HDR会相互竞争,在真核细胞中,NHEJ往往比HDR更高效地修复DNA的DSB,所以在编辑的细胞中经常会发生小片段DNA插入或缺失[9]。
Cas9蛋白有两个核酸酶结构域,分别是RuvC和HNH结构域,其中HNH结构域切割与sgRNA互补配对的DNA链,而RuvC切割另一条未与sgRNA配对的DNA链[8]。Qi等通过突变Cas9核酸酶结构域的2个活性位点(D10A与H840A),获得仅能结合DNA而无切割活性的dCas9 (Dead Cas9)。利用dCas9的这一特性,构建了CRISPR interference (CRISPRi)技术,实现sgRNA介导的靶基因转录抑制,目前该技术已被广泛应用[10]。另外,如将dCas9与具有转录激活作用的蛋白质结构域(如VP16)进行融合,可以激活靶基因的表达[11];而将dCas9与DNA甲基化酶进行融合,可以对靶基因进行甲基化修饰[12]。2016年,Komor等创造性地将dCas9与胞嘧啶核苷脱氨基酶进行融合,首次实现了精确的DNA碱基编辑,从胞嘧啶(C)到胸腺嘧啶(T)的转换[13](图 1A)。DNA碱基编辑与Cas9切割介导的基因编辑有明显的区别,前者不依赖于DNA的双链断裂,而Cas9介导的基因编辑依赖于DNA的双链断裂。此外,相比于使用Cas9进行HDR介导的精确基因编辑,碱基编辑不需要引入外源同源序列。

|
| 图 1 胞嘧啶碱基编辑器和腺嘌呤碱基编辑器的作用机理 Figure 1 The mechanism of cytosine base editing and adenine base editing 注:A:胞嘧啶核苷脱氨基酶催化的化学反应及编辑原理;B:腺嘌呤核苷脱氨基酶催化的化学反应及编辑原理. Note: A: The chemical reaction catalyzed by the cytidine deaminase and the mechanism of cytosine base editing; B: The chemical reaction catalyzed by the adenosine deaminase and the mechanism of adenine base editing. |
|
|
目前,已开发的DNA碱基编辑器有两种,一种是上面提及的胞嘧啶碱基编辑器(Cytosine base editor,CBE),依赖于胞嘧啶核苷脱氨基酶,通过将胞嘧啶核苷脱氨转换为尿嘧啶核苷,尿嘧啶核苷在DNA复制和修复过程中会转换为胸腺嘧啶核苷,从而实现C到T的转换[13](图 1A)。另一种是腺嘌呤碱基编辑器(Adenine base editor,ABE),依赖于腺嘌呤核苷脱氨基酶,通过将腺嘌呤核苷脱氨转换为次黄苷,然后在DNA复制和修复过程中会转换为鸟嘌呤核苷,从而实现腺嘌呤(A)到鸟嘌呤(G)的转换[14](图 1B)。这两种DNA碱基编辑器可以实现所有四种碱基的转换,包括C到T,A到G,T到C以及G到A[15]。除了DNA碱基编辑器以外,目前还开发了CRISPR/Cas系统辅助的RNA碱基编辑器,将Cas13与腺嘌呤核苷脱氨基酶融合为复合体,可以将目标腺嘌呤核苷脱氨转换为次黄苷[16]。碱基编辑器常用的蛋白主要包括:胞嘧啶核苷脱氨基酶、腺嘌呤核苷脱氨基酶、CRISPR/Cas蛋白(如第二类Ⅱ型的Cas9、Ⅴ型的Cpf1)、尿嘧啶DNA糖苷酶抑制子、DNA末端结合蛋白Gam (表 1)。本文将就DNA和RNA碱基编辑器的最新研究进展进行简单综述,并简要介绍其在细菌基因组编辑研究中的应用。
| 蛋白 Protein |
简写 Abbreviation |
物种 Species |
功能或特点 Function or characteristics |
| 胞嘧啶核苷脱氨基酶Cytidine deaminase | rAPOBEC1 | Rattus norvegicus | 对编辑位点具有偏好性,对TC或CC位点的编辑效率较高 Base editing exhibit sequence context preference and the efficiency is higher for TC or CC sites |
| hAPOBEC3A | Homo sapiens | 可以对甲基化区域的胞嘧啶进行编辑 Edit cytosine found in methylated DNA regions |
|
| PmCDA1 | Petromyzon marinus | 没有明显编辑位点偏好性 No strong context preference for base editing |
|
| hAID*P182X | Homo sapiens | 编辑过程会使胞嘧啶转换为其它3种碱基 Convert cytosine to other three bases |
|
| 腺嘌呤核苷脱氨基酶 Adenosine deaminase |
TadA* | Escherichia coi | 由tRNA腺嘌呤核苷脱氨酶TadA改造得到的突变体,可以将DNA上的腺嘌呤核苷转换为次黄苷 Evolved from tRNA adenosine deaminase TadA, could convert adenosine to inosine in DNA |
| ADAR2 | Homo sapiens | 将RNA上的腺嘌呤核苷转换为次黄苷 Convert adenosine to inosine in RNA |
|
| Sp dCas9 | Streptococcus pyogenes | 识别NGG PAM位点,不切割DNA链 Recognize NGG PAM site and do not cut DNA |
|
| Cas9 | Sp nCas9 (D10A) | Streptococcus pyogenes | 识别NGG PAM位点,仅切割与sgRNA互补的DNA链 Recognize NGG PAM site and nick the DNA strand complementary to sgRNA |
| Sa nCas9 (D10A) | Staphylococcus aureus | 识别NNGRRT PAM位点,仅切割与sgRNA互补的DNA链 Recognize NNGRRT PAM site and nick the DNA strand complementary to sgRNA |
|
| Cpf1 | dLbCpf1 | Lachnospiraceae bacterium | 识别TTTV PAM位点,不切割DNA链 Recognize TTTV PAM site and do not cut DNA |
| 尿嘧啶DNA糖苷酶抑制子 Uracil DNA glycosylase inhibitor |
UGI | Bacteriophage PBS1 | 抑制尿嘧啶DNA糖苷酶的活性,抑制尿嘧啶的切除 Repress the activity of uracil DNA glycosylase and thereby inhibit the removal of uracil in DNA |
| DNA末端结合蛋白Gam DNA end binding Gam protein |
Gam | Bacteriophage Mu | 结合于DNA双链断裂的切口,减少Indel的形成 Bind to the ends of DSBs and reduce indel formation |
CBE可以实现C到T的转换,由Komor等率先开发[13]。他们将dCas9与鼠源APOBEC1脱氨基酶进行融合,开发了第一代碱基编辑器BE1。当dCas9和sgRNA形成的复合物与目标DNA结合时,会打开DNA双链结构,置换出一条单链DNA;而APOBEC1只能以单链DNA作为底物,不能以双链DNA作为底物[17]。BE1可以有效地对单链DNA上的碱基进行编辑,编辑窗口约为5 bp,位于PAM序列上游−13到−17。虽然BE1在体外可以对胞嘧啶进行有效编辑,效率约为25%−40%,但其在体内的编辑效率却很低,仅为0.8%−7.7%,限制该技术在体内的应用。作者推测可能是体内的DNA修复机制导致编辑效率下降。前期研究表明,当DNA双链上出现尿嘧啶时,尿嘧啶N-糖苷酶(Uracil N-glycosylase,UNG)就会启动碱基切除修复机制,将尿嘧啶切除,进而修复DNA。为了抑制UNG的活性,Komor等将来源于噬菌体PBS的UNG抑制子UGI (Uracil DNA glycosylase inhibitor)[18]引入BE1,构建了第二代碱基编辑器BE2[13]。结果显示,BE2可以在细菌中进行有效地碱基编辑,但在哺乳动物细胞中编辑效率仍不高。为了进一步提高编辑效率,他们将BE2中的dCas9替换为nCas9(D10A),开发了第三代碱基编辑器BE3[13]。nCas9(D10A)可以在未被编辑的DNA单链引入切口,DNA倾向于以已发生编辑的DNA单链作为模板进行修复,使目标碱基的编辑效率提高2−6倍,在哺乳动物细胞中编辑效率达到37%左右。值得一提的是,虽然BE3相较于BE2编辑效率明显提高,但是Indel出现的频率也明显提高,说明该版本的碱基编辑器仍需进一步优化完善。
与此同时,Nishida等采用来自七鳃鳗的胞嘧啶核苷脱氨基酶PmCDA1与nCas9(D10A)进行融合,在酵母和哺乳动物细胞中开发了类似的CBE,命名为Target-AID[19]。由于使用了不同的胞嘧啶核苷脱氨基酶以及融合蛋白的组织结构不同,Target-AID与BE3存在以下几点不同:(1)编辑窗口与BE3明显不同,Target-AID编辑窗口位于PAM序列的−15到−19;(2)编辑位点邻近的碱基对编辑效率的影响不同,编辑位点邻近的碱基对BE3的编辑效率有很大影响。例如,某些编辑位点胞嘧啶的5'端为鸟嘌呤时,BE3的编辑效率较低;而Target-AID对编辑位点邻近的碱基没有明显的偏好性;(3) Target-AID在某些位点会将胞嘧啶转换为鸟嘌呤,且突变频率与胞嘧啶的位置有关;当胞嘧啶距离PAM越近时,突变为鸟嘌呤的频率越高。BE3也存在胞嘧啶突变为鸟嘌呤的现象,但突变频率与胞嘧啶的位置无关[19]。
为了使胞嘧啶高效率转换为胸腺嘧啶,而不是鸟嘌呤和腺嘌呤,Komor等在BE3的基础上,通过优化APOBEC1与nCas9(D10A)的Linker区长度、nCas9(D10A)与UGI的Linker区长度以及增加一个拷贝的UGI,获得了第四代碱基编辑器BE4[20]。相比于BE3,BE4的编辑效率提高了1.5倍,胞嘧啶转换为鸟嘌呤和腺嘌呤的频率与Indel出现的频率均降低230%。由于Indel的出现可能破坏基因的功能,Komor等在BE4的氨基端引入来自Mu噬菌体的Gam蛋白,构建了BE4-Gam。Gam蛋白可以结合在DNA双链断裂的末端,保护DNA不被降解。相比于BE4,BE4-Gam使Indel出现的频率再降低210%。
当编辑窗口存在多个胞嘧啶时,胞嘧啶核苷脱氨基酶会使其它非目标胞嘧啶也发生突变。为了减少这种情况的出现,Kim等又在BE3的基础上开发了编辑窗口更小的碱基编辑器,他们将BE3中的APOBEC1进行点突变,筛选到的突变体使编辑窗口减小到1−2 bp,从而显著增加了碱基编辑的精确性[21]。此外,现有碱基编辑器在识别编辑单链DNA上的胞嘧啶时需依赖PAM序列的存在,但往往很多编辑位点的附近没有合适的PAM序列。为了解决这一问题,Kim等将BE3中的nCas9(D10A)引入各种点突变,或替换为来源于金黄色葡萄球菌的Sa nCas9(D10A),改良版BE3识别的PAM序列更加多样,进而增加了DNA上的可编辑位点(约2.5倍)[21]。Hu等采用噬菌体辅助的连续进化(PACE)技术,筛选到可以识别更多PAM序列的Cas9,命名为xCas9[22]。将BE3中的nCas9(D10A)替换为xCas9(D10A)构建的xBE3,可以有效识别并编辑附近存在PAM序列为NGN、GAA、GAT的胞嘧啶碱基。xCas9不仅可以识别更多的PAM,而且它的使用还进一步降低了编辑的脱靶率[22]。Nishimasu等进行了类似的研究工作,他们采用理性设计的方法获得了一种Cas9突变体,命名为NG-Cas9[23]。基于NG-dCas9的编辑器可以有效编辑某些PAM序列为NG附近的胞嘧啶位点[23]。最近,研究发现DNA的甲基化修饰会降低BE3的编辑效率,Wang等将鼠源的APOBEC1替换为来自人类的APOBEC3A (hA3A),构建的hA3A-BE3可以明显减少DNA甲基化对编辑效率的影响,实现对甲基化胞嘧啶的有效编辑[24]。Ma等将Human activation-induced deaminase (hAID)的C端去除得到AIDx,将dCas9与AIDx进行融合开发了一种Targeted AID-mediated mutagenesis (TAM),这种方法可以将胞嘧啶转化为其它3种碱基[25]。此外,Gehrke等开发的eA3A-BE3可以在不改变编辑窗口的情况下,优先编辑“TC”位点中的胞嘧啶,这种方法使碱基编辑更加精准[26](表 2)。以上研究工作,尤其是哈佛大学David R. Liu实验室的工作[13, 20-22],一方面显著提升了CBE的效率以及精确性,另一方面也大幅度增加了基因组上的可编辑位点,这些努力为其应用于哺乳动物、植物细胞基因组的精准编辑打下了良好的基础。
| 胞嘧啶碱基编辑器 Cytosine base editor |
组织结构 Architecture |
编辑窗口 Editing window |
PAM | 参考文献 References |
| BE1 | rAPOBEC1-Sp dCas9 | −13–−17 | NGG | [13] |
| BE2 | rAPOBEC1-Sp dCas9-UGI | −13–−17 | NGG | [13] |
| BE3 | rAPOBEC1-Sp nCas9(D10A)-UGI | −13–−17 | NGG | [13] |
| Target-AID | Sp nCas9(D10A)-PmCDA1-UGI | −15–−19 | NGG | [19] |
| BE4 | rAPOBEC1-Sp nCas9(D10A)-UGI-UGI | −13–−17 | NGG | [20] |
| BE4-Gam | Gam-rAPOBEC1-Sp nCas9(D10A)-UGI-UGI | −13–−17 | NGG | [20] |
| SaBE3 | rAPOBEC1-Sa nCas9(D10A)-UGI | −9–−18 | NNGRRT | [21] |
| Sa(KKH)-BE3 | rAPOBEC1-Sa KKH nCas9(D10A)-UGI | −9–−18 | NNNRRT | [21] |
| VQR-BE3 | rAPOBEC1-Sp VQR nCas9(D10A)-UGI | −10–−17 | NGAN | [21] |
| VRER-BE3 | rAPOBEC1-Sp VRER nCas9(D10A)-UGI | −11–−18 | NGCG | [21] |
| YE1-BE3 | rAPOBEC1 W90Y R126E-Sp nCas9(D10A)-UGI | −14–−17 | NGG | [21] |
| YE2-BE3 | rAPOBEC1 W90Y R132E-Sp nCas9(D10A)-UGI | −15–−16 | NGG | [21] |
| EE-BE3 | rAPOBEC1 R126E R132E-Sp nCas9(D10A)-UGI | −15–−16 | NGG | [21] |
| YEE-BE3 | rAPOBEC1 W90Y R126E R132E-Sp nCas9(D10A)-UGI | −15–−16 | NGG | [21] |
| SaBE4 | rAPOBEC1-Sa nCas9(D10A)-UGI-UGI | −9–−18 | NNGRRT | [20] |
| SaBE4-Gam | Gam-rAPOBEC1-Sa nCas9(D10A)-UGI-UGI | −9–−18 | NNGRRT | [20] |
| xBE3 | rAPOBEC1-Sp xCas9(D10A)-UGI | −13–−17 | NG | [22] |
| Target-AID-NG | Sp nCas9(D10A)-NG-PmCDA1-UGI | −13–−19 | NG | [23] |
| TAM | Sp dCas9-hAID*-P182X | −12–−16 | NGG | [25] |
目前,除了Cas9被广泛应用于基因组编辑以外,属于第二类Ⅴ型CRISPR/Cas系统的Cpf1 (又称Cas12a)也被开发用于基因组编辑。最近,Li等将来源于Lachnospiraceae bacterium切割活性丧失的Cpf1 (dLbCpf1)与APOBEC1和UGI进行融合,构建了dLbCpf1-BE0,可以对PAM下游8−13 bp的胞嘧啶进行编辑,编辑效率约为20%−22%,且Indel出现的频率和脱靶率都很低[27]。该编辑器的开发为细胞基因组的编辑(C到T或G到A碱基的转换)提供了一个备选工具。
1.2 腺嘌呤碱基编辑器(ABE)ABE是由Gaudelli等开发,他们首先提出了一个假设,如将DNA双链上的腺嘌呤核苷脱氨基后可生成次黄苷,次黄苷在DNA复制和修复过程中能与胞嘧啶核苷配对,进而实现腺嘌呤到鸟嘌呤的转换[14](图 1B)。如果将CBE中的胞嘧啶核苷脱氨基酶替换为腺嘌呤核苷脱氨基酶,就可以开发出相应的ABE。然而,目前尚未发现以单链DNA作为底物的腺嘌呤核苷脱氨基酶。所以,开发ABE的最大困难就是寻找以单链DNA作为底物的腺嘌呤核苷脱氨基酶[15]。已经报道的腺嘌呤核苷脱氨基酶的作用底物包括:游离的腺嘌呤、游离的腺嘌呤核苷、RNA中的腺嘌呤核苷、RNA和DNA的异源二聚体中错配的腺嘌呤核苷[28]。Gaudelli等[14]首先尝试将BE3中的APOBEC1替换为已报道的腺嘌呤核苷脱氨基酶,包括:大肠杆菌来源的TadA[29-30]、人类来源的ADAR2[31]/ADAT2[32]与小鼠来源的ADA[33],但均不能实现腺嘌呤到鸟嘌呤的转换。作者推测这些酶都不能以单链DNA作为底物进行脱氨基反应,于是希望通过人工改造的方法获得可以作用于DNA的腺嘌呤核苷脱氨基酶。选择大肠杆菌来源的TadA作为改造的靶点,主要出于以下三方面的考虑:(1) TadA是一个tRNA腺嘌呤核苷脱氨基酶,可以将tRNAArg反密码子环上单链RNA中的腺嘌呤核苷脱氨基生成次黄苷。TadA和APOBEC1有一定的同源性,并且TadA与tRNA结合的构象和APOBEC1结合单链DNA的构象类似[17, 34];(2) TadA催化的脱氨基反应不需要小分子激活剂[35];(3) TadA作用的底物是核酸链上的腺嘌呤核苷,而不是游离的腺嘌呤核苷和腺嘌呤[33]。作者首先构建了一个TadA的突变体(TadA*)库,将其与dCas9进行融合后,导入大肠杆菌中进行筛选。为了便于筛选,作者将氯霉素酰基转移酶(CmR)的第193位活性位点组氨酸突变为酪氨酸,导入大肠杆菌中作为筛选压力。只有当突变的TadA*-dCas9和sgRNA使活性位点的腺嘌呤转换为鸟嘌呤,才能使菌株恢复对氯霉素的抗性。经过第一轮筛选,找到了2个关键的TadA点突变A106V和D108N。将突变的TadA*与密码子优化的nCas9(D10A)进行融合,并引入核定位信号(NLS),构建出第一版腺嘌呤碱基编辑器ABE1.2 (TadA*-XTEN-nCas9-NLS)。然而,ABE1.2在哺乳动物细胞中的编辑效率非常低,仅为3.2%;通过使用更高浓度的氯霉素进行第二轮筛选,找到2个新的TadA点突变D147Y和E155V,将这2个点突变引入ABE1.2得到ABE2.1 (ABE1.2+D147Y+ E155V),编辑效率提高至11%。由于原始的TadA是以同源二聚体形式发挥功能,其中一个单体催化脱氨基反应,另一个单体结合tRNA底物[36]。于是,作者在ABE2.1的氨基端引入另一个TadA*得到ABE2.9,编辑效率提高至20%。为了进一步提高编辑效率,作者进行了第三轮筛选,找到3个新的点突变位点L84F、H123Y和I157F。将这些点突变引入ABE2.9得到ABE3.1,编辑效率提高至29%。然而,ABE3.1偏好编辑YAC位点的腺嘌呤,对某些位点(如GAG)的编辑效率比较低。为了消除ABE3.1的偏好性,作者进行了第四轮和第五轮筛选,找到4个点突变H36L、R51L、S146C和K157N。将其引入ABE3.1得到ABE5.1,但编辑效率反而降低170%,作者推测这些点突变的引入可能破坏了负责底物供应的氨基端TadA的结构,于是将ABE5.1中氨基端TadA*替换为原始的TadA得到ABE5.3,编辑效率提高至39%,且在非YAC位点编辑效率明显提高。为了去除不利点突变并提高编辑效率,作者再次进行了第六、第七轮筛选,分别得到ABE6.3和ABE7.10,编辑效率分别提高至47%与58%。ABE7.10的编辑窗口为PAM序列上游的−14到−17,且对含有多个腺嘌呤位点的编辑效率明显提高。此外,相比于使用Cas9进行的基因编辑,ABE7.10的脱靶率更低,Indel出现的频率也明显降低。
与CBE类似,通过替换ABE7.10中的nCas9(D10A),开发了多种其它的ABE碱基编辑工具,如xABE[22]、ABESa[37]、Sa(KKH)-ABE[38]等(表 3)。这些ABE工具可以识别不同的PAM位点,且编辑窗口有所差异,使基因组中可被编辑的位点进一步增多。
与CBE和ABE依赖于CRISPR/Cas系统进行DNA的碱基编辑不同,许多RNA碱基编辑器不依赖于CRISPR系统,而是利用已经报道的脱氨基酶的特性进行编辑。研究较多的RNA脱氨基酶是ADAR (Adenosine deaminase acting on RNA)家族蛋白[39-40]。ADAR可以催化RNA链上的腺嘌呤核苷转换为次黄苷,次黄苷在翻译和RNA剪接过程中会被误读为鸟嘌呤核苷,进而实现RNA的碱基编辑[39-40]。ADAR蛋白有2个结构域,氨基端的双链RNA结合结构域和羧基端的脱氨基酶结构域,脱氨基酶结构域的底物是RNA双链结构中的腺嘌呤核苷[31, 41]。人类基因组中有2个ADAR蛋白:ADAR1和ADAR2。此外,ADAR的关键活性位点谷氨酸突变为谷氨酰胺,可提高ADAR的碱基编辑效率[28]。ADAR对RNA双链结构中腺嘌呤核苷与胞嘧啶核苷的错配(A-C错配)编辑效率较高[42]。利用ADAR的这些特点,开发了许多RNA碱基编辑器,可大致分为两类:反义RNA介导的碱基编辑器和CRISPR介导的碱基编辑器。
反义RNA介导的碱基编辑器主要采用3种策略:(1)将ADAR结合的双链RNA结构与反义RNA进行融合,利用反义RNA靶向目标RNA,并使需要突变的腺嘌呤核苷与反义RNA中的胞嘧啶核苷配对,形成A-C错配,从而提高编辑效率[43];(2)将ADAR的脱氨基酶结构域(ADARDD)与SNAP标签(改造的O6-烷基鸟嘌呤-DNA-烷基转移酶,具有与5′-O-苄基鸟嘌呤修饰的RNA结合的活性)进行融合[44],形成SNAP-ADARDD融合蛋白,并将反义RNA的5′端用苄基鸟嘌呤进行修饰,利用SNAP标签和苄基鸟嘌呤的特异性结合,将ADARDD引导至反义RNA和目标RNA形成的双链区进行碱基编辑[45-46];(3)将λ噬菌体的N蛋白与ADARDD进行融合,并将反义RNA与17 nt的BoxB发卡结构进行融合,利用λ噬菌体的N蛋白与BoxB发卡结构的相互作用将ADARDD引导至目标位点进行编辑[47-48]。然而,由于反义RNA介导的碱基编辑仅仅依赖于反义RNA和目标RNA的碱基互补配对进行识别和编辑,所以其特异性往往较低。此外,反义RNA介导的碱基编辑无法克服ADAR的底物偏好性,对某些位点(如GAN位点)的编辑效率较低[16]。
最近,Cox等开发了一种CRISPR/Cas介导的RNA碱基编辑器REPAIR (RNA editing for Programmable A to I Replacement)[16]。与反义RNA介导的碱基编辑不同,REPAIR系统利用失活的Cas13蛋白的辅助进行识别,不仅可以编辑更多的位点,特异性也得到明显提升。Cas13蛋白属于第二类第Ⅵ型CRISPR核酸酶,具有RNA核酸内切酶结构域,能够在RNA引导下切割RNA,主要包括3个家族:Cas13a、Cas13b和Cas13c[5]。Cox等[16]通过筛选不同来源的Cas13蛋白,发现来自Prevotella sp. P5-125的Cas13b (PspCas13b)对靶基因的干扰活性较强,且特异性较好。于是,将失活的PspCas13b蛋白(dPspCas13b)与高活性的ADAR2DD(E488Q)进行融合,构建了第一版的RNA碱基编辑器REPAIRv1,编辑效率高,但脱靶率也很高。作者推测是由于ADAR2DD(E488Q)的活性过强所致。随后,他们通过结构指导的蛋白改造策略对REPAIRv1系统中的ADAR2DD(E488Q)进行点突变筛选,双位点突变体ADAR2DD(E488Q+T375G)的脱靶率大幅降低(接近3个数量级),命名为REPAIRv2。相对于反义RNA介导的碱基编辑,REPAIRv2特异性大幅提升,脱靶率降低一个数量级以上[16];不过,与REPAIRv1相比,它的靶向编辑活性明显下降。尽管如此,由于REPAIRv2具有高特异性、低脱靶率的优势,它将是一种非常有应用潜力的RNA编辑工具。
3 碱基编辑器在细菌基因组编辑研究中的应用鉴于碱基编辑器介导的基因组编辑技术操作简便、特异性高,因此倍受科研工作者的青睐。目前该技术已被广泛应用于人类疾病相关的点突变修复研究或治疗、植物分子育种、疾病动物模型的建立与高通量功能基因组的研究等[49-57]。最近,CBE碱基编辑器也被应用于细菌的基因组编辑[58],然而这方面的应用研究尚处于起步阶段,目前仅在以下一些细菌中得到了应用,包括大肠杆菌[59-60]、谷氨酸棒状杆菌[61],以及多个致病菌,如金黄色葡萄球菌[62]、肺炎克雷伯菌[63]、假单胞菌[64]和羊布鲁氏杆菌[60](表 4)。下面进行简单介绍。
| 细菌 Bacteria |
质粒 Plasmid |
组织结构 Architecture |
编辑窗口 Editing window |
多位点编辑效率 Multiple site editing efficiency |
参考文献 References |
| Escherichia coli | pScI-dCas9-CDA-UL | Sp dCas9-PmCDA1-UGI-LVA | −16–−20 | 3 sites (62.5%) 6 sites (87.5%) | [59] |
| Escherichia coli | pEcBE3 | rAPOBEC1-Sp nCas9(D10A)-UGI-NLS | ND | ND | [60] |
| Corynebacterium glutamicum | pnCas9(D10A)-AIDTS | Sp nCas9(D10A)-PmCDA1 | −16–−20 | 2 sites (85.0%) 3 sites (23.3%) | [61] |
| Staphylococcus aureus | pnCasSA-BEC | rAPOBEC1-Sp nCas9(D10A) | −13–−17 | ND | [62] |
| Pseudomonas | pnCasPA-BEC | rAPOBEC1-Sp nCas9(D10A) | −13–−18 | ND | [64] |
| Klebsiella pneumoniae | pBECKP | rAPOBEC1-Sp nCas9(D10A) | −13–−18 | ND | [63] |
| Brucella melitensis | pBmBE3 | rAPOBEC1-Sp nCas9(D10A)-UGI-NLS | ND | ND | [60] |
| 注:ND:编辑窗口或效率没有测定. Note: ND: The editing window or efficiency was not determined. |
|||||
Banno等将之前在哺乳动物细胞和酵母中研发的Target-AID系统应用于大肠杆菌,开发了基于CBE的多重基因组编辑技术[59]。开始,他们尝试使用nCas9(D10A)-PmCDA1进行碱基编辑,但因其对大肠杆菌存在较大毒性而改用dCas9-PmCDA1 (简写为dCas9-CDA)。结果显示,dCas9-CDA可以实现大肠杆菌的基因组编辑(C到T),但编辑效率差异较大,有些位点的编辑效率高达61.7%−95.1%,而有些位点的效率仅为1.4%−9.2%,最佳编辑窗口为5 bp (在PAM序列上游−16到−20 bp)。为了提高编辑效率,作者在dCas9-CDA的C端融合了UNG抑制子UGI以及蛋白降解标签LVA两种元件,构建了编辑器dCas9-CDA-UL。LVA的引入可以降低dCas9-CDA-UGI过量表达造成的细胞毒性[65]。结果显示,改良版的编辑器可以对所有测试的靶点进行高效编辑。同时,他们发现调整sgRNA的长度可以使编辑窗口发生改变;当sgRNA延长时,编辑窗口会向远离PAM的5'端偏移。通过全基因组测序得知,dCas9-CDA-UL的脱靶率会高于dCas9-CDA,且脱靶位点没有明显的位置偏好性。随后,作者开展多位点编辑的尝试,结果显示当3个位点同时编辑时,dCas9-CDA无法一次获得三位点突变株,但dCas9-CDA-UL可以一次获得三位点突变株,而且效率高达62.5%;当运用dCas9-CDA-UL进行6个位点的同时突变,效率更是高达87.5%。预示基于dCas9-CDA-UL的碱基编辑技术非常高效。最后,他们使用4个sgRNA引导dCas9-CDA-UL靶向多拷贝的转座酶编码基因(分别是10、12、5与14个拷贝,总计41个位点),实现一次对41个位点胞嘧啶的碱基编辑,充分证明dCas9-CDA-UL碱基编辑器的高效率[59]。该dCas9-CDA-UL介导的基因组编辑技术适合用于多重及高通量基因突变,并且同样适用于其他细菌的基因组编辑,甚至包括遗传操作困难的细菌。因此,它的广泛使用必将极大推动细菌的功能基因组研究。另外,值得一提的是,Gandelli等在开发适用于哺乳动物细胞的ABE过程中,对腺嘌呤核苷脱氨基酶TadA的改造均是在大肠杆菌中完成[14],证明ABE系统也可以在大肠杆菌中有效发挥作用。
3.2 谷氨酸棒杆菌之前的研究工作中,科研人员已在重要工业微生物谷氨酸棒状杆菌建立了基于CRISPR/Cas9或Cpf1的基因组编辑技术,但这些技术的多基因同步编辑能力较弱,一次最多只能编辑2个基因[66-68]。最近,来自中国科学院天津工业生物技术研究所的研究团队开发了一种多重、自动化的碱基编辑器MACBETH (Multiplex automated Corynebacterium glutamicum base editing method)[61]。他们首先将dCas9(D10A/H840A)与PmCDA1进行融合,构建了CBE编辑器dCas9-AID,其编辑效率仅约11.2%。为了提高编辑效率,作者将编辑器中的dCas9替换成nCas9(D10A),构建了nCas9(D10A)-AID,其编辑效率显著提升,达到62.4%±8.2%。随后,他们通过连续传代,延长编辑时间,进一步提高了编辑效率,单基因的编辑效率最高可达90%−100% (3−5代);两基因同时编辑效率达到85% (5代),三基因同时编辑效率达到23.3% (8代)。在该研究中,他们还将碱基编辑系统用于谷氨酸棒杆菌的代谢工程改造,通过同时失活pyk和ldhA基因使谷氨酸产量提高3倍。最后,作者将碱基编辑器与自动化系统进行整合,运用MACBETH技术成功实现了94个转录因子的高通量失活(引入终止密码子),其中超过86%靶基因的编辑效率(C到T的转换)超过50%。该技术与之前谷氨酸棒杆菌中建立的基于CRISPR/Cas9或Cpf1编辑技术相比,更加简便、高效,并且编辑通量更高。
3.3 金黄色葡萄球菌、肺炎克雷伯氏菌等致病菌为加速病原菌的相关研究,包括致病与耐药机制、新型药靶筛选等,开发适用于病原菌的高效快速基因组编辑技术十分重要。最近,来自上海科技大学的Ji研究团队在多种病原菌或重要工业微生物中开发了胞嘧啶碱基编辑器[62-64]。他们将鼠源的APOBEC1 (位于N端)与nCas9(D10A) (C端)通过XTEN linker进行融合,构建CBE编辑系统pnCasSA-BEC。运用该编辑系统,在金黄色葡萄球菌临床分离株中,如ST239 (770)和ST398 (1059),实现了高效的单基因编辑,效率最高可达100%。由于金黄色葡萄球菌的同源重组效率较低,限制了CRISPR系统介导的基因缺失技术的应用。因此,该技术的建立必将极大地改善致病性金黄色葡萄球菌的遗传操作[62]。采用相同的策略,他们又相继在多种假单胞菌(包括绿脓假单胞菌、恶臭假单胞菌、荧光假单胞菌和丁香假单胞菌)中开发了基于pnCasPA-BEC的基因编辑技术[64]。Wang等在肺炎克雷伯菌(既是一种重要工业微生物,同时也是一种人类病原菌)中开发类似的CBE编辑系统pBECKP,实现了高效的碱基编辑[63]。此外,来自华中农业大学的Zheng等将在哺乳动物细胞中开发的BE3编辑器应用于大肠杆菌和羊布鲁氏杆菌(Brucella melitensis)的单基因编辑[60],效率也高达100%。这些基于CBE的高效基因编辑技术的开发大幅提高了相关病原菌的基因组改造效率,必将推动病原菌致病与耐药机制、药靶筛选的研究工作。
3.4 链霉菌在重要工业微生物链霉菌中已相继开发了基于CRISPR/Cas9或Cpf1的高效基因组编辑技术,可以实现单基因/双基因、单基因簇/双基因簇以及定点突变等形式的基因组高效编辑。这些技术的研发大幅提高了链霉菌基因组的编辑效率,极大促进了此类细菌的功能基因组以及代谢工程的研究[69-74]。但总体来看,这些技术的通量相对较低,一般情况下一次遗传操作仅能完成1−2个基因或位点的缺失或突变;另外,这些技术需要依赖于HDR或NHEJ进行DSB的修复,进而完成基因的缺失或突变,所以现有技术并不适用于DNA同源重组能力弱的链霉菌的基因组编辑。为此,本实验室开发了适用于链霉菌基因组多重编辑的胞嘧啶碱基编辑器,并在天蓝色链霉菌(模式菌)与雷帕链霉菌(免疫抑制剂-雷帕霉素产生菌)中进行了功能及效率测试,证实该技术可以在这两种链霉菌很好地发挥功能,实现基因组的高效多重编辑。该技术的研发为链霉菌的基因组编辑提供了一种很好的替代工具;同时,由于碱基编辑器介导的基因组改造不依赖于同源重组,因此该方法的建立必将推动DNA重组能力较弱链霉菌中功能基因的筛选鉴定以及代谢工程研究。
4 总结与展望碱基编辑器分成两大类,分别是DNA和RNA碱基编辑器,它们可以对生物体遗传信息(DNA或RNA)进行高效、高分辨率地精准改造(单一碱基对)。与CRISPR/Cas系统介导的基因改造不同,碱基编辑器不依赖于宿主细胞自身的NHEJ和HDR途径,而是借助于脱氨基酶的催化活性实现碱基的转换,进而完成基因编辑[50-51]。DNA碱基编辑器有2种,分别是将DNA上C∙G碱基对转换成T∙A的CBE和将A∙T转换成G∙C的ABE;而RNA碱基编辑器在RNA水平上实现腺嘌呤核苷到次黄苷的转换,可以在不改变DNA的情况下,实现基因的可逆编辑[15]。目前,碱基编辑器在真核生物中已被广泛应用,主要用于人类疾病相关的点突变修复研究或治疗、植物分子育种、疾病动物模型的建立、高通量功能基因组研究等[49-57]。两年来,针对碱基编辑器(尤其是CBE)存在的一些问题,包括编辑效率低、可编辑位点少以及脱靶效应等,科研人员开展了卓有成效的研究工作,通过各种策略,包括优化Cas蛋白或脱氨基酶(理性设计或连续进化等)、引入UNG抑制子UGI、优化Cas蛋白与脱氨基酶之间连接区的长度、控制碱基编辑器的作用时间等,获得多种改良版的CBE。与第一版本的BE1相比,它们呈现出编辑效率高、可编辑位点多、脱靶效应低等优势。为最终用于点突变引起的人类遗传疾病的精准治疗奠定了良好的基础。
在原核生物中,碱基编辑器的开发应用尚处于起步阶段,至今仅在少数一些细菌中得到了应用,包括大肠杆菌[59-60]、谷氨酸棒状杆菌[61],以及多个致病菌,如金黄色葡萄球菌[62]、肺炎克雷伯菌[63]、假单胞菌[64]与羊布鲁氏杆菌[60]等,而且使用的都是CBE。通过CBE在基因内部引入终止密码子,引起基因表达终止及蛋白功能的失活,以此来进行功能基因的鉴定以及代谢工程研究。该技术同样适用于其他细菌的基因组编辑,尤其可以为同源重组效率低下、遗传操作困难的细菌的基因组编辑提供很好的解决方案。由于碱基编辑器存在简单、高效率、高特异性以及低脱靶率等优势,相信在不久的将来必将在更多细菌中建立碱基编辑器介导的基因组编辑技术,进而促进众多细菌的功能基因组研究。必须指出的是,从现有结果来看,细菌中使用的CBE的脱靶率相对较高,将来可以通过优化sgRNA设计以及使用更加严谨的CRISPR/Cas系统,或通过蛋白质工程等来优化碱基编辑器,以达到降低脱靶率的效果。另外,在细菌中ABE及RNA编辑器还有待开发,这些技术的发展将进一步丰富现有细菌基因组编辑的工具箱,从而为细菌功能基因组的研究提供更多备选方案。最后,从编辑通量来看,在大肠杆菌中开发的dCas-CDA-UL通量最高,一次可以同时对6个位点进行高效的碱基编辑[59]。如能在众多细菌中开发更高通量的碱基编辑器将进一步促进功能基因组研究,而且将为工业微生物的分子育种提供强大的基因突变工具。
| [1] |
McGinn J, Marraffini LA. Molecular mechanisms of CRISPR-Cas spacer acquisition[J]. Nature Reviews Microbiology, 2019, 17(1): 7-12. DOI:10.1038/s41579-018-0071-7 |
| [2] |
Knott GJ, Doudna JA. CRISPR-Cas guides the future of genetic engineering[J]. Science, 2018, 361(6405): 866-869. DOI:10.1126/science.aat5011 |
| [3] |
Shah SA, Erdmann S, Mojica FJM, et al. Protospacer recognition motifs: mixed identities and functional diversity[J]. RNA Biology, 2013, 10(5): 891-899. DOI:10.4161/rna.23764 |
| [4] |
Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2015, 13(11): 722-736. DOI:10.1038/nrmicro3569 |
| [5] |
Koonin EV, Makarova KS, Zhang F. Diversity, classification and evolution of CRISPR-Cas systems[J]. Current Opinion in Microbiology, 2017, 37: 67-78. DOI:10.1016/j.mib.2017.05.008 |
| [6] |
Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell, 2014, 157(6): 1262-1278. DOI:10.1016/j.cell.2014.05.010 |
| [7] |
Deltcheva E, Chylinski K, Sharma CM, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ[J]. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886 |
| [8] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-921. DOI:10.1126/science.1225829 |
| [9] |
Paquet D, Kwart D, Chen A, et al. Efficient introduction of specific homozygous and heterozygous mutations using CRISPR/Cas9[J]. Nature, 2016, 533(7601): 125-129. DOI:10.1038/nature17664 |
| [10] |
Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [11] |
La Russa MF, Qi LS. The new state of the art: Cas9 for gene activation and repression[J]. Molecular and Cellular Biology, 2015, 35(22): 3800-3809. DOI:10.1128/MCB.00512-15 |
| [12] |
Xu XS, Qi LS. A CRISPR-dCas toolbox for genetic engineering and synthetic biology[J]. Journal of Molecular Biology, 2019, 431(1): 34-47. |
| [13] |
Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946 |
| [14] |
Gaudelli NM, Komor AC, Rees HA, et al. Programmable base editing of A∙T to G∙C in genomic DNA without DNA cleavage[J]. Nature, 2017, 551(7681): 464-471. DOI:10.1038/nature24644 |
| [15] |
Rees HA, Liu DR. Base editing: precision chemistry on the genome and transcriptome of living cells[J]. Nature Reviews Genetics, 2018, 19(12): 770-788. DOI:10.1038/s41576-018-0059-1 |
| [16] |
Cox DBT, Gootenberg JS, Abudayyeh OO, et al. RNA editing with CRISPR-Cas13[J]. Science, 2017, 358(6366): 1019-1027. DOI:10.1126/science.aaq0180 |
| [17] |
Harris RS, Petersen-Mahrt SK, Neuberger MS. RNA editing enzyme APOBEC1 and some of its homologs can act as DNA mutators[J]. Molecular Cell, 2002, 10(5): 1247-1253. DOI:10.1016/S1097-2765(02)00742-6 |
| [18] |
Mol CD, Arvai AS, Sanderson RJ, et al. Crystal structure of human uracil-DNA glycosylase in complex with a protein inhibitor: protein mimicry of DNA[J]. Cell, 1995, 82(5): 701-708. DOI:10.1016/0092-8674(95)90467-0 |
| [19] |
Nishida K, Arazoe T, Yachie N, et al. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems[J]. Science, 2016, 353(6305): aaf8729. DOI:10.1126/science.aaf8729 |
| [20] |
Komor AC, Zhao KT, Packer MS, et al. Improved base excision repair inhibition and bacteriophage Mu Gam protein yields C: G-to-T: A base editors with higher efficiency and product purity[J]. Science Advances, 2017, 3(8): eaao4774. DOI:10.1126/sciadv.aao4774 |
| [21] |
Kim YB, Komor AC, Levy JM, et al. Increasing the genome-targeting scope and precision of base editing with engineered Cas9-cytidine deaminase fusions[J]. Nature Biotechnology, 2017, 35(4): 371-376. |
| [22] |
Hu JH, Miller SM, Geurts MH, et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity[J]. Nature, 2018, 556(7699): 57-63. DOI:10.1038/nature26155 |
| [23] |
Nishimasu H, Shi X, Ishiguro S, et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space[J]. Science, 2018, 361(6408): 1259-1262. DOI:10.1126/science.aas9129 |
| [24] |
Wang X, Li JN, Wang Y, et al. Efficient base editing in methylated regions with a human APOBEC3A-Cas9 fusion[J]. Nature Biotechnology, 2018, 36(10): 946-949. |
| [25] |
Ma YQ, Zhang JY, Yin WJ, et al. Targeted AID-mediated mutagenesis (TAM) enables efficient genomic diversification in mammalian cells[J]. Nature Methods, 2016, 13(12): 1029-1035. DOI:10.1038/nmeth.4027 |
| [26] |
Gehrke JM, Cervantes O, Clement MK, et al. An APOBEC3A-Cas9 base editor with minimized bystander and off-target activities[J]. Nature Biotechnology, 2018, 36(10): 977-982. |
| [27] |
Li XS, Wang Y, Liu YJ, et al. Base editing with a Cpf1-cytidine deaminase fusion[J]. Nature Biotechnology, 2018, 36(4): 324-327. DOI:10.1038/nbt.4102 |
| [28] |
Zheng YX, Lorenzo C, Beal PA. DNA editing in DNA/RNA hybrids by adenosine deaminases that act on RNA[J]. Nucleic Acids Research, 2017, 45(6): 3369-3377. |
| [29] |
Wolf J, Gerber AP, Keller W. TadA, an essential tRNA-specific adenosine deaminase from Escherichia coli[J]. The EMBO Journal, 2002, 21(14): 3841-3851. DOI:10.1093/emboj/cdf362 |
| [30] |
Kim J, Malashkevich V, Roday S, et al. Structural and kinetic characterization of Escherichia coli TadA, the wobble-specific tRNA deaminase[J]. Biochemistry, 2006, 45(20): 6407-6416. DOI:10.1021/bi0522394 |
| [31] |
Matthews MM, Thomas JM, Zheng YX, et al. Structures of human ADAR2 bound to dsRNA reveal base-flipping mechanism and basis for site selectivity[J]. Nature Structural & Molecular Biology, 2016, 23(5): 426-433. |
| [32] |
Gerber AP, Keller W. An adenosine deaminase that generates inosine at the wobble position of tRNAs[J]. Science, 1999, 286(5442): 1146-1149. DOI:10.1126/science.286.5442.1146 |
| [33] |
Grunebaum E, Cohen A, Roifman CM. Recent advances in understanding and managing adenosine deaminase and purine nucleoside phosphorylase deficiencies[J]. Current Opinion in Allergy and Clinical Immunology, 2013, 13(6): 630-638. DOI:10.1097/ACI.0000000000000006 |
| [34] |
Shi K, Carpenter MA, Banerjee S, et al. Structural basis for targeted DNA cytosine deamination and mutagenesis by APOBEC3A and APOBEC3B[J]. Nature Structural & Molecular Biology, 2017, 24(2): 131-139. |
| [35] |
Macbeth MR, Schubert HL, van Demark AP, et al. Inositol hexakisphosphate is bound in the ADAR2 core and required for RNA editing[J]. Science, 2005, 309(5740): 1534-1539. DOI:10.1126/science.1113150 |
| [36] |
Losey HC, Ruthenburg AJ, Verdine GL. Crystal structure of Staphylococcus aureus tRNA adenosine deaminase TadA in complex with RNA[J]. Nature Structural & Molecular Biology, 2006, 13(2): 153-159. |
| [37] |
Hua K, Tao XP, Zhu JK. Expanding the base editing scope in rice by using Cas9 variants[J]. Plant Biotechnology Journal, 2018. DOI:10.1111/pbi.12993 |
| [38] |
Yang L, Zhang XH, Wang LR, et al. Increasing targeting scope of adenosine base editors in mouse and rat embryos through fusion of TadA deaminase with Cas9 variants[J]. Protein & Cell, 2018, 9(9): 814-819. |
| [39] |
Nishikura K. Functions and regulation of RNA editing by ADAR deaminases[J]. Annual Review of Biochemistry, 2010, 79: 321-349. DOI:10.1146/annurev-biochem-060208-105251 |
| [40] |
Tan MH, Li Q, Shanmugam R, et al. Dynamic landscape and regulation of RNA editing in mammals[J]. Nature, 2017, 550(7675): 249-254. DOI:10.1038/nature24041 |
| [41] |
Bass BL, Weintraub H. An unwinding activity that covalently modifies its double-stranded RNA substrate[J]. Cell, 1988, 55(6): 1089-1098. DOI:10.1016/0092-8674(88)90253-X |
| [42] |
Wong SK, Sato S, Lazinski DW. Substrate recognition by ADAR1 and ADAR2[J]. RNA, 2001, 7(6): 846-858. |
| [43] |
Wettengel J, Reautschnig P, Geisler S, et al. Harnessing human ADAR2 for RNA repair - Recoding a PINK1 mutation rescues mitophagy[J]. Nucleic Acids Research, 2017, 45(5): 2797-2808. |
| [44] |
Stafforst T, Schneider MF. An RNA-deaminase conjugate selectively repairs point mutations[J]. Angewandte Chemie International Edition, 2012, 51(44): 11166-11169. DOI:10.1002/anie.201206489 |
| [45] |
Vogel P, Schneider MF, Wettengel J, et al. Improving site-directed RNA editing in vitro and in cell culture by chemical modification of the guideRNA[J]. Angewandte Chemie International Edition, 2014, 53(24): 6267-6271. DOI:10.1002/anie.201402634 |
| [46] |
Vogel P, Moschref M, Li Q, et al. Efficient and precise editing of endogenous transcripts with SNAP-tagged ADARs[J]. Nature Methods, 2018, 15(7): 535-538. DOI:10.1038/s41592-018-0017-z |
| [47] |
Montiel-González MF, Vallecillo-Viejo I, Yudowski GA, et al. Correction of mutations within the cystic fibrosis transmembrane conductance regulator by site-directed RNA editing[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(45): 18285-18290. DOI:10.1073/pnas.1306243110 |
| [48] |
Montiel-González MF, Vallecillo-Viejo IC, Rosenthal JJC. An efficient system for selectively altering genetic information within mRNAs[J]. Nucleic Acids Research, 2016, 44(21): e157. |
| [49] |
Hess GT, Tycko J, Yao D, et al. Methods and applications of CRISPR-mediated base editing in eukaryotic genomes[J]. Molecular Cell, 2017, 68(1): 26-43. |
| [50] |
Kim JS. Precision genome engineering through adenine and cytosine base editing[J]. Nature Plants, 2018, 4(3): 148-151. DOI:10.1038/s41477-018-0115-z |
| [51] |
Eid A, Alshareef S, Mahfouz MM. CRISPR base editors: genome editing without double-stranded breaks[J]. Biochemical Journal, 2018, 475(11): 1955-1964. DOI:10.1042/BCJ20170793 |
| [52] |
Komor AC, Badran AH, Liu DR. Editing the genome without double-stranded DNA breaks[J]. ACS Chemical Biology, 2018, 13(2): 383-388. |
| [53] |
Wu WY, Yang YH, Lei HT. Progress in the application of CRISPR: From gene to base editing[J]. Medicinal Research Reviews, 2018. DOI:10.1002/med.21537 |
| [54] |
May A. Base editing on the rise[J]. Nature Biotechnology, 2017, 35(5): 428-429. DOI:10.1038/nbt.3871 |
| [55] |
Plosky BS. CRISPR-mediated base editing without DNA double-strand breaks[J]. Molecular Cell, 2016, 62(4): 477-478. DOI:10.1016/j.molcel.2016.05.006 |
| [56] |
Bjerke JN, Beardslee PC, McNaughton BR. Recent advances in CRISPR base editing: from A to RNA[J]. Biochemistry, 2018, 57(6): 886-887. |
| [57] |
Seo H, Kim JS. Towards therapeutic base editing[J]. Nature Medicine, 2018, 24(10): 1493-1495. DOI:10.1038/s41591-018-0215-3 |
| [58] |
Arazoe T, Kondo A, Nishida K. Targeted nucleotide editing technologies for microbial metabolic engineering[J]. Biotechnology Journal, 2018, 13(9): 1700596. DOI:10.1002/biot.v13.9 |
| [59] |
Banno S, Nishida K, Arazoe T, et al. Deaminase-mediated multiplex genome editing in Escherichia coli[J]. Nature Microbiology, 2018, 3(4): 423-429. DOI:10.1038/s41564-017-0102-6 |
| [60] |
Zheng K, Wang Y, Li N, et al. Highly efficient base editing in bacteria using a Cas9-cytidine deaminase fusion[J]. Communications Biology, 2018, 1(1): 32. DOI:10.1038/s42003-018-0035-5 |
| [61] |
Wang Y, Liu Y, Liu J, et al. MACBETH: Multiplex automated Corynebacterium glutamicum base editing method[J]. Metabolic Engineering, 2018, 47: 200-210. DOI:10.1016/j.ymben.2018.02.016 |
| [62] |
Gu TN, Zhao SQ, Pi YS, et al. Highly efficient base editing in Staphylococcus aureus using an engineered CRISPR RNA-guided cytidine deaminase[J]. Chemical Science, 2018, 9(12): 3248-3253. DOI:10.1039/C8SC00637G |
| [63] |
Wang Y, Wang SS, Chen WZ, et al. Precise and efficient genome editing in Klebsiella pneumoniae using CRISPR-Cas9 and CRISPR-assisted cytidine deaminase[J]. Applied and Environmental Microbiology, 2018, 84(23): e01834-18. |
| [64] |
Chen WZ, Zhang Y, Zhang YF, et al. CRISPR/Cas9-based genome editing in Pseudomonas aeruginosa and cytidine deaminase-mediated base editing in Pseudomonas species[J]. iScience, 2018, 6: 222-231. DOI:10.1016/j.isci.2018.07.024 |
| [65] |
Andersen JB, Sternberg C, Poulsen LK, et al. New unstable variants of green fluorescent protein for studies of transient gene expression in bacteria[J]. Applied and Environmental Microbiology, 1998, 64(6): 2240-2246. |
| [66] |
Jiang Y, Qian FH, Yang JJ, et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum[J]. Nature Communications, 2017, 8: 15179. DOI:10.1038/ncomms15179 |
| [67] |
Liu J, Wang Y, Lu YJ, et al. Development of a CRISPR/Cas9 genome editing toolbox for Corynebacterium glutamicum[J]. Microbial Cell Factories, 2017, 16(1): 205. DOI:10.1186/s12934-017-0815-5 |
| [68] |
Cho JS, Choi KR, Prabowo CPS, et al. CRISPR/Cas9-coupled recombineering for metabolic engineering of Corynebacterium glutamicum[J]. Metabolic Engineering, 2017, 42: 157-167. DOI:10.1016/j.ymben.2017.06.010 |
| [69] |
Cobb RE, Wang YJ, Zhao HM. High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system[J]. ACS Synthetic Biology, 2015, 4(6): 723-728. DOI:10.1021/sb500351f |
| [70] |
Tong YJ, Charusanti P, Zhang LX, et al. CRISPR-Cas9 based engineering of actinomycetal genomes[J]. ACS Synthetic Biology, 2015, 4(9): 1020-1029. DOI:10.1021/acssynbio.5b00038 |
| [71] |
Huang H, Zheng GS, Jiang WH, et al. One-step high-efficiency CRISPR/Cas9-mediated genome editing in Streptomyces[J]. Acta Biochimica et Biophysica Sinica, 2015, 47(4): 231-243. DOI:10.1093/abbs/gmv007 |
| [72] |
Zeng H, Wen S, Xu W, et al. Highly efficient editing of the actinorhodin polyketide chain length factor gene in Streptomyces coelicolor M145 using CRISPR/Cas9-CodA(sm) combined system[J]. Applied Microbiology and Biotechnology, 2015, 99(24): 10575-10585. DOI:10.1007/s00253-015-6931-4 |
| [73] |
Li L, Wei KK, Zheng GS, et al. CRISPR-Cpf1-assisted multiplex genome editing and transcriptional repression in Streptomyces[J]. Applied and Environmental Microbiology, 2018, 84(18): e00827-18. |
| [74] |
Tao WX, Yang AN, Deng ZX, et al. CRISPR/Cas9-based editing of Streptomyces for discovery, characterization, and production of natural products[J]. Frontiers in Microbiology, 2018, 9: 1660. DOI:10.3389/fmicb.2018.01660 |
 2019, Vol. 46
2019, Vol. 46