扩展功能
文章信息
- 占米林, 阚宝军, 张辉, 董晋军, 许国超, 韩瑞枝, 倪晔
- ZHAN Mi-Lin, KAN Bao-Jun, ZHANG Hui, DONG Jin-Jun, XU Guo-Chao, HAN Rui-Zhi, NI Ye
- 谷氨酸棒状杆菌CRISPR-Cpf1和Cre/loxP基因敲除技术的比较
- Comparison of CRISPR-Cpf1 with Cre/loxP for gene knockout in Corynebacterium glutamicum
- 微生物学通报, 2019, 46(2): 278-291
- Microbiology China, 2019, 46(2): 278-291
- DOI: 10.13344/j.microbiol.china.180225
-
文章历史
- 收稿日期: 2018-03-21
- 接受日期: 2018-05-08
- 网络首发日期: 2018-06-05
谷氨酸棒状杆菌是一种非致病性的、革兰氏阳性菌株[1-2],作为一种重要的底盘微生物广泛用于工业发酵生产氨基酸及有机酸等,如L-谷氨酸、L-谷氨酰胺、L-赖氨酸、L-缬氨酸和L-精氨酸[3-5]。目前,通过传统诱变育种的策略可获得生产性能改良的谷氨酸棒状杆菌,然而由于突变的随机性及传代的不稳定性[6],需要巨大的筛选和繁琐的菌种复壮工作,且代谢流向不明确难以实现精细化调控和经济化生产。随着谷氨酸棒状杆菌ATCC 13032及相关亚种的全基因组测序[7],科研工作者可以利用代谢工程或合成生物学的策略对谷氨酸棒状杆菌进行定向改造,使其具有更高的生产效率。
目前,谷氨酸棒状杆菌中的基因敲除载体或系统非常有限。其中最经典的敲除系统是基于同源重组及蔗糖致死基因sacB的反向筛选技术[8],但该系统存在一些明显不足,包括筛选转化子费时费力、易发生转座子的插入而使sacB基因失活的现象等[9]。因此,迫切需要可适用于谷氨酸棒状杆菌的高效基因编辑技术。近些年开发了包括基于同源重组及位点特异性重组的Cre/loxP敲除系统和基于CRISPR-Cpf1的同源重组敲除系统。Cre/loxP敲除系统[10]通过两次同源重组实现靶向基因的敲除,首先利用同源重组将基因组上的靶向基因替换成两端带有重组位点loxP的kanR片段,第二步通过pDTW109表达的重组酶Cre识别重组位点loxP的序列并发生重组反应[11-12],消除替换到基因组上kanR片段,并利用pDTW109温敏特性实现自我消除,最终完成整个基因敲除过程。继大肠杆菌的基因组编辑技术CRISPR-Cas[13-14]的成功构建,最近报道了基于土拉热弗朗西丝菌(Francisella novicida) Cpf1的谷氨酸棒状杆菌基因组编辑方法CRISPR-Cpf1[15],该系统主要由FnCpf1、CRISPR RNA和同源臂组成,在特异性的序列上切割靶DNA,然后通过同源重组去除靶向基因,同样利用质粒的温敏特性消除自身的存在,最后获得基因敲除菌株。本研究以谷氨酸棒状杆菌ATCC 13032的argR和argF两个基因[16]的敲除为例,比较研究了CRISPR-Cpf1和Cre/loxP两种基因敲除系统的优缺点,并对CRISPR-Cpf1技术进行了展望。
1 材料与方法 1.1 菌株和质粒实验所用菌株和质粒见表 1。
| 菌株和质粒 Strains and plasmids |
特征 Characteristic |
来源 Source |
| Strains | ||
| E. coli JM109 | E. coli cloning strain | TaKaRa |
| C. glutamicum ATCC 13032 | C. glutamicum model strain | ATCC |
| JML01 | C. glutamicum ATCC 13032ΔargR | This work |
| JML02 | C. glutamicum ATCC 13032ΔargF | This work |
| JML03 | C. glutamicum ATCC 13032ΔargR | This work |
| JML04 | C. glutamicum ATCC 13032ΔargF | This work |
| Plasmids | ||
| pJYS3_ΔcrtYf | Kanamycin resistance (kanR), shuttle vector | [15] |
| pJYS3_ΔargR | kanR, shuttle vector | This work |
| pJYS3_ΔargF | kanR, shuttle vector | This work |
| pBlueScript Ⅱ SK | Cloning vector, ColE1, lacZ, Ampicillin resistance | [17] |
| pDTW202 | pBlueScript harboring a kanR gene flanking with loxPLE/loxPRE sites | [17] |
| pDTW109 | pDTW109 harboring a cre gene, Chloramphenicol resistance | [17] |
| pDTW203 | pBlueScript harboring argR gene flanking regions and loxPLE/PRE-kanR | This work |
| pDTW204 | pBlueScript harboring argF gene flanking regions and loxPLE/PRE-kanR | This work |
LB培养基(g/L):胰蛋白胨10.0,氯化钠10.0,酵母粉5.0。
大肠杆菌培养温度为37 ℃,摇床转速120 r/min,抗生素添加浓度:氯霉素34 μg/mL,氨苄青霉素100 μg/mL,卡那霉素50 μg/mL。
BHI培养基(g/L):胰蛋白胨10.0,氯化钠5.0,脑心浸液18.5,酵母粉5.0,pH 7.2。
感受态细胞培养基为EPO培养基(g/L):甘氨酸30.0,胰蛋白胨10.0,酵母粉5.0,氯化钠5.0,脑心浸液18.5,吐温80 1.0,异烟肼0.4。
转化恢复培养基为LB-HIS培养基(g/L):胰蛋白胨10.0,脑心浸液18.5,酵母粉5.0,氯化钠5.0,D-山梨醇92.0。
谷氨酸棒状杆菌培养温度为30 ℃,摇床转速120 r/min,抗生素添加浓度:氯霉素10 μg/mL,卡那霉素30 μg/mL。
如需配制固体培养基,则向上述培养基中加入18.0 g/L的琼脂粉。所有培养基于1×105 Pa灭菌20 min。
1.3 主要试剂和仪器IPTG、氨苄青霉素、卡那霉素、氯霉素、琼脂糖凝胶回收试剂盒、质粒小量提取试剂盒、细菌基因组DNA快速提取试剂盒、DNA Marker,上海捷瑞生物工程有限公司;限制性内切酶、Dpn Ⅰ、Primer STAR DNA聚合酶、2×Taq PCR Master Mix,TaKaRa宝生物工程(大连TaKaRa)有限公司;胰蛋白胨、酵母粉、脑心浸液,OXOID公司;引物合成和测序服务由苏州泓迅生物科技股份有限公司提供。电转仪,Bio-Rad公司。
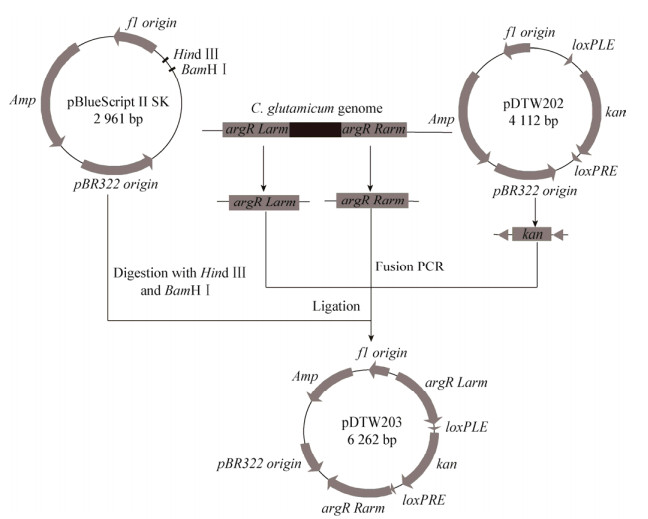
1.4 方法 1.4.1 Cre/loxP系统敲除质粒的构建以敲除谷氨酸棒状杆菌ATCC 13032基因组的argR为例,Cre/loxP系统敲除质粒的构建步骤如下。首先以谷氨酸棒状杆菌ATCC 13032基因组为模板,分别以argR-U-F/argR-U-R和argR-D-F/argR-D-R为引物扩增得到argR的上、下游同源片段,其中在上游同源片段的5′末端和下游同源片段的3′末端分别引入Hind Ⅲ和BamH Ⅰ酶切位点序列。然后以pDTW202质粒为模板,以argR-K-F/argR-K-R为引物,扩增两端含有loxP位点的kanR片段。通过融合PCR使上述3个片段串联融合,同时pBlueScript Ⅱ SK经过Hind Ⅲ/BamH Ⅰ酶切后即可获得线性化片段,然后利用Gibson法将融合片段与线性化载体进行连接,并取5 μL连接液通过化学转化法转化至E. coli JM109感受态细胞,待其长出菌落后进行菌落PCR和基因测序验证,其中验证正确的打靶质粒命名为pDTW203,具体流程如图 1所示。其中引物设计见表 2。
|
| 图 1 敲除质粒pDTW203的构建 Figure 1 Construction of knockout plasmid pDTW203 |
|
|
| 引物Primers | 引物序列Primers sequence (5′→3′) |
| argR-U-F | GTCGACGGTATCGATAAGCTTAGGACTCAAACTTATGACTTCACAACCA |
| argR-U-R | CGCCCTATAGTGAGTCGTATTGGGATTTAAGTTTTCCGGTGTTGACG |
| argR-K-F | CGTCAACACCGGAAAACTTAAATCCCAATACGACTCACTATAGGGCG |
| argR-K-R | TGCCTGCATTAACAAGCGATTAACGCGCAATTAACCCTCACTAAAG |
| argR-D-F | CTTTAGTGAGGGTTAATTGCGCGTTAATCGCTTGTTAATGCAGGCA |
| argR-D-R | CGCTCTAGAACTAGTGGATCCCAAAGCCTCGTGAGCCTTAATC |
| argR-F | GTGACTCGCACTGCACGC |
| argR-R | TTAAGTGGTGCGCCCGCT |
| A-F | CTATTGCCGAGACAATCGCATA |
| A-R | CGCTCGATGGTGACATCTTC |
| argF-U-F | GTCGACGGTATCGATAAGCTTTGCACGCTTGACTGGTCGT |
| argF-U-R | GCCCTATAGTGAGTCGTATTAAGTTTGAGTCCTTTATGCGATTG |
| argF-K-F | AATACGACTCACTATAGGGCGAATT |
| argF-K-R | CTGGGTGATATCGCGCAATTAACCCTCACTAAAGG |
| argF-D-F | AATTGCGCGATATCACCCAGGCCACCTTG |
| argF-D-R | CGCTCTAGAACTAGTGGATCCAGCCGACCTCGAAACGAACC |
| 注:下划线表示限制性酶切位点. Note: The underlined sequences are the restriction enzymes sites. |
|
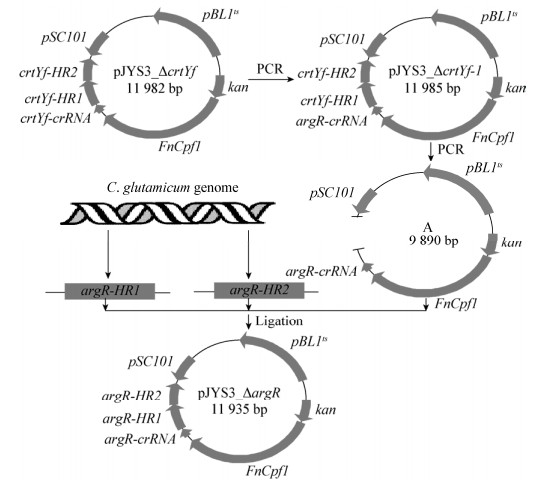
以敲除谷氨酸棒状杆菌ATCC 13032基因组的argR为例,CRISPR-Cpf1系统敲除质粒的构建步骤如下。首先以pJYS3_ΔcrtYf质粒为模板,以crtYf-F-1/crtYf-R-1为引物进行定点突变,通过全质粒PCR将crtYf基因的crRNA序列(5′-GAATTTCT ACTGTTGTAGATCAGGCAACCATAGGGCAGGAA-3′)替换为argR基因的crRNA序列(5′-GAATTTC TACTGTTGTAGATTACAGATCATTCCGGCAACATCGC-3′)以及argF的crRNA序列(5′-GAATTTCTA CTGTTGTAGATGTCCCTCGAGTGGACGCTCCG-3′),然后用Dpn Ⅰ限制性内切酶消化母本质粒。取3 μL消化后的PCR产物通过化学转化法转化到E. coli JM109感受态细胞中,待其长出菌落进行PCR验证并测序验证,得到质粒pJYS3_ΔcrtYf-1。再以pJYS3_ΔcrtYf-1为模板,以crRNA_argR-F/crRNA_ argR-R为引物,通过PCR扩增得到除去crtYf基因上下游同源臂的A片段。再以谷氨酸棒状杆菌ATCC 13032基因组为模板,分别以argR-F-F/argR-F-R和argR-R-F/argR-R-R为引物扩增argR的上游和下游同源片段,并将A片段、argR的上游和下游同源片段进行连接,取5 μL连接液热转化至E. coli JM109感受态细胞,待其长出菌落后,以argR-F-F/argR-F-R为引物进行菌落PCR和基因测序验证,其中验证正确的打靶质粒命名为pJYS3_ΔargR。具体构建流程如图 2所示。其中引物设计见表 3。
|
| 图 2 敲除质粒pJYS3_ΔargR的构建 Figure 2 Construction of knockout plasmid pJYS3_ΔargR |
|
|
| 引物Primers | 引物序列Primers sequence (5′→3′) |
| crtYf-F-1 | CATTATACCTAGGACTGAGCTAGCTGTCAATCTAGCCTATCCAGCAGTCTTCCTGTC |
| crtYf-R-1 | CTAACAAACAAAAGTAGAACAACTGTTCACCGGGCCCGTGGGTGGCTAGGCAAGTTAC |
| crRNA_argR-R | CGTCAACACCGGAAAACTTAAATCCCAATACGACTCACTATAGGGCG |
| crRNA_argR-F | TGCCTGCATTAACAAGCGATTAACGCGCAATTAACCCTCACTAAAG |
| argR-F-R | GGGATTTAAGTTTTCCGGTGT |
| argR-F-F | CTAACAAACAAAAGTAGAACAACTGTTCACCGGGCCGGACTCAAACTTATGACTTCACAACC |
| argR-R-F | TCAACCCCGTCAACACCGGAAAACTTAAATCCCAGCGCCCCTAGTTCAAGGCTT |
| argR-R-R | CATTATACCTAGGACTGAGCTAGCTGTCAATCTAGCAAAGCCTCGTGAGCCTTAATC |
| crtYf-F-2 | GTTGTAGATGTCCCTCGAGTGGACGCTCCGATTTAAATAAAACGAAAGGCTCAGTCGAAAGA |
| crtYf-R-2 | CCACTCGAGG GACATCTACAACAGTAGAAATTCGGATCCATTA |
| crRNA_argF-R | GGCCCGGTGAACAGTTGTT |
| crRNA_argF-F | CTAGATTGACAGCTAGCTCAGTCCTAG |
| argF-F-F | GGCCTGGGTGATATCAAGTTTGAGTCCTTTATGCGATTG |
| argF-F-R | TGAGCTAGCTGTCAATCTAGTGCACGCTTGACTGGTCGT |
| argF-R-F | GAACAACTGTTCACCGGGCCAGCCGACCTCGAAACGAACC |
| argF-R-R | AACTTGATATCACCCAGGCCACCTTG |
| F-F | GTTTGGATCGGTGTCCATGA |
| F-R | ACGAGGCTGCTTTCAAGAT |
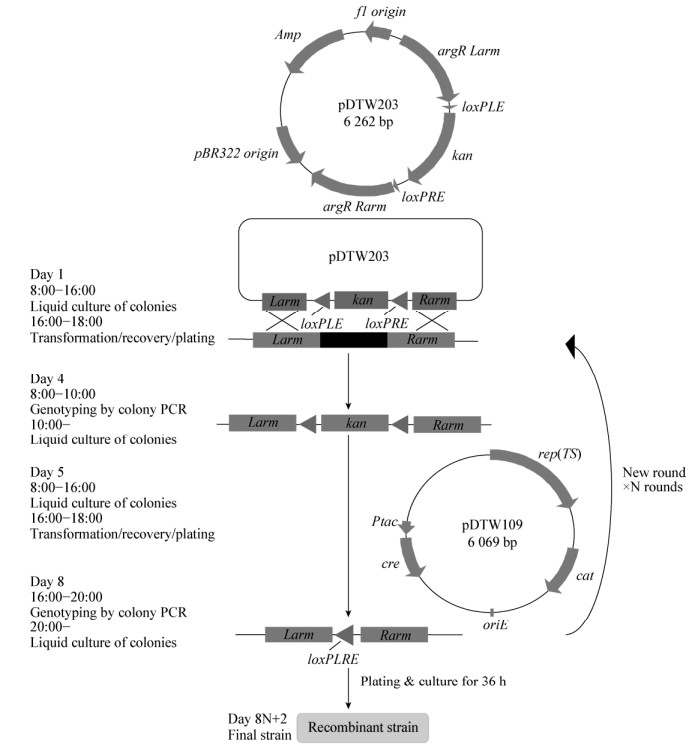
以敲除谷氨酸棒状杆菌ATCC 13032基因组的argR为例,Cre/loxP系统敲除菌株的构建步骤如图 3所示。首先将敲除质粒pDTW203电转入谷氨酸棒状杆菌ATCC 13032的感受态细胞进行基因打靶,在同源重组的作用下原始菌株的argR基因被替换为两端含有loxP位点的kanR片段,获得菌株谷氨酸棒状杆菌ATCC 13032ΔargR::kanR。然后将Cre重组酶表达载体pDTW109电转化到突变株的感受态细胞中,涂布于含有氯霉素的恢复培养基平板,在Cre重组酶的作用下,kanR基因两端的loxP相互识别并发生重组,从而将kanR片段消除,只留下一个34 bp左右的小片段loxPLR,挑选转化子进行菌落PCR验证。由于pDTW109质粒复制子的温敏特性,在37 ℃条件下培养即可消除质粒。为了确保菌株的正确性,最后还需要验证其抗性,将PCR验证正确的菌株分别转接到含有30 μg/mL卡那霉素的LB-HIS平板、10 μg/mL氯霉素的LB-HIS平板和LB-HIS平板上,其中能在LB-HIS平板上生长,但对卡那霉素和氯霉素敏感的菌株即是基因被敲除成功的菌株。
|
| 图 3 Cre/loxP辅助的谷氨酸棒状杆菌基因敲除的流程图 Figure 3 Flow chart of Cre/loxP assisted gene knockout in C. glutamicum |
|
|
以敲除谷氨酸棒状杆菌ATCC 13032基因组的argR为例,CRISPR-Cpf1系统敲除菌株的构建步骤如图 4所示。首先将敲除质粒pJYS3_ΔargR电转入谷氨酸棒状杆菌ATCC 13032的感受态细胞进行基因打靶,然后Cpf1对crRNA进行加工,成熟的crRNA引导Cpf1结合到DNA的特异位点上切割靶DNA,在菌体内重组酶的作用下,通过同源重组实现基因argR的缺失,菌落PCR验证选出正确的突变株。然后利用质粒pJYS3_ΔargR自身的温敏特性,34 ℃条件下培养即可将敲除质粒消除。为了确保突变株的正确性,还需要验证其抗性,分别将转化子转接到含有30 μg/mL卡那霉素的LB-HIS平板和LB-HIS平板上,其中能在LB-HIS平板上生长,但对卡那霉素敏感的菌株即敲除成功的菌株。

|
| 图 4 CRISPR-Cpf1辅助的谷氨酸棒状杆菌基因编辑的流程图 Figure 4 Flow chart of CRISPR-Cpf1 assisted gene knockout in C. glutamicum |
|
|
谷氨酸棒状杆菌感受态细胞的制备采用电转化方法[18],取约1-2 μg打靶质粒与100 μL感受态细胞轻轻混匀,加入电击杯中,连续电击2次(电击条件为2.5 kV/cm电压、5.9 ms、25 mF电容、2 mm电击杯)。然后置于合适温度下、100 r/min培养3 h,涂布于含有相对应的抗生素固体培养基,培养至长出较大的单菌落时,挑取单菌落进行验证。
2 结果与分析 2.1 Cre/loxP系统敲除质粒的构建参考实验方法1.4.1,分别以谷氨酸棒状杆菌ATCC 13032基因组、pDTW202质粒为模板,通过PCR扩增出argR基因上下同源片段和两端含有loxP位点的kanR片段,再利用融合PCR技术获得目的片段,最后与线性化载体pBlueScript Ⅱ SK连接,得到敲除质粒pDTW203。以argR-U-F/argR-D-R为引物对重组菌株进行PCR验证,如图 5D所示,融合片段(3 377 bp)已经成功连接到载体pBlueScript Ⅱ SK上,说明敲除质粒pDTW203构建成功。

|
| 图 5 质粒pDTW203琼脂糖凝胶电泳验证 Figure 5 Construction and verification of plasmid pDTW203 注:M:Marker. A:PCR扩增argR基因的上游、下游同源片段和kanR盒;1-2:argR基因的上游同源片段;3-5:kanR盒;6-7:argR基因的下游同源片段. B:argR基因的上游、下游同源片段和kanR盒的融合PCR;1-2:融合片段. C:pBlueScript Ⅱ SK的BamH Ⅰ/Hind Ⅲ酶切;1-2:线性化pBlueScript Ⅱ SK. D:转化子的菌落PCR验证;1-2:融合片段. Note: M: Marker. A: Amplification of upstream and downstream homologous fragments of argR gene and kanR cassette by PCR; 1-2: Upstream homology arms of argR gene; 3-5: kanR cassette; 6-7: Downstream homologous arms of argR gene. B: Fusion PCR of upstream and downstream homology arms and kanR box of argR gene; 1-2: Fusion fragment. C: Digestion of pBlueScript Ⅱ SK with BamH Ⅰ and Hind Ⅲ; 1-2: Linearized pBlueScript Ⅱ SK. D: Validation of transformants by colony PCR; 1-2: Fusion fragment. |
|
|
参考实验方法1.4.3,将pDTW203质粒电转化至谷氨酸棒状杆菌ATCC 13032感受态细胞中,通过菌落PCR验证阳性克隆,其中基因组上argR基因被替换为两端带有loxP位点的kanR基因的正确转化子命名为谷氨酸棒状杆菌ATCC 13032ΔargR::kanR。制作突变株的感受态细胞,并将kanR消除质粒pDTW109电转入其感受态细胞中,菌落PCR验证结果如图 6所示。然后通过提高温度使质粒pDTW109丢失,验证抗性无误后的菌株命名为JML01。

|
| 图 6 JML01的菌落PCR验证 Figure 6 Verification of JML01 by colony PCR 注:M:Marker. A:谷氨酸棒状杆菌ATCC 13032ΔargR::kanR的菌落PCR验证;1-2:以A-F/A-R为引物谷氨酸棒状杆菌ATCC 13032的菌落PCR;3-4:以A-F/A-R为引物谷氨酸棒状杆菌ATCC 13032ΔargR::kanR的菌落PCR. B:JML01菌株的菌落PCR验证;1-2:以A-F/A-R为引物谷氨酸棒状杆菌ATCC 13032的菌落PCR;3-4:以A-F/A-R为引物JML01的菌落PCR. C:JML01菌株的菌落PCR验证;1-2:以argR-F/argR-R为引物谷氨酸棒状杆菌ATCC 13032的菌落PCR;3-4:以argR-F/argR-R为引物JML01的菌落PCR. Note: M: Marker. A: Verification of C. glutamicum ATCC 13032ΔargR::kanR by colony PCR; 1-2: Colony PCR product of C. glutamicum ATCC 13032 with primers A-F/A-R; 3-4: Colony PCR product of C. glutamicum ATCC 13032ΔargR::kanR with primers A-F/A-R. B: Verification of JML01 by colony PCR; 1-2: Colony PCR product of C. glutamicum ATCC 13032 with primers A-F/A-R; 3-4: Colony PCR product of JML01 with primers A-F/A-R. C: Verification of JML01 by colony PCR; 1-2: Colony PCR product of C. glutamicum ATCC 13032 with primers argR-F/argR-R; 3-4: Colony PCR product of JML01 with primers argR-F/argR-R. |
|
|
以A-F/A-R为引物进行菌落PCR验证,其中图 6A中泳道1–2是用谷氨酸棒状杆菌ATCC 13032为模板,泳道3–4是用谷氨酸棒状杆菌ATCC 13032ΔargR::kanR的基因组为模板,可以发现kanR盒已成功替换基因组的argR基因。然后以A-F/A-R为引物进行菌落PCR验证,其中图 6B中泳道1–2是用谷氨酸棒状杆菌ATCC 13032菌株为模板,泳道3–4是用JML01菌株为模板,并以argR-F/argR-R为引物进行菌落PCR验证,其中图 6C中泳道1–2是用谷氨酸棒状杆菌ATCC 13032菌株为模板,泳道3–4是用JML01菌株为模板,可以发现kanR基因被成功消除。挑取单菌落分别验证其卡那霉素及氯霉素抗性,其中有些菌株可以在无抗LB-HIS平板能够生长(图 7A),而在卡那霉素(图 7B)和氯霉素抗性LB-HIS平板(图 7C)不能生长的即是目的菌株。以上结果显示,成功构建了敲除菌株JML01。同理,按照同样的方法构建了argF的敲除菌株JML02,此处不在赘述。

|
| 图 7 JML01菌株的抗性验证 Figure 7 Antibiotic verification of JML01 strain 注:A:LB-HIS;B:携带卡那霉素的LB-HIS;C:携带氯霉素的LB-HIS. Note: A: LB-HIS; B: LB-HIS with kanamycin; C: LB-HIS with chloramphenicol. |
|
|
在同源重组Cre/loxP系统中,敲除质粒是由pBlueScript Ⅱ SK构建而来,其中pBlueScript Ⅱ SK在E. coli中是高拷贝的,质粒大小只有2 961 bp左右,容易提取出纯净且高浓度的质粒,有利于同源重组的进行。由图 6可看出,构建的JML01菌落PCR结果无误。pDTW109是Cre重组酶高效表达的温敏型载体,具有双复制子可以在大肠杆菌和谷氨酸棒状杆菌中穿梭复制,还有和敲除质粒不同的抗性标记氯霉素抗性,这些都有利于质粒的转化。由于质粒的温敏特性,提高培养温度即可使质粒pDTW109丢失[19],由图 7可知,少数菌株还具有卡那霉素抗性(3/16),氯霉素抗性已被消除。由此可以看出pDTW109可以高效表达Cre重组酶,并除去滞留在基因组上的kanR片段,同时温敏载体pDTW109也容易通过提高培养温度使其丢失。最终,根据实验统计,将1 μg敲除质粒导入到感受态细胞中,长出的转化子约100个。再经过kanR片段消除后,随机挑取40个转化子,以A-F/A-R为引物经过PCR验证有25%的转化子是argR成功敲除的菌株(图 8A)。在敲除argF的过程中长出的转化子约150个,随机挑取40个转化子,以F-F/F-R为引物经过PCR验证有33%的转化子是argF成功敲除的菌株(图 8B)。然而,其周期相对较长,成功敲除一个基因需要10 d。

|
| 图 8 通过Cre/loxP敲除系统和CRISPR-Cpf1敲除系统获得的转化子的PCR验证 Figure 8 PCR validation of colonies obtained by Cre/loxP assisted system and CRISPR-Cpf1 assisted system 注:A:Cre/loxP系统敲除argR获得的转化子;B:Cre/loxP系统敲除argF获得的转化子;C:CRISPR-Cpf1系统敲除argR获得的转化子;D:CRISPR-Cpf1系统敲除argF获得的转化子. argR和argF缺失的阳性转化子在琼脂糖凝胶上用三角形标记. Note: A: Transformants for the deletion of argR by Cre/loxP assisted system; B: Transformants for the deletion of argF by Cre/loxP assisted system; C: Transformants for the deletion of argR by CRISPR-Cpfl assisted system; D: Transformants for the deletion of argF by CRISPR-Cpfl assisted system. The editing-positive transformants for the deletion of argR and argF were marked with triangles in agarose gel. |
|
|
参考实验方法1.4.2,首先以pJYS3_ΔcrtYf质粒进行定点突变,通过全质粒PCR将crtYf基因的crRNA替换为argR基因的crRNA,并转化到E. coli JM109感受态细胞中,经验证后得到质粒pJYS3_ΔcrtYf-1。再以质粒pJYS3_ΔcrtYf-1为模板通过PCR扩增得到片段A,同时以谷氨酸棒状杆菌ATCC 13032基因组为模板扩增出argR基因的上下游同源片段,通过Gibson法将3个片段进行组装,并转化至E. coli JM109感受态细胞,最后以argR-F-F/argR-F-R为引物进行菌落PCR验证并测序验证,如图 9E所示,3个片段已经成功连接,说明敲除质粒pJYS3_ΔargR构建成功。
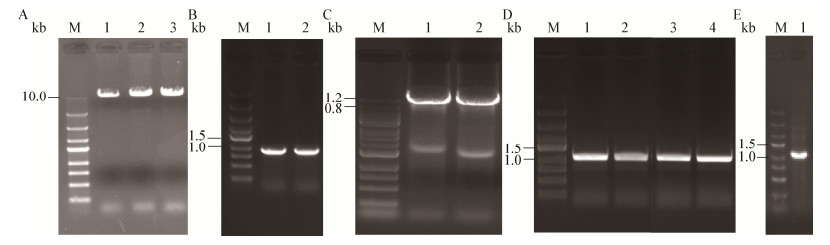
|
| 图 9 质粒pJYS3_ΔargR琼脂糖凝胶电泳验证 Figure 9 Construction and verification of plasmid pJYS3_ΔargR 注:M:Marker. A:pJYS3_ΔcrtYf全质粒PCR;1-3:pJYS3_ΔcrtYf-1. B:转化子的菌落PCR验证;1-2:pJYS3_ΔcrtYf-1突变后的片段. C:片段A的扩增;1-2:片段A. D:转化子的菌落PCR验证;1-2:argR基因的上游同源片段;3-4:argR基因的下游同源片段. E:转化子的菌落PCR验证;1:上游同源片段(1 020 bp). Note: M: Marker. A: Full-plasmid of pJYS3_ΔcrtYf PCR; 1-3: pJYS3_ΔcrtYf-1. B: Validation of transformants by colony PCR; 1-2: Fragments of the pJYS3_ΔcrtYf-1 mutation. C: Amplification of fragment A; 1-2: Fragment A. D: Validation of transformants by colony PCR; 1-2: Upstream homology arm of argR; 3-4: Downstream homologous arm of the argR. E: Validation of transformants by Colony PCR; 1: Upstream homology fragment (1 020 bp). |
|
|
参考实验方法1.4.4,通过电转化法将pJYS3_ΔargR敲除质粒转入谷氨酸棒状杆菌ATCC 13032的感受态细胞进行基因打靶,在胞内重组酶的作用下通过同源重组反应实现基因argR的敲除。挑出转化子进行菌落PCR验证,结果如图 10所示。以A-F/A-R为引物进行菌落PCR验证,其中泳道1–2是用谷氨酸棒状杆菌ATCC 13032菌株为模板,泳道3–6是用JML03菌株为模板。并以argR-F/argR-R为引物进行菌落PCR验证,泳道7–8是用谷氨酸棒状杆菌ATCC 13032菌株为模板,泳道9–12是用JML03菌株为模板,均显示敲除成功。再挑取平板上单菌落验证其卡那霉素抗性,由图 11可以发现有部分菌株可以在无抗LB-HIS平板生长,而对卡那霉素抗性敏感,这部分菌株的argR被成功敲除,命名为JML03。同理,按照同样的方法构建了argF的敲除菌株JML04,此处不在赘述。

|
| 图 10 JML03菌株的菌落PCR验证 Figure 10 Verification of JML03 strain by colony PCR 注:M:Marker;1-2:以A-F/A-R为引物谷氨酸棒状杆菌ATCC 13032的菌落PCR;3-6:以A-F/A-R为引物JML03的菌落PCR;7-8:以argR-F/argR-R为引物谷氨酸棒状杆菌ATCC 13032的菌落PCR;9-12:以argR-F/argR-R为引物JML03的菌落PCR. Note: M: Marker; 1-2: Colony PCR product of C. glutamicum ATCC 13032 with primers A-F/A-R; 3-6: Colony PCR product of JML03 with primers A-F/A-R; 7-8: Colony PCR product of C. glutamicum ATCC 13032 with primers argR-F/argR-R; 9-12: Colony PCR product of JML03 with primers argR-F/argR-R. |
|
|
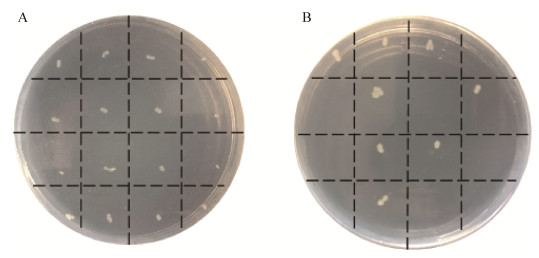
|
| 图 11 JML02菌株的抗性验证 Figure 11 Antibiotic verification of JML02 strain 注:A:LB-HIS;B:携带卡那霉素的LB-HIS. Note: A: LB-HIS; B: LB-HIS with kanamycin. |
|
|
在CRISPR-Cpf1敲除系统中,打靶质粒pJYS3_ΔargR是由pJYS3_ΔcrtYf构建而来,其基本结构如图 4所示。该质粒主要包括FnCpf1、crRNA、上下游同源臂和两个复制子[20]。质粒pJYS3_ΔargR大小超过10 000 bp,质粒过大对细胞负荷较高,因而拷贝数较低。将敲除质粒导入细胞后发现,其基因编辑效率较低,有些菌株基因组上仍然含有argR基因。在利用pJYS3_ΔargR自身的温敏特性消除抗生素标记时,还有较多菌株具有卡那霉素抗性(8/16),这些不利因素都严重影响了该敲除系统的效率(图 11)。据统计,将1 μg敲除质粒导入到感受态细胞中,约产生300个转化子,随机挑取40个转化子,以A-F/A-R为引物经过PCR验证有18%的转化子是argR成功敲除的菌株(图 8C)。在敲除argF的过程中,约产生300个转化子,随机挑取40个转化子,以F-F/F-R为引物经过PCR验证有18%的转化子是argF成功敲除的菌株(图 8D)。对于CRISPR-Cpf1敲除系统,仅需要7 d即可完成单个基因的无痕敲除,与Cre/loxP系统相比具有明显优势。
3 结论与讨论本研究立足于谷氨酸棒状杆菌代谢工程领域的最新研究进展,以谷氨酸棒状杆菌ATCC 13032作为出发菌株,选取了两个靶基因argR和argF,分别利用CRISPR-Cpf1和Cre/loxP两种基因编辑系统成功敲除了这两个靶基因。其中,Cre/loxP敲除系统基因敲除的重组率略高,在诸多报道中也得到广泛的应用[21-22]。经过同源置换和loxP位点重组后,仅在靶基因位置残留34 bp的loxPLR位点。当再次敲除时,Cre重组酶并不识别残留的loxPLR位点[23],不影响基于loxP位点的新一轮重组,因此可实现谷氨酸棒状杆菌基因的连续敲除。可能由于培养基中添加的卡那霉素浓度不高,且抗性筛选所需的时间较长(至少需要3 d,如图 3所示),培养基中卡那霉素的抗性作用减弱,因而容易产生假阳性菌株。经过数据统计发现,argR和argF敲除阳性率分别达到25%和33%。综上,在Cre/loxP敲除系统中通过两步同源重组过程可实现基因的敲除,其基因编辑效率略低于sacB的反向筛选技术[19],需要经过8N+2 d才可完成N轮迭代基因敲除,而后者仅需8 d即可完成一轮敲除。
由于Cre/loxP系统并不是一种无痕敲除技术,可能会对宿主细胞产生一定的影响,且周期较长,仍然需要开发更加高效和无痕的敲除技术。CRISPR是最新开发的基因组编辑技术,可以实现对多种原核和真核生物基因组DNA的定向敲除和整合,并于2017年首次在谷氨酸棒状杆菌得以应用[15]。与Cre/loxP敲除系统相比,CRISPR-Cpf1系统可以实现基因的无痕敲除,不会留下任何辅助识别位点,同时该敲除系统的操作周期比较短,正如相关报道所示CRISPR-Cpf1敲除系统是迄今为止发现的一种最简单的CRISPR免疫系统[24],仅需一次同源重组即可完成靶基因的敲除。理论上,采用该系统可在5N+2 d内完成N轮迭代基因敲除(图 4),比Cre/loxP敲除系统和sacB反向筛选技术更加简单、快捷。除此之外,CRISPR-Cpf1敲除系统还可一次作用于多个靶位点,实现多重编辑,这将大大提高多基因的敲除效率。然而可能由于Cpf1表达不足或crRNA转录盒突变导致假阳性,因此CRISPR/Cpf1敲除系统的argR和argF阳性敲除率略低于Cre/loxP敲除系统。
综上所述,CRISPR-Cpf1系统在谷氨酸棒状杆菌的基因敲除中更具优势,仅需一次同源重组即可实现基因的无痕敲除,缩短时间和降低工作量。尽管如此,CRISPR-Cpf1系统在某些方面也有待进一步完善,如敲除质粒偏大、假阳性菌株数目较多,给质粒构建和菌株筛选工作带来一定的压力。因而,在CRISPR-Cpf1同源重组敲除系统的后续研究中,应从提高Cpf1的表达、减少crRNA转录盒突变以及优化同源臂的长度等方面入手,使该基因编辑系统的效率更高。
| [1] |
Siebert D, Wendisch VF. Metabolic pathway engineering for production of 1, 2-propanediol and 1-propanol by Corynebacterium glutamicum[J]. Biotechnology for Biofuels, 2015, 8: 91. DOI:10.1186/s13068-015-0269-0 |
| [2] |
Shi AQ, Zhu XN, Lu J, et al. Activating transhydrogenase and NAD kinase in combination for improving isobutanol production[J]. Metabolic Engineering, 2013, 16: 1-10. DOI:10.1016/j.ymben.2012.11.008 |
| [3] |
Lindner SN, Niederholtmeyer H, Schmitz K, et al. Polyphosphate/ATP-dependent NAD kinase of Corynebacterium glutamicum: biochemical properties and impact of ppnK overexpression on lysine production[J]. Applied Microbiology and Biotechnology, 2010, 87(2): 583-593. DOI:10.1007/s00253-010-2481-y |
| [4] |
Park SH, Kim HU, Kim TY, et al. Metabolic engineering of Corynebacterium glutamicum for L-arginine production[J]. Nature Communications, 2014, 5: 4618. DOI:10.1038/ncomms5618 |
| [5] |
Kim SY, Lee J, Lee SY. Metabolic engineering of Corynebacterium glutamicum for the production of L-ornithine[J]. Biotechnology and Bioengineering, 2015, 112(2): 416-421. |
| [6] |
Man ZW, Xu MJ, Rao ZM, et al. Systems pathway engineering of Corynebacterium crenatum for improved L-arginine production[J]. Scientific Reports, 2016, 6: 28629. DOI:10.1038/srep28629 |
| [7] |
Kalinowski J, Bathe B, Bartels D, et al. The complete Corynebacterium glutamicum ATCC 13032 genome sequence and its impact on the production of L-aspartate-derived amino acids and vitamins[J]. Journal of Biotechnology, 2003, 104(1/3): 5-25. |
| [8] |
Jäger W, Schäfer A, Pühler A, et al. Expression of the Bacillus subtilis sacB gene leads to sucrose sensitivity in the gram-positive bacterium Corynebacterium glutamicum but not in Streptomyces lividans[J]. Journal of Bacteriology, 1992, 174(16): 5462-5465. DOI:10.1128/jb.174.16.5462-5465.1992 |
| [9] |
Schäfer A, Tauch A, Jäger W, et al. Small mobilizable multi-purpose cloning vectors derived from the Escherichia coli plasmids pK18 and pK19: selection of defined deletions in the chromosome of Corynebacterium glutamicum[J]. Gene, 1994, 145(1): 69-73. |
| [10] |
Hu JY, Tan YZ, Li YY, et al. Construction and application of an efficient multiple-gene-deletion system in Corynebacterium glutamicum[J]. Plasmid, 2013, 70(3): 303-313. DOI:10.1016/j.plasmid.2013.07.001 |
| [11] |
Xu DQ, Tan YZ, Huan XJ, et al. Construction of a novel shuttle vector for use in Brevibacterium flavum, an industrial amino acid producer[J]. Journal of Microbiological Methods, 2010, 80(1): 86-92. DOI:10.1016/j.mimet.2009.11.003 |
| [12] |
Xu DQ, Tan YZ, Shi F, et al. An improved shuttle vector constructed for metabolic engineering research in Corynebacterium glutamicum[J]. Plasmid, 2010, 64(2): 85-91. DOI:10.1016/j.plasmid.2010.05.004 |
| [13] |
Pyne ME, Moo-Young M, Chung DA, et al. Coupling the CRISPR/Cas9 system with lambda red recombineering enables simplified chromosomal gene replacement in Escherichia coli[J]. Applied and Environmental Microbiology, 2015, 81(15): 5103-5114. DOI:10.1128/AEM.01248-15 |
| [14] |
Jiang WY, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology, 2013, 31(3): 233-239. |
| [15] |
Jiang Y, Qian FH, Yang JJ, et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum[J]. Nature Communications, 2017, 8: 15179. DOI:10.1038/ncomms15179 |
| [16] |
Theron G, Reid SJ. ArgR-promoter interactions in Corynebacterium glutamicum arginine biosynthesis[J]. Biotechnology and Applied Biochemistry, 2011, 58(2): 119-127. DOI:10.1002/bab.v58.2 |
| [17] |
Chen C, Li YY, Hu JY, et al. Metabolic engineering of Corynebacterium glutamicum ATCC13869 for L-valine production[J]. Metabolic Engineering, 2015, 29: 66-75. DOI:10.1016/j.ymben.2015.03.004 |
| [18] |
van der Rest ME, Lange C, Molenaar D. A heat shock following electroporation induces highly efficient transformation of Corynebacterium glutamicum with xenogeneic plasmid DNA[J]. Applied Microbiology and Biotechnology, 1999, 52(4): 541-545. |
| [19] |
Okibe N, Suzuki N, Inui M, et al. Efficient markerless gene replacement in Corynebacterium glutamicum using a new temperature-sensitive plasmid[J]. Journal of Microbiological Methods, 2011, 85(2): 155-163. DOI:10.1016/j.mimet.2011.02.012 |
| [20] |
Nakamura J, Kanno S, Kimura E, et al. Temperature-sensitive cloning vector for Corynebacterium glutamicum[J]. Plasmid, 2006, 56(3): 179-186. DOI:10.1016/j.plasmid.2006.05.003 |
| [21] |
Huang YY, Li L, Xie S, et al. Recombineering using RecET in Corynebacterium glutamicum ATCC 14067 via a self-excisable cassette[J]. Scientific Reports, 2017, 7(1): 7916. DOI:10.1038/s41598-017-08352-9 |
| [22] |
Tsuge Y, Suzuki N, Inui M, et al. Random segment deletion based on IS31831 and Cre/loxP excision system in Corynebacterium glutamicum[J]. Applied Microbiology and Biotechnology, 2007, 74(6): 1333-1341. DOI:10.1007/s00253-006-0788-5 |
| [23] |
Suzuki N, Inui M, Yukawa H. Site-directed integration system using a combination of mutant lox sites for Corynebacterium glutamicum[J]. Applied Microbiology and Biotechnology, 2007, 77(4): 871-878. DOI:10.1007/s00253-007-1215-2 |
| [24] |
Hur JK, Kim K, Been KW, et al. Targeted mutagenesis in mice by electroporation of Cpf1 ribonucleoproteins[J]. Nature Biotechnology, 2016, 34(8): 807-808. DOI:10.1038/nbt.3596 |
 2019, Vol. 46
2019, Vol. 46