扩展功能
文章信息
- 庄淼, 张智敏, 王宝腾, 金锋杰
- ZHUANG Miao, ZHANG Zhi-Min, WANG Bao-Teng, JIN Feng-Jie
- 应用双分子荧光互补技术分析米曲霉Fus3与Ste12之间的蛋白互作
- Interaction between Fus3 and Ste12 in Aspergillus oryzae analysed by Bimolecular fluorescence complementation
- 微生物学通报, 2019, 46(2): 269-277
- Microbiology China, 2019, 46(2): 269-277
- DOI: 10.13344/j.microbiol.china.180951
-
文章历史
- 收稿日期: 2018-11-25
- 接受日期: 2019-01-02
- 网络首发日期: 2019-01-08
米曲霉(Aspergillus oryzae)是一种丝状真菌,在发酵工业中具有重要的作用,是我国传统酿造食品酱、酱油和酒类的生产菌株[1],也可用于工业生产蛋白酶、淀粉酶、纤维素酶、植酸酶等[2]。米曲霉在食品工业中有着上千年的安全应用历史,是美国食品与药品管理局(Food and Drug Administration,FDA)公布的安全微生物菌种(GRAS级)[3],也是世界保健组织公布的绝对安全性食品[4]。在众多曲霉属家族中,米曲霉有着十分重要的地位。尽管米曲霉在发酵工业等应用领域有着重要的地位,但由于它属于多核细胞且没有有性生殖等因素,导致它的基因操作手段受到了一定的限制,与其他模式菌株相比其基础研究进展缓慢,需要进一步开发新的研究手段,为米曲霉基础研究和工业生产应用提供技术支持[5]。
细胞内蛋白质之间的相互作用或形成蛋白复合物在细胞的生命活动过程中起着重要的作用。比如:转录因子常通过蛋白间相互作用形成二聚体或多聚体调节下游蛋白的表达[6];蛋白质磷酸化或脱磷酸化等反应也都需要蛋白之间的相互作用[7]。目前对蛋白质相互作用研究的实验方法也十分多样,主要可分为体外实验和体内实验。体外实验如免疫共沉淀、表面等离子共振、蛋白质芯片等;体内实验如酵母双杂交、荧光共振能量转移、蛋白质片段互补、双分子荧光互补技术(Bimolecular fluorescence complementation,BiFC)等[8]。其中BiFC技术起源于蛋白质片段互补技术[7],是近年来发展的在活细胞中研究蛋白相互作用的一种新技术。相比其他体外实验或酵母双杂交等手段,BiFC技术可以更直观地观察活体细胞中两种蛋白质的相互作用以及胞内作用位点,可以更好地把蛋白之间互作关系和它们的功能联系起来。但是截至目前,在米曲霉中蛋白质相互作用的研究主要还是依赖于酵母双杂交体系[9-10],尚未应用BiFC实验体系。
生物信号传递是指细胞感知胞外刺激后,通过细胞内信号分子的逐级传递作用,最终诱导产生一系列的细胞质、细胞核内事件和各种生理生化反应的过程。而促分裂原活化蛋白激酶(Mitogen-activated protein kinase,MAPK)是一组能被不同的细胞外刺激,如细胞因子、神经递质、激素、细胞应激及细胞黏附等激活的丝氨酸-苏氨酸蛋白激酶。MAPK信号通路是利用逐级磷酸化过程将信号放大传至细胞核内调节转录因子的活性,调控相应基因的表达,进而引起细胞反应的一类重要信号系统。MAPK级联激活是多种信号通路的中心,参与到细胞的分裂、分化、凋亡等多种生命过程[11-12]。这一途径广泛存在于各种真核生物(酵母、植物、哺乳动物)中,起到承上启下及整合、放大和传递信号的作用[13]。Fus3蛋白是这一级联途径的最后一个激酶,在酵母细胞中被上游的激酶磷酸化后,活化的Fus3进入细胞核,与细胞核中的转录因子Ste12相互作用,致使Ste12进一步磷酸化启动下游的有性繁殖[14]。而在丝状真菌中,情况似乎更复杂,目前在丝状真菌模式菌株构巢曲霉(Aspergillus nidulans)中发现,Fus3 (MpkB)与Ste12 (SteA)通过相互作用来启动有性发育,同时Fus3还可以使VeA磷酸化,VeA主要负责调节有性发育和次级代谢产物的生成[15-16]。为了探究属于无性繁殖系的米曲霉中Fus3与Ste12蛋白是否也存在相互作用,从而探究它们进一步的功能,本研究通过双分子荧光互补(BiFC)实验,验证米曲霉中的Fus3与Ste12蛋白存在的相互作用关系,为深入研究米曲霉其他蛋白相互作用与功能研究提供新的实验方法并奠定基础。
1 材料与方法 1.1 材料 1.1.1 引物、质粒和菌株实验中使用的引物、质粒和米曲霉菌株见表 1、2和3。米曲霉野生株Aspergillus oryzae RIB40的营养缺陷型宿主菌niaD300 (niaD-),由日本东京大学微生物实验室提供。大肠杆菌(Escherichia coli) DH5α购自TaKaRa公司。以上菌株分别用于米曲霉和大肠杆菌的转化实验。
| 引物名称 Primers name |
引物序列 Primers sequence (5′→3′) |
| Fus3(qPCR)-F | TCACGAAAGATCTCCTTCAACG |
| Fus3(qPCR)-R | CACGCAAAGTTCGCAAGCAGAA |
| Ste12(qPCR)-F | CCTCCTCAGAAACCGGAAACAT |
| Ste12(qPCR)-R | GTTGATCTGGTTGCCAGTCTAC |
| histone H2A(qPCR)-F | GTTCACCGTTTGCTTCGTAAGG |
| histone H2A(qPCR)-R | GGTCTTCTTGTTATCACGAGCAG |
| aB4-PamyB-F | GGGGACAACTTTGTATAGAAAAGTTGCAGGAAACAGCTATGACCATGATTACGATATC |
| Fusion-PamyB-R | CTTGCTCACCATGGACTCACGAATAGCAAGGAATTCGG |
| Fusion-nEGFP-F | CTATTCGTGAGTCCATGGTGAGCAAGGGCGAGGAG |
| nEGFP-linker-R | CGTGGCGATGGAGCGCATGATATAGACGTTGTGGCT |
| aB1-TamyB-F | GGGGACAAGTTTGTACAAAAAAGCAGGCTGATCTGTAGTAGCTCGTGAAGGGTG |
| Fusion-TamyB-R | CATAGCTGTTTCCTGTTTCCTATAATAGACTAGCGTGCTTGGCG |
| Fusion-PamyB-F | CTAGTCTATTATAGGAAACAGGAAACAGCTATGACCATGATTACGATATC |
| aB2-PamyB-R | GGGGACCACTTTGTACAAGAAAGCTGGGTGGACTCACGAATAGCAAGGAATTCGG |
| aB2-cEGFP-F | GGGGACAGCTTTCTTGTACAAAGTGGATGGCCGACAAGCAGAAGAACGG |
| cEGFP-linker-R | GTGGTTCATGACCTTCTGTTTCAGGTCGTTCGGGATCTTGCAGGCCGGGCGCTTGTACAGCTCGTCCATGCC |
| aB1-Sma I-nEGFP-R | GGGGACTGCTTTTTTGTACAAACTTGTCAcccgggCGTGGCGATGGAGCGCATGATA |
| aB3-Sma I-cEGFP-R | GGGGACAACTTTGTATAATAAAGTTGTCAcccgggGTGGTTCATGACCTTCTGTTTCAGGTC |
| Fus3-F | ATGGTGCAGCAACTTCCTCCC |
| Fus3-R | TCACCGCATGATCTCCTCGTAG |
| Ste12-F(IF)3' | GGTCATGAACCACCCC-ATGTTTCCGCAACATGGTGCTCCCATGGCAC |
| Ste12-R(IF)3' | AATAAAGTTGTCACCC-CTACATCTGCTGTTGTAGAAGTTGCGGAGCA |
| 质粒 Plasmids |
描述 Description |
| pg5'PaNG | amyB promoter+negfp (Sma I) |
| pgETPaB | amyB terminator+amyB promoter |
| pg3'CG | cegfp (Sma I) |
| pg5'PaNGFus3 | amyB promoter+negfp+Fus3 |
| pg3'CGSte12 | cegfp+Ste12 |
| pgDFuStG | amyB promoter+negfp+Fus3+amyB terminator+amyB promoter+cegfp+Ste12 |
| pgDNGCG | amyB promoter+negfp+amyB terminator+ amyB promoter+cegfp |
| 菌株Strains | 描述Description | 样本来源Source |
| Aspergillus oryzae niaD300 | niaD- | From the University of Tokyo |
| FuSt | niaD-, pgDFuStG::niaD | This study |
| NGCG | niaD-, pgDNGCG::niaD | This study |
MultiSite Gateway克隆系统,Invitrogen公司;RNA提取试剂盒、RNA反转录试剂盒、In-Fusion HD克隆试剂盒、质粒提取试剂盒、荧光染料SYBR® Premix Ex Taq II和ROX Reference Dye,TaKaRa公司;常规化学试剂均为国产分析纯,国药集团化学试剂有限公司。
定量PCR仪,Applied Biosystems公司;PCR仪,Bio-Rad公司;荧光显微镜,Olympus公司。
1.1.3 主要培养基CD(Glu)培养基(g/L):硝酸钠3.00,氯化钾2.00,磷酸二氢钾1.00,七水硫酸镁0.50,七水硫酸亚铁0.02,葡萄糖20.00,琼脂粉20.00,加蒸馏水至1 L,pH 5.5。
CD(Dex)培养基(g/L):硝酸钠3.00,氯化钾2.00,磷酸二氢钾1.00,七水硫酸镁0.50,七水硫酸亚铁0.02,糊精20.00,琼脂粉20.00,加蒸馏水至1 L,pH 5.5。
CD(Sta)培养基(g/L):硝酸钠3.00,氯化钾2.00,磷酸二氢钾1.00,七水硫酸镁0.50,七水硫酸亚铁0.02,淀粉20.00,加蒸馏水至1 L,pH 5.5。
YPD培养基(g/L):酵母提取物10.0,蛋白胨20.0,葡萄糖20.0,琼脂粉20.0,加蒸馏水至1 L,pH 5.5。
LB培养基(g/L):蛋白胨10.00,酵母粉5.00,氯化钠10.00,琼脂粉20.00,加蒸馏水至1 L,pH自然。
1.2 方法 1.2.1 米曲霉RNA的提取与cDNA的合成分别接种米曲霉到液体培养基(40 mL液体培养基中接种106个分生孢子)和固体培养基,30 ℃培养2 d,液体培养的摇床转速为150 r/min,培养结束后使用漏斗和滤纸过滤菌体,并使用灭菌超纯水冲洗后称取50 mg菌体,同时刮取约50 mg在固体培养基上培养2 d的菌丝体,按RNA提取试剂盒说明分别提取米曲霉总RNA。利用NanoDrop2000C检测RNA浓度,并调成同一浓度100 ng/μL,同时利用1.2%琼脂凝胶电泳检测RNA的完整性和质量。
RNA反转录按照试剂盒说明书操作,cDNA反转录条件为:37 ℃ 15 min,85 ℃ 5 s。反应结束后将浓度调成统一浓度。
1.2.2 实时荧光定量PCR实验使用表 1中的引物Fus3(qPCR)-F/Fus3(qPCR)-R、Ste12(qPCR)-F/Ste12(qPCR)-R分别对AO090003000402、AO090009000638进行实时荧光定量PCR,并使用histone H2A作为内参基因。实时荧光定量PCR的反应体系,参照SYBR® Premix Ex Taq进行,PCR反应体系:2×SYBR Premix Ex Taq 10 μL,上、下游引物(10 μmol/L)各0.8 μL,50×ROX Reference Dye 0.4 μL,cDNA模板1 μL,加灭菌超纯水补足20 μL。PCR反应条件:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s,40个循环;95 ℃ 15 s;60 ℃ 60 s;95 ℃ 15 s。
1.2.3 质粒构建使用MultiSite Gateway克隆系统通过BP/LR重组反应来构建质粒,并使用In-Fusion HD克隆试剂盒通过In-Fusion重组反应构建质粒。
为了表达融合蛋白nEGFP-Fus3和cEGFP-Ste12,构建质粒pg5'PaNG:使用引物aB4-PamyB-F和Fusion-PamyB-R扩增amyB启动子,获得amyB(p)。使用引物Fusion-nEGFP-F和nEGFP-linker-R扩增egfp的N端;并以此为模板,使用引物Fusion- nEGFP-F和aB1-Sma I-nEGFP-R进行扩增,获得negfp(Sma I)。这两段扩增的片段存在同源区段,可通过Fusion PCR技术进行融合,获得amyB(p)- negfp(Sma I)。通过BP重组反应,amyB(p)-negfp(Sma I)可以克隆到质粒pDONRTMP4-P1R中,产生质粒pg5'PaNG。使用引物Fus3-F和Fus3-R扩增编码Fus3的DNA获得fus3,然后通过DNA连接酶插入pg5'PaNG的Sma I酶切位点,得到pg5'NGFus3。
使用引物aB1-TamyB-F和Fusion-TamyB-R,以及Fusion-PamyB-F和aB2-PamyB-R分别扩增amyB终止子和启动子,获得amyB(t)和amyB(p)。这两段扩增的片段存在同源区段,可以通过融合PCR连接获得amyB(t)-amyB(p)。通过BP重组反应,amyB(t)-amyB(p)可以克隆到载体pDONRTM221中,产生质粒pgETPaB。
使用引物aB2-cYFP-F和cYFP-linker-R扩增egfp的C端,并以此为模板使用引物aB2-cEGFP-F和aB3-Sma I-cEGFP-R进行扩增,获得cegfp(Sma I)。然后通过BP重组反应克隆到载体pDONRTM P2R-P3中,产生质粒pg3'CG。使用引物Ste12-F (IF)3'和Ste12-R(IF)3'扩增编码Ste12的DNA,获得ste12,然后使用In-Fusion克隆系统将其插入pg3'CG的Sma I位点,产生质粒pg3'CGSte12。
将产生的3个质粒(pg5'NGFus3,pgETPaB,pg3'CGSte12)与含有niaD选择标记和amyB终止子的目的载体pgDN[17]混合,用于LR重组反应。产生的质粒pgDFuStG用于在米曲霉菌株中转化表达融合蛋白nEGFP-Fus3和cEGFP-Ste12。将产生的3个质粒(pg5'PaNG,pgETPaB,pg3'CG)与含有niaD选择标记和amyB终止子的目的载体pgDN混合,用于LR重组反应。产生的质粒pgDNGCG转化米曲霉获得的菌株用于实验的阴性对照。
1.2.4 重组米曲霉菌株的构建和培养为了产生表达融合蛋白nEGFP-Fus3和cEGFP-Ste12的菌株FuSt,将质粒pgDFuStG导入米曲霉菌株niaD300[18]中,使用选择培养基CD(Glu)筛选出转化菌株FuSt。与此同时,将质粒pgDNGCG导入米曲霉菌株niaD300中,使用选择培养基CD(Glu)获得对照株NGCG。将获得的菌株FuSt和NGCG分别接种到CD(Dex)液体培养基,30 ℃静置培养17 h,荧光显微镜观察。
2 结果与分析 2.1 识别米曲霉中Fus3和Ste12的同源基因前期研究表明构巢曲霉中的Fus3(MpkB)与Ste12(SteA)存在相互作用关系,通过BLAST在米曲霉中检索到了这两个的同源基因fus3 (AO090003000402)和ste12 (AO090009000638)。BLASTp分析结果表明,米曲霉的Fus3与构巢曲霉的MpkB中氨基酸相似性高达98.6%,米曲霉的Ste12与构巢曲霉的SteA中氨基酸相似性也有80%。以Fus3-F和Fus3-R、Ste12-F (IF)3'和Ste12-R(IF)3'为扩增引物,米曲霉野生菌株RIB40基因组为模板,通过PCR分别扩增出约1.2 kb的fus3 (AO090003000402)和2.4 kb的ste12 (AO090009000638)。
2.2 米曲霉中fus3和ste12基因的表达水平采用qRT-PCR比较米曲霉菌株fus3和ste12基因在CD(Glu)琼脂培养基、CD(Glu)液体培养基、CD(Dex)琼脂培养基、CD(Dex)液体培养基、CD(Sta)液体培养基、YPD琼脂培养基、YPD液体培养基这7种培养基中的表达情况,结果如图 1所示。fus3和ste12基因在不同培养基有相似的表达趋势,推测这两个基因有相近的表达背景,其编码蛋白有可能参与到相似的细胞功能;另外,两个基因在CD琼脂培养基均表现出较高的表达水平,表达水平没有因为培养基中碳源的改变而改变,即当CD培养基中的碳源由葡萄糖变成多糖糊精时,表达量没有明显变化。但是,当氮源由无机氮变为有机氮时,两个基因的表达量显著降低,说明在琼脂培养基中,相对饥饿的条件下有助于两个基因的表达,而有机氮源对两个基因的表达有抑制作用。然而这个趋势在液体培养条件下正好是相反的。两个基因在液体CD(Glc)最小培养基培养条件下表现相对低的表达水平,而在多糖和有机氮作为碳、氮源的培养条件下表达水平均有所提高。

|
| 图 1 不同营养成分的培养基对米曲霉菌fus3和ste12基因表达的影响 Figure 1 Relative expression level of fus3 and ste12 genes in Aspergillus oryzae under different culture conditions 注:A:fus3基因的表达;B:ste12基因的表达. Note: A: fus3 gene expression; B: ste12 gene expression. |
|
|
选用在米曲霉中表达能力较强的淀粉酶启动子amyB(p)驱动nEGFP与cEGFP组合的融合蛋白。将amyB(p)-negfp(Sma I)、amyB(t)-amyB(p)和cegfp(Sma I)通过BP重组反应分别克隆到质粒pDONR™P4-P1R、pDONRTM221和pDONRTM P2R-P3中,转化E. coli DH5α后挑取阳性克隆,采用试剂盒法提取质粒,经DNA测序验证获得质粒pg5'PaNG、pgETPaB和pg3'CG。将fus3和ste12基因扩增的片段分别连接至经Sma I酶切的质粒pg5'PaNG和pg3'CG上,然后转化E. coli DH5α并挑取阳性克隆,采用试剂盒法提取质粒,经DNA测序验证获得质粒pg5'NGFus3和pg3'CGSte12。pg5'PaNG、pgETPaB、pg3'CG和pgDN通过LR重组反应产生质粒pgDNGCG (图 2A)。pg5'NGFus3、pgETPaB、pg3'CGSte12和pgDN通过LR重组反应产生质粒pgDFuStG (图 2B)。
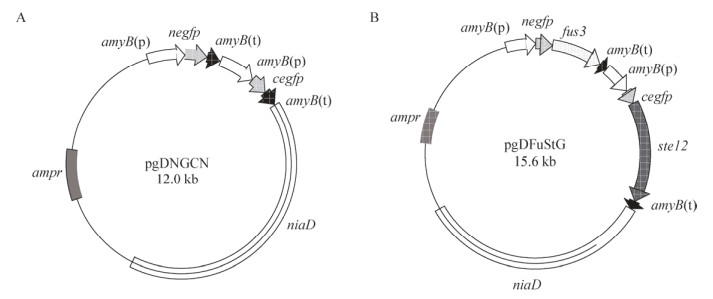
|
| 图 2 重组质粒pgDNGCG和pgDFuStG的构建 Figure 2 Construction of recombinant plasmid pgDNGCG and pgDFuStG 注:A:对照株载体pgDNGCG;B:实验株载体pgDFuStG. Note: A: Plasmid pgDNGCG of control strain; B: Plasmid pgDFuStG of experimental strain. |
|
|
将构建好的重组质粒pgDFuStG和pgDNGCG分别转化米曲霉niaD300,使用选择培养基CD(Glc)筛选出转化菌株FuSt和对照株NGCG。将获得的菌株FuSt和NGCG分别接种到CD(Dex)液体培养基,30 ℃静置培养17 h后进行荧光观察(图 3)。在绿色荧光视野可以看出,FuSt菌株部分菌丝中明显观察到EGFP绿色荧光,而在阴性对照中全视野内没有观察到绿色荧光,进一步验证了米曲霉Fus3和Ste12蛋白互作的真实性以及米曲霉蛋白互作BiFC实验体系的成功建立。由于绿色荧光视野较暗无法观察菌丝内部结构,使用DIG微分干涉可以观察到菌丝内部的形态结构,以此可大致推断出两蛋白相互作用的位置。在DIG视野中可观察到的较大气泡为液泡,绿色荧光的位置基本与液泡位置不重合。实验使用的菌丝仅培养17 h,菌丝处于早期生长状态,绿色荧光部位的形态和位置与早期细胞核形态相似,而且由于转录因子Ste12已被证实定位在细胞核,因此推测相互作用发生的位点在细胞核,但还需要进一步的实验验证。

|
| 图 3 双分子荧光互补实验验证米曲霉中Fus3和Ste12蛋白之间的互作 Figure 3 Confirm the interactions between Fus3 and Ste12 in Aspergillus oryzae by BiFC 注:DIC:微分干涉;GFP:绿色荧光视野;NGCG:对照菌株菌丝;FuSt:Fus3-Ste12转化菌株菌丝. Note: DIC: Differential interference; GFP: Field of green fluorescence; NGCG: Control strain hyphae; FuSt: Fus3-Ste12 transformed strain hyphae. |
|
|
在绿色荧光视野虽然可以观察到部分菌丝的绿色荧光,但并不是所有菌丝都能观察到,这说明Fus3和Ste12蛋白在特定的条件下才会发生相互作用。使用DIG微分干涉进行观察发现,Fus3和Ste12蛋白会发生相互作用的菌丝大多是液泡存在较多且较大的菌丝,即状态不是很好的菌丝。由此推测菌丝在状态不够良好或受到某些外界压力的条件下,这两个蛋白会产生相互作用。因此设计了一些压力实验,例如在培养基中加入1 mol/L NaCl或将培养温度升高到37 ℃等,通过荧光观察发现发出绿色荧光的菌丝并没有明显增多(数据未显示)。综上,Fus3和Ste12蛋白存在相互作用,但相互作用的激发条件还需进一步研究。
3 讨论BiFC技术是能够快速且直观地判断目标蛋白在活细胞中的定位和相互作用的新技术[19]。目前虽然已经开发了一系列实验方法用于分析蛋白质之间的相互作用,但大多数方法需要破坏细胞来检测分子间相互作用,有些方法只能间接判断两蛋白的相互作用。BiFC技术利用荧光蛋白本身的一个特点:荧光蛋白活性被重建后能够自我催化形成荧光活性中心,重新恢复荧光蛋白的特征光谱发出荧光,自身作为报告蛋白,直接反映蛋白质之间的相互作用[20]。BiFC技术为活细胞和生物体中分子相互作用的可视化提供了最直接的方法。2002年,Hu等[21]利用被切开的黄色荧光蛋白(YFP)的2个非荧光片段来研究bZIP与Rel家族转录因子之间的相互作用,并且首次提出了BiFC的概念。Michielse等[22]利用该技术研究水稻恶苗病菌(Fusarium fujikuroi)中GATA转录因子AreA和AreB相互关系的结果表明,在氮饥饿的环境下AreA和AreB定位于细胞核且会发生相互作用。已有研究表明酿酒酵母(Saccharomyces cerevisiae)中的Fus3与转录因子Ste12存在相互作用[14];Bayram等[16]利用BiFC技术验证了构巢曲霉Fus3与Ste12之间的相互作用,进一步证实了激酶和转录因子之间的相互作用从酵母到丝状真菌是保守的。Okabe等[23]利用BiFC技术研究了米曲霉的自融合能力以及异核体的不亲和性。本研究成功建立了米曲霉BiFC系统,并且验证了Fus3与转录因子Ste12存在相互作用,由于转录因子Ste12已被证实定位在细胞核,因此推测相互作用发生的位点在细胞核,这需要进一步的验证。
由于米曲霉与构巢曲霉不同,米曲霉中并不存在有性生殖,因此无法直接进行杂交等实验。此外,米曲霉中可选用的选择标记基因有限,因此我们通过使用MultiSite Gateway克隆系统构建米曲霉的BiFC质粒,即在一个载体质粒中同时表达两种融合蛋白,从而为米曲霉蛋白互作功能研究提供一种新的检测技术和方法。尽管在构巢曲霉中Fus3与Ste12已被验证存在相互作用,但研究发现它们是通过相互作用来启动有性发育。然而米曲霉中至今未发现有性生殖,因此在米曲霉中确认它们之间的相互作用仍然非常具有功能上的研究意义。
4 结论本研究将2个蛋白分别连接到EGFP截断的N末端和C末端,当这2个蛋白相互作用靠近时,EGFP截断的N末端和C末端便形成完整的EGFP发出绿色荧光。本试验成功构建了荧光表达载体,并且应用空BiFC载体为对照,得到转化株后在CD(Dex)液体培养17 h,然后通过激光共聚焦显微镜观察,FuSt检测到绿色荧光信号而NGCG未检测到荧光,暗示Fus3与转录因子Ste12在米曲霉中存在相互作用。
| [1] |
Liu LP, Liu LH. Study and application of Aspergillus oryzae in different domains[J]. China Condiment, 2008, 33(4): 28-32. (in Chinese) 刘丽萍, 刘丽华. 米曲霉研究进展与应用[J]. 中国调味品, 2008, 33(4): 28-32. DOI:10.3969/j.issn.1000-9973.2008.04.003 |
| [2] |
Chang SJ, Zu YP, Tao B, et al. Construction of a full-length cDNA library from the Aspergillus oryzae RIB40[J]. Microbiology China, 2014, 41(8): 1485-1490. (in Chinese) 常淑君, 祖永平, 陶波, 等. 米曲霉(Aspergillus oryzae) RIB40全长cDNA文库的构建[J]. 微生物学通报, 2014, 41(8): 1485-1490. |
| [3] |
Yamada O, Ikeda R, Ohkita Y, et al. Gene silencing by RNA interference in the Koji mold Aspergillus oryzae[J]. Bioscience, Biotechnology, and Biochemistry, 2007, 71(1): 138-144. DOI:10.1271/bbb.60405 |
| [4] |
Abe K, Gomi K, Hasegawa F, et al. Impact of Aspergillus oryzae genomics on industrial production of metabolites[J]. Mycopathologia, 2006, 162(3): 143-153. DOI:10.1007/s11046-006-0049-2 |
| [5] |
Zhang ZM, Zhuang M, Jin FJ. Advances in gene engineering technologies for Aspergillus oryzae[J]. Biotechnology Bulletin, 2018, 34(9): 170-176. (in Chinese) 张智敏, 庄淼, 金锋杰. 米曲霉基因工程技术的进展[J]. 生物技术通报, 2018, 34(9): 170-176. |
| [6] |
Wang L, Tang T, Ouyang WY, et al. A study on the interaction of transcription factor AP-2beta and p53 protein[J]. Acta Laser Biology Sinica, 2017, 26(2): 189-192. (in Chinese) 王琳, 汤婷, 欧阳文英, 等. 转录因子AP-2beta和p53蛋白相互作用的研究[J]. 激光生物学报, 2017, 26(2): 189-192. DOI:10.3969/j.issn.1007-7146.2017.02.016 |
| [7] |
Gao X. The mechanism of myosin phosphorylation to actin-myosin interaction[D]. Yangling: Masterʼs Thesis of Northwest Agriculture and Forestry University, 2016 (in Chinese) 高星.肌球蛋白磷酸化调控肌动-肌球蛋白相互作用的机理研究[D].杨淩: 西北农林科技大学硕士学位论文, 2016 |
| [8] |
Fan JY, Cui ZQ, Zhang XE. Bimolecular fluorescence complementation technique[J]. Chinese Journal of Biochemistry and Molecular Biology, 2008, 24(8): 767-774. (in Chinese) 樊晋宇, 崔宗强, 张先恩. 双分子荧光互补技术[J]. 中国生物化学与分子生物学报, 2008, 24(8): 767-774. DOI:10.3969/j.issn.1007-7626.2008.08.013 |
| [9] |
Jin FJ, Nishida M, Hara S, et al. Identification and characterization of a putative basic helix-loop-helix transcription factor involved in the early stage of conidiophore development in Aspergillus oryzae[J]. Fungal Genetics and Biology, 2011, 48(12): 1108-1115. DOI:10.1016/j.fgb.2011.10.001 |
| [10] |
Zhuang M, Jin FJ. Research progress of Aspergillus bHLH transcription factors[J]. Microbiology China, 2018, 45(10): 2243-2253. (in Chinese) 庄淼, 金锋杰. 曲霉属bHLH转录因子的研究进展[J]. 微生物学通报, 2018, 45(10): 2243-2253. |
| [11] |
Mu JY, Chen XG. The progress of the study on the mitogen-activated protein kinase pathways[J]. Chinese Bulletin of Life Sciences, 2002, 14(4): 208-211, 203. (in Chinese) 牟金叶, 陈晓光. 促分裂原激活的蛋白激酶(MAPK)信号传导通路的研究进展[J]. 生命科学, 2002, 14(4): 208-211, 203. DOI:10.3969/j.issn.1004-0374.2002.04.005 |
| [12] |
Hagiwara D, Sakamoto K, Abe K, et al. Signaling pathways for stress responses and adaptation in Aspergillus species: stress biology in the post-genomic era[J]. Bioscience, Biotechnology, and Biochemistry, 2016, 80(9): 1667-1680. DOI:10.1080/09168451.2016.1162085 |
| [13] |
Yang XL, Jiang SJ, Ma J, et al. Identification of MAPK cascade signal pathway and prediction of the model in rice sheath blight pathogen Rhizoctonia solani[J]. Journal of Agricultural Science and Technology, 2017, 19(12): 24-33. (in Chinese) 杨系玲, 姜述君, 马婧, 等. 水稻纹枯病菌MAPK级联信号途径分析及信号通路模型预测[J]. 中国农业科技导报, 2017, 19(12): 24-33. |
| [14] |
Saito H. Regulation of cross-talk in yeast MAPK signaling pathways[J]. Current Opinion in Microbiology, 2010, 13(6): 667-683. |
| [15] |
Paoletti M, Seymour FA, Alcocer MJC, et al. Mating type and the genetic basis of self-fertility in the model fungus Aspergillus nidulans[J]. Current Biology, 2007, 17(16): 1384-1389. DOI:10.1016/j.cub.2007.07.012 |
| [16] |
Bayram Ö, Bayram ÖS, Ahmed YL, et al. The Aspergillus nidulans MAPK module AnSte11-Ste50-Ste7-Fus3 controls development and secondary metabolism[J]. PLoS Genetics, 2012, 8(7): e1002816. DOI:10.1371/journal.pgen.1002816 |
| [17] |
Mabashi Y, Kikuma T, Maruyama JI, et al. Development of a versatile expression plasmid construction system for Aspergillus oryzae and its application to visualization of mitochondria[J]. Bioscience, Biotechnology, and Biochemistry, 2006, 70(8): 1882-1889. DOI:10.1271/bbb.60052 |
| [18] |
Minetoki T, Nunokawa Y, Gomi K, et al. Deletion analysis of promoter elements of the Aspergillus oryzae agdA gene encoding α-glucosidase[J]. Current Genetics, 1996, 30(5): 432-438. |
| [19] |
Cui XY, Zhang JX, Dou Y, et al. Interactions between SSⅠ and PPDK1 of maize using bimolecular fluorescence complementation (BiFC) assay[J]. Journal of Northwest A & F University (Natural Science Edition), 2013, 41(7): 49-53. (in Chinese) 崔喜艳, 张继晓, 窦瑶, 等. 双分子荧光互补技术(BIFC)分析玉米SSⅠ与PPDK1之间的蛋白互作[J]. 西北农林科技大学学报:自然科学版, 2013, 41(7): 49-53. |
| [20] |
Miller KE, Kim Y, Huh WK, et al. Bimolecular fluorescence complementation (BiFC) analysis: advances and recent applications for genome-wide interaction studies[J]. Journal of Molecular Biology, 2015, 427(11): 2039-2055. DOI:10.1016/j.jmb.2015.03.005 |
| [21] |
Hu CD, Chinenov Y, Kerppola TK. Visualization of interactions among bZIP and Rel family proteins in living cells using bimolecular fluorescence complementation[J]. Molecular Cell, 2002, 9(4): 789-798. DOI:10.1016/S1097-2765(02)00496-3 |
| [22] |
Michielse CB, Pfannmüller A, Macios M, et al. The interplay between the GATA transcription factors AreA, the global nitrogen regulator and AreB in Fusarium fujikuroi[J]. Molecular Microbiology, 2014, 91(3): 472-493. |
| [23] |
Okabe T, Katayama T, Mo TN. BiFC-based visualisation system reveals cell fusion morphology and heterokaryon incompatibility in the filamentous fungus Aspergillus oryzae[J]. Scientific Reports, 2018, 8(1): 2922. DOI:10.1038/s41598-018-21323-y |
 2019, Vol. 46
2019, Vol. 46