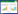扩展功能
文章信息
- 严若冰, 王威, 张雪洪
- YAN Ruo-Bing, WANG Wei, ZHANG Xue-Hong
- 洛蒙德链霉菌S015中cutR/cutS双组分调控系统对洛蒙真菌素合成的调控
- Function of cutR/cutS two component system in lomofungin biosynthesis in Streptomyces lomondensis S015
- 微生物学通报, 2019, 46(2): 261-268
- Microbiology China, 2019, 46(2): 261-268
- DOI: 10.13344/j.microbiol.china.180341
-
文章历史
- 收稿日期: 2018-04-28
- 接受日期: 2018-05-28
土壤中分离的链霉菌、假单胞菌等微生物可以生产具有广谱抗菌、抑菌活性的吩嗪类次级代谢产物,吩嗪及其衍生物对引起小麦全蚀病、水稻枯萎病等的植物病原菌有显著的抑制作用[1],也可用于肺结核及麻风病等疾病的治疗[2]。因此,这类化合物在农业和医药领域越来越受到人们的重视。
大部分天然吩嗪类化合物是以前体物质吩嗪一羧酸(Phenazine-1-carboxylic acid,PCA)或吩嗪-1, 6-二羧酸(Phenazine-1, 6-dicarboxylate,PDC)为基础,经过不同的修饰酶添加侧链基团形成。假单胞菌中吩嗪类化合物生物合成前体为PCA,而在链霉菌中合成的吩嗪类化合物除了以PCA为前体,还可以由PDC作为前体物质[3]。研究表明,作为吩嗪化合物合成前体的PCA和PDC主要来源于莽草酸途径[4]。
我们实验室前期在洛蒙德链霉菌(Streptomyces lomondensis) S015中分离鉴定出一种新型吩嗪类次级代谢产物——洛蒙真菌素(Lomofungin)。与假单胞菌相比,链霉菌中吩嗪合成基因簇相关研究较少。在洛蒙德链霉菌中鉴定得到核心吩嗪合成基因簇lphzGFEDCB,经证明与假单胞菌的吩嗪合成基因簇具有高度相似性[5]。在假单胞菌的莽草酸代谢途径中,由phzABCDEFG 7个基因组成的核心基因簇对吩嗪化合物合成起重要作用。在吩嗪化合物生物合成途径中,phzC基因编码的DAHP合成酶PhzC催化3-脱氧-D-阿拉伯庚酮糖酸-7-磷酸(3-deoxy- D-arabinoheptulosonate-7-phosphate,DAHP)的生成。DAHP进一步经莽草酸途径形成分支酸(Chorismic acid),经PhzE催化[6]转变为2-氨基-4-脱氧分支酸(2-amino-2-desoxyisochorismic acid,ADIC)。ADIC经phzD编码的异构分支酶催化水解得到2, 3-二氢-3-羟基邻氨基苯甲酸(Trans-2, 3-dihydro-3-hydroxyanthranilic acid,DHHA),DHHA经PhzG、PhzF等蛋白催化,随后发生一系列二聚化反应、氧化反应和脱羧反应,最终得到PCA和PDC[7-9]。洛蒙德链霉菌S015的洛蒙真菌素生物合成过程中,分支酸经吩嗪合成基因簇作用得到吩嗪合成前体PDC,推测在这个过程中lphzB和lphzG与吩嗪化合物前体物质成环相关,lphzC作用于分支酸形成,lphzE则编码蛋白催化分支酸生成ADIC。中间产物PDC经侧链修饰反应获得各类吩嗪化合物。前期研究表明,lomo10和lomo14为洛蒙真菌素侧链修饰过程中的侧链羟基化基因,PDC经一系列反应生成洛蒙真菌素生物合成过程中的前体物质,1-甲酯-6-甲酰-4, 9-二羟基吩嗪(1-carbomethoxy-6-formyl-4, 9-dihydroxy- phenazine,CMFDP)[5],该前体经lomo10和lomo14的侧链修饰及其他反应生成洛蒙真菌素,推测的洛蒙德链霉菌S015中洛蒙真菌素合成途径见图 1。
|
| 图 1 推测的洛蒙德链霉菌S015中洛蒙真菌素合成途径 Figure 1 Proposed lomofungin biosynthetic pathway in Streptomyces lomondensis S015 |
|
|
自然界中链霉菌、假单胞菌合成吩嗪类衍生物的产量较低。因此,本实验希望通过基因工程手段改变菌种次级代谢调控网络,以提高次级代谢产物的生物合成。双组分调控系统普遍存在于链霉菌次级代谢产物调控系统中,这些双组分体系通常由膜相关组氨酸激酶、传感器以及在细胞质中作用的反应调节剂组成。传感器检测环境信号或压力,调节蛋白触发细胞通过基因转录调节完成应答[10]。双组分调控系统广泛参与细胞的次级代谢调控过程,cutR/cutS双组分调控系统是链霉菌中最早被鉴定的双组分调控系统,首先在变青链霉菌(Streptomyces lividans)中被发现并鉴定[11]。研究表明,cutR/cutS双组分调控系统在链霉菌模式菌株天蓝色链霉菌(S. coelicolor)中对其主要次级代谢产物放线紫红素(Actinorhodin,ACT)起负调控作用[12],在变青链霉菌(S. lividans)中同样会抑制次级代谢产物的生成[13]。目前关于cutR/cutS双组分调控系统的研究报道较少,关于该系统对链霉菌次级代谢的调控机制尚未有详细阐述。本研究发现在洛蒙德链霉菌S015中该双组分调控系统对洛蒙真菌素的合成起负调控作用。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒本研究所使用的菌株及质粒见表 1。
| 菌株和质粒 Strains & plasmids |
描述 Characteristics |
来源 References |
| Streptomyces lomondensis | ||
| S015 | Wild type | Our lab |
| S015ΔcutR | In frame deletion of cutR | This study |
| S015ΔcutS | In frame deletion of cutS | This study |
| Escherichia coli | ||
| DH5α | For transformation | TransGen biotech |
| ET12567(pUZ8002) | For conjugation, KmR, ChlR | [14] |
| Plasmids | ||
| pKC1139 | AprR | [15] |
| pKC1139-RH | Fused cutR homologous arms inserted in to pKC1139 | This study |
| pKC1139-SH | Fused cutS homologous arms inserted in to pKC1139 | This study |
本研究所使用的引物见表 2。
| 引物名称 Primers name |
引物序列 Primers sequence (5′→3′) |
用途 Purpose |
| R-UF | AAATTTAAGCTTTGAGGAGCCCTGTGACCGAC | cutR left homologous arm clone |
| R-UR | CACCGCATCGGCGAGCAGTT | |
| R-DF | CTGCTCGCCGATGCGGTGCGCCCGTCATCGTCACCGTC | cutR right homologous arm clone |
| R-DR | AAATTTTCTAGATCGACACCCAACCGGACTCCTC | |
| S-UF | AAATTTAAGCTTCCCGCTTCGTACGGCCTGAT | cutS left homologous arm clone |
| S-UR | TGCCGCCGACGATCTTGAAG | |
| S-DF | TCAAGATCGTCGGCGGCACGGTCACATCTACGCCCAGC | cutS right homologous arm clone |
| S-DR | AAATTTTCTAGATCTCGTCTCGCTGCGGTTCAG |
LB培养基(g/L):蛋白胨10.0,酵母提取物5.0,NaCl 10.0,琼脂15.0 (固体培养基),pH 7.5。
YEME液体发酵培养基(g/L):酵母提取物4.0,麦芽糖提取物10.0,葡萄糖4.0,pH 7.0。
MS固体培养基:黄豆饼粉30.0 g,加适量水加热至沸腾,煮沸1 h后用6层纱布过滤,向滤液中加入甘露醇和琼脂各20.0 g,定容至1 L。
2×YT孢子预萌发培养基(g/L):氯化钠5.0,酵母提取物10.0,胰蛋白胨15.0,pH 7.5。
抗生素浓度(mg/L)如下:氨苄青霉素(Ampicillin,Am) 100,安普霉素(Apramycin,Apr) 50,卡那霉素(Kanamycin,Km) 50,氯霉素(Chloramphenicol,Chl) 25,萘啶酮酸(Nalidixic acid) 40。
1.2 主要试剂及仪器乙腈、2-丁酮、甲酸、丙三醇,上海国药集团;蛋白胨、酵母提取物,Oxoid公司;氯化钠、甘露醇、氯化钙,上海凌峰化学试剂有限公司;黄豆饼粉,上海五四有限公司;麦芽糖提取物,北京索莱宝有限公司;限制性内切酶,大连宝生物工程有限公司。高效液相色谱分析仪、色谱柱,安捷伦科技有限公司。
1.3 实验方法 1.3.1 菌株培养所有大肠杆菌的培养均采用LB培养基。洛蒙德链霉菌S015的培养方法参照文献[16]:液体培养或发酵采用YEME培养基,培养温度为28 ℃,摇床转速为220 r/min;固体培养采用MS固体培养基,培养温度为28 ℃。
1.3.2 基因缺失菌株的构建及筛选验证(1) 洛蒙德链霉菌S015全基因组DNA的提取
根据Tiangen试剂盒说明书进行基因组DNA的提取。
(2) 接合转移
接合转移供体菌为E. coli ET12567(pUZ8002),受体菌为S. lomondensis S015。接合转移操作方法参照文献[17]。
(3) 双交换基因缺失突变菌株的筛选和验证
参照文献[17]进行基因缺失突变株的筛选及验证。PCR反应体系:ddH2O 8.5 μL,DMSO 1.25 μL,上、下游引物(10 μmol/L)各0.75 μL,DNA模板1.25 μL,PrimerSTAR Max Premix 12.5 μL。PCR反应条件:98 ℃ 1 min;98 ℃ 20 s,59 ℃ 45 s,72 ℃ 60 s,30个循环;72 ℃ 10 min。
1.3.3 生长曲线测定及产物分析(1) 生长曲线测定
每24 h对发酵液进行取样测定生长曲线。取200 μL发酵菌液置于EP管中,12 000×g离心5 min,弃去上清,以1 mL ddH2O重悬。稀释3倍后以ddH2O为空白对照,利用紫外分光光度计测定OD450值。以发酵时间为横坐标,以OD450值为纵坐标绘制菌株生长曲线。
(2) HPLC分析
发酵液预处理:取培养4 d的发酵液2 mL,12 000×g离心3 min,吸取上层清液1 mL,以6 mol/L的HCl调节pH至2.0,加入1 mL 2-丁酮,振荡混匀5 min萃取,以12 000×g离心5 min。取上层有机相至洁净1.5 mL离心管中,33 ℃真空旋转蒸发仪旋转蒸干后,溶于1 mL乙腈和0.1%甲酸水1:1 (体积比)混合液中,振荡使之充分溶解,使用0.22 μm有机相滤头过滤至洁净HPLC小瓶中,用于产物的HPLC分析。
产物的HPLC分析参照文献[18]。以0.1%甲酸溶液(A)和乙腈(B)为流动相,1-4 min,A:B=8:2;4-20 min,A:B=6:4;20-30 min,A:B=8:2。检测波长为270 nm,流速1 mL/min,柱温30 ℃,使用Agilent Eclipse Plus C18色谱柱(250 mm×4.6 mm,5 μm)检测洛蒙真菌素,保留时间为18 min左右时的产物为洛蒙真菌素。依据峰面积和洛蒙真菌素标准曲线(y=0.015 4x-6.936 4,R2=0.991 8;其中y为产物浓度,单位为mg/L,变量x为峰面积)进行产物定量。
(3) qPCR分析
取1 mL发酵48 h的菌液4 ℃、8 000×g离心5 min收集菌丝体,用带有直径为1 μm玻璃珠的Redzol溶液悬浮菌丝体,在细胞破碎仪上将菌丝体破碎(6 500 r/min, 20 s,间歇20 s,振荡2-3个循环),室温放置2-3 min。匀浆后样品8 000×g离心3 min,向上清中加入200 μL苯酚:氯仿:异戊醇=25:24:1 (体积比)的混合液以去除蛋白,12 000×g离心10 min并向上清中加入200 μL氯仿混匀;12 000×g离心10 min,向上清中加入200 μL无水乙醇混匀并加入离心柱中;12 000×g离心2 min后向离心柱中加50 μL DEPC-water溶解RNA,12 000×g离心1 min收集样品。使用Fermentas公司的RevertAidTM H Minus First Strand cDNA Synthesis Kit进行RNA反转录操作,cDNA的Real-time PCR用SYBR Green Real-time PCR方法[19]进行。对lomo14、lomo10、lphzB、lphzC、lphzE和lphzG 6个基因设计引物,以野生型S015的16S rRNA基因为内参基因,进行qPCR实验。
(4) 统计学分析方法
显著性计算采用双总体t检验方法进行。
2 结果与分析 2.1 S015ΔcutR和S015ΔcutS突变株的构建使用同框敲除的原理构建cutR、cutS单基因缺失突变菌株。使用UF/UR和DF/DR引物对进行PCR扩增分别获取目的基因的上、下游同源臂片段,对获得的上、下游同源臂片段进行融合PCR,通过内切酶Hind III和Xba I双酶切体系同时酶切片段和pKC1139载体,酶切产物使用LigationMix酶连体系进行连接,构建了敲除质粒。将敲除质粒转化至大肠杆菌ET12567(pUZ8002)作为接合转移供体菌,与野生型S015进行接合转移,挑取长出的接合子培养,并用对应引物进行验证,获得正确的目的基因缺失菌株,其基因敲除原理如图 2所示。

|
| 图 2 cutR及cutS基因敲除原理 Figure 2 Mechanism of cutR and cutS knock-out |
|
|
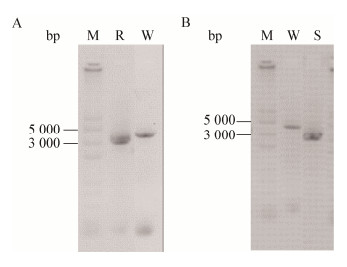
设计引物时,在cutR上游同源臂引物R-UF和下游同源臂引物R-DR的5′端分别引入Hind III和Xba I酶切位点及保护碱基,在下游同源臂引物R-DF的5′端引入一段上游同源臂引物R-UR的Overlap序列以进行后续的融合PCR实验。上游同源臂片段为1 597 bp,下游同源臂片段为1 587 bp,同源臂总长度为3 184 bp,预计敲除cutR基因片段长度为562 bp。
按照同样的方法设计了cutS引物。cutS上游同源臂片段长度为1 517 bp,下游同源臂片段长度为1 566 bp,同源臂总长度为3 083 bp,预计敲除cutS基因片段长度为952 bp。
其中cutR基因缺失菌株采用R-UF/R-DR引物对进行验证,预计野生型菌株的PCR扩增产物条带长度为3 746 bp,cutR基因缺失菌株对应条带长度为3 184 bp,其PCR扩增验证电泳图谱如图 3A所示;cutS基因缺失菌株采用S-UF/S-DR引物对进行PCR验证,预计野生型菌株的PCR扩增产物条带长度为4 035 bp,cutS基因缺失菌株对应条带长度为3 083 bp,其PCR扩增验证电泳图谱如图 3B所示。电泳结果与预期相符,cutR和cutS单基因敲除成功,敲除菌株分别命名为S015ΔcutR和S015ΔcutS。
|
| 图 3 cutR (A)及cutS (B)基因敲除PCR扩增验证电泳图谱 Figure 3 PCR validation of cutR (A) and cutS (B) gene deletion 注:M:DNA marker 2K Plus II;W:Wild-type S015;R: S015ΔcutR突变株;S:S015ΔcutS突变株. Note: M: DNA marker 2K Plus II; W: Wild-type S015; R: S015ΔcutR mutant; S: S015ΔcutS mutant. |
|
|
将洛蒙德链霉菌S015野生型菌株、S015ΔcutR及S015ΔcutS突变株接种至YEME液体培养基中,28 ℃、220 r/min发酵条件下培养,在洛蒙真菌素产量达到最高(培养至4 d)时取样进行HPLC分析,3株菌株发酵产物对应的HPLC实测图见图 4。

|
| 图 4 菌株的发酵产物HPLC实测图 Figure 4 HPLC profiles of fermentation products from different strains |
|
|
每24 h对发酵液取样测定细胞生长曲线,如图 5A所示,S015ΔcutS突变菌株与S015野生型菌株细胞生长没有明显区别,说明cutS基因对细胞生长没有显著影响;S015ΔcutR发酵培养前4天的细胞生长较S015野生型菌株有所下降,说明cutR基因的敲除对前期细胞生长有一定抑制作用。

|
| 图 5 基因敲除对细胞生长(A)和洛蒙真菌素产量(B)的影响 Figure 5 Effects of gene deletion on cell growth (A) and lomofungin production (B) |
|
|
依据洛蒙真菌素标准曲线对发酵4 d时的产物进行定量,如图 5B所示,发酵4 d时S015ΔcutR和S015ΔcutS洛蒙真菌素产量分别达到128.1±25.4 mg/L和61.8±4.5 mg/L,分别是野生型菌株产量的11.5倍和5.5倍。
2.3 cutR和cutS对洛蒙真菌素合成基因的转录影响为考察cutR和cutS两个基因在转录水平上对链霉菌次级代谢的影响。取发酵48 h菌液,用前述方法提取RNA并转录cDNA,以野生型S015的16S rRNA为内参基因,对lomo14、lomo10、lphzB、lphzC、lphzE和lphzG合成基因进行qPCR分析,所得结果如图 6所示。S015ΔcutR突变株中lomo14、lomo10、lphzB、lphzC、lphzE和lphzG的表达量分别达到野生型的1 151.7±88.8、110.5±5.8、129.3±7.7、380.2±34.6、348.2±42.1和299.8±38.2倍;S015ΔcutS突变株中lomo14、lomo10、lphzB、lphzC、lphzE和lphzG的表达量分别达到野生型的4.3±0.5、2.2±0.2、9.3±0.9、10.3±0.6、20.7±1.5和20.4±0.8倍,均较野生型菌株有所提高。

|
| 图 6 敲除株S015ΔcutR (A)及S015ΔcutS (B)中合成基因的相对转录活性 Figure 6 Relatively transcriptional level of biosynthesis genes in strain S015ΔcutR (A) and S015ΔcutS (B) 注:**:t检验结果极显著. Note: **: The results of t test were extremely significant. |
|
|
本文研究了cutR和cutS两个基因对洛蒙德链霉菌S015洛蒙真菌素合成的影响。发现cutR和cutS这两个基因对洛蒙德链霉菌洛蒙真菌素的合成起负调控作用。为进一步研究cutR和cutS对洛蒙真菌素合成的调控机制,我们研究了这两个基因对洛蒙真菌素合成基因lomo14、lomo10、lphzB、lphzC、lphzE和lphzG转录水平的影响。其中lphzB、lphzC、lphzE和lphzG是lphz基因簇上基因,该基因簇作用于吩嗪类化合物合成重要前体物质PDC的生物合成;lomo10和lomo14则是洛蒙真菌素合成过程中其侧链羟基修饰相关的基因。S015ΔcutR和S015ΔcutS突变株洛蒙真菌素合成过程中lomo14、lomo10、lphzB、lphzC、lphzE和lphzG表达水平均相对上调,说明cutR和cutS对这些基因的表达有抑制作用。推测cutR和cutS均可抑制吩嗪中间产物合成及洛蒙真菌素合成的侧链修饰过程,从而抑制洛蒙真菌素合成,其调控机制有待进一步研究。
双组分调控系统普遍存在于链霉菌次级代谢产物调控系统中,并广泛参与细胞次级代谢产物的合成调控。cutR/cutS双组分调控系统已被证明分别在S. coelicolor[8]和S. lividans[9]中抑制其次级代谢产物ACT的生成,但关于该双组分调控系统对链霉菌次级代谢的调控机制尚未有详细阐明。本文首次在洛蒙德链霉菌中鉴定出cutR/cutS双组分调控系统,研究发现其对次级代谢产物洛蒙真菌素具较强的调控作用,初步阐释了该双组分系统在洛蒙德链霉菌S015中调控洛蒙真菌素合成的机理,为进一步对洛蒙德链霉菌进行基因工程改造奠定了基础。
| [1] |
Xu S, Pan XY, Luo JY, et al. Effects of phenazine-1-carboxylic acid on the biology of the plant-pathogenic bacterium Xanthomonas oryzae pv. oryzae[J]. Pesticide Biochemistry and Physiology, 2015, 117: 39-46. DOI:10.1016/j.pestbp.2014.10.006 |
| [2] |
Laursen JB, Nielsen J. Phenazine natural products: biosynthesis, synthetic analogues, and biological activity[J]. Chemical Reviews, 2004, 104(3): 1663-1686. DOI:10.1021/cr020473j |
| [3] |
Kim CG, Yu TW, Fryhle CB, et al. 3-Amino-5-hydroxybenzoic acid synthase, the terminal enzyme in the formation of the precursor of mC7N units in rifamycin and related antibiotics[J]. The Journal of Biological Chemistry, 1998, 273(11): 6030-6040. DOI:10.1074/jbc.273.11.6030 |
| [4] |
Qu L, Liu HZ, Hu HB, et al. The biosynthesis of natural phenazine[J]. Chinese Journal of Antibiotics, 2010, 35(3): 168-174. (in Chinese) 屈丽, 刘宏志, 胡洪波, 等. 天然吩嗪化合物的生物合成[J]. 中国抗生素杂志, 2010, 35(3): 168-174. |
| [5] |
Zhang CX, Sheng CL, Wang W, et al. Identification of the lomofungin biosynthesis gene cluster and associated flavin-dependent monooxygenase gene in Streptomyces lomondensis S015[J]. PLoS One, 2015, 10(8): e136228. |
| [6] |
Parsons JF, Greenhagen BT, Shi K, et al. Structural and functional analysis of the pyocyanin biosynthetic protein PhzM from Pseudomonas aeruginosa[J]. Biochemistry, 2007, 46(7): 1821-1828. DOI:10.1021/bi6024403 |
| [7] |
Gibson J, Sood A, Hogan DA. Pseudomonas aeruginosa-Candida albicans interactions: localization and fungal toxicity of a phenazine derivative[J]. Applied and Environmental Microbiology, 2009, 75(2): 504-513. |
| [8] |
Mentel M, Ahuja EG, Mavrodi DV, et al. Of two make one: the biosynthesis of phenazines[J]. Chembiochem, 2009, 10(14): 2295-2304. DOI:10.1002/cbic.v10:14 |
| [9] |
Mavrodi DV, Peever TL, Mavrodi OV, et al. Diversity and evolution of the phenazine biosynthesis pathway[J]. Applied and Environmental Microbiology, 2010, 76(3): 866-879. |
| [10] |
Cui CY, Yang CX, Song SH, et al. A novel two-component system modulates quorum sensing and pathogenicity in Burkholderia cenocepacia[J]. Molecular Microbiology, 2018, 108(1): 32-44. DOI:10.1111/mmi.13915 |
| [11] |
Tseng HC, Chen CW. A cloned ompR-like gene of Streptomyces lividans 66 suppresses defective melC1, a putative copper-transfer gene[J]. Molecular Microbiology, 1991, 5(5): 1187-1196. DOI:10.1111/mmi.1991.5.issue-5 |
| [12] |
Rodríguez H, Rico S, Díaz M, et al. Two-component systems in Streptomyces: key regulators of antibiotic complex pathways[J]. Microbial Cell Factories, 2013, 12: 127. DOI:10.1186/1475-2859-12-127 |
| [13] |
Chang HM, Chen MY, Shieh YT, et al. The cutRS signal transduction system of Streptomyces lividans represses the biosynthesis of the polyketide antibiotic actinorhodin[J]. Molecular Microbiology, 1996, 21(5): 1075-1085. |
| [14] |
Macneil DJ, Gewain KM, Ruby CL, et al. Analysis of Streptomyces avermitilis genes required for avermectin biosynthesis utilizing a novel integration vector[J]. Gene, 1992, 111(1): 61-68. DOI:10.1016/0378-1119(92)90603-M |
| [15] |
Bierman M, Logan R, O'Brien K, et al. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp.[J]. Gene, 1992, 116(1): 43-49. |
| [16] |
Wang W, Wang HS, Hu HB, et al. Overexpression of afsR and optimization of metal chloride to improve lomofungin production in Streptomyces lomondensis S015[J]. Journal of Microbiology and Biotechnology, 2015, 25(5): 672-680. DOI:10.4014/jmb.1409.09091 |
| [17] |
Sheng CL, Wang W, Hu HB, et al. Function of a methyltransferase gene lomo3 involved in the biosynthesis of lomofungin[J]. Microbiology China, 2016, 43(3): 575-582. (in Chinese) 盛超兰, 王威, 胡洪波, 等. 一个甲基转移酶基因lomo3在洛蒙真菌素生物合成途径中的功能[J]. 微生物学通报, 2016, 43(3): 575-582. |
| [18] |
Li S, Ye QM, Wang W, et al. High-performance liquid chromatography analysis of lomofungin in Streptomyces lomondensis[J]. Journal of Liquid Chromatography & Related Technologies, 2013, 36(15): 2059-2068. |
| [19] |
Xie K, Peng HS, Hu HB, et al. OxyR, an important oxidative stress regulator to phenazines production and hydrogen peroxide resistance in Pseudomonas chlororaphis GP72[J]. Research in Microbiology, 2013, 168(10): 646-653. DOI:10.1016/j.micres.2013.05.001 |
 2019, Vol. 46
2019, Vol. 46