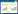扩展功能
文章信息
- 王九儒, 赵飞扬, 李曼莉, 裴广倩, 范航, 张湘莉兰, 米志强, 童贻刚
- WANG Jiu-Ru, ZHAO Fei-Yang, LI Man-Li, PEI Guang-Qian, FAN Hang, ZHANG Xiang-Li-Lan, MI Zhi-Qiang, TONG Yi-Gang
- 一株肺炎克雷伯菌噬菌体的生物学特性及全基因组分析
- Characterization and genome analysis of a novel Klebsiella pneumoniae phage
- 微生物学通报, 2019, 46(12): 3402-3413
- Microbiology China, 2019, 46(12): 3402-3413
- DOI: 10.13344/j.microbiol.china.190032
-
文章历史
- 收稿日期: 2019-01-10
- 接受日期: 2019-04-02
- 网络首发日期: 2019-04-24
2. 军事科学院军事医学研究院微生物流行病研究所 病原微生物生物安全国家重点实验室 北京 100071;
3. 青岛农业大学动物医学院 山东 青岛 266109
2. State Key Laboratory of Pathogen and Biosecurity, Beijing Institute of Microbiology and Epidemiology, Beijing 100071, China;
3. College of Veterinary Medicine, Qingdao Agricultural University, Qingdao, Shandong 266109, China
肺炎克雷伯菌(K. peneumoniae)属于肠杆菌科(Enterobacteriaceae)克雷伯氏菌属(Klebsiella),为革兰氏阴性杆菌。肺炎克雷伯菌对人致病性较强,能导致败血症、肺炎及尿路感染,严重时可能致人死亡,是重要的条件致病菌和医源性病原体之一。随着各类广谱抗生素的广泛应用,肺炎克雷伯菌出现耐药性的现象也越来越普遍[1]。据世界卫生组织全球抗微生物耐药性监测系统(Global antimicrobial resistance surveillance system,GLASS)报道[2],肺炎克雷伯菌已成为七类主要的耐药性致病菌之一,其耐药性问题已经引起了世界范围的关注。在临床环境中过量使用头孢菌素和β-内酰胺酶是导致肺炎克雷伯菌耐药的主要原因。近年来,由于第三代头孢菌素耐药肺炎克雷伯菌的出现,碳青霉烯类抗生素被广泛用于治疗多重耐药性肺炎克雷伯菌引起的严重感染[3],这加速了碳青霉烯类耐药菌株的出现。据CHINET报道,截至2017年,肺炎克雷伯菌对碳青霉烯类的耐药率均大于20%,较2014年上升8倍之多[4-5]。而且相关研究表明[6]致病菌产生耐药性的速度已远大于新抗生素研发速度。
噬菌体作为一类特异性感染细菌的病毒,是一种天然抗菌物。在全球医疗领域面临日益严峻的细菌耐药性威胁时,噬菌体治疗逐渐显现出潜力。陈松建等[7]使用一株肺炎克雷伯菌噬菌体治疗感染了产超广谱β-内酰胺酶(Extended spectrum β-lactamase,ESBLs)耐药菌的小鼠,实验组败血症小鼠存活率为87.5%,对照组败血症小鼠24 h后全部死亡。一些研究[8-9]表明,对于噬菌体的个体化治疗手段,鸡尾酒疗法可能会比使用单一噬菌体治疗有更好的疗效。噬菌体与其宿主菌是共同进化的,有时噬菌体进化速度甚至要大于其宿主产生抗性的速度,这都使得噬菌体治疗重新成为人们关注的热点。
本研究通过对一株新发现的肺炎克雷伯菌噬菌体进行分离鉴定并分析其生物学特性,为耐药性肺炎克雷伯菌的噬菌体治疗提供依据。此外,本研究还通过高通量测序及基因组分析对噬菌体基因功能进行预测,有助于人们更加深刻地认识其生物学行为。
1 材料与方法 1.1 材料 1.1.1 菌种从医院污水中分离得到的一株肺炎克雷伯菌噬菌体,由军事医学科学院微生物流行病研究所分离鉴定并保存;从病人尿液中分离出的一株具有多重耐药性的肺炎克雷伯菌,由军事医学科学院微生物流行病研究所分离鉴定并保存;20株肺炎克雷伯菌,由307医院提供并于军事医学科学院微生物流行病研究所分离鉴定并保存。
1.1.2 培养基肺炎克雷伯菌培养基为LB培养基[10] (固体培养基加1.5%琼脂粉,半固体培养基加0.7%琼脂粉)。
1.1.3 主要试剂和仪器酚:氯仿:异戊醇液(25:24:1,体积比),北京索莱宝科技有限公司;蛋白酶K、RNase A、DNase Ⅰ,Fermentas公司。Amicon Ultra-15离心超滤管(截留分子量100 kD),AMRESCO公司;MiSeq测序仪,Illumina公司。
1.2 方法 1.2.1 宿主菌的准备将分离出的肺炎克雷伯菌接种于普通LB琼脂平板上,37 ℃培养过夜,挑取单菌落接种于5 mL液体LB培养基中,37 ℃、220 r/min振荡培养6 h,OD600约为0.5时用于分离肺炎克雷伯菌噬菌体。
1.2.2 宿主菌的药物敏感性实验通过测定药物的最小抑菌浓度(Minimum inhibitory concentration,MIC)确定宿主菌的药敏性。
1.2.3 噬菌体的分离纯化将医院取回的污水于10 000 r/min离心20 min取上清,经0.2 μm微孔滤器过滤,取滤液100 μL加入5 mL对数期(OD600约为0.5)的肺炎克雷伯菌中,在37 ℃、220 r/min培养过夜,8 000 r/min离心10 min后,经0.2 μm微孔滤器过滤,收集上清即为噬菌体原液。将上述噬菌体原液进行梯度稀释10-1-10-8,分别取100 μL不同浓度的稀释液铺双层板,37 ℃倒置培养直至长出透亮的单个噬菌斑。用枪头挑取单斑加入5 mL LB液体培养基,再加入100 μL菌液后于37 ℃、220 r/min振荡培养5-8 h,8 000 r/min离心取上清,经0.2 μm微孔滤器过滤,取滤液梯度稀释,如此重复3次,以达到纯化的效果。
1.2.4 噬菌体的大量培养及浓缩将细菌接种至500 mL LB液体培养基中,培养至OD600约为0.5,加入5 μL噬菌体原液,37 ℃振荡培养直至液体变澄清。12 000 r/min离心10 min取上清,将该液体经0.2 μm微孔滤器过滤,按照参考文献[10]的方法,使用超滤管进行浓缩。
1.2.5 透射电镜观察噬菌体形态采用磷钨酸负染法观察噬菌体的形态,将镀碳支持膜铜网放在封口膜上,吸取1滴纯化的噬菌体悬液滴在铜网上,放置2-5 min,用带尖的滤纸从铜网边缘吸去多余的噬菌体悬液,干燥1 min。加1滴2%磷钨酸溶液,染色1 min。用滤纸吸去多余的染液,室温放置30 min后置于电镜下观察。
1.2.6 噬菌体最佳感染复数(Multiplicity of infection,MOI)测定将宿主菌培养至对数期,按照噬菌体浓度(PFU/mL)/细菌浓度(CFU/mL)为0.01、0.1、1、10、100的比例混合,37 ℃孵育5 min后,8 000 r/min离心5 min弃去上清,去除未吸附的噬菌体,用5 mL液体LB将沉淀重悬,37 ℃振荡培养2 h,12 000 r/min离心过滤取上清,利用双层平板法测定噬菌体效价并统计结果[9, 11]。
1.2.7 噬菌体裂解谱的测定通过点板试验,将受试菌(共20株肺炎克雷伯菌)培养至OD600约为0.5,取出400 μL铺双层平板,室温静置5 min,将3 μL噬菌体浓缩液滴于双层平板上,室温静置至噬菌体浓缩液被吸收后,倒置于37 ℃温箱中9 h后观察有无噬菌斑产生。
1.2.8 一步生长曲线的测定在最佳MOI条件下,将噬菌体和宿主菌混合,总体积20 mL,37 ℃温孵5 min,12 000 r/min离心30 s,弃去上清;加入适量LB (37 ℃)培养基,12 000 r/min离心1 min后弃去上清,重复3次,以除去未吸附的宿主菌噬菌体;加入20 mL液体LB培养基,37 ℃振荡培养,从0时刻起每隔10 min取样,4 ℃、12 000 r/min离心10 min,收集上清液,测定噬菌体的滴度,试验重复3次。以感染时间(t)为横坐标,噬菌体滴度(PFU/mL)为纵坐标,绘制一步生长曲线,得出噬菌体的潜伏期、裂解期,并计算裂解量。裂解量=裂解末期噬菌体滴度/感染初期宿主菌浓度。
1.2.9 提取噬菌体基因组参考文献[9, 12-13]的蛋白酶K/SDS法提取噬菌体核酸,方法略有改动,具体方法:取600 μL已纯化的噬菌体,加入DNase Ⅰ和RNase A至终浓度1 mg/L,在37 ℃金属浴孵育过夜,80 ℃灭活15 min,使酶失活。然后加入0.5 mol/L EDTA至终浓度0.02 mol/L,20 g/L蛋白酶K至终浓度50 mg/L,10% SDS至终浓度0.5%,56 ℃水浴1 h。加入650 μL平衡酚溶液抽提,振荡混匀1 min,10 000 r/min离心5 min,转移上层水相到新的1.5 mL离心管中。向上述液体中加入620 μL的酚-氯仿-异戊醇(25:24:1,体积比),温和振荡1 min后10 000 r/min离心10 min。将上述离心管中上层水相移到一个新的离心管中,并加入550 μL异戊醇。在-20 ℃放置2 h后于4 ℃、12 000 r/min离心15 min,弃去所有液体。用75%的-20 ℃预冷乙醇洗涤上述沉淀,4 ℃、12 000 r/min离心20 min弃上清,将沉淀置于室温至完全干燥,用无核酸水重悬干燥的DNA沉淀,置于-20 ℃保存。
1.2.10 噬菌体的全基因组测序及生物信息学分析使用NEBNext® Ultra™ Ⅱ DNA Library Prep Kit构建600 bp测序文库。主要流程:基因组DNA超声打断(160 W,工作30 s,停歇90 s,共11 min),末端补平,加上特异接头,扩增纯化筛选DNA即为构建的测序文库。使用Illumina MiSeq测序仪对噬菌体进行全基因组测序。使用软件Newbler V3.0 (Roche diagnostics company,Indianapolis,IN)对噬菌体原始测序数据进行组装和拼接。使用RAST (http://rast.nmpdr.org/)在线注释对全基因组进行快速注释,使用在线BLASTp工具(http://www.ncbi.nlm.nih.gov/BLAST)进一步对注释蛋白进行功能预测[14]。用软件MEGA 6.0 (http://www.megasoftware.net)对噬菌体衣壳蛋白进行进化分析。
2 结果与分析 2.1 宿主菌的药敏性宿主菌药敏性检测结果如表 1所示。
| 抗生素名称 Antimicrobial drugs name |
最小抑菌浓度 MIC (μg/mL) |
药敏性 Sensitivity |
| 丁胺卡那霉素Amikacin | 16 | S |
| 头孢呋辛纳Cefuroxime-Sodium | ≥64 | R |
| 环丙沙星Ciprofloxacim | ≥4 | R |
| 庆大霉素Gentamicin | ≥16 | R |
| 亚胺培南Imipenem | ≤1 | S |
| 美洛培南Meropenem | ≤0.25 | S |
| 哌拉西林Piperacillin | ≥128 | R |
| 妥布霉素Tobramycin | ≥16 | R |
| 复方新诺明Cotrimoxazole | ≥320 | R |
| 左旋氧氟沙星Levofloxacin | ≥8 | R |
| 呋喃妥因Nitrofurantoin | 64 | I |
| 氨苄西林/舒巴坦Ampicillin/Sulbactam | ≥32 | R |
| 氨曲南Aztreonam | ≥64 | R |
| 头孢吡肟Cefepime | 16 | I |
| 头孢替坦Cefotetan | ≤4 | S |
| 头孢他啶Ceftazidime | 16 | R |
| 头孢曲松Ceftriaxone | ≥64 | R |
| 注:S:敏感;I:中介度;R:耐药. Note: S: Sensitive; I: Intermediary; R: Resistant. | ||
从医院污水中分离得到一株肺炎克雷伯菌噬菌体,命名为vB_KpnP_IME279。噬菌体vB_KpnP_IME279的透射电镜照片如图 1所示,衣壳呈正20面体,直径约60 nm,尾鞘长约15 nm,无收缩性,可隐约看到尾丝。依据最新国际病毒分类方法[15],vB_KpnP_IME279属于有尾噬菌体目短尾噬菌体科(Podoviridae)。
|
| 图 1 噬菌体vB_KpnP_IME279的形态 Figure 1 The morphology of phage vB_KpnP_IME279 |
|
|
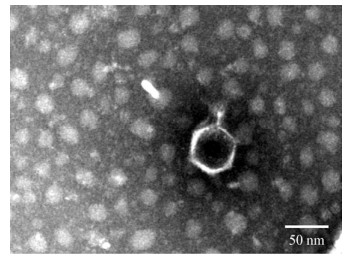
结果如图 2所示,当MOI为0.1时,噬菌体感染宿主后释放的子代噬菌体的数量最多,即噬菌体vB_KpnP_IME279的最佳感染复数为0.1。
|
| 图 2 最佳感染复数的测定结果 Figure 2 Determination of optimal multiplicity of infection (MOI) |
|
|
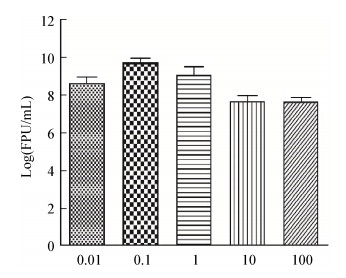
依据实验数据绘制噬菌体的一步生长曲线,如图 3所示。噬菌体vB_KpnP_IME279的潜伏期(Latent period)是20 min,其暴发期持续约50 min,随后进入平稳期,理论上裂解释放应该是瞬时出现的,但由于每个细菌细胞的裂解无法同步,所以暴发期较长。噬菌体的裂解量约为噬菌体滴度/细菌的浓度=(1.4×109)/(1×107)=140。
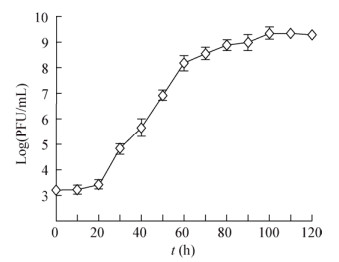
|
| 图 3 噬菌体vB_KpnP_IME279的一步生长曲线 Figure 3 One-step growth curve of phage vB_KpnP_IME279 |
|
|
通过对共计20株肺炎克雷伯菌的双层板点板实验表明,该噬菌体对其中10株有裂解能力,结果如表 2所示。
| 肺炎克雷伯菌编号 Klebsiella pneumoniae No. |
裂解性 Lytic ability |
| 1731 | - |
| 1732 | + |
| 1733 | - |
| 1734 | - |
| 1735 | - |
| 1736 | - |
| 1737 | + |
| 1738 | + |
| 1739 | + |
| 1740 | + |
| 465 | - |
| 474 | + |
| 497 | - |
| 501 | - |
| 510 | + |
| 529 | + |
| 530 | + |
| 551 | + |
| 553 | - |
| 554 | - |
| 注:+:裂解;-:不裂解. Note: +: Indicates cleavage; -: Indicates no cleavage. | |
噬菌体vB_KpnP_IME279全基因组长度为42 518 bp,基因组为线性,其核酸序列NCBI登录号为MF614100。基因组A、C、G和T碱基含量分别为18.7%、29.7%、29.6%和22.0%,(G+C)mol%为59.3%。BLASTn分析结果如表 3所示,共有4株噬菌体与噬菌体vB_KpnP_IME279基因组进行了对比,覆盖率为22%-70%,相似度在69%-80%之间,基因相似性较低。噬菌体vB_KpnP_IME279与其中2株相似度最高的噬菌体的基因组对比分析结果见图 4。RAST基因注释结果显示共有59个开放阅读框(Open reading frame,ORF),其中有2个ORF起始密码为GTG,56个ORF起始密码为ATG,仅有1个ORF起始密码为GCG。CDS核酸序列长度在174-3 144 bp之间,对应的蛋白质序列长度在57-1 047 aa之间。全部的ORF共含39 765 bp的碱基,基因组基因密度高达93.5%。
| Description | Query cover (%) | E value | Identity (%) | Accession No. |
| Salmonella phage Lumpael | 70 | 0 | 71 | MK125141.1 |
| Enterobacteria phage IME_EC2 | 70 | 0 | 80 | KF591601.1 |
| Escherichia phage Skarpretter | 26 | 0 | 72 | MK105855.1 |
| Escherichia phage C130_2 | 22 | 2.00E-153 | 69 | MH363708.1 |
| Citrobacter farmeri strain AUSMDU00008141 | 0 | 2.00E-77 | 80 | CP022695.1 |
| Citrobacter amalonaticus Y19 | 0 | 1.00E-73 | 79 | CP011132.1 |
| Citrobacter amalonaticus strain FDAARGOS_122 | 0 | 4.00E-73 | 79 | CP014015.2 |
| Citrobacter freundii complex sp. CFNIH2 chromosome | 0 | 4.00E-73 | 79 | CP025757.1 |

|
| 图 4 vB_KpnP_IME279与其近源噬菌体同源分析 Figure 4 The homology comparison between vB_KpnP_IME279 and its proximal phages |
|
|
使用BLASTp对蛋白序列进行在线分析,共得到59个开放阅读框(Open reading frame,ORF),已知功能的ORF如表 4所示。其中ORF1为AAA家族三磷酸腺苷酶,该类酶通常与各类细胞活动有着密切关系[16]。ORF16为Dcm甲基化酶,可将CCAGG和CCTGG序列中的胞嘧啶变为5-甲基胞嘧啶,该酶确切功能尚不清楚[17]。与噬菌体结构相关的蛋白为ORF12 (头尾连接处蛋白)、ORF25 (基底板相关蛋白)、ORF28 (尾部纤维蛋白)、ORF37 (噬菌体衣壳蛋白)、ORF38 (支架蛋白)和ORF44 (噬菌体门蛋白)。噬菌体门蛋白可以在衣壳中形成通道并有助于噬菌体基因组注入宿主细胞[18]。
| ORF | Top BLAST hita | Identity (aa)b (%) | E value | Accession No. |
| 1 | AAA family ATPase [Enterobacter bugandensis] | 527/553 (95%) | 0 | WP_047367159.1 |
| 2 | Hypothetical protein DI533_21445 [Rhodobacter sphaeroides] | 149/151 (99%) | 2.00E-101 | PZQ94814.1 |
| 3 | No hits | |||
| 4 | Hypothetical protein DI533_21165 [Rhodobacter sphaeroides] | 147/151 (97%) | 2.00E-105 | PZQ94762.1 |
| 5 | Hypothetical protein [Enterobacter bugandensis] | 75/81 (93%) | 1.00E-44 | WP_047367155.1 |
| 6 | Hypothetical protein DI533_21175 [Rhodobacter sphaeroides] | 114/115 (99%) | 1.00E-79 | PZQ94764.1 |
| 7 | Hypothetical protein DI533_21180 [Rhodobacter sphaeroides] | 72/73 (99%) | 1.00E-43 | PZQ94765.1 |
| 8 | Hypothetical protein DI533_21185 [Rhodobacter sphaeroides] | 91/92 (99%) | 1.00E-60 | PZQ94766.1 |
| 9 | Hypothetical protein [Enterobacter bugandensis] | 112/116 (97%) | 2.00E-79 | WP_082131788.1 |
| 10 | Lysin [Rhodobacter sphaeroides] | 150/154 (97%) | 2.00E-106 | PZQ94768.1 |
| 11 | DUF2514 domain-containing protein [Rhodobacter sphaeroides] | 170/172 (99%) | 4.00E-114 | PZQ94817.1 |
| 12 | Neck whiskers protein [Rhodobacter sphaeroides] | 322/333 (97%) | 0 | PZQ94769.1 |
| 13 | Hypothetical protein DI533_21210 [Rhodobacter sphaeroides] | 129/130 (99%) | 6.00E-89 | PZQ94770.1 |
| 14 | No hits | |||
| 15 | No hits | |||
| 16 | Dcm methylase [Rhodobacter sphaeroides] | 244/245 (99%) | 0 | PZQ94774.1 |
| 17 | Hypothetical protein DI533_21235 [Rhodobacter sphaeroides] | 188/194 (97%) | 5.00E-139 | PZQ94775.1 |
| 18 | Phage N-6-adenine-methyltransferase [Rhodobacter sphaeroides] | 229/233 (98%) | 2.00E-169 | PZQ94776.1 |
| 19 | DNA primase/helicase [Escherichia phage C130_2] | 140/279 (50%) | 3.00E-87 | AXC34353.1 |
| 20 | DNA helicase, phage-associated [Rhodobacter sphaeroides] | 680/684 (99%) | 0 | PZQ94778.1 |
| 21 | Hypothetical protein [Enterobacter bugandensis] | 147/148 (99%) | 2.00E-104 | WP_052954301.1 |
| 22 | Putative exodeoxyribonuclease Ⅷ [Enterobacteria phage IME_EC2] | 293/341 (86%) | 0 | AGZ17810.1 |
| 23 | Recombinase [Escherichia phage C130_2] | 157/238 (66%) | 2.00E-100 | AXC34349.1 |
| 24 | Single-stranded DNA-binding protein [Rhodobacter sphaeroides] | 180/182 (99%) | 8.00E-125 | PZQ94782.1 |
| 25 | Baseplate wedge protein [Enterobacteria phage IME_EC2] | 110/146 (75%) | 3.00E-78 | AGZ17813.1 |
| 26 | No hits | |||
| 27 | No hits | |||
| 28 | Phage tail fibers [Enterobacter bugandensis] | 751/787 (95%) | 0 | WP_052954300.1 |
| 29 | Lytic transglycosylase domain-containing protein [Enterobacter bugandensis] | 889/976 (91%) | 0 | WP_120247335.1 |
| 30 | Prophage DNA injection protein [Enterobacteria phage IME_EC2] | 175/232 (75%) | 1.00E-103 | AGZ17819.1 |
| 31 | DNA transfer protein [Rhodobacter sphaeroides] | 218/220 (99%) | 1.00E-151 | PZQ94785.1 |
| 32 | Hypothetical protein DI533_21305 [Rhodobacter sphaeroides] | 128/131 (98%) | 8.00E-91 | PZQ94786.1 |
| 33 | Hypothetical protein DI533_21310 [Rhodobacter sphaeroides] | 185/192 (96%) | 3.00E-129 | PZQ94787.1 |
| 34 | DNA stabilization protein [Enterobacteria phage IME_EC2] | 328/481 (68%) | 0 | AGZ17823.1 |
| 35 | DNA stabilization protein [Enterobacteria phage IME_EC2] | 91/149 (61%) | 3.00E-61 | AGZ17824.1 |
| 36 | Hypothetical protein [Enterobacter bugandensis] | 157/181 (87%) | 1.00E-104 | WP_047367130.1 |
| 37 | Phage capsid protein [Rhodobacter sphaeroides] | 418/420 (99%) | 0 | PZQ94791.1 |
| 38 | Scaffold protein [Enterobacteria phage IME_EC2] | 163/339 (48%) | 8.00E-83 | AGZ17827.1 |
| 39 | Hypothetical protein DI533_21345 [Rhodobacter sphaeroides] | 148/149 (99%) | 6.00E-103 | PZQ94794.1 |
| 40 | Hypothetical protein DI533_21350 [Rhodobacter sphaeroides] | 131/138 (95%) | 2.00E-82 | PZQ94795.1 |
| 41 | No hits | |||
| 42 | Hypothetical protein DI533_21355 [Rhodobacter sphaeroides] | 141/145 (97%) | 5.00E-99 | PZQ94796.1 |
| 43 | Phage portal protein [Rhodobacter sphaeroides] | 743/744 (99%) | 0 | PZQ94797.1 |
| 44 | Phage terminase, large subunit [Enterobacter bugandensis] | 523/540 (97%) | 0 | KLQ32567.1 |
| 45 | Hypothetical protein ABR33_06125 [Enterobacter bugandensis] | 140/146 (96%) | 2.00E-101 | KLQ32566.1 |
| 46 | Hypothetical protein [Enterobacter bugandensis] | 163/173 (94%) | 3.00E-114 | WP_047367174.1 |
| 47 | Hypothetical protein DI533_21380 [Rhodobacter sphaeroides] | 96/97 (99%) | 1.00E-66 | PZQ94801.1 |
| 48 | Hypothetical protein DI533_21385 [Rhodobacter sphaeroides] | 95/138 (69%) | 1.00E-33 | PZQ94802.1 |
| 49 | No hits | |||
| 50 | Hypothetical protein DI533_21395 [Rhodobacter sphaeroides] | 296/312 (95%) | 0 | PZQ94804.1 |
| 51 | No hits | |||
| 52 | Hypothetical protein [Enterobacter bugandensis] | 83/91 (91%) | 1.00E-52 | WP_047367168.1 |
| 53 | No hits | |||
| 54 | Hypothetical protein DI533_21415 [Rhodobacter sphaeroides] | 121/126 (96%) | 2.00E-83 | PZQ94808.1 |
| 55 | Hypothetical protein DI533_21420 [Rhodobacter sphaeroides] | 95/104 (91%) | 2.00E-64 | PZQ94809.1 |
| 56 | Hypothetical protein DI533_21425 [Rhodobacter sphaeroides] | 80/85 (94%) | 2.00E-51 | PZQ94810.1 |
| 57 | No hits | |||
| 58 | Hypothetical protein DI533_21430 [Rhodobacter sphaeroides] | 72/78 (92%) | 5.00E-33 | PZQ94811.1 |
| 59 | No hits | |||
| 注:a:最相近基因组微生物物种的基因和名称;No hits:没有比对到相近的氨基酸序列;b:BLASTp搜索到比对百分比. Note: a: The most closely related genes and names of the organisms; No hits: Indicates no significant homology detected for a particularamino acid sequence; b: Percent identity for top hits in BLASTp searches. | ||||
ORF34和ORF35是DNA稳定蛋白,属于噬菌体稳定超家族(Phage_stabilize superfamily),研究表明该蛋白可能参与了噬菌体包装过程中的DNA凝缩过程[19]。
在噬菌体DNA复制及调控相关基因中包括:ORF19 (引物酶/解旋酶)、ORF20 (解旋酶)、ORF23 (重组酶)、ORF24 (DNA单链结合蛋白)。由于DNA复制的半不连续性,导致后随链的复制过程需要引物酶的参与[20]。此外,引物酶可以与解旋酶的N-末端结合形成复制子,其在DNA复制、修复和转录中具有广泛作用。
ORF10 (溶菌酶)能诱导宿主菌的裂解,从而释放出子代噬菌体。ORF44(末端酶大亚基)基因是终止酶全酶的重要组成部分,据相关研究[21]表明,末端酶复合物可以与门蛋白组装到一起,形成一个基因组包装马达,在噬菌体的包装过程中发挥重要的作用。ORF29 (糖基转移酶)可以催化糖基与核酸或者蛋白质等物质之间形成糖苷键,进而形成具有独特生物学活性的糖基化合物[22]。
使用Inkscape软件制作噬菌体全基因组模式图,如图 5所示,共4圈。

|
| 图 5 IME279全基因组图谱 Figure 5 Genome map of IME279 注:从外到里分别是:最外侧代表基因组正向阅读框,其次内侧代表负向阅读框;黑色代表(G+C)mol%,向外表示含量大于全基因组平均(G+C)mol%,向内表示含量低于平均(G+C)mol%;最里面圈代表G+C偏嗜性G-C/G+C,向外代表大于0,向内代表小于0. Note: Patterns are divided into four circles. From the outside to the inside: The outermost represents the forward reading frame, followed by the outermost circle represents the reverse reading frame; Black represents the (G+C)mol% (outward is more than the whole genome average (G+C)mol% and inwardly is the opposite); The innermost green and purple circles represent G+C skew of G-C/G+C (outward represents greater than 0, inward represents less than 0). |
|
|
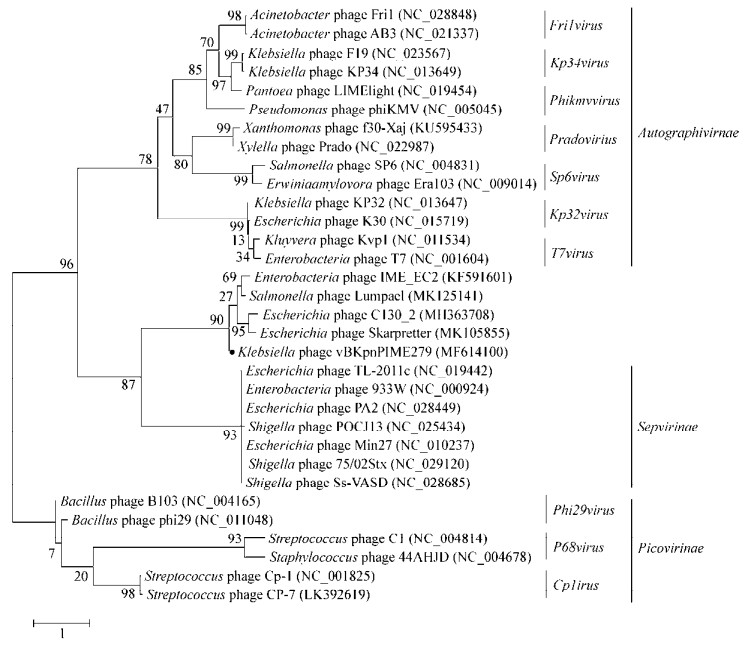
为了更好地分析噬菌体IME279与其它噬菌体之间的关系。选取具有进化意义且保守的噬菌体主要衣壳蛋白(ORF39)和其他短尾科噬菌体的主要衣壳蛋白氨基酸序列构建系统进化树[23],如图 6所示。进化树结果显示噬菌体IME279与IME EC2、C130-2、Lumpael和Skarpretter位于Sepvirinae亚科中一个新的分支,而且不属于已知属,我们建议组成一个新的属。
|
| 图 6 使用噬菌体主要衣壳蛋白制作的进化树 Figure 6 Phylogenetic tree was formed by major capsid protein of phage 注:分支点上的数字代表可信度,数值越接近100,可信度越强;标尺:代表遗传距离,标尺越短代表亲缘关系越近;黑色圆点:vB_KpnP_IME279;括号内:GenBank序列号. Note: The number at the branch point: The confidence level; Scale: The genetic distance; Black ball: vB_KpnP_IME279; In brackets: GenBank accession No.. |
|
|
在抗生素广泛应用的今天,致病菌产生耐药性的问题越发严重,世界卫生组织更是认为细菌耐药性威胁是21世纪的最大威胁之一。在1921年,Bruynoghe等[24]首次运用噬菌体治疗细菌感染之后,噬菌体治疗曾经受到了一定程度的关注,但是由于当时技术所限,噬菌体的培养和提取受到了很大程度的限制。在抗生素诞生后,其高效便捷的特性更使得噬菌体治疗被人们所遗忘。但是由于当前全球面临的细菌耐药性提升的严峻形势,而抗生素的开发速度远远低于抗药性的产生速度,从而使噬菌体治疗手段再次受到了全世界范围的关注。目前噬菌体治疗已经取得了很多成果[25]。2009年,Wright等[26]首次用噬菌体治疗多重耐药铜绿假单胞菌感染导致的慢性耳炎患者,一段时间后患者症状均显著改善,且未发现任何毒副作用。
本实验利用从医院污水中分离得到的肺炎克雷伯菌噬菌体进行研究,命名为vB_KpnP_IME279。通过透射电镜观察,按照噬菌体分类学中的分类标准,判断该噬菌体属于短尾噬菌体科(Podoviridae),进一步进行生物学特性研究发现该噬菌体最佳感染复数为0.1,一步生长曲线显示IME279的潜伏期20 min,暴发量为140 PFU/cell。此外,本研究利用Illumina MiSeq高通量测序仪进行全基因组测序,利用BLASTn进行在线分析,发现IME279与目前已知的噬菌体一致性均较低。通过BLASTp预测了ORF功能,为后续对噬菌体进行更深入的基因研究奠定了基础。在ORF分析中,并未找到穿孔蛋白(Holin)与DNA聚合酶(DNA polymerase)的CDS序列,通过与噬菌体IME EC2和Lumpael基因组同源分析得知,ORF11可能是穿孔蛋白,利用TMHMM server (http://www.cbs.dtu.dk/services/ TMHMM-2.0/)在线分析得知该ORF有一段跨膜区,可能为Ⅲ型穿孔蛋白,其结果还需要进行进一步实验验证。在IME 279与另外两株参与对比的噬菌体基因组中均未找到末端酶小亚基的CDS序列。ORF16 (Dcm甲基化酶)与ORF18 (N-6-腺嘌呤甲基转移酶)可能参与了宿主基因组的修饰,ORF44 (重组酶)可以催化链的断裂与连接。
尽管噬菌体治疗被给予了很高的期望来抗争所谓“超级耐药菌”,但是仍然有很多亟待解决的问题,尤其是噬菌体制剂的质量评价标准难以确定。为了系统评价噬菌体治疗的安全性,欧盟曾在2015年启动了Phagoburn计划[27]。我们相信,随着科学家对噬菌体研究的不断深入,噬菌体制剂标准化问题会得到解决,噬菌体治疗会更好地应用于临床治疗,以解决当前的耐药性问题。
| [1] |
Shi ZY, Liu XH, Li G, et al. Drug resistance changes of clinical isolation of Escherichia coli and Klebsiella pneumoniae[J]. Laboratory Medicine and Clinic, 2018, 15(22): 3349-3352. (in Chinese) 师志云, 刘香花, 李刚, 等. 临床分离大肠埃希菌和肺炎克雷伯菌耐药性变迁[J]. 检验医学与临床, 2018, 15(22): 3349-3352. DOI:10.3969/j.issn.1672-9455.2018.22.008 |
| [2] |
Organization WH. Antimicrobial resistance global report on surveillance: 2014 summary[C]. World Health Organization, 2014
|
| [3] |
Cheng GY, Li J, Gu YF, et al. Antimicrobial resistance surveillance systems of WHO, EU and China[J]. Chinese Journal of Antibiotics, 2018, 43(6): 665-674. (in Chinese) 程古月, 李俊, 谷宇锋, 等. 世界卫生组织、欧盟和中国抗生素耐药性监测现状[J]. 中国抗生素杂志, 2018, 43(6): 665-674. DOI:10.3969/j.issn.1001-8689.2018.06.004 |
| [4] |
Hu FP, Zhu DM, Wang F, et al. CHINET 2014 surveillance of bacterial resistance in China[J]. Chinese Journal of Infection and Chemotherapy, 2015, 15(5): 401-410. (in Chinese) 胡付品, 朱德妹, 汪复, 等. 2014年CHINET中国细菌耐药性监测[J]. 中国感染与化疗杂志, 2015, 15(5): 401-410. DOI:10.3969/j.issn.1009-7708.2015.05.001 |
| [5] |
Hu FP, Guo Y, Zhu DM, et al. Antimicrobial resistance profile of clinical isolates in hospitals across China:report from the CHINET Surveillance Program, 2017[J]. Chinese Journal of Infection and Chemotherapy, 2018, 18(3): 241-251. (in Chinese) 胡付品, 郭燕, 朱德妹, 等. 2017年CHINET中国细菌耐药性监测[J]. 中国感染与化疗杂志, 2018, 18(3): 241-251. |
| [6] |
Wang S, Tong YG. Recent advance in bacteriophage therapy[J]. Microbiology China, 2009, 36(7): 1019-1024. (in Chinese) 王盛, 童贻刚. 噬菌体治疗研究进展[J]. 微生物学通报, 2009, 36(7): 1019-1024. |
| [7] |
Chen SJ, Wang SW, Zhang G, et al. Characterization and application of a novel phage F20 in the rescue septicemia mice caused by extend[J]. Microbiology China, 2019, 46(6): 1502-1509. (in Chinese) 陈松建, 王书伟, 张改, 等. 超广谱β-内酰胺酶肺炎克雷伯菌噬菌体F20的分离鉴定及其对小鼠败血症治疗效果[J]. 微生物学通报, 2019, 46(6): 1502-1509. |
| [8] |
Oliveira A, Sereno R, Azeredo J. In vivo efficiency evaluation of a phage cocktail in controlling severe colibacillosis in confined conditions and experimental poultry houses[J]. Veterinary Microbiology, 2010, 146(3/4): 303-308. |
| [9] |
Xing SZ, Zhang XLL, Shu P, et al. Characterization and complete genome sequence analysis of Enterococcus faecalis bacteriophage vB_E. faecalis_IME196[J]. Microbiology China, 2016, 43(9): 2040-2048. (in Chinese) 邢少贞, 张湘莉兰, 舒鹏, 等. 粪肠球菌噬菌体vB_E. faecalis_IME196的生物学特性及其全基因组分析[J]. 微生物学通报, 2016, 43(9): 2040-2048. |
| [10] |
Han CY, Zhang FX, Tong YG. Comparison of different bacteriophage concentration methods[J]. Letters in Biotechnology, 2013, 24(5): 695-697, 721. (in Chinese) 韩传银, 张飞雄, 童贻刚. 噬菌体浓缩方法的比较[J]. 生物技术通讯, 2013, 24(5): 695-697, 721. DOI:10.3969/j.issn.1009-0002.2013.05.023 |
| [11] |
Zhu JM, Rao XC, Tan YL, et al. Identification of lytic bacteriophage MmP1, assigned to a new member of T7-like phages infecting Morganella morganii[J]. Genomics, 2010, 96(3): 167-172. DOI:10.1016/j.ygeno.2010.06.001 |
| [12] |
Zhang WH, An XP, Fan H, et al. Isolation and characterization of an Enterococcus faecalis phage[J]. Letters in Biotechnology, 2013, 24(4): 484-487. (in Chinese) 张文惠, 安小平, 范航, 等. 一株粪肠球菌噬菌体的分离及其生物学特性研究[J]. 生物技术通讯, 2013, 24(4): 484-487. DOI:10.3969/j.issn.1009-0002.2013.04.009 |
| [13] |
Lu SG, Le S, Tan YL, et al. Genomic and proteomic analyses of the terminally redundant genome of the Pseudomonas aeruginosa phage PaP1: establishment of genus PaP1-like phages[J]. PLoS One, 2013, 8(5): e62933. DOI:10.1371/journal.pone.0062933 |
| [14] |
Altschul SF, Madden TL, Schäffer AA, et al. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs[J]. Nucleic Acids Research, 1997, 25(17): 3389-3402. DOI:10.1093/nar/25.17.3389 |
| [15] |
Lefkowitz EJ, Dempsey DM, Hendrickson RC, et al. Virus taxonomy: the database of the International Committee on Taxonomy of Viruses (ICTV)[J]. Nucleic Acids Research, 2018, 46(D1): D708-D717. DOI:10.1093/nar/gkx932 |
| [16] |
Wang H, Jin S, Chen X, et al. Target deletion of the AAA ATPase PpCDC48Ⅱ in Physcomitrella patens results in freezing sensitivity after cold acclimation[J]. Science China Life Sciences, 2012, 55(2): 156-157. |
| [17] |
Gao J, Han TS, Feng J, et al. Pleiotropic effects on Streptomyces lividans growth by Dcm expression[J]. Microbiology China, 2014, 41(9): 1925-1931. (in Chinese) 高婕, 韩铁生, 丰俊, 等. 大肠杆菌甲基转移酶dcm基因的表达对变铅青链霉菌的多效性影响[J]. 微生物学通报, 2014, 41(9): 1925-1931. |
| [18] |
Prevelige PE, Cortines JR. Phage assembly and the special role of the portal protein[J]. Current Opinion in Virology, 2018, 31: 66-73. |
| [19] |
Eppler K, Wyckoff E, Goates J, et al. Nucleotide sequence of the bacteriophage P22 genes required for DNA packaging[J]. Virology, 1991, 183(2): 519-538. DOI:10.1016/0042-6822(91)90981-G |
| [20] |
Guilliam TA, Keen BA, Brissett NC, et al. Primase-polymerases are a functionally diverse superfamily of replication and repair enzymes[J]. Nucleic Acids Research, 2015, 43(14): 6651-6664. DOI:10.1093/nar/gkv625 |
| [21] |
Sun SY, Rao VB, Rossmann MG. Genome packaging in viruses[J]. Current Opinion in Structural Biology, 2010, 20(1): 114-120. DOI:10.1016/j.sbi.2009.12.006 |
| [22] |
Gloster TM. Advances in understanding glycosyltransferases from a structural perspective[J]. Current Opinion in Structural Biology, 2014, 28: 131-141. DOI:10.1016/j.sbi.2014.08.012 |
| [23] |
Benson SD, Bamford JKH, Bamford DH, et al. Viral evolution revealed by bacteriophage PRD1 and human adenovirus coat protein structures[J]. Cell, 1999, 98(6): 825-833. DOI:10.1016/S0092-8674(00)81516-0 |
| [24] |
Bruynoghe R, Maisin J. Essais de the rapeutique au moyen du bacteriophage[J]. Comptes Rendus des Séances de la Société de Biologie et de Ses Filiales, 1921, 85: 1120-1121. |
| [25] |
Fu LN, Wang LL, Li XY, et al. Advances in phage control of three drug-resistant strains[J]. Chinese Journal of Antibiotics, 2017, 42(10): 836-841. (in Chinese) 付丽娜, 王丽丽, 李晓宇, 等. 噬菌体对三大耐药菌的防控作用研究进展[J]. 中国抗生素杂志, 2017, 42(10): 836-841. DOI:10.3969/j.issn.1001-8689.2017.10.005 |
| [26] |
Wright A, Hawkins CH, Änggård EE, et al. A controlled clinical trial of a therapeutic bacteriophage preparation in chronic otitis due to antibiotic-resistant Pseudomonas aeruginosa; a preliminary report of efficacy[J]. Clinical Otolaryngology, 2009, 34(4): 349-357. DOI:10.1111/j.1749-4486.2009.01973.x |
| [27] |
Yang YH, Le S. Progress on Pseudomonas aeruginosa bacteriophage therapy[J]. Chinese Journal of Antibiotics, 2017, 42(10): 814-820. (in Chinese) 杨雨卉, 乐率. 铜绿假单胞菌噬菌体治疗的研究进展[J]. 中国抗生素杂志, 2017, 42(10): 814-820. DOI:10.3969/j.issn.1001-8689.2017.10.002 |
 2019, Vol. 46
2019, Vol. 46