扩展功能
文章信息
- 赵飞燕, 刘亚华, 李枝, 李伟程, 徐海燕, 孙志宏, 孟和毕力格, 张和平
- ZHAO Fei-Yan, LIU Ya-Hua, LI Zhi, LI Wei-Cheng, XU Hai-Yan, SUN Zhi-Hong, Menghe Bilige, ZHANG He-Ping
- 锡林郭勒地区鲜马奶中细菌多样性分析
- Bacterial diversity of fresh mare's milk in Xilingol
- 微生物学通报, 2019, 46(12): 3295-3304
- Microbiology China, 2019, 46(12): 3295-3304
- DOI: 10.13344/j.microbiol.china.190096
-
文章历史
- 收稿日期: 2019-02-02
- 接受日期: 2019-03-27
- 网络首发日期: 2019-04-23
马奶是一种具有鲜明地域特色和深厚文化底蕴的草原资源,其营养物质丰富且易于被人体消化吸收,其中乳清蛋白与酪蛋白的比例接近1:1,饱和脂肪酸、单不饱和脂肪酸和多不饱和脂肪酸的比例接近联合国粮农组织和世界卫生组织(FAO/WHO)推荐比例(1:1:1)。此外,马奶还具有促进身体解毒,缓解细胞老化,及杀菌、抗病毒和抗炎的作用[1-4]。长期以来,草原牧民就有饮用鲜马奶及利用马奶制作酸马奶、马奶酒的传统[5]。
作为一种传统的自然发酵乳制品,酸马奶中蕴含着丰富的微生物资源。本团队早期利用纯培养技术对内蒙古酸马奶中乳酸菌进行分离鉴定,发现干酪乳杆菌(Lactobacillus casei)和乳酸乳球菌(Lactococcus lactis)是该地区酸马奶中优势菌[6-9]。纯培养技术是获得微生物菌种的有效途径,但可培养的微生物数量十分有限,且不能很好地显示酸马奶中微生物多样性。随着宏基因组学理论的提出,使得从非培养角度全面解析酸马奶中微生物多样性成为可能。Li等[10]利用PacBio SMRT测序技术发现酸马奶中乳酸菌的多样性和丰度很高,其中瑞士乳杆菌(Lactobacillus helveticus)为优势菌种,但也发现酸马奶中含有克雷伯氏菌(Klebsiella)、副乳房链球菌(Streptococcus parauberis)等条件致病菌。基于单细胞扩增结合高通量测序技术,姚国强[11]发现了酸马奶中一些低丰度的乳酸菌,如柠檬明串珠菌(Leuconostoc citreum)、类布氏乳杆菌(Lactobacillus parabuchneri)、Lactobacillus otakiensis等。基于以上研究发现,酸马奶中蕴含着丰富的乳酸菌资源,但鲜马奶中微生物组成是怎样的,以及在马奶发酵过程中是如何变化的,仍有待研究。
传统的酸马奶是将未杀菌过滤的鲜马奶倒入木头或动物皮革制成的容器内,同时加入酸马奶“引子”(发酵好的酸马奶)发酵1−3 d而成[11],研究者认为引子对酸马奶的菌群有很大的影响[12],但鲜马奶中的微生物也是发酵过程中的关键因素之一,并且对酸马奶的品质有影响。然而,目前对于鲜马奶中微生物组成的深入研究较少。An等[13]利用纯培养技术分析了14份采自内蒙古鲜马奶中的细菌多样性,发现鲜马奶中的微生物主要由球菌组成,包括肠膜明串珠菌(Leuconostoc mesenteroides)、假肠膜明串珠菌(Leuconostoc pseudomesenteroides)、格氏乳球菌(Lactococcus garvieae)、乳酸乳球菌和屎肠球菌(Enterococcus faecium)等。但基于纯培养的方法无法完整地揭示鲜马奶的菌群组成。
PacBio SMRT (Single molecule real-time)测序技术采用宏基因组学的研究策略,通过将环境样品中微生物的基因组混合作为一个整体进行研究,打破了传统微生物学基于纯培养研究的局限性。而且相比于Illumina、454焦磷酸等高通量测序技术,PacBio SMRT技术在测序过程中无需进行PCR扩增,可以直接读取目标序列,同时测序读长较长,可在细菌种的水平上揭示样品的微生物菌群结构及丰度信息[14-16]。
本研究以6份新鲜马奶样品为研究对象,采用PacBio SMRT测序技术,在种水平上全面地揭示鲜马奶的菌群结构。同时,与之前报道的锡林郭勒地区酸马奶的菌群结构[17]作比较,探究鲜马奶与酸马奶菌群之间的关联。
1 材料与方法 1.1 材料 1.1.1 样品采集2017年6月至8月于锡林郭勒盟白音锡勒牧场一连的6户牧民家共采集了6份新鲜马奶样品(Z1−Z6),其中6户牧民家距离很近,并且所有马匹放牧于同一草场。每份样品采集30 mL,具体采样信息见表 1。样品采集后首先进行pH值的测定,然后加入10 mL DNA保护液(Sample protector for RNA/DNA)。将样品混匀后置于液氮中冷冻3 min,并尽快以干冰冷藏寄回实验室,于−80 ℃冰箱冷冻储存备用。
| 样品编号 Sample number |
采样时间 Time |
采样地 Site |
| Z1 | 2017.06 | 锡林郭勒盟白音锡勒牧场一连 Baiyin Xile ranch in Xilingol |
| Z2 | 2017.06 | 锡林郭勒盟白音锡勒牧场一连 Baiyin Xile ranch in Xilingol |
| Z3 | 2017.06 | 锡林郭勒盟白音锡勒牧场一连 Baiyin Xile ranch in Xilingol |
| Z4 | 2017.08 | 锡林郭勒盟白音锡勒牧场一连 Baiyin Xile ranch in Xilingol |
| Z5 | 2017.08 | 锡林郭勒盟白音锡勒牧场一连 Baiyin Xile ranch in Xilingol |
| Z6 | 2017.08 | 锡林郭勒盟白音锡勒牧场一连 Baiyin Xile ranch in Xilingol |
Sample Protector for RNA/DNA,大连宝生物工程有限公司;OMEGA DNA Isolation Kit,OMEGA公司;KAPA HiFi HotStart ReadyMix PCR Kit,KAPA公司;5×TBE电泳缓冲液(Tris碱54 g,Na2EDTA·2H2O 3.72 g,硼酸27.5 g,定容至1 L,pH 8.0),1.0%的琼脂糖胶(1.0 g琼脂糖溶于100 mL 0.5×TBE缓冲液),天津基准化学试剂公司。PacBio SMRTbellTM Template Prep Kit 1.0、DNA/Polymerase Binding Kit P6 V2、DNA Sequencing Kit 4.0 V2,Life Technologies公司。
台式高速冷冻离心机,Eppendorf公司;UPV凝胶成像分析系统,北京赛智创业科技有限公司;电泳仪,北京六一生物科技有限公司;微量紫外分光光度计,NanoDrop公司;PCR仪、PacBio SMRT RS II测序平台,Life Technologies公司。
1.2 方法 1.2.1 pH值测定在采样地点现场,用便携式pH计测定6份鲜马奶的pH值。
1.2.2 鲜马奶中微生物DNA的提取取2 mL鲜马奶样品,采用OMEGA DNA Isolation Kit,按照说明书的要求提取鲜马奶宏基因组DNA,利用1.0%琼脂糖凝胶电泳和微量紫外分光光度计检验DNA的纯度和浓度。将DNA纯度符合OD260/OD280为1.8−2.0,浓度大于20 ng/μL,且片段化程度小的DNA置于−20℃冰箱备用。
1.2.3 细菌16S rRNA基因序列全长扩增使用KAPA HiFi HotStart ReadyMix PCR Kit将满足要求的DNA作为模板进行PCR扩增,16S rRNA基因序列引物为27F (5′-AGAGTTTGATCM TGGCTCAG-3′)和1492R (5′-ACCTTGTTACGACT T-3′)。在引物中加入16个核苷酸标签(Barcode)以区分各样品。PCR反应体系(50 μL):正、反向引物(10 μmol/L)各1.5 μL,模板DNA (< 100 ng) 1.5 μL,KAPA Mix 25.0 μL,ddH2O补足至50 μL。PCR反应条件:95℃ 3 min;98 ℃ 20 s,60 ℃ 15 s,72 ℃ 30 s,30个循环;72 ℃ 2 min。
1.2.4 PacBio SMRT三代测序PCR产物经纯化后通过Pacific Biosciences SMRTbellTM Template Prep Kit 1.0试剂盒构建文库,使用DNA/Polymerase Binding Kit P6 V2,DNA Sequencing Kit 4.0 V2等试剂上机测序,具体操作按照厂家提供的说明书进行。
1.2.5 高质量序列的提取使用RS_ReadsOfinsert.1对测序得到的原始序列进行质量控制,具体质控条件为:(1)最小循环测序次数为5;(2)最小预测精确度为90;(3)最小插入序列长度为1 400 bp;(4)最大序列长度为1 800 bp。
1.2.6 生物信息学分析应用QIIME (V1.7.0)平台,对高质量序列进行生物信息学分析。首先采用PyNAST[18]将序列校准排齐后,在97%的相似性下进行UCLUST[19]归并划分分类操作单元(Operational taxonomic units,OTU);从各OTU中选取1条代表性序列,使用RDP (Ribosomal database project)、Greengenes (V13.8)和Silva V128数据库对其进行序列同源性比对,整合3个数据的比对结果确定各OTU的分类学地位;使用FastTree[20]构建基于OTU代表性序列的系统发育进化树,在此基础上计算各样品的超1指数(Chao1 index)、发现物种数(Observed species index)、香农指数(Shannon index)和辛普森指数(Simpson index)用于评估各样品的细菌丰度和多样性。
为了对比分析同一地区酸马奶和鲜马奶菌群之间的关联,文中引用之前本团队利用PacBio SMRT技术对锡林郭勒地区的酸马奶中菌群结构的测序结果[17] (A5−E5)。
1.2.7 数据处理利用Spearman分析检验鲜马奶细菌之间的相关性。采用R软件(V3.3.2)和Origin 8.6软件对QIIME产生的结果进行分析和可视化处理。本研究中涉及的序列数据已提交至NCBI数据库,序列号为SRP142492。
2 结果与分析 2.1 鲜马奶pH值测定6份鲜马奶的pH值分别为6.82、6.80、6.74、7.07、6.79、6.76 (平均值±标准差:6.83±0.12)。酸马奶发酵后pH约为3.8[21],因此本试验中所有鲜马奶样品均处于未发酵状态。
2.2 鲜马奶中细菌多样性分析 2.2.1 测序结果评估6份鲜马奶样品共产生38 656条高质量的16S rRNA基因序列,平均每个样品产生6 442条(表 2)。根据序列相似度划分OTU后,共得到3 789个OTU,样品具体OTU数见表 2。
| Sample | Number of reads | Number of OTUs | Observed species index | Chao1 index | Shannon index | Simpson index |
| Z1 | 4 963 | 585 | 481.29 | 1 493.740 | 6.02 | 0.94 |
| Z2 | 5 939 | 551 | 420.05 | 935.618 | 5.91 | 0.96 |
| Z3 | 7 024 | 554 | 373.24 | 973.424 | 4.95 | 0.85 |
| Z4 | 8 166 | 717 | 452.84 | 1 050.090 | 6.14 | 0.96 |
| Z5 | 7 622 | 673 | 435.38 | 1 098.160 | 6.22 | 0.97 |
| Z6 | 5 351 | 622 | 488.20 | 1 462.770 | 6.00 | 0.95 |
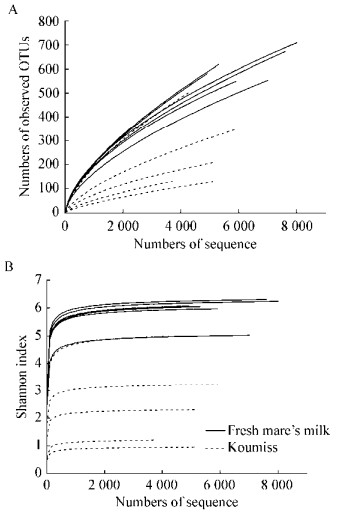
采用稀疏曲线和香农曲线评估各样品测序量是否足够,结果如图 1所示。由图 1A可知,各样品的稀疏曲线均未进入平台期,因此随着测序量的增加有可能发现新的细菌种系型,但图 1B中则显示随着测序深度的增加香农曲线已进入平台期,说明在该测序深度下,样品中的细菌的多样性可以被充分展示。因此本研究中细菌测序量满足后续生物信息学分析的要求。利用Mann-Whitney U检验对比两类样品中微生物多样性和丰度的差异,发现鲜马奶微生物的辛普森指数(P=0.014)和香农指数(P=0.014)显著高于酸马奶,说明鲜马奶中微生物多样性显著高于酸马奶。各样品的具体测序序列信息和α多样性指数如表 2所示。
|
| 图 1 稀疏曲线图(A)和香农多样性图(B) Figure 1 Rarefaction curves (A) and Shannon diversity index curves (B) |
|
|
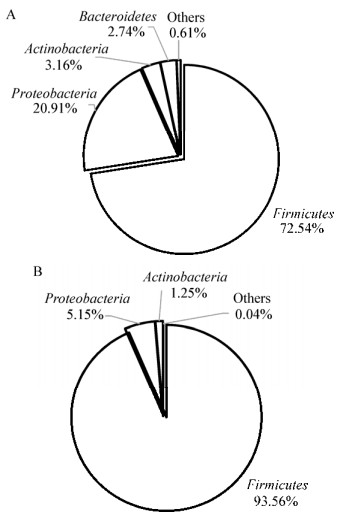
鲜马奶中的细菌主要隶属于硬壁菌门(Firmicutes)和变形菌门(Proteobacteria),其平均相对含量分别为72.42%和21.06%。此外,相对含量大于1%的细菌门还包括放线菌门(Actinobacteria,3.14%)和拟杆菌门(Bacteroidetes,2.73%)。本团队之前对酸马奶菌群组成的研究结果显示硬壁菌门占酸马奶菌群中的93.56%,此外酸马奶中还检测到变形菌门和放线菌门,平均相对含量分别为5.15%和1.25%。在两类样品中,未鉴定到门水平的菌群相对含量均不超过0.1%。两类样品中细菌在门水平的组成如图 2所示。
|
| 图 2 鲜马奶(A)和酸马奶(B)在门水平主要细菌相对含量 Figure 2 The relative abundance of major bacteria in fresh mare's milk (A) and koumiss (B) at phylum level |
|
|
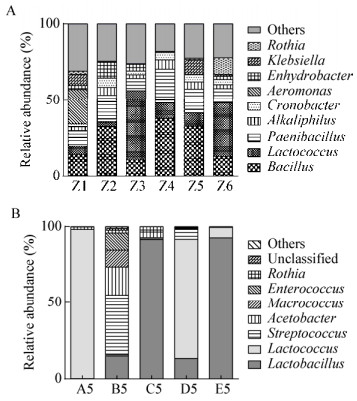
在细菌属水平,6份鲜马奶中共检测到152个细菌属,平均相对含量大于1%的菌属包括芽孢杆菌属(Bacillus,23.44%)、乳球菌属(Lactococcus,18.18%)、类芽孢杆菌属(Paenibacillus,13.80%)、嗜碱菌属(Alkaliphilus,3.96%)、气单胞菌属(Aeromonas,3.88%)、克罗诺菌属(Cronobacter,3.88%)、水栖菌属(Enhydrobacter,3.28%)、克雷伯氏菌属(3.11%)、罗氏菌属(Rothia,2.41%)、肠球菌属(Enterococcus,2.40%)、乳杆菌属(Lactobacillus,2.06%)、明串珠菌属(Leuconostoc,1.98%)、微杆菌属(Exiguobacterium,1.93%)、金黄杆菌属(Chryseobacterium,1.71%)、巨型球菌属(Macrococcus,1.64%)、假单胞菌属(Pseudomonas,1.19%)和链球菌属(Streptococcus,1.07%)。酸马奶中共检出25个细菌属,其中乳杆菌的平均相对含量高达42.48%,其次为乳球菌属(36.86%)、链球菌属(9.36%)、醋酸杆菌属(Acetobacter,4.9%)、肠球菌属(2.24%)和罗氏菌属(1.13%)的平均相对含量大于1%。各样品在种水平的相对含量如图 3所示。
|
| 图 3 鲜马奶(A)和酸马奶(B)在属水平主要细菌相对含量 Figure 3 The relative abundance of major bacteria in fresh mare's milk (A) and koumiss (B) at genus level |
|
|
6份鲜马奶样品中细菌种水平平均含量大于1%的菌种有20个,具体包括蜡样芽孢杆菌(B. cereus,15.52%)、乳酸乳球菌(12.43%)、环状芽孢杆菌(B. circulans,5.46%)、A. oremlandii (3.95%)、阪崎肠杆菌(C. sakazakii,3.86%)、嗜水气单胞菌(A. hydrophila,3.64%)、格氏乳球菌(3.53%)、E. aerosaccus (3.28%)、R. endophytica (2.40%)、P. barcinonensis (1.92%)、乙酰微小杆菌(E. acetylicum,1.85%)、肠膜明串珠菌(1.77%)、M. caseolyticus (1.60%)、产酸克雷伯氏菌(K. pneumoniae,1.60%)、鱼乳球菌(L. piscium,1.56%)、瑞士乳杆菌(1.51%)、解淀粉类芽孢杆菌(P. amylolyticus,1.51%)、K. oxytoca (1.49%)、P. odorifer (1.36%)和C. bovis (1.16%)。而酸马奶中仅有6个菌种的平均相对含量大于1%,包括瑞士乳杆菌(41.20%)、乳酸乳球菌(31.2%)、副乳房链球菌(9.02%)、M. caseolyticus (2.28%)和R. endophytica (1.07%)。两类样品在种水平的相对含量如图 4所示。
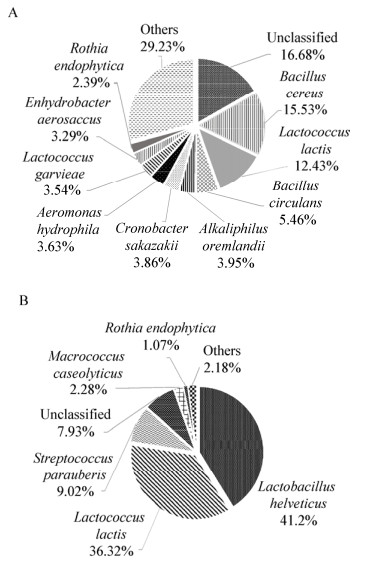
|
| 图 4 鲜马奶(A)和酸马奶(B)在种水平主要细菌相对含量 Figure 4 The relative abundance of major bacteria in fresh mare's milk (A) and koumiss (B) at species level |
|
|
鲜马奶和酸马奶样品在细菌属和种水平结构相差较大,鲜马奶发酵后乳杆菌或乳球菌占绝对优势,同时一些低丰度的细菌被抑制,如粪肠球菌(E. faecalis,0.25%:0.07%)。
2.2.3 OTU水平菌群结构分析OTU对于无论是各分类水平的划分还是α多样性分析都具有重要意义,因此本研究在OTU水平上对鲜马奶和酸马奶菌群进行进一步分析。鲜马奶样品共产生2 150个OTU,酸马奶样品中共有1 088个OTU (图 5)。在6份鲜马奶样品中共有的OTU为89个,所包含的序列数为27186条,分别占OTU总数和质控后合格序列数的4.01%和69.43%。把6份样品中均出现的OTU定义为鲜马奶的核心OTU,这些OTU隶属于芽孢杆菌属(24个OTU)、类芽孢杆菌(21个OTU)、乳球菌属(7个OTU)、乳杆菌属(4个OTU)。进一步将鲜马奶的核心OTU与酸马奶的核心OTU进行对比分析,发现仅有3个OTU出现在酸马奶中,分别为乳酸乳球菌(Denovo3211、Denovo636)和瑞士乳杆菌(Denovo1518),同时未发现芽孢杆菌属、类芽孢杆菌等菌属的OTU,进一步说明某些细菌会随着发酵的进行成为优势菌,而另一些则会被抑制。

|
| 图 5 鲜马奶和酸马奶样品中OTU出现次数的统计 Figure 5 Frequency of occurrences of OTU in the fresh mare's milk and koumiss |
|
|
本研究利用Spearman相关性检验分析了鲜马奶中细菌之间的相互关系。图 6A展示了鲜马奶中优势细菌属之间的相关关系,结果显示乳球菌属与链球菌属呈显著正相关(P=0.005,r=0.94),乳杆菌属与肠球菌属呈显著负相关(P=0.005,r=−0.94),巨球菌与假单胞菌属呈显著负相关(P=0.005,r=−0.94),与链球菌属呈显著正相关(P=0.042,r=0.83)。图 6B展示了鲜马奶中优势细菌种之间的相关关系,结果显示肠膜明串珠菌与格氏乳球菌(P=0.049,r=0.81)、乳酸乳球菌(P=0.005,r=0.94)呈显著正相关关系,与嗜水气单胞菌呈显著负相关(P=0.02,r=−0.88)。

|
| 图 6 鲜马奶中属水平(A)和种水平(B)细菌之间的相关性 Figure 6 The correlation of bacterial genus level (A) and bacteria species level (B) in fresh mare's milk Note: *: P < 0.05; **: P < 0.01; ***: P < 0.001. |
|
|
鲜马奶作为一种具有地域民族特色的食物,一直以来深受草原牧民的喜爱。有学者对鲜马奶的营养成分进行检测[22-24],但很少有人探究鲜马奶的微生物组成,本文首次利用三代测序技术在“种”水平解析鲜马奶中细菌的组成,并与酸马奶的菌群结构作了比较,不仅全面揭示了鲜马奶中微生物组成,同时也展示了鲜马奶和酸马奶菌群之间的演替变化。
本研究中发现蜡样芽孢杆菌是鲜马奶含量最多的菌种。蜡样芽孢杆菌是潜在的致病菌,其毒性取决于特定的胞外分泌物,如磷脂酶和脑苷脂,蜡样芽孢杆菌在破坏细胞膜及诱导坏死性小肠结肠炎中发挥作用[25-26]。乳头是原料乳中微生物来源之一,并且农场放牧行为也会对原料乳中菌群多样性产生影响[27]。由于鲜马奶采集于露天环境且牧场均属于牧民自己管理,因此鲜马奶中的蜡样芽孢杆菌可能是环境或青贮饲料对其污染而导致。鲜马奶中还检测出阪崎肠杆菌、Alkaliphilus oremlandii等条件致病菌。阪崎肠杆菌是婴幼儿配方奶粉中危害较大的食源性致病菌,其感染新生儿可致其患脑膜炎等疾病[28]。因此直接饮用鲜马奶存在一定的风险。
An等[13]曾提出鲜马奶中的乳酸菌是主要由球菌组成的,本研究结果显示鲜马奶中不仅有肠膜明串珠菌、格氏乳球菌和乳酸乳球菌等球菌,还有一些低丰度的乳杆菌种,如瑞士乳杆菌、德式乳杆菌、类干酪乳杆菌。并且本研究中检测出了乳球菌属和明串珠菌属的部分低丰度菌种,如棉籽糖乳球菌(0.58%)、柠檬明串珠菌(0.18%)等。这与本研究中使用的三代测序技术在检测微生物多样性方面的优势有关,同时也为样品中低丰度物种的研究提供了新思路。Zhang等[29]曾指出乳球菌与乳糖呈正相关,而本研究中检测到鲜马奶中乳球菌属的含量可达18.18%,因此也可以间接表明鲜马奶中的乳糖含量很高。
本研究发现鲜马奶有着复杂的微生物结构,但发酵成为酸马奶后微生物多样性显著降低。Zhong等和李伟程等基于高通量测序技术揭示了新疆和俄罗斯等地酸马奶中的微生物多样性,发现乳杆菌为主要的优势菌群,同时存在少量的志贺菌属[30-31]。本文中引用对比的5份酸马奶中,2份以乳酸乳球菌为绝对优势菌,相对含量分别为97.90%和76.12%,2份以瑞士乳杆菌为最优势菌(89.61%和91.44%),而本研究中鲜马奶的优势菌种为蜡样芽孢杆菌,其次为乳酸乳球菌,同时存在着Alkaliphilus oremlandii、粪肠球菌等条件致病菌。Gesudu等[17]和布仁其其格等[12]分别指出乳杆菌和乳球菌在马奶开始发酵后迅速繁殖逐渐成为环境中的优势菌,产生高浓度乳酸,使得酸马奶的pH值降低,抑制了致病菌和条件致病菌的生长,降低了酸马奶中微生物多样性[32]。此外,殷文政等[33]研究表明在酸马奶发酵过程中,乳酸菌代谢的抑菌物质也会抑制大量有害菌的生长。孙天松等[34]基于纯培养方法发现新疆酸马奶中瑞士乳杆菌(51.3%)、嗜酸乳杆菌(18.4%)和干酪乳杆菌假植物亚种(8.5%)为优势菌,未分离到粪肠球菌等条件致病菌,这可能是由于酸马奶发酵过程中乳杆菌、乳球菌等优势菌群产生大量乳酸和抑菌物质,抑制了条件致病菌的生长。本文中相关性分析也发现了乳酸菌与条件致病菌呈显著的负相关,暗示着马奶发酵过程中明串珠菌、乳酸乳球菌等乳酸菌抑制蜡样芽孢杆菌、Alkaliphilus oremlandii等条件致病菌的生长,导致其多样性显著降低。
在马奶发酵过程中,同一菌种不同菌株的发酵特性可能有所不同。马玉珠[35]研究表明瑞士乳杆菌的不同菌株之间发酵特性具有很大差异,并且对酸表现出了不同的耐受性。本研究中发现6份鲜马奶的核心OTU中4个属于乳酸乳球菌,但与酸马奶对比后发现,仅有2个乳酸乳球菌的OTU为11份样品所共有,因此在酸马奶的发酵过程中可能是几株菌共同发酵,并且由一株或几株作为发酵剂而占主导地位,但仍需后续验证。
4 结论研究发现,鲜马奶的微生物多样性显著高于酸马奶。在马奶自然发酵过程中,乳酸菌利用乳糖快速生长,逐渐成为优势菌种,同时抑制了其它菌株,包括条件致病菌的生长,使得酸马奶中的微生物多样性显著降低。由此可见,鲜马奶中的微生物组成,特别是乳酸菌的组成对酸马奶的品质具有重要影响。
| [1] |
Kong HR, Tang SH, Hu Y, et al. Present situation and progress of koumiss[J]. China Dairy Industry, 2016, 44(5): 32-35. (in Chinese) 孔杭如, 唐善虎, 胡洋, 等. 酸马奶研究现状及进展[J]. 中国乳品工业, 2016, 44(5): 32-35. DOI:10.3969/j.issn.1001-2230.2016.05.008 |
| [2] |
Jastrzębska E, Wadas E, Daszkiewicz T, et al. Nutritional value and health-promoting properties of mare's milk − a review[J]. Czech Journal of Animal Science, 2017, 62(12): 511-518. DOI:10.17221/61/2016-CJAS |
| [3] |
Gu XY, Guo J, Li SS, et al. Comparison on milk fat fatty acids profile of Inner Mongolian cow, horse and Bactrian camel[J]. China Dairy Industry, 2016, 44(3): 16-19. (in Chinese) 顾翔宇, 郭军, 李莎莎, 等. 内蒙古牛马驼乳脂肪中脂肪酸构成的比较[J]. 中国乳品工业, 2016, 44(3): 16-19. DOI:10.3969/j.issn.1001-2230.2016.03.004 |
| [4] |
Abdel-Salam AM, Al-Dekheil A, Babkr A, et al. High fiber probiotic fermented mare's milk reduces the toxic effects of mercury in rats[J]. North American Journal of Medical Sciences, 2010, 2(12): 569-575. |
| [5] |
Yang WH, De H, Zhang M. The use of mare's milk and its products[J]. Journal of Inner Mongolia University for Nationalities, 2008, 14(2): 89-90. (in Chinese) 杨文华, 德慧, 张珉. 马乳及乳产品利用[J]. 内蒙古民族大学学报, 2008, 14(2): 89-90. |
| [6] |
Liu HX, Ren Y, Zhang DL, et al. Isolation and identification of lactic acid bacteria from koumiss from Xilingol in Inner Mongolia[J]. China Dairy Industry, 2015, 43(10): 27-30. (in Chinese) 刘红新, 任艳, 张冬蕾, 等. 内蒙古锡林郭勒地区酸马奶中乳酸菌的分离鉴定[J]. 中国乳品工业, 2015, 43(10): 27-30. DOI:10.3969/j.issn.1001-2230.2015.10.006 |
| [7] |
Menghe BLG, Wu RN, Wang LP, et al. Isolation and identification of Lactobacillus from koumiss collected in Inner Mongolia and People's Republic of Mongolia[J]. China Dairy Industry, 2004, 32(11): 6-11. (in Chinese) 孟和毕力格, 乌日娜, 王立平, 等. 不同地区酸马奶中乳杆菌的分离及其生物学特性的研究[J]. 中国乳品工业, 2004, 32(11): 6-11. DOI:10.3969/j.issn.1001-2230.2004.11.002 |
| [8] |
Sun ZH, Liu WJ, Zhang JC, et al. Identification and characterization of the dominant lactobacilli isolated from koumiss in China[J]. The Journal of General and Applied Microbiology, 2010, 56(3): 257-265. DOI:10.2323/jgam.56.257 |
| [9] |
Hu SL, Liu HX, Yu J, et al. Diversity of lactic acid bacteria associated with traditional fermented milk from Hulun Buir in Inner Mongolia[J]. Microbiology China, 2016, 43(5): 984-990. (in Chinese) 呼斯楞, 刘红新, 于洁, 等. 内蒙古呼伦贝尔地区传统发酵乳中乳酸菌的多样性分析[J]. 微生物学通报, 2016, 43(5): 984-990. |
| [10] |
Li CK, Hou QC, Duolana, et al. Koumiss consumption alleviates symptoms of patients with chronic atrophic gastritis: a possible link to modulation of gut Microbiota[J]. Journal of Nutritional Oncology, 2017, 2(4): 48-63. |
| [11] |
Yao GQ. Study on bacterial diversity and functional genes in traditional fermented dairy products[D]. Hohhot: Doctoral Dissertation of Inner Mongolia Agricultural University, 2017 (in Chinese) 姚国强.传统发酵乳中细菌多样性及其功能基因研究[D].呼和浩特: 内蒙古农业大学博士学位论文, 2017 |
| [12] |
Burenqiqige, Gao YH, Ren XJ, et al. Dynamic changes of bacteria community structure during koumiss fermentation[J]. Food Science, 2016, 37(11): 108-113. (in Chinese) 布仁其其格, 高雅罕, 任秀娟, 等. 不同发酵时期酸马奶细菌群落结构[J]. 食品科学, 2016, 37(11): 108-113. DOI:10.7506/spkx1002-6630-201611019 |
| [13] |
An Y, Adachi Y, Ogawa Y. Classification of lactic acid bacteria isolated from Chigee and mare milk collected in Inner Mongolia[J]. Animal Science Journal, 2004, 75(3): 245-252. DOI:10.1111/j.1740-0929.2004.00183.x |
| [14] |
Cao CX, Han W, Zhang HP. Application of third generation sequencing technology to microbial research[J]. Microbiology China, 2016, 43(10): 2269-2276. (in Chinese) 曹晨霞, 韩琬, 张和平. 第三代测序技术在微生物研究中的应用[J]. 微生物学通报, 2016, 43(10): 2269-2276. |
| [15] |
Mosher JJ, Bowman B, Bernberg EL, et al. Improved performance of the PacBio SMRT technology for 16S rDNA sequencing[J]. Journal of Microbiological Methods, 2014, 104: 59-60. DOI:10.1016/j.mimet.2014.06.012 |
| [16] |
Shin SC, Ahn DH, Kim SJ, et al. Advantages of single-molecule real-time sequencing in high-GC content genomes[J]. PLoS One, 2013, 8(7): e68824. DOI:10.1371/journal.pone.0068824 |
| [17] |
Gesudu Q, Zheng Y, Xi XX, et al. Investigating bacterial population structure and dynamics in traditional koumiss from Inner Mongolia using single molecule real-time sequencing[J]. Journal of Dairy Science, 2016, 99(10): 7852-7863. DOI:10.3168/jds.2016-11167 |
| [18] |
Caporaso JG, Bittinger K, Bushman FD, et al. PyNAST: a flexible tool for aligning sequences to a template alignment[J]. Bioinformatics, 2010, 26(2): 266-267. |
| [19] |
Edgar RC. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [20] |
Price MN, Dehal PS, Arkin AP. Fasttree: computing large minimum evolution trees with profiles instead of a distance matrix[J]. Molecular Biology and Evolution, 2009, 26(7): 1641-1650. DOI:10.1093/molbev/msp077 |
| [21] |
Qimu G. Dynamics of bacterial diversity and functional genes during koumiss fermentation[D]. Hohhot: Doctoral Dissertation of Inner Mongolia Agricultural University, 2017 (in Chinese) 其木格苏都.自然发酵酸马奶细菌多样性及其基因动态变化研究[D].呼和浩特: 内蒙古农业大学博士学位论文, 2017 |
| [22] |
Gulibahar K, Gao XL, Chang ZY, et al. Comparative the basic properties and composition of mare's milk and camel milk, donkey milk, milk[J]. Food Science and Technology, 2017, 42(7): 123-127. (in Chinese) 古丽巴哈尔卡吾力, 高晓黎, 常占瑛, 等. 马乳与驼乳、驴乳、牛乳基本理化性质及组成比较[J]. 食品科技, 2017, 42(7): 123-127. |
| [23] |
Gao DL, Mu QE, Liu LM, et al. Determination and comparison on protein and amino acids profile of four kinds of livestock milk in Inner Mongolian[J]. Food Science and Technology, 2017, 42(2): 267-272. (in Chinese) 高玎玲, 木其尔, 刘莉敏, 等. 内蒙古4种家畜乳蛋白质和氨基酸检测与比较[J]. 食品科技, 2017, 42(2): 267-272. |
| [24] |
Liu YD, Song Q, Zhi X, et al. Fatty acid composition and distribution of triacylglycerol fraction from mare and mature breast milk[J]. Science and Technology of Food Industry, 2012, 33(18): 171-173, 189. (in Chinese) 刘亚东, 宋秋, 支潇, 等. 马奶和成熟母乳甘油三酯中脂肪酸组成及分布[J]. 食品工业科技, 2012, 33(18): 171-173, 189. |
| [25] |
Berthold-Pluta A, Pluta A, Garbowska M. The effect of selected factors on the survival of Bacillus cereus in the human gastrointestinal tract[J]. Microbial Pathogenesis, 2015, 82: 7-14. DOI:10.1016/j.micpath.2015.03.015 |
| [26] |
Elshaghabee FMF, Rokana N, Gulhane RD, et al. Bacillus as potential probiotics: status, concerns, and future perspectives[J]. Frontiers in Microbiology, 2017, 8: 1490. DOI:10.3389/fmicb.2017.01490 |
| [27] |
Zheng Y, Xi XX, Xu HY, et al. Using PacBio long-read high-throughput microbial gene amplicon sequencing to evaluate infant formula safety[J]. Journal of Agricultural and Food Chemistry, 2016, 64(37): 6993-7001. DOI:10.1021/acs.jafc.6b01817 |
| [28] |
Joseph S, Sonbol H, Hariri S, et al. Diversity of the Cronobacter genus as revealed by multilocus sequence typing[J]. Journal of Clinical Microbiology, 2012, 50(9): 3031-3039. DOI:10.1128/JCM.00905-12 |
| [29] |
Zhang FX, Wang ZX, Lei FY, et al. Bacterial diversity in goat milk from the Guanzhong area of China[J]. Journal of Dairy Science, 2017, 100(10): 7812-7824. DOI:10.3168/jds.2017-13244 |
| [30] |
Zhong Z, Hou Q, Kwok L, et al. Bacterial microbiota compositions of naturally fermented milk are shaped by both geographic origin and sample type[J]. Journal of Dairy Science, 2016, 99(10): 7832-7841. DOI:10.3168/jds.2015-10825 |
| [31] |
Li WC, Hou QC, Yu J, et al. Study on the diversity of microbial in traditional fermented dairy products[J]. Science and Technology of Food Industry, 2018, 39(1): 131-136. (in Chinese) 李伟程, 侯强川, 于洁, 等. 传统发酵乳制品中微生物多样性研究[J]. 食品工业科技, 2018, 39(1): 131-136. |
| [32] |
Blaya J, Barzideh Z, LaPointe G. Symposium review: interaction of starter cultures and nonstarter lactic acid bacteria in the cheese environment[J]. Journal of Dairy Science, 2018, 101(4): 3611-3629. DOI:10.3168/jds.2017-13345 |
| [33] |
Yin WZ, Wu N, Qian JW, et al. Study on biological activity dynamic of Kumys from Xilinguole (1)[J]. Journal of Inner Mongolia Agricultural University, 2002, 23(1): 9-16. (in Chinese) 殷文政, 乌尼, 钱建伟, 等. 锡林郭勒牧区马奶酒生物活性动态的研究(一)[J]. 内蒙古农业大学学报, 2002, 23(1): 9-16. |
| [34] |
Sun TS, Wang JG, Zhang LB, et al. The biodiversity of lactic acid bacteria isolated from koumiss-a traditional fermented mare milk product in Xinjiang of China[J]. Microbiology China, 2007, 34(3): 451-454. (in Chinese) 孙天松, 王俊国, 张列兵, 等. 中国新疆地区酸马奶中乳酸菌生物多样性研究[J]. 微生物学通报, 2007, 34(3): 451-454. DOI:10.3969/j.issn.0253-2654.2007.03.013 |
| [35] |
Ma YZ. Screening of Lactobacillus helveticus applied in active lactic acid bacteria beverages and its metabolomics studies[D]. Hohhot: Master's Thesis of Inner Mongolia Agricultural University, 2017 (in Chinese) 马玉珠.适用于活性乳酸菌饮料生产瑞士乳杆菌的筛选及其代谢组学研究[D].呼和浩特: 内蒙古农业大学硕士学位论文, 2017 |
 2019, Vol. 46
2019, Vol. 46