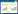扩展功能
文章信息
- 李静, 杨弢, 李玥, 朱成艳, 姚远, 赵语夏, 张潇月, 周潇, 肖嶙, 赵珂
- LI Jing, YANG Tao, LI Yue, ZHU Cheng-Yan, YAO Yuan, ZHAO Yu-Xia, ZHANG Xiao-Yue, ZHOU Xiao, XIAO Lin, ZHAO Ke
- 金沙土遗址土壤细菌群落结构和推测的代谢特征
- Diversity and speculative metabolism of bacteria from soil of Jinsha earthen site
- 微生物学通报, 2019, 46(12): 3205-3215
- Microbiology China, 2019, 46(12): 3205-3215
- DOI: 10.13344/j.microbiol.china.190080
-
文章历史
- 收稿日期: 2019-01-25
- 接受日期: 2019-04-25
- 网络首发日期: 2019-05-31
2. 成都文物考古研究院 四川 成都 610072
2. Chengdu Institute of Cultural Relics and Archaeology, Chengdu, Sichuan 610072, China
金沙土遗址位于四川省成都市青羊区青羊大道西侧,是我国进入21世纪的第一项重大考古发现。迄今为止,金沙土遗址出土了全球同时期遗址中最密集的象牙及数量最丰富的金器和玉器[1]。金沙土遗址的考古发现将成都的历史从距今2 500年提前到距今3 200年前后,表明成都平原是长江上游文明的起源中心,对研究古蜀历史文化具有极其重要的意义[2]。为保护和展示金沙土遗址商周时期大型祭祀场所,在原址上建成了遗迹馆。遗迹馆的修建虽有效缓解了风吹、日晒、雨淋等对土遗址的破坏,但土遗址与大地相连,不能脱离它所依附的环境,且长期处于半开放的自然环境中,土壤与环境因子之间发生着错综复杂的相互作用,在物理、化学、生物等因素的影响下,金沙土遗址的部分位置已出现了不同程度的酥碱、黄斑、裂隙和霉变等劣化问题,严重威胁金沙土遗址的长期保存。目前,人们对土遗址劣化原因的研究主要通过测定遗址土壤中的盐分含量、可溶盐的反复结晶作用、水分含量、温度变化等[3-5]进行,这些分析主要集中在物理、化学作用上,而对生物因素却鲜有关注。因此,本研究采用PCR-DGGE与Biolog相结合的方法分析不同劣化状态下金沙土遗址中土壤微生物群落结构组成和功能,为科学有效地保护金沙土遗址提供理论依据。
1 材料与方法 1.1 样品采集2017年10月30日在金沙土遗址遗迹馆,根据土遗址的保存状态,选择目前保存状况最差的象牙坑区域作为研究对象,并针对该区域土遗址劣化状态和劣化程度的不同,选择4个具有代表性位置(4个采样位置的劣化程度依次为J4 > J3 > J2 > J1)进行样品采集。为了尽可能使所采样品具有代表性,每个采样位置选择5个点进行样品采集并充分混合后装于无菌离心管中,立即置于装有液氮的保温箱,带回实验室即刻进行相关分析。
1.2 主要试剂和仪器Biolog-Eco板,Biolog公司;FastDNA® SPIN Kit for Soil,MP Biomedicals公司;RNA PowerSoil® Total RNA Isolation Kit,Qiagen公司;RevertAid First Strand cDNA Synthesis Kit,Thermo公司;PCR引物、凝胶回收试剂盒-UNIQ-10 DNA纯化试剂盒,生工生物工程(上海)股份有限公司;PCR Mix、pMD19-T,大连宝生物工程有限公司。
冷冻离心机,Eppendorf公司;涡旋匀仪,Scilogex公司;PCR仪、凝胶成像仪、电泳仪、DCode DGGE System,Bio-Rad公司;多功能酶标仪,Molecular Devices公司。
1.3 土遗址细菌功能多样性测定 1.3.1 Biolog-Eco操作方法采用Biolog公司生产的Biolog-Eco微平板分析不同劣化程度土壤中细菌群落的代谢特征。Biolog-Eco板是有31种碳源的生态板,每种碳源3个重复,从4 ℃冰箱中取出后于室温预热30 min备用。称10 g土壤样品,加入90 mL无菌生理盐水,摇床振荡30 min后,用移液枪吸取5 mL溶液,加入装有45 mL无菌水的三角瓶中进行100倍稀释。在超净工作台内向Biolog-Eco平板各小孔中加入150 μL的样品,置于25 ℃恒温培养,每隔24 h于波长为590 nm和750 nm的Biolog读数器上读数,整个培养过程共持续7 d[6-7]。
1.3.2 微生物碳源代谢水平各土壤样品的平均光密度值(Average well color development,AWCD)的计算方法:AWCD=∑[(Ci− R)590−(Ci−R)750]/31,其中Ci是第i反应孔的吸光值,R是对照孔的吸光值,31是Biolog-Eco板中的底物数量[8]。
1.3.3 微生物多样性计算方法及统计分析选取72 h平均光密度值(AWCD)计算不同土壤样品中微生物群落多样性指数[6, 9]:
Shannon指数(H ′)=−∑(Pi×lnPi);
Simpson多样性指数(D)=1−∑(Pi)2;
McIntosh多样性指数
式中:Pi为第i孔相对光密度值与整个平板相对吸光度值总和的比值;ni是第i孔的相对吸光值(Ci−R);选取72 h平均光密度值(AWCD)应用Canoco 5.0进行微生物群落碳源代谢特征的主成分分析(Principal component analysis,PCA)。
1.4 PCR-DGGE分析土遗址土壤微生物多样性 1.4.1 总DNA和RNA提取称取0.5 g土壤样品按照操作手册使用FastDNA® SPIN Kit for Soil提取土壤总DNA,获得的DNA于−20 ℃保存备用。按照操作手册使用RNA PowerSoil® Total RNA Isolation Kit提取土壤总RNA,对获得的总RNA按照Thermo公司试剂盒RevertAid First Strand cDNA Synthesis Kit说明书进行反转录,所得的cDNA和RNA于−80 ℃保存备用。
1.4.2 PCR扩增采用巢氏PCR对目标片段进行扩增。第一轮PCR扩增的引物为21f (5′-AGAGTTTGATCMTGG CTCAG-3′)和1492r (5′-TACGGYTACCTTGTTACG ACTT-3′)[10],第二轮所用的引物为F-968-GC (5′-CG CCCGGGGCGCGCCCCGGGCGGGGCGGGGGCACGGGGGGAACGCGAAGAACCTTAC-3′)和R-1401 (5′-CGGTGTGTACAAGACCC-3′)[11-12]。两轮的PCR反应体系与条件均参照Gu等[13]的方法进行。PCR产物用1.0%的琼脂糖电泳检测,目标片段大小约为500 bp。
1.4.3 PCR-DGGE产物检测与指纹图谱分析DGGE丙烯酰胺凝胶强度为8%,变性剂梯度为30%−70%。凝胶板制作好之后,组装放入含有1×TAE的电泳槽中,预热到60 ℃时点样。每孔加入20 μL PCR产物,点样完成后,先50 V预电泳30 min,然后在60 ℃、180 V下电泳6 h。电泳结束后取出凝胶,使用银染法进行染色[14]后进行拍照。采用Bio-Rad公司的Quantity One软件(V4.5)对DGGE条带进行数字化分析,评价金沙土遗址不同劣化土壤微生物的群落结构。
1.4.4 DGGE条带测序及系统发育分析将DGGE条带中的优势条带和特异性的条带切割后,用无菌去离子水清洗3次,并用20 μL去离子水浸泡过夜,再以浸液为模板进行PCR扩增。用第2次PCR扩增时用的引物(无GC发夹)进行PCR扩增,体系和条件同1.4.2。PCR产物用UNIQ-10 DNA纯化试剂盒进行纯化,之后用大连宝生物公司的pMD19-T Vector试剂盒进行克隆。挑选白色菌落进一步测序,测序由生工生物工程(上海)股份有限公司完成。测序结果通过BLAST程序在GenBank数据库中获取相似度较高的序列,然后使用MEGA 7.0采用Neighbor-Joining方法构建系统发育树[15]。将测序得到的序列提交GenBank数据库,登录号为MK379976−MK379996。
2 结果与分析 2.1 Biolog试验结果 2.1.1 微生物整体代谢活性AWCD值反映了样品所含微生物在群落水平上对不同类型碳源的利用能力,从功能代谢方面显示微生物群落结构多样性及其功能差异。由图 1可知,最初的24 h,4个土壤样品的AWCD值差异不明显,培养48 h后AWCD值均呈现不断上升的趋势,各土壤微生物群落AWCD值差异逐渐加大,J2土壤样品的AWCD值快速上升,在所有样品中始终保持最大,而样品J4和J3的AWCD值呈现出相互交叉的变化过程,且AWCD值均高于J1土壤样品。来自金沙土遗址的4个土壤样品AWCD值变化趋势不同,表明不同劣化状态下土壤微生物碳源利用能力存在差异,J2样品中的土壤微生物代谢活性最高,J4和J3代谢活性相近但低于J2,J1样品中微生物活性最低。

|
| 图 1 不同劣化状态下土壤微生物AWCD变化特征随时间的动态变化 Figure 1 AWCD changes with incubation time of different degradation samples |
|
|
按化学性质把Biolog-Eco微平板上的31种碳源分成6类,即多聚物类、碳水化合物类、氨基酸类、酚酸类、羧酸类和胺类[16]。图 2为4个样品对微孔板中6大类碳源的相对利用率分析结果。
|
| 图 2 遗址土壤微生物群落对6类碳源的相对利用率 Figure 2 Relative utilization ratios of 6 kinds of carbon sources by microbial community in four soil samples |
|
|
结果显示,不同样品土壤微生物对6大类碳源的相对利用率较为平均,但都对碳水化合物类、多聚物类碳源表现出较高的利用率。样品J2对各类碳源的代谢水平均高于其它土壤样品,其碳源代谢的优势群落依次为碳水化合物类代谢群落、多聚物类代谢群落、酚酸类代谢群落等,J3和J4样品土壤中土壤微生物对各类碳源的利用率较为相似,但对碳源代谢的优势群落有差异,J4样品土壤中微生物对氨基酸类、胺类、酚酸类的利用率高于J3样品,而J3样品土壤中微生物对其它3种碳源的利用率则高于J4样品土壤中。相较于其它3个土壤样品,J1样品微生物对各类碳源的利用率最低,表明不同劣化状态下土壤微生物群落代谢功能类群存在差异。
2.1.3 遗址土壤微生物群落功能多样性分析采用丰富度指数、Shannon指数、Simpson指数和McIntosh指数对不同劣化状态下土壤微生物群落的功能多样性特征进行比较。如表 1所示,土壤微生物群落代谢功能多样性指数在不同劣化状态下有一定变化。J2样品土壤微生物碳源利用丰富度指数(S)、Shannon指数(H′)、Simpson指数(D)和McIntosh指数(U)均表现为最高,与AWCD值随培养时间的变化曲线表现结果一致,其中丰富度指数(S)和McIntosh指数(U)显著高于其它样品,J4和J3样品土壤微生物丰富度指数(S)显著高于J1 (P < 0.05)。表明J2土壤样品中的微生物种类丰富,碳源利用度较高,活性较强,不同劣化状态下土壤微生物群落代谢特征存在差异,也说明金沙土遗址土壤微生物群落代谢功能受到土壤劣化的影响。
| 样品编号 Sample No. |
丰富度指数 Richness index (S) |
多样性指数 Shannon index (H ′) |
多样性指数 Simpson index (D) |
多样性指数 McIntosh index (U) |
| J4 | 11.33±2.517c | 1.470 5±0.086 04a | 0.995 1±0.003 37a | 0.338 4±0.017 92b |
| J3 | 18.01±1.732b | 1.489 5±0.087 80a | 0.995 9±0.000 87a | 0.420 0±0.043 45b |
| J2 | 28.67±1.528a | 1.500 6±0.528 40a | 0.996 2±0.000 40a | 0.651 4±0.167 57a |
| J1 | 6.67±3.215d | 1.468 1±0.205 80a | 0.994 9±0.002 69a | 0.346 8±0.118 18b |
| 注:n=3,计算结果为平均值±标准差,字母不同表示样品之间存在显著性差异(P < 0.05). Note: n=3, the value is the average±standard deviation, the difference in capital letters indicates that there are significant differences among samples (P < 0.05). |
||||
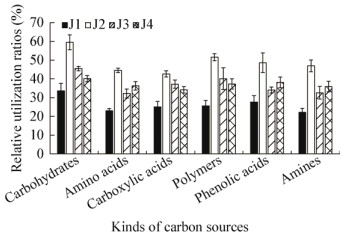
主成分分析能够直观地表现出微生物群落代谢对一些碳源利用的偏好性,箭头所指的正方向即为正相关,说明微生物比较偏好利用此类碳源。
由图 3可以看出,不同样品对碳源底物的利用情况不同,4个样品大致可以分成2种类型。一类为J2样品,比较偏好利用的碳源有:E2 (N-乙酰-D-葡萄糖胺)、D2 (D-甘露醇)、G1 (D-纤维二糖)、C1 (吐温40)、D1 (吐温80),其中E2、D2、G1为碳水化合物类,C1、D1为多聚物类。另一类为J4、J3、J1样品,这3个样品的箭头方向较为一致,比较偏好利用的碳源有:E2 (N-乙酰-D-葡萄糖胺)、G1 (D-纤维二糖)、B2 (D-木糖)、H1 (α-D-乳糖)、B3 (D-半乳糖醛酸)、A2 (β-甲基-D-葡萄糖苷)、C1 (吐温40)、D1 (吐温80)、F1 (肝糖),其中E2、G1、B2、H1、B3、A2为碳水化合物类,C1、D1、F1为多聚物类。4个遗址土壤样品所含微生物均对碳水化合物类以及多聚物类碳源利用率高,结果与各样品中微生物群落碳源利用水平分析结果一致,说明样品中利用碳水化合物和多聚物的微生物数量和代谢活性均高于其他微生物类群。
|
| 图 3 4个土壤样品所含微生物对31种碳源利用情况的主成分分析 Figure 3 Principal components analysis (PCA) of different carbon source utilization by microbial in four soil samples |
|
|
微生物群落的遗传多样性在一定程度上可以通过DGGE条带的数量反映出来[17]。从金沙土遗址不同劣化状态下土壤中分别提取DNA和RNA,PCR扩增产物进行DGGE分析结果显示(图 4),从各样品中分离出数目不等的条带,不同样品各条泳道的强度和迁移率不相同。从整体上的条带数量来看,J2土壤样品在DNA和RNA水平上均有较多的条带处于同一位置,而其它样品条带位置差异较大(图 4),样品J1的条带数量在DNA和RNA水平上都是最少的,RNA水平上的条带明显少于DNA水平,说明该样品中其具有活性的细菌多样性较为单一(表 2)。

|
| 图 4 4个土壤样品微生物16S rRNA基因片段DGGE电泳结果 Figure 4 DGGE banding patterns of 16S rRNA fragment from four soil samples 注:其中J4−J1为DNA,J4#−J1#为RNA. Note: J4−J1 belong to DNA; J4#−J1# belong to RNA. |
|
|
| 样品编号 Sample No. |
条带数 Band |
多样性指数 Shannon index (H′) |
均匀度指数 Evenness index (J) |
辛普森指数 Simpson index (D) |
优势度指数 Dominance index (C) |
|
| DNA-level | J4 | 23 | 3.22 | 0.996 0 | 0.955 4 | 0.044 6 |
| J3 | 23 | 3.12 | 0.995 4 | 0.955 4 | 0.044 6 | |
| J2 | 26 | 3.36 | 0.996 1 | 0.961 9 | 0.042 9 | |
| J1 | 22 | 2.87 | 0.992 1 | 0.952 2 | 0.047 8 | |
| RNA-level | J4# | 20 | 2.95 | 0.998 7 | 0.948 7 | 0.051 3 |
| J3# | 19 | 2.54 | 0.995 7 | 0.947 0 | 0.053 0 | |
| J2# | 24 | 3.08 | 0.998 9 | 0.957 1 | 0.038 7 | |
| J1# | 6 | 1.58 | 0.993 2 | 0.829 1 | 0.170 9 | |
依据DGGE电泳图谱中条带的数字化结果进行多样性指数计算,通过多样性指数(H′)、均匀度指数(J)、辛普森指数(D)以及优势度指数(C)等指数对不同劣化状态下各样品细菌多样性进行了综合分析(表 2)。结果表明,在DNA和RNA水平上,各土壤样品中细菌的各个指标都存在着不同程度的差异。在DNA水平上,J2样品的细菌多样性指数(H′)、均匀度指数(J)、辛普森指数(D)皆为最高,其次为J4、J3,而J1样品的细菌多样性指数(H′)、均匀度指数(J)、辛普森指数(D)均为最低。RNA水平上样品中细菌的各项指标呈现出与DNA水平一致的结果,表现为J2 > J4 > J3 > J1。综合各项指标来看,发生劣化的土壤中细菌多样性升高,表明金沙土遗址中细菌多样性和群落结构受土壤劣化的影响,可能对土遗址的保存具有潜在危害。
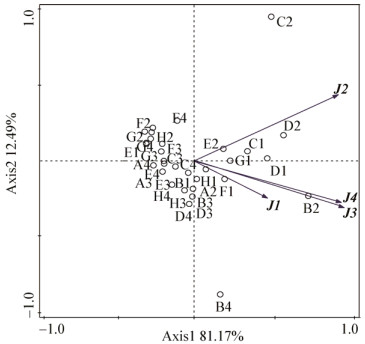
运用CANOCO软件对各个样品的细菌群落结构进行PCA分析,结果如图 5所示,J2样品的DNA和RNA水平的点聚在了一起,其余各样点在DNA水平上聚在了一起,而在RNA水平上则较为分散,其中J1样品的差异最大,说明J2样品中总体细菌群落结构和活性细菌群落结构具有较高的一致性,而其它样品的总体细菌群落结构和活性细菌群落结构一致性较低,J1样品的一致性最低。
|
| 图 5 不同样品土壤微生物群落DGGE条带的主成分分析 Figure 5 Principal component analysis (PCA) of DGGE banding patterns of soil microbial community in different samples |
|
|
电泳结束后,对主条带进行切胶回收,克隆测序后,共获得21条序列,所有序列与NCBI数据库中已知序列相似性均在98%−100%之间(表 3)。结果如表 3所示,土遗址样品中的细菌分属4个门:放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)、异常球菌-栖热菌门(Deinococcus-Thermus)、变形菌门(Proteobacteria);7个属分别为红色杆菌属(Rubrobacter)、北里孢菌属(Kitasatospora)、短状杆菌属(Brachybacterium)、Tellurimicrobium、异常球菌属(Deinococcus)、伯克氏菌属(Burkholderia)、短波单胞菌属(Brevundimonas),以上7个属的细菌在DNA水平上均有检出,而伯克氏菌属、Tellurimicrobium和Uncultured Burkholderiales bacterium没有在RNA水平上检出。此外,在DNA水平上,红色杆菌属、异常球菌属、短波单胞菌属细菌在所有供试样品中均有检出,短状杆菌属细菌仅在J2和J3样品中检出,Tellurimicrobium仅在J2样品中检出;伯克氏菌属细菌仅在J3样品中检出。在RNA水平上,红色杆菌属和短波单胞菌属细菌也在所有供试样品中均有检出,北里孢菌属细菌仅在J3样品中检出,短状杆菌属细菌仅在J2样品中检出。回收的21条序列有11条归属于红色杆菌属,占回收序列的52.83%,且在劣化样品中检出较多(图 5,表 3)。结果表明,不同样品中细菌种群组成的差异可能与其劣化状态和劣化程度不同有关,而在RNA水平检测出的菌属值得关注。
| 条带编号 Bands No. |
不同样品中的条带 Band appearance in different samples |
归属类群(门) Phylogenetic group (Phylum) |
基因库中比对出最相似序列(属) Closest relative sequence in GenBank (Genus) |
相似度 Similarity (%) |
|||||||
| J4 | J4# | J3 | J3# | J2 | J2# | J1 | J1# | ||||
| 1-2 | + | + | + | + | + | + | + | - | Actinobacteria | Rubrobacter sp. (EU512989) | 100.0 |
| 5-1 | - | - | + | + | + | + | + | - | Rubrobacter sp . (EU512989) | 99.0 | |
| 6-1 | - | - | + | + | + | + | + | - | Rubrobacter sp . (EU512989) | 99.0 | |
| 6-6 | + | + | + | + | + | + | + | - | Rubrobacter sp.(EU512989) | 99.0 | |
| 8-1 | - | - | - | - | - | - | - | + | Rubrobacter sp . (EU512989) | 99.0 | |
| 5-2 | - | - | + | + | + | + | - | - | Rubrobacter bracarensis (NR117640) | 100.0 | |
| 5-3 | - | - | - | - | + | - | - | - | Rubrobacter bracarensis (NR117640) | 100.0 | |
| 5-6 | - | + | + | - | + | + | - | - | Rubrobacter bracarensis (NR117640) | 100.0 | |
| 6-2 | - | - | + | + | + | + | - | - | Rubrobacter bracarensis (NR117640) | 99.0 | |
| 6-4 | - | + | + | - | + | + | - | - | Rubrobacter bracarensis (NR117640) | 100.0 | |
| 6-5 | + | + | + | + | + | + | + | - | Rubrobacter bracarensis (NR117640) | 99.0 | |
| 7-1 | - | - | + | + | - | - | + | - | Kitasatospora aburaviensis (MK138636) | 100.0 | |
| 5-4 | - | - | + | - | + | + | - | - | Brachybacterium paraconglomeratum (MK249686) | 100.0 | |
| 6-3 | - | - | + | - | + | + | - | - | Brachybacterium paraconglomeratum (MK249686) | 100.0 | |
| 5-5 | - | - | - | - | + | - | - | - | Acidobacteria | Tellurimicrobium multivorans (NR146026) | 99.0 |
| 2-2 | + | + | + | - | + | + | - | - | Deinococcus-Thermus | Deinococcus metallilatus (NR137265) | 98.0 |
| 8-2 | - | - | - | - | - | - | + | + | Deinococcus metallilatus (NR137265) | 100.0 | |
| 3-2 | - | - | + | - | - | - | - | - | Proteobacteria | Burkholderia sp . (KU323 02) | 100.0 |
| 1-1 | + | + | + | + | + | + | + | + | Brevundimonas vesicularis (MK045801) | 100.0 | |
| 2-1 | + | + | + | + | + | + | + | + | Brevundimonas vesicularis (MK045801) | 100.0 | |
| 3-1 | + | - | + | - | + | - | + | - | Uncultured Burkholderiales bacterium (JX493018) | 100.0 | |
土遗址是以土作为主要建筑材料的人类历史上生产、生活等各种活动遗留下来的遗迹,是一种重要的文物资源[17]。随着科技的发展和人们对土遗址认识的逐渐加深,土遗址保护越来越受到人们的重视和普遍关注,研究也逐步深入。然而,目前有关土遗址劣化的研究主要集中在物理、化学因素对土遗址劣化的影响,且主要针对北方干旱地区的土遗址[3, 18-19],对南方潮湿地区土遗址的研究较少,生物因素,特别是微生物对土遗址劣化的潜在作用更是鲜有关注。金沙土遗址是南方潮湿地区土遗址的典型代表,目前已出现不同程度的劣化现象,而金沙土遗址中微生物类群及潜在功能如何,对金沙土遗址劣化有何影响,这都值得人们去探究。因此,本研究采用Biolog与PCR-DGGE相结合的方法研究金沙土遗址中微生物群落结构组成,解析微生物菌群代谢特征,揭示对金沙土遗址具有潜在危害的微生物菌群,为金沙土遗址的科学保护提供依据。
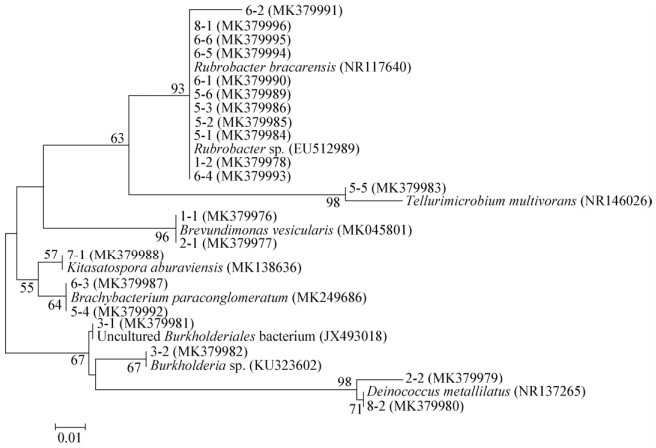
|
| 图 6 金沙土遗址土壤样品16S rRNA基因序列的系统进化树 Figure 6 Phylogenetic tree of 16S rRNA gene sequences of soil samples from Jinsha earthen site 注:菌株后括号内为基因库中比对最相似序列登录号,编号后括号内为回收条带在基因库中的登录号. Note: After the strain in parentheses is the closest relative sequence in GenBank, after the number of band in parentheses is the sequence of band in GenBank. |
|
|
Biolog微平板法作为一种方便快速的微生物检验技术,已广泛应用于环境微生物检测、微生物生态研究等方面,发挥着越来越重要的作用。在本研究中,Biolog实验结果表明,4个样品所含微生物共同偏好碳水化合物类以及多聚物类碳源,说明利用这2类碳源的土壤细菌最多,且这些土壤细菌的生理代谢活动较强。张焕军等[20]研究发现碳水化合物是土壤中最重要的有机质成分之一,约占土壤有机质总量的10%−20%,是土壤微生物的主要能源和碳源,与微生物活性显著相关[21-22],盛玉钰等[23]研究表明多聚物类是微生物利用的优势碳源。主成分分析结果显示,各样品偏好的碳源具体种类虽有所差异,但所偏好的碳源类型均为碳水化合物和多聚物类,表明微生物利用碳源的偏好与所生存的环境息息相关,而不同样品偏好的碳源种类不同可能是劣化土壤的微环境发生了变化所致。由于Biolog技术表征的只是土壤中生长快速或富营养微生物的活性,而不能反映土壤中生长缓慢或不可培养微生物活性[24-25],因此Biolog技术解析的只能是土壤中部分微生物群落特征,要想全面解析土壤微生物群落结构,还需要结合其他研究方法[26]。
PCR-DGGE实验结果表明,不同劣化情况下土壤样品中的细菌种群组成存在差异。DNA和RNA的水平都表现为劣化的土壤所含细菌在种类和丰度相对未发生劣化的土壤较为丰富,这种变化表明随着土遗址的劣化,微生物的多样性和活性菌有所增高,而这种增高反过来又可能加速了土遗址的劣化。通过条带测序,发现劣化土壤中细菌以变形菌门(Proteobacteria)和放线菌门(Actinobacteria)为主,这与变形菌门和放线菌门在土壤中广泛分布有着密切关系[27]。样品中检出率较高的细菌类型为短波单胞菌属(Brevundimonas)、红色杆菌属(Rubrobacter)、Tellurimicrobium和短状杆菌属(Brachybacterium),其中短波单胞菌属以及红色杆菌属在所有样品中均有检出,短波单胞菌属在DNA和RNA样品中均有检出,可能与该属的细菌广泛存在于土壤当中有关,而红色杆菌属在劣化土壤样品中的RNA的检出率很高,说明劣化样品中该菌的活性高。Laiz等[28]在研究中也发现红色杆菌属细菌与砖石和石灰壁画表面的粉化及玫瑰色变色有关,揭示了红色杆菌属细菌加速了文物的生物降解的进程,表明红色杆菌属细菌可能与金沙土遗址的劣化有关。此外,酸杆菌门(Acidobacteria)细菌Tellurimicrobium仅在劣化的J2样品检出,研究发现,酸杆菌门在各种生境中都存在,特别是在土壤环境中,数量占到细菌总量的20%左右,并在土壤物质循环和生态环境构建过程中起到了重要作用[29],且在富铁的酸性环境条件下含量高[30],在各种生态环境的铁循环中起到一定的作用,Lu等[31]研究发现菌株Acidobacterium capsulatum DSM 11244T在极端厌氧或微氧条件下具有异化铁还原能力。杜烨[32]在莫高窟壁画表面发现酸杆菌门细菌,Zimmermann等[33]在Altamira洞穴、Pašić等[34]在Pajsarjeva洞穴均发现酸杆菌门细菌,表明酸杆菌门细菌可能参与了文物的生物降解过程。酸杆菌门细菌仅在J2样品中检出,说明该门细菌可能与土遗址劣化和出现泛黄现象有关。短状杆菌属细菌是一类异养微生物,在生长过程中会分泌一些代谢产物[32],如其在生长过程中产酸[35],而酸性物质可能会破坏土壤结构。本研究仅在出现劣化的J2、J3样品的DNA和J2样品的RNA样品中检测到了短状杆菌属细菌,表明短状杆菌属细菌对土遗址的保护产生一定影响。付彤彤等[36]采用克隆文库手段对李渡酒窖土遗址中的微生物种群进行鉴定和分析,结果表明土遗址中优势细菌为盐单胞菌属(Halomonas)、甲基盐单胞菌属(Methylohalomonas)和乳杆菌属(Lactobacillus),其中含有较多具有耐盐碱特性的细菌,与酒窖的现场环境相一致;吕春爽等[37]通过纯培养技术从泥河湾藉箕滩分离获得130株细菌,主要归属于芽孢杆菌属(Bacillus)、短波单胞菌属(Brevundimonas)和链霉菌属(Streptomyces)。以上结果表明,不同的土遗址的微生物群落结构存在差异,这与土遗址所处的环境不同、研究者所采用的研究手段不同有关。虽然PCR-DGGE技术存在检测通量有限、高估特种丰度以及低估微生物群落大小和多样性的可能[38-39],但与目前流行的高通量测序技术相比,DGGE技术仍具有一些不可替代的优点,复杂微生物群落可以利用DGGE达到可视化,能够直观地比较和分析微生物群落结构的变化规律和快速检出样本中优势微生物[40]。基于以上技术特征,DGGE技术应用于文物微生物群落结构的分析为实现文物“病害”微生物的快速检测具有可行性和合理性,同时也为文物保护提供了相关的微生物学依据。
综上所述,本研究通过对金沙土遗址细菌群落结构组成和代谢特征的分析发现,随着土遗址劣化程度的增加,土壤微生物类群及代谢功能多样性都随之增大,其中仅在劣化土壤样品中检出或者具有高表达活性的红色杆菌属、Tellurimicrobium、短状杆菌属细菌可能参与了土壤劣化过程,揭示金沙土遗址土壤中存在多种可能对土遗址产生破坏性的微生物,为认识金沙土遗址微生物损害机制提供了理论依据。
| [1] |
Zhu ZY, Zhang Q, Wang F, et al. Discovery, excavation and significance of Jinsha Site in Chengdu[J]. Sichuan Cultural Relics, 2002(2): 3-10. (in Chinese) 朱章义, 张擎, 王方, 等. 成都金沙遗址的发现、发掘与意义[J]. 四川文物, 2002(2): 3-10. DOI:10.3969/j.issn.1003-6962.2002.02.001 |
| [2] |
Yang RX. Chengdu plain is the center of ancient civilization in the upper reaches of the Yangtze river[J]. Forum on Chinese Culture, 2003(4): 21-24. (in Chinese) 杨荣新. 成都平原是长江上游的古代文明中心[J]. 中华文化论坛, 2003(4): 21-24. DOI:10.3969/j.issn.1008-0139.2003.04.004 |
| [3] |
Wu SM, Chen L. Research on the factors of damage and preservation measures of Earthen Ruins—Case of Linzhou Ancient City[J]. Research on Heritages and Preservation, 2017, 2(1): 112-117. (in Chinese) 吴少敏, 陈雳. 土遗址的病害因素与保护措施研究——以麟州故城为例[J]. 遗产与保护研究, 2017, 2(1): 112-117. DOI:10.3969/j.issn.2096-0913.2017.01.022 |
| [4] |
Qian L, Xia Y, Hu HY, et al. A study on the soluble salt contents and occurrence state in the soil of the Earthen site at Xiongjiazhong[J]. Dunhuang Research, 2017(5): 138-140. (in Chinese) 钱玲, 夏寅, 胡红岩, 等. 熊家冢含盐情况调查与分析[J]. 敦煌研究, 2017(5): 138-140. |
| [5] |
Yu XM. A case study on the ruins of the city wall and its influencing factors—Take Suijing Ruins as an example[J]. Research on Heritages and Preservation, 2017, 2(5): 183-185. (in Chinese) 于晓敏. 浅析土遗址病害成因与保护策略——以塔城市绥靖城城墙遗址为例[J]. 遗产与保护研究, 2017, 2(5): 183-185. DOI:10.3969/j.issn.2096-0913.2017.05.049 |
| [6] |
Yan FJ, Tian XL, Dong SL, et al. Seasonal variation of functional diversity of aquatic microbial community in Apostichopus japonicus cultural pond[J]. Chinese Journal of Applied Ecology, 2014, 25(5): 1499-1505. (in Chinese) 闫法军, 田相利, 董双林, 等. 刺参养殖池塘水体微生物群落功能多样性的季节变化[J]. 应用生态学报, 2014, 25(5): 1499-1505. |
| [7] |
Zhan XY, Wu DX, Zhang L, et al. Response of microbial community functional diversity in the rhizosphere of Caragana microphylla to environmental change[J]. Acta Ecologica Sinica, 2010, 30(12): 3087-3097. (in Chinese) 展小云, 吴冬秀, 张琳, 等. 小叶锦鸡儿根际微生物群落功能多样性对环境变化的响应[J]. 生态学报, 2010, 30(12): 3087-3097. |
| [8] |
Classen AT, Boyle SI, Haskins KE, et al. Community-level physiological profiles of bacteria and fungi: plate type and incubation temperature influences on contrasting soils[J]. FEMS Microbiology Ecology, 2003, 44(3): 319-328. DOI:10.1016/S0168-6496(03)00068-0 |
| [9] |
Liu CR, Ma KP. Measurement of biotic community diversity V. methods for estimating the number of species in a community[J]. Acta Ecologica Sinica, 1997, 17(6): 601-610. (in Chinese) 刘灿然, 马克平. 生物群落多样性的测度方法V.生物群落物种数目的估计方法[J]. 生态学报, 1997, 17(6): 601-610. DOI:10.3321/j.issn:1000-0933.1997.06.003 |
| [10] |
Devereux R, Willis SG. Amplification of ribosomal RNA sequences[A]//Akkermans ADL, van Elsas JD, de Bruijn FJ. Molecular Microbial Ecology Manual[M]. Dordrecht: Springer, 1995: 277-287
|
| [11] |
Nubel U, Engelen B, Felske A, et al. Sequence heterogeneities of genes encoding 16S rRNAs in Paenibacillus polymyxa detected by temperature gradient gel electrophoresis[J]. Journal of Bacteriology, 1996, 178(19): 5636-5643. DOI:10.1128/jb.178.19.5636-5643.1996 |
| [12] |
Lane DJ, Field KG, Olsen GJ, et al. Reverse transcriptase sequencing of ribosomal RNA for phylogenetic analysis[J]. Methods in Enzymology, 1988, 167: 138-144. DOI:10.1016/0076-6879(88)67015-7 |
| [13] |
Gu YF, Zhang XP, Tu SH, et al. Soil microbial biomass, crop yields, and bacterial community structure as affected by long-term fertilizer treatments under wheat-rice cropping[J]. European Journal of Soil Biology, 2009, 45(3): 239-246. DOI:10.1016/j.ejsobi.2009.02.005 |
| [14] |
Sanguinetti CJ, Dias Neto E, Simpson AJ. Rapid silver staining and recovery of PCR products separated on polyacrylamide gels[J]. BioTechniques, 1994, 17(5): 914-921. |
| [15] |
Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis Version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [16] |
Liu YH, Wu YX, Liu CM, et al. Analysis of soil microbial diversity by Biolog-Eco technology in Maotianshan and Jiaolongtan[J]. Ecological Science, 2014, 33(6): 1047-1052. (in Chinese) 刘云华, 吴毅歆, 刘春明, 等. 帽天山、蛟龙潭土壤微生物多样性的Biolog-Eco分析[J]. 生态科学, 2014, 33(6): 1047-1052. |
| [17] |
Sun ML. Research status and development of the conservation of earthen sites[J]. Sciences of Conservation and Archaeology, 2007, 19(4): 64-70. (in Chinese) 孙满利. 土遗址保护研究现状与进展[J]. 文物保护与考古科学, 2007, 19(4): 64-70. DOI:10.3969/j.issn.1005-1538.2007.04.014 |
| [18] |
Zhao HY, Li ZX, Han WF, et al. Main diseases and their causes of earthen ruins in arid region of Northwestern China[J]. Chinese Journal of Rock Mechanics and Engineering, 2003, 22(S2): 2875-2880. (in Chinese) 赵海英, 李最雄, 韩文峰, 等. 西北干旱区土遗址的主要病害及成因[J]. 岩石力学与工程学报, 2003, 22(S2): 2875-2880. |
| [19] |
Cui K, Chong XC, Chen WW, et al. Different effects of shady and sunny slopes in undercutting area of linear earthen ruin in the Hexi corridor[J]. Journal of Engineering Geology, 2017, 25(2): 547-555. (in Chinese) 崔凯, 崇显灿, 谌文武, 等. 河西走廊地区线性土遗址阴阳坡掏蚀区差异效应研究[J]. 工程地质学报, 2017, 25(2): 547-555. |
| [20] |
Zhang HJ, Yu HY, Ding WX. Progress in the study on transformation and accumulation of carbohydrates in soil[J]. Acta Pedologica Sinica, 2013, 50(6): 1200-1206. (in Chinese) 张焕军, 郁红艳, 丁维新. 土壤碳水化合物的转化与累积研究进展[J]. 土壤学报, 2013, 50(6): 1200-1206. |
| [21] |
Larson WE, Pierce FJ, Dowdy RH. The threat of soil erosion to long-term crop production[J]. Science, 1983, 219(4584): 458-465. DOI:10.1126/science.219.4584.458 |
| [22] |
Rasmussen PE, Allmaras RR, Rohde CR, et al. Crop residue influences on soil carbon and nitrogen in a wheat-fallow system[J]. Soil Science Society of America Journal, 1980, 44(3): 596-600. DOI:10.2136/sssaj1980.03615995004400030033x |
| [23] |
Sheng YY, Lu H, Yu HM, et al. Altitude distribution of soil microbial function diversity in Alpine Meadow in Sanjiangyuan Region, Qinghai province[J]. Progress in Modern Biomedicine, 2018, 18(1): 37-42, 107. (in Chinese) 盛玉钰, 卢慧, 喻海茫, 等. 青海三江源高寒草甸土壤微生物功能多样性的海拔分布[J]. 现代生物医学进展, 2018, 18(1): 37-42, 107. |
| [24] |
Li CG, Li XM, Wang JG. Effect of soybean continuous cropping on bulk and rhizosphere soil microbial community function[J]. Acta Ecologica Sinica, 2006, 26(4): 1144-1150. (in Chinese) 李春格, 李晓鸣, 王敬国. 大豆连作对土体和根际微生物群落功能的影响[J]. 生态学报, 2006, 26(4): 1144-1150. DOI:10.3321/j.issn:1000-0933.2006.04.023 |
| [25] |
Konopka A, Oliver L, Turco Jr RF. The use of carbon substrate utilization patterns in environmental and ecological microbiology[J]. Microbial Ecology, 1998, 35(2): 103-115. |
| [26] |
Wang GH, Liu JJ, Qi XN, et al. Effects of fertilization on bacterial community structure and function in a black soil of Dehui region estimated by Biolog and PCR-DGGE methods[J]. Acta Ecologica Sinica, 2008, 28(1): 220-226. (in Chinese) 王光华, 刘俊杰, 齐晓宁, 等. Biolog和PCR-DGGE技术解析施肥对德惠黑土细菌群落结构和功能的影响[J]. 生态学报, 2008, 28(1): 220-226. |
| [27] |
Li Z, Xu ZH. Assessing bacterial diversity in soil[J]. Journal of Soils and Sediments, 2008, 8(6): 379-388. DOI:10.1007/s11368-008-0043-z |
| [28] |
Laiz L, Miller AZ, Jurado V, et al. Isolation of five Rubrobacter strains from biodeteriorated monuments[J]. Naturwissenschaften, 2009, 96(1): 71-79. DOI:10.1007/s00114-008-0452-2 |
| [29] |
Wang GH, Liu JJ, Yu ZH, et al. Research progress of Acidobacteria ecology in soils[J]. Biotechnology Bulletin, 2016, 32(2): 14-20. (in Chinese) 王光华, 刘俊杰, 于镇华, 等. 土壤酸杆菌门细菌生态学研究进展[J]. 生物技术通报, 2016, 32(2): 14-20. |
| [30] |
Barns SM, Cain EC, Leslie S, et al. Acidobacteria phylum sequences in uranium-contaminated subsurface sediments greatly expand the known diversity within the phylum[J]. Applied and Environmental Microbiology, 2007, 73(9): 3113-3116. DOI:10.1128/AEM.02012-06 |
| [31] |
Lu SP, Gischkat S, Reiche M, et al. Ecophysiology of Fe-cycling bacteria in acidic sediments[J]. Applied and Environmental Microbiology, 2010, 76(24): 8174-8183. DOI:10.1128/AEM.01931-10 |
| [32] |
Du Y. The study of microorganisms diversity on discolored fresco in Mogao Grottoes[D]. Lanzhou: Master's Thesis of Lanzhou University, 2013 (in Chinese) 杜烨.莫高窟色变壁画表面微生物多样性研究[D].兰州: 兰州大学硕士学位论文, 2013 |
| [33] |
Zimmermann J, Gonzalez J, Saiz-Jimenez C, et al. Detection and phylogenetic relationships of highly diverse uncultured acidobacterial communities in altamira cave using 23S rRNA sequence analyses[J]. Geomicrobiology Journal, 2005, 22(7/8): 379-388. |
| [34] |
Pašić L, Kovče B, Sket B, et al. Diversity of microbial communities colonizing the walls of a Karstic cave in Slovenia[J]. FEMS Microbiology Ecology, 2010, 71(1): 50-60. |
| [35] |
Takeuchi M, Fang CX, Yokota A. Taxonomic study of the genus Brachybacterium: proposal of Brachybacterium conglomeratum sp. nov., nom. rev., Brachybacterium paraconglomeratum sp. nov., and Brachybacterium rhamnosum sp. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 1995, 45(1): 160-168
|
| [36] |
Fu TT, Wang Y, Ge QY, et al. Identification and analysis of the microbe in Lidu ancient earthen cellar[J]. Microbiology China, 2017, 44(1): 49-58. (in Chinese) 付彤彤, 王宇, 葛琴雅, 等. 李渡酒窖土遗址中微生物种群的鉴定与分析[J]. 微生物学通报, 2017, 44(1): 49-58. |
| [37] |
Lyu CS, Cui YM, Liang SY, et al. Separation and phylogenetic analysis of soil microorganisms in Jijitan ancient site of Nihewan Basin[J]. Journal of Hebei Normal University (Natural Science Edition), 2017, 41(5): 440-447. (in Chinese) 吕春爽, 崔艳美, 梁思源, 等. 泥河湾藉箕滩遗址土壤微生物的分离与系统发育分析[J]. 河北师范大学学报:自然科学版, 2017, 41(5): 440-447. |
| [38] |
Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
| [39] |
Rettedal EA, Clay S, Br zel VS. GC-clamp primer batches yield 16S rRNA gene amplicon pools with variable GC clamps, affecting denaturing gradient gel electrophoresis profiles[J]. FEMS Microbiology Letters, 2010, 312(1): 55-62. DOI:10.1111/j.1574-6968.2010.02097.x |
| [40] |
Xu W, Dai MZ, Yang K, et al. Application of polymerase chain reaction denaturing gradient gel electrophoresis in environmental microbiology[J]. Environmental Research and Monitoring, 2017, 30(1): 1-6. (in Chinese) 许伟, 戴明忠, 杨凯, 等. 聚合酶链式反应-变性梯度凝胶电泳在环境微生物中的应用[J]. 环境研究与监测, 2017, 30(1): 1-6. |
 2019, Vol. 46
2019, Vol. 46