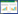扩展功能
文章信息
- 雍玮, 乔梦凯, 石利民, 王璇, 何敏, 丁洁
- YONG Wei, QIAO Meng-Kai, SHI Li-Min, WANG Xuan, HE Min, DING Jie
- 一株人感染高致病性禽流感(H5N1)病毒基因组分子特征分析
- Genomic molecular characterization of a highly pathogenic avian influenza A (H5N1) virus
- 微生物学通报, 2019, 46(11): 3058-3069
- Microbiology China, 2019, 46(11): 3058-3069
- DOI: 10.13344/j.microbiol.china.190578
-
文章历史
- 收稿日期: 2019-07-17
- 接受日期: 2019-08-20
- 网络首发日期: 2019-09-25
禽流感病毒H5N1亚型是属于正黏病毒科流感病毒属的A型禽流感病毒。病毒基因组为单股负链RNA,分为8个节段,为高致病性禽流感病毒。H5N1病毒于1996年在我国广东省的病鹅体内首次被发现[1]。1997年H5N1毒株A/HongKong/156/97在我国香港的活禽养殖业内引发了严重的疫病和病禽的高死亡率,随后跨种传播传染给了人类,导致18人感染,其中6人死亡,这也是全球首次H5N1感染人类的报道[2]。自此以后,高致病性禽流感病毒H5N1开始在亚洲和北非地区散发,尤其是在中国、越南、印度、印度尼西亚、柬埔寨和埃及等地多见,并且不断有人感染病例的报道[3]。截至2019年6月24日,全球共有861人感染H5N1禽流感病毒,其中死亡455人,死亡率高达52%[4]。我国内陆首例确诊的人感染H5N1患者于2006年在湖南被发现[5],此后陆续有人感染病例的报道[6]。
2015年1月22日,我中心确诊一例人感染H5N1病例,这是江苏省被确诊的第3例H5N1人感染病例,之前的两位患者为2007年确诊的一例家庭聚集性病例[7]。为进一步了解该病毒的特点,为人感染H5N1禽流感病毒的防控提供数据,考虑到病毒分离培养后得到的子代病毒可能会与亲代病毒在遗传特点上产生差异,本研究直接对病人原始呼吸道样本中的H5N1病毒A/Nanjing/1/2015进行全基因组序列测定,并对其分子特征进行分析和阐述。
1 材料与方法 1.1 标本来源2015年1月南京市疾病预防控制中心收到一例快速进展的急性重症呼吸道感染病人痰液样本。患者为女性,年龄37岁。
1.2 主要试剂和仪器病毒RNA小提试剂盒(QIAamp Viral RNA Mini Kit),QIAGEN公司;甲型/乙型流感病毒核酸检测试剂盒、流感H1N1亚型核酸检测试剂盒、禽流感H7N9亚型核酸检测试剂盒、禽流感H5N1亚型核酸检测试剂盒,江苏硕世生物科技有限公司;普通DNA产物纯化试剂盒,天根生化科技(北京)有限公司;Nextera XT DNA文库制备试剂盒,Illumina公司。MiSeq测序仪,Illumina公司;实时荧光定量PCR仪,赛默飞世尔科技(中国)有限公司;荧光定量仪,QIAGEN公司。
1.3 病毒核酸检测取4 mL D-Hank’s液稀释痰液样本,剧烈振荡30 s,取140 μL稀释后的痰液标本上清,按照试剂盒说明书手工进行核酸抽提,抽提结束后以60 μL洗脱液溶解核酸。各取RNA提取液5 μL,使用荧光PCR法对病毒核酸进行甲型/乙型流感、甲型H1N1亚型、禽流感H7N9亚型和H5N1亚型检测。
1.4 测序文库构建及高通量测序使用流感病毒逆转录引物Uni12 (M)[8] (江苏省疾控中心提供)将病毒RNA反转录成单链cDNA,再通过甲型流感病毒通用引物扩增流感病毒8个片段,PCR产物纯化后用荧光定量仪进行定量。纯化后的DNA作为测序模板按照Nextera XT DNA文库制备试剂盒说明构建测序文库,使用MiSeq测序仪进行全基因组序列测定。
1.5 序列分析及进化树构建测序所得原始序列数据使用CLC Genomics Workbench 9.0进行质控、拼接,所得序列使用NCBI BLASTn进行相似性检索,在MEGA 5.22中使用邻近归并法(Neighbor-Joining)构建系统进化树并分析重要氨基酸位点的变异情况,进化树用Bootstrap重复抽样1 000次进行验证。
2 结果与分析 2.1 病毒基因组序列基本特征和遗传进化特征分析对A/Nanjing/1/2015病毒进行全基因组测序,序列整理并拼接后获得8个基因片段(PB2、PB1、PA、HA、NP、NA、M、NS)的全长序列,长度分别为2 280、2 274、2 151、1 704、1 497、1 350、982、823 bp。此8个片段的基因序列已经在线提交至GenBank,序列号分别为PB2 (MF598576)、PB1 (MF598577)、PA (MF598578)、HA (MF598579)、NP (MF598580)、NA (MF598581)、M (MF598582)、NS (MF598583)。
在NCBI中对H5N1禽流感病毒8个基因片段进行检索,使用MEGA 5.22软件对A/Nanjing/1/ 2015病毒和GenBank中下载的病毒序列构建进化树(图 1)。通过和分属不同家系的H5亚型血凝素HA基因进行聚类分析发现(图 1A),A/Nanjing/ 1/2015病毒的HA基因属于H5的2.3.2.1c家系,2007年江苏发现的两例人感染H5N1病毒的HA基因属于2.3.4家系。A/Nanjing/1/2015病毒的HA基因在进化树上同江苏H5N1分离株A/chicken/ Jiangsu/YB7/2015和温州H5N1分离株A/duck/ Wenzhou/YJYF33/2015的遗传距离最近,形成一个紧密的独立分支,而同时期分离的2.3.2.1c家系里的其他毒株则聚类成另一个大的分支。神经氨酸酶NA基因与温州H5N1分离株A/duck/Wenzhou/ YJYF33/2015的遗传距离最近。其余的6个基因也是与江苏H5N1分离株A/chicken/Jiangsu/YB7/ 2015和/或温州H5N1分离株A/duck/Wenzhou/ YJYF33/2015的亲缘关系最近。
|
| 图 1 A/Nanjing/1/2015病毒基因系统进化树 Figure 1 Phylogenetic trees of A/Nanjing/1/2015 virus 注:A:HA;B:NA;C:PB2;D:PB1;E:PA;F:NP;G:M;H:NS. “●”标注的为本研究中的A/Nanjing/1/2015病毒;“■”标注的为2007年江苏分离到的两株人感染H5N1毒株;“▲”为H5亚型2.3.2.1c家系的疫苗株;括号内为GenBank序列号,疫苗株为GISAID数据库序列号;分支节点上的数字表示构建系统树时在1 000次计算中推算出此节点的百分比;标尺长度代表进化距离. Note: A: HA; B: NA; C: PB2; D: PB1; E: PA; F: NP; G: M; H: NS. "●" indicates the present strain A/Nanjing/1/2015; "■" indicates the H5N1 strains isolated from Jiangsu in 2007; "▲" indicates the vaccine strain of H5 subtype clade 2.3.2.1c;Numbers in parentheses are GenBank accession numbers, and the vaccine strain accession number was obtained from GISAID database; The bootstrap values (%) located on the nodes are inferred from 1 000 replications; The scale bars indicate the evolutionary distance. |
|
|
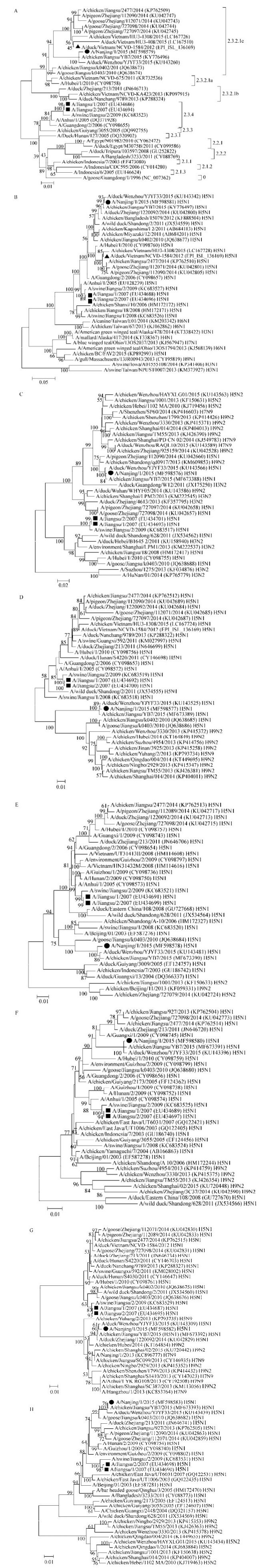
A/Nanjing/1/2015病毒的HA裂解位点氨基酸序列为PQRERRRR/G (图 2),与另外5株同属2.3.2.1c家系的H5病毒株序列一致。在裂解位点处存在连续4个碱性氨基酸精氨酸(R),符合高致病性禽流感病毒特征[9]。

|
| 图 2 不同进化支H5N1病毒HA裂解位点比较 Figure 2 Comparison of HA protein cleavage sites of H5N1 in different clades 注:“.”表明该位点上的氨基酸与A/Goose/Guangdong/1/96一致;“–”表明与A/Goose/Guangdong/1/96相比该位点上的氨基酸缺失. Note: "." indicates that the amino acid in the present position is consistent with A/Goose/Guangdong/1/96; "–" indicates the deletion of amino acid in the present position compared with A/Goose/Guangdong/1/96. |
|
|
如图 3所示,HA蛋白受体结合位点第222位(H5亚型编号)氨基酸为Q,第224位为G,未发生突变,保持禽类受体特征[10]。HA蛋白94位点出现D→A突变,133位点出现S→A突变,188位点出现T→I突变,这些突变被认为可以增加禽流感病毒的毒力和对人类受体的结合能力[11-12]。此外,在HA1氨基酸序列中,有6个潜在的N型糖基化位点,可能成为流感病毒逃脱宿主免疫的重要因素[13]。
|
| 图 3 血凝素HA1蛋白氨基酸序列比对结果 Figure 3 Comparison of amino acids sequences in HA1 protein 注:灰色底纹表示关键性的氨基酸置换位点,下划线表示潜在糖基化位点. “.”表明该位点上的氨基酸与A/Goose/Guangdong/1/96一致;“–”表明与A/Goose/Guangdong/1/96相比该位点上的氨基酸缺失. Note: Grey shade indicates the positions of key amino acids substitution; Underlines indicate the potential glycosylation sites. "." indicates that the amino acid in the present position is consistent with A/Goose/Guangdong/1/96; "–" indicates the deletion of amino acid in the present position compared with A/Goose/Guangdong/1/96. |
|
|
与H5N1病毒2.3.2.1c家系的疫苗株A/duck/ Vietnam/NCVD-1584/2012相比,两者在树图上分属两个较大的分支(图 1 HA),并且该病毒HA1蛋白中存在16个氨基酸置换的位点(L66W、T71I、A86V、D88G、L129K、V131G、N140K、D154N、N155D、A156T、G163D、I174V、E184K、T188I、R189T、I282V),提示其相对于疫苗株来说已经发生了较大的进化和变异(图 3)。
2.3 神经氨酸酶NA蛋白中氨基酸位点对致病性和传播性的影响NA蛋白颈部的长度与病毒的传播和致病力相关。A/Nanjing/1/2015病毒的NA蛋白颈部在49–68位缺失20个氨基酸,更易于表现出高致病性[9, 14]。在NA蛋白中出现了神经氨酸酶类抗病毒药物耐药性突变H274Y[15]。其他常见的耐药位点中,116位为V,150位为K,222位为I,并未发生突变。
2.4 其他蛋白重要氨基酸位点分析禽流感病毒内部基因的某些关键位点对病毒的致病性、传播能力和耐药性也有着重要影响。在A/Nanjing/1/2015病毒的PB2蛋白中,第627位氨基酸为E,未发生突变,但是PB2蛋白中出现了L89V、G309D、T339K、R477G和I495V置换;PB1蛋白第598位出现P→L突变,622位出现S→G突变;NS1蛋白中80-84位的氨基酸缺失,同时存在P42S置换;M1蛋白中出现N30D和T215A突变。这些突变均能在一定程度上增强病毒的毒力和致病性(表 1)。此外,NS1蛋白中第149位为丙氨酸时提示该病毒拮抗干扰素生成的能力较强;M2蛋白第31位氨基酸为S,未发生耐药突变。
| 蛋白Protein | 关键位点Key sites | 作用Function |
| PB2 | L89V G309D T339K R477G I495V | 增强聚合酶活性和病毒毒力[16] Enhancing polymerase activity and virus virulence[16] |
| PB1 | P598L | 在没有发现PB2蛋白E627K突变的H5N1病毒中修复PB2在人类细胞中的聚合酶活性,使其仍易于感染人类细胞[17] Repairing the polymerase activity of H5N1 PB2 protein without E627K mutations in order to keep it easy to infect human cells[17] |
| S622G | 增强病毒致病性[18] Enhancing the pathogenicity of the virus[18] | |
| NS1 | P42S 80-84 deletion | 增强病毒毒力[19-20] Increasing virus virulence[19-20] |
| M1 | 149A N30D T215A | 149位为丙氨酸的重组病毒拮抗干扰素生成的能力较强[21] Recombinant viruses with 149A have a strong ability to antagonize interferon production[21] 这两者的累加效应是H5N1病毒对哺乳动物高致病力所必需[22] The cumulative effect of the two mutations is necessary for H5N1 virus to be highly pathogenic to mammals[22] |
本研究采用高通量测序技术对一株人感染H5N1禽流感病毒A/Nanjing/1/2015进行全基因组测序。有研究显示,流感病毒在分离培养过程中,其关键性的氨基酸位点会发生选择性变异,子代病毒往往会丢失一些亲代病毒的特征或出现新的突变[23-24]。因此在本研究中,用于测序的模板是患者痰液样本中直接提取的病毒核酸,病毒没有经过人工的分离培养过程,能够更加直接准确地反映其遗传特点。
经测序分析,A/Nanjing/1/2015病毒属于H5亚型禽流感病毒2.3.2.1c家系。H5亚型禽流感病毒2.3.2.1c家系自2009年起在我国和日本、韩国等国家流行[25]。2013年该家系在北美首例人感染H5N1病毒中发现新的重组,其PB2基因片段来源于H9N2病毒,其余片段来源于H5N1病毒,此病患者发病前曾到过中国旅游[26]。随后,这一新的重组病毒在中国山东株和河南三门峡野鸟疫情中均被检测到[27]。随着鸟类迁徙,该病毒在中国、亚洲其他国家、西非以及欧洲的野鸟及家禽中被传播,并在印度的家禽中引起了数次禽流感的暴发,提示这一亚型的H5N1病毒传播广泛,致病力较强。本研究中的A/Nanjing/1/2015病毒8个片段均来自于江浙地区的H5N1病毒,但在其PB2树图中仍可看到,该片段与2013-2014年间江浙地区H9N2病毒PB2片段的同源性很高,高度提示这一病毒的PB2片段初始来源可能为H9N2病毒,并且这一重组病毒已成为江浙地区禽类中流行的主要病毒类型。
在禽流感病毒中,HA蛋白受体结合部位(Receptor-binding site,RBS)的氨基酸序列对病毒的宿主特异性、致病力、复制能力和传播能力有着重要的影响。H5N1禽流感病毒的RBS由3个二级结构元件组成:190螺旋(184-196)、130环(129-133)和220环(217-224)[28],其中位于220环的两个重要突变Q222L和G224S是禽流感病毒能够跨种传播感染哺乳动物的关键性因素[29]。对A/Nanjing/1/2015病毒的分子特征分析表明,该病毒HA蛋白中受体结合决定性位点Q222和G224没有发生突变,仍保持着与禽类唾液酸α-2, 3型受体结合的特点。但是,该病毒同时出现了D94N、S133A和T188I的氨基酸置换,其中D94N可以降低病毒对禽类唾液酸α-2, 3型受体的亲和性而增强对人型唾液酸α-2, 6半乳糖受体的结合[11],而S133A和T188I的双位点突变也能使病毒对唾液酸α-2, 6半乳糖受体的亲和性增强[12],从而增强病毒从禽类向人传播的能力。此外,在病毒HA1氨基酸序列中还存在6个N型糖基化位点。糖基化是病毒可以逃脱宿主免疫的一个重要方式,它能够帮助病毒遮蔽抗原表位、抑制宿主的体液免疫应答,从而使病毒逃避宿主免疫系统的识别和清理[30]。
另一个禽流感病毒跨种传播的决定性突变,位于PB2蛋白中的E627K,在A/Nanjing/1/2015病毒中也未被发现。有研究表明,H5N1病毒PB1蛋白中的P598L置换可以在未出现E627K突变的病毒株中增强病毒聚合酶的活性,修复病毒在人类细胞中的复制能力,使之仍然易于感染人类细胞并增殖[17]。A/Nanjing/1/2015病毒PB1蛋白中存在P598L置换,增强了该毒株感染人类的致病力。同时,PB1蛋白中的S622G位点,PB2蛋白中的L89V、G309D、T339K、R477G和I495V突变,NS1蛋白中的P42S置换和80-84位的氨基酸缺失以及M1蛋白中的N30D和T215A置换,这些突变均被报道可以在一定程度上增强病毒的毒力和致病性,提高了该病毒从禽类传播给人的概率。
在对A/Nanjing/1/2015病毒耐药位点的分析中发现,NA蛋白中存在神经氨酸酶抑制剂类抗病毒药物关键的耐药性突变H274Y,说明病毒已对奥司他韦产生耐药[15]。不过,M2蛋白第31位氨基酸仍为S,未发生耐药突变,提示该病毒对M2离子通道抑制剂类抗病毒药物金刚烷胺仍然敏感[31]。
自1997年H5N1禽流感病毒感染人以来,该亚型一直在通过重配来进行活跃的基因进化。H5基因片段会与多个不同的NA片段进行重配组合,产生不同亚型的多种禽流感病毒,如H5N2、H5N5、H5N6和H5N8等,在禽鸟中广泛传播[32]。自人感染H5N1以来,2014年5月四川又出现了全球第一例人感染H5N6病例,随后在广东、福建、江西等地散发出现H5N6感染病例,病死率高达50%[6]。卫生部门对活禽养殖、屠宰及交易等场所的外环境禽流感病毒监测发现,H5亚型长期存在于活禽体内和禽类环境中,并存在多种NA亚型的组合,如H5N1、H5N6等[7]。这些病毒常会在养殖家禽中引起大规模的疫情,仅2015年农业部就报告了30多起H5N6疫情。虽然近年来国家已在家禽养殖业中开展禽流感疫苗注射,但禽流感疫情仍是家禽养殖的巨大威胁,如2018年11月扬州家禽H5N6高致病性禽流感疫情[33]、2019年2月辽宁新民家禽H5N1禽流感疫情[34]、2019年5月新疆霍尔果斯市家禽H5N6亚型禽流感疫情[35]等,扑杀家禽均以万计。家禽携带病毒的增多,在一定程度上加大了病毒跨种传播给人类的风险。因此,有必要持续强化对外环境中禽流感病毒的监测,加强对禽流感病毒分子进化趋势的分析和动态监测,及早发现新的重组型别,及时提出流行预警,为防控策略的制定提供依据。
| [1] |
Xu XY, Subbarao, Cox NJ, et al. Genetic characterization of the pathogenic influenza A/Goose/Guangdong/1/96 (H5N1) virus: similarity of its hemagglutinin gene to those of H5N1 viruses from the 1997 outbreaks in Hong Kong[J]. Virology, 1999, 261(1): 15-19. DOI:10.1006/viro.1999.9820 |
| [2] |
Claas ECJ, de Jong JC, van Beek R, et al. Human influenza virus A/HongKong/156/97 (H5N1) infection[J]. Vaccine, 1998, 16(9/10): 977-978. |
| [3] |
Dhingra MS, Artois J, Robinson TP, et al. Global mapping of highly pathogenic avian influenza H5N1 and H5Nx clade 2.3.4.4 viruses with spatial cross-validation[J]. eLife, 2016, 5: e19571. DOI:10.7554/eLife.19571 |
| [4] |
WHO. Cumulative number of confirmed human cases for avian influenza A (H5N1) reported to WHO, 2003-2019[EB/OL]. (2019-06-24). https://www.who.int/influenza/human_animal_interface/2019_06_24_tableH5N1.pdf?ua=1
|
| [5] |
Yu HJ, Shu YL, Hu SX, et al. The first confirmed human case of avian influenza A (H5N1) in Mainland China[J]. The Lancet, 2006, 367(9504): 84. DOI:10.1016/S0140-6736(05)67894-4 |
| [6] |
Wan XF, Dong LB, Lan Y, et al. Indications that live poultry markets are a major source of human H5N1 influenza virus infection in China[J]. Journal of Virology, 2011, 85(24): 13432-13438. DOI:10.1128/JVI.05266-11 |
| [7] |
Wang H, Feng ZJ, Shu YL, et al. Probable limited person-to-person transmission of highly pathogenic avian influenza A (H5N1) virus in China[J]. The Lancet, 2008, 371(9622): 1427-1434. DOI:10.1016/S0140-6736(08)60493-6 |
| [8] |
Hoffmann E, Stech J, Guan Y, et al. Universal primer set for the full-length amplification of all influenza A viruses[J]. Archives of Virology, 2001, 146(12): 2275-2289. DOI:10.1007/s007050170002 |
| [9] |
Xu XL, Bao HM, Chen HL, et al. Research progress on key amino acid sites affectting pathogenicity and transmission of avian influenza virus[J]. Chinese Journal of Preventive Veterinary Medicine, 2014, 36(2): 165-168. (in Chinese) 徐晓龙, 包红梅, 陈化兰, 等. 影响禽流感病毒致病性和传播的关键氨基酸位点的研究进展[J]. 中国预防兽医学报, 2014, 36(2): 165-168. DOI:10.3969/j.issn.1008-0589.2014.02.19 |
| [10] |
Zhou JF, Wang DY, Gao RB, et al. Biological features of novel avian influenza A (H7N9) virus[J]. Nature, 2013, 499(7459): 500-503. DOI:10.1038/nature12379 |
| [11] |
Su Y, Yang HY, Zhang BJ, et al. Analysis of a point mutation in H5N1 avian influenza virus hemagglutinin in relation to virus entry into live mammalian cells[J]. Archives of Virology, 2008, 153(12): 2253-2261. DOI:10.1007/s00705-008-0255-y |
| [12] |
Yang ZY, Wei CJ, Kong WP, et al. Immunization by avian H5 influenza hemagglutinin mutants with altered receptor binding specificity[J]. Science, 2007, 317(5839): 825-828. DOI:10.1126/science.1135165 |
| [13] |
Wu CL, Lu X, Wang X, et al. Clinical symptoms, immune factors, and molecular characteristics of an adult male in Shenzhen, China infected with influenza virus H5N1[J]. Journal of Medical Virology, 2013, 85(5): 760-768. DOI:10.1002/jmv.23492 |
| [14] |
Li KS, Guan Y, Wang J, et al. Genesis of a highly pathogenic and potentially pandemic H5N1 influenza virus in eastern Asia[J]. Nature, 2004, 430(6996): 209-213. DOI:10.1038/nature02746 |
| [15] |
Yusuf M, Mohamed N, Mohamad S, et al. H274Y's effect on oseltamivir resistance: what happens before the drug enters the binding site[J]. Journal of Chemical Information and Modeling, 2016, 56(1): 82-100. DOI:10.1021/acs.jcim.5b00331 |
| [16] |
Li JW, Ishaq M, Prudence M, et al. Single mutation at the amino acid position 627 of PB2 that leads to increased virulence of an H5N1 avian influenza virus during adaptation in mice can be compensated by multiple mutations at other sites of PB2[J]. Virus Research, 2009, 144(1/2): 123-129. |
| [17] |
Xu C, Hu WB, Xu K, et al. Amino acids 473V and 598P of PB1 from an avian-origin influenza A virus contribute to polymerase activity, especially in mammalian cells[J]. Journal of General Virology, 2012, 93(3): 531-540. DOI:10.1099/vir.0.036434-0 |
| [18] |
Feng XX, Wang Z, Shi JZ, et al. Glycine at position 622 in PB1 contributes to the virulence of H5N1 avian influenza virus in mice[J]. Journal of Virology, 2016, 90(4): 1872-1879. DOI:10.1128/JVI.02387-15 |
| [19] |
Jiao PR, Tian GB, Li YB, et al. A single-amino-acid substitution in the NS1 protein changes the pathogenicity of H5N1 avian influenza viruses in mice[J]. Journal of Virology, 2008, 82(3): 1146-1154. DOI:10.1128/JVI.01698-07 |
| [20] |
Trapp S, Soubieux D, Marty H, et al. Shortening the unstructured, interdomain region of the non-structural protein NS1 of an avian H1N1 influenza virus increases its replication and pathogenicity in chickens[J]. Journal of General Virology, 2014, 95(6): 1233-1243. |
| [21] |
Li ZJ, Jiang YP, Jiao PR, et al. The NS1 gene contributes to the virulence of H5N1 avian influenza viruses[J]. Journal of Virology, 2006, 80(22): 11115-11123. DOI:10.1128/JVI.00993-06 |
| [22] |
Fan SH, Deng GH, Song JS, et al. Two amino acid residues in the matrix protein M1 contribute to the virulence difference of H5N1 avian influenza viruses in mice[J]. Virology, 2009, 384(1): 28-32. DOI:10.1016/j.virol.2008.11.044 |
| [23] |
Takemae N, Ruttanapumma R, Parchariyanon S, et al. Alterations in receptor-binding properties of swine influenza viruses of the H1 subtype after isolation in embryonated chicken eggs[J]. Journal of General Virology, 2010, 91(4): 938-948. DOI:10.1099/vir.0.016691-0 |
| [24] |
Gambaryan AS, Robertson JS, Matrosovich MN. Effects of egg-adaptation on the receptor-binding properties of human influenza A and B viruses[J]. Virology, 1999, 258(2): 232-239. DOI:10.1006/viro.1999.9732 |
| [25] |
Sakoda Y, Ito H, Uchida Y, et al. Reintroduction of H5N1 highly pathogenic avian influenza virus by migratory water birds, causing poultry outbreaks in the 2010-2011 winter season in Japan[J]. Journal of General Virology, 2012, 93(3): 541-550. DOI:10.1099/vir.0.037572-0 |
| [26] |
Pabbaraju K, Tellier R, Wong S, et al. Full-genome analysis of avian influenza A (H5N1) virus from a human, North America, 2013[J]. Emerging Infectious Diseases, 2014, 20(5): 887-891. |
| [27] |
Wang HM, Chen ZQ, Zhang ZJ, et al. Continuing spread and evolution of clade 2.3.2.1c H5N1 highly pathogenic avian influenza viruses in China[J]. Journal of Pathogen Biology, 2016, 11(2): 97-102, 115. (in Chinese) 王海明, 陈占强, 张振杰, 等. 2.3.2.1c家系H5N1禽流感病毒在我国的持续传播与进化[J]. 中国病原生物学杂志, 2016, 11(2): 97-102, 115. |
| [28] |
Skehel JJ, Wiley DC. Receptor binding and membrane fusion in virus entry: the influenza hemagglutinin[J]. Annual Review of Biochemistry, 2000, 69: 531-569. DOI:10.1146/annurev.biochem.69.1.531 |
| [29] |
Herfst S, Schrauwen EJA, Linster M, et al. Airborne transmission of influenza A/H5N1 virus between ferrets[J]. Science, 2012, 336(6088): 1534-1541. DOI:10.1126/science.1213362 |
| [30] |
Abe Y, Takashita E, Sugawara K, et al. Effect of the addition of oligosaccharides on the biological activities and antigenicity of influenza A/H3N2 virus hemagglutinin[J]. Journal of Virology, 2004, 78(18): 9605-9611. DOI:10.1128/JVI.78.18.9605-9611.2004 |
| [31] |
Wang J, Wu YB, Ma CL, et al. Structure and inhibition of the drug-resistant S31N mutant of the M2 ion channel of influenza A virus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(4): 1315-1320. DOI:10.1073/pnas.1216526110 |
| [32] |
Jhung MA, Nelson DI. Outbreaks of avian influenza A (H5N2), (H5N8), and (H5N1) among birds-United States, December 2014-January 2015[J]. Morbidity and Mortality Weekly Report, 2015, 64(4): 111. |
| [33] |
Sina Jiangsu. An epidemic of H5N6 highly pathogenic avian influenza occurred in Yangzhou and more than 30, 000 poultry were culled[EB/OL]. (2018-11-23). http://news.sina.com.cn/c/2018-11-23/doc-ihpevhck2514268.shtml (in Chinese) 新浪江苏.扬州发生H5N6亚型高致病性禽流感疫情三万余家禽被扑杀[EB/OL]. (2018-11-23). http://news.sina.com.cn/c/2018-11-23/doc-ihpevhck2514268.shtml |
| [34] |
Ministry of Agriculture and Rural Affairs of the People's Republic of China. An outbreak of highly pathogenic H5N1 avian influenza in poultry occurred in Xinmin city, Liaoning Province[EB/OL]. (2019-04-02). http://www.moa.gov.cn/gk/yjgl_1/yqfb/201904/t20190402_6177777.htm (in Chinese) 中华人民共和国农业农村部.辽宁省新民市发生一起家禽H5N1高致病性禽流感疫情[EB/OL]. (2019-04-02). http://www.moa.gov.cn/gk/yjgl_1/yqfb/201904/t20190402_6177777.htm |
| [35] |
Ministry of Agriculture and Rural Affairs of the People's Republic of China. An outbreak of highly pathogenic H5N6 avian influenza in poultry occurred in Horgos, Xinjiang[EB/OL]. (2019-05-30). http://www.moa.gov.cn/gk/yjgl_1/yqfb/201906/t20190603_6316030.htm (in Chinese) 中华人民共和国农业农村部.新疆霍尔果斯市发生一起家禽H5N6亚型高致病性禽流感疫情[EB/OL]. (2019-05-30). http://www.moa.gov.cn/gk/yjgl_1/yqfb/201906/t20190603_6316030.htm |
 2019, Vol. 46
2019, Vol. 46